Method Article
Ökaryotik Hücrelerde Protein Kompleksleri Tandem Affinity Arıtma
* Bu yazarlar eşit katkıda bulunmuştur
Bu Makalede
Özet
We describe here a novel, robust, and efficient tandem affinity purification (TAP) method for the expression, isolation, and characterization of protein complexes from eukaryotic cells. This protocol could be utilized for the biochemical characterization of discrete complexes as well as the identification of novel interactors and post-translational modifications that regulate their function.
Özet
Aktif protein-protein, protein-nükleik asit komplekslerinin saflaştırılması enzimsel faaliyetlerin ve yeni alt birimleri ve post-translasyonel modifikasyonlar de novo tanımlanması karakterizasyonu için çok önemlidir. Bakteriyel sistemlerde bir polipeptidler ve protein kompleksleri çok çeşitli ekspresyon ve saflaştırma için izin verir. Bununla birlikte, bu sistem, post-translasyonel modifikasyonları (örneğin, fosforilasyon ve asetilasyon) içeren protein alt birimlerinin temizleme, ve ökaryotik sisteminde ifade, sadece mevcut olan yeni düzenleyici alt birimlerin / belirlenmesini sağlamaz. Burada, ökaryotik hücrelerden geçici veya stabil ifade epitop etiketli proteinler ile protein komplekslerinin saflaştırılması kolaylaştıran bir roman, sağlam ve streptokok kullanarak verimli tandem yakınlık arıtma (TAP) yöntemiyle ve FLAG-etiketli proteinlerin ayrıntılı bir açıklama sağlar. Bu protokol, Prot karakterize uygulanabilirein karmaşık işlevsellik, kompleks alt birimlerin üzerine post-translasyonel modifikasyonlar keşfetmek ve kütle spektrometresi ile yeni düzenleyici kompleks bileşenleri tanımlamak için. Özellikle, bu TAP yöntemi ve böylece alt deney imkanları geniş bir dizi elde ökaryotik veya patojenik (viral ve bakteriyel) bileşenlerinin oluşturduğu protein kompleksleri çalışma uygulanabilir. Biz protein kompleksleri ile çalışan araştırmacılar, çok farklı şekillerde bu yaklaşımı kullanmak olabilir öneriyoruz.
Giriş
Protein-protein etkileşimleri (PPİ) işlevleri yaklaşık 2 bilgilendirebilir biyolojik süreçler 1, bu PPI üzerinde ileri çalışmalar hassas düzenlenmesi için kritik öneme sahiptir. Çeşitli yaklaşımlar PPI çalışma ve karakterizasyonu için, ayrıca bu yeni düzenleyici protein bileşenlerinin de novo tanımlanması için geliştirilmiştir. 1989 yılında, Stanley Alanlar ve arkadaşları maya iki hibrit (Y2H) tahlil 3 bildirdi. Bu yaklaşım, Saccharomyces cerevisiae 'interactors oranı (yem) tanımlanmış bir protein için (avlayan) tarafsız ve kapsamlı bir tanımlanmasına izin verir. PPIs keşfetmek için dikkate değer yardımcı ek olarak, Y2H deneyi, maya hücrelerinde protein çiftleri karakterize az etkileşim alanları tanımlamak ve bu etkileşimi ortadan kaldırmak başkalaşımların teşhis edilmesi için kullanılabilir. Y2H deneyi değiştirerek, PPIs, aynı zamanda, memeli hücrelerinde incelenebilirclass = "xref"> 4. Y2H tahlilinde (örneğin, maya, üç-hibrid sistemi) varyasyonları da protein-RNA ve hücrelerde protein küçük organik ligand etkileşimlerini incelemek için uygulanabilir.
Homolog bir sistemde PPIs incelemek için bir başka genel olarak kullanılan bir araç ko-immünopresipitasyon (ko-IP) deney 5'tir. ilgili bir proteini immünopresipitasyon bir antikor kullanılarak, CO-IP deney araştırmacılar, çeşitli çevre koşulları ve deneysel durumlarda hücrelere PPI izlemesine izin verir. Epitop etiketli proteinler (örneğin, FLAG, myc, Strep ve HA, diğerleri arasında) afinite temizliği (AP) yöntemleri, batı lekesi de dahil olmak üzere çok sayıda alt-tahliller için kompleks protein karışımları proteinlerin izole edilmesini kolaylaştırmıştır, gümüş leke kullanımı ve enzimatik analizi. Bununla birlikte, bu daha önceki yaklaşımların hiçbiri, in vitro bir de dahil olmak üzere daha ayrıntılı karakterizasyon için protein kompleksleri büyük miktarlarda izole edilmesini sağlamakssays, kütle spektrometresi, ve post-translasyonel modifikasyonlar tanımlanması ile düzenleyici alt birimlerin bulunması. AP yöntemin geliştirilmiş bir versiyonu, iki takip eden AP 6, 7 aracılığıyla saflaştırılır iki epitop bir füzyon proteini oluşturarak PPIs çalışmak için bir saflaştırma tekniğidir Tandem AP (TAP) olarak adlandırılır. Bu makalede, iki alt birimi farklı epitoplar ile etiketlenir ve daha sonra iki ardışık AP (STREP AP FLAG IP elde edilmiş) ile saflaştırılmış edildiği arıtma protein kompleksleri TAP yöntemin bir varyasyonu sunulmuştur. Araştırmacılar kendi ilgi protein kompleksine bunları uygulamak, böylece Biz ilk, TAP minimalist bakış (Şekil 1) ve sonra tüm deneysel adımlar ayrıntılı bir açıklama (Şekil 2) sağlarlar.
TAP yöntemin uygulanabilirliğini göstermek için, (karmaşık, iyi karakterize edilmiş siklin cdk PT şu şekilde de ifade seçtikDüzenleyici alt birim, siklin T1 (CycT1) ve bir kinaz (CDK9) oluşmaktadır, ve RNA polimeraz II (pol II), 8, 9, 10 ile transkripsiyonun düzenlenmesinde görevli EFP kinaz). P-TEFb Pol II ve promoter de transkripsiyon duraklatma hafifletir ilişkili olumsuz uzama faktörleri, C-terminal sahasını fosforilleyen ve böylece transkripsiyon uzama 11, 12, 13 kolaylaştırır. Bu akılda bilinen etkileşim sayesinde, CycT1 STREP-etiketli ve FLAG-etiketli CDK9 HEK293T hücrelerinde ifade üzerindeydi. Bir karşılıklı TAP deney daha protein etkileşim kullanılan epitopların bağımsız olduğunu doğrulamak için STREP etiketli CDK9 ve FLAG-etiketli CycT1 ile gerçekleştirildi. Hücreler toplandı ve 48 saat sonra transfeksiyon parçalanmıştır. Çözünür lizat TAP ile saflaştırılmıştır (STREP AP FLAG ardındanIP). Giriş ve saflaştırılmış proteinler Western blot ve gümüş leke (Şekil 3) ile analiz edilmiştir.
Protokol
1. Kaplama Hücreler
- Bir gün, transfeksiyondan önce,% 10 (h / h) cenin sığır serumu (FBS) ve% 1 (h / h), penisilin / streptomisin ile takviye edilmiş Dulbecco Modified Eagle Medium (DMEM) içinde 10 mL 3.5 x 10 6 HEK293T hücreleri plaka 100 mm tabak başına (10.000 U / ml stok). Plaka 3 - Deney / durum başına 5 100 mm yemekleri yeterli protein iyileşme sağlamak için.
Not: plakaların sayısı ilgili protein kompleksi ifade seviyeleri ve çözünürlüğüne bağlı olacaktır, her bir deney / durum için tespit edilmesi gerekir. Daha büyük ölçekli saflaştırma gerektiğinde, alternatif olarak, hücre çizgileri döndürücü şişelere 2 süspansiyonlar büyümeye adapte edilebilir, ancak bu yöntem, geçici nakiller için çalışmaz.
2. Kararlı Hücre Hatları hücrelerin transfekte ya da indükleyici
- Ertesi gün, mikroskop altında hücrelerin kontrol edin. proce önce% 80 konfluent - Hücreler 70 olmalıEding.
- 100 mm tabak başına transfeksiyon reaktifi 7 plasmid DNA ug ve 21 ul toplam karışımı hücreleri transfekte. Transfeksiyon etkinliğindeki için bir kontrol olarak, bir vektör ifade GFP (Tablo 1) ile transfekt hücre.
Not: doksisiklin 14, 15 karşılık olarak iki veya daha fazla kompleks alt birimleri ekspresyonunu indükleyen bir HEK293-T-Rex veya benzer sabit bir hücre doğrusu kullanılıyorsa, indükleyici hücrelere ilave edildi ve 48 saat süre ile inkübe edilmesi gerekecektir. Benzer şekilde, indükleyici ilave edilmesi üzerine GFP ekspresyonunu indükleyen bir dengeli hücre çizgisi, aynı zamanda, doğrulama için gerekli olacaktır. 2.7 adıma geçin. - 100 mm tabak başına transfeksiyon karışımı hazırlayın. 1.5 ml mikrosantrifüj tüpü içinde, (iki veya daha fazla plazmidin tekabül eder) toplam plazmid DNA 7 ug katkısız DMEM 500 ul karıştırın. Ayrı bir mikrosantrifüj tüpü içinde, katkısız DMEM wi 500 ul karışımıtransfeksiyon reaktifi inci 21 ul. Her transfeksiyon reaksiyon için tekrarlayın.
- plazmid DNA çözeltisi içine transfeksiyon reaktif çözüm pipetle (ters çözüm karışmaz). en az 4 kez pipetleme karıştırın.
- 10, oda sıcaklığında, bu karışım inkübe - 15 dakika, ancak artık, 15 dakika daha.
- Bir medya rezervuar oluşturmak ve dikkatle hücreleri bozmadan plaka iç yüzüne damla damla transfeksiyon karışımı pipetle için plaka yatırın. orijinal düz konumuna çanak dönün ve karışım dağıtmak için hafifçe döndürün.
- (Bu durum, bundan sonra "doku kültür inkübatörü" olarak da adlandırılır),% 5 CO2 ile nemlendirilmiş bir hücre kültürü inkübatöründe 37 ° C'de inkübe hücreleri. Medya 5 saat sonrası transfeksiyon (opsiyonel) değiştirin.
3. kontrolü Transfeksiyon veya İndüksiyon Verimlilik
- 24 saat sonrası transfeksiyon at, bir grip altında hücrelerin görselleştirmekFlüoresan mikroskop transfeksiyon verimliliği (ya da GFP ifade kontrol HEK293T-rex istikrarlı bir hücre hattı indüksiyonu) kontrol etmek için. bir 24 saat daha inkübe edilir.
4. STREP Affinity saflaştırma (STREP AP)
- hücrelerin toplanması
- 48 saat sonra transfeksiyon da ortamını çıkarın ve soğuk 1 x fosfat tamponlu tuzlu su (PBS) içinde 8 ml ekleyin. plaka hücreleri ayırmak için aşağı yukarı pipet ve.
- Toplayın ve 15 ml tüp içine hücre süspansiyonu aktarmak ve 4 ° C'de 4 dakika boyunca 800 xg'de hücreleri aşağı doğru döndürün.
- PBS süpernatantı. herhangi bir artık PBS ortadan kaldırmak için (4 ° C 'de 1 dakika boyunca 800 x g) döndürme aşağı tekrarlayın. ileride kullanmak üzere -80 ° C'de hücre pelletini Mağaza veya protein saflaştırma devam etmek için aşağıdaki prosedürü uygulayın.
- hücrelerin çözülmesi
- Pasif Lizis Tamponu hücre peleti hacmi x 5 kullanılarak hücre peletleri (~ plaka başına 250 ul) yeniden süspanse edin(PLB: 20 mM Tris-HCI pH 7.5, 150 mM NaCl, 1.5 mM MgCl2, 1 mM DTT, proteaz önleyici kokteyl tablet,% 5 h / h gliserol, ve% 1.0 NP-40) ile bir hücre süspansiyonu aktarmak 1.5 ml mikrosantrifüj tüp.
- Kısaca, 4 ° C'de 30 dakika süre ile süspansiyon ve nutate girdap.
- 4 ° C'de 10 dakika boyunca 10,000 x g'de hücre süspansiyonu dönerler.
- Yeni 1.5 ml mikrosantrifüj tüp içine çözünür lizatları aktarın ve hücre pelletleri atın. gibi çözülebilir lizat son hacminin% 10 Kaydet "STREP AP girişi." 4 ° C'de saklayın.
- STREP boncuk dengeleme
NOT: İlk AP adım için kullanın STREP boncuk! STREP boncuk BAYRAK boncuklar ile aşağı çekti göre anlamlı olarak daha fazla protein kurtarmak ile kompleksler aşağı çekti.- çıkartın (n 40 ul ×) + n% 50 STREP boncuk bulamacı ul (n = örnek sayısı) ve 4 ° C'de 2 dakika süreyle 1500 xg'de aşağı doğru döndürün.
NOT: Geniş m kullanınpipet tanelerine outhed ipuçları. - Süpernatantı ve boncuk PLB 500 ul ekleyin. 4 ° C'de 5 dakika boyunca boncuk karışımı Nutate. 4 ° C'de 2 dakika boyunca 1500 x g'de Spin.
- 3 yıkama toplam tekrarlayın. 40 ul x n nihai bir hacme kadar PLB boncuk tekrar.
- 4 ° C'de 2 saat boyunca her bir hücre lizatı numunesi ve nutate% 50 STREP boncuk çamuru 40 ul ekle.
- çıkartın (n 40 ul ×) + n% 50 STREP boncuk bulamacı ul (n = örnek sayısı) ve 4 ° C'de 2 dakika süreyle 1500 xg'de aşağı doğru döndürün.
- STREP AP
- 4 ° C'de 2 dakika süreyle 1500 xg'de çözüm aşağı doğru döndürün.
- 4 ° C'de "STREP AP Akış-through (FT)" olarak supernatant ve mağaza çıkarın.
- I (20 mM Tris-HCI pH 7.5, 250 mM NaCI, 1.5 mM MgCl2, 1 mM DTT,% 5 gliserol ve% 0.2 NP-40) STREP AP Yıkama Tamponu tanelere 500 ul ekle. 4 ° C'de 2 dakika boyunca 1500 x g'de 5, 4 ° C'de dakikada santrifüj için boncuk karışımı Nutate. süpernatant atın.
- 4 yıkar toplam için tekrarlayın.
NOT: Dördüncü yıkamadan önce, protein arka plan azaltmak için yeni bir 1.5 ml mikrosantrifüj tüp boncuk çözüm aktarın. Bu adım gereklidir.
- 4 yıkar toplam için tekrarlayın.
- 500 ul yıkama tamponu II (100 mM Tris-HCI, pH 8.0, 150 mM NaCl ve 1 mM EDTA) ilave edilir ve tüpler ters çevrilerek boncuk dengeye. 4 ° C'de 2 dakika süreyle 1500 xg'de Spin ve süpernatant atın.
- STREP elüsyon tamponu (100 mM Tris-HCI, pH 8.0, 150 mM NaCI, 1 mM EDTA ve 2.5 mM destiobiyotin) 50 ul ilgili proteini Zehir. 4 ° C'de 15 dakika boyunca 800 rpm'de vorteksleyin.
- 4 ° C'de 1 dakika için 1500 x g'de boncuk Spin. süpernatant toplayın. Bu fraksiyon "STREP AP elüsyon" örnek karşılık gelir.
- İlk elüsyon ile elde edilen protein miktarının yarısı kadar verim olabilir ikinci elüsyon, 4 ° C 'de boncuk fraksiyonu kaydedin. gümüş boyama ve / veya batı blot STREP AP Tasfiye Kısım 10%16.
- BAYRAK boncuk dengeleme
- (Nx 16 ul) + n BAYRAK boncuk çözümünün ul (numune n = sayı) çıkarın ve 4 ° C'de 2 dakika süreyle 1500 xg'de aşağı doğru döndürün.
NOT: pipet boncuk geniş ağızlı ipuçlarını kullanın. - Süpernatantı ve FLAG yıkama tamponu 500 ul ekle (100 mM Tris-HCI, pH 8.0, 150 mM NaCI, 1 mM EDTA ve% 0.1 NP-40) ile boncuklara. 4 ° C'de 2 dakika boyunca 1500 x g'de Spin. 3 yıkama toplam tekrarlayın.
- nx 16 ul bir son hacme FLAG yıkama tamponu boncuk tekrar.
- 4 ° C 'de geri kalan STREP AP elüsyon ve nutate gece boyunca FLAG boncuk çamuru 16 ul ekle.
- (Nx 16 ul) + n BAYRAK boncuk çözümünün ul (numune n = sayı) çıkarın ve 4 ° C'de 2 dakika süreyle 1500 xg'de aşağı doğru döndürün.
5. BAYRAK IP
- 4 ° C'de 2 dakika boyunca 1500 x g'de FLAG boncuk süspansiyonu dönerler. 4 ° C'de "BAYRAK IP Akış-through" (FT) olarak süpernatant ve mağaza kaydedin.
- 500 ekle81; çözeltiye FLAG yıkama tamponu. 4 ° C'de 2 dakika boyunca 1500 x g'de 5, 4 ° C'de dakikada santrifüj için Nutate. 4 yıkar toplam tekrarlayın ve her yıkamadan sonra süpernatant kaldırmak.
NOT: Dördüncü yıkamadan önce, arka plan azaltmak için yeni bir 1.5 ml mikrosantrifüj tüpüne protein boncuk çözüm aktarın. Bu adım gereklidir. - Final spin süpernatantı ve boncuk haline FLAG peptit 200 ng / ul içeren BAYRAK yıkama tamponu 30 uL ekleyin. 4 ° C'de 2 saat boyunca 800 rpm'de vorteksleyin.
- 4 ° C'de 2 dakika boyunca 1500 x g'de süspansiyon Spin. olarak 4 ° C'de supernatant ve mağaza Hasat "FLAG IP Elüsyon."
NOT: elüsyon numuneleri standart protokoller 16 kullanılarak gümüş leke ve / veya western blot ile teyit ve daha fazla protein deneyleri için hazır olabilir. (1) STREP AP Giriş, (2) STREP AP FT, (3) STREP AP Eluti: Batı leke kurarken, aşağıdaki örneklerin hepsi koşmak(4) BAYRAK IP FT, ve (5) BAYRAK IP Tasfiye üzerinde. Yüklenen Birimleri her bir deney için belirlenecek gerekecektir. Toplanan tüm örnekler ve boncuk kısa süreli depolama ve uzun süreli depolama için -80 ° C 'de 4 ° C' de muhafaza edilebilir. TAP örnekleri, bileşenlerinin kimliğini doğrulamak için yeni bir post-translasyonel modifikasyonlar (PTMS) tespit etmek, ve / veya saflaştırılmış kompleksi 2, 17 ile herhangi bir yeni protein etkileşimi keşfetmek için kütle spektrometresi ile analiz edilmiştir. Bu amaçla, Coomassie jel veya gümüş leke çalıştırdıktan sonra, bantlar, temiz bir neşter kullanılarak jelden edilebilir. Kütle spektrometresi yöntemlerini tartışmak birkaç mükemmel değerlendirmeleri daha önce, 19 18 yayınlandı.
Sonuçlar
Bu yazıda, (aynı zamanda P-TEFb kinaz olarak da bilinir), karmaşık iyi karakterize CycT1-CDK9 TAP yönteminin uygulanabilirliğini göstermek.
Ve CDK9,-FLAG (CDK9: F) ya da CDK9-Strep (CDK9: G) ve siklin T1 bayrağı (CycT1: F): Sikline T1 Strep (s CycT1) kodlayan plazmidler (Tablo 1), HEK293T hücrelerine transfekte edildi . F ya da CycT1: (sırasıyla Şekil 3A ve B,) F plazmitleri Negatif kontroller, boş vektör ve CDK9 transfeksiyon içermektedir. Hücreler toplandı ve önce proteinler, 48 saat boyunca ifade edildi. Hücrelerin çok tabakayı, protein üretimi ve geri kazanımı arttırmak, (örnek başına beş levhalar, Şekil 3'te sunulan veriler için kullanıldı) deneyde için kullanılmıştır. hücreleri bireysel mikrosantrifüj tüpleri içinde parçalanmıştır sonra aynı numunenin hücre lizatları bir microcentrif birleştirildiSTREP boncuk eklemeden önce uge tüp. boncuklar bir kısım protein örnekleri birleştiren ifade etmek zordur proteinlerden protein geri kazanımı artırabilir son protein konsantrasyonunu artırır. İlk elüsyon ardından, ikinci elüsyon FLAG IP için toplam protein girişini artırmak amacıyla yapılabilir. Ihtiyaç, ileride giderme (Tablo 2) boncukların önce her saflaştırma aşaması için giriş kaydetmek önemlidir.
Ve S CDK9: F, Western blot ile analiz Şekil 3A'da veriler CycT1 TAP yöntemin sonuçlarını göstermektedir. CDK9 F CycT1 ile birlikte yıkandı: S STREP boncuklardan ancak CycT1 yoksun negatif kontrol AP:, Bu CDK9 gösteren: F CycT1: S, protein-protein kompleksi oluştururlar. Beklendiği gibi, her iki proteinin de daha kompleks oluşumunu teyit FLAG elüsyon olarak tespit edildi. Şekil 3B resu göstermektedir(: S ve CycT1: CDK9 F) karşılıklı TAP iki proteinin epitop takıları takas edilmiştir yöntemin LTS ve western blot ile analiz edilmiştir.
Şekil 3C ve 3D, sırasıyla, gümüş boyama ile analiz TAP ve ters TAP sonuçlarını göstermektedir. F CycT1 ile birlikte yıkandı: S STREP boncuk kapalı, fakat kontrol AP (şerit 1) ile ilgili, ve böylece CDK9 belirten: F CycT1: S, protein-protein kompleksi meydana Şekil 3C, şerit 2 CDK9 olduğunu göstermiştir. daha sonra FLAG akıtmada olarak hat 4'te gösterildiği gibi, CycT1: FLAG boncuk rahatsız F: S CDK9 ile birlikte yıkandı. STREP boncuk rahatsız S, fakat protein-protein kompleksi etkin ikinci bir FLAG IP adımından sonra geri kazanılmıştır olmayan kontrol AP ve bu F CDK9 ile birlikte yıkandı: Benzer şekilde, Şekil 3B CycT1 gösterdi. Hem batı leke ve gümüş lekeler TAP protokol purificatio için verimli bir şekilde çalıştı doğruladı n, izolasyon ve ilgi protein kompleksinin karakterizasyonu.

Şekil 1: TAP Arıtma Stratejisinin genel bakış. TAP adımlarını açıklayan basit bir şema. Her iki bant veya FLAG epitopu etiketi ile ilgili proteini kodlayan plasmidler ko-transfekte edilmiş memeli hücrelerine bulunmaktadır. 48 saat indirgemeden sonra, hücreler toplandı ve lize edilmiştir. Diğer proteinler ile birlikte ilgili proteini ihtiva eden çözünebilir hücre lizatı STREP AP STREP boncuklar üzerine yüklenir. Her iki STREP etiketi içeren veya boncuk bağlı STREP-etiketli proteine bağlı proteinler daha sonra yıkanır. FLAG Kürecikler daha sonra STREP elüsyon eklenmiştir. Her iki BAYRAK etiketi içeren veya boncuk bağlı FLAG etiketli proteine bağlı proteinler daha sonra yıkanır. Elüsyon belirtilen yöntemlerle analiz edilebilir.ttp: //ecsource-jove-com.remotexs.ntu.edu.sg/files/ftp_upload/55236/55236fig1large.jpg "target =" _ blank "> bu rakamın daha büyük bir versiyonunu görmek için lütfen buraya tıklayınız.
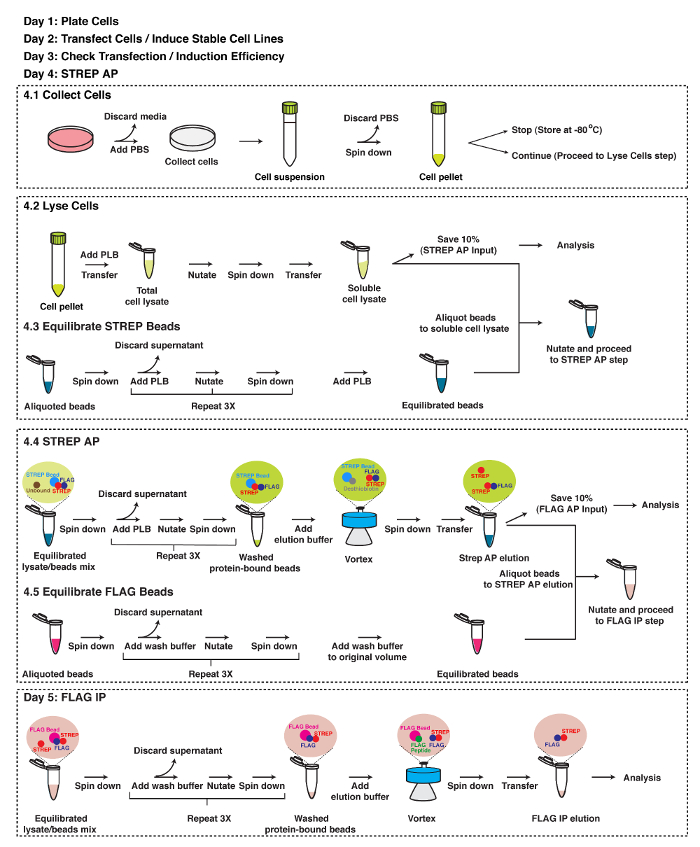
Şekil 2: TAP Strateji şematik gösterimi. TAP ayrıntılı bir protokol. ayrıntılar için metne bakınız. Bu rakamın büyük halini görmek için lütfen buraya tıklayınız.
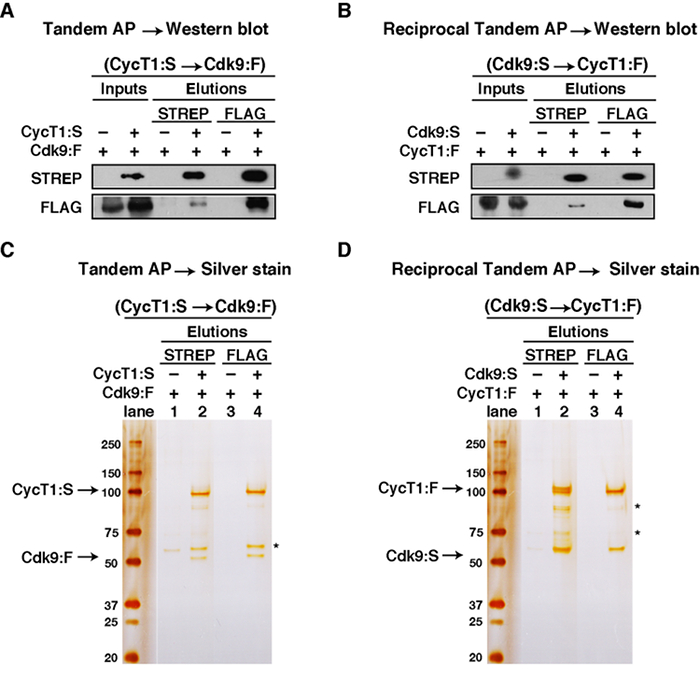
Şekil 3: TAP Arıtma Adım adım Analizi Örneği. (A) TAP Western blot analizi (CycT1: S ve CDK9: F). (B) karşılıklı TAP Western blot analizi (CDK9: S ve CycT1: F). froBütün hücre lizatı, 5 ul (% 0.2) m 1 ml olarak yüklenen "STREP AP Giriş." Geri kalan bütün hücre lizatı 975 ul STREP STREP AP boncuklar ve "STREP AP elüsyonu" 100 ul toplandı ilave edildi. Bu kaynaktan, 5 ul (% 2) yüklendi. "STREP AP elüsyonu" 80 ul FLAG FLAG IP boncuklar ve "FLAG IP elüsyonu" 30 ul toplandı ilave edildi. "BAYRAK IP Tasfiye" 30 ul itibaren, 7 ul (% 23.3) yüklenmiştir. (C) TAP Gümüş leke analizi (CycT1: S ve CDK9: F). (: S ve CycT1: CDK9 F) karşılıklı TAP (D) Gümüş leke analizi. "STREP AP Tasfiye" ve "BAYRAK IP Tasfiye" 7 ul (% 23.3) 5 ul (% 2) yüklendi. Yıldız birlikte yıkandı yabancı maddeleri belirtir. Bu rakamın büyük halini görmek için lütfen buraya tıklayınız.
| Protein | Kalıntıları | Vektör | Etiket | Klonlama siteleri | Referans |
| CycT1 | 1-708 | pcDNA / 4tuşuna | STREP | Hind - EcoRI | McNamara ve diğ. 2013 10 |
| CycT1 | 1-708 | pcDNA / 4tuşuna | BAYRAK | Hind - EcoRI | Bu makale |
| CDK9 | 1-372 | pcDNA / 4tuşuna | STREP | Hind - Xhol | Bu makale |
| CDK9 | 1-372 | pcDNA / 4tuşuna | FLAG | Hind - Xhol | McNamara ve ark. 2013 10 |
| GFP | 1-238 | pcDNA / 4tuşuna | Strep-FLAG | BamHI - Xhol | Bu makale |
Tablo 1: Bu çalışmasında kullanılan Plazmidler. CycT1 ve CDK9 pcDNA / 4tuşuna-Strep ve pcDNA / 4tuşuna-FLAG plazmid vektörlerine lige edilmiştir. Bu yapılar DH5α uyumlu hücrelere transforme ve LB-ampisilin levhalar üzerine kaplandı. Koloniler, seçilen büyüme ve elenmiştir. Başarıyla lige klonlar Sanger sekanslama ve sınırlama sindirimi ve agaroz jel elektroforezi kombinasyonu ile doğrulanmıştır. yerine, tam uzunlukta (1-726) zengin son 18 tortuları, bir PEST sırası içerdiğinden (bir peptit sekansı - CycT1 için bir kısa bir versiyonunu (708 1) kullanılanprotein yıkımı 20 için bir sinyal peptidi olarak hareket prolin, glutamik asit, serin ve treonin). CDK9 tam uzunluktadır. aşağıdaki gibi tandem STREP etiketinin dizisidir: GGGGWSHPQFEKGGGSGGGSGGGSWSHPQFEK. aşağıdaki gibi tandem FLAG etiketi dizisidir: GGGGDYKDHDGDYKDHDIDYKDDDDK.
Tablo 2: Sorun Giderme Tablosu. sorun giderme başlamadan önce, çift kontrol her önemli bir adım takip edilmiştir emin olmak için. Bu protokol bazı adımlar beklenen sonuca ulaşmak için diğerlerinden daha fazla optimizasyon gerektirir. Bu dosyayı indirmek için tıklayınız.
Tartışmalar
İfade ve ökaryotik hücrelerde protein komplekslerinin izolasyonu için açıklanan protokol, moleküler düzeneklerinin biyokimyasal karakterizasyonu ile sınırlı değildir, aynı zamanda işlevini düzenleyen olabilir yeni interactors ve post-translasyonel modifikasyonlar tanımlanması için de kullanılabilir. afinite etiketleri kullanımı Bu protokolde belirtilen ne ile sınırlı değildir; ancak deneyimi ilk AP adım olarak Strep kullanımı ile önemli ölçüde nihai protein-protein kompleksinin verimi artırır göstermektedir. Hem ve STREP boncuklar bağlayıcı ve elüsyon adımlar (örneğin, FLAG) diğer epitop etiketleri ile karşılaştırıldığında son derece verimlidir. Buna ek olarak, teorik olarak, TAP, tampon koşulları, (her ikisi de geçici ve kararlı ifade sistemleri için) deney başına levha sayısı, vb Ayrıca bazı ufak optimizasyonlar ile başka proteinler veya etki tatbik edilebilir, TAP yöntemi de olabilir uygulamalı tdiğer hücre hatları o spesifik protein kompleksleri kendi işlevi veya benzersiz faktörler ile bunların karşılıklı etkileşimi için özel bir sistem gerektiriyorsa. Sorun kolaylaştırmak için, yüksek giriş ve FT örnekler her iki (Tablo 2) kaydedilir önerilir. beklenen protein bantları elüsyon görüntülenmiştir edilemediğinde, FT örnekleri deney giderilir ve numune bağlı ve / veya boncuk akıtılır olan olup olmadığını belirlemek için kullanılabilir.
Başarılı TAP protein ekspresyonu ve kurtarma yüksek düzeylerde sıkı sıkıya bağlıdır. Böylece, araştırmacılar bir vaka-by-case bazında başlangıç malzemesi miktarını optimize etmesi gerekir. Geçici transfeksiyon protein bileşenlerinin ifade etmek için kullanılıyorsa, transfeksiyon verimi TAP ikinci aşamasına geçmeden önce, en az% 70 olması gerekir. (Epitoplar iki yemler arasında takas edildiği) karşılıklı TAP yöntem genellikle hangi deneysel strateji belirlemede yardımcı olurEn iyi saflık ve verim elde etmek için kullanılmalıdır. Yine, bu bir vaka ile ayrı ayrı araştırıldı gerekecektir. Önemli bir şekilde, karşılıklı TAP performans da PPI kullanılan afinite etiketi bağımsız olduğunu teyit etmektedir.
sunulan TAP yönteminin bazı yönleri burada sınırlamaları vardır. Örneğin, FLAG- ve STREP-etiketli proteinlerini aşın ektopik ifade proteini kendisi ya da interactors için eserler yaratabilir. Farklı epitop takıları, protein sentezleme seviyeleri, bir protein konformasyonu ve / veya hücre içi lokalizasyonu etkileyebilir. hücre dizisi ve transfeksiyon verimliliği sınırlama yanı sıra, komplekslerin boyutu, bir sorun olabilir arındırılacak. Bu TAP yöntemi çok alt birim protein kompleksleri için de çalışıyor. Deneyimlerimize göre, dört madde gümüş boya ile tespit edilebilir (veri gösterilmemiştir). İki etiketli bileşenler kullanılarak, ancak, diğer Uygulayıcılar eş saflaştınldı benzer stokiyometrik seviyelerde olabilir. Bu nedenle,Bu özel amaç için, karmaşık bileşenlerin ifade düzeyi düşük uyaran hücre hatlarının kullanılması tavsiye edilir. Başka bir sınırlama ÜFE gücü olacaktır. etkileşimleri yeterince kararlı değilse, bazı bileşenler (özellikle son BAYRAK IP aşamasında) saflaştırma adımları sırasında kaybolmuş olabilir.
TAP yöntemi daha önce açıklanan ve birkaç farklı epitop etiketleri, test edilmiş, doğrulanmış ve 6 karşılaştırıldı. Ancak, bu makalede sunulan TAP yöntemi roman ve PPIs, karmaşık derleme çalışma, ve protein ağı bileşimi ve işlevini anlaması için kolay, verimli, sağlam ve alternatif bir strateji sağlar. Önceki TAP yöntemleri ile karşılaştırıldığında, bu Strep-FLAG TAP stratejisi kompleks bileşim, etkinlik ya da fonksiyonunun bir ön bilgi olmaksızın daha fazla karakterizasyon kompleksler hızlı saflaştırılmasına olanak; ve protein, yıkama için proteaz bölünme (örneğin, TEV) gerek kalmadan (WHich önemli ölçüde) protein verimi azaltabilir. Çünkü artan protein kompleksi iyileşme, Strep-FLAG TAP yöntemi de tandem kütle spektrometresi analizi 10, 15 ile hücrelerde ilgili protein kompleksi ile ilişkilendirmek proteinlerin sağlam tanımlanmasına izin verir. Daha sonra, diğer yöntemler, konfokal mikroskopi, boyut dışlama kromatografisi, ve ortak bir IP deneylerinde olduğu gibi, aynı zamanda PPIs doğrulamak için kullanılabilir. Son olarak, biz TAP protokolünün alternatif versiyonu arındırmak ve müthiş RNA biyoloji alanında araştırmacılar yardımcı olacak ayrık protein-RNA kompleksleri, montajı çalışmaya adapte olabilir öneriyoruz.
Özetle, saflaştırmak ve memeli hücre çizgilerinde PPIs karakterize etmek için bir yöntem sunmaktadır. Biz bu yöntem gelecek karakterizasyonu ve birçok yeni protein kompleksleri fonksiyonel analiz temelini oluşturmaktadır öneriyoruz.
Açıklamalar
The authors have nothing to disclose.
Teşekkürler
Research reported in this publication was supported by the National Institute of Allergy and Infectious Diseases (NIAID) of the NIH under award number R01AI114362 and Welch Foundation grant I-1782 to Iván D'Orso.
Malzemeler
| Name | Company | Catalog Number | Comments |
| Dulbecco's Modified Eagle Medium (DMEM) | GE Healthcare Life Sciences/Hyclone | SH3002.2FS | |
| Fetal bovine serum | GE Healthcare Life Sciences/Hyclone | SH30071 | |
| Penicillin/Streptomycin | MP Biomedicals | MP091670049 | |
| PolyJet | SignaGen Laboratories | SL100688 | |
| Protease inhibitor cocktail | Roche | 11836153001 | |
| STREP-Tactin Superflow | IBA Lifesciences | 2-1208-010 | |
| STREP-tag elution buffer | IBA Lifesciences | 2-1000-025 | |
| EZview Red ANTI-FLAG M2 Affinity Gel | Sigma-Aldrich | F2426 | |
| Corning 100 mm × 20 mm style dish cell culture treated nonpyrogenic polystyrene 20/sleeve | Corning | 430167 | |
| Protein Lo-Bind Eppendorf | Eppendorf | 022431081 | |
| Digital Vortex Mixer | Fisher Scientific | 02-215-370 | |
| 48 hole micro tube foam rack | Fisher Scientific | 02-215-386 | |
| Labquake shaker rotisserie | Thermo | 415110 |
Referanslar
- Sharan, R., Ulitsky, I., Shamir, R. Network-based prediction of protein function. Mol Syst Biol. 3, 88-99 (2007).
- Jager, S., et al. Global landscape of HIV-human protein complexes. Nature. 481, 365-370 (2012).
- Fields, S., Song, O. A novel genetic system to detect protein-protein interactions. Nature. 340, 245-246 (1989).
- Luo, Y., Batalao, A., Zhou, H., Zhu, L. Mammalian two-hybrid system: a complementary approach to the yeast two-hybrid system. Biotechniques. 22, 350-352 (1997).
- Lee, C. Coimmunoprecipitation assay. Methods Mol Biol. 362, 401-406 (2007).
- Puig, O., et al. The tandem affinity purification (TAP) method: a general procedure of protein complex purification. Methods. 24, 218-229 (2001).
- Jager, S., et al. Purification and characterization of HIV-human protein complexes. Methods. 53, 13-19 (2011).
- Price, D. H. P-TEFb, a cyclin-dependent kinase controlling elongation by RNA polymerase II. Mol Cell Biol. 20, 2629-2634 (2000).
- Peterlin, B. M., Price, D. H. Controlling the elongation phase of transcription with P-TEFb. Mol Cell. 23, 297-305 (2006).
- McNamara, R. P., McCann, J. L., Gudipaty, S. A., D'Orso, I. Transcription factors mediate the enzymatic disassembly of promoter-bound 7SK snRNP to locally recruit P-TEFb for transcription elongation. Cell Rep. 5, 1256-1268 (2013).
- Adelman, K., Lis, J. T. Promoter-proximal pausing of RNA polymerase II: emerging roles in metazoans. Nat Rev Genet. 13, 720-731 (2012).
- D'Orso, I. 7SKiing on chromatin: move globally, act locally. RNA Biol. 13, 545-553 (2016).
- McNamara, R. P., Bacon, C. W., D'Orso, I. Transcription Elongation Control by the 7SK snRNP Complex: Releasing the Pause. Cell Cycle. 15, 2115-2123 (2016).
- Yao, F., Svensjo, T., Winkler, T., Lu, M., Eriksson, C., Eriksson, E. Tetracycline repressor, tetR, rather than the tetR-mammalian cell transcription factor fusion derivatives, regulates inducible gene expression in mammalian cells. Hum Gene Ther. 9, 1939-1950 (1998).
- McNamara, R. P., et al. KAP1 Recruitment of the 7SK snRNP Complex to Promoters Enables Transcription Elongation by RNA Polymerase II. Mol Cell. 61, 39-53 (2016).
- Ausubel, F. M., et al. Current Protocols in Molecular Biology. , Greene Publishing Associates and Wiley-Interscience. New York. (1994).
- Trudgian, D. C., et al. Comparative evaluation of label-free SINQ normalized spectral index quantitation in the central proteomics facilities pipeline. Proteomics. 11, 2790-2797 (2011).
- Choudhary, C., Mann, M. Decoding signalling networks by mass spectrometry-based proteomics. Nat Rev Mol Cell Biol. 11, 427-439 (2010).
- Bensimon, A., Heck, A. J., Aebersold, R. Mass spectrometry-based proteomics and network biology. Annu Rev Biochem. 81, 379-405 (2012).
- Rogers, S., Wells, R., Rechsteiner, M. Amino acid sequences common to rapidly degraded proteins: the PEST hypothesis. Science. 234, 364-368 (1986).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır