Method Article
La purification d'affinité en tandem de complexes protéiques à partir des cellules eucaryotes
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
We describe here a novel, robust, and efficient tandem affinity purification (TAP) method for the expression, isolation, and characterization of protein complexes from eukaryotic cells. This protocol could be utilized for the biochemical characterization of discrete complexes as well as the identification of novel interactors and post-translational modifications that regulate their function.
Résumé
La purification de la protéine-protéine active et des complexes protéine-acide nucléique est cruciale pour la caractérisation des activités enzymatiques et la détermination de novo de nouvelles sous - unités et les modifications post-traductionnelles. Les systèmes bactériens permettent l'expression et la purification d'une grande variété de polypeptides simples et complexes protéiques. Cependant, ce système ne permet pas la purification des sous - unités protéiques qui contiennent des modifications post-traductionnelles (par exemple, la phosphorylation et acétylation), et l'identification de nouvelles sous - unités régulatrices qui sont seulement présents / exprimées dans le système eucaryote. Ici, nous fournissons une description détaillée d'un roman, robuste et méthode efficace de purification par affinité en tandem (TAP) en utilisant streptomycine et des protéines FLAG-tagged qui facilite la purification de complexes protéiques avec transitoirement ou exprimé de manière stable des protéines de marquage épitopique à partir de cellules eucaryotes. Ce protocole peut être appliqué pour caractériser protein fonctionnalité complexe, pour découvrir les modifications post-traductionnelles sur des sous-unités complexes, et d'identifier de nouveaux composants réglementaires complexes par spectrométrie de masse. En particulier, cette méthode TAP peut être appliquée à l'étude des complexes protéiques formés par des composants ou eucaryotes pathogènes (virus et bactéries), ce qui donne ainsi un large éventail de possibilités expérimentales en aval. Nous proposons que les chercheurs travaillant avec des complexes de protéines pourraient utiliser cette approche dans de nombreuses façons différentes.
Introduction
Les interactions protéine-protéine (IPP) sont essentiels pour la régulation précise des processus biologiques 1, et d' autres études sur ces IPP peut indiquer leur fonction 2. Plusieurs approches ont été mises au point pour l'étude et la caractérisation des IPP, ainsi que pour l'identification de novo de nouveaux composants régulateurs protéiques. En 1989, les champs et les collègues Stanley a signalé la levure à deux hybrides (Y2H) dosage 3. Cette approche permet l'identification impartiale et complète de interacteurs (proies) pour une protéine d'intérêt définie (appât) dans Saccharomyces cerevisiae. En plus de son utilité pour la découverte remarquable PPIs, le dosage Y2H peut être utilisé pour caractériser des paires de protéines dans des cellules de levure, en définissant des domaines d'interaction minimale, et l'identification des mutations qui suppriment ces interactions. En modifiant le dosage de Y2H, IPP peut également être étudié dans des cellules de mammifèresclass = "xref"> 4. Les variations du dosage Y2H (par exemple, le système à trois hybrides de levure) peuvent également être appliqués à l' étude de protéines et d' ARN interactions ligand organique à petites protéines dans les cellules.
Un autre outil couramment utilisé pour étudier les IPP dans un système homologue est le co-immunoprécipitation (co-IP) dosage 5. En utilisant un anticorps à immunoprécipiter une protéine d'intérêt, le test de co-IP permet aux chercheurs de surveiller IPP dans le traitement des cellules pour diverses conditions environnementales et des situations expérimentales. L'utilisation de protéines de marquage épitopique (par exemple, FLAG, Myc, STREP, et HA, entre autres) dans la purification d'affinité (AP) méthodes a facilité l'isolement de protéines à partir de mélanges de protéines complexes pour plusieurs essais en aval, y compris western blot, tache d' argent et l'analyse enzymatique. Cependant, aucune de ces approches antérieures permettent l'isolement de grandes quantités de complexes de protéines pour une caractérisation ultérieure in vitro , y compris unssays, découverte de sous-unités régulatrices par spectrométrie de masse, et l'identification des modifications post-traductionnelles. Une version améliorée du procédé est appelé AP AP tandem (TAP), qui est une technique de purification pour l' étude des IPP en créant une protéine de fusion avec deux epitopes qui est purifié par deux points d' accès ultérieurs 6, 7. Dans cet article, nous présentons une variante de la méthode de TAP pour les complexes protéiques de purification dans lequel deux sous-unités sont marqués avec différents épitopes, puis purifiés par deux points d'accès séquentiels (STREP AP suivi par FLAG IP). Nous fournissons d' abord un aperçu de la TAP minimalistic (Figure 1), puis une description détaillée de toutes les étapes expérimentales (Figure 2), de sorte que les chercheurs puissent les appliquer à leur complexe de protéine d'intérêt.
Pour démontrer l'applicabilité de la méthode TAP, nous avons choisi une cycline-CDK bien caractérisé complexe (appelé PTEFB kinase), qui est composé de la cycline T1 de sous - unité régulatrice (CycT1) et une kinase (CDK9), et est impliqué dans la régulation de la transcription par l' ARN polymérase II (Pol II) 8, 9, 10. P-TEFb phosphoryle le domaine C-terminal de Pol II et ses associés des facteurs d'élongation négative, ce qui soulage pause de la transcription au niveau du promoteur et facilite ainsi la transcription d' allongement 11, 12, 13. Avec cette interaction connue à l'esprit, STREP-tagged CycT1 et CDK9 FLAG marqués étaient surexprimés dans les cellules HEK293T. Une expérience TAP réciproque a été réalisée avec CDK9 et étiquetée FLAG étiquette strep CycT1 pour valider en outre que l'interaction des protéines est indépendante des épitopes utilisés. Les cellules ont été recueillies et lysées 48 heures après transfection. Le lysat soluble a été purifié par TAP (STREP AP suivie par FLAGIP). D' entrée et de protéines purifiées ont été analysées par Western Blot et coloration à l' argent (figure 3).
Protocole
1. Cellules Placage
- Un jour avant la transfection, la plaque de 3,5 x 10 6 cellules HEK293T dans 10 ml de milieu de Eagle modifié par Dulbecco (DMEM) supplémenté avec 10% (v / v) de sérum de fœtus bovin (FBS) et 1% (v / v) de pénicilline / streptomycine (10 000 U / ml stock) par boîte de 100 mm. Planche 3 - 5 boîtes de 100 mm par expérience / la condition pour assurer la récupération des protéines suffisante.
REMARQUE: Le nombre de plaques doit être déterminé pour chaque expérience / condition, qui reposera sur les niveaux d'expression et de la solubilité du complexe de protéine d'intérêt. Par ailleurs, si la purification à plus grande échelle est nécessaire, les lignées cellulaires peuvent être adaptées pour se développer dans des suspensions dans des flacons spinner 2, mais cette méthode ne fonctionnera pas pour les transfections transitoires.
2. transfection de cellules ou Induire lignées cellulaires stables
- Le lendemain, vérifier les cellules au microscope. Les cellules doivent être de 70 - 80% confluentes avant proceeding.
- Transfecter les cellules avec un mélange total de 7 pg d'ADN de plasmide et 21 ul de réactif de transfection par boîte de 100 mm. Transfecter des cellules avec un vecteur exprimant la GFP (Tableau 1) en tant que contrôle pour l' efficacité de la transfection.
Remarque: Si l'on utilise un HEK293 T-Rex ou une ligne semblable cellulaire stable qui induit l'expression de deux ou plus complexes des sous - unités en réponse à la doxycycline 14, 15, l'inducteur devra être ajouté aux cellules et incubé pendant 48 heures. De même, une lignée cellulaire stable qui induit l'expression de la GFP lors de l'addition de l'inducteur serait également requis à des fins de validation. Passez à l'étape 2.7. - Préparer le mélange de transfection par boîte de 100 mm. Dans un tube de 1,5 ml, mélanger 500 pi de DMEM non supplémenté avec 7 pg d'ADN plasmidique totale (correspondant à deux ou plusieurs plasmides). Dans un tube de microcentrifugeuse, mélanger 500 pi de DMEM non supplémenté wie 21 pi du réactif de transfection. Répétez l'opération pour chaque réaction de transfection.
- Pipeter la solution de réactif de transfection dans la solution d'ADN du plasmide (ne solutions dans l'ordre inverse de mélange). Mélanger par pipetage au moins 4 fois.
- Faire incuber ce mélange à la température ambiante pendant 10 à 15 minutes, mais pas plus de 15 min.
- Inclinez la plaque pour créer un réservoir de médias et de la pipette le mélange de transfection goutte à goutte sur le côté intérieur de la plaque sans déranger les cellules avec soin. Remettre le plat à sa position originale plat et agiter doucement pour distribuer le mélange.
- Incuber les cellules à 37 ° C dans un incubateur humidifié de culture cellulaire avec 5% de CO 2 (cette condition est appelé ci - après "incubateur de culture tissulaire»). Remplacer les médias 5 heures après la transfection (en option).
3. Vérification Transfection ou Induction efficacité
- À 24 h post-transfection, visualiser les cellules sous une grippemicroscope orescent pour vérifier l'efficacité de transfection (ou l'induction de contrôle HEK293T-REx lignée cellulaire stable exprimant la GFP). Incuber pendant 24 h.
4. STREP Affinity Purification (STREP AP)
- La collecte des cellules
- A 48 heures post-transfection, retirez le support et ajouter 8 ml de solution saline 1x froide tamponnée au phosphate (PBS). Pipeter vers le haut et vers le bas pour détacher les cellules de la plaque.
- Recueillir et transférer la suspension de cellules dans un tube de 15 ml et tourner les cellules vers le bas à 800 g pendant 4 min à 4 ° C.
- Retirer le surnageant PBS. Répéter la rotation vers le bas (800 g pendant 1 min à 4 ° C) pour éliminer tout PBS résiduel. Stocker le culot cellulaire à -80 ° C pour une utilisation ultérieure ou suivez la procédure ci-dessous pour continuer la purification des protéines.
- Lyser les cellules
- Remettre en suspension les culots cellulaires à l'aide de 5 x le volume du culot cellulaire (~ 250 ul par plaque) de tampon de lyse passive(PLB: 20 mM Tris-HCl pH 7,5, 150 mM de NaCl, 1,5 mM de MgCl2, 1 mM de DTT, de la protéase comprimé inhibiteur de cocktail, 5% v / v glycérol et 1,0% de NP-40) et à transférer la suspension cellulaire à un 1,5 ml de tube de microcentrifugeuse.
- vortex brièvement la suspension et nutation pendant 30 min à 4 ° C.
- Isoler la suspension cellulaire à 10.000 xg pendant 10 min à 4 ° C.
- Transférer les lysats solubles dans un nouveau tube de 1,5 ml et jeter les culots cellulaires. Économisez 10% du volume final du lysat soluble "STREP AP entrée." Conserver à 4 ° C.
- Équilibrant les perles de STREP
NOTE: perles Utilisez STREP pour la première étape AP! Complexes tiré vers le bas avec des perles de STREP récupérer beaucoup plus de protéines que celles tiré vers le bas avec des perles de FLAG.- Sortez (n × 40 pi) + n pl de la STREP perle bouillie à 50% (n = nombre d'échantillons) et centrifuger à 1500 g pendant 2 min à 4 ° C.
REMARQUE: Utilisez l'échelle mconseils outhed à perles de pipette. - Retirer le surnageant et ajouter 500 pi de PLB aux billes. Nutation le mélange des perles pendant 5 min à 4 ° C. Isoler à 1500 g pendant 2 min à 4 ° C.
- Répétez l'opération pour un total de 3 lavages. Remettre en suspension les billes dans PLB à un volume final de 40 ul nx.
- Ajouter 40 ul de 50% STREP bourrelet suspension pour chaque échantillon de lysat cellulaire et nutation pendant 2 heures à 4 ° C.
- Sortez (n × 40 pi) + n pl de la STREP perle bouillie à 50% (n = nombre d'échantillons) et centrifuger à 1500 g pendant 2 min à 4 ° C.
- STREP AP
- Isoler solution à 1500 g pendant 2 min à 4 ° C.
- Retirer le surnageant et le magasin comme «STREP AP accréditives (FT)" à 4 ° C.
- Ajouter 500 pi de tampon de lavage STREP AP I (Tris-HCl 20 mM , pH 7,5, 250 mM de NaCl, 1,5 mM de MgCl2, 1 mM de DTT, 5% de glycerol et 0,2% de NP-40) pour les billes. Nutation le mélange des perles pendant 5 min à 4 ° C et centrifuger à 1500 g pendant 2 min à 4 ° C. Jeter le surnageant.
- Répétez l'opération pour un total de 4 lavages.
NOTE: Avant le quatrième lavage, transférer la solution de perles à un nouveau tube de 1,5 ml pour réduire l'arrière-plan de la protéine. Cette étape est essentielle.
- Répétez l'opération pour un total de 4 lavages.
- Ajouter 500 pi de tampon de lavage II (100 mM de Tris-HCl, pH 8,0, NaCl 150 mM et EDTA 1 mM) et équilibrer les billes en inversant les tubes. Isoler à 1500 g pendant 2 min à 4 ° C et jeter le surnageant.
- Éluer la protéine d'intérêt avec 50 pi de tampon d'élution STREP (Tris-HCl 100 mM, pH 8,0, NaCl 150 mM, EDTA 1 mM et 2,5 mM desthiobiotine). Vortex à 800 rpm pendant 15 min à 4 ° C.
- Isoler les perles à 1500 g pendant 1 min à 4 ° C. Recueillir le surnageant. Cette fraction correspond à l'échantillon "STREP AP Elution».
- Enregistrer la fraction de perles à 4 ° C pendant une deuxième élution, ce qui pourrait conduire à la moitié de la quantité de protéine obtenue avec la première élution. Aliquote de 10% du PA STREP Elution pour la coloration d'argent et / ou le transfert de western16.
- Équilibrant les perles FLAG
- Sortez (nx 16 pi) + n pl de solution de perles de FLAG (n = nombre d'échantillons) et centrifuger à 1500 g pendant 2 min à 4 ° C.
REMARQUE: Utilisez des conseils à large ouverture à des billes de pipettes. - Eliminer le surnageant et ajouter 500 pi de tampon de lavage FLAG (100 mM de Tris-HCl, pH 8,0, NaCl 150 mM, EDTA 1 mM et 0,1% de NP-40) pour les billes. Isoler à 1500 g pendant 2 min à 4 ° C. Répétez l'opération pour un total de 3 lavages.
- Resuspendre les perles dans un tampon de lavage FLAG à un volume final de 16 ul nx.
- Ajouter 16 ul du FLAG bourrelet suspension pour le reste élution STREP AP et nuit nutation à 4 ° C.
- Sortez (nx 16 pi) + n pl de solution de perles de FLAG (n = nombre d'échantillons) et centrifuger à 1500 g pendant 2 min à 4 ° C.
5. FLAG IP
- Isoler la suspension des perles de FLAG à 1500 g pendant 2 min à 4 ° C. Enregistrez le surnageant et stocker le «FLAG IP accréditives" (FT) à 4 ° C.
- Ajouter 50081; l de lavage FLAG tampon à la solution. Nutation pendant 5 min à 4 ° C et centrifuger à 1500 g pendant 2 min à 4 ° C. Répétez l'opération pour un total de 4 lavages et retirer le surnageant après chaque lavage.
NOTE: Avant le quatrième lavage, transférer la solution de protéines-billes à un nouveau tube de 1,5 ml pour réduire fond. Cette étape est essentielle. - Retirer le surnageant de l'essorage final et ajouter 30 pi de tampon de lavage FLAG contenant 200 ng / ul de peptide FLAG dans les perles. Vortex à 800 rpm pendant 2 heures à 4 ° C.
- Isoler la suspension à 1500 g pendant 2 min à 4 ° C. Récolter le surnageant et conserver à 4 ° C comme le "FLAG Elution IP."
REMARQUE: Les échantillons d'élution peut être confirmée par coloration à l' argent et / ou Western Blot en utilisant des protocoles standard 16, et sont prêts pour d' autres essais de protéines. Lors de la mise en place d'un western blot, exécutez tous les échantillons suivants: (1) STREP AP Entrée, (2) STREP AP FT, (3) STREP AP Elutisur, (4) FLAG IP FT, et (5) FLAG IP Elution. Les volumes chargés devront être déterminées pour chaque expérience. Tous les échantillons collectés et billes peuvent être conservées à 4 ° C pour le stockage à court terme et à -80 ° C pour le stockage à long terme. Des échantillons de TAP peuvent être analysés par spectrométrie de masse pour vérifier l'identité de leurs composants, afin d' identifier de nouvelles modifications post-traductionnelles (PTM) et / ou pour découvrir toute nouvelle interaction de la protéine purifiée avec le complexe 2, 17. A cet effet, après l'exécution d'un gel ou d'argent Coomassie, les bandes peuvent être excisées à partir du gel en utilisant un scalpel propre. Plusieurs excellentes critiques qui traitent des méthodes de spectrométrie de masse ont déjà été publiés 18, 19.
Résultats
Dans cet article, nous démontrons l'applicabilité de la méthode TAP pour la CycT1-CDK9 bien caractérisé complexe (également connu sous le nom P-TEFb kinase).
Plasmides codant pour la cycline T1-STREP (CycT1: S) et CDK9-FLAG (CDK9: F), ou CDK9-STREP (CDK9: S) et cycline T1-FLAG (CycT1: F) (tableau 1), ont été transfectées dans des cellules HEK293T . Les contrôles négatifs inclus transfections avec un vecteur vide et CDK9: F ou CycT1: F plasmides (Figures 3A et B, respectivement). Les protéines ont été exprimées pendant 48 heures avant que les cellules ont été recueillies. Plaques multiples de cellules ont été utilisées par expérience (cinq plaques par échantillon ont été utilisées pour les données présentées dans la figure 3) pour augmenter la production de protéines et de récupération. Après que les cellules ont été lysées dans des microtubes individuels, les lysats cellulaires du même échantillon ont été combinées en une microcentrifuge tube avant l'addition des billes de STREP. Combiner les échantillons de protéines avec une partie des perles augmente la concentration finale en protéine, ce qui peut améliorer la récupération des protéines à partir de protéines qui sont difficiles à exprimer. À la suite de l'élution initiale, une deuxième élution peut être effectuée afin d'augmenter l'apport en protéines totales pour le drapeau IP. Il est important d'enregistrer l'entrée pour chaque étape de purification avant d' ajouter les perles pour le dépannage futur (tableau 2), si nécessaire.
Les données sur la figure 3A montre les résultats de la méthode de la TAP pour CycT1: S et CDK9: F, analysée par Western Blot. CDK9 F co-élué avec CycT1: S à partir des perles de STREP, mais pas dans le PA témoin négatif dépourvu CycT1: S, ce qui indique que CDK9: F et CycT1: S forment un complexe protéine-protéine. Expectedly, les deux protéines ont été détectées dans l'élution de FLAG, qui a confirmé en outre la formation d'un complexe. La figure 3B montre l'results de la méthode réciproque de TAP, où les marqueurs d'épitopes des deux protéines ont été échangés (CDK9: S et CycT1: F), et analysé par western blot.
Les figures 3C et 3D montrent les résultats de la TAP et TAP réciproque analysé par coloration à l' argent, respectivement. La figure 3C piste 2 montre que CDK9 F co-élué avec CycT1: S hors des perles de STREP, mais pas à partir du point d' accès de commande (voie 1), indiquant ainsi que CDK9: F et CycT1: S forment un complexe protéine-protéine. Dans le FLAG élution subséquente, comme le montre la ligne 4, CycT1: S co-élue avec CDK9: F off des perles de FLAG. De même, la figure 3D montre que CycT1 F co-élué avec CDK9: S hors des perles de STREP, mais pas à partir du point d' accès de commande, et que le complexe protéine-protéine est récupérée de manière efficace après la deuxième étape DRAPEAU IP. Tant les western blots et les taches d'argent ont confirmé que le protocole TAP travaille efficacement pour la purificatio n, l'isolement et la caractérisation du complexe de protéine d'intérêt.

Figure 1: Vue d' ensemble de la stratégie TAP Purification. Un schéma simple décrivant les étapes de TAP. Plasmides codant pour la protéine d'intérêt avec soit l'épitope balise STREP ou FLAG sont co-transfectés dans des cellules de mammifères. Après 48 h d'induction, les cellules sont collectées et lysées. lysat cellulaire soluble contenant la protéine d'intérêt ainsi que d'autres protéines sont chargées sur des billes de STREP pour STREP AP. Les protéines qui contiennent soit la balise STREP ou sont liés à la protéine STREP-tagged attaché aux billes sont ensuite élues. Les perles de FLAG ont ensuite été ajoutés dans l'élution de STREP. Les protéines qui contiennent soit l'étiquette FLAG ou sont liés à la protéine FLAG-tagged attaché aux billes sont ensuite élues. Elutions pourrait être analysé par les méthodes indiquées.ttp: //ecsource-jove-com.remotexs.ntu.edu.sg/files/ftp_upload/55236/55236fig1large.jpg "target =" _ blank "> S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
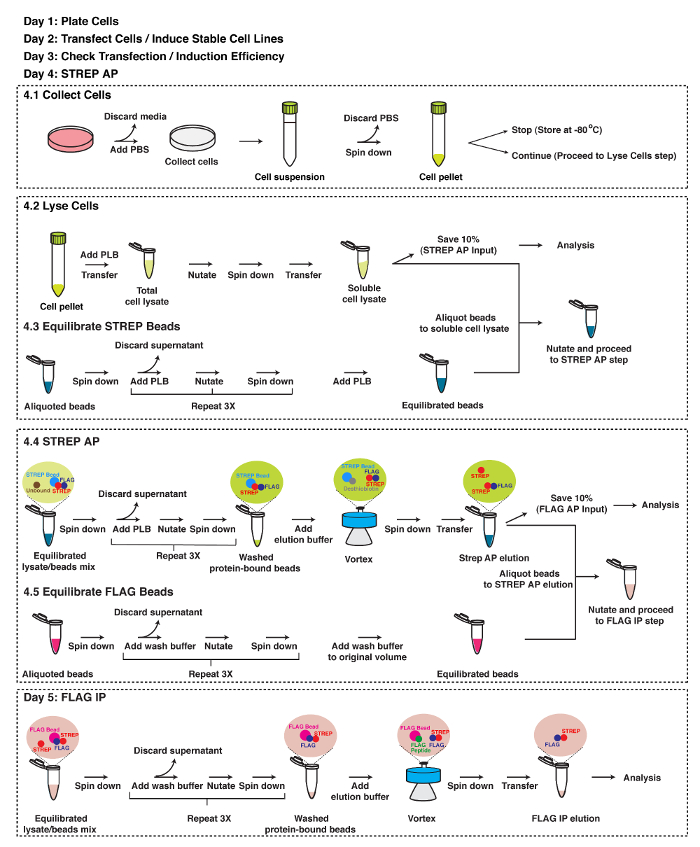
Figure 2: Représentation schématique de la stratégie de TAP. Un protocole détaillé de la TAP. Voir le texte pour les détails. S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
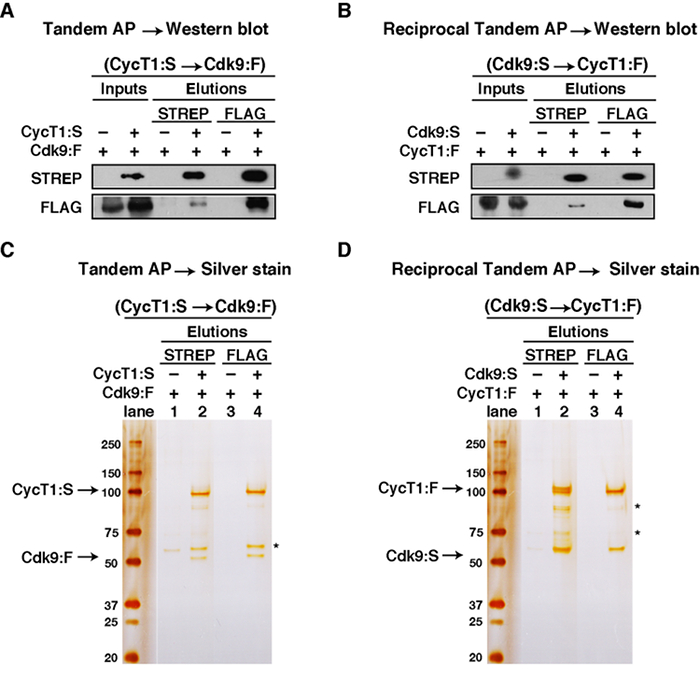
Figure 3: Exemple d'analyse étape par étape de TAP Purification. (A) d'analyse par Western blot de TAP (CycT1: S et CDK9: F). (B) Western analyse par transfert de TAP réciproque (CDK9: S et CycT1: F). From 1 ml de lysat de cellules entières, 5 ul (0,2%) ont été chargés comme "STREP AP Input". 975 pi de lysat de cellules entières restantes ont été ajoutés aux billes de STREP pour STREP AP et 100 pi de «STREP AP élution" a été recueillie. De là, 5 ul (2%) a été chargé. 80 ul de la "STREP AP Elution" a été ajouté à des billes de FLAG pour FLAG IP et 30 pl de "DRAPEAU IP Elution" a été collectées. A partir de 30 pi de «DRAPEAU IP Elution", 7 pi (23,3%) a été chargé. (C) Silver analyse des taches de TAP (CycT1: S et CDK9: F). Analyse (D) Argent tache de TAP réciproque (CDK9: S et CycT1: F). 5 ul (environ 2%) de "STREP AP Elution» et 7 ul (23,3%) de "DRAPEAU IP Elution» ont été chargés. Les astérisques indiquent les impuretés co-élués. S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
| Protéine | résidus | Vecteur | Marque | Des sites de clonage | Référence |
| CycT1 | 1-708 | pcDNA / 4TO | STREP | HindIII - EcoRI | McNamara et al. 2013 10 |
| CycT1 | 1-708 | pcDNA / 4TO | DRAPEAU | HindIII - EcoRI | Cet article |
| CDK9 | 1-372 | pcDNA / 4TO | STREP | HindIII - Xhol | Cet article |
| CDK9 | 1-372 | pcDNA / 4TO | FLAG | HindIII - Xhol | McNamara et al., 2013 10 |
| GFP | 1-238 | pcDNA / 4TO | STREP-FLAG | BamHI - Xhol | Cet article |
Tableau 1: Plasmides utilisés dans cette étude. CycT1 et CDK9 ont été ligaturés dans le pcDNA / 4TO-STREP et pcDNA / 4TO-FLAG vecteurs plasmidiques. Ces constructions ont été transformées en cellules compétentes DH5a et plaquées sur des plaques LB-ampicilline. Les colonies ont été sélectionnées, cultivées, et criblées. Les clones avec succès ligaturés ont été vérifiés par séquençage de Sanger, et la combinaison d'une digestion de restriction et électrophorèse sur gel d'agarose. Pour CycT1, nous avons utilisé une version plus courte (1 - 708) au lieu de la pleine longueur (1 - 726) parce que les 18 derniers résidus contiennent une séquence PEST (une séquence peptidique riche enla proline, l' acide glutamique, la serine et la threonine), qui agit comme un peptide signal pour la dégradation des protéines 20. CDK9 était pleine longueur. La séquence de l'étiquette tandem STREP est la suivante: GGGGWSHPQFEKGGGSGGGSGGGSWSHPQFEK. La séquence de l'étiquette tandem FLAG est la suivante: GGGGDYKDHDGDYKDHDIDYKDDDDK.
Tableau 2: Tableau de dépannage. Avant de commencer le dépannage, une double vérification pour faire en sorte que chaque étape essentielle a été suivie. Certaines étapes de ce protocole nécessitent plus d'optimisation que d'autres pour obtenir le résultat escompté. S'il vous plaît cliquer ici pour télécharger ce fichier.
Discussion
Le protocole décrit ici pour l'expression et l'isolement des complexes de protéines à partir de cellules eucaryotes ne se limite pas à la caractérisation biochimique de ces assemblages moléculaires, mais il peut également être utilisé pour l'identification de nouveaux interacteurs et les modifications post-traductionnelles qui pourraient réguler leur fonctionnement. L'utilisation de marqueurs d'affinité ne se limite pas à ce qui est mentionné dans ce protocole; mais notre expérience montre que l'utilisation de STREP comme la première étape AP améliore significativement le rendement de la finale complexe protéine-protéine. Les deux étapes de liaison et d' élution vers et à partir des perles de STREP sont extrêmement efficaces par rapport aux autres marqueurs d'épitope (par exemple, FLAG). En outre, en théorie, la TAP pourrait être appliquée à d' autres protéines ou domaines avec certaines optimisations mineures, telles que des conditions de tampon, le nombre de plaques par expérience (pour les deux systèmes exprimant transitoires et stables), etc. En outre, le procédé TAP peut également être appliquée to d'autres lignées cellulaires si complexes protéiques spécifiques nécessitent un système spécial pour leur fonction ou leurs interactions avec des facteurs uniques. Pour faciliter le dépannage, il est fortement recommandé que les échantillons d'entrée et de FT deux être enregistrés (tableau 2). Lorsque les bandes de protéines attendues ne peuvent pas être visualisées dans l'élution, les échantillons FT peuvent être utilisés pour résoudre les problèmes de l'expérience et de déterminer si oui ou non l'échantillon était lié à et / ou élué hors des perles.
Une TAP réussie est strictement dépendante des niveaux élevés d'expression de la protéine et la récupération. Ainsi, les chercheurs doivent optimiser la quantité de matière de départ sur une base au cas par cas. Si la transfection transitoire est utilisée pour exprimer les composants protéiques, l'efficacité de transfection doit être d'au moins 70% avant de procéder à la deuxième étape du TAP. La méthode TAP réciproque (dans lequel les épitopes sont échangés entre les deux amorces) permet généralement de déterminer quelle stratégie expérimentaledoit être utilisé pour obtenir le meilleur rendement et la pureté. Encore une fois, cela devra être étudié au cas par cas. Fait important, la réalisation TAP réciproque confirme également que le PPI est indépendant de l'étiquette d'affinité utilisé.
Certains aspects de la méthode TAP présentés ici ont des limites. Par exemple, surexprimant le FLAG et les protéines de STREP marqués pourrait générer des artefacts pour la protéine ectopique exprimé lui-même ou leurs interacteurs. marqueurs épitopiques différents peuvent affecter les niveaux d'expression des protéines, la conformation des protéines, et / ou la localisation subcellulaire. En plus de la limitation de la lignée cellulaire et de l'efficacité de la transfection, la taille des complexes à purifier peut être un problème. Cette méthode TAP fonctionne bien pour des complexes de protéines sous-unités multiples. D'après notre expérience, quatre composants peuvent être détectés par coloration à l'argent (données non présentées). Cependant, lors de l'utilisation de composants à deux marqués, les autres interacteurs peuvent ne pas être co-purifié à des niveaux stoechiométriques identiques. Donc,pour atteindre cet objectif particulier, l'utilisation de lignées cellulaires qui induit des niveaux inférieurs d'expression des composants complexes est conseillé. Une autre limitation serait la force du PPI. Si les interactions ne sont pas assez stables, certains composants pourraient être perdus au cours des étapes de purification (en particulier dans l'étape finale IP FLAG).
La méthode TAP a été décrit précédemment et plusieurs marqueurs d'épitopes différents ont été testés, validés et comparés 6. Cependant, la méthode TAP présentée dans cet article est nouveau et offre une stratégie simple, efficace, robuste et alternative à étudier PPIs, assemblage complexe, et en déduire la composition des réseaux de protéines et la fonction. Par rapport aux méthodes précédentes de TAP, cette stratégie de TAP STREP-FLAG permet la purification rapide de complexes pour une caractérisation plus poussée sans connaissance préalable du complexe composition, activité, ou de la fonction; et sans qu'il soit nécessaire de clivage de la protéase (par exemple, TEV) pour l' élution des protéines (which peut diminuer de façon significative le rendement en protéines). En raison de la récupération accrue de protéine complexe, le procédé TAP STREP-FLAG permet également l'identification robuste des protéines qui associent avec le complexe de protéine d'intérêt dans des cellules par une analyse par spectrométrie de masse en tandem 10, 15. Par la suite, d'autres procédés, comme la microscopie confocale, la chromatographie d'exclusion stérique, et les essais de co-IP, peuvent également être utilisées pour valider les IPP. Enfin, nous proposons qu'une version alternative du protocole TAP pourrait être adapté pour purifier et étudier l'assemblage des complexes protéines-ARN discrets, qui énormément aider les chercheurs dans le domaine de la biologie ARN.
En résumé, nous proposons un procédé pour purifier et caractériser les IPP dans des lignées cellulaires de mammifères. Nous proposons que cette méthode pose les bases pour la caractérisation future et de l'analyse fonctionnelle de nombreux complexes protéiques nouveaux.
Déclarations de divulgation
The authors have nothing to disclose.
Remerciements
Research reported in this publication was supported by the National Institute of Allergy and Infectious Diseases (NIAID) of the NIH under award number R01AI114362 and Welch Foundation grant I-1782 to Iván D'Orso.
matériels
| Name | Company | Catalog Number | Comments |
| Dulbecco's Modified Eagle Medium (DMEM) | GE Healthcare Life Sciences/Hyclone | SH3002.2FS | |
| Fetal bovine serum | GE Healthcare Life Sciences/Hyclone | SH30071 | |
| Penicillin/Streptomycin | MP Biomedicals | MP091670049 | |
| PolyJet | SignaGen Laboratories | SL100688 | |
| Protease inhibitor cocktail | Roche | 11836153001 | |
| STREP-Tactin Superflow | IBA Lifesciences | 2-1208-010 | |
| STREP-tag elution buffer | IBA Lifesciences | 2-1000-025 | |
| EZview Red ANTI-FLAG M2 Affinity Gel | Sigma-Aldrich | F2426 | |
| Corning 100 mm × 20 mm style dish cell culture treated nonpyrogenic polystyrene 20/sleeve | Corning | 430167 | |
| Protein Lo-Bind Eppendorf | Eppendorf | 022431081 | |
| Digital Vortex Mixer | Fisher Scientific | 02-215-370 | |
| 48 hole micro tube foam rack | Fisher Scientific | 02-215-386 | |
| Labquake shaker rotisserie | Thermo | 415110 |
Références
- Sharan, R., Ulitsky, I., Shamir, R. Network-based prediction of protein function. Mol Syst Biol. 3, 88-99 (2007).
- Jager, S., et al. Global landscape of HIV-human protein complexes. Nature. 481, 365-370 (2012).
- Fields, S., Song, O. A novel genetic system to detect protein-protein interactions. Nature. 340, 245-246 (1989).
- Luo, Y., Batalao, A., Zhou, H., Zhu, L. Mammalian two-hybrid system: a complementary approach to the yeast two-hybrid system. Biotechniques. 22, 350-352 (1997).
- Lee, C. Coimmunoprecipitation assay. Methods Mol Biol. 362, 401-406 (2007).
- Puig, O., et al. The tandem affinity purification (TAP) method: a general procedure of protein complex purification. Methods. 24, 218-229 (2001).
- Jager, S., et al. Purification and characterization of HIV-human protein complexes. Methods. 53, 13-19 (2011).
- Price, D. H. P-TEFb, a cyclin-dependent kinase controlling elongation by RNA polymerase II. Mol Cell Biol. 20, 2629-2634 (2000).
- Peterlin, B. M., Price, D. H. Controlling the elongation phase of transcription with P-TEFb. Mol Cell. 23, 297-305 (2006).
- McNamara, R. P., McCann, J. L., Gudipaty, S. A., D'Orso, I. Transcription factors mediate the enzymatic disassembly of promoter-bound 7SK snRNP to locally recruit P-TEFb for transcription elongation. Cell Rep. 5, 1256-1268 (2013).
- Adelman, K., Lis, J. T. Promoter-proximal pausing of RNA polymerase II: emerging roles in metazoans. Nat Rev Genet. 13, 720-731 (2012).
- D'Orso, I. 7SKiing on chromatin: move globally, act locally. RNA Biol. 13, 545-553 (2016).
- McNamara, R. P., Bacon, C. W., D'Orso, I. Transcription Elongation Control by the 7SK snRNP Complex: Releasing the Pause. Cell Cycle. 15, 2115-2123 (2016).
- Yao, F., Svensjo, T., Winkler, T., Lu, M., Eriksson, C., Eriksson, E. Tetracycline repressor, tetR, rather than the tetR-mammalian cell transcription factor fusion derivatives, regulates inducible gene expression in mammalian cells. Hum Gene Ther. 9, 1939-1950 (1998).
- McNamara, R. P., et al. KAP1 Recruitment of the 7SK snRNP Complex to Promoters Enables Transcription Elongation by RNA Polymerase II. Mol Cell. 61, 39-53 (2016).
- Ausubel, F. M., et al. Current Protocols in Molecular Biology. , Greene Publishing Associates and Wiley-Interscience. New York. (1994).
- Trudgian, D. C., et al. Comparative evaluation of label-free SINQ normalized spectral index quantitation in the central proteomics facilities pipeline. Proteomics. 11, 2790-2797 (2011).
- Choudhary, C., Mann, M. Decoding signalling networks by mass spectrometry-based proteomics. Nat Rev Mol Cell Biol. 11, 427-439 (2010).
- Bensimon, A., Heck, A. J., Aebersold, R. Mass spectrometry-based proteomics and network biology. Annu Rev Biochem. 81, 379-405 (2012).
- Rogers, S., Wells, R., Rechsteiner, M. Amino acid sequences common to rapidly degraded proteins: the PEST hypothesis. Science. 234, 364-368 (1986).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon