Method Article
Эндогенного белка, пометки в человека индуцированных плюрипотентных стволовых клеток с использованием ТРИФОСФАТЫ/Cas9
В этой статье
Резюме
Описанные здесь является протокол для пометки эндогенно выразил белки с флуоресцентные метки в человеческое индуцированных плюрипотентных стволовых клеток с использованием ТРИФОСФАТЫ/Cas9. Предположительно отредактированных ячеек обогащены сортировки клеток активированных флуоресцированием и клоновых клеточных линий генерируются.
Аннотация
Протокол представляется для генерации человеческое индуцированных плюрипотентных стволовых клеток (hiPSCs), которые Экспресс эндогенного белки сливается с N - или C-терминал флуоресцентные метки в кадр. Прокариот ТРИФОСФАТЫ/Cas9 системы (кластерный регулярно interspaced короткие палиндром повторяется/ТРИФОСФАТЫ связанные 9) может использоваться для внедрения крупных внешних последовательности в геномной локусов через гомологии направлены ремонт (HDR). Для достижения желаемого забивные, этот протокол использует рибонуклеопротеида (RNP)-на основе подхода, где дикого типа Streptococcus pyogenes Cas9 белков, синтетические 2-часть руководство РНК (gRNA) и доноров шаблон плазмида доставляются в клетки через Электропорация. Предположительно измененные клетки, выражая дневно тегами белки обогащены клеток активированных флуоресцированием сортируя (FACS). Клоновых линий затем генерируются и могут быть проанализированы для точного редактирования результатов. Путем введения флуоресцентных тег в геномной Локус гена интереса, результирующий субцеллюлярные локализации и динамика синтез белка могут быть изучены под эндогенного регулирования, ключевым улучшение по сравнению с обычными гиперэкспрессия систем. Использование hiPSCs как модель системы для маркировки генов предоставляет возможность изучить тегами белков в клетках диплоидных, nontransformed. Поскольку hiPSCs может быть дифференцирована на несколько типов клеток, такой подход обеспечивает возможность создания и изучения тегами белков в различных isogenic клеточных контекстах.
Введение
Использование изменения генома стратегий, особенно ТРИФОСФАТЫ/Cas9, изучение клеточных процессов становится все более доступным и ценные в1,2,3,4,5, 6 , 7. один из многих приложений ТРИФОСФАТЫ/Cas9 является введение (через гомологии направлены ремонт (HDR)) больших внешних последовательности, например GFP в конкретных геномной локусов, которые затем служить репортеров для активности гена или протеин продукт8 . Этот метод может использоваться присоединиться флуоресцентный белок последовательность кадре эндогенного открытом чтения где результате эндогенно регулируемых синтез белка может использоваться для визуализации субцеллюлярные локализации и динамика протеина интереса5 ,6,9,10,11. В то время как эндогенно тегами белки предлагают много преимуществ по сравнению с системами гиперэкспрессия, вставки больших последовательностей генома человека является неэффективным процесс, обычно требуют выделения или обогащения стратегию для получения популяция клеток, которые могут быть легко изучал5,12.
Этот протокол описывает вставку последовательности ДНК кодирования флуоресцентный белок (FP) в желаемой геномной Локус. Протокол включает в себя проектирование и поставка плазмида шаблон доноров и рибонуклеопротеида (RNP) комплекс (дикого типа S. pyogenes Cas9 белка в сочетании с синтетическими ТРИФОСФАТЫ РНК (crRNA) и транс активация crRNA (tracrRNA)). Также описано это обогащение предположительно отредактированных ячеек через клеток активированных флуоресцированием сортируя (FACS) и процесс создания линии клоновых ячейки. На сегодняшний день, этот метод был использован для создания hiPSC линии с monoallelic или (редко) Би аллельные Зеленый флуоресцентный белок (ГПУП) Теги маркировки двадцать пять белки, представляющих основных клеточных структур. Результате отредактированных ячеек из этих усилий были подтверждены ожидаемых генетических вставки, Экспресс правильно локализация синтез белка, и поддержания плюрипотентности и стабильной кариотипа12 (и неопубликованные данные). Этот метод также используется для создания нескольких других одним и двумя (два различных белков, отмеченных в той же ячейке) редактировать популяций hiPSCs (неопубликованные данные).
Человеческого iPSCs, полученные от здоровых доноров были выбраны для эти изменения генома усилия, потому что, в отличие от многих обычных клеточных линий, они являются диплоидными, karyotypically стабильной, -трансформированных и пролиферативной. Эти свойства предоставляют привлекательная модель для изучения фундаментальных клеточной биологии и моделирование болезней. Кроме того дифференциация потенциал hiPSCs предоставляет возможность изучить несколько этапы развития параллельно различных линий и типы клеток с использованием isogenic клеток, включая organoids, тканей и моделей «болезни в блюдо»13 ,14,15. Хотя этот протокол был разработан для hiPSCs (WTC линия), это может быть информативным для разработки протоколов, с помощью других линий клеток млекопитающих.
протокол
1. в Silico дизайн crRNA и доноров шаблоне плазмиды для FP стук в
- Получить последовательность аннотированный справочник NCBI16 или геноме UCSC Browser17 (например, GenBank формат) гена интереса и импортировать его в биоинформатике программного выбора. Если известно, что последовательность генома узла содержат варианты относительно ссылки, включают те, теперь, регулируя ссылка последовательности в биоинформатике программного обеспечения (см. обсуждение).
- Найдите требуемый FP вставки сайт. Для C-терминала тегов последовательность для FP тега будут введены между последней базе последнего кодон и первой базы стоп-кодон. Для N-терминальный пометки последовательность для FP тега обычно будут вводиться между последней база начала кодон и первой базы следующего кодон. В некоторых случаях, например когда начала кодон экзона один кодон, или где последовательность сигнала существует рядом с конечной остановкой белка желаемый сайт FP вставки могут быть расположены в более 3ʹ позиции, при условии, что она продолжает быть в кадре.
- Использование 50 bp на каждой стороне требуемой вставки сайт как входной последовательности для любой общедоступной crRNA дизайн инструмента. После 2-4 crRNA цели определяются рядом вставки сайте, аннотируйте сайтов связывания crRNA и protospacer прилегающих мотив (PAM) последовательностей (НЭС) в программном обеспечении биоинформатики. Эти crRNAs будут использоваться побудить двойной мель перерывы (см. обсуждение для более руководства на crRNA дизайн).
Примечание: Пользовательские crRNA последовательности могут быть представлены для синтеза с коммерческим поставщиком (рекомендуется), или последовательность может использоваться как отправной точки для разработки клонирования или в пробирке синтеза стратегию, которая выходит за рамки настоящего Протокола (см. обсуждение ). - Инициировать доноров шаблоне плазмиды, использовать 1 КБ последовательности вверх по течению требуемой вставки сайт как 5ʹ гомологии руки (это должно включать начала кодоном для N-терминальный вставок) и использовать 1 КБ последовательности по течению требуемой вставки сайт как 3ʹ гомологии руки (это должно включать остановки кодоном для C-терминала вставок). Обычно базы между двумя ветвями гомология не пропускаются. Включая линии клетки конкретные варианты в руках гомологии сохранит эти генетические варианты в результате отредактированных ячейках.
- Между двумя ветвями гомологии, вставьте в последовательности для FP (или другие стук в последовательности) и компоновщик последовательности (см. обсуждение для более руководства на линкеры). Для N-терминальный тегов компоновщик последовательность должна быть непосредственно 3ʹ FP; для C-терминала тегов компоновщик последовательность должна быть непосредственно 5ʹ FP.
- Нарушить сайтов связывания crRNA в шаблон плазмида доноров для предотвращения Cas9 резки доноров последовательности (см. обсуждение соображений при изменении привязки сайтов crRNA). Если возможно нарушение PAM в последовательность помимо NGG или NAG является предпочтительным. Кроме того представляя точечные мутации до трех баз в регионе семян crRNA (10 баз проксимальнее PAM) прогнозируется достаточно сорвать crRNA привязки. Некоторые сайты связывания crRNA нарушена введением FP последовательности в шаблон плазмида доноров; Убедитесь, что ни PAM, или нетронутыми привязки регион по-прежнему существует в таких случаях.
Примечание: В silico доноров шаблоне плазмиды могут представляться для синтеза гена коммерческим поставщиком, или может использоваться в качестве отправной точки для клонирования стратегию, которая выходит за рамки настоящего Протокола. Достаточно простой позвоночника, таких как pUC19 или pUC57.
2. рибонуклеопротеида (RNP) Transfection для ТРИФОСФАТЫ/Cas9 при посредничестве стук в в hiPSCs
Примечание: В настоящем Протоколе, термин «gRNA» описывает синтетическими crRNA и tracrRNA должным образом вновь приостановлено, количественные и pre-complexed за инструкциями производителя (см. Таблицу материалы). Дополнить все СМИ с 1% стрептомицин, пенициллин. Общие руководящие принципы культивирования линии hiPSC WTC подробно описаны в более Аллен клеток Explorer18,19. WTC hiPSCs используются в настоящем Протоколе, но с надлежащей трансфекции оптимизации, электропорация RNP и доноров плазмида шаблон может быть успешно адаптирована для других типов клеток.
- Подготовка 10 мкм рабочие запасы gRNA и дикого типа S. pyogenes Cas9 белка2,20; Держите на льду. Подготовка 1 мкг/мкл работает запас доноров шаблоне плазмиды; Храните при комнатной температуре (RT). Используйте буфер рН 8,0 TE для всех разведениях.
- Подготовка матрицы покрытием культуры ткани 6-ну плита с 5 мл свежих питательных сред с 10 мкм рок ингибитора (Ri) в колодец. Держите пластины с СМИ в инкубаторе при 37 ° C и 5% CO2 до готовности плита клетки после процедуры трансфекции (максимум 2 h).
Примечание: Все матрицы покрытием пластины, используемые в настоящем Протоколе сделаны, добавив, что объем ледяной Matrigel разводят 1:30 в холодной среде DMEM/F12 СМИ по данным Института Аллен для клеток науки протокол для культивирования WTC hiPSC линия19. - С помощью реагента нежный диссоциации одной ячейки как рекомендовано в Таблице материалов, проход hiPSCs в одну ячейку подвеска и count клетки, используя счетчик автоматизированных клеток, или Горяева.
Примечание: Подробный протокол для ЦМТ hiPSC линии здесь можно найти в Аллен клеток Explorer19. Вкратце вымыть клетки один раз с RT DPBS и лечения с диссоциации реагент для 3-5 минут. Затем нарезанных ячеек в одну ячейку подвеска нежный закупорить и Пелле центрифугированием. Ресуспензируйте Пелле живой клетки в СМИ роста с 10 мкм ри.- Подготовьте Алиготе 1,84 х 106 клеток для каждого экспериментальные условия для transfected в отдельном 1.5 мл пробирок.
Примечание: Все тома рассчитываются для тома общая реакция 4.5 мкл в томе 100 µL электропорация, раз 2.3 реакций. Это составляет для повторяющихся transfection и избыток для дозирования ошибки.
- Подготовьте Алиготе 1,84 х 106 клеток для каждого экспериментальные условия для transfected в отдельном 1.5 мл пробирок.
- Подготовить рибонуклеопротеида комплекс трубы (RNP) для каждого экспериментальные условия, добавив 2,88 мкл 10 мкм gRNA и 2,88 мкл 10 мкм Cas9 в 1,5 мл трубку. Инкубируйте на RT как минимум 10 мин (максимум 1 ч).
- Пелле одну ячейку аликвота (подготовленных на шаге 2.3.1) на 211 x g 3 мин на RT. аспирата супернатант и Ресуспензируйте Пелле клеток в 220 мкл буфера электропорации производителя.
- Мкл 220 ресуспензированы клеток с шагом 2,5 в 1,5 мл RNP комплекс трубку, подготовленный на шаге 2.4.
- 4.60 мкл 1 мкг/мкл доноров шаблоне плазмиды, 1,5 мл пробирку, подготовленный на шаге 2.4.
- Nucleofection наконечник и пипетки смешать содержимое трубки 2 - 3 раза, а затем передачи 100 мкл суспензии на подготовленный электропорации устройство. Избегайте введения любые пузыри в подсказке. Примените 1300 V 1 импульса 30 мс.
- Аккуратно передачи подвеска в пластину 6-хорошо подготовленные шаге 2.2 с закрученного движения. Разогнать клетки осторожно передвигая пластину side-to-side и передней и задней.
- Используя новый наконечник nucleofection, повторите шаги 2.8-2.9 с оставшиеся 100 мкл подвеска и передачи в второй хорошо подготовленных пластины 6-хорошо.
- Повторите шаги 2.4 через 2.10 для каждой gRNA и доноров шаблон плазмида комбинации, включая ненацеливания gRNA, доноров шаблоне плазмиды только и буфер только элементы управления. Позаботьтесь, чтобы изменить пипетки и nucleofection советы, чтобы избежать перекрестного загрязнения.
- Инкубируйте transfected клеток при 37 ° C и 5% CO2. Измените СМИ на постоянный рост средств массовой информации (не Ri) в 24 ч и продолжать кормление hiPSCs каждые 24 ч для 72-96 h, мониторинг confluency. Когда hiPSCs достигает 60-80% слияния, перейдите к шагу 3.
Примечание: Тяжелые клеточную смерть (> 70%, по оценкам) является нормальной 24-48 ч после transfection.
3. СУИМ-обогащение предположительно редактировать hiPSCs
Примечание: При сортировке стволовых клеток, адаптировать параметры инструмента содействия выживаемость клеток, как это было предложено в ходе обсуждения. Кратко, используйте крупнейших сопла возможно (130 мкм), низкая скорость потока (≤ 24 мкл/мин), платье консервантов жидкости (например физраствора, см. Таблицу материалы) и низкого образец давление (10 psi).
- До начала эксперимента СУИМ, измените СМИ на рост СМИ с 10 мкм Ри и инкубации клеток при 37 ° C и 5% CO2 на 2-4 ч для поощрения выживания после СУИМ.
- Использование нежный одноклеточных диссоциации реагента, как это рекомендовано в Таблице материалов, hiPSCs проход в одну ячейку подвеска в СМИ роста с 10 мкм ри19.
- Фильтр hiPSC подвеска через сетчатый фильтр 35 мкм в полистирола круглодонные трубы.
- Сортировка клеток с использованием вперед разброс и разброс стороне (включая высоту и ширину) для исключения мусора и Дуплеты. Использование живой, буфер только контролировать клетки для задания FP-положительные ворота, что < 0,1% клеток буфер только входят в ворота.
- Сортировать все население FP-положительных клеток в 1,5-15 мл полипропиленовые трубы, содержащих 0,5-2 мл RT питательных сред с 10 мкм ри.
Примечание: Полипропилен уменьшает потенциал клеточной адгезии на пластик. - Центрифуга собранные клетки на 211 x g 3 мин на RT.
- Тщательно удалить супернатант и Ресуспензируйте Пелле клеток в 200 мкл питательных сред с 10 мкм ри. До 3000 отсортированных клетки перехода к одной хорошо свежий матрица покрытием 96-луночных пластины19.
Примечание: При установке соответствующего инструмента, клетки также могут быть отсортированы (навалом) непосредственно в один колодец матрицы покрытием культуры ткани 96-луночных пластины, содержащие 200 мкл питательных сред с 10 мкм ри на рекомендуемой плотности 1000-3000 клеток на хорошо для hiPSC. - Инкубируйте сортировки клеток при 37 ° C и 5% CO2. Измените СМИ роста СМИ дополнена 5 мкм ри в 24 ч. На 48 ч Начните кормление СМИ постоянный рост клеток (без ри) каждые 24 ч для 72-96 h, мониторинг confluency. Выживание после СУИМ оценивается в более чем 50% если минимум 500 клеток являются семенами в одной скважине 96-луночных плиты.
- Когда hiPSCs достичь слияния 60-80% и показать Зрелые морфологии (гладкая, хорошо упакованные колонии центры), проход в крупных формат плиты например 24-ну пластины, а затем от 24-ну пластины в 6-ну плиту.
- Когда hiPSCs в пластине 6-ну достигают 60-80% слияния и показать Зрелые морфологии, разверните 100 мм пластины, повторно пластины для обработки изображений, криоконсервировать или семян на клон собирание плотность (шаг 4)19.
4. Создание предположительно редактировать клоновых линий hiPSC
- С помощью реагента нежный диссоциации одной ячейки как рекомендовано в Таблице материалов, проход hiPSCs в одну ячейку подвеска и определить количество клеток / мл19.
- Клетки семян 10000 отредактированный населения hiPSCs на блюдо культуры ткани свежий матрица покрытием 100 мм, с помощью средства роста дополнена 10 мкм ри19. Измените СМИ на рост СМИ без ри 24 ч после посева и кормить hiPSCs с свежими роста СМИ каждые 24 ч для 5-7 дней.
- Когда hiPSCs сформировали колоний, которые видны макроскопически (примерно 500 мкм), они являются достаточно большой, чтобы быть изолированы. Подготовьте плиту 96-луночных матрицы покрытием, аспирационных избыток матрицы и добавив 100 мкл питательных сред с 10 мкм ри за хорошо19.
- На рассечения Пипетка использования P-200 Микроскоп, или аналогичные, осторожно очистите и аспирационная отдельных колоний от поверхности пластины. Объем трафика (~ 20-100 мкл) содержащий колонии в один колодец 96-луночных пластины, подготовленный на шаге 4.3.
- После того, как были переданы все колонии, Инкубируйте пластину в инкубатор культуры ткани на 37˚с и 5% CO2. Измените СМИ на постоянный рост средств массовой информации (не Ri) в 24 ч и продолжать кормление клетки каждые 24 ч для 72-96 h до колонии примерно три раза в размер (около 1500 мкм).
Примечание: Выбор 24-96 колоний за crRNA, используемых в трансфекции рекомендуется. Выживание изолированных клонов обычно более чем на 95%.
- После того, как были переданы все колонии, Инкубируйте пластину в инкубатор культуры ткани на 37˚с и 5% CO2. Измените СМИ на постоянный рост средств массовой информации (не Ri) в 24 ч и продолжать кормление клетки каждые 24 ч для 72-96 h до колонии примерно три раза в размер (около 1500 мкм).
- С помощью нежный одноклеточных диссоциации реагента, как рекомендовано в Таблице материалов, проход hiPSC клонов в новую плиту 96-луночных матрицы покрытием следующим19.
- С помощью 8-канальный аспиратор, снимите и выбросьте СМИ из первого столбца 96-луночных пластины.
- С помощью P-200 многоканальные пипетки, добавьте ~ 200 мкл DPBS в первый столбец 96-луночных пластины для мытья клетки. С помощью 8-канальный аспиратор, снимите и выбросьте мыть DPBS из первого столбца 96-луночных пластины.
- С помощью P-200 многоканальные пипетки, добавьте 40 мкл Реагента диссоциации в первый столбец 96-луночных пластины.
- Повторите шаги 4.5.1-4.5.3 для до в общей сложности шесть столбцов 96-луночных пластины, изменяя советы чтобы не кросс загрязнить скважин. Место пластину в инкубаторе 37 ° C для 3-5 минут от времени диссоциации реагент был добавлен к первому столбцу (шаг 4.5.3).
Примечание: Рекомендуется только проход максимум шесть колонн (48 скважин) в то время, из-за то время, необходимое для выполнения этих шагов. При выполнении этот протокол для в первый раз, начните с только что один или два столбца в то время. Ограничение на количество столбцов, пассированные одно время гарантирует, что клетки не оставил в диссоциации реагент для слишком долго, которые могут быть вредны для hiPSCs. - Когда ячейки в первом столбце плиты начали поднять от нижней пластины, используйте P-200 многоканальные пипетки для 160 мкл DPBS в первый столбец 96-луночных пластины и аккуратно нарезанных клетки в «12:00» , «3:00», «6:00», а «9:00» позиции каждой скважины. Передать весь объем суспензии клеток (200 мкл) V-дно 96-луночных пластины.
- Повторите шаг 4.5.5 для оставшихся столбцов ячеек, которые имеют диссоциации реагента в них; Измените советы о том, не кросс загрязняют колодцы.
- Спиновые V-днище в центрифуге на 385 x g 3 мин на RT.
- С помощью P-200 многоканальные пипетки, аккуратно удалить супернатант и Ресуспензируйте клетки в 200 мкл свежих питательных сред с 10 мкм ри за хорошо. Повторите для всех скважин, изменяя советы о том, не загрязнять крест.
- Передать все суспензию клеток свежим покрытием матрицу 96-луночных пластины19. Инкубируйте пластину в инкубатор культуры ткани при 37 ° C и 5% CO2. Измените СМИ на постоянный рост средств массовой информации (не Ri) в 24 ч и продолжать кормление клетки каждые 24 ч для 72-96 ч, до тех пор, пока большинство клонов достичь слияния 60-80%.
Примечание: Этот отрывок помогает разложить клетки и позволить больше роста по всей поверхности 96-луночных пластины.
- Соблюдать клоны и определить соответствующие Сплит коэффициент для каждого индивидуального клона в 96-луночных плиты (например, 1:10 или 1:8). С помощью реагента нежный диссоциации одной ячейки как предлагается в Таблице материалов, проход hiPSC клонов в новой матрицы покрытием 96-луночных плиту (шаги 4.5.1-4.5.8) передачу отношение суспензии клеток, подходящие для каждого клона. Инкубируйте пластину в инкубатор культуры ткани на 37˚с и 5% CO2. Измените СМИ на постоянный рост средств массовой информации (не Ri) в 24 ч и продолжать питание клеток каждые 24 ч для 72-96 ч, до тех пор, пока большинство клонов достичь слияния 60-80% и показать Зрелые морфологии.
Примечание: Каждый клон могут иметь соотношение различных Сплит из-за немного отличается роста или выживание от предыдущего прохода, поэтому этот отрывок помогает нормализовать количество ячеек на хорошо каждого клона для замораживания шаг, чтобы следовать. Из-за различной ставки выживания и роста некоторые клоны могут перерасти или не расти в течение этих пассированый шаги.- Сохраните оставшуюся часть суспензию клеток и гранулы для изоляции геномная ДНК путем центрифугирования клетки в V-дно 96-луночных пластины на 385 x g 3 мин на RT. удалить супернатант и приступить к изоляции геномная ДНК с помощью комплекта 96-луночных или хранить тарелку гранулированных клеток на - 20˚с для до Чет ee недель.
5. криоконсервирования клоновых клеточных линий в формат 96-луночных пластины
- С использованием одной ячейки диссоциации реагент, hiPSC проход клоны как описано выше (4.5.1-4.5.7 меры). Аспирационная супернатанта, используя P-200 многоканальные пипетки и вновь приостановить в 60 мкл питательных сред с 10 мкм ри. Повторите для всех скважин; Измените советы о том, не загрязнять крест.
- Передача 30 мкл суспензии клеток к пластине-матрица с покрытием 96-луночных тканевой культуры. Затем быстро добавить 170 мкл замораживания буфера (см. Таблицу материалы) для каждой скважины, без перемешивания. Повторите путем передачи оставшихся 30 мкл суспензии в сестра пластины и добавление замораживания 170 мкл буфера.
Примечание: Этот процесс делается в повторяющихся сестра тарелки так что резервные популяция клеток существует после оттаивания одной из отдельных пластин. Ввод криоконсервированных клеток в только каждый другой столбец, 96-луночных плиты позволяет быстрее оттаивания (шаг 5.6). - Обернуть тарелку с парафина и место в RT пенополистирола коробки с крышкой. Место целый ящик в морозильной камере-80 ° С.
- После 24 часов пластины могут передаваться из пенополистирола коробки и хранятся на - 80˚с на срок до четырех недель.
Примечание: в то время как клетки временно хранятся при температуре-80 ° C, генетического контроля качества анализов могут быть выполнены с геномная ДНК, добываемых из клеток, полученных на шаге 4.6.1 с целью выявления клонов оттепель и распространять дальше, как говорилось в ранее опубликованной работе 12. кратко, копии номер капелька цифровой ПЦР пробирного может использоваться для идентификации клоны, которые содержат одну или две копии гена GFP и не доноров шаблон плазмида основу интеграции. Сочетание концевой анализы ПЦР и Сэнгер последовательности могут затем определить клоны, которые содержат точное вставки. - Растаять, принести всю тарелку до 37 ° C в инкубатор культуры ткани, внимательно наблюдая за гранулы льда таять. Скважины на краю пластины, как правило, первые оттепели.
- Когда тает лед Пелле желаемого клона, мягко передавать весь 200 мкл 15 мл конические трубка, содержащая 3 мл RT питательных сред с 10 мкм Ри и центрифуги на 211 x g 3 мин на RT.
- Аспирационная супернатант и Ресуспензируйте гранулированных клеток в 1 мл RT питательных сред с 10 мкм ри. Трансфер в свежий матрица покрытием 24-ну тарелку и Инкубируйте на 37˚с и 5% CO2. Изменить СМИ на постоянный рост средств массовой информации (не Ri) в 24 ч и продолжать кормление клетки каждые 24 ч для 72-96 h, пока клон достигает 60-80% confluency и зрелые морфология19.
Результаты
Целью этого эксперимента было предохранитель mEGFP (односегментную расширенной GFP) ядерных lamin B1 белка путем введения mEGFP последовательность в конце 5ʹ гена LMNB1 (N-конечная белка). Компоновщик (аминокислотной последовательности SGLRSRAQAS) был выбран на основании предыдущих cDNA конструкции из Майкл Дэвидсон флуоресцентный белок коллекция21. Потому что crRNA привязки региона в плазмиду доноров шаблон для каждого кандидата crRNA было нарушено после вставки в silico mEGFP и компоновщик последовательности, не точечные мутации необходимо сделать сорвать потенциальных crRNA признание и расщепление Cas9 доноров последовательности (рис. 1). Доноров последовательности содержал 1 КБ гомологии оружия фланговые обоих концах mEGFP компоновщик последовательности. Результате 2,734 bp ДНК был клонирован в pUC57 позвоночника, последовательность проверки, и результирующий шаблон плазмида доноров был очищен с помощью бесплатно эндотоксина макси преп. Доноров шаблоне плазмиды и RNP комплекса были transfected, предположительно отредактированных ячеек обогатились и локализация mEGFP ядерных lamin B1 синтез белка была подтверждена микроскопии флуоресцирования (рис. 2). Только результаты из crRNA1 transfection описаны здесь, хотя обе crRNA последовательности производства предположительно отредактированный населения12.
По сравнению с отрицательного контроля, который не содержит gRNA, Cas9 белков или доноров шаблон плазмида в реакции электропорации (только буфер), LMNB1 crRNA1 transfected клеток содержится 0,95% mEGFP положительных клеток, представляющие предположительно редактировать mEGFP ядерная Ламина B1 населения (рис. 3a). Этот результат был в пределах стук в эффективности наблюдается во многих геномной локусов, используя этот метод, как сообщалось ранее12.
MEGFP положительных клеток были изолированные FACS и образы живой микроскопии для подтверждения ожидаемых локализация mEGFP ядерных lamin B1 синтез белка в ядерная оболочка. После обогащения СУИМ примерно 90% сортировки клеток от LMNB1 crRNA1 населения были mEGFP позитивные (как определяется микроскопии), о том, что некоторые mEGFP отрицательные клетки совместно очищенный с GFP-положительных клеток во время процедуры сортировки. Это был приемлемый уровень обогащения, что позволило для комплектации 96 клонов, которые могли бы затем генетически проверяться для успешного редактирования. Как правило, отсчета для успешного обогащения составляет 50% положительных GFP.
Большинство населения отсортированных ячейки отображается флуоресценции в ядерная оболочка (ядерной периферии) в nondividing клетках и для расширенного ядерного пластинки в цитоплазму во время митоза обеспечение доверия в правильной геномной редактировать в LMNB1 Локус. Обогащенный населения содержатся клетки с яркие или слабом сигнал. Эта разница в интенсивности сигнала может отражать сочетание правильных и неправильных результатов и подчеркивается полезность создания генетически проверенных клоновых линия для дальнейшего исследования (см. обсуждение) редактирования (рис. 3b)12. После генерации клоновых строки генетически проверяются клеток показали равномерное GFP интенсивности в микроскопии экспериментов (рис. 3 c).
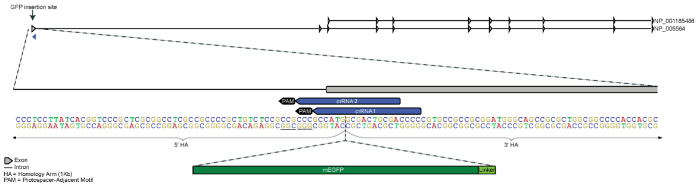
Рисунок 1 . Разработка стратегии для N-конечная GFP пометки гена LMNB1. GFP тег был разработан для 5ʹ N-терминальный вставки первого экзона LMNB1, расположенный на хромосоме 5. 5ʹ и 3ʹ гомологии вооружений являются 1 kb и встретиться между началом кодон (ГПТ) и второй кодон (гомологии оружия лишь частично представлена на рисунке). Два кандидата crRNAs были разработаны для руководства Cas9 к расщеплять как можно ближе к предполагаемой вставки сайт как можно, сохраняя при этом уникальную в геноме. Последовательность для mEGFP и компоновщик аминокислоты были вставленной просто 3ʹ начала кодон (mEGFP и компоновщик последовательность не в масштабе). Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.

Рисунок 2 . Рабочий процесс для производства эндогенно меткой клоновых линий hiPSC. Трансфекция компонентов, включая комплекс RNP Cas9/crRNA/tracrRNA (показано как красный Cas9 белка с золотой crRNA и фиолетовый tracrRNA), плазмида доноров шаблон, содержащий гомологии оружия (HAs, показано в золоте) и FP + компоновщика последовательности (показано зеленым), были electroporated. После 4 дней предположительно редактировать FP-положительных клеток были обогащенный СУИМ и расширил как населения путем заполнения всех сортировки ячеек в одну скважину 96-луночных плиты (~ 1000 ячеек) и затем расширена в культуре до работающего населения несколько миллионов клетки могут быть assayed как «обогащенный населения» (см. Протокол шаг 3,8). Доходность FP-положительных клеток отличается эксперимент из-за переменной ставки HDR12; успешный обогащения может обычно включают в себя ~ 300-5000 клеток после трансфекции приблизительно 1.6 x 106 клеток бедра. Предварительные тепловизионные исследования подтвердили сигнала и локализации синтез белка обогащенного населения. Колонии были вручную выбрал в 96-луночных пластина для расширения и криоконсервации. Далее геномной контроля качества скрининга с помощью дроплета цифровой ПЦР (ddPCR) и другие на основе ПЦР анализов затем использовался для выявления надлежащим образом отредактированного клоны, как описано выше12. Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.

Рисунок 3 . Обогащение населения предположительно редактируемой ячейки. (A) потока цитометрии участков LMNB1 редактировать пост transfection клетки четыре дня. Ось y отображает интенсивность GFP и на оси x отображается вперед скаттер. Сортировки ворота были установлены на основе только для буфера элемента управления. Поскольку hiPSCs чувствительны к возмущений, жить/мертвые пятна был опущен и весьма консервативных FSC/SSC ворота был использован вместо. (B) после обогащения, население LMNB1 Cr1 редактировать клетки показали ~ 90% клеток с GFP локализации для ядерная оболочка (ожидаемые LMNB1 локализации). Населения содержала клетки различной интенсивности GFP, а также некоторых GFP-отрицательные клетки. Масштаб гистограммы являются 10 мкм. (C) после генерации клоновых строки, клетки показали равномерное GFP интенсивности, с некоторыми отличиями зависимых клеточного цикла. Шкалы бар-20 мкм.
Обсуждение
Метод, представленные здесь для генерации эндогенно регулируется флуоресцентный протеин сплавливания в hiPSCs это универсальный и мощный подход для генерации гена редактировать клеточных линий с приложениями, начиная от живых клеток различных функциональных исследований и» болезни в блюдо» модели с помощью пациент производные hiPSC линии13,14,15. Хотя этот метод был использован ввести большие FP теги к N - или C-конечная эндогенного белков, он потенциально может использоваться представить другие теги или небольших генетические изменения модели или правильный болезнетворные мутации22,23 . Для небольших вставок размер гомологии рука может быть уменьшена, но по-прежнему применять общий подход к редактированию, представленные в этом методе могут24,25. В то время как использование hiPSCs настоятельно рекомендуется для их обширной Утилита, с тщательной оптимизации, этот протокол может быть адаптирована к редактировать геномов других линий клеток млекопитающих.
При определении ген интереса для FP пометки, Стенограмма изобилие оценок (от microarray дна или RNA-Seq данных) являются хорошей отправной точкой для оценки ли ген или изоформы интерес выражается, хотя Стенограмма уровнях не всегда коррелирует с уровни белка. СУИМ обогащение стратегии, описанные здесь будут работать лучше для генов, которые по крайней мере умеренно хорошо выражены в тип ячейки интереса. Эта стратегия также была успешной в выборе для слияний, которые показывают пунктата или дискретных локализации шаблонов, таких как centrin, десмоплакином и paxillin, где фон соотношение сигнал является очень низким12,19. Гены, которые не выражаются высоко или только выражаются в типы производных клеток может потребовать дополнительного отбора стратегий.
Отправной точкой для crRNA и доноров шаблон плазмида конструкций, используемых в линий клеток человека должно быть геном человека ссылки (GRCh38). Потому что геномов различных клеточных линий может варьироваться в пределах одного вида, и потому что ТРИФОСФАТЫ/Cas9 зависит от последовательности, это очень полезно для выявления клеток линии конкретных вариантов (единичных нуклеотидных полиморфизмов или вставки/удаления (indels)) которые отличаются от ссылка генома и включить их в дизайн. Это гарантирует что crRNAs будет совместима с геном хост и что доноров шаблон плазмида гомологии оружия будет сохранять любые конкретные варианты линии клетки. Предложенная стратегия является включение гомозиготных варианты в crRNAs и доноров шаблон плазмида гомологии оружия во время процесса проектирования. Включение гетерозиготных вариантов является необязательным. Конкретные реагенты используются для больших забивные экспериментов и другие ключевые соображения для настоящего Протокола, рассматриваются ниже.
Cas9 белка
Основное преимущество использования Cas9 белок является то, что введение Cas9 и привести к ограниченной продолжительности нуклеиназы активности по сравнению с подходы, основанные на плазмида, где выражение Cas9 и gRNA может затянуться на несколько дней и привести было показано gRNA как RNP комплекс для более на и вне цель деятельности26,27. Дополнительное преимущество использования Cas9 белок является доступной для расщеплять раз внутри клетки. Это контрастирует с более традиционными методами использования мРНК Cas9 или Cas9/gRNA плазмиды, которые требуют транскрипции, перевод и белка, обработка26,28. Белок wildtype S. pyogenes Cas9 теперь доступен из многих коммерческих источников.
Руководство РНК
Есть много публично доступные инструменты для поиска целей crRNA неподалеку от желаемого места вставки FP, содержащих ноль или несколько прогнозируемых вне цели в геном31,29,30,хост32. Эффективность в HDR и точность результатов HDR различаются между crRNA задач, используемых в данном локусе12. По этой причине, тестирование несколько crRNAs (2-4 и, желательно, в течение 50 bp требуемой вставки сайт) в локусе рекомендуется, поскольку это может увеличить вероятность успешного редактирования эксперимента. Текущие возможности для доставки gRNA включают синтетические 2-часть crRNA и tracrRNA, синтетические одного оформления (sgRNAs), в пробирке транскрипции sgRNAs, или доставку плазмида клетки, выражая sgRNA от промотор U6. Этот протокол не был оптимизирован для высокой расщепления деятельности. Неизмененное 2-часть crRNA и tracrRNA (см. Таблицу материалы) были использованы с целью получения моно аллельные FP-тегами клеточных линий причиняя наименее потенциальных возмущений в клетки.
Доноров шаблоне плазмиды
Потому что некоторые из гомологии руку последовательности условии в донора плазмида шаблона будут включены в хост геномов во время события HDR, точечные мутации crRNA признание сайты следует ввести для предотвращения дальнейшего расщепления по Cas9 после HDR. Часто простой подрывной изменяется мутировать PAM последовательности. Потому что некоторые нестандартные PAM последовательности все еще могут быть признаны дикого типа S. pyogenes Cas9, то лучше избегать использования NGG, ПИЛИТЬ или Нга33. Когда мутирует гомологии руку, Избегайте не синоним перегласовок и введение редких кодонов. Если синоним изменения последовательности PAM не возможен, рассмотреть возможность сделать три синонимом точечные мутации в регионе семян (10 bp проксимальнее PAM) crRNA привязки сайта. Особое внимание следует проявлять при внесении этих изменений в регионе непереведенные 5ʹ (утр), поскольку эти регионы могут содержать важные нормативные последовательности. Консультации сохранения генетических ресурсов базы данных такие, как браузер геноме UCSC сравнительной геномики треков может обеспечить руководство в таких случаях, как изменения сохраняясь баз может лучше переносится, чем изменения весьма сохранившихся баз17. Иногда простой последовательности FP является достаточно, чтобы нарушить привязки сайта crRNA (см. рис. 1); Однако недавно присоединенных последовательности должны быть проверены для сохранения привязки crRNA и Пэм последовательности.
Аминокислота линкеры между FP и родном протеине, рекомендуется сохранить функции синтеза белка34. Часто компоновщика аминокислот могут быть выбраны для его конкретного обвинения или размер. Если cDNA сплавливание с дизайн похож на целевых эндогенного синтез белка хорошо изучено, что же компоновщика последовательность может использоваться для ТРИФОСФАТЫ/Cas9 стучите в эксперимент12,19. Если такая информация недоступна, короткий компоновщика как GTSGGS также был успешно использован12. Другие исследования продемонстрировали успех с последовательностью универсальный маленький 3-амино кислоты компоновщика для различных целей35.
Трансфекция и обогащения СУИМ
Многие коммерчески доступных трансфекции реагентов сформулированы для доставки некоторых типов молекул клетки, тогда как электропорации система может использоваться для доставки реагентов с широкий спектр размер, заряд и состава. Помимо того, что общий метод трансфекции для жестких transfect клетки как hiPSCs, электропорация также несет благо доставки всех трех компонентов для ТРИФОСФАТЫ/Cas9-опосредованной FP-стук, как описано в этом методе. Электропорация было установлено производить лучшие результаты по сравнению с другими коммерчески доступных реагентов при разработке этого метода (данные не показаны) и также используется другими RNP доставки26,28,36 .
При использовании данного протокола для редактирования hiPSCs, особое внимание следует обеспечить нежный обработки клеток до и после гена, процесс для оптимального ячейку выживания и минимальным спонтанное дифференциации редактирования. В частности, методы обогащения СУИМ должны быть адаптированы для сортировки стволовых клеток с помощью крупнейших сопла возможно (130 мкм), низкая скорость потока (≤24 мкл/мин), платье консервантов жидкости (например физраствора, см. Таблицу материалы) и низкого образца давление (10 psi). Вместо одной ячейке сортировки, который приводит к неоптимальной жизнеспособности в стволовых клеток, обогащенный СУИМ hiPSCs сортируются навалом и расширена как население оптимизировать жизнеспособность и стволовых клеток целостность клеток. Тем не менее одну ячейку Сортировка может подходить для менее чувствительных клеток. Для поощрения выживания клетки, клетки вернулся в культуре не больше чем один час после сбора урожая для обогащения СУИМ и хранятся при комнатной температуре на протяжении всего процесса сортировки. Для некоторых типов клеток выживание клетки также могут быть расширены путем выращивания клеток на льду (4° C) на протяжении всего процесса сортировки.
Основная расширение FP-положительных клеток обеспечивает возможность оценить населения путем анализа изображений для синтеза белка локализации до создания клоновых линий. В то время как результате обогащенного популяция клеток может быть достаточно для некоторых исследований, эти группы населения часто отображения сигнала FP различной интенсивности. Изолированные клоновых линий имеют единый сигнал (рис. 3), что делает их более подходящими для функциональных эксперименты12.
Клоновых клеток линии поколение
В процессе редактирования и клоновых линии поколения важно контролировать морфологии клеток. колонии hiPSC, выращенные в условиях свободной подачи должны exhibit гладкими краями и12,18,даже, хорошо упакованные центра19. Дифференцированных клеток должны соблюдаться в менее чем 5% культуры. При выборе отдельных колоний, выберите те, что выставка хорошего морфологии. В течение 96-луночных пластины, пассированый события проверьте клоны для морфологии и прекратить те, которые заросший, как это может привести к дифференциации или свидетельствовать о генетической нестабильности.
Поколение клоновых клеточных линий позволяет Генетическое подтверждение точного редактирования, который имеет важное значение, потому что Cas9-индуцированной двойной нити влезает в геноме часто устраняются неточно несмотря на включение тега в желаемой локусе. Описано на основе ПЦР анализов показали, что совокупности через десять уникальных геномной локусов многих (45%) FP-выражая клонов страдал от доноров плазмида основой интеграции в целевых локусе или (редко) случайно в геном12. Кроме того, 23% GFP-позитивных клоны (n = 177) через десять уникальных локусов были найдены в гавани мутации на или вблизи ожидаемого crRNA резки сайт в неразмеченных аллель, скорее всего, из-за NHEJ12. Этот генетический анализ многих клоновых линий (~ 100 клонов и редактировать) подчеркнул важность генетической проверки, который не возможен в популяции клеток, поскольку FP-выражение и ожидаемых синтеза белка локализации только не гарантируют точное редактирование12 . Кроме того эти анализы ПЦР основе не может быть выполнена на обогащенный популяция клеток с уверенностью, оправдывающих необходимость генерации клоновых строки перед значимый анализ может быть завершена. Генетическое подтверждение вставляемых тегов FP и проверки целостности генетического неотредактированных аллеля (в моно аллельные отредактированный клон), так необходимые для обеспечения точного редактирования в целевых Локус за пределами тегов выражение.
На сегодняшний день с помощью этого метода (неопубликованные данные)12наблюдается низкий уровень Би аллельные правок и отсутствие пробить мутаций (как Оксиметрический анализ Сэнгер и exome последовательности). Это согласуется с предыдущих исследований, описывающих использование недолго RNP для экспериментов ТРИФОСФАТЫ/Cas926,27. Отсутствие клоновых клеточных линий с Би аллельные изменения могут также быть Локус конкретных или вследствие неспособности клетки переносят два помечены копии основных белков как предлагалось ранее опубликованные экспериментов где предполагаемого Би аллельные измененные клетки были наблюдается на один локус (LMNB1), но не другой (TUBA1B)12. BI аллельные полностью проверенных клоновых клеточных линий были получены с помощью этого метода для тега ST6 бета galactoside альфа-2, 6-sialyltransferase 1 (ST6GAL1) и RAB5A член семьи онкогена РАН (RAB5A) с mEGFP19.
Помимо подтверждения точности редактирования в геноме, существует целый ряд анализов качества контроля, которые могут использоваться для дальнейшего характеризуют клоновых линии и выявления клонов, которые выполняют все стволовых клеток, геномные, и клеток биологические критерии для использования в будущих исследованиях . Биологической и функциональной assays клетки может использоваться для подтверждения соответствующее выражение, локализации и функции синтеза белка12. Сравнение неотредактированных родительского контроля поможет оценить влияние процесса редактирования на локализации, динамика и функции. Другие анализы такие, как анализ роста и тесты для геномная стабильность также могут помочь определить, является ли протеин тегами отмерена к ячейке. При использовании hiPSC в этом протоколе, Оценка маркеров плюрипотентности и дифференциации потенциал может быть решающее значение в определении клона, что ценно для вниз по течению исследования12. Потому что продлен культуры hiPSC было показано, что приведет к генетической нестабильности, мониторинг темпы роста и кариотип клоновых клеточных линий является также важным12,37. Однако финал предназначено использование отредактированных ячейках в конечном итоге будет определять уровень и масштабы контроля качества анализа и будет зависеть от приложения.
Раскрытие информации
Авторы не имеют ничего сообщать.
Благодарности
Мы благодарим Дафна Dambournet за многие глубокие обсуждения и консультации по гена, редактирование, ДУ Thao для иллюстрации, Нельсон Angelique для критических чтении рукописи и Эндрю Такер для создания линии mEGFP тегами ячейки B1 Ламин. Мы хотим отметить стволовых клеток и Джин редактирования и команды разработчиков Assay в институте Аллен клеток науки за их вклад в ген редактирования и процесс контроля качества. Линии ЦМТ, который мы использовали для создания нашего гена редактировать клеточной линии была оказана Брюс р. Conklin лабораторией в Гладстон институтах и при Калифорнийском университете. Мы благодарим институт Аллен основатель науки клеток, G. Пол Аллен, за его видение, поощрение и поддержки.
Материалы
| Name | Company | Catalog Number | Comments |
| Geneious R9 | Biomatters, or similar | bioinformatics software for in silico donor plasmid design | |
| TE Buffer pH 8.0 | IDT, or similar | 11-01-02-05 | |
| HERAcell VIOS 160i CO2 incubator, or similar | ThermoFisher Scientific, or similar | 51030408 | |
| Pipettes (1000 µL, 200 µL, 20 µL, 10 µL, 2 µL) | Rainin, or similar | ||
| Pipette tips (1000 µL, 200 µL, 20 µL) | Rainin, or similar | ||
| Multi-channel Pipette (200 µL) | Rainin, or similar | ||
| Serological pipetes (25 mL, 10 mL, 5 mL) | Costar, or similar | ||
| BRAND 8-Channel Manifold for Quiksip, Autoclavable | Millipore Sigma, or similar | BR704526-1EA | use with non-filtered pipet tips, such as Molecular BioProducts Low Retention Pipet Tips, Pure 10, below |
| Molecular BioProducts Low Retention Pipet Tips, Pure 10 | Thermo Fisher, or similar | 3501-05 | |
| XP2 Pipette Controller | Drummond, or similar | 4-000-501 | |
| Disposable Pasteur Pipets | VWR, or similar | 53300-567 | |
| Class II, Type A2 Biological Safety Cabinet | CELLGARD, or similar | NU-481 | |
| Matrigel Matrix, Growth Factor Reduced | Corning | 354230 | lot tested before use with hiPSC |
| DMEM/F12 (-phenol red) | Gibco | 11039-021 | cold, for diluting Matrigel 1:30 |
| mTeSR1 Complete Media | StemCell Technologies | 85850 | recommended growth media for WTC hiPSC line |
| Penicillin-streptomycin | Gibco | 15070-063 | |
| WTC hiPSC line | Coriell | GM25256 | the hiPSC line used in this protocol is available through Coriell |
| Tissue culture dish 100 mm | Falcon | 353003 | |
| 6-well Cell Culture Plate | CELLSTAR | 657160 | |
| StemPro Accutase | Gibco | A11105-01 | |
| Dulbecco's Phosphate Buffered Saline (DPBS) no calcium, no magnesium | Gibco | 14190144 | |
| 15 mL polystyrene conical | Sarstedt | 62.554.100 | |
| Y-27632 (ROCK Inhibitor) | StemCell Technologies | 72308 | |
| Edit-R CRISPR-Cas9 Synthetic crRNA, unmodified (custom sequence) | Dharmacon | Custom0247 | |
| Edit-R CRISPR-Cas9 Synthetic tracrRNA | Dharmacon | U-002005-05 | |
| Recombinant wild-type Streptococcus pyogenes Cas9-NLS purified protein, 40 µM | University of California-Berkeley QB3 Macrolab | ||
| Custom donor plasmid (PriorityGENE) | Genewiz | donor insert design was synthesized and cloned into pUC57 backbone by Genewiz | |
| DNA LoBind Tube 1.5 mL | Eppendorf | 22431021 | |
| NucleoBond Xtra Maxi EF | Clontech | 740424.50 | |
| Neon Transfection System | ThermoFisher Scientific | MPK5000 | |
| Neon Transfection System, 100 µL kit | ThermoFisher Scientific | MPK10096 | |
| 5 mL Polystyrene Round-bottom Tube with Cell Strainer Cap | Falcon | 352235 | |
| 15 mL High Clarity Polyproylene Conical Tube | Falcon | 352196 | |
| FACSAriaIII Fusion | BD Biosciences | 656700 | |
| FACSDiva software | BD Biosciences | ||
| FlowJo version 10.2 | TreeStar | ||
| NERL Blood Bank Saline | ThermoFisher Scientific | 8504 | used as preservative-free FACS Buffer |
| Olympus SZX7 Stereo Microscope, or similar | Olympus, or similar | ||
| Tissue culture plate, 96-well | Falcon | 353072 | |
| 96 well Cell Culture Plate, V-Bottom | CELLSTAR | 351180 | |
| CryoStor CS10 | Sigma | C2874-100ML | used as cryopreservation buffer for cells in 96-well plate format |
| Parafilm | Bemis | PM-996 | |
| 24 well Cell Culture Plate | CELLSTAR | 662160 |
Ссылки
- Wood, A. J., et al. Targeted genome editing across species using ZFNs and TALENs. Science. 333 (6040), 307 (2011).
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Cong, L., et al. Multiplex genome engineering using CRISPR/Cas systems. Science. 339 (6121), 819-823 (2013).
- Mali, P., et al. RNA-guided human genome engineering via Cas9. Science. 339 (6121), 823-826 (2013).
- Dambournet, D., Hong, S. H., Grassart, A., Drubin, D. G. Tagging endogenous loci for live-cell fluorescence imaging and molecule counting using ZFNs, TALENs, and Cas9. Methods in Enzymology. , 139-160 (2014).
- Ratz, M., Testa, I., Hell, S. W., Jakobs, S. CRISPR/Cas9-mediated endogenous protein tagging for RESOLFT super-resolution microscopy of living human cells. Sci Rep. 5, 9592 (2015).
- Hendriks, W. T., Warren, C. R., Cowan, C. A. Genome Editing in Human Pluripotent Stem Cells: Approaches, Pitfalls, and Solutions. Cell Stem Cell. 18 (1), 53-65 (2016).
- Hockemeyer, D., Jaenisch, R. Induced Pluripotent Stem Cells Meet Genome Editing. Cell Stem Cell. 18 (5), 573-586 (2016).
- Doyon, J. B., et al. Rapid and efficient clathrin-mediated endocytosis revealed in genome-edited mammalian cells. Nature Cell Biology. 13 (3), 331-337 (2011).
- Cho, W. K., et al. Super-resolution imaging of fluorescently labeled, endogenous RNA Polymerase II in living cells with CRISPR/Cas9-mediated gene editing. Sci Rep. 6, 35949 (2016).
- White, C. W., Vanyai, H. K., See, H. B., Johnstone, E. K. M., Pfleger, K. D. G. Using nanoBRET and CRISPR/Cas9 to monitor proximity to a genome-edited protein in real-time. Sci Rep. 7 (1), 3187 (2017).
- Roberts, B., et al. Systematic gene tagging using CRISPR/Cas9 in human stem cells to illuminate cell organization. Mol Biol Cell. 28 (21), 2854-2874 (2017).
- Soldner, F., et al. Generation of isogenic pluripotent stem cells differing exclusively at two early onset Parkinson point mutations. Cell. 146 (2), 318-331 (2011).
- Young, J. E., Goldstein, L. S. Alzheimer's disease in a dish: promises and challenges of human stem cell models. Human Molecular Genetics. 21 (R1), R82-R89 (2012).
- Soares, F. A., Sheldon, M., Rao, M., Mummery, C., Vallier, L. International coordination of large-scale human induced pluripotent stem cell initiatives: Wellcome Trust and ISSCR workshops white paper. Stem Cell Reports. 3 (6), 931-939 (2014).
- . . Gene, NCBI- National Center for Biotechnology Information. , (2017).
- . . Gene, NCBI- National Center for Biotechnology Information. , (2017).
- . . Allen Cell Methods: Single cell passaging human iPS cells. , (2018).
- . . Allen Cell Explorer. , (2017).
- Lingeman, E., Jeans, C., Corn, J. E. Production of Purified CasRNPs for Efficacious Genome Editing. Curr Protoc Mol Biol. 120, 31-31 (2017).
- . . Michael Davidson Fluorescent Protein Collection. , (2017).
- Cox, D. B., Platt, R. J., Zhang, F. Therapeutic genome editing: prospects and challenges. Nature Medicine. 21 (2), 121-131 (2015).
- Haas, S. A., Dettmer, V., Cathomen, T. Therapeutic genome editing with engineered nucleases. Hamostaseologie. 37 (1), 45-52 (2017).
- Beumer, K. J., Trautman, J. K., Mukherjee, K., Carroll, D. Donor DNA Utilization During Gene Targeting with Zinc-Finger Nucleases. G3 (Bethesda). 3 (4), 657-664 (2013).
- Orlando, S. J., et al. Zinc-finger nuclease-driven targeted integration into mammalian genomes using donors with limited chromosomal homology. Nucleic Acids Research. 38 (15), e152 (2010).
- DeWitt, M. A., Corn, J. E., Carroll, D. Genome editing via delivery of Cas9 ribonucleoprotein. Methods. 121-122, 9-15 (2017).
- Kim, S., Kim, D., Cho, S. W., Kim, J., Kim, J. S. Highly efficient RNA-guided genome editing in human cells via delivery of purified Cas9 ribonucleoproteins. Genome Research. 24 (6), 1012-1019 (2014).
- Lin, S., Staahl, B. T., Alla, R. K., Doudna, J. A. Enhanced homology-directed human genome engineering by controlled timing of CRISPR/Cas9 delivery. Elife. 3, e04766 (2014).
- Labun, K., Montague, T. G., Gagnon, J. A., Thyme, S. B., Valen, E. CHOPCHOP v2: a web tool for the next generation of CRISPR genome engineering. Nucleic Acids Research. 44 (W1), W272-W276 (2016).
- Montague, T. G., Cruz, J. M., Gagnon, J. A., Church, G. M., Valen, E. CHOPCHOP: a CRISPR/Cas9 and TALEN web tool for genome editing. Nucleic Acids Research. 42 (Web Server issue), W401-W407 (2014).
- Bae, S., Park, J., Kim, J. S. Cas-OFFinder: a fast and versatile algorithm that searches for potential off-target sites of Cas9 RNA-guided endonucleases. Bioinformatics. 30 (10), 1473-1475 (2014).
- . . CRISPOR V4.3. , (2017).
- Zhang, Y., et al. Comparison of non-canonical PAMs for CRISPR/Cas9-mediated DNA cleavage in human cells. Sci Rep. 4, 5405 (2014).
- Chen, X., Zaro, J. L., Shen, W. C. Fusion protein linkers: property, design and functionality. Adv Drug Deliv Rev. 65 (10), 1357-1369 (2013).
- Leonetti, M. D., Sekine, S., Kamiyama, D., Weissman, J. S., Huang, B. A scalable strategy for high-throughput GFP tagging of endogenous human proteins. Proc Natl Acad Sci U S A. 113 (25), E3501-E3508 (2016).
- Liang, X., et al. Rapid and highly efficient mammalian cell engineering via Cas9 protein transfection. Journal of Biotechnology. 208, 44-53 (2015).
- Baker, D., et al. Detecting Genetic Mosaicism in Cultures of Human Pluripotent Stem Cells. Stem Cell Reports. 7 (5), 998-1012 (2016).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены