Method Article
Protéine endogène de marquage dans l’homme induites par les cellules souches pluripotentes à l’aide de CRISPR/Cas9
Dans cet article
Résumé
Décrite ici, c’est un protocole pour le marquage de façon endogène des protéines avec des étiquettes fluorescentes dans les cellules souches humaines pluripotentes induites à l’aide de CRISPR/Cas9 exprimées. Cellules putativement édités sont enrichis en triant les cellules fluorescence activé et lignées clonales sont générées.
Résumé
Un protocole est présenté pour générer des cellules souches humaines pluripotentes induites (hiPSCs) que les protéines endogènes express fusionnés pour étiquettes fluorescentes N - ou C-terminale dans le cadre. Le système CRISPR/Cas9 procaryote (cluster court régulièrement dois‑je palindromique 9 répétitions/CRISPR-associés) peut-être servir d’y introduire les grandes séquences exogènes locus génomiques par réparation homologie réalisée (HDR). Pour atteindre le désiré knock-in, ce protocole utilise les ribonucléoprotéines (RNP)-approche où les protéines de type sauvage Cas9 Streptococcus pyogenes , synthétique 2 parties guide RNA (gRNA) et un plasmide de modèle de donateurs sont livrées aux cellules par l’intermédiaire de base électroporation. Putativement édités cellules exprimant les protéines fluorescent étiquetées sont enrichis par cellule à fluorescence activé triant (FACS). Lignées clonales sont alors générées et peuvent être analysées pour résultats de montage précis. En introduisant la balise fluorescente au locus génomique du gène d’intérêt, la localisation subcellulaire qui en résulte et la dynamique de la protéine de fusion peut être étudiée sous contrôle réglementaire endogène, une amélioration clé sur la surexpression classique systèmes. L’utilisation de hiPSCs comme un système modèle pour le gène marquage fournit l’occasion d’étudier les protéines étiquetées dans les cellules diploïdes, non transformées. Étant donné que les hiPSCs peuvent être différenciés dans plusieurs types de cellules, cette approche offre la possibilité de créer et d’étudier les protéines étiquetées dans une variété de contextes cellulaires isogéniques.
Introduction
L’utilisation de stratégies de modification du génome, surtout CRISPR/Cas9, à étudier les processus cellulaires devient de plus en plus accessible et plus précieux1,2,3,4,5, 6 , 7. une des nombreuses applications de CRISPR/Cas9 est l’introduction (via la réparation (HDR) de l’homologie réalisée) des grandes séquences exogènes comme GFP dans des loci génomiques spécifiques qui servent ensuite de reporters pour l’activité d’un gène ou une protéine produit8 . Cette technique peut être utilisée pour joindre une séquence de la protéine fluorescente à un cadre de lecture ouvert endogène où la protéine de fusion endogène réglementé qui en résulte peut être utilisée pour visualiser la localisation subcellulaire et la dynamique de la protéine d’intérêt5 ,6,9,10,11. Alors que les protéines étiquetées de façon endogène offrent de nombreux avantages par rapport aux systèmes de surexpression, insérant des grandes séquences du génome humain est un processus inefficace exigent généralement une sélection ou une stratégie d’enrichissement pour obtenir une population de cellules qui peuvent être facilement étudié5,12.
Ce protocole décrit l’insertion d’une séquence d’ADN codant pour une protéine fluorescente (FP) dans un locus génomique désiré. Le protocole prévoit la conception et la livraison du plasmide modèle donateur et le complexe ribonucléoprotéique (RNP) (protéine de type sauvage S. pyogenes Cas9 combiné avec l’ARN synthétique de CRISPR (crRNA) et activation de trans crRNA (tracrRNA)). Également décrit est l’enrichissement des cellules putativement édités par cellule à fluorescence activé triant (FACS) et le processus de génération de ligne de cellules clonales. A ce jour, cette méthode a été utilisée pour générer des lignes de hiPSC avec monoallelic ou balises (rarement) bi-alléliques protéine fluorescente verte (GFP), étiquetage des vingt-cinq protéines représentant les principales structures cellulaires. Les cellules édités résultants de ces efforts ont été confirmés ont l’insertion génétique attendue, exprimer une protéine de fusion localiser correctement, et maintenir la pluripotence et un caryotype stable12 (et des données non publiées). Cette méthode a également été utilisée pour générer plusieurs autre single et double (deux différentes protéines marquées dans la même cellule) sous la direction des populations de hiPSCs (données non publiées).
CISP humains provenant d’un donneur sain ont été choisis pour ces efforts de modification du génome car, contrairement à nombreuses lignées classiques, ils sont diploïdes, caryotype stable, non transformées et prolifératives. Ces propriétés fournissent un modèle attrayant pour l’étude de la biologie cellulaire fondamentale et modélisation de la maladie. En outre, le potentiel de différenciation des hiPSCs offre la possibilité d’étudier plusieurs stades de développement en parallèle à travers diverses lignées et des types de cellules en utilisant des cellules isogéniques, y compris les organoids, les tissus et les modèles « maladie dans un plat »13 ,14,15. Alors que ce protocole a été développé pour hiPSCs (ligne WTC), il peut être instructif pour l’élaboration de protocoles à l’aide d’autres lignées cellulaires de mammifères.
Protocole
1. conception in Silico de crRNA et de donateurs modèle plasmide pour FP Knock-in
- Obtenir la séquence de référence annoté du NCBI16 ou l' UCSC Genome Browser17 (p. ex. format de GenBank) du gène d’intérêt et l’importer dans un logiciel bioinformatique de choix. Si la séquence du génome hôte est connue pour contenir des variantes par rapport à la référence, sont maintenant en ajustant la séquence de référence dans le logiciel bioinformatique (voir Discussion).
- Localiser le site d’insertion FP désiré. Pour les balises de C-terminal, la séquence pour le tag FP sera introduite entre la dernière base du codon dernière et la première base du codon stop. Pour N-terminale de marquage, la séquence pour le tag FP sera généralement introduite entre la dernière base du codon start et la première base du codon suivant. Dans certains cas, notamment lorsque le codon de départ est un exon codon unique, ou lorsqu’une séquence signal existe près du terminus de la protéine, le site d’insertion FP désiré peut être situé dans une position 3ʹ plus, tant qu’il continue d’être dans le cadre.
- Utilisation 50 bp sur chaque côté du site d’insertion souhaité comme la séquence d’entrée pour n’importe quel outil de conception de crRNA accessibles au public. Une fois 2-4 crRNA cibles identifiés près du site d’insertion, annoter les accepteurs crRNA et séquences adjacentes protospacer motif (PAM) (NGG) dans le logiciel bioinformatique. Ces crRNAs serviront pour induire des doubles sauts échoués (voir la Discussion pour obtenir des directives sur la conception de crRNA).
Remarque : Des séquences de crRNA Custom peuvent être présentées pour la synthèse avec un fournisseur commercial (recommandé), ou la séquence peut servir comme point de départ pour concevoir une stratégie de synthèse clonage ou in vitro , qui déborde le cadre du présent protocole (voir la Discussion ). - Pour lancer le plasmide de modèle de donateurs, utilisez 1 kb de séquences en amont du site d’insertion souhaité comme le bras d’homologie de 5ʹ (cela doit inclure le codon de début pour les insertions de N-terminal) et 1 Ko de séquence en aval du site d’insertion souhaité comme la 3ʹ bras d’homologie (cela doit inclure le codon d’insertions C-terminal). Entre les deux branches de l’homologie des bases ne sont généralement pas omis. Y compris les variantes spécifiques de la lignée cellulaire dans les bras de l’homologie préservera ces variants génétiques dans les cellules modifiés qui en résulte.
- Entre les deux branches de l’homologie, insérez la séquence pour la FP (ou autre séquence knock-in) et la séquence de l’éditeur de liens (voir la Discussion pour obtenir des directives sur les linkers). Pour les balises de N-terminal, la séquence de l’éditeur de liens doit être directement 3ʹ du FP ; pour les balises de C-terminal, la séquence de l’éditeur de liens doit être directement 5ʹ du FP.
- Perturber des sites de fixation de crRNA dans le plasmide de modèle de donateurs pour empêcher Cas9 couper des séquence de donateurs (voir la Discussion pour des considérations lors de la modification des sites de liaison crRNA). Si possible, la perturbation de la PAM à une séquence autre que NGG ou NAG est préférée. Alternativement, l’introduction de mutations ponctuelles à trois bases dans la région de la semence de la crRNA (10 bases proximales à la PAM) prévoit suffisamment perturber la liaison crRNA. Certains sites de liaison crRNA sont perturbés par l’introduction de la séquence FP dans le plasmide de modèle donateur ; Assurez-vous qu’aucun PAM, ou région de liaison intacte n’existe toujours dans ces cas.
NOTE : In silico le plasmide modèle donateur peut être présenté pour la synthèse de gènes par un fournisseur commercial, ou il peut être utilisé comme point de départ pour concevoir une stratégie de clonage qui déborde le cadre du présent protocole. Un squelette simple comme pUC19 ou pUC57 est suffisant.
2. ribonucléoprotéines (RNP) de Transfection pour CRISPR/Cas9 médiée par Knock-in en hiPSCs
Remarque : Dans le présent protocole, le terme « gRNA » décrit crRNA synthétique et tracrRNA correctement remises en suspension, quantifié et pré-complexé par les instructions du fabricant (voir Table des matières). Compléter tous les médias avec 1 % pénicilline streptomycine. Directives générales de culture de la ligne de hiPSC WTC sont décrites plus en détail à l’Explorateur de cellules de Allen18,19. WTC hiPSCs sont utilisés dans le présent protocole, mais avec optimisation de transfection correcte, électroporation de RNP et plasmide modèle donneur peut être adaptée avec succès à d’autres types de cellules.
- Préparer 10 stocks de travail µM de gRNA et de type sauvage S. pyogenes Cas9 protéine2,20; rester sur la glace. Préparer 1 µg/µL travail stock du plasmide de donneur de modèle ; conserver à température ambiante (RT). Utiliser le tampon TE pH 8.0 pour toutes les dilutions.
- Préparer une plaque de culture de tissu enduit de matrice 6 puits avec 5 mL de milieux de culture fraîche additionné d’inhibiteur de ROCK 10 µM (Ri) / puits. Garder la plaque avec les médias dans l’incubateur à 37 ° C et 5 % de CO2 jusqu'a aux cellules de la plaque après l’intervention de transfection (maximum 2 h).
Remarque : Toutes les plaques de revêtement matrice utilisées dans le présent protocole sont faits en ajoutant qu'un volume de Matrigel glacé dilué 01:30 dans les médias DMEM/F12 froids selon l’Institut Allen pour le protocole de la cellule de la Science pour cultiver le WTC hiPSC ligne19. - Utilisant un réactif douce unicellulaire dissociation tel que recommandé dans la Table des matières, le passage des hiPSCs dans les cellules à l’aide d’un compteur de cellules automatisées ou un hémocytomètre de la suspension et comte unicellulaires.
Remarque : Un protocole détaillé pour la ligne de hiPSC WTC utilisé ici se trouvent à l' Explorateur de cellules de Allen19. En bref, laver les cellules avec RT SPD et traiter avec le réactif de dissociation pendant 3-5 minutes. Puis triturer les cellules en suspension monocellulaire de pipetage doux et pellet par centrifugation. Resuspendre le culot de cellules vivantes dans des milieux de culture additionnés de 10 µM Ri.- Préparer une partie aliquote de 1,84 x 106 cellules pour chaque condition expérimentale à transfecter dans des tubes distincts 1,5 mL.
Remarque : Tous les volumes sont calculés pour un volume de réaction totale 4,5 µL dans un volume d’électroporation de 100 µL, temps 2,3 réactions. Cela représente pour la transfection en double et excès pour erreur de pipettage.
- Préparer une partie aliquote de 1,84 x 106 cellules pour chaque condition expérimentale à transfecter dans des tubes distincts 1,5 mL.
- Préparer les ribonucléoprotéines (RNP) tubes complexes pour chaque condition expérimentale en ajoutant 2,88 µL de 10 µM gRNA et 2,88 µL de 10 µM Cas9 dans un tube de 1,5 mL. Incuber à RT pendant au moins 10 minutes (maximum 1 h).
- Une cellule aliquote (préparée à l’étape 2.3.1) à 211 x g pendant 3 min à RT. aspirer surnageant de granule et Resuspendre le culot dans 220 µL de tampon d’électroporation du fabricant.
- Ajouter 220 µL de cellules resuspendues étape 2.5 dans le tube de complexe RNP 1,5 mL préparé à l’étape 2.4.
- Ajouter 4,60 µL de 1 µg/µL donateur modèle plasmide dans le tube de 1,5 mL préparé à l’étape 2.4.
- Utiliser la pointe de nucleofection et la pipette pour mélanger le contenu du tube de 2 - 3 fois, puis transférer 100 µL de suspension à l’appareil d’électroporation préparés. Éviter d’introduire les bulles dans la pointe. Appliquer 1300 V pour 1 impulsion de 30 ms.
- Doucement, transférer la suspension dans la plaque de 6 puits préparée de l’étape 2.2 avec un mouvement tourbillonnant. Disperser les cellules en déplaçant légèrement la plaque à côté et front-to-back.
- Utilisant un nouvel embout de nucleofection, répétez les étapes 2,8 et 2,9 avec le reste 100 µL de la suspension et le transfert dans un deuxième puits de la plaque 6 puits préparé.
- Répétez les étapes 2.4 par 2.10 pour chaque combinaison de plasmide du modèle gRNA et donateurs, dont les seuls contrôles tampon ne pointeraient gRNA et plasmide de modèle donneur seulement. Prenez soin de changer de pipette et nucleofection conseils pour éviter la contamination croisée.
- Incuber les cellules transfectées à 37 ° C et 5 % de CO2. Changer les médias aux milieux de croissance régulière (pas Ri) à 24 h, puis continuer alimentation hiPSCs toutes les 24 h pendant 72-96 h, suivi de confluence. Quand hiPSCs atteignent 60 à 80 % confluence, passez à l’étape 3.
NOTE : La mort des cellules lourde (> 70 %, estimée) est normal 24-48 h après la transfection.
3. FACS-enrichissement de hiPSCs putativement édité
Remarque : Lors du tri des cellules souches, adapter les paramètres de l’instrument afin de promouvoir la survie des cellules comme suggéré dans la Discussion. En bref, utilisez la buse plus grande possible (130 µm), un faible débit (≤ 24 µL/min), liquide de gaine sans conservateur (comme le sérum physiologique, voir la Table des matières) et pression faible échantillon (10 lb/po2).
- Avant de commencer une expérience de FACS, changer des médias à des milieux de culture additionnés de 10 µM Ri et incuber les cellules à 37 ° C et 5 % de CO2 pour 2-4 h pour favoriser la survie après FACS.
- Utilisant un réactif douce unicellulaire dissociation tel que recommandé dans la Table des matières, le passage des hiPSCs en suspension monocellulaire dans les milieux de culture additionnés de 10 µM Ri19.
- Filtrer hiPSC suspension filtre à tamis 35 µm dans des tubes en polystyrène à fond rond.
- Trier les cellules à l’aide de forward scatter et diffusion latérale (y compris les hauteur et largeur) d’exclure les débris et les doublets. Usage direct, tampon seulement contrôlent les cellules pour définir la porte FP-positifs, tels que < 0,1 % de cellules seul tampon relèvent de la porte.
- Trier l’ensemble de la population de FP-positifs des cellules dans un tube en polypropylène de 1,5 à 15 mL contenant de 0,5 à 2 mL de milieux de culture RT additionné de 10 µM Ri.
NOTE : Polypropylène réduit le potentiel d’adhérence cellulaire au plastique. - Centrifuger les cellules recueillies à 211 g pendant 3 min à température ambiante.
- Avec précaution, aspirer le surnageant et Resuspendre le culot dans 200 µL de milieux de culture additionné de 10 µM Ri. Jusqu'à 3 000 cellules triées de transfert d’un seul puits d’une plaque de 96 puits de matrice-enduit frais19.
Remarque : Avec réglage de l’instrument approprié, les cellules peuvent également être triés (en vrac) directement dans un seul puits d’une plaque de culture de tissu enduit de matrice 96 puits contenant 200 µL de milieux de culture additionné de 10 µM Ri à une densité recommandée de 1 000-3 000 cellules / puits pour hiPSC. - Incuber les cellules triées à 37 ° C et 5 % de CO2. Modifier les médias à des milieux de culture additionnés de 5 µM Ri après 24 h. 48 h commence à se nourrir des milieux de croissance régulière des cellules (aucun Ri) toutes les 24 h pendant 72-96 h, suivi de confluence. Survie après que FACS est estimée à plus de 50 % si un minimum de 500 cellules est ensemencé dans un puits d’une plaque de 96 puits.
- Lorsque hiPSCs atteindre 60 à 80 % confluence, apparaissent des mature morphologie (centres de colonie lisse, bien emballé), passage à une plaque de format plus grande comme une plaque 24 puits, puis d’une plaque 24 puits dans une plaque de 6 puits.
- Une fois les hiPSCs dans une plaque 6 puits atteignent la confluence de 60 à 80 % et matures morphologie, étendre sur une plaque de 100 mm, re-plaque d’imagerie, cryoconservé ou graines à clone cueillette densité (étape 4)19.
4. générer putativement clonale édité hiPSC lignes
- Utilisant un réactif douce unicellulaire dissociation tel que recommandé dans la Table des matières, hiPSCs de passage en suspension monocellulaire et déterminer le nombre de cellules par mL,19.
- Cellules de semences 10 000 de la population éditée de hiPSCs sur un plateau de culture de tissus frais 100 mm enduit de matrice à l’aide de milieux de culture additionné de 10 µM Ri19. Modifier les médias à des milieux de culture sans Ri 24 h après l’ensemencement et nourrir les hiPSCs avec les milieux de culture fraîche toutes les 24 h pendant 5 à 7 jours.
- Quand les hiPSCs ont formé des colonies qui sont visibles à le œil nu (environ 500 µm) ils sont assez grands pour être isolé. Préparer une plaque à 96 puits matrice-enduit en aspirant de l’excès matrice et en ajoutant 100 µL de milieux de culture additionné de 10 µM Ri par bien19.
- Sur une dissection pipette d’utiliser un P-200 de microscope, ou similaire, pour gratter et aspirer des colonies individuelles de la surface de la plaque en douceur. Volume de transfert (~ 20-100 µL) contenant la colonie à un seul puits de la plaque à 96 puits préparé à l’étape 4.3.
- Après que toutes les colonies ont été transférées, incuber la plaque dans un incubateur de culture tissulaire à 37˚C et 5 % de CO2. Modifier les médias aux milieux de croissance régulière (pas Ri) à 24 h et continuer à nourrir les cellules toutes les 24 h pendant 72-96 h jusqu'à ce que les colonies ont environ triplé dans la taille (environ 1500 µm).
Remarque : Cueillette des colonies 24 96 par crRNA utilisé dans la transfection est recommandé. Survie des clones isolés est généralement supérieure à 95 %.
- Après que toutes les colonies ont été transférées, incuber la plaque dans un incubateur de culture tissulaire à 37˚C et 5 % de CO2. Modifier les médias aux milieux de croissance régulière (pas Ri) à 24 h et continuer à nourrir les cellules toutes les 24 h pendant 72-96 h jusqu'à ce que les colonies ont environ triplé dans la taille (environ 1500 µm).
- Utilisant un réactif douce unicellulaire dissociation tel que recommandé dans la Table des matières, passage hiPSC clones dans une plaque à 96 puits nouvelle matrice-enduit comme suit19.
- À l’aide d’un aspirateur de 8 canaux, retirez et jetez les médias de la première colonne de la plaque à 96 puits.
- À l’aide d’une pipette multicanaux P-200, ajouter ~ 200 µL de SPD à la première colonne de la plaque à 96 puits à laver les cellules. À l’aide d’un aspirateur de 8 canaux, retirez et jetez le SPD lavage de la première colonne de la plaque à 96 puits.
- À l’aide d’une pipette multicanaux P-200, ajouter 40 µL de réactif de dissociation de la première colonne de la plaque à 96 puits.
- Répétez les étapes 4.5.1-4.5.3 pour un maximum de six colonnes de la plaque à 96 puits, changer la pointe pour être sûr de ne pas croisée puits. Placer la plaque dans un incubateur à 37 ° C pendant 3 à 5 minutes de l’époque, que le réactif de dissociation a été ajouté à la première colonne (étape 4.5.3).
Remarque : Il est recommandé de passage seulement un maximum de six colonnes (48 puits) à la fois en raison du temps qu’il faut pour effectuer ces étapes. Lorsque vous effectuez ce protocole pour la première fois, commencez par passage seulement une ou deux colonnes à la fois. Limiter le nombre de colonnes repiquées en même temps s’assure que les cellules ne soient pas laissés dans le réactif de dissociation pendant trop longtemps, ce qui pourrait nuire à la hiPSCs. - Lorsque les cellules de la première colonne de la plaque ont commencé à décoller du fond de la boîte, utilisez une pipette multicanaux de P-200 pour ajouter 160 µL de SPD dans la première colonne de la plaque à 96 puits et triturer doucement les cellules à la « 12:00 » , « 03:00 », « 06:00 "et « 09:00" des positions de chaque puits. Transférer la totalité du volume de suspension cellulaire (200 µL) dans une plaque de fond en V 96 puits.
- Répétez l’étape 4.5.5 pour les autres colonnes de cellules qui ont réactif de dissociation en eux ; changer des conseils quant à la non contamination croisée puits.
- Faites tourner la plaque de fond V dans une centrifugeuse à 385 x g pendant 3 min à température ambiante.
- À l’aide d’une pipette multicanaux P-200, retirez délicatement le surnageant et remettre en suspension des cellules dans 200 µL de milieux de culture fraîche additionné de 10 µM Ri / puits. Répétez pour tous les puits, changeant des conseils quant à ne pas la contamination croisée.
- Transférer l’ensemble de la suspension cellulaire à une plaque de 96 puits de matrice-enduit frais19. Incuber la plaque dans un incubateur de culture tissulaire à 37 ° C et 5 % de CO2. Modifier les médias aux milieux de croissance régulière (pas Ri) à 24 h et continuer à nourrir les cellules toutes les 24 h pendant 72-96 heures, jusqu'à ce que la majorité des clones atteindre la confluence de 60 à 80 %.
NOTE : Ce passage permet d’étaler les cellules et permettre une croissance plus sur toute la surface de la plaque à 96 puits.
- Observer des clones et de déterminer un ratio de partage approprié pour chaque clone individuel dans la plaque à 96 puits (p. ex., 01:10, ou 1:8). En utilisant un réactif douce unicellulaire dissociation comme suggéré dans la Table des matières, passage hiPSC clones dans une nouvelle matrice-enduit plaque à 96 puits (mesures 4.5.1-4.5.8) transférant un ratio de la suspension cellulaire appropriée pour chaque clone. Incuber la plaque dans un incubateur de culture tissulaire à 37˚C et 5 % de CO2. Modifier les médias aux milieux de croissance régulière (pas Ri) à 24 h et continuer à nourrir les cellules toutes les 24 h pendant 72-96 heures, jusqu'à ce que la majorité des clones atteindre la confluence de 60 à 80 % et montrent une morphologie mature.
Remarque : Chaque clone peut avoir un ratio de partage différents en raison des taux de croissance légèrement différent ou la survie du passage précédent, donc ce passage contribue à normaliser le nombre de cellules par puits de chaque clone pour l’étape de congélation à suivre. En raison des différents taux de survie et de croissance, certains clones peuvent proliférer ou ne pas se développer au cours de ces étapes de passage.- Sauver le reste de la suspension cellulaire et granule pour ADNg isolation par centrifugation des cellules dans une plaque de fond en V 96 puits à 385 x g pendant 3 min à RT. Remove surnageant et procéder à l’isolement de génomique à l’aide d’un kit de 96 puits ou stocker des plaque de cellules granulés à - 20 ° c pour jusqu'à thr semaines, EE.
5. cryoconservation des lignées clonales dans le format de la plaque à 96 puits
- À l’aide d’un réactif de dissociation unicellulaire, le passage des clones hiPSC comme décrits précédemment (pas 4.5.1-4.5.7). Aspirer le surnageant à l’aide d’une pipette multicanaux P-200 et remettre en suspension dans 60 µL de milieux de culture additionné de 10 µM Ri. Répétez pour tous les puits ; changer des conseils quant à ne pas la contamination croisée.
- Transférer 30 µL de suspension cellulaire à une plaque de matrice non couché vitroplants à 96 puits. Puis ajouter rapidement 170 µL de tampon de gel (voir Table des matières) dans chaque puits, sans mélange. Répéter en transférant le reste 30 µL de suspension dans une sœur plaque et ajouter 170 µL de congélation de la mémoire tampon.
Remarque : Ce processus se fait en duplicate soeur plaques afin qu’une population de sauvegarde des cellules existe après décongélation une des plaques individuelles. Mettre des cellules cryoconservés en seulement toutes les autres colonnes d’une plaque 96 puits permet de dégel plus rapide (point 5.6). - Enrouler la plaque avec du parafilm et placer dans une boîte de Styrofoam RT avec couvercle. Placez la boîte entière dans un congélateur à-80 ° C.
- Après 24h, plaques peuvent être transférés hors de la boîte de styromousse et conservés à - 80 ˚c jusqu'à quatre semaines.
Remarque : Alors que les cellules sont temporairement stockées à-80 ° C, des essais de contrôle de la qualité génétique peuvent être effectuées avec l’ADNg récoltée à partir des cellules obtenues à l’étape 4.6.1 afin d’identifier les clones pour décongeler et propager de plus, comme nous le verrons dans les travaux déjà publié 12. brièvement, une copie numéro gouttelette numérique PCR peut être utilisée pour identifier les clones qui contiennent un ou deux exemplaires de la GFP et aucune intégration donateur modèle plasmide épine dorsale. Une combinaison de tests de PCR point final et Sanger séquençage peut alors identifier les clones contenant un insert précis. - Pour décongeler, porter la plaque entière à 37 ° C dans un incubateur de culture tissulaire, regarder attentivement pour les pellets de glace à fondre. Puits au bord de la plaque ont tendance à fondre tout d’abord.
- Lorsque les pellets de glace du clone désiré fond, doucement transférer toute 200 µL dans un tube conique 15 mL contenant 3 mL de milieu de culture de RT additionné de 10 µM Ri et centrifuger à 211 x g pendant 3 min à température ambiante.
- Aspirer le surnageant et remettre en suspension les cellules boulettes dans 1 mL de milieux de culture RT additionné de 10 µM Ri. Transférer sur une plaque de 24 puits matrice-enduit frais et incuber à 37˚C et 5 % de CO2. Modifier les médias aux milieux de croissance régulière (pas Ri) à 24 h et continuer à nourrir les cellules toutes les 24 h pendant 72-96 h jusqu'à ce que le clone atteint confluence de 60 à 80 % et a la morphologie mature19.
Résultats
L’objectif de cette expérience était de fusible mEGFP (monomère GFP renforcée) à la protéine nucléaire de B1 lagoutte en introduisant la séquence de mEGFP à la fin de la 5ʹ du gène LMNB1 (extrémité N-terminale de la protéine). Un linker (séquence d’acides aminés SGLRSRAQAS) a été choisi en fonction des constructions précédentes de cDNA de la Collection Michael Davidson Fluorescent Protein21. Parce que la région de liaison de crRNA dans le plasmide de modèle du donneur pour chaque crRNA de candidat a été interrompue après l’insertion in silico de mEGFP et la séquence de l’éditeur de liens, il fallait pas mutations ponctuelles de perturber les potentiels crRNA reconnaissance et clivage de Cas9 de la séquence de donateurs (Figure 1). La séquence de donateurs contenait des armes d’homologie 1KO flanquant les deux extrémités de la séquence mEGFP-linker. La résultante 2 734 bp d’ADN a été cloné dans un squelette pUC57, séquence vérifié, et le plasmide de modèle donneur qui en résulte a été purifié à l’aide d’une préparation exempte d’endotoxine Maxie. Le plasmide modèle donneur et RNP complexe ont été transfectées, cellules putativement édités ont été enrichies et la localisation de la protéine de fusion B1 lagreve mEGFP nucléaire a été confirmée par microscopie de fluorescence (Figure 2). Seuls les résultats de la transfection de crRNA1 sont décrits ici, bien que les deux séquences de crRNA produit des populations putativement édité12.
Comparativement au témoin négatif, qui ne contenait aucune gRNA, protéine Cas9 ou plasmide modèle donateur dans la réaction d’électroporation (tampon seulement), le crRNA1 LMNB1 transfectées les cellules contiennent des cellules positives à le mEGFP de 0,95 % représentant la putativement édité population de B1 lagreve mEGFP-nucléaire (Figure 3 a). Ce résultat a été portée de knock-in efficacité observée à travers plusieurs locus génomiques à l’aide de cette méthode, comme indiqué précédemment12.
Les cellules mEGFP séropositifs ont été isolés par FACS et imagés par microscopie direct pour confirmer la localisation prévue de la mEGFP nucléaire lagoutte B1 protéine de fusion à l’enveloppe nucléaire. Après enrichissement de FACS, environ 90 % des cellules triées de la population de crRNA1 de LMNB1 ont été mEGFP-positive (tel que déterminé par microscopie), ce qui suggère que certaines cellules mEGFP-négatif co purifiés avec les cellules de GFP-positifs au cours de la procédure de tri. Il s’agissait d’un niveau acceptable d’enrichissement qui a permis à la récolte de 96 clones qui pourrait ensuite être génétiquement projetés pour l’édition réussie. Généralement, un seuil pour l’enrichissement de réussite est de 50 % positifs GFP.
La majorité de la population cellulaire trié affichée de fluorescence à l’enveloppe nucléaire (nuclear périphérie) dans les cellules ne se divisant pas et à une lamina nucléaire étendu dans le cytoplasme au cours de la mitose donnant confiance dans le bon génomique éditer à la LMNB1 locus. La population enrichie contenue des cellules avec signal lumineux ou dim. Cette différence d’intensité du signal peut refléter une combinaison de correctes et incorrectes d’éditer les résultats et les faits saillants de l’utilitaire de génération d’une lignée clonale génétiquement validée pour davantage d’études (voir Discussion) (Figure 3 b)12. Après la génération de la lignée clonale, génétiquement validé les cellules montrés l’intensité uniforme de GFP dans des expériences de microscopie (Figure 3C).
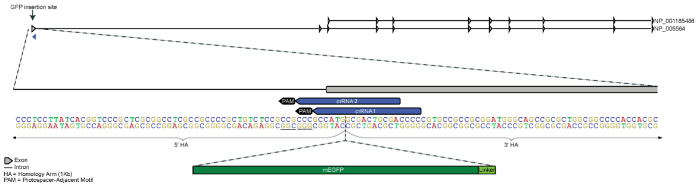
Figure 1 . Concevoir la stratégie pour le marquage de GFP de l’extrémité N-terminale du gène LMNB1. La balise de la GFP a été conçue pour N-terminale d’insertion 5ʹ du premier exon de LMNB1 situé sur le chromosome 5. Homologie des 5ʹ et 3ʹ d’armes sont 1 Ko chacun et se rencontrent entre le codon de début (ATG) et le second codon (armes d’homologie que partiellement représentées dans la figure). Deux candidats crRNAs ont été conçus pour guider Cas9 s’attacher aussi près du site d’insertion prévu que possible, tout en étant unique dans le génome. Séquence pour mEGFP et un éditeur de liens des acides aminés ont été insérées juste 3ʹ du codon start (séquence mEGFP et linker ne pas à l’échelle). S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.

Figure 2 . Workflow de production endogène tag hiPSC lignées clonales. Composants de transfection, y compris le complexe RNP Cas9/crRNA/tracrRNA (montré aussi rouge Cas9 protéine avec crRNA or et pourpre tracrRNA), le plasmide de modèle donneur contenant les armoiries d’homologie (HAs, montrés en or) et FP + séquence linker (indiqué en vert), ont été électroporation. Après 4 jours, les cellules positives FP putativement édité ont été enrichies par FACS élargis comme une population de toutes les cellules triées ensemencement dans un seul puits d’une plaque de 96 puits (~ 1 000 cellules) et ensuite élargis en culture jusqu’au travail plusieurs millions d’habitants cellules pourraient être dosés comme la « population enrichie » (voir protocole étape 3,8). Le rendement des cellules positives FP se distingue par l’expérience grâce à des taux variables de HDR12; un enrichissement réussi peut inclure généralement ~ 300-5 000 cellules après la transfection d’environ 1,6 x 106 hanches. Les études d’imagerie préliminaires a confirmé le signal et la localisation de la protéine de fusion dans la population enrichie. Colonies ont été cueillies manuellement dans une plaque à 96 puits pour l’expansion et la cryoconservation. Outre le dépistage génomique contrôle de la qualité à l’aide de gouttelettes digital PCR (ddPCR) et autres analyses PCR a ensuite été utilisé pour identifier des clones édités correctement, comme décrit précédemment12. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.

Figure 3 . Enrichissement des populations cellulaires putativement edited. (A) écoulement cytometry parcelles de le LMNB1 édité après transfection de cellules quatre jours. L’axe y affiche intensité GFP et l’axe des abscisses avant dispersion. Gates tris ont été définies selon la commande tampon seulement. Puisque les hiPSCs sont sensibles aux perturbations, live/dé tache a été omis et une porte très conservatrice de FSC/SSC a été utilisée à la place. (B) après l’enrichissement, la population de LMNB1 Cr1 édition cellules ont montré environ 90 % des cellules avec GFP localisation à l’enveloppe nucléaire (localisation de LMNB1 attendue). La population contenait des cellules d’intensité variable de GFP ainsi que certaines cellules de GFP-négatif. Barreaux de l’échelle est de 10 microns. (C) après la génération de la lignée clonale, cellules montrent une intensité uniforme de GFP, avec quelques différences dépendant du cycle cellulaire. Barre d’échelle est de 20 microns.
Discussion
La méthode présentée ici pour générer de façon endogène sont réglementés par des fusions de protéine fluorescente dans hiPSCs est une approche souple et puissante pour générer des lignées cellulaires de gène édité avec des applications allant de l’imagerie de cellules vivantes à diverses études fonctionnelles et » modèles de maladies dans un plat » à l’aide de dérivés de patient hiPSC lignes13,14,15. Bien que cette méthode a servi à introduire de grandes étiquettes FP à la N - ou C-terminale de protéines endogènes, il susceptibles de servir à présenter autres balises ou des petits changements génétiques de modèle ou pathogènes correcte des mutations22,23 . Pour les insertions plus petites, la taille du bras homologie peut être réduite, mais l’approche générale présentée dans cette méthode d’édition peut-être toujours s’appliquer24,25. Tandis que l’utilisation de hiPSCs est fortement encouragée pour leur grande utilité, avec optimisation de l’attention, ce protocole peut être adapté afin de modifier le génome d’autres lignées cellulaires de mammifères.
Lorsqu’on veut identifier un gène d’intérêt pour le marquage de FP, estimations de l’abondance de la transcription (à partir de « microarray » ou données de RNA-Seq) est un bon point de départ pour déterminer si un gène ou une isoforme d’intérêt est exprimé, bien que les niveaux de transcription ne concordent pas toujours avec taux de protéines. La stratégie de FACS-enrichissement décrite ici qui fonctionnera le mieux pour les gènes qui sont exprimés au moins modérément bien dans le type de cellules d’intérêt. Cette stratégie a également été réussie en choisissant des fusions qui montrent des phénomènes de localisation ponctuée et/ou discrètes comme centrine, desmoplakine et paxillin où le rapport fond signal est très faible12,19. Les gènes qui ne sont pas fortement exprimées ou sont exprimés uniquement dans les types dérivés de cellules peuvent exiger des stratégies de sélection supplémentaires.
Le point de départ pour crRNA et donateurs plasmide modèles utilisés dans des lignées cellulaires humaines devrait être le génome humain de référence (GRCh38). Parce que les génomes de différentes lignées cellulaires peuvent varier au sein de la même espèce, et que CRISPR/Cas9 est séquence-spécifique, il est très utile identifier les variantes de ligne spécifique de cellule (polymorphismes mononucléotidiques ou insertions/délétions (indels)) qui diffèrent du génome de référence et incorporer dans la conception. Cela garantit que crRNAs sera compatible avec le génome de l’hôte et que les bras d’homologie donateur modèle plasmide seront conservé pour les éventuelles variantes spécifiques de la lignée cellulaire. Une stratégie suggérée est d’intégrer les variantes homozygotes dans bras d’homologie du plasmide de modèle de crRNAs et donateur au cours du processus de conception. Incorporant des variantes hétérozygotes est facultative. Les réactifs spécifiques utilisés pour grandes expériences knock-in et autres considérations clés de ce protocole sont examinées ci-dessous.
Protéine de Cas9
Le principal avantage de l’utilisation de protéines Cas9 est celle présentant le Cas9 et gRNA comme un complexe RNP s’est avéré résulter en une durée limitée de l’activité nucléasique par rapport aux approches axées sur le plasmide où expression des Cas9 et gRNA peut continuer pendant des jours et conduire sur - et hors-cible à une plus grande activité26,27. Un autre avantage de l’utilisation de protéines Cas9, c’est qu’il est facilement disponible en conjonction avec une fois à l’intérieur des cellules. Cela contraste avec les méthodes plus conventionnelles d’utiliser Cas9 ARNm ou Cas9/gRNA plasmide nécessitant une transcription, traduction et protéine traitement26,28. Protéine S. pyogenes Cas9 type sauvage est maintenant disponible auprès de nombreuses sources commerciales.
Guide de l’ARN
Il existe de nombreux outils accessibles au public pour trouver les cibles de crRNA près du site d’insertion FP désiré qui ont zéro ou peu estimées hors-cibles dans l’hôte du génome29,30,31,32. Gains d’efficience en HDR et la précision des résultats HDR varient considérablement de crRNA cibles utilisées à un locus donné12. Pour cette raison, les tests crRNAs plusieurs (2-4 et de préférence moins de 50 bp du site d’insertion souhaité) par locus est recommandé car cela peut augmenter la probabilité d’une expérience d’édition réussie. Possibilités de livraison gRNA incluent synthétiques 2-partie crRNA et tracrRNA, synthétique unique gRNAs (sgRNAs), in vitro transcrit sgRNAs, ou livrer un plasmide de cellules exprimant le sgRNA partir d’un promoteur U6. Ce protocole ne était pas optimisé pour l’activité élevée de clivage. CrRNA de 2 parties non modifiée et tracrRNA (voir la Table des matières) ont été utilisés dans le but de générer des lignées de cellules mono-alléliques de FP-tag tout en causant la moins possible perturbation aux cellules.
Donateur modèle plasmide
Parce que certains de l’homologie arm séquence autant chez le donneur plasmide modèle est incorporée dans le génome hôte pendant l’événement HDR, mutations ponctuelles pour les sites de reconnaissance de crRNA devrait être introduites pour prévenir d’autres clivage par Cas9 suite HDR. Souvent, le changement de perturbateur plus simple consiste à muter la séquence de PAM. Parce que certaines séquences non canonique de PAM se distingues encore par sauvage S. pyogenes Cas9, il est préférable d’éviter d’utiliser NGG, NAG ou NGA33. Lorsque la mutation le bras d’homologie, éviter les mutations non-synonymes et l’introduction des codons rares. Si un changement synonyme à la séquence de PAM n’est pas possible, pensez à faire trois mutations synonymes de point dans la région de semences (10 bp proximal à PAM) du site de fixation crRNA. Doit être très prudent lorsque vous effectuez ces modifications dans la région non traduite de 5ʹ (UTR) car ces régions peuvent contenir des séquences régulatrices importants. Consulter une base de données de conservation génétique tels que les titres de génomique Comparative du navigateur UCSC Genome peuvent guider dans ces cas, comme changements aux bases non conservée peuvent être mieux tolérées que devient hautement conservée bases17. Parfois la simple insertion de la séquence de la FP est suffisant pour perturber le site de liaison de crRNA (comme dans la Figure 1) ; Cependant, la séquence nouvellement ajoutée doit être vérifiée pour la persistance des séquences de PAM et de la liaison crRNA.
Acide aminé linkers entre FP et la protéine native sont recommandés pour conserver la fonction de la protéine de fusion34. Souvent un linker acide aminé peut être choisi pour sa charge particulière ou de taille. Si une fusion d’ADNc avec un design similaire à le ciblées endogène protéine de fusion a été bien étudiée, que même linker séquence peut être utilisée pour le CRISPR/Cas9 knock-in expérience12,19. Si cette information n’est pas disponible, un éditeur de liens court tels que GTSGGS a également été utilisée avec succès12. Autres études ont démontré le succès avec une séquence de générique petit éditeur de liens acide amino-3 pour une variété d’objectifs35.
La transfection et l’enrichissement des FACS
De nombreux réactifs de transfection disponibles dans le commerce sont formulés pour la livraison de certains types de molécules, de cellules, alors qu’un système d’électroporation permet de livrer des réactifs avec un large éventail de taille, charge et composition. En plus d’être une méthode commune de transfection de cellules de dur-à-transfecter comme hiPSCs, électroporation réalise également l’avantage de fournir tous les trois composants pour FP CRISPR/Cas9-mediated knock-in tel que décrit dans cette méthode. Électroporation a été trouvée pour produire les meilleurs résultats par rapport aux autres réactifs disponibles dans le commerce dans l’élaboration de cette méthode (données non présentées) et a également été utilisé par d’autres pour le RNP livraison26,28,36 .
Lorsque vous utilisez ce protocole pour l’édition hiPSCs, spécial il faut pour assurer une manipulation douce des cellules avant et après le gène processus de survie optimale cellulaire et différenciation spontanée minimale de montage. En particulier, les méthodes d’enrichissement FACS doivent être adaptés pour le tri des cellules souches à l’aide de la buse plus grande possible (130 µm), un faible débit (≤24 µL/min), liquide de gaine sans agent de conservation (comme le sérum physiologique, voir la Table des matières) et l’échantillon faible pression (10 lb/po2). Au lieu de cellule unique tri, qui se traduit par viabilité sous-optimale dans les cellules souches, les hiPSCs enrichie en FACS sont triés en vrac et élargis comme une population pour optimiser l’intégrité de cellules souches et de la viabilité cellulaire. Cependant, il peut être approprié pour moins de types de cellules sensibles d’unicellulaire tri. Afin de favoriser la survie des cellules, cellules sont retournés à la culture n’est plus d’une heure après la récolte pour l’enrichissement de FACS et conservés à température ambiante pendant tout le processus de tri. Pour certains types de cellules, la survie des cellules peut également être améliorée par incubation des cellules sur la glace (4° C) pendant tout le processus de tri.
L’expansion en vrac des cellules positives FP fournit une occasion d’évaluer la population par l’analyse d’imagerie pour la localisation de protéine de fusion avant de générer des lignées clonales. Alors que la population enrichie résultante de cellules peut être suffisante pour certaines études, ces populations affichent fréquemment FP signal d’intensité variable. Les lignées clonales isolées ont signal uniforme (Figure 3), ce qui les rend plus approprié pour les expériences fonctionnelles12.
Génération de ligne de cellules clonales
Tout au long du processus de génération de ligne montage et clonale, il est important de surveiller la morphologie cellulaire. hiPSC colonies cultivées dans des conditions sans chargeur devraient montrer des bords lisses et un centre même, bien emballé12,18,19. Les cellules différenciées doivent être observées dans moins de 5 % de la culture. Lors de la cueillette des colonies individuelles, choisissez ceux que la morphologie bonne exposition. Au cours de la plaque à 96 puits passage des événements, vérifiez les clones pour la morphologie et arrêter ceux qui ont envahi car cela pourrait conduire à la différenciation ou être l’indice d’instabilité génétique.
Génération de lignées clonales permettant la confirmation génétique de montage précis, qui est important parce qu’induite par le Cas9 double brins dans le génome sont souvent réparé imprécisément malgré l’incorporation de la balise du locus désirée. Tests PCR décrites précédemment ont montré que cumulativement à travers dix locus génomiques uniques nombreux (45 %) des clones exprimant le FP a souffert de l’intégration de colonne vertébrale plasmide donneur au locus ciblé ou (rarement) au hasard dans le génome de12. En outre, 23 % des séropositifs au GFP clones (n = 177) dans dix lieux uniques trouvées pour héberger les mutations au ou près du site de coupe crRNA prévus dans l’allèle non balisé, probablement en raison de NHEJ12. Cette analyse génétique de nombreuses lignées clonales (~ 100 clones/edit) a souligné l’importance de validation génétique qui n’est pas possible dans une population de cellules puisque FP-expression et localisation des protéines fusion attendue seul ne garantit pas un montage précis12 . En outre, ces analyses basées sur la PCR ne peuvent être effectuées sur une population enrichie de cellules avec certitude, justifiant la nécessité pour la génération de la lignée clonale avant une analyse significative peut être complétée. Confirmation génétique de la balise insérée de FP et de vérification de l’intégrité génétique de l’allèle non éditée (dans un clone édité mono-alléliques) sont toutes deux nécessaires pour assurer un montage précis du locus ciblées au-delà l’expression de l’étiquette.
Un faible taux de modifications bi-alléliques et absence de mutations hors-cible (comme analysée par Sanger et séquençage de l’exome) ont été observés à ce jour à l’aide de cette méthode (données non publiées)12. Cela concorde avec les études précédentes décrivant l’utilisation du RNP de courte durée pour CRISPR/Cas9 expériences26,27. L’absence de lignées clonales avec bi-alléliques modifications peut aussi être locus spécifique ou en raison de l’incapacité de la cellule à tolérer deux tag copies d’une protéine essentielle, comme le suggèrent des expériences publiées antérieurement où étaient les cellules édités bi-alléliques putatifs observées pour un locus (LMNB1), mais pas un autre (TUBA1B)12. Bi-alléliques entièrement validée lignées clonales ont été générées à l’aide de cette méthode pour tag ST6 bêta-galactoside alpha-2, 6-sialyltransférase 1 (ST6GAL1) et membre de le RAB5A famille d’oncogène RAS (RAB5A) avec mEGFP19.
Au-delà de confirmer la précision du montage dans le génome, il existe une variété de tests de contrôle de la qualité qui peuvent être utilisés pour caractériser la lignée clonale et identifier les clones qui répondent à toutes les cellules souches, génomique, et critères biologiques de cellule pour utilisent à l’avenir des études . Analyses biologiques et fonctionnels des cellules peuvent être utilisés pour confirmer l’expression appropriée, localisation et fonction de la protéine de fusion12. La comparaison avec les contrôles parentaux aidera à évaluer l’influence du processus d’édition sur la localisation, la dynamique et la fonction. Autres tests tels que l’analyse de la croissance et des tests de stabilité génomique contribue également à déterminent si la protéine étiquetée est perturbante à la cellule. Lorsque vous utilisez hiPSC dans le présent protocole, évaluation des marqueurs pluripotence et potentiel de différenciation peut être critique dans la détermination d’un clone qui est précieux pour en aval d’études12. Parce que l’étendue de la culture de hiPSC a démontré que conduire à une instabilité génétique, suivi du taux de croissance et caryotype des lignées clonales est aussi important de12,37. Toutefois, la finale destinée utilisation des cellules édités déterminera en fin de compte le niveau et la profondeur de l’analyse de contrôle qualité et variera selon l’application.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Nous remercions Dambournet Daphné pour de nombreuses discussions perspicaces et conseils sur le gène édition, Thao à titre d’illustration, Angelique Nelson pour une lecture critique du manuscrit et Andrew Tucker pour la génération de la lignée de cellules B1 Lamin-le tag mEGFP. Nous tenons à souligner les cellules souches et les gène Editing et les équipes de développement de l’analyse à l’Institut Allen pour la Science des cellules pour leur contribution au processus de contrôle de la qualité et de gène d’édition. La ligne WTC qui nous permet de créer notre lignée génétique-édité a été fournie par le Bruce R. Conklin Laboratory aux instituts de Gladstone et UCSF. Nous remercions l’Institut Allen pour le fondateur de la Science des cellules, Paul G. Allen, pour sa vision, encouragement et appuyer.
matériels
| Name | Company | Catalog Number | Comments |
| Geneious R9 | Biomatters, or similar | bioinformatics software for in silico donor plasmid design | |
| TE Buffer pH 8.0 | IDT, or similar | 11-01-02-05 | |
| HERAcell VIOS 160i CO2 incubator, or similar | ThermoFisher Scientific, or similar | 51030408 | |
| Pipettes (1000 µL, 200 µL, 20 µL, 10 µL, 2 µL) | Rainin, or similar | ||
| Pipette tips (1000 µL, 200 µL, 20 µL) | Rainin, or similar | ||
| Multi-channel Pipette (200 µL) | Rainin, or similar | ||
| Serological pipetes (25 mL, 10 mL, 5 mL) | Costar, or similar | ||
| BRAND 8-Channel Manifold for Quiksip, Autoclavable | Millipore Sigma, or similar | BR704526-1EA | use with non-filtered pipet tips, such as Molecular BioProducts Low Retention Pipet Tips, Pure 10, below |
| Molecular BioProducts Low Retention Pipet Tips, Pure 10 | Thermo Fisher, or similar | 3501-05 | |
| XP2 Pipette Controller | Drummond, or similar | 4-000-501 | |
| Disposable Pasteur Pipets | VWR, or similar | 53300-567 | |
| Class II, Type A2 Biological Safety Cabinet | CELLGARD, or similar | NU-481 | |
| Matrigel Matrix, Growth Factor Reduced | Corning | 354230 | lot tested before use with hiPSC |
| DMEM/F12 (-phenol red) | Gibco | 11039-021 | cold, for diluting Matrigel 1:30 |
| mTeSR1 Complete Media | StemCell Technologies | 85850 | recommended growth media for WTC hiPSC line |
| Penicillin-streptomycin | Gibco | 15070-063 | |
| WTC hiPSC line | Coriell | GM25256 | the hiPSC line used in this protocol is available through Coriell |
| Tissue culture dish 100 mm | Falcon | 353003 | |
| 6-well Cell Culture Plate | CELLSTAR | 657160 | |
| StemPro Accutase | Gibco | A11105-01 | |
| Dulbecco's Phosphate Buffered Saline (DPBS) no calcium, no magnesium | Gibco | 14190144 | |
| 15 mL polystyrene conical | Sarstedt | 62.554.100 | |
| Y-27632 (ROCK Inhibitor) | StemCell Technologies | 72308 | |
| Edit-R CRISPR-Cas9 Synthetic crRNA, unmodified (custom sequence) | Dharmacon | Custom0247 | |
| Edit-R CRISPR-Cas9 Synthetic tracrRNA | Dharmacon | U-002005-05 | |
| Recombinant wild-type Streptococcus pyogenes Cas9-NLS purified protein, 40 µM | University of California-Berkeley QB3 Macrolab | ||
| Custom donor plasmid (PriorityGENE) | Genewiz | donor insert design was synthesized and cloned into pUC57 backbone by Genewiz | |
| DNA LoBind Tube 1.5 mL | Eppendorf | 22431021 | |
| NucleoBond Xtra Maxi EF | Clontech | 740424.50 | |
| Neon Transfection System | ThermoFisher Scientific | MPK5000 | |
| Neon Transfection System, 100 µL kit | ThermoFisher Scientific | MPK10096 | |
| 5 mL Polystyrene Round-bottom Tube with Cell Strainer Cap | Falcon | 352235 | |
| 15 mL High Clarity Polyproylene Conical Tube | Falcon | 352196 | |
| FACSAriaIII Fusion | BD Biosciences | 656700 | |
| FACSDiva software | BD Biosciences | ||
| FlowJo version 10.2 | TreeStar | ||
| NERL Blood Bank Saline | ThermoFisher Scientific | 8504 | used as preservative-free FACS Buffer |
| Olympus SZX7 Stereo Microscope, or similar | Olympus, or similar | ||
| Tissue culture plate, 96-well | Falcon | 353072 | |
| 96 well Cell Culture Plate, V-Bottom | CELLSTAR | 351180 | |
| CryoStor CS10 | Sigma | C2874-100ML | used as cryopreservation buffer for cells in 96-well plate format |
| Parafilm | Bemis | PM-996 | |
| 24 well Cell Culture Plate | CELLSTAR | 662160 |
Références
- Wood, A. J., et al. Targeted genome editing across species using ZFNs and TALENs. Science. 333 (6040), 307 (2011).
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Cong, L., et al. Multiplex genome engineering using CRISPR/Cas systems. Science. 339 (6121), 819-823 (2013).
- Mali, P., et al. RNA-guided human genome engineering via Cas9. Science. 339 (6121), 823-826 (2013).
- Dambournet, D., Hong, S. H., Grassart, A., Drubin, D. G. Tagging endogenous loci for live-cell fluorescence imaging and molecule counting using ZFNs, TALENs, and Cas9. Methods in Enzymology. , 139-160 (2014).
- Ratz, M., Testa, I., Hell, S. W., Jakobs, S. CRISPR/Cas9-mediated endogenous protein tagging for RESOLFT super-resolution microscopy of living human cells. Sci Rep. 5, 9592 (2015).
- Hendriks, W. T., Warren, C. R., Cowan, C. A. Genome Editing in Human Pluripotent Stem Cells: Approaches, Pitfalls, and Solutions. Cell Stem Cell. 18 (1), 53-65 (2016).
- Hockemeyer, D., Jaenisch, R. Induced Pluripotent Stem Cells Meet Genome Editing. Cell Stem Cell. 18 (5), 573-586 (2016).
- Doyon, J. B., et al. Rapid and efficient clathrin-mediated endocytosis revealed in genome-edited mammalian cells. Nature Cell Biology. 13 (3), 331-337 (2011).
- Cho, W. K., et al. Super-resolution imaging of fluorescently labeled, endogenous RNA Polymerase II in living cells with CRISPR/Cas9-mediated gene editing. Sci Rep. 6, 35949 (2016).
- White, C. W., Vanyai, H. K., See, H. B., Johnstone, E. K. M., Pfleger, K. D. G. Using nanoBRET and CRISPR/Cas9 to monitor proximity to a genome-edited protein in real-time. Sci Rep. 7 (1), 3187 (2017).
- Roberts, B., et al. Systematic gene tagging using CRISPR/Cas9 in human stem cells to illuminate cell organization. Mol Biol Cell. 28 (21), 2854-2874 (2017).
- Soldner, F., et al. Generation of isogenic pluripotent stem cells differing exclusively at two early onset Parkinson point mutations. Cell. 146 (2), 318-331 (2011).
- Young, J. E., Goldstein, L. S. Alzheimer's disease in a dish: promises and challenges of human stem cell models. Human Molecular Genetics. 21 (R1), R82-R89 (2012).
- Soares, F. A., Sheldon, M., Rao, M., Mummery, C., Vallier, L. International coordination of large-scale human induced pluripotent stem cell initiatives: Wellcome Trust and ISSCR workshops white paper. Stem Cell Reports. 3 (6), 931-939 (2014).
- . . Gene, NCBI- National Center for Biotechnology Information. , (2017).
- . . Gene, NCBI- National Center for Biotechnology Information. , (2017).
- . . Allen Cell Methods: Single cell passaging human iPS cells. , (2018).
- . . Allen Cell Explorer. , (2017).
- Lingeman, E., Jeans, C., Corn, J. E. Production of Purified CasRNPs for Efficacious Genome Editing. Curr Protoc Mol Biol. 120, 31-31 (2017).
- . . Michael Davidson Fluorescent Protein Collection. , (2017).
- Cox, D. B., Platt, R. J., Zhang, F. Therapeutic genome editing: prospects and challenges. Nature Medicine. 21 (2), 121-131 (2015).
- Haas, S. A., Dettmer, V., Cathomen, T. Therapeutic genome editing with engineered nucleases. Hamostaseologie. 37 (1), 45-52 (2017).
- Beumer, K. J., Trautman, J. K., Mukherjee, K., Carroll, D. Donor DNA Utilization During Gene Targeting with Zinc-Finger Nucleases. G3 (Bethesda). 3 (4), 657-664 (2013).
- Orlando, S. J., et al. Zinc-finger nuclease-driven targeted integration into mammalian genomes using donors with limited chromosomal homology. Nucleic Acids Research. 38 (15), e152 (2010).
- DeWitt, M. A., Corn, J. E., Carroll, D. Genome editing via delivery of Cas9 ribonucleoprotein. Methods. 121-122, 9-15 (2017).
- Kim, S., Kim, D., Cho, S. W., Kim, J., Kim, J. S. Highly efficient RNA-guided genome editing in human cells via delivery of purified Cas9 ribonucleoproteins. Genome Research. 24 (6), 1012-1019 (2014).
- Lin, S., Staahl, B. T., Alla, R. K., Doudna, J. A. Enhanced homology-directed human genome engineering by controlled timing of CRISPR/Cas9 delivery. Elife. 3, e04766 (2014).
- Labun, K., Montague, T. G., Gagnon, J. A., Thyme, S. B., Valen, E. CHOPCHOP v2: a web tool for the next generation of CRISPR genome engineering. Nucleic Acids Research. 44 (W1), W272-W276 (2016).
- Montague, T. G., Cruz, J. M., Gagnon, J. A., Church, G. M., Valen, E. CHOPCHOP: a CRISPR/Cas9 and TALEN web tool for genome editing. Nucleic Acids Research. 42 (Web Server issue), W401-W407 (2014).
- Bae, S., Park, J., Kim, J. S. Cas-OFFinder: a fast and versatile algorithm that searches for potential off-target sites of Cas9 RNA-guided endonucleases. Bioinformatics. 30 (10), 1473-1475 (2014).
- . . CRISPOR V4.3. , (2017).
- Zhang, Y., et al. Comparison of non-canonical PAMs for CRISPR/Cas9-mediated DNA cleavage in human cells. Sci Rep. 4, 5405 (2014).
- Chen, X., Zaro, J. L., Shen, W. C. Fusion protein linkers: property, design and functionality. Adv Drug Deliv Rev. 65 (10), 1357-1369 (2013).
- Leonetti, M. D., Sekine, S., Kamiyama, D., Weissman, J. S., Huang, B. A scalable strategy for high-throughput GFP tagging of endogenous human proteins. Proc Natl Acad Sci U S A. 113 (25), E3501-E3508 (2016).
- Liang, X., et al. Rapid and highly efficient mammalian cell engineering via Cas9 protein transfection. Journal of Biotechnology. 208, 44-53 (2015).
- Baker, D., et al. Detecting Genetic Mosaicism in Cultures of Human Pluripotent Stem Cells. Stem Cell Reports. 7 (5), 998-1012 (2016).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon