Method Article
Всеобъемлющий рабочий процесс для идентификации генома и выражение мета анализа семьи ATL E3 убиквитин лигаза гена в Грейпвайне
* Эти авторы внесли равный вклад
В этой статье
Резюме
Эта статья описывает процедуру идентификации и характеристика гена семьи в виноградной лозы, применяется к семье ligases убиквитин E3 Arabidopsis Tóxicos в Levadura (библиотека ATL).
Аннотация
Классификация и номенклатура генов в семье может значительно способствовать к описанию разнообразия закодированные белков и предсказание семейных функций на основе нескольких функций, таких как присутствие последовательности мотивы или особое сайты для столб-поступательные изменения и выражение профиль членов семьи в различных условиях. Эта работа описывает подробный протокол для семьи характеристика генов. Здесь эта процедура применяется к характеристике Arabidopsis Tóxicos в Levadura (ATL) E3 убиквитин лигаза семьи в виноградной лозы. Методы включают генома идентификации членов семьи, характеристика гена локализации, структуры и дублирования, анализа сохраненных белков мотивы, предсказание сайтов локализации и фосфорилирование белков, а также ген выражение профилирование по всей семьи в разных наборов данных. Такая процедура, которая может быть расширен для дальнейшего анализа в зависимости от экспериментальных целей, могут применяться к любой семье гена в любых видов растений, для которого имеются геномных данных, и предоставляет ценную информацию для выявления интересных кандидатов для функциональных исследований давая понимание молекулярных механизмов адаптации растений к их окружающей среде.
Введение
В течение последнего десятилетия много исследований было проведено в Грейпвайн геномики. Грейпвайн – является признанным экономически значимых культур, который стал моделью для исследований на развитие плода и на реакции древесных растений к биотическим и абиотическим стрессовым. В этом контексте освобождение геном Vitis vinifera cv. PN40024 в 2007 году1 и его обновленную версию в 20112 привели к быстрому накоплению «омику»-масштаба данных и всплеск высок объём исследований. На основе опубликованных последовательности данных, всесторонний анализ данного гена семьи (обычно состоит из белков, обмена сохраняется мотивы, структурные или функциональные сходства и эволюционных отношений), теперь могут быть выполнены раскрыть его молекулярные функции, эволюция и профили выражения гена. Эти анализы могут способствовать пониманию как ген семей контролировать физиологические процессы в масштабах всего генома.
Многие аспекты жизненного цикла растений регулируются убиквитин опосредованного деградации ключевых белков, которые требуют оборачиваемостью доработаны для обеспечения регулярного клеточных процессов. Важные компоненты процесса деградации убиквитин опосредованного ligases убиквитин E3, которые отвечают за гибкость системы, благодаря набору конкретные цели3. Соответственно эти ферменты представляют собой семейство огромный гена, с около 1400 E3 лигаза кодирование генов предсказал в проростках Arabidopsis thaliana генома4, каждая E3 убиквитин лигаза, действуя для ubiquitination конкретных целевых белков. Несмотря на важность конкретного субстрата ubiquitination в клеточной регуляции в растениях мало что известно о как регулируется ubiquitination путь и белков-мишеней были выявлены только в нескольких случаях. Расшифровка такой специфики и регулирование механизмов зависит сначала идентификации и характеризации различных компонентов системы, в частности E3 ligases. Среди ligases убиквитин ATL подсемейство характеризуется 91 членов, определенных в A. thaliana показано кольцо-H2 палец домена5,6, некоторые из них играет свою роль в обороне и гормон ответы7.
Первым важным шагом для определения членов нового гена семьи является точное определение семейных функций, таких как консенсус мотивы, ключевые домены и характеристики последовательности белка. Действительно надежный поиск всех членов семьи гена, на основе анализа взрыва требует некоторые обязательные последовательности характеристики, в частности белка доменах ответственным за белка функция/деятельность, выступающей в качестве подписи белка. Это может быть облегчено предыдущих характеристика той же семьи гена в других видов растений или достигнуто путем анализа различных генов, предположительно принадлежащих к той же семье в различных видов растений, чтобы изолировать общих последовательностей. Члены семьи могут затем быть индивидуально названы следующие общие правила, заселен международных консорциумов для данного растения. В виноградной лозе к примеру, такая процедура подвергается рекомендации Комитета супер-номенклатура для винограда гена аннотации (sNCGGa), создание строительство филогенетическое дерево, включая V. vinifera и A. thaliana членов семьи гена разрешить гена аннотации на основе нуклеотидной последовательности8.
Хромосома локализация генов дублирования обследования и членов семьи позволяют, подчеркнув наличие всего генома или тандем дублируется генов. Такая информация представляется полезным разгадать предполагаемого гена функции, поскольку он может показать функциональные избыточности или выявить различные ситуации, то есть, не функционализации, нео функционализации или суб функционализации9. Обе нео - и суб - functionalization являются важными событиями, которые создают генетические новизны, предоставления новых клеточных компонентов для адаптации растений к меняющимся среды10. В частности дублирования предков генов и производство новых генов были очень часто в ходе эволюции генома виноградной лозы и недавно сформированной генов, возникая от проксимальных и тандем дублирования в grapevine чаще производить новые 11функции.
Еще одним ключевым фактором в расшифровке семьи функции гена-транскриптомики профиль. Наличие общедоступных баз данных, предоставляя доступ к огромное количество транскриптомики данных могут быть использованы таким образом назначить предполагаемые функции гена членов семьи с использованием крупномасштабных в silico анализа выражения. Действительно своеобразное выражение некоторых генов в органов конкретных растений или в ответ на определенные подчеркивает может дать некоторые подсказки относительно предполагаемых ролей соответствующих белков в определенных условиях и оказывать поддержку гипотезы о возможных суб функционализация дублируется генов реагировать на различные вызовы. Для этой цели, важно рассмотреть несколько наборов данных: они могут быть уже имеющихся ген выражение матриц, таких как Атлас генома всей транскриптомики органов виноградной лозы и этапы развития12, или может быть построен ad hoc по получение транскриптомики наборов данных для конкретных растений, подвергается определенным напряжений. Кроме того простой подход с использованием двух матриц, один с попарных сходство данных и один с коэффициентами попарных Сопредседатель выражение может применяться для оценки взаимосвязи между последовательности подобия и выражения шаблоны в семейство генов.
Цель этой работы-обеспечить глобальный подход, определяя структуру гена, сохраненных белков мотивы, хромосомных местоположение, Джин дублирования и шаблоны выражений, как также предсказание белка локализации и фосфорилирование сайтов, для достижения исчерпывающую характеристику семейство генов растений. Такой комплексный подход применяется здесь к характеристике ATL E3 убиквитин лигаза семьи в виноградной лозы. Согласно новой роли членов подсемейство ATL в регулировании основных клеточных процессов7, эта работа может также содействовать выявлению сильных кандидатов для функциональных исследований и в конечном итоге разгадать молекулярные механизмы, регулирующие адаптация этой важной культурой для своей окружающей среды.
протокол
1. Определение предполагаемого ATL семейство генов участника(ов)
- PSI-ДОМЕННАЯ веб-версия
- Откройте веб-страницу взрыва13 и нажмите на раздел белка взрыва.
- В поле «Ввод запроса последовательности» введите последовательность аминокислот белка (здесь VIT_05s0077g01970), который будет использоваться в качестве зонда для определения других членов семьи.
Примечание: Хороший представитель белков должны быть использованы (белок, отображение всех важных особенностей, которые характеризуют семьи). - В поле «Выбрать Поиск набора» выберите базу данных «Ссылка белка» (refseq_protein) и организм интерес (V. vinifera - taxid:29760).
- В поле «Выбор программы» выберите алгоритм PSI-взрыв и нажмите кнопку «взрыв» для запуска анализа.
Примечание: Нажав на «параметры алгоритма» можно настроить некоторые дополнительные параметры (Макс последовательности, матрица скоринга, PSI-ДОМЕННАЯ порог и т.д.). - Первый раунд взрыв извлекает все последовательности, отображение соответствующих матчи с запросом (e значение выше выбранного порога - по умолчанию 0,005; 0,001 в этом эксперименте). Отменить выбор всех записей, которые явно не принадлежат к семейству рассмотрении нажатием на галочку в столбце «выберите для PSI-BLAST» и запустить второй итерации PSI-взрыв, нажав кнопку взрыва как шаг 1.1.4.
- Недавно выявленные последовательности будут выделены желтым цветом. Отменить выбор явно неправильно проверено хитов и раскрыть дополнительные итерации, как описано в шаге 1.1.5.
- Продолжите итераций, пока алгоритм не найти каких-либо соответствующих запись или достигнет конвергенции (найдено никаких новых записей). Скачайте список предполагаемых генов членов семьи для дальнейшего анализа. Осмотрите проверено хитов в каждой итерации, чтобы избежать наличия ложных срабатываний.
- PSI-ДОМЕННАЯ автономная версия
- Скачайте отдельную версию взрыва, нажав кнопку «Загрузить взрыв» на домашней странице взрыва13.
Примечание: Взрыв автономное программное обеспечение является командной строки версии Web-интерфейса, описано ранее. Он позволяет выполнение поиска PSI-взрыва против пользовательской локальной или удаленной базе данных. Кроме того она позволяет осуществлять поиск с предварительно определенные положения конкретных Оценка матрица (запасами).
- Скачайте отдельную версию взрыва, нажав кнопку «Загрузить взрыв» на домашней странице взрыва13.
2. ручной проверки PSI-взрыв определенных членов семьи
- Несколько выравнивания
- Собирать амино кислой последовательностей, ранее определенных в файле FASTA-формате и загрузить его в программного обеспечения мега14 приступить несколько выравнивания.
- Откройте программное обеспечение MEGA, нажмите кнопку «Выровнять», нажмите «Edit/построить выравнивание», нажмите кнопку «Создать новый выравнивание», нажмите «Белка».
- Нажмите кнопку «Редактировать» от меню выравнивание и «Вставка последовательности из файла». Перейдите к файлу FASTA, созданные до и подтвердите загрузку всех исследуемых последовательностей.
- Нажмите кнопку «Выравнивание» от меню выравнивание и «Выровнять по мышц». Использовать параметры по умолчанию, нажмите кнопку «Вычислить» и дождитесь завершения несколько выравнивания.
- Осмотрите несколько выравнивания исключить неверно предсказал членов семьи. Канонический CxxC (13 x) PxCxHxxHxxCxxxW (7 x) CxxCW мотив, (в частности присутствие остатков пролина перед третьим цистеин), является ключевой особенностью, необходимые для определения членов семьи ATL.
- Анализ конкретных логотип
- Представить окончательный список членов семьи (96 Грейпвайн последовательности выполнения требований будет считаться ATL) несколько Em мотив заключения (мем)15 для определения сохраняемой мотивы по всей семьи.
- На домашней странице MEME нажмите на кнопку «Мем» и завершить «Данных представления формы» с конкретной информацией относительно семьи интерес.
- Использование анализа MEME подтвердить наличие двух ожидаемых мотивов внутри членов семьи ATL виноградной лозы, т.е., кольцо-H2 и мотивы ООВ.
- Кроме того, выполните шаги 2.1 и 2.2, одновременно используя набор программного обеспечения биоинформатики (см. Таблицу материалы).
- Загрузить файл FASTA (см. шаг 2.1.1) в suite. Выберите «Файл» из меню, а затем «Импорт» и нажмите «из файла». FASTA файл и нажмите «Открыть».
- Выберите все импортированные последовательностей в списке и нажмите на кнопку «Выровнять/сборка» в панели инструментов, а затем нажмите кнопку «Pairwise несколько выравнивание». Выберите «Мышцы выравнивание» и нажмите кнопку «ОК», чтобы запустить выравнивание с помощью параметров по умолчанию.
- Чтобы визуализировать логотип выравнивание, нажмите на «Графики» → «параметры» и выберите «Последовательность логотип».
3. Анализ белков физических параметров и домены
- Как определение различных физических параметров опрошенных членов семьи важно иметь всеобъемлющее описание семьи, представить список членов семьи к конкретным веб-инструменты.
- Для Изоэлектрическая точка (pI) и молекулярного веса (кДа) используйте инструмент ProtParam16 на веб-сайте Expasy с параметрами по умолчанию.
- Для локализации внутриклеточных белков используйте различные инструменты для получения более надежных предсказание например ngLOC v1.017 с параметрами по умолчанию, targetP v1.118 с параметрами по умолчанию и белка Мародер субцеллюлярные локализации v1.219 с сокращения вероятности 0.5. Фосфорилирование сайтов используйте MUsite v1.0 веб инструмент20 с параметрами по умолчанию.
- Исследуйте дополнительные белка доменов в членов семьи.
- Откройте веб-страницу базы данных Pfam21, выберите инструмент «Поиск последовательности», представить белковых последовательностей в поле запроса и нажмите кнопку «Go», чтобы запустить анализ.
Примечание: Каждая последовательность белка анализируется отдельно. E значение 1.0 в параметр по умолчанию позволяет проводить дискриминацию между значительным и незначительных хиты. - Открывайте TMHMM сервера22 из центра для анализа биологических последовательностей для расследования присутствия предполагаемого трансмембранного регионов.
- Откройте веб-страницу базы данных Pfam21, выберите инструмент «Поиск последовательности», представить белковых последовательностей в поле запроса и нажмите кнопку «Go», чтобы запустить анализ.
4. хромосомных распределение, дублирования и Организация ЭКЗОН Интрон
- Карта ATL членов семьи на хромосомы, основанные на данных, полученных из виноградной лозы генома CRIBI Biotech центр веб-сайт24.
- Просмотр главной страницы сайта PhenoGram25. Запись «входной файл» как табуляцией текстовый файл с особенностями генов для сопоставления на хромосомы, согласно исчерпывающие рекомендации и примеры относительно компиляции файл после путь «Phenogram» →» Документация» → «Опции» → «входной файл».
- Запись «Титул» работы. Выберите геном быть выписанным. Для геномов, не реализованы в программном обеспечении, например геноме виноградной лозы выберите «другое» в раскрывающемся меню. Запись файла генома согласно рекомендации и примеры, следуя по пути «Phenogram» → «Документация» → «Опции» → «Геном» и загрузить его.
- Использовать параметры по умолчанию «Фенотип интервалы», «Фенотипом цвета», «Формат изображения» или выберите альтернатив в соответствующих меню и нажмите кнопку «Печать» для получения визуализации генов в хромосомах за гены.
- Определите состояние дублирования с использованием программного обеспечения MCScanX26членов семьи.
- Скачайте и распакуйте копию MCscanX на локальном компьютере запуск командной строки 1 (дополнительный файл 1). Введите папку MCscanX и создать необходимые исполняемые файлы запуска командной строки 2 (дополнительный файл 1).
Примечание: Установка MCscanX известно неудачу на некоторых машинах Linux 64 bit вследствие относительно функция chdir. Если относящиеся к этой функции после make возвращается сообщение об ошибке выполнения команды, следует запускать в командной строки 3 (дополнительный файл 1) и команду «make», должно выполняться после этого. - Скачайте V. vinifera белков и аннотации файл запуска командной строки 4 (дополнительный файл 1).
Примечание: Файл аннотации Грейпвайн нужно быть unzipped и одной хромосомы информации Кот в уникальный файл, запустив командные строки 5 (дополнительный файл 1). - Запуск «все против всех» blastp Поиск с использованием файла V. vinifera белка как запрос, так и предметом.
- Создайте базу данных для поиска взрыв с использованием V. vinifera белка файл запуска командной строки 6 (дополнительный файл 1). Выполните поиск blastp с помощью файла белки V. vinifera как запрос к базе данных, созданный ранее, запустив командные строки 7 (дополнительный файл 1).
- Преобразуйте файл аннотации в подходящий формат для MCScanX. Запуск командной строки 8 (дополнительный файл 1) для загрузки пользовательских perl скрипт parseMSCanXgff.pl. Выполните анализ выполнения командной строки 9 (дополнительный файл 1).
Примечание: Файл vitis.gff создается содержащий координаты гена в следующем формате:
SP # гена, начальная позиция ending положение
где «СП» — двухбуквенный код для видов (Vv для виноградной лозы), тогда как «#» — это имя леса. Обратите внимание, что предоставленный пользовательский perl-скрипт подходит для большинства преобразования, хотя в некоторых конкретных случаях вследствие многообразия информации, представленной в файле доступны аннотации могут потребоваться некоторые изменения кода. - Запуск MCScanX запуск командной строки 10 (дополнительный файл 1).
Примечание: «Витис» является префиксом аннотацию и взрыва выходного файла. Это является обязательным требованием для программного обеспечения для запуска. - Анализ результатов MCScanX. MCScanX производит один текстовый файл «vitis.collinearity», который содержит коллинеарных блоков. Такой файл может быть проверен любым текстовым редактором (см. Пример вывода 1 дополнительный файл 1).
Примечание: В «mcscaxOutput.html» каталог создается, содержащий html-файлы, показывая несколько рядов блоков коллинеарных против каждой эталонной хромосомы. Эти файлы могут быть проверены через веб-браузер. - Классифицируйте paralogous генов, основанный на их относительные позиции в хромосомах запуск командной строки 11 (дополнительный файл 1).
Примечание: Paralogous гена классификации описан в Дополнительной таблице II. Сгенерированный выходной файл «vitis.gene_type» содержит все сведения происхождения с форматом простой с разделителями табуляции. - Выполните анализ обогащения для оценки ли семейство генов бытовой возникла на конкретный механизм запуска командной строки 12 (дополнительный файл 1).
Примечание: Файл «vitis.gene_type» создается на этапе 4.2.8, тогда как файл «gene_family_file» представляет собой одну строку текстового файла, в котором имя семейства (например, ATL_genes) следуют Локус имена для всех генов, принадлежащих к семейству разделенные вкладки. Прикладной статистический тест для обогащения является точный тест Фишера и p-значения различного происхождения, хранятся в файле «outputFile.txt».
- Скачайте и распакуйте копию MCscanX на локальном компьютере запуск командной строки 1 (дополнительный файл 1). Введите папку MCscanX и создать необходимые исполняемые файлы запуска командной строки 2 (дополнительный файл 1).
- Визуализируйте Экзон Интрон организации генов с помощью интерактивных Древо жизни (iTOL)27, он-лайн инструмент для отображения, аннотации и управления филогенетических деревьев.
- Загрузите филогенетическое дерево в разделе «Загрузки» на сайте iTOL. Дерево строится согласно статье 5 ниже. Для каждого члена семьи гена получить Предсказание структуры генов из V1 аннотации генома виноградной лозы (сайт CRIBI цитируется выше). Рассчитайте длину (в bp) предполагаемого экзонов, интронов и непереведенные регионов (необычных).
- Используйте набор данных, «Доменов протеина» для графической визуализации Экзон Интрон шаблона.
5. филогенетический анализ и номенклатура
- Проанализируйте отношения между членами семьи ATL путем строительства филогенетическое дерево высокого качества и определение семьи номенклатуры.
- Для семьи гена виноградной лозы следуйте правилам, установленным в Grapevine Super номенклатура Комитета8.
- Извлечение последовательности A. thaliana ATL, как ссылку для виноградной лозы гена номенклатуры8, от базы данных UniProt28 .
- Напишите файл FASTA, включая все нуклеотидные последовательности виноградной лозы и A. thaliana гена членов семьи должны быть включены в филогенетический анализ. Нуклеотидные последовательности позволяют максимум изменчивости среди членов семьи (по сравнению с белковых последовательностей).
- Филогенетическое дерево
Примечание: Использование конвейера 29 Phylogeny.fr рекомендуется получить филогенетическое дерево высокого качества, но не является обязательным.- Просмотр главной страницы Phylogeny.fr29и выберите трубопровода «Филогения анализ».
Примечание: «Один клик» подходит в большинстве случаев, но при необходимости его можно выбрать конкретные дополнительные параметры («дополнительно») или даже полностью настроить анализ («а La Carte»; см. шаг 5.2.5). - Записи «Имя анализа», загрузить файл FASTA, созданный ранее (шаг 5.2.1 и нажмите кнопку «Отправить», чтобы запустить анализ.
- Кроме того если процедура описана выше (шаги 5.2.1, 5.2.2) результаты в сообщение об ошибке, завершайте каждый шаг трубопровода филогении люкс индивидуально, следующим образом.
- МЫШЦЫ программного обеспечения домашней странице30загрузить файл FASTA в «Шаг 1», выберите «Пирсон/FASTA» как «Формат вывода» в «Шаг 2» и нажмите кнопку «Отправить» в «Шаг 3» для выравнивания последовательности запроса.
- Нажмите кнопку «Загрузить файл выравнивание» и сохраните как файл FASTA для дальнейших шагов.
- Процесс выравнивания FASTA файл, чтобы устранить плохо согласованы позиции, с помощью сервера Gblocks инструмент31. Загрузить файл FASTA выравнивание, выберите «ДНК» как «Тип последовательности» и выбрал вариант(ы) жесткости, который лучше всего подходит с анализа (например, для виноградной лозы ATL гена семьи выберите все три варианта предложил «менее строгого отбора», потому что высокая последовательность расхождения). Нажмите «Получить блоки» чтобы запустить анализ.
- Нажмите кнопку «Результирующая выравнивание» в нижней части страницы выходных данных и сохранить результаты в новом файле FASTA.
- Главная Phylogeny.fr29выберите «A la Carte» как «Филогения анализ» трубопровода. Затем, снимите флажок «Несколько выравнивание» и «Выравнивание курирование». Нажмите кнопку «Создать рабочий процесс», загрузите файл Gblocks куратор FASTA (шаг 5.2.5.4), выберите «Bootstrapping процедура» с параметрами по умолчанию в «Настройки» и нажмите «Отправить», чтобы запустить анализ.
- Крах плохо поддерживает ветви (например, загрузчик значения < 70%), нажав кнопку «Сворачивать ветви» в разделе «Выбор и действия» и скачать окончательные результаты в формате Невик для дальнейшего анализа.
- Просмотр главной страницы Phylogeny.fr29и выберите трубопровода «Филогения анализ».
- Назначьте имя гена, основанный на филогении.
- Обзор филогенетическое дерево, чтобы оценить надежность структуры дерева, загрузив его в iTOL люкс, упомянутых выше (раздел 4.3).
- Назначьте вручную ген имя каждого члена семьи. В случае индивидуальных orthologues, назначить Arabidopsis-как имя (например, AtATL3 → VviATL3). Дифференцировать Грейпвайн генов (два или более) вытекающие из одного Arabidopsis гомолога с же филогенетических расстояние, используя цифры или буквы если геном арабидопсиса заканчивается с числом (например, AtATL23 → VviATL23a, VviATL23b).
- В случае orthologues один ко многим или многие ко многим, назначить новое имя ген состоит из Arabidopsis-как имя (здесь, «Атл») в паре с номером выше, чем наибольшее число уже используется для V. vinifera и арабидопсиса (например., VviATL83).
- Полное номенклатуру недавно определенные семьи, спуская от верхней к нижней части филогенетическое дерево.
6. Грейпвайн органа и стадии выражение профилирования
- Создание рабочих данных матрицы содержащие выражения данных для членов семьи.
- Скачайте по ссылке, распределенных на платформе ResearchGate32 V. vinifera cv. Корвина ген выражение Атлас datamatrix. Этот файл содержит значения выражения RMA нормированный использоваться в следующие шаги.
- Извлечь значения выражения для каждой семьи генов из атлас datamatrix и написать «Рабочая datamatrix», содержащий же строка заголовка как datamatrix Атлас. Сохраните «Рабочая datamatrix» как табуляцией текстового файла.
- Выполните иерархических Би кластерный анализ, используя Multi эксперимент Viewer (МэВ) программного обеспечения.
- Скачать и установить программное обеспечение МЭВ33.
- Загрузить «Рабочая datamatrix» (шаг 6.1.2) по пути «Файл» → «Загрузка данных» → «Обзор» и выберите текстовый файл. Выберите «Single цвет массив» и снять галочку «Load аннотации» когда автоматической аннотации не предоставляется. Выберите значение верхнего левого выражения предварительного просмотра таблицы выражение и нажмите кнопку «Загрузить».
- Корректировка данных, применения преобразований Log2 («Корректировать данные» → «Журнала преобразований» → «Log2 преобразования») и гена/строки нормализации («Корректировать данные» → «Джин/ряд корректировок» → «медиана центр гена/строки»). Установите предел надлежащего масштаба («Отобразить» → «задать цвет границы шкалы»).
- Вычислить иерархическая кластеризация по пути «Анализ» → «Группирование» → «HCL».
7. выражение профилирование в ответ к биотическим и абиотическим стрессовым
- Повторите шаг 6.1 с идентификатором присоединения GSE получены из соответствующих публикаций и исследований, исследование биотического и абиотического стресса на виноградной лозы. Например эксперименты, обеспечивая транскриптом профиль виноградной ягоды инфицированных грибкового патогена Botrytis cinerea с помощью microarray винограда NimbleGen целом генома могут просматриваться с ГСИ ID GSE52586. Повторите шаги 6.1.1 и 6.1.2.
- Поиск Архив считывает последовательность NCBI34 с SRA/BioProject ID (например, SRP055458 или PRJNA275778 для «виноградной лозы цветка затенение» экспериментов) и скачать все связанные считывает необработанные последовательности. РНК seq наборов данных из многих различных исследований, обрабатываются с помощью единого конвейера для обеспечения согласованности.
- Вкратце обрезать сырой последовательности FASTQ чтений (одно - и пара конец) и фильтр качества с Trimmomatic35. Использование AVGQUAL и MINLEN фильтр 20 и 40, соответственно и все параметры по умолчанию.
- Индекс 12 X Грейпвайн ссылка генома1 с помощью Bowtie236. Скачайте 12 X Грейпвайн ссылка генома (например, bowtie2-строить) перед запуском команды bowtie2 .
- Получите количество матрица таблиц с htseq граф37 с помощью файла аннотации (GFF/GTF) модель гена V1 виноградной лозы.
- Выполните анализ дифференциального ген выражение (ре-) в R38 с40 Лимма39 библиотеки для RMA-нормированный матрицы и DESeq2 для игр матрица таблиц, полученных из шагов 7.1.1 и 7.2.1, соответственно.
- Выполнить стандартное сравнение «два групп» (то есть, «лечение» / «контроль»). Убедитесь, что дизайн матрица/группировки «управляет» и «лечение» условий указаны правильно.
Примечание: Типичный дизайн для microarray анализ дифференциального выражения (GSE52586) для сравнения Эль-33 ягоды, инфицированных с Botrytis cinerea против управления (здоровых) ягоды на той же стадии развития Лимма запуск командной строки 13 показано в дополнительных файлов 1. Типичный дизайн для анализа дифференциальной выражение РНК seq (SRP055458 или PRJNA275778), для сравнения цветов (на 7 дней после Кап осень) под тени лечения против управления с DESeq2 запуск командной строки 14 показан на дополнительных файлов 1 . - Получение списков дифференциально выраженной генов (град) в каждом контраст, для Лимма, использовать функции lmFit(), а затем eBayes()и затем topTable() функции, в то время как для DESeq2, используйте DESeqDataSetFromMatrix(), DESeq()и results() функции. Ниже, типичный рабочий процесс для подражания.
- Microarray анализ дифференциального выражения командные строки 15 (дополнительный файл 1) см. РНК seq анализ дифференциального выражения см командные строки 16 (дополнительный файл 1). Повторите эти шаги для всех других контрастирует с другой соответствующей конструкции схемы (см. примеры на шаге 7.3.1)
- Выполнить стандартное сравнение «два групп» (то есть, «лечение» / «контроль»). Убедитесь, что дизайн матрица/группировки «управляет» и «лечение» условий указаны правильно.
- Из списков DEGs генерируется, извлекать все строки, которые не соответствуют ATL V1 присоединения, сохранить столбцы, содержащие log2 сбросить изменения (лечение/управления) > | 0,5 | и скорректированы p-значения (ФДР) < 0,05 и объединить их соответственно в матричную таблицу, ли исследование попадает в «абиотические» или «биотические/возбудителя взаимодействия» конспекты.
- Постройте иерархический кластерный карты (абиотических и биотических конспекты) в R с использованием библиотеки gplots.
Примечание: Вызов функции heatmap.2 создает heatmap наряду с dendrograms строки из таблиц, соответствующих матрицы. Дополнительные аргументы, с помощью cellnote функции помогает различать дифференциально выраженной (log2FC > 0.5, Рузвельт < 0,05) ATL генов в каждой сравнение через широкий спектр экспериментальных условий * символ. Применить типичный рабочий процесс в R запуск командной строки 17 (дополнительный файл 1), или альтернативно, повторите шаги 6.2.2 для 6.2.5 для построения диаграммы, с помощью программного обеспечения МэВ.
8. анализ отношений между Paralogous последовательность расхождения и Сопредседатель экспрессии генов
- Создание матрицы, содержащие парные сходство. Элементы матрицы подобия значений последовательность сходства рассчитывается из рядов попарных белка.
- Используйте EMBOSS иглы веб сервера41 с параметрами по умолчанию выравниваний последовательности попарных и сохранить как текстовый файл. Открыть выходной текстовый файл и удалите все строки комментариев, а также имена столбцов и строк для создания файла под названием «similarityTable.txt».
Примечание: Такая таблица имеет линию для каждого гена ATL отчетности подобия значения, вычисленные в каждом из попарных выравнивание. Локусов в строках и столбцах заказ же так, что симметричная матрица создается с уважением диагональных значений.
- Используйте EMBOSS иглы веб сервера41 с параметрами по умолчанию выравниваний последовательности попарных и сохранить как текстовый файл. Открыть выходной текстовый файл и удалите все строки комментариев, а также имена столбцов и строк для создания файла под названием «similarityTable.txt».
- Постройте матрицу с данными совместного выражения путем расчета коэффициента корреляции Пирсона. Следующая процедура требует R и perl-модуль PDL.
- Скачайте выражение значения для 96 ATL гены работает командные строки 18 (дополнительный файл 1) в пределах терминала. Выполните анализ совместного выражения с помощью пользовательских perl-скрипт, который может быть загружен, запустив командные строки 19 (дополнительный файл 1). Такой сценарий будет вычислить коэффициент корреляции Пирсона между парами ATL локусов как сообщалось ранее.
- Запуск скрипта командной строки 20 (дополнительный файл 1) и следуйте инструкциям вывода.
- Запустите тест Mantel, загрузки данных двух матриц и выполняя статистики запуска командной строки 21 (дополнительный файл 1), с «nrep», представляющий количество перестановок. Тест состоит из расчета корреляции между элементами этих матриц, permuting матриц и затем снова расчета же тестовая статистика.
Примечание: Все полученные значения статистика теста используются для построения ссылка распределения статистика теста, который будет использоваться для вычисления p-значение для проверки на значение. Количество перестановок определяет точность, с которой p-значение может быть получено.
Результаты
VIT_05s0077g01970 гена, определены как наиболее близок к A. thaliana ATL2 (At3g16720) через поиск BLASTp, был использован как зонд для обследования членов семьи ATL в геноме виноградной лозы (V. vinifera cv Pinot Noir PN40024). PSI-BLAST анализ сходились после нескольких циклов, раскрывая список предполагаемых генов, принадлежащих к семье гена ATL виноградной лозы (Рисунок 1A). Присутствие каноническим доменом кольцо-H2 для каждого кандидата оценивалась путем визуального осмотра мышц выравнивание всех элементов, выявленных в ходе анализа (рис. 1B). Только эти гены, содержащие правильно расположенными сохранены аминокислоты, два гистидина остатков, а также остатков пролина до третьего хвоща рассматривались как атласом согласно исходное определение ATL в проростках Arabidopsis5. В общей сложности 96 Грейпвайн генов выполнила требования и были рассмотрены для дальнейшей характеризации. Каждый член семьи ATL были проанализированы для определения конкретных характеристик гена и соответствующие кодировке белка, т.е., присутствие других известных домена(ов) помимо кольцо-H2, трансмембранного или гидрофобные богатые регионы, субцеллюлярные Локализация и предполагаемый фосфорилирование сайты (Таблица 1 и Таблица 2).

Рисунок 1: PSI-ДОМЕННАЯ обследования и выравнивание предполагаемого Грейпвайн атласом. (A) скриншот 10 лучших хитов первого PSI-ДОМЕННАЯ итерации поиска с помощью последовательности белка VIT_05s0077g01970 как приманку. (B) часть выравнивание 96 выбранных виноградной лозы, предполагаемый атласом, показаны их кольцо-H2 домена и соответствующий логотип полученные с помощью набора молекулярной биологии (см. Таблицу материалы). Воспроизводится из "Ariani" et al. по лицензии Creative Commons Attribution 4.0 международного42лицензии.Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.
| Имя | Джин ID | Джин Длина (bp) | Интрон номер | UniProt ID | Длина белка (aa) | КОЛЬЦО-H2 мотив | Число доменов TM/Ч | Другие домены | ||
| VviATL3 | VIT_09s0002g00220 | 1245 | 0 | F6HXK6 | 304 | PxC | 1 | |||
| VviATL4 [VviRHX1A] | VIT_15s0021g00890 | 1827 | 3 | D7SM36 | 203 | PxC | 0 | |||
| VviATL18 | VIT_11s0118g00780 | 1113 | 2 | F6HCI8 | 193 | PC | 0 | |||
| VviATL23a | VIT_18s0001g01060 | 935 | 0 | F6H0E4 | 114 | PxC | 0.5 | |||
| VviATL23b | VIT_18s0001g01050 | 399 | 0 | E0CQX3 | 132 | PxC | 1 | |||
| VviATL24 | VIT_17s0000g06460 | 4466 | 4 | D7SI89 | 217 | PxC | 1 | |||
| VviATL27 | VIT_00s0264g00020 | 2554 | 4 | D7T1R5 | 235 | PxC | 1 | |||
| VviATL43 | VIT_11s0052g00530 | 1576 | 2 | D7SQD9 | 457 | PxC | 3 | |||
| VviATL54a | VIT_18s0001g06640 | 3221 | 1 | F6H0Y5 | 405 | PxC | 1 | |||
| VviATL54b | VIT_03s0017g00670 | 2774 | 1 | F6HTI0 | 427 | PxC | 1 | |||
| VviATL55 [VviRING1] | VIT_07s0191g00230 | 1844 | 0 | F6HRP9 | 372 | PxC | 1 | |||
| VviATL63 | VIT_06s0004g06930 | 804 | 0 | D7SJU6 | 267 | PxC | 1 | |||
| VviATL65 | VIT_03s0063g01890 | 2068 | 0 | F6HQI8 | 396 | PxC | 1 | |||
| VviATL82 | VIT_01s0026g02540 | 820 | 0 | F6HPQ9 | 233 | PC | 0.5 | |||
| VviATL83 | VIT_17s0000g08400 | 1887 | 0 | F6GSQ4 | 143 | PC | 0 | |||
| VviATL84 | VIT_06s0004g00120 | 1853 | 0 | F6GUP5 | 368 | PC | 0.5 | ZF-RING_3 | ||
| VviATL85 | VIT_12s0034g01400 | 786 | 0 | F6H965 | 261 | PC | 0.5 | |||
| VviATL86 | VIT_12s0034g01390 | 1434 | 1 | D7T016 | 451 | PC | 0.5 | |||
| VviATL87 | VIT_18s0001g03270 | 1002 | 0 | F6H0T2 | 333 | PC | 0.5 | ZF-RING_3 | ||
| VviATL88 | VIT_08s0040g00590 | 1320 | 0 | F6HQR2 | 314 | PC | 0 | ZF-RING_3 | ||
Таблица 1: первые 20 VviATL генов и последовательности характеристики соответствующих белков. ТМ: трансмембранный; H: гидрофобное; 0.5 указывает на наличие одного или более гидрофобных регионов. Воспроизводится из "Ariani" et al. по лицензии Creative Commons Attribution 4.0 международного42лицензии.

Таблица 2: данные о первых 20 VviATL Джин позиции в V. vinifera генома, дублирования государства и ATL белка физико химические характеристики и местоположение. () количество мест фосфорилирования предсказано Musite; (b) аналогичные прогнозы, полученные с по крайней мере две программы, выделены жирным шрифтом; ngLOC используется с параметрами по умолчанию, тогда как TargetP v1.1 и белка Prowler субцеллюлярные локализации были использованы с сокращения вероятности 0.5. КНУ, ядро; MIT, митохондрий; КХЛ, хлоропластов; ПЛА, плазматической мембраны; S, секреторную путь (наличие сигнала пептид); M, митохондрий; C, хлоропластов; O или -, других местах; ой, не определено (то есть, значение ниже порога). Воспроизводится из "Ariani" et al. по лицензии Creative Commons Attribution 4.0 международного42лицензии. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Филогенетический анализ, включая нуклеотидные последовательности выявленных Грейпвайн ATL-кодирование генов совместно с последовательностями ссылки A. thaliana ATL гена семьи был использован для виноградной лозы ATL номенклатуры, согласно руководящим принципам sNCGGa8. Девяносто шесть и 83 нуклеотидные последовательности от V. vinifera и A. thaliana, соответственно, были подвергнуты в Phylogeny.fr конвейер для получения надежного филогенетическое дерево.Последний последовательности были впоследствии использованы для аннотирования и имя Грейпвайн генов на основе прочных связей (рис. 2). После этого подхода 13 из 96 Грейпвайн атласом получил определенный идентификатор, учитывая их индивидуальные Гомология с A. thaliana библиотеки ATL. Имена других генов, 83 были назначены, на основе филогенетическое дерево, с последовательной нумерации сверху вниз, начиная с номером гена ATL, выше, чем высокий число используемых в A. thaliana.
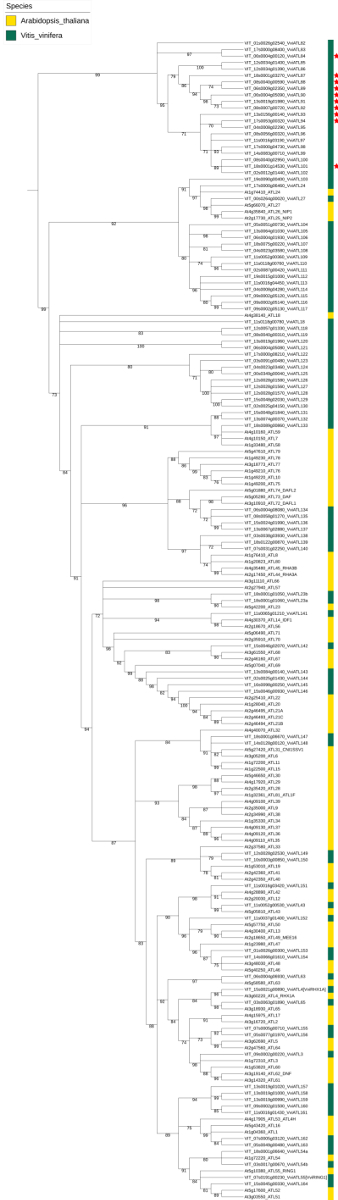
Рисунок 2: Филогенетическое дерево V. vinifera A. thaliana ATL E3 убиквитин лигаза кодирование генов и. Некорневых дерево был сгенерирован с Phylogeny.fr люкс (V. vinifera (в зеленом) и 83 ATL гены A. thaliana сообщили в UniProt базе (желтый). Отделение поддержки значения были получены из 100 загрузчик реплицирует. Красные звезды указывают на наличие BCA2 цинка палец (БЗФ) домена в соответствующем белков. Воспроизводится из "Ariani" et al. по лицензии Creative Commons Attribution 4.0 международного42лицензии. Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.
Сопоставление ATL-кодирование генов хромосом Грейпвайн показал широкое распространение во всем генома, предлагая всего генома дублирования как основных эволюционных сил в расширении ATL семейство генов в виноградной лозы. Действительно 31 атласом были найдены в гомологичных хромосом регионах, потенциально возникая от сегментарный или целого генома дублирования мероприятий. Кроме того тот же анализ подчеркнул 13 tandemly дублированный генов, один проксимальный дублировать и 51 дисперсной дубликаты (рис. 3). Учитывая очень большое количество повторяющихся генов в семье ATL мы провели тест обогащения (точный тест Фишера) проверить льготные удержания дублируется генов в геноме фракционирования. С p-значение < 0,001, этот тест подтвердил гипотезу, что дублируется ATL гены были сохранены более чем случайным образом ожидается, предложив определенную роль для семьи гена ATL во время адаптации виноградной лозы и эволюции.
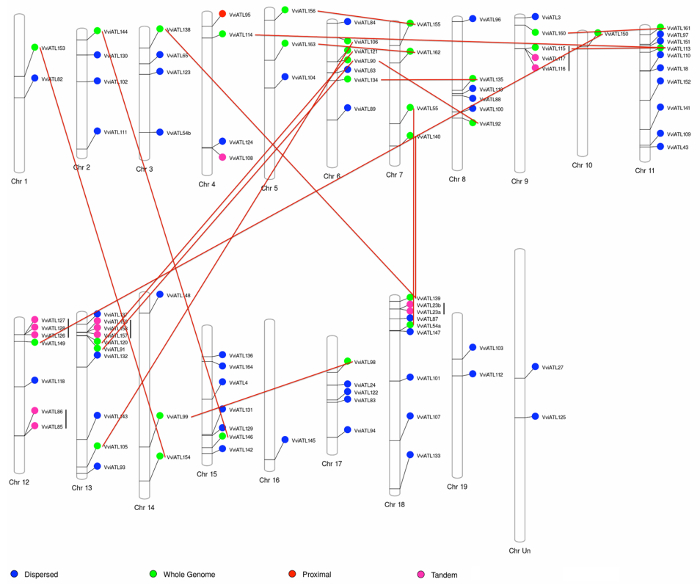
Рисунок 3: Грейпвайн ATL-кодирование гена распределение V. vinifera хромосом и дублирования государству. Гены ATL 96 виноградной лозы с точным хромосомных информации, имеющейся в базе данных были сопоставлены 19 V. vinifera хромосом. Цвета указывают исходное событие дублирования. Вертикальные черные и красные линии определения пар, производные от тандемных дупликаций и весь геном дублирования, соответственно. Воспроизводится из "Ariani" et al. по лицензии Creative Commons Attribution 4.0 международного42лицензии. Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.
Для дальнейшего расследования предполагаемого биологические функции атласом в виноградной лозы, мета анализ был проведен на V. vinifera cv. Корвина глобальной ген выражение Атлас12. Набор данных включает в себя всего генома выражение значения 54 виноградной лозы различных органов и этапы развития и был использован для выполнения иерархического Би кластерного анализа. Результаты не только подтвердил, что все 96 атласом были выражена в по крайней мере один из 54 тканей/этапов, но также отметил наличие пяти основных групп выражение профилей (рис. 4A). Вкратце кластеры A и E показал напротив поведения, в частности первый характеризуется общей Даунрегуляция ATL генов в несовершеннолетних образцы, в том числе ранних стадиях Берри, молодые листья, усики, соцветия и большинство этапов бутон. С другой стороны в том же кластере А зрелые образцы таких ягод в созревания и послеуборочной увядание этапов, древесных тканей и поздних стадиях развития семян ATL генов показали преобладающим upregulation. Гены в кластере C были главным образом downregulated в большинстве образцов, в то время как ATL генов в кластере D были часто upregulated на поздних стадиях развития Берри. Наконец кластер B не показали любые соответствующие изменения в профилях выражение.
Аналогичный подход был применен для изучения выражение Грейпвайн ATL членов семьи в ответ к биотическим и абиотическим стрессам, с использованием определенных наборов данных, построил для этой цели. Огромное количество данных выражение, вытекающих из microarray дна и RNA-seq эксперименты доступны общественности доступа к базам данных например Омнибус выражение гена (GEO) и ArrayExpress. После того, как собраны и удобно нормализуется, информация была использована для дальнейшего понимание потенциальной функции атласом в реакцию растений к стрессам. Анализируя выражение профили Грейпвайн атласом в ответ биотических стрессов показал, что 62 из 96 стенограмм показал значительный модуляции (log2 фолд изменения (FC) > | 0,5 |) в по крайней мере два условия, с частотой ложных обнаружения (ДРД) < 0,05 () Рисунок 4B). Число увеличивается до 81, учитывая только ФДР порог в одно условие. Эти результаты настоятельно предложил непосредственное участие семьи гена ATL в ответ на патогены также в виноградной лозы. В частности, Группа 12 генов (VviATL3-27-54b-55-90-97-123-144-148-149-156) были сильно upregulated в ответ на большинство патогенных микроорганизмов, включая biotrophic и necrotrophic грибов и травоядных животных и таким образом, заслуживают внимания для дальнейшей функциональной анализ.
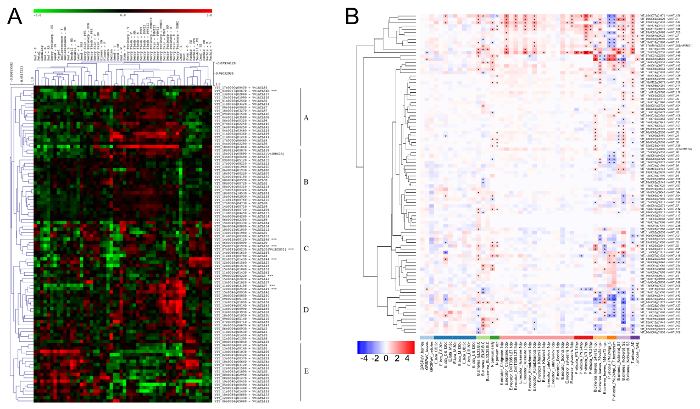
Рисунок 4: иерархических clusteringof ATL экспрессии генов в Грейпвайн Атлас и Грейпвайн биотических стрессовых dataset. (A) журнала превращается выражение значения генов ATL виноградной лозы в grapevine Atlas12 были использованы для иерархических кластерный анализ, основанный на Пирсона расстояние метрики. Цвет шкалы представляет выше (красный) или нижней (зеленый) выражение уровнях с уважением к изобилию средний транскрипт каждого гена во всех образцах. Буквы A – E на правой стороне указывают различные кластеры определены.AB: после взрыва; B: взрыв; Бад W: Зимние почки; F: цветения; FB: цветение начинается; FS: фруктов набор; G: зеленый; Г-н: середине созревания; PFS: после фруктов набор; PHWI-II-III: послепромысловых вянуть 1, 2 и 3 месяцев; R: созревания; S: стареющей; Вуди стволовых стволовых W:; V: созревания; WD: хорошо развиты; Y: Янг. (B) цвет шкалы представляет увеличение (красный) или уменьшить (синий) раз изменения экспрессии генов ATL виноградной лозы в зараженных проб, по сравнению с элементов управления для каждого условия. Звездочки показывают значительных дифференциальных выражение (ФДР < 0,05) каждой библиотеки ATL при соответствующих условиях. Воспроизводится из "Ariani" et al. по лицензии Creative Commons Attribution 4.0 международного42лицензии. Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.
Дополнительная таблица 1: ATL генов кандидатов для альтернативного сплайсинга. () ATL гена ID согласно V1 винограда гена прогнозирования и аннотации, (b) ATL гена ID согласно V2 винограда гена прогнозирования и аннотации43, (c) количество предполагаемых ATL альтернативного сплайсинга вариантов, (d) Информация о последовательности каждый предполагаемый вариант ATL кодирования. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Дополнительная таблица 2: Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Дополнительный файл 1: Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Обсуждение
В эпоху геномной многие семьи гена глубоко характеризовались в нескольких видов растений. Эта информация является предварительным условием для функциональных исследований и обеспечить рамки для дальнейшего изучения роли различных членов в семье. В этом контексте существует также потребность в номенклатуре системы позволяет уникально идентифицировать каждый член семьи, избегая дублирования и путаницы, которые могут возникнуть, когда имена назначаются независимо различных генов в различных исследовательских групп.
После вдумчивого рассмотрения научное сообщество Грейпвайн решила имя Грейпвайн генов в семье на основе сходства с Arabidopsis генов и создала ряд правил, которые должны быть применены для описания новых генов семей в виноградной лозы, в основном начиная с филогенетической сравнение нуклеотидные последовательности между виноградной лозы и арабидопсиса членов семьи8. Таким образом только гены, которые уже аннотацией и правильно названный в проростках Arabidopsis может использоваться в номенклатуре виноградной лозы. Поэтому процедура применяется для определения orthologues ATL виноградной лозы в проростках Arabidopsis, описанные здесь проводилась исключительно для удовлетворения требований назначения правильный Грейпвайн гена семьи номенклатуры. Тем не менее для других видов растений, альтернативные подходы могут быть вариантом. Например, могут быть выведены Гомология, с помощью двунаправленной взрыв хитов (BBH), где orthologues определяются как пар генов в двух видов, которые больше похожи (т.е., с высокий балл выравнивание) друг с другом, чем любой другой ген в другой видов44. Однако этот метод может пропустить много orthologues в случае высокий уровень дублирования гена, такие как в45растений и животных. Кроме того, в случае ATL-кодирование генов, ВВН может получить гены, не хватает точных ATL-тип кольцо-H2 структуры (включая остатков пролина) или гены, которые не Аннотированная и называют атласом в арабидопсиса. Хотя с эволюционной точки зрения этот поиск может быть соответствующим, получение orthologues, не аннотированные не выполнили сферы Грейпвайн ATL гена семьи аннотацию и номенклатуры и orthologues, который не помечен как атласом нельзя использовать имя Грейпвайн членам семьи. Другой возможностью является вывести Гомология, основанные на аминокислоты вместо нуклеотидных последовательностей с использованием InParanoid46, или самых последних Hieranoid 247, хотя такие процессы не рекомендуются прямо со стороны научного сообщества.
Выражение мета анализ, который может быть определен как систематический подход к изучению и комбинировать различные публично доступных данных хранилищ данных выражение, позволяет, выделение общих и различных молекулярных механизмов в различных условиях. Таким образом, интеграция информации выражение гена из нескольких крупных транскриптомики экспериментов может улучшить характеристика гена семьи, путем определения выражения профили членов семьи через эксперименты, таким образом минимизируя влияние эксперимент специфические факторы и поддержки более надежным предположение функции предполагаемого гена в частности процессов. Однако использование microarray данных требует интеграции выражение данных, полученных с различных платформ, учитывая свои собственные ограничения. Например, в платформе Nimblegen microarray виноградной лозы, значительная часть probesets для соответствующих генов, представленных в массиве (~ 13 000 генов) имеют потенциально крест гибридизации вопросы48. В случае семьи ATL виноградной лозы 15 гены могут быть затронуты такие явления. Тем не менее, как обсужено Крамер и др. 48, кросс выявление весьма аналогичные Джин членов семьи же зонд может предоставить интересную информацию относительно выражение в конкретных условиях, не только из одного гена, но из двух более генов, Обмен высокой последовательности сходство и таким образом потенциально обмена целей и функций. Другой потенциальной проблемой, относящиеся к microarray наборов данных является предел обнаружения выражение microarray платформ, которые не очень чувствительны. Оба решения касается, т.е., крест гибридизации и сигнал чувствительности, возможным решением может быть рассматривать только RNAseq выражения наборов данных. Однако мета анализ данных RNAseq очень больших наборов данных из многих различных исследований может стать весьма много времени и может потребовать много вычислительных ресурсов и высокий профессионализм.
Хотя подход здесь представлены стремится быть исчерпывающим, он может быть конечно еще дополнить другие анализы. Во-первых для достижения дальнейшего взглянуть на молекулярной эволюции и Филогенетические отношения между членами семьи генов растений, филогенетический анализ может быть продлен построения филогенетического дерева с использованием множественных выравниваний последовательности членов семьи от нескольких видов растений. Это также можно рассчитать время эволюционной семьи генов, оценки их синонимами и не являются синонимами замены ставок в ходе эволюции, определяя значения Ks (количество синонимом замен сайтаКупить синонимами в данной период времени) и Ка (количество nonsynonymous замены на не синонимами сайт в тот же период). Соотношения Ka/Ks используется для определения механизмов гена дублирование событий после отклонения от их предков. Значение ка/Ks = 1 предлагает нейтральные выбор, значение ка/Ks < 1 предлагает очищающий отбор и значение ка/Ks > 1 предлагает позитивного выбора49. Кроме того если анализ структуры гена показывает наличие интронов, характеристика семьи ген может быть продлен на обнаружение альтернативного сплайсинга варианты. На основе глубокого обзора РНК seq данных из различных тканей, стрессовых состояний и генотипов43, 21 (из 96) атласом действительно сильных кандидатов для альтернативного сплайсинга события, с потенциальное количество изоформ, от 2 до 16 для этих атласом (см. Дополнительная таблица 1). Альтернативные стенограммы часто производят изоформ белка, что различаются в аминокислотных последовательностей, и эти изменения могут изменить свойства клеточных белков и может вызвать изменения от тонкие модуляции к потере функции продукта гена. По этой причине альтернативного сплайсинга события были причастны функций важных растений, в том числе реакции на стресс, болезням, фотосинтез и цветения50,51.Интеграция информации промоутера генов ATL, содержащего предполагаемый СНГ-регуляторных элементов52 или нахождения молекулы (например, микроРНК и длиной некодирующих РНК), потенциально таргетинга атласом53 может также дополняться для выявить системы понимание сложных молекулярных регулирования и взаимодействие Грейпвайн атласом.
В заключение выбор анализы должны быть выполнены, а также процедуры для применения к характеризуют новое семейство генов в видов растений главным движителем правила научного сообщества, а также сферу семейных идентификации генов. Это важно иметь в виду возможные последующие расследования шаги, которые будет использовать набор данных, среди которых включает гена эволюции среди видов растений, описание структуры генома или надежных кандидатов для отбора в функциональных исследования.
Раскрытие информации
Авторы не имеют ничего сообщать.
Благодарности
Работа была поддержана Веронский университет – в рамках совместного проекта 2014 (характеристика семьи гена ATL в виноградной лозы и ее участия в устойчивость к милдью винограда).
Материалы
| Name | Company | Catalog Number | Comments |
| Personal computer | |||
| Basic Local Alignment Search Tool (BLAST) | https://blast.ncbi.nlm.nih.gov/Blast.cgi | ||
| Molecular Evolutionary Genetics Analysis (MEGA) | http://www.megasoftware.net/ | ||
| Motif-based sequence analysis tools (MEME) | http://meme-suite.org/ | ||
| Geneious | Biomatters Limited | http://www.geneious.com/ | |
| ProtParam Tool | http://web.expasy.org/protparam/ | ||
| ngLOC | http://genome.unmc.edu/ngLOC/index.html | ||
| TargetP v1.1 Server | http://www.cbs.dtu.dk/services/TargetP/ | ||
| Protein Prowler | http://bioinf.scmb.uq.edu.au:8080/pprowler_webapp_1-2/ | ||
| MUsite | http://musite.sourceforge.net/ | ||
| Pfam | http://pfam.xfam.org/ | ||
| TMHMM Server v. 2.0 | http://www.cbs.dtu.dk/services/TMHMM/ | ||
| ProtScale | http://web.expasy.org/protscale/ | ||
| Grape Genome Database (CRIBI) | http://genomes.cribi.unipd.it/grape/ | ||
| PhenoGram | http://visualization.ritchielab.psu.edu/phenograms/plot | ||
| MCScanX | http://chibba.pgml.uga.edu/mcscan2/ | ||
| Interactive Tree Of Life (iTOL) | http://itol.embl.de/ | ||
| UniProt | http://www.uniprot.org/ | ||
| Phylogeny.fr | http://www.phylogeny.fr/index.cgi | ||
| MUSCLE | http://www.ebi.ac.uk/Tools/msa/muscle/ | ||
| Gblocks Server | http://molevol.cmima.csic.es/castresana/Gblocks_server.html | ||
| Vitis vinifera cv. Corvina gene expression Atlas datamatrix | https://www.researchgate.net/publication/273383414_54sample_ datamatrix_geneIDs_Fasoli2012 | ||
| Multi Experiment Viewer (MeV) | http://mev.tm4.org/#/welcome | ||
| Sequence Read Archive (SRA) | https://www.ncbi.nlm.nih.gov/sra | ||
| R | https://www.r-project.org/ | ||
| EMBOSS Needle (EMBL-EBI) | http://www.ebi.ac.uk/Tools/psa/emboss_needle/ |
Ссылки
- Jaillon, O., et al. The grapevine genome sequence suggests ancestral hexaploidization in major angiosperm phyla. Nature. 449 (7161), 463-467 (2007).
- Adam-Blondon, A. -F., et al. Genetics, Genomics, and Breeding of Grapes. , Science Publishers. 211-234 (2011).
- Chen, L., Hellmann, H. Plant E3 Ligases: Flexible Enzymes in a Sessile World. Mol. Plant. 6 (5), 1388-1404 (2013).
- Vierstra, R. D. The ubiquitin-26S proteasome system at the nexus of plant biology. Nat. Rev. Mol. Cell Biol. 10 (6), 385-397 (2009).
- Serrano, M., Parra, S., Alcaraz, L. D., Guzmán, P. The ATL Gene Family from Arabidopsis thaliana and Oryza sativa Comprises a Large Number of Putative Ubiquitin Ligases of the RING-H2 Type. J. Mol. Evol. 62 (4), 434-445 (2006).
- Aguilar-Hernández, V., Aguilar-Henonin, L., Guzmán, P. Diversity in the Architecture of ATLs, a Family of Plant Ubiquitin-Ligases, Leads to Recognition and Targeting of Substrates in Different Cellular Environments. PLoS One. 6 (8), e23934(2011).
- Guzmán, P. The prolific ATL family of RING-H2 ubiquitin ligases. Plant Signal Behav. 7 (8), 1014-1021 (2012).
- Grimplet, J., et al. The grapevine gene nomenclature system. BMC Genomics. 15, 1077(2014).
- Prince, V. E., Pickett, F. B. Splitting pairs: the diverging fates of duplicated genes. Nat. Rev. Genet. 3 (11), 827-837 (2002).
- Magadum, S., Nerjee, U., Murugan, P., Gangapur, D., Ravikesavan, R. Gene duplication as a major force in evolution. J. Gen. 92 (1), 155-161 (2013).
- Wang, N. Patterns of Gene Duplication and Their Contribution to Expansion of Gene Families in Grapevine. Plant Mol. Biol. Rep. 31 (4), 852-861 (2013).
- Fasoli, M. The Grapevine Expression Atlas Reveals a Deep Transcriptome Shift Driving the Entire Plant into a Maturation Program. Plant Cell. 24 (9), 3489-3505 (2012).
- BLAST. BLAST2.6.0. , Available from: https://blast.ncbi.nlm.nih.gov/Blast.cgi (2016).
- MEGA. MEGA7.0.25 build 7170412. , Available from: http://www.megasoftware.net/ (2017).
- MEME. MEME Suite Version 4.11.4. , Available from: http://meme-suite.org/ (2017).
- ProtParam. ExPASy Server. , Available from: http://web.expasy.org/protparam/ (2005).
- ngLOC v1.0. , Available from: http://genome.unmc.edu/ngLOC/index.html (2007).
- TargetP v1.1 Server. , Available from: http://www.cbs.dtu.dk/services/TargetP/ (2000).
- Prowler v1.2. , Available from: http://bioinf.scmb.uq.edu.au:8080/pprowler_webapp_1-2/ (2005).
- MuSite v1.0. , Available from: http://musite.sourceforge.net/ (2010).
- Pfam. Pfam version 31.0. , Available from: http://pfam.xfam.org/ (2016).
- TMHMM v2.0c. , Available from: http://www.cbs.dtu.dk/services/TMHMM/ (2007).
- ExPASy. ProtScale. , Available from: http://web.expasy.org/protscale/ (2005).
- CRIBI. Grape genome database. , Available from: http://genomes.cribi.unipd.it/grape/ (2012).
- PhenoGram. , Available from: http://visualization.ritchielab.psu.edu/phenograms/plot (2012).
- ScanX v0.8. , Available from: http://chibba.pgml.uga.edu/mcscan2/ (2013).
- Interactive Tree Of Life (iTOL). Version3.5.3. , Available from: http://itol.embl.de/ (2016).
- UniProt. , Available from: http://www.uniprot.org/ (2016).
- Phylogeny.fr. , Available from: http://www.phylogeny.fr/index.cgi (2008).
- MUSCLE. , Available from: http://www.ebi.ac.uk/Tools/msa/muscle/ (2017).
- Gblocks Server. Version 0.91b. , Available from: http://molevol.cmima.csic.es/castresana/Gblocks_server.html (2002).
- Vitis vinifera cv. Corvina gene expression Atlas. , Available from: https://www.researchgate.net/publication/273383414_54sample_datamatrix_geneIDs_Fasoli2012 (2015).
- Multiple Experiment Viewer (MeV). Version 4.8.1. , Available from: http://mev.tm4.org/ (2017).
- Sequence Read Archive (SRA). , Available from: https://www.ncbi.nlm.nih.gov/sra (2017).
- Bolger, A. M., Lohse, M., Usadel, B. Trimmomatic: a flexible trimmer for Illumina sequence data. Bioinformatics. 30 (15), 2114-2120 (2014).
- Langmead, B., Salzberg, S. L. Fast gapped-read alignment with Bowtie 2. Nat Meth. 9 (4), 357-359 (2012).
- Anders, S., Pyl, P. T., Huber, W. HTSeq-a Python framework to work with high-throughput sequencing data. Bioinformatics. 31 (2), 166-169 (2015).
- R. Version 3.4.1. , Available from: https://www.r-project.org/ (2017).
- Ritchie, M. E. limma powers differential expression analyses for RNA-sequencing and microarray studies. Nucleic Acids Res. 43 (7), e47(2015).
- Love, M. I., Huber, W., Anders, S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2. Genome Biology. 15 (12), 550(2014).
- EMBL-EBI. EMBOSS Needle. , Available from: http://www.ebi.ac.uk/Tools/psa/emboss_needle/ (2017).
- Ariani, P. Genome-wide characterisation and expression profile of the grapevine ATL ubiquitin ligase family reveal biotic and abiotic stress-responsive and development-related members. Sci. Rep. 6, 38260(2016).
- Vitulo, N., et al. A deep survey of alternative splicing in grape reveals changes in the splicing machinery related to tissue, stress condition and genotype. BMC Plant Biol. 14 (1), 99(2014).
- Overbeek, R., Fonstein, M., D'Souza, M., Pusch, G. D., Maltsev, N. The use of gene clusters to infer functional coupling. Proc. Natl. Acad. Sci. USA. 96 (6), 2896-2901 (1999).
- Dalquen, D. A., Dessimoz, C. Bidirectional Best Hits Miss Many Orthologs in Duplication-Rich Clades such as Plants and Animals. Genome Biol. Evol. 5 (10), 1800-1806 (2013).
- Remm, M., Storm, C. E. V., Sonnhammer, E. L. L. Automatic clustering of orthologs and in-paralogs from pairwise species comparisons1. J. Mol. Biol. 314 (5), 1041-1052 (2001).
- Kaduk, M., Sonnhammer, E. Improved orthology inference with Hieranoid 2. Bioinformatics. 33 (8), (2017).
- Cramer, G. R., et al. Transcriptomic analysis of the late stages of grapevine (Vitis vinifera cv. Cabernet Sauvignon) berry ripening reveals significant induction of ethylene signaling and flavor pathways in the skin. BMC Plant Biol. 14, 370(2014).
- Juretic, N., Hoen, D. R., Huynh, M. L., Harrison, P. M., Bureau, T. E. The evolutionary fate of MULE-mediated duplications of host gene fragments in rice. Genome Res. 15 (9), 1292-1297 (2005).
- Filichkin, S. A. Genome-wide mapping of alternative splicing in Arabidopsis thaliana. Genome Res. 20 (1), 45-58 (2010).
- Quesada, V., Macknight, R., Dean, C., Simpson, G. G. Autoregulation of FCA pre-mRNA processing controls Arabidopsis flowering time. EMBO J. 22 (12), 3142-3152 (2003).
- Wong, D. C. J., Gutierrez, R. L., Gambetta, G. A., Castellarin, S. D. Genome-wide analysis of cis-regulatory element structure and discovery of motif-driven gene co-expression networks in grapevine. DNA Res. 24 (3), 311-326 (2017).
- Wong, D. C. J., Matus, J. T. Constructing Integrated Networks for Identifying New Secondary Metabolic Pathway Regulators in Grapevine: Recent Applications and Future Opportunities. Front. Plant Sci. 8, 505(2017).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены