Method Article
Flux de travail complet pour le génome-large d’Identification et de la Meta-analyse de l’Expression de la famille des gènes ATL E3 ubiquitine Ligase à Grapevine
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Cet article décrit la procédure pour l’identification et la caractérisation d’une famille de gènes dans la vigne à cette famille d' Arabidopsis Tóxicos dans Levadura (ATL) E3 ubiquitine ligases.
Résumé
Classification et nomenclature des gènes dans une famille peuvent contribuer significativement à la description de la diversité des protéines et à la prévision de fonctions familiales selon plusieurs caractéristiques, telles que la présence de motifs de séquence ou de particulier sites de modification post-traductionnelle et le profil d’expression des membres de la famille dans des conditions différentes. Cet ouvrage décrit un protocole détaillé pour la caractérisation des gènes familiaux. Ici, la procédure est appliquée à la caractérisation de la famille Arabidopsis Tóxicos dans Levadura (ATL) E3 ubiquitine ligase à grapevine. Les méthodes comprennent l’identification de tout le génome des membres de la famille, la caractérisation de la localisation du gène, la structure et les doubles emplois, l’analyse des motifs de protéine conservée, la prédiction des sites de localisation et de la phosphorylation des protéines ainsi que Profil d’expression génique au sein de la famille dans différents ensembles de données. Telle procédure, qui pourrait être étendu à de nouvelles analyses selon des fins expérimentales, pourrait être appliquée à toute famille de gènes à des espèces végétales pour lesquelles il existe des données génomiques, et il fournit des renseignements précieux pour identifier les candidats intéressants pour des études fonctionnelles, donnant un aperçu des mécanismes moléculaires de l’adaptation des plantes à leur environnement.
Introduction
Au cours de la dernière décennie, beaucoup de recherche a été effectuée en génomique de la vigne. Vigne est une culture économiquement pertinente reconnue, qui est devenu un modèle pour la recherche sur le développement de fruits et sur les réponses des plantes ligneuses aux stress biotiques et abiotiques. Dans ce contexte, la libération du Vitis vinifera CV PN40024 génome en 20071 et sa version mise à jour en 20112 conduit à une accumulation rapide de données à l’échelle « omiques » et à un éclatement des études de haut débit. Selon les données publiées de séquence, l’analyse détaillée d’une famille de gène donné (généralement composée de protéines partageant des motifs conservés, les similitudes structurelles ou fonctionnelles et les relations évolutives), peut désormais être réalisée afin de découvrir ses les fonctions moléculaires, évolution et profils d’expression génique. Ces analyses peuvent contribuer à comprendre comment familles de gènes contrôlent les processus physiologiques à une échelle du génome.
Plusieurs aspects du cycle de vie végétale sont réglementées par dégradation ubiquitine-mediated de protéines clés, qui exigent un chiffre d’affaires adaptée pour garantir des processus cellulaires réguliers. Important les composants du processus de dégradation induite par l’ubiquitine sont l’E3 ubiquitine ligases, qui sont responsables de la souplesse du système, grâce au recrutement de cibles spécifiques3. En conséquence, ces enzymes représentent une famille de gènes énorme, avec environ 1 400 E3 ligase-codage gènes prévues dans Arabidopsis thaliana génome4, chaque ubiquitine ligase E3 agissant pour l’ubiquitination des protéines cibles spécifiques. Malgré l’importance de l’ubiquitination de substrat spécifique dans la régulation cellulaire chez les plantes, on connaît mal comment la voie de l’ubiquitination est réglementée et protéines cibles ont été identifiées que dans de rares cas. Le déchiffrage de ces mécanismes de la spécificité et la régulation s’appuie d’abord sur l’identification et la caractérisation des différentes composantes du système, en particulier l’E3 ligases. Entre ubiquitine ligases, la sous-famille des ATL se caractérise par 91 membres identifiés chez a. thaliana affiche un anneau-H2 doigt domaine5,6, certains d'entre eux jouant un rôle dans la défense et l’hormone réponses7.
La première étape cruciale pour définir les membres d’une nouvelle famille de gènes est la définition précise des caractéristiques familiales, tels que les motifs de consensus, les domaines clés et caractéristiques de séquences de protéines. En effet, la récupération fiable de tous les membres de famille de gène BLAST analyse nécessite certaines caractéristiques de la séquence obligatoire, dans les domaines de protéine responsables de la protéine fonction/activité, servant de signature de la protéine. Cela peut être facilité par la caractérisation antérieure de la même famille de gènes chez d’autres espèces végétales ou obtenu en analysant les différents gènes putativement appartenant à la même famille dans différentes espèces de plantes, d’isoler les séquences communes. Les membres de la famille peuvent alors être individuellement nommés suivant des règles communes, a été réglées par un consortium international pour une espèce végétale donnée. Dans la vigne, par exemple, cette procédure est soumis aux recommandations du Comité de Nomenclature Super pour l’Annotation de gène de raisin (sNCGGa), établissant la construction d’un arbre phylogénétique y compris V. vinifera et a. thaliana membres de famille de gène pour permettre l’annotation de gène issu des séquences de nucléotides8.
Localisation chromosomique des membres de la famille et enquête sur la duplication génique permettent mettant en évidence la présence de gènes dupliqués génome entier ou tandem. Cette information semble utile pour démêler les fonctions des gènes putatifs, puisqu’elle pourrait voir la redondance fonctionnelle ou révéler des situations différentes, c'est-à-dire, non-fonctionnalisation, neo-fonctionnalisation ou subsidiaire fonctionnalisation9. Les deux néo - et sous-sous - functionalization sont des événements importants qui créent la nouveauté génétique, fournissant de nouveaux composants cellulaires pour l’adaptation des plantes à l’évolution des environnements10. En particulier, les duplications de gènes ancestraux et production de nouveaux gènes ont été très fréquentes au cours de l’évolution du génome vigne et nouvellement formé de gènes provenant des duplications proximales et tandem à grapevine étaient plus susceptibles de produire une nouvelle fonctions11.
Un autre facteur clé à déchiffrer la fonction des gènes familiaux est le profil de transcriptomique. La disponibilité des bases de données publiques donnant accès à une quantité énorme de données transcriptomiques peut être ainsi exploitée pour attribuer des fonctions aux membres de famille de gène à grande échelle en silico analyses d’expression. En effet, l’expression particulière de certains gènes dans les organes de la plante spécifique ou en réponse à certaines contraintes peut donner quelques conseils au sujet des rôles présumés des protéines correspondantes dans les conditions définies et apporter un soutien à des hypothèses sur la possible auxiliaire de fonctionnalisation de gènes dupliqués pour répondre aux différents défis. À cette fin, il est important de tenir compte de plusieurs ensembles de données : ceux-ci peuvent être gène déjà disponible des matrices d’expression, tels que l’atlas du génome transcriptomique des organes de la vigne et des stades de développement,12, ou peut être construits ad hoc par récupération des ensembles de données transcriptomiques pour les espèces de plante particulière soumises à des contraintes définies. En outre, une approche simple à l’aide de deux matrices, avec données de similitude par paires et l’autre avec des coefficients de co-expression par paire peuvent être appliqués afin d’évaluer les relations entre modèles de similitude et d’expression de séquence au sein d’une famille multigénique.
Le but de ce travail est de fournir une approche globale, définissant la structure des gènes, motifs de protéine conservée, localisation chromosomique, duplications géniques et profils d’expression, comme bien la prédiction des sites de localisation et de la phosphorylation des protéines, pour atteindre un caractérisation exhaustive d’une famille de gènes chez les plantes. Une telle approche globale est appliquée ici à la caractérisation de la famille ATL E3 ubiquitine ligase vigne. Selon le rôle émergent des ATL sous-famille des membres dans la régulation des processus cellulaires clés7, ce travail peut bien aider l’identification des candidats solides pour des études fonctionnelles et éventuellement élucider les mécanismes moléculaires régissant la adaptation de cette culture importante à son environnement.
Protocole
1. identification des ATL putatif famille multigénique membre (s)
- Version web PSI-BLAST
- Ouvrez la page web BLAST13 et cliquez sur la section BLAST de protéine.
- Dans le champ « Enter Query sequence », entrez la séquence d’acides aminés de la protéine (ici VIT_05s0077g01970) qui sera utilisée comme sonde pour identifier les autres membres de la famille.
Remarque : Une bonne protéine représentative doit être utilisée (une protéine affichant toutes les caractéristiques importantes qui caractérisent la famille). - Dans le champ « Jeu de recherche de choisir », sélectionnez la base de données « Protéine de référence » (refseq_protein) et l’organisme d’intérêt (V. vinifera - taxid:29760).
- Dans la champ « programme selection », sélectionnez algorithme PSI-BLAST et cliquez sur le bouton BLAST pour exécuter l’analyse.
NOTE : En cliquant sur les « paramètres de l’algorithme », il est possible d’ajuster certains paramètres avancés (séquences cibles Max, notation matricielle, seuil de PSI-BLAST, etc.). - La première explosion ronde récupère toutes les séquences affichant des correspondances pertinentes avec la requête (e-valeur supérieure au seuil sélectionné - par défaut 0,005 ; 0,001 dans cette expérience). Désélectionner toutes les entrées, ce qui clairement ne pas appartenir à la famille en cours d’examen en cliquant sur la coche dans la colonne « sélectionner pour PSI-BLAST » et exécuter la deuxième itération de PSI-BLAST en cliquant sur le bouton BLAST comme au point 1.1.4.
- Séquences nouvellement identifiés sont surlignés en jaune. Désélectionner les hits récupérées manifestement erronées et découvrir les autres itérations comme indiqué au point 1.1.5.
- Continuer avec les itérations jusqu'à ce que l’algorithme ne trouve pas n’importe quelle entrée pertinente ou qu’elle atteigne la convergence (aucuns nouvelles entrées ne sont trouvés). Télécharger la liste des membres de la famille des gènes putatifs pour approfondir les analyses. Inspecter visuellement les hits récupérées à chaque itération pour éviter la présence de faux positifs.
- Version autonome de PSI-BLAST
- Télécharger la version autonome de BLAST en cliquant sur le bouton « Télécharger BLAST » sur la page d’accueil BLAST13.
Remarque : Le logiciel BLAST autonome est une version de ligne de commande de l’interface web décrit précédemment. Il permet l’exécution de la recherche de PSI-BLAST contre une base de données locale ou distante personnalisée. En outre, il permet la recherche avec une matrice prédéfinis des Score spécifique en Position (PSSM).
- Télécharger la version autonome de BLAST en cliquant sur le bouton « Télécharger BLAST » sur la page d’accueil BLAST13.
2. manuelle Inspection des membres des familles PSI-BLAST-identifiés
- Alignement multiple
- Collecter les séquences d’acides aminés précédemment identifiés dans un fichier au format FASTA et transférez-le dans le méga logiciel14 de procéder à l’alignement multiple.
- Ouvrez le logiciel MEGA, cliquez sur le bouton « Aligner », cliquez sur « Edit/Build alignement », « Créer un nouveau tracé », « Protéines ».
- Cliquez sur « Editer » dans le menu alignement « Insérez la séquence from File ». Recherchez le fichier FASTA créé avant et confirmer le téléchargement de toutes les séquences étudiées.
- Cliquez sur « Alignement » dans le menu alignement et « Aligner par MUSCLE ». Utilisez les paramètres par défaut, cliquez sur le bouton « Calculer » et attendre l’achèvement de l’alignement multiple.
- Inspecter visuellement l’alignement multiple pour exclure les membres de la famille mal prédites. Le canonique CxxC (x 13) PxCxHxxHxxCxxxW (x 7) CxxCW motif, (en particulier la présence du résidu proline avant la troisième cystéine), est l’élément clé nécessaire pour définir les membres de la famille ATL.
- Analyse du LOGO spécifique
- Présenter la liste définitive des membres de la famille (96 séquences de vigne satisfont les exigences à prendre en considération ATL) à l’Em multiples pour Motif Elicitation (MEME)15 pour définir les motifs conservés au sein de la famille.
- De la MEME page d’accueil, cliquez sur le bouton « MEME » et compléter le « données formulaire de soumission » avec notamment les renseignements concernant la famille d’intérêt.
- Utiliser l’analyse de MEME pour confirmer la présence des deux motifs attendus dans les membres de la famille vigne ATL, c'est-à-dire, l’anneau-H2 et les motifs GLD.
- Sinon, procédez comme 2.1 et 2.2 simultanément à l’aide de la suite de logiciels de bioinformatique (voir Table des matières).
- Télécharger fichier FASTA (voir étape 2.1.1) dans la suite. Sélectionnez « Fichier » dans le menu, puis « Importer » et cliquez sur « fichier ». Recherchez le fichier FASTA et cliquez sur « Ouvrir ».
- Sélectionner toutes les séquences importées dans la liste et cliquez sur le bouton « Aligner/Assemble » dans la barre d’outils, puis cliquez sur « Pairwise alignement Multiple ». Sélectionnez « Alignement de Muscle » et cliquez sur « OK » pour lancer l’alignement en utilisant les paramètres par défaut.
- Pour visualiser le LOGO de l’alignement, cliquez sur « Graphs » → « options » et sélectionnez « Logo de la séquence ».
3. analyse des paramètres physiques de protéine et domaines
- Comme la définition des différents paramètres physiques des membres de la famille interrogés est importante d’avoir une description complète de la famille, présenter la liste des membres de la famille d’outils web spécifiques.
- Point isoélectrique (pI) et poids moléculaire (kDa), utilisez l' outil de ProtParam16 sur le site Expasy avec les paramètres par défaut.
- Pour la localisation subcellulaire des protéines, utiliser différents outils pour obtenir une prévision plus fiable comme ngLOC v1.017 avec les paramètres par défaut, targetP v1.118 avec les paramètres par défaut et la protéine prowler localisation sous-cellulaire v1.219 avec un seuil de probabilité de 0,5. Pour les sites de phosphorylation, utilisez le MUsite v1.0 web outil20 avec les paramètres par défaut.
- Enquêter sur des domaines de protéine supplémentaire dans les membres de la famille.
- Ouvrez la page Web de base de données Pfam21, sélectionnez l’outil « Recherche de séquence », soumettre des séquences de protéines dans la boîte de requête et cliquez sur « Go » pour lancer l’analyse.
Remarque : Chaque séquence protéique est analysée individuellement. Une e-valeur de 1,0 dans le paramètre par défaut permet de distinguer des succès significatifs et non significatifs. - Ouvrez le serveur TMHMM22 du Centre pour l’analyse des séquences biologiques enquêter sur la présence de régions transmembranaires putatives.
- Ouvrez la page Web de base de données Pfam21, sélectionnez l’outil « Recherche de séquence », soumettre des séquences de protéines dans la boîte de requête et cliquez sur « Go » pour lancer l’analyse.
4. chromosomique Distribution, Duplications et organisation Exon-intron
- Carte des membres de la famille ATL sur les chromosomes basés sur les informations récupérées du site Web de vigne Genome CRIBI Biotech Center24.
- Parcourir le phénogramme site Web page d’accueil25. Écrire le « Input File » comme un fichier texte délimité par des tabulations avec les caractéristiques spécifiques des gènes à être localisés sur les chromosomes, conformément aux lignes directrices exhaustives et exemples concernant la compilation du fichier fourni suivant le chemin « Phénogramme » → " Documentation » → « Options » → « Input file ».
- Écrire le « titre » de l’ouvrage. Sélectionnez le génome à tirer. Pour les génomes non implémentées dans le logiciel, telles que le génome de la vigne, sélectionnez « autre » dans le menu déroulant. Écrire le fichier de génome selon les lignes directrices et les exemples fournis, suivant le chemin « Phénogramme » → « Documentation » → « Options » → « Génome » et de le transférer.
- Utilisez les paramètres par défaut de « Espacement de phénotype », « Couleur de phénotype », « Format de l’Image » ou sélectionner des solutions de rechange dans les menus respectifs, puis cliquez sur « Tracer » obtenir la visualisation des gènes sur les chromosomes.
- Définir l’état de la duplication des membres de la famille en utilisant le logiciel de MCScanX26.
- Téléchargez et décompressez une copie de MCscanX sur un ordinateur local exécutant les lignes de commande 1 (supplémentaire 1 fichier). Entrez dans le dossier MCscanX et créer des exécutables requis exécute les lignes de commande 2 (supplémentaire 1 fichier).
Remarque : Installation de MCscanX est connue pour échouer sur certains Linux 64 bit machines suite à un problème concernant la fonction chdir. Si un message d’erreur est retourné lié à cette fonction sur la marque exécution de la commande, les lignes de commande 3 (fichier complémentaire 1) doit être exécutés et la commande « make » devrait être tentée par la suite. - Télécharger les protéines de V. vinifera et le fichier d’annotation exécutant les lignes de commande 4 (supplémentaire 1 fichier).
Remarque : La vigne annotation fichier doit être décompressé et le chat d’informations de chromosomes simples dans un unique fichier en exécutant les lignes de commande 5 (supplémentaire 1 fichier). - Exécuter un blastp « tous contre tous » une recherche en utilisant le fichier de protéine V. vinifera en tant que la requête et le sujet.
- Créer une base de données interrogeable souffle en utilisant le fichier protéine V. vinifera lignes 6 (fichier complémentaire 1) de la commande en cours d’exécution. Effectuer la recherche blastp en utilisant le fichier de protéines de V. vinifera comme une requête contre la base de données créé précédemment en exécutant les lignes de commande 7 (supplémentaire 1 fichier).
- Convertissez le fichier d’annotation dans un format approprié pour MCScanX. Exécutez les lignes de commande 8supplémentaire 1 fichierpour télécharger le parseMSCanXgff.pl de script perl personnalisé. Effectuer l’analyse en exécutant les lignes de commande 9 (supplémentaire 1 fichier).
Remarque : Un fichier vitis.gff est généré qui contient les coordonnées de gène dans le format suivant :
position de départ de gène en SP # position de fin
« sp » où est un code à deux lettres pour l’espèce (Vv pour la vigne) alors que le « # » est le nom de l’échafaudage. Notez que le script perl personnalisé fourni est aménageable plus, bien que quelques modifications de code peuvent être nécessaire dans certains cas particuliers, en raison de la diversité de l’information fournie dans le fichier d’annotation disponibles. - Lancer MCScanX lignes de commande 10supplémentaire 1 fichieren cours d’exécution.
NOTE : Le « vitis » est le préfixe de l’annotation et le fichier de sortie de souffle. Il s’agit d’une condition obligatoire pour le logiciel de fonctionner. - Analyser les résultats de le MCScanX. MCScanX produit un fichier texte « vitis.collinearity », qui contient les blocs colinéaires. Un tel fichier peut être inspecté par n’importe quel éditeur de texte (voir exemple 1 supplémentaire 1 fichierde sortie).
Remarque : Un répertoire « mcscaxOutput.html » contenant des fichiers html comportant plusieurs alignements de blocs colinéaires contre chaque chromosome de référence est généré. Ces dossiers peuvent être consultés via un navigateur web. - Classer des gènes paralogues basés sur leur position relative dans les chromosomes lignes 11 (fichier complémentaire 1) de la commande en cours d’exécution.
NOTE : Classification de gènes paralogues est décrite au Tableau complémentaire II. Le fichier de sortie généré « vitis.gene_type » contient toutes les informations d’origine avec un format simple délimité par des tabulations. - Effectuer une analyse de l’enrichissement d’évaluer si la famille de gènes est à l’origine principalement par un mécanisme spécifique en exécutant les lignes de commande 12 (supplémentaire 1 fichier).
Remarque : Le fichier « vitis.gene_type » est généré pendant l’étape 4.2.8, tandis que le fichier « gene_family_file » représente un fichier de texte d’une ligne dans laquelle le nom de la famille (p. ex., ATL_genes) est suivi par les noms de lieu pour l’ensemble des gènes appartenant à la famille séparées par une tabulation. Le test statistique appliqué pour l’enrichissement est un test exact de Fisher et les p-valeurs d’origines différentes sont stockées dans le fichier « outputFile.txt ».
- Téléchargez et décompressez une copie de MCscanX sur un ordinateur local exécutant les lignes de commande 1 (supplémentaire 1 fichier). Entrez dans le dossier MCscanX et créer des exécutables requis exécute les lignes de commande 2 (supplémentaire 1 fichier).
- Visualiser l’organisation exon-intron des gènes à l’aide Interactive Tree Of Life (iTOL)27, un outil en ligne pour l’affichage, annotation et gestion des arbres phylogénétiques.
- Télécharger un arbre phylogénétique dans la section « Téléchargement » du site iTOL. L’arbre est construit selon la Section 5 ci-dessous. Pour chaque gène membre de la famille, récupérer la prédiction de structure génétique de l’annotation de la V1 du génome de la vigne (site de le CRIBI citée plus haut). Calculer la longueur (en PB) de régions non traduites (RTNS), introns et exons putatifs.
- Utiliser le dataset « Domaines protéiques » pour une visualisation graphique du modèle exon-intron.
5. nomenclature et analyse phylogénétique
- Analyser les relations entre les membres de la famille à travers la construction d’un arbre phylogénétique de haute qualité et la définition d’une nomenclature famille ATL.
- Pour une famille de gènes de vigne, suivre les règles établies par le Comité de Nomenclature Grapevine Super8.
- Récupérer les séquences chez a. thaliana ATL, nécessaires comme référence pour la vigne gène nomenclature8, de base de données UniProt28 .
- Écrire un fichier FASTA, y compris toutes les séquences de nucléotides de la vigne et les membres de famille de gène a. thaliana à inclure dans l’analyse phylogénétique. Les séquences nucléotidiques accepte le maximum de variabilité entre les membres de la famille (par rapport aux séquences de protéines).
- Arbre phylogénétique
Remarque : L’utilisation de l’oléoduc de 29 Phylogeny.fr est recommandée pour obtenir un arbre phylogénétique de haute qualité, mais non obligatoire.- Accédez à la page d’accueil de Phylogeny.fr29, puis sélectionnez le pipeline « Analyse de phylogénie ».
NOTE : « One Click » convient dans la plupart des cas, mais si nécessaire, il est possible de sélectionner les paramètres avancés spécifiques (« avancé ») ou même une analyse entièrement personnalisée (« a la Carte » ; Voir l’étape 5.2.5). - Écrire le « nom de l’analyse, » Télécharger le fichier FASTA créé précédemment (étape 5.2.1 et cliquez sur « Soumettre » pour exécuter l’analyse.
- Par ailleurs, si la procédure décrite plus haut (étapes 5.2.1, 5.2.2) entraîne un message d’erreur complet chaque étape de l’oléoduc de suite de phylogénie individuellement, comme suit.
- Dans le MUSCLE logiciel Accueil30, upload le fichier FASTA « Étape 1 », sélectionnez « Pearson/FASTA » comme « Format de sortie » à le « étape 2", cliquez sur « Soumettre » à le « Étape 3 » pour aligner les séquences de la requête.
- Cliquez sur « Télécharger le fichier d’alignement » et enregistrer en format FASTA pour nouvelles mesures à prendre.
- Processus du fichier FASTA alignement afin d’éliminer le mal aligné des positions à l’aide de Gblocks serveur outil31. Télécharger le fichier FASTA alignement, sélectionnez « ADN » comme « Type de séquence » et a choisi l’ou les options d’une rigueur qui correspond le mieux à l’analyse (par exemple, pour sélectionner famille de gène ATL de vigne tous les trois options proposées pour « moins stricte sélection » parce que haute de divergence des séquences). Cliquez sur « Get blocs » pour exécuter l’analyse.
- Cliquez sur « Alignement résultant » au bas de la page de sortie et enregistrer les résultats dans un nouveau fichier FASTA.
- Depuis la page d’accueil de Phylogeny.fr29, sélectionnez « A la Carte » pipeline « Analyse de phylogénie ». Ensuite, décochez l’option « Alignement Multiple » et « Curation d’alignement ». Cliquez sur « Créer un flux de travail », téléchargez le fichier Gblocks-curated FASTA (étape 5.2.5.4), sélectionnez « Procédure de Bootstrapping » avec les paramètres par défaut dans « Paramètres » et cliquez « Valider » pour lancer l’analyse.
- Branches d’effondrement mal pris en charge (c'est-à-dire, les valeurs « bootstrap » < 70 %) en cliquant sur « Effondrement des branches » dans la section « Select et action » et télécharger les résultats finaux au format Newick d’analyses plus poussées.
- Accédez à la page d’accueil de Phylogeny.fr29, puis sélectionnez le pipeline « Analyse de phylogénie ».
- Attribuez un nom de gène basé sur la phylogénie.
- Examiner l’arbre phylogénétique pour évaluer la fiabilité de la structure de l’arbre en le téléchargeant dans la suite iTOL citée ci-dessus (section 4.3).
- Attribuer manuellement un nom de gène pour chaque membre de la famille. Dans le cas des orthologues biunivoque, attribuer l' Arabidopsis-comme nom (p. ex., AtATL3 → VviATL3). Différencier les gènes de la vigne (deux ou plus) dérivant d’un homologue d’Arabidopsis unique avec la même distance phylogénétique à l’aide de nombres ou lettres si les gènes de l’Arabidopsis se termine par un nombre (par exemple, AtATL23 → VviATL23a, VviATL23b).
- Dans le cas d’un-à-plusieurs ou plusieurs-à-plusieurs orthologues, attribuer un nouveau nom de gènes de l' Arabidopsis-comme nom (ici, « ATL ») associé à un nombre plus élevé que le plus grand nombre, déjà utilisé pour V. vinifera et Arabidopsis (e.g., VviATL83).
- Compléter la nomenclature de la famille nouvellement définie, descendant du haut vers le bas de l’arbre phylogénétique.
6. vigne orgue et Stage profilage de l’Expression
- Générer les données d’expression contenant travail données matrice pour les membres de la famille.
- Téléchargez le V. vinifera CV Corvina gene expression Atlas datamatrix du lien distribué sur la plate-forme de ResearchGate32. Ce fichier contient les valeurs d’expression de RMA normalisé à être utilisés en suivant les étapes.
- Extrait les valeurs de l’expression pour chaque gène familial le datamatrix Atlas et écrire un « travail datamatrix » contenant la même ligne d’en-tête que le datamatrix Atlas. Sauver le datamatrix « travail » comme un fichier texte délimité par des tabulations.
- Effectuer l’analyse hiérarchique de bi-cluster utilisant le logiciel Multi expérience Viewer (MeV).
- Télécharger et installer le MeV logiciel33.
- Télécharger le « travail datamatrix » (étape 6.1.2) suivant le chemin « Fichier » → « Load Data » → « Parcourir » et sélectionnez le fichier texte. Sélectionnez « tableau de simple-couleur » et décochez la case de « Charge Annotation » lorsqu’une annotation automatique n’est pas fournie. Sélectionnez la valeur d’expression coin supérieur gauche de l’aperçu de table expression et cliquez sur le bouton « Load ».
- Ajuster les données application transformation Log2 (« Ajuster les données » → « Journal des Transformations » → « Log2 transformer ») et normalisation de gène/Row (« Ajuster les données » → « Gene/ligne ajustements » → « gène/rangée médiane centrale »). Définissez la limite de l’échelle appropriée (« Display » → « Set couleur échelle limites »).
- Calculer le Clustering hiérarchique suivant le chemin « Analyse » → « Clustering » → « HCL ».
7. profilage de l’expression en réponse aux stress biotiques et abiotiques
- Répétez l’étape 6.1 avec l’ID d’adhésion GSE provenant de publications respectives et des études sur le stress biotiques et abiotiques sur vigne. Par exemple, les expériences fournissant le profil de transcriptome de vigne baies infectées par le champignon pathogène Botrytis cinerea utilisant le raisin NimbleGen Whole-genome microarray peuvent être parcourus avec GSE ID de GSE52586. Répétez les étapes 6.1.1 et 6.1.2.
- Rechercher le NCBI séquence lit Archive34 avec le SRA/BioProject ID (par exemple, SRP055458 ou PRJNA275778 pour des expériences de « ombrage de fleur de vigne ») et télécharger toutes les lectures de séquence brute. RNA-seq datasets de nombreuses différentes études sont traitées à l’aide d’un seul tuyau par souci de cohérence.
- En bref, couper la séquence brute FASTQ se lit comme suit (single - et paire-end) et filtre qualité avec Trimmomatic35. Utilisez qu'un AVGQUAL et le DIFOK filtrent de 20 et 40, respectivement et tous les paramètres par défaut.
- Index des 12 X vigne référence génome1 à l’aide de Bowtie236. Télécharger 12 X vigne référence génome (par exemple, bowtie2-build) avant d’exécuter la commande bowtie2 .
- Obtenir des tableaux matrice avec htseq-comte de37 en utilisant le fichier vigne V1 gène modèle annotation (GFF/FTE).
- Effectuer une analyse différentielle des gènes expression (re-) R38 avec Leduc39 bibliothèques pour les matrices de RMA normalisé et DESeq240 pour tableaux matrice obtenues à partir des étapes 7.1.1 et 7.2.1, respectivement.
- Effectuer une comparaison entre les « deux groupes » standard (c'est-à-dire, le « traitement » / « commande »). S’assurer que les conception matrice/groupements de conditions « contrôle » et « traitement » sont correctement spécifiées.
Remarque : Un design typique pour l’analyse de l’expression différentielle de microarray (GSE52586) pour comparer les EL-33 baies infectées avec Botrytis cinerea contre baies (sain) de contrôle au même stade de développement par Leduc en exécutant les lignes de commande 13 apparaît en supplémentaire 1 fichier. Un design typique pour l’analyse de l’expression différentielle de RNA-seq (SRP055458 ou PRJNA275778) pour comparer les fleur (à 7 jours après la chute de cap) sous traitement ombre contre le contrôle avec DESeq2 en exécutant les lignes de commande 14 apparaît en supplémentaire 1 fichier . - Obtenir les listes des gènes différentiellement exprimés (DEG) dans chaque contraste, Leduc, utilisez les fonctions lmFit(), suivie eBayes()et puis par topTable() fonctions, tandis que pour DESeq2, utilisez le DESeqDataSetFromMatrix(), DESeq()et fonctions results() . Ci-dessous, un flux de production type à suivre.
- Pour l’analyse de l’expression différentielle de microarray, voir les lignes de commande 15 (supplémentaire 1 fichier). Pour l’analyse de l’expression différentielle de RNA-seq voir les lignes de commande 16 (supplémentaire 1 fichier). Répétez les étapes ci-dessus pour toutes les autres contrastes avec le schéma de conception appropriée différente (voir exemples dans étape 7.3.1)
- Effectuer une comparaison entre les « deux groupes » standard (c'est-à-dire, le « traitement » / « commande »). S’assurer que les conception matrice/groupements de conditions « contrôle » et « traitement » sont correctement spécifiées.
- Dans les listes de DEGs générées, extraire toutes les lignes qui ne correspondent à l’adhésion de l’ATL V1, conserver les colonnes contenant la modification de plier log2 (traitement/contrôle) > | 0,5 | et ajusté les p-valeurs (FDR) < 0,05 et fusion eux en conséquence dans une table de matrice, si une étude correspond à « abiotique » ou recueils des « interactions biotiques/pathogène ».
- Construire les heatmaps hiérarchique en cluster (recueils abiotiques et biotiques) en R en utilisant les bibliothèques gplots.
NOTE : Appel de la fonction de heatmap.2 construit le heatmap avec ligne dendrogrammes des tables respectives de matrice. Des arguments supplémentaires à l’aide de cellnote fonctionnent permet de distinguer différentiellement exprimés (log2FC > 0,5, FDR < 0,05) gènes ATL dans chaque comparaison à travers une large gamme de conditions expérimentales par une * symbole. Appliquer le flux de travail typique dans R exécution de lignes de commande 17 (supplémentaire 1 fichier) ou sinon, répétez les étapes 6.2.2 à 6.2.5 pour construire les heatmaps à l’aide de logiciels de MeV.
8. l’analyse des relations entre la Divergence des séquences paralogues et gène la co-expression
- Construire la matrice contenant similitude par paires. Les éléments de la matrice de similarité sont les valeurs de similarité de séquence calculée à partir des alignements de protéine par paires.
- Utilisez le BOSSELAGE aiguille web serveur41 avec les paramètres par défaut pour faire des alignements de séquences par paires et enregistrer en tant que fichier texte. Ouvrez le fichier texte de sortie et supprimer toutes les lignes de commentaires, ainsi que les noms de colonne et de ligne pour générer un fichier appelé « similarityTable.txt ».
Remarque : Une telle table dispose d’une ligne pour chaque gène ATL rapports les valeurs de similarité calculées dans chacune de l’alignement par paires. L’ordre des locus en lignes et colonnes est le même, afin qu’une matrice symétrique est générée dans le respect des valeurs diagonales.
- Utilisez le BOSSELAGE aiguille web serveur41 avec les paramètres par défaut pour faire des alignements de séquences par paires et enregistrer en tant que fichier texte. Ouvrez le fichier texte de sortie et supprimer toutes les lignes de commentaires, ainsi que les noms de colonne et de ligne pour générer un fichier appelé « similarityTable.txt ».
- Construire la matrice avec les données de la co-expression en calculant le coefficient de corrélation de Pearson. La procédure suivante requiert R et le module perl PDL.
- Télécharger les valeurs de l’expression pour les gènes ATL 96 lignes de commande 18 (supplémentaire 1 fichier) dans un terminal en cours d’exécution. Effectuer une analyse de la co-expression en utilisant un script perl personnalisé qui peut être téléchargé en exécutant les lignes de commande 19 (supplémentaire 1 fichier). Ce script va calculer le coefficient de corrélation de Pearson entre paires de loci ATL comme indiqué précédemment.
- Lancez le script exécutant les lignes de commande 20supplémentaire 1 fichieret suivez les instructions de sortie.
- Exécutez le test de Mantel en chargeant les matrices de deux données et produire les statistiques en exécutant les lignes de commande 21 (fichier complémentaire 1), avec « nrep » représentant le nombre de permutations. Le test se compose de calcul de la corrélation entre les éléments de ces matrices, permutation des matrices et ensuite calculer la statistique de test même encore une fois.
Remarque : Toutes les valeurs obtenues du test statistique sont utilisés pour construire une distribution de référence du test statistique, qui servira à calculer une p-valeur pour tester la signification. Le nombre de permutations définit la précision avec laquelle la p-valeur peut être obtenue.
Résultats
Le gène VIT_05s0077g01970, identifié comme étant le plus semblable à a. thaliana ATL2 (At3g16720) grâce à une recherche de BLASTp, a été utilisé comme sonde pour sonder les membres de la famille ATL dans le génome de la vigne (Vitis vinifera cv Pinot Noir PN40024). L’analyse de PSI-BLAST ont convergé après quelques cycles révélant une liste de gènes putatifs appartenant à la famille de gène ATL de vigne (Figure 1 a). La présence du domaine canonique RING-H2 pour chaque candidat a été évaluée par l’examen visuel de l’alignement de MUSCLE de toutes les entrées définies dans l’analyse (Figure 1 b). Seulement ces gènes contenant des acides aminés conservés correctement espacées, les deux résidus d’histidine, ainsi que les résidus de proline avant la troisième cystéine ont été considérés comme ATLs selon la définition originale de ATL dans Arabidopsis5. Un total de 96 vigne gènes rempli les conditions et ont été considérés pour davantage de caractérisation. Chaque membre de la famille ATL a été analysée pour définir les caractéristiques spécifiques du gène et la correspondante protéine codée, c'est-à-direla présence d’autres domaines connus en plus de l’anneau-H2, régions riches transmembranaires ou hydrophobes, subcellulaires localisation et sites de phosphorylation putatif (tableau 1 et tableau 2).

Figure 1 : enquête PSI-BLAST et alignement de la vigne putatif ATLs. (A) capture d’écran des hits top 10 de la première recherche d’itération PSI-BLAST à l’aide de la séquence de la protéine VIT_05s0077g01970 comme appât. (B) partie de l’alignement de la vigne sélectionné 96 putatifs ATLs montrant leur domaine RING-H2 et le LOGO correspondant obtenus à l’aide d’une suite de biologie moléculaire (voir Table des matières). Reproduit de Ariani et al. , sous une licence Creative Commons Attribution 4.0 International42.S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
| Nom | ID de gène | Longueur de gène (bp) | Numéro de l’intron | UniProt ID | Longueur de protéine (aa) | Motif de l’anneau-H2 | Nombre de domaine TM/H | Autres domaines | ||
| VviATL3 | VIT_09s0002g00220 | 1245 | 0 | F6HXK6 | 304 | PxC | 1 | |||
| VviATL4 [VviRHX1A] | VIT_15s0021g00890 | 1827 | 3 | D7SM36 | 203 | PxC | 0 | |||
| VviATL18 | VIT_11s0118g00780 | 1113 | 2 | F6HCI8 | 193 | PC | 0 | |||
| VviATL23a | VIT_18s0001g01060 | 935 | 0 | F6H0E4 | 114 | PxC | 0,5 | |||
| VviATL23b | VIT_18s0001g01050 | 399 | 0 | E0CQX3 | 132 | PxC | 1 | |||
| VviATL24 | VIT_17s0000g06460 | 4466 | 4 | D7SI89 | 217 | PxC | 1 | |||
| VviATL27 | VIT_00s0264g00020 | 2554 | 4 | D7T1R5 | 235 | PxC | 1 | |||
| VviATL43 | VIT_11s0052g00530 | 1576 | 2 | D7SQD9 | 457 | PxC | 3 | |||
| VviATL54a | VIT_18s0001g06640 | 3221 | 1 | F6H0Y5 | 405 | PxC | 1 | |||
| VviATL54b | VIT_03s0017g00670 | 2774 | 1 | F6HTI0 | 427 | PxC | 1 | |||
| VviATL55 [VviRING1] | VIT_07s0191g00230 | 1844 | 0 | F6HRP9 | 372 | PxC | 1 | |||
| VviATL63 | VIT_06s0004g06930 | 804 | 0 | D7SJU6 | 267 | PxC | 1 | |||
| VviATL65 | VIT_03s0063g01890 | 2068 | 0 | F6HQI8 | 396 | PxC | 1 | |||
| VviATL82 | VIT_01s0026g02540 | 820 | 0 | F6HPQ9 | 233 | PC | 0,5 | |||
| VviATL83 | VIT_17s0000g08400 | 1887 | 0 | F6GSQ4 | 143 | PC | 0 | |||
| VviATL84 | VIT_06s0004g00120 | 1853 | 0 | F6GUP5 | 368 | PC | 0,5 | ZF-RING_3 | ||
| VviATL85 | VIT_12s0034g01400 | 786 | 0 | F6H965 | 261 | PC | 0,5 | |||
| VviATL86 | VIT_12s0034g01390 | 1434 | 1 | D7T016 | 451 | PC | 0,5 | |||
| VviATL87 | VIT_18s0001g03270 | 1002 | 0 | F6H0T2 | 333 | PC | 0,5 | ZF-RING_3 | ||
| VviATL88 | VIT_08s0040g00590 | 1320 | 0 | F6HQR2 | 314 | PC | 0 | ZF-RING_3 | ||
Tableau 1 : première 20 VviATL gènes et caractéristiques de la séquence des protéines correspondantes. TM : transmembranaire ; H: hydrophobes ; 0,5 indique la présence d’une ou plusieurs régions hydrophobes. Reproduit de Ariani et al. , sous une licence Creative Commons Attribution 4.0 International42.

Tableau 2 : détails sur les 20 premiers VviATL position du gène dans V. vinifera génome, état de dédoublement et caractéristiques physico-chimiques ATL protéine et emplacement. (a) nombre de sites de phosphorylation prédite par Musite ; (b) des prévisions similaires obtenues avec au moins deux logiciels sont mises en évidence en caractères gras ; ngLOC a été utilisée avec les paramètres par défaut, tandis que TargetP v1.1 et la localisation intracellulaire de la protéine Prowler ont été utilisés avec un seuil de probabilité de 0,5. NUC, noyau ; MIT, mitochondries ; CHL, chloroplaste ; PLA, membrane plasmique ; S, voie de sécrétion (présence d’un peptide signal) ; M, mitochondries ; C, chloroplaste ; O ou -, autres localités ; ND, ne pas déterminé (par exemple, valeur inférieure au seuil). Reproduit de Ariani et al. , sous une licence Creative Commons Attribution 4.0 International42. S’il vous plaît cliquez ici pour télécharger ce fichier.
Une analyse phylogénétique, y compris les séquences nucléotidiques des gènes de codage de ATL vigne identifiés ainsi que les séquences de la famille de gènes de référence a. thaliana ATL a été utilisée pour la nomenclature de vigne ATL, conformément aux directives de le sNCGGa8. Quatre-vingt-seize et 83 des séquences de nucléotides de V. vinifera et a. thaliana, respectivement, ont été soumis à l’oléoduc de Phylogeny.fr pour obtenir un arbre phylogénétique fiable.Les séquences de ce dernier furent ensuite utilisées pour annoter et nommez les gènes de la vigne sur la base de relations solides (Figure 2). Suivant cette approche, 13 des 96 vigne ATLs a reçu un identifiant spécifique compte tenu de leur niveau un à un avec un a. thaliana ATL Les noms des 83 autres gènes ont été assignés basés sur l’arbre phylogénétique, avec un progressif numérotation de haut en bas, à partir d’un nombre de gènes ATL plus élevé que la plus élevée utilisée dans numéro a. thaliana.
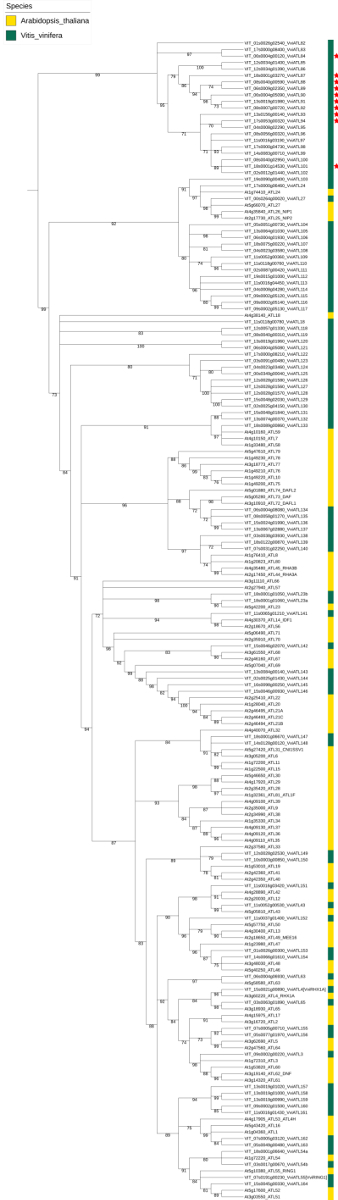
Figure 2 : Arbre phylogénétique de V. vinifera et a. thaliana ATL E3 ubiquitine ligase-codage gènes. L’arbre des s’est amorcé à la suite de Phylogeny.fr (V. vinifera (en vert) et les gènes ATL 83 de a. thaliana signalés dans la base de données UniProt (en jaune). Les valeurs de support Branch proviennent de 100 répétitions bootstrap. Les étoiles rouges indiquent la présence d’un domaine de doigt (BZF) de zinc BCA2 en protéines correspondantes. Reproduit de Ariani et al. , sous une licence Creative Commons Attribution 4.0 International42. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
Cartographie des gènes codant ATL aux chromosomes vigne ont montré une large distribution dans tout le génome, ce qui suggère de duplication du génome entier comme la plus grande force évolutive dans l’expansion de la famille de gènes ATL à grapevine. En effet, 31 ATLs ont été trouvés dans des régions chromosomiques homologues potentiellement provenant d’évènements de duplication du génome entier ou segmentaire. En outre, la même analyse a souligné 13 gènes dupliqués en tandem, un duplicata proximale et 51 doublons dispersées (Figure 3). Compte tenu du très grand nombre de gènes dupliqués dans la famille ATL, nous avons effectué un test d’enrichissement (test exact de Fisher) pour vérifier la conservation préférentielle des gènes dupliqués lors du fractionnement du génome. Avec une p-valeur < 0,001, cet essai confirmé l’hypothèse selon laquelle dupliqué gènes ATL ont été retenus prévu plus de façon aléatoire, ce qui suggère un rôle pour la famille de gènes ATL pendant l’adaptation de la vigne et de l’évolution.
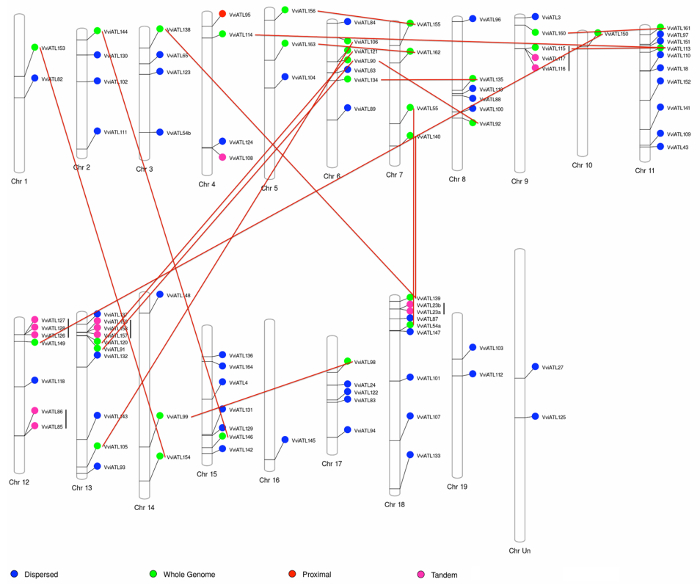
Figure 3 : Vigne ATL-encodage la distribution des gènes sur les chromosomes de V. vinifera et état de dédoublement. Les gènes ATL 96 vigne avec informations chromosomiques précises disponibles dans la base de données ont été localisés aux 19 V. vinifera chromosomes. Les couleurs indiquent l’événement original de la duplication. Les lignes noires verticales et des lignes rouges identifient des paires de dérivés de duplications en tandem et duplications du génome entier, respectivement. Reproduit de Ariani et al. , sous une licence Creative Commons Attribution 4.0 International42. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
Afin d’étudier les fonctions biologiques putatives de l’ATLs à grapevine, une méta-analyse a été effectuée sur le CV de V. vinifera l’expression des gènes global Corvina Atlas12. Le dataset comprend des valeurs de l’expression du génome entier de 54 vigne différents organes et stades de développement et a été utilisé pour effectuer une analyse hiérarchique de bi-groupé. Résultats non seulement confirment que tous les 96 ATLs étaient exprimés dans au moins un des tissus 54/étapes, mais faits aussi remarquer la présence de cinq grands groupes de profils d’expression (Figure 4 a). Brièvement, groupes A et E ont montré face à des comportements, en particulier le premier est caractérisé par une régulation générale négative des gènes ATL dans des échantillons de juvéniles, y compris les débuts berry, jeunes feuilles, vrilles, inflorescence et la plupart des stades bourgeon. En revanche, dans le même groupe A, matures échantillons tels que les baies à maturité et post-récolte flétrissement stades, tissus ligneux et stades de développement de semences ATL gènes ont montré une augmentation prédominante. Gènes en Cluster C étaient principalement diminué dans la plupart des échantillons, tandis que les gènes ATL du groupe D ont été souvent surexprimés aux derniers stades de développement de la baie. Enfin, cluster B ne montrent aucune variation significative dans les profils d’expression.
Une approche similaire a été appliquée à l’étude de l’expression de vigne ATL membres de la famille en réponse aux stress biotiques et abiotiques, à l’aide d’ensembles de données spécifique, construit à cet effet. Il existe une énorme quantité de données d’expression dérivant de microarray et expériences de RNA-seq de bases de données public access comme Gene Expression Omnibus (GEO) et ArrayExpress. Une fois collectées et idéalement normalisé, l’information a été exploitée pour encore mieux comprendre la fonction potentielle de ATLs dans la réponse des plantes aux stress. Analyser les profils d’expression de la vigne ATLs en réponse aux stress biotiques a révélé que 62 sur 96 transcriptions ont montré une modulation importante (log2 pli-changement (FC) > | 0,5 |) au moins deux conditions, avec un taux de fausse découverte (FDR) < () 0,05 Figure 4 b). Le nombre atteint 81 ne considérant que le seuil FDR à une seule condition. Ces résultats suggèrent fortement une participation directe de la famille de gènes ATL dans la réponse aux agents pathogènes aussi dans la vigne. En particulier, un groupe de 12 gènes (VviATL3-27-54b-55-90-97-123-144-148-149-156) ont fortement augmenté en réponse à la plupart des agents pathogènes, y compris biotrophe et champignons nécrotrophe et herbivores et donc, méritent l’attention des plus fonctionnelles analyses.
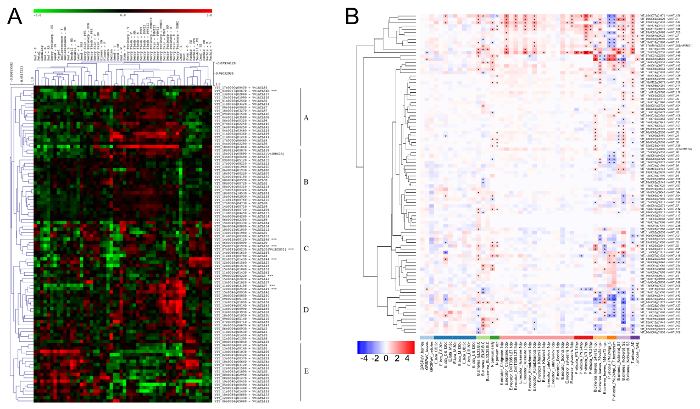
Figure 4 : expression des gènes ATL classification hiérarchique à grapevine Atlas et vigne biotiques liés au stress DataSet. (A) l’expression journal transformé valeurs des gènes ATL vigne dans la vigne Atlas12 ont été utilisées pour l’analyse de groupement hiérarchique basée sur distance métrique de Pearson. La couleur échelle représente plus élevé (rouge) ou bas (vert) expression niveaux en ce qui concerne l’abondance de transcription médian de chaque gène dans l’ensemble de tous les échantillons. Lettres de A à E du côté droit indiquent les différents clusters identifiés.AB : après l’éclatement ; B: rafale ; bourgeon-w : bourgeon d’hiver ; F: floraison ; FB : début de la floraison ; FS : nouaison ; G : vert ; MR : mi-maturation ; PFS : après nouaison ; PHWI-II-III : après récolte, dépérissement 1, 2 et 3 mois ; R : maturation ; S: sénescentes ; tige-w : tige ligneuse ; V : véraison ; WD : bien développées ; Y: le jeune. Échelle (B) la couleur représente une augmentation (rouge) ou diminue (bleu) pli changements vigne ATL d’expression de gène dans les échantillons infectés par rapport aux témoins pour chaque condition. Les astérisques indiquent l’expression différentielle importante (FDR < 0,05) de chaque ATL dans les conditions correspondantes. Reproduit de Ariani et al. , sous une licence Creative Commons Attribution 4.0 International42. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
Supplémentaire tableau 1 : candidats gènes ATL à l’épissage alternatif. (un) ATL ID gène selon la prédiction de raisin gène V1 et l’annotation, (b), ATL gène ID selon la V2 raisin gène prédiction et annotation43, (c) nombre de présumés ATL épissage variantes, (d) informations sur la séquence de chaque variante ATL putatif de codage. S’il vous plaît cliquez ici pour télécharger ce fichier.
Supplémentaires Tableau 2 : S’il vous plaît cliquez ici pour télécharger ce fichier.
Fichier supplémentaire 1 : S’il vous plaît cliquez ici pour télécharger ce fichier.
Discussion
Dans l’ère de la génomique, de nombreuses familles de gènes ont été profondément caractérisés chez plusieurs espèces de plantes. Cette information est préliminaire à des études fonctionnelles et fournissent un cadre pour étudier davantage le rôle des différents membres d’une famille. Dans ce contexte, il y a également un besoin pour un système de nomenclature permettant d’identifier de manière unique chaque membre d’une famille, en évitant la redondance et les confusions qui peuvent survenir lorsque les noms sont attribués indépendamment de différents gènes par différents groupes de recherche.
Après un examen réfléchi, la communauté scientifique de la vigne a accepté de gènes de vigne de nom dans une famille basée sur les similitudes avec les gènes de l’Arabidopsis et établi une série de règles qui doivent être appliquées pour décrire de nouvelles familles de gènes dans la vigne, essentiellement à partir de la comparaison phylogénétique des séquences de nucléotides entre vigne et Arabidopsis membres de la famille8. Par conséquent, seuls gènes qui se trouvent déjà annotés et nommés correctement chez Arabidopsis peuvent être utilisés dans la nomenclature de la vigne. La procédure appliquée pour l’identification de vigne ATL orthologues chez Arabidopsis décrit ici a été réalisée par conséquent uniquement pour combler le besoin d’assigner la nomenclature famille de gène de vigne correct. Néanmoins, pour les autres espèces de plantes, approches alternatives pourraient être une option. Par exemple, niveau pouvait être déduite en utilisant un hits BLAST bidirectionnel (BBH), où les orthologues sont définis sous forme de paires de gènes chez les deux espèces qui sont plus proches (c'est-à-direavec le meilleur score de l’alignement) les uns que pour n’importe quel autre gène dans l’autre espèces,44. Toutefois, cette méthode pourrait manquer plusieurs orthologues dans le cas de taux élevé de la duplication de gènes, tels que dans les plantes et les animaux45. En outre, dans le cas de gènes codant ATL, BBH peut récupérer des gènes manquent la structure précise de ATL-type anneau-H2 (y compris les résidus de proline) ou des gènes qui ne sont pas annotées et nommés comme ATLs dans Arabidopsis. Bien que du point de vue évolutif, cette recherche peut être pertinente, la récupération des orthologues qui ne sont pas annotés n'aurait pas répondu à la portée de vigne ATL gène familiale annotation et nomenclature et orthologues qui ne sont pas annotées comme ATLs ne peut servir à des membres de la famille nom vigne. Une autre possibilité consiste à déduire le niveau issu des acides aminés au lieu des séquences de nucléotides à l’aide de InParanoid46, ou le plus récent de 2 Hieranoid47, quoique ces flux de travail n’est pas expressément recommandés par la communauté scientifique.
Meta-analyse de l’expression, qui peut être définie comme une approche systématique pour étudier et combiner différents référentiels de données accessibles au public des données d’expression, permet en soulignant partagés et différents mécanismes moléculaires dans une variété de conditions. Ainsi, l’intégration des informations d’expression de gène de multiples expériences de transcriptomique à grande échelle peut améliorer la caractérisation d’une famille de gènes, en définissant les profils d’expression des membres de la famille à travers des expériences, minimisant ainsi la impact des facteurs propres à expérience et soutenir une hypothèse plus robuste de la fonction des gènes putatifs dans des procédés particuliers. Cependant, l’utilisation de données microarray nécessite l’intégration de données d’expression obtenues avec différentes plates-formes, compte tenu de leurs propres limites. Par exemple, dans la plateforme microarray Nimblegen de vigne, une proportion importante des probesets pour les gènes correspondants représentées sur le tableau (~ 13 000 gènes) ont potentiellement hybridation croisée questions48. Dans le cas de la famille ATL vigne, 15 gènes peuvent être affectées par ce phénomène. Néanmoins, comme nous l’avons par Cramer et al. 48, la croix-identification des proches très similaires de gène par la même sonde pourrait fournir des informations intéressantes concernant l’expression, dans des conditions particulières, non seulement d’un seul gène, mais de deux à plus de gènes partage séquence très similitudes et donc potentiellement partage les objectifs et fonctions. Un autre problème potentiel lié aux ensembles de données microarray est la limite de détection d’expression des plates-formes de microarray, qui ne sont pas très sensibles. Pour résoudre les deux préoccupations, i.e., hybridation croisée et de signal, une solution possible consisterait à envisager seulement RNAseq expression datasets. Toutefois, la méta-analyse des données de RNAseq de très grands ensembles de données de plusieurs études différentes peut devenir très fastidieux et peut nécessiter beaucoup de ressources de calcul et d’une expertise de haut.
Bien que l’approche présentée ici vise à être exhaustive, elle peut être certainement encore complétée par d’autres analyses. Tout d’abord, pour réaliser d’autres aperçus de l’évolution moléculaire et les relations phylogénétiques entre les membres de famille de gènes chez les plantes, l’analyse phylogénétique pourrait être étendu construire un arbre phylogénétique en utilisant des alignements de séquences multiples des membres de la famille de plusieurs espèces de plantes. Il est également possible de calculer le temps évolutionnaire des gènes de la famille, une estimation de leur taux de substitutions synonymes et non-synonymes au cours de l’évolution, en déterminant les valeurs Ks (nombre de substitutions synonymes par site synonyme dans une donnée période de temps) et Ka (nombre de substitutions non-synonymes par site non-synonymes dans la même période). Le ratio de Ka/Ks est utilisé pour déduire les mécanismes des évènements de duplication de gène après la divergence de leurs ancêtres. Une valeur de Ka/Ks = 1 suggère la sélection neutre, une valeur de Ka/Ks de < 1 suggère purifiant de sélection et une valeur de Ka/Ks de > 1 suggère une sélection positive,49. En outre, si l’analyse de la structure génétique révèle la présence d’introns, la caractérisation famille de gène peut être étendue à la détection des variantes d’épissage alternatif. En effet, selon une étude profonde de RNA-seq données provenant de différents tissus, des conditions de stress et de génotypes43, 21 (des 96) ATLs sont des candidats pour des événements d’épissage alternatif, avec le nombre potentiel d’isoformes allant de 2 à 16 pour ces ATLs (voir Supplémentaire tableau 1). Autres transcriptions produisent fréquemment des isoformes protéiques qui varient dans les séquences d’acides aminés et ces changements peuvent modifier les propriétés cellulaires des protéines et peuvent provoquer des altérations de la subtile modulation à la perte de la fonction du produit du gène. Pour cette raison, événements d’épissage alternatif ont été impliqués dans les fonctions végétales importantes, y compris la réponse au stress, la résistance aux maladies, la photosynthèse et floraison50,,51.Intégration de l’information du promoteur du gène ATL contenant putatif cis-éléments régulateurs52 ou trouver des molécules (par exemple, micro-ARN et ARN de long non codantes) potentiellement ciblage ATLs53 peut également être complété à révèlent des aperçus de système de la régulation moléculaire complexe et l’interaction de la vigne ATLs.
En conclusion, le choix des analyses à effectuer ainsi que les procédures à appliquer pour caractériser une nouvelle famille de gènes dans une espèce de plante sont alimentées principalement par les règles de la communauté scientifique, ainsi que par l’étendue de l’identification des gènes familiaux. Il est important de garder à l’esprit les étapes de l’enquête ultérieure possible, qui vont exploiter l’ensemble des informations, parmi qui comprend évolution des gènes chez les espèces végétales, description de structure du génome ou candidats fiables pour la sélection dans le fonctionnel études.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Le travail a été soutenu par l’Université de Vérone, dans le cadre de Joint projet 2014 (caractérisation de la famille de gènes ATL dans la vigne et de son implication dans la résistance au Plasmopara viticola).
matériels
| Name | Company | Catalog Number | Comments |
| Personal computer | |||
| Basic Local Alignment Search Tool (BLAST) | https://blast.ncbi.nlm.nih.gov/Blast.cgi | ||
| Molecular Evolutionary Genetics Analysis (MEGA) | http://www.megasoftware.net/ | ||
| Motif-based sequence analysis tools (MEME) | http://meme-suite.org/ | ||
| Geneious | Biomatters Limited | http://www.geneious.com/ | |
| ProtParam Tool | http://web.expasy.org/protparam/ | ||
| ngLOC | http://genome.unmc.edu/ngLOC/index.html | ||
| TargetP v1.1 Server | http://www.cbs.dtu.dk/services/TargetP/ | ||
| Protein Prowler | http://bioinf.scmb.uq.edu.au:8080/pprowler_webapp_1-2/ | ||
| MUsite | http://musite.sourceforge.net/ | ||
| Pfam | http://pfam.xfam.org/ | ||
| TMHMM Server v. 2.0 | http://www.cbs.dtu.dk/services/TMHMM/ | ||
| ProtScale | http://web.expasy.org/protscale/ | ||
| Grape Genome Database (CRIBI) | http://genomes.cribi.unipd.it/grape/ | ||
| PhenoGram | http://visualization.ritchielab.psu.edu/phenograms/plot | ||
| MCScanX | http://chibba.pgml.uga.edu/mcscan2/ | ||
| Interactive Tree Of Life (iTOL) | http://itol.embl.de/ | ||
| UniProt | http://www.uniprot.org/ | ||
| Phylogeny.fr | http://www.phylogeny.fr/index.cgi | ||
| MUSCLE | http://www.ebi.ac.uk/Tools/msa/muscle/ | ||
| Gblocks Server | http://molevol.cmima.csic.es/castresana/Gblocks_server.html | ||
| Vitis vinifera cv. Corvina gene expression Atlas datamatrix | https://www.researchgate.net/publication/273383414_54sample_ datamatrix_geneIDs_Fasoli2012 | ||
| Multi Experiment Viewer (MeV) | http://mev.tm4.org/#/welcome | ||
| Sequence Read Archive (SRA) | https://www.ncbi.nlm.nih.gov/sra | ||
| R | https://www.r-project.org/ | ||
| EMBOSS Needle (EMBL-EBI) | http://www.ebi.ac.uk/Tools/psa/emboss_needle/ |
Références
- Jaillon, O., et al. The grapevine genome sequence suggests ancestral hexaploidization in major angiosperm phyla. Nature. 449 (7161), 463-467 (2007).
- Adam-Blondon, A. -F., et al. Genetics, Genomics, and Breeding of Grapes. , Science Publishers. 211-234 (2011).
- Chen, L., Hellmann, H. Plant E3 Ligases: Flexible Enzymes in a Sessile World. Mol. Plant. 6 (5), 1388-1404 (2013).
- Vierstra, R. D. The ubiquitin-26S proteasome system at the nexus of plant biology. Nat. Rev. Mol. Cell Biol. 10 (6), 385-397 (2009).
- Serrano, M., Parra, S., Alcaraz, L. D., Guzmán, P. The ATL Gene Family from Arabidopsis thaliana and Oryza sativa Comprises a Large Number of Putative Ubiquitin Ligases of the RING-H2 Type. J. Mol. Evol. 62 (4), 434-445 (2006).
- Aguilar-Hernández, V., Aguilar-Henonin, L., Guzmán, P. Diversity in the Architecture of ATLs, a Family of Plant Ubiquitin-Ligases, Leads to Recognition and Targeting of Substrates in Different Cellular Environments. PLoS One. 6 (8), e23934(2011).
- Guzmán, P. The prolific ATL family of RING-H2 ubiquitin ligases. Plant Signal Behav. 7 (8), 1014-1021 (2012).
- Grimplet, J., et al. The grapevine gene nomenclature system. BMC Genomics. 15, 1077(2014).
- Prince, V. E., Pickett, F. B. Splitting pairs: the diverging fates of duplicated genes. Nat. Rev. Genet. 3 (11), 827-837 (2002).
- Magadum, S., Nerjee, U., Murugan, P., Gangapur, D., Ravikesavan, R. Gene duplication as a major force in evolution. J. Gen. 92 (1), 155-161 (2013).
- Wang, N. Patterns of Gene Duplication and Their Contribution to Expansion of Gene Families in Grapevine. Plant Mol. Biol. Rep. 31 (4), 852-861 (2013).
- Fasoli, M. The Grapevine Expression Atlas Reveals a Deep Transcriptome Shift Driving the Entire Plant into a Maturation Program. Plant Cell. 24 (9), 3489-3505 (2012).
- BLAST. BLAST2.6.0. , Available from: https://blast.ncbi.nlm.nih.gov/Blast.cgi (2016).
- MEGA. MEGA7.0.25 build 7170412. , Available from: http://www.megasoftware.net/ (2017).
- MEME. MEME Suite Version 4.11.4. , Available from: http://meme-suite.org/ (2017).
- ProtParam. ExPASy Server. , Available from: http://web.expasy.org/protparam/ (2005).
- ngLOC v1.0. , Available from: http://genome.unmc.edu/ngLOC/index.html (2007).
- TargetP v1.1 Server. , Available from: http://www.cbs.dtu.dk/services/TargetP/ (2000).
- Prowler v1.2. , Available from: http://bioinf.scmb.uq.edu.au:8080/pprowler_webapp_1-2/ (2005).
- MuSite v1.0. , Available from: http://musite.sourceforge.net/ (2010).
- Pfam. Pfam version 31.0. , Available from: http://pfam.xfam.org/ (2016).
- TMHMM v2.0c. , Available from: http://www.cbs.dtu.dk/services/TMHMM/ (2007).
- ExPASy. ProtScale. , Available from: http://web.expasy.org/protscale/ (2005).
- CRIBI. Grape genome database. , Available from: http://genomes.cribi.unipd.it/grape/ (2012).
- PhenoGram. , Available from: http://visualization.ritchielab.psu.edu/phenograms/plot (2012).
- ScanX v0.8. , Available from: http://chibba.pgml.uga.edu/mcscan2/ (2013).
- Interactive Tree Of Life (iTOL). Version3.5.3. , Available from: http://itol.embl.de/ (2016).
- UniProt. , Available from: http://www.uniprot.org/ (2016).
- Phylogeny.fr. , Available from: http://www.phylogeny.fr/index.cgi (2008).
- MUSCLE. , Available from: http://www.ebi.ac.uk/Tools/msa/muscle/ (2017).
- Gblocks Server. Version 0.91b. , Available from: http://molevol.cmima.csic.es/castresana/Gblocks_server.html (2002).
- Vitis vinifera cv. Corvina gene expression Atlas. , Available from: https://www.researchgate.net/publication/273383414_54sample_datamatrix_geneIDs_Fasoli2012 (2015).
- Multiple Experiment Viewer (MeV). Version 4.8.1. , Available from: http://mev.tm4.org/ (2017).
- Sequence Read Archive (SRA). , Available from: https://www.ncbi.nlm.nih.gov/sra (2017).
- Bolger, A. M., Lohse, M., Usadel, B. Trimmomatic: a flexible trimmer for Illumina sequence data. Bioinformatics. 30 (15), 2114-2120 (2014).
- Langmead, B., Salzberg, S. L. Fast gapped-read alignment with Bowtie 2. Nat Meth. 9 (4), 357-359 (2012).
- Anders, S., Pyl, P. T., Huber, W. HTSeq-a Python framework to work with high-throughput sequencing data. Bioinformatics. 31 (2), 166-169 (2015).
- R. Version 3.4.1. , Available from: https://www.r-project.org/ (2017).
- Ritchie, M. E. limma powers differential expression analyses for RNA-sequencing and microarray studies. Nucleic Acids Res. 43 (7), e47(2015).
- Love, M. I., Huber, W., Anders, S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2. Genome Biology. 15 (12), 550(2014).
- EMBL-EBI. EMBOSS Needle. , Available from: http://www.ebi.ac.uk/Tools/psa/emboss_needle/ (2017).
- Ariani, P. Genome-wide characterisation and expression profile of the grapevine ATL ubiquitin ligase family reveal biotic and abiotic stress-responsive and development-related members. Sci. Rep. 6, 38260(2016).
- Vitulo, N., et al. A deep survey of alternative splicing in grape reveals changes in the splicing machinery related to tissue, stress condition and genotype. BMC Plant Biol. 14 (1), 99(2014).
- Overbeek, R., Fonstein, M., D'Souza, M., Pusch, G. D., Maltsev, N. The use of gene clusters to infer functional coupling. Proc. Natl. Acad. Sci. USA. 96 (6), 2896-2901 (1999).
- Dalquen, D. A., Dessimoz, C. Bidirectional Best Hits Miss Many Orthologs in Duplication-Rich Clades such as Plants and Animals. Genome Biol. Evol. 5 (10), 1800-1806 (2013).
- Remm, M., Storm, C. E. V., Sonnhammer, E. L. L. Automatic clustering of orthologs and in-paralogs from pairwise species comparisons1. J. Mol. Biol. 314 (5), 1041-1052 (2001).
- Kaduk, M., Sonnhammer, E. Improved orthology inference with Hieranoid 2. Bioinformatics. 33 (8), (2017).
- Cramer, G. R., et al. Transcriptomic analysis of the late stages of grapevine (Vitis vinifera cv. Cabernet Sauvignon) berry ripening reveals significant induction of ethylene signaling and flavor pathways in the skin. BMC Plant Biol. 14, 370(2014).
- Juretic, N., Hoen, D. R., Huynh, M. L., Harrison, P. M., Bureau, T. E. The evolutionary fate of MULE-mediated duplications of host gene fragments in rice. Genome Res. 15 (9), 1292-1297 (2005).
- Filichkin, S. A. Genome-wide mapping of alternative splicing in Arabidopsis thaliana. Genome Res. 20 (1), 45-58 (2010).
- Quesada, V., Macknight, R., Dean, C., Simpson, G. G. Autoregulation of FCA pre-mRNA processing controls Arabidopsis flowering time. EMBO J. 22 (12), 3142-3152 (2003).
- Wong, D. C. J., Gutierrez, R. L., Gambetta, G. A., Castellarin, S. D. Genome-wide analysis of cis-regulatory element structure and discovery of motif-driven gene co-expression networks in grapevine. DNA Res. 24 (3), 311-326 (2017).
- Wong, D. C. J., Matus, J. T. Constructing Integrated Networks for Identifying New Secondary Metabolic Pathway Regulators in Grapevine: Recent Applications and Future Opportunities. Front. Plant Sci. 8, 505(2017).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon