Method Article
Fluxo de trabalho abrangente para a identificação de todo o genoma e Meta-análise da expressão da ATL E3 ubiquitina Ligase Gene família em videira
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
Este artigo descreve o procedimento para a identificação e caracterização de uma família de gene em videira aplicada à família de Arabidopsis Tóxicos em Levadura (ATL) E3 ubiquitina ligases.
Resumo
Classificação e nomenclatura dos genes em uma família podem contribuir de forma significativa para a descrição da diversidade das proteínas codificadas e a previsão da família funções com base em diversas características, tais como a presença de motivos de sequência ou de particular sites para modificação pós-traducional e o perfil de expressão dos membros da família em diferentes condições. Este trabalho descreve um protocolo detalhado para a caracterização de família do gene. Aqui, o procedimento é aplicado para a caracterização da Arabidopsis Tóxicos em Levadura (ATL) E3 ubiquitina ligase família na videira. Os métodos incluem a identificação de todo o genoma dos membros da família, a caracterização da localização do gene, estrutura e duplicação, a análise dos motivos da proteína conservada, a previsão dos sites de localização e fosforilação de proteínas, bem como Gene expressão perfila através da família em diferentes conjuntos de dados. Tal procedimento, que poderia ser alargado a novas análises dependendo fins experimentais, poderia ser aplicado a qualquer família de gene em quaisquer espécies de plantas para as quais dados genomic estão disponíveis, e fornece informações valiosas para identificar candidatos interessantes para estudos funcionais, dando insights sobre os mecanismos moleculares de adaptação da planta para seu ambiente.
Introdução
Durante a última década, muita pesquisa realizada na genômica de videira. Videira é uma reconhecida cultura economicamente relevante, que se tornou um modelo para a investigação sobre o desenvolvimento do fruto e sobre as respostas de plantas lenhosas a estresses bióticos e abióticos. Neste contexto, o lançamento do genoma Vitis vinifera CV. PN40024 em 2007-1 e sua versão atualizada em 20112 levou a uma rápida acumulação de dados "Omics"-escala e a uma explosão de estudos de alto rendimento. Baseado nos dados de sequência publicada, a análise abrangente de uma família de determinado gene (geralmente composta por proteínas partilha motivos conservados, semelhanças estruturais e/ou funcionais e as relações evolutivas), agora pode ser realizada para descobrir sua perfis de expressão genética, evolução e funções moleculares. Essas análises podem contribuir para a compreensão de como as famílias gene controlam processos fisiológicos em um nível de todo o genoma.
Muitos aspectos do ciclo de vida da planta são regulados pela mediada por ubiquitina de degradação de proteínas chaves, que requerem um volume de negócios de aperfeiçoá-lo para assegurar o regulares processos celulares. Importantes componentes do processo de degradação mediada por ubiquitina são os E3 ubiquitina ligases, que são responsáveis pela flexibilidade do sistema, graças ao recrutamento de alvos específicos3. Por conseguinte, estas enzimas representam uma família enorme de gene, com cerca de 1.400 E3 ligase-codificação genes previstos em Arabidopsis thaliana genoma4, cada ligase de ubiquitina E3 atuando para o ubiquitination de proteínas alvo específico. Apesar da importância da ubiquitination substrato específico no Regulamento celular em plantas, pouco se sabe sobre como o caminho da ubiquitination é regulamentado e proteínas alvo foram identificadas somente em alguns casos. O deciframento de tais mecanismos de especificidade e regulamentação depende primeiramente a identificação e caracterização das diferentes componentes do sistema, em particular as ligases E3. Entre ubiquitina ligases, subfamília ATL é caracterizada por 91 Membros identificados em a. thaliana , exibindo um anel-H2 dedo domínio5,6, alguns deles desempenhando um papel na defesa e hormônio respostas7.
O primeiro passo crucial para definir os membros de uma família nova do gene é a definição precisa das características familiares, tais como motivos de consenso, domínios-chave e características da sequência da proteína. Com efeito, a recuperação confiável de todos os membros da família gene com base na análise de explosão requer algumas características de sequência obrigatória, em domínios de particular da proteína responsáveis pela proteína função/atividade, servindo como assinatura de proteína. Isto pode ser facilitado pela caracterização anterior da mesma família gene em outras espécies de plantas ou alcançado através da análise de diferentes genes presumidamente pertencentes à família mesma em diferentes espécies de plantas, para isolar as sequências comuns. Os membros da família podem então ser individualmente nomeados seguindo regras comuns colonizadas por consórcios internacionais para uma espécie de determinada planta. Em videira, por exemplo, tal procedimento é submetido às recomendações do Comité de nomenclatura super para anotação de Gene de uva (sNCGGa), que estabelece a construção de uma árvore filogenética, incluindo V. vinifera e a. thaliana membros da família para permitir a anotação de gene gene baseiam em sequências de nucleotídeos8.
Localização de cromossomo de membros da família e pesquisa de duplicação do gene permitir destacando a presença de genes duplicados do inteiro-genoma ou em tandem. Essa informação aparece útil para desvendar as funções do gene putativo, desde que pode mostrar a redundância funcional ou revelar situações diferentes, ou seja, não-functionalization, neo-functionalization ou functionalization sub9. Ambos os neo - e sub - functionalization é eventos importantes que criam novidade genética, fornecendo novos componentes celulares para adaptação da planta às mudanças ambientes10. Em particular, as duplicações de genes ancestrais e produção de novos genes eram muito frequentes durante a evolução do genoma de videira e recém-formado genes originários de duplicações em tandem e proximais em videira eram mais propensos a produzir novos funções de11.
Outro fator chave em decifrar a função dos genes familiares é o perfil de transcriptomic. A disponibilidade de bases de dados públicas, dando acesso a uma enorme quantidade de dados de transcriptomic pode ser explorada, assim, para atribuir funções putativos para membros da família gene usando análises de expressão em larga escala em silico . Com efeito, a expressão peculiar de alguns genes em órgãos da planta específica ou em resposta a certas tensões pode dar algumas dicas sobre os putativos papéis das proteínas correspondentes em condições definidas e dar suporte a hipóteses sobre possíveis sub functionalization dos genes duplicados para responder aos desafios diferentes. Para esse efeito, é importante considerar os vários conjuntos de dados: estas podem ser gene já disponível matrizes de expressão, tais como o atlas transcriptomic de todo o genoma da videira órgãos e estádios de desenvolvimento,12, ou podem ser construídas ad hoc por recuperação de conjuntos de dados transcriptomic para as espécies de planta particular sujeitados a tensões definidas. Além disso, uma abordagem simples, usando duas matrizes, uma com dados de similaridade emparelhadas e outro com os coeficientes emparelhadas expressão co podem ser aplicados para avaliar as relações entre padrões de semelhança e a expressão de sequência dentro de uma família de gene.
O objetivo deste trabalho é apresentar uma abordagem global, definindo a estrutura do gene, motivos de proteína conservada, localização cromossômica, duplicações do gene e padrões de expressão, como também a previsão de sites localização e fosforilação da proteína, para atingir uma caracterização exaustiva de uma família de genes em plantas. Uma abordagem abrangente é aplicada aqui, para a caracterização da família ATL E3 ubiquitina ligase em videira. De acordo com o papel emergente de membros da subfamília ATL na regulação de processos celulares chave7, este trabalho pode também auxiliar a identificação dos fortes candidatos para estudos funcionais e eventualmente a desvendar os mecanismos moleculares que regem o adaptação desta cultura importante para seu ambiente.
Protocolo
1. identificação da família do Gene ATL putativo membro (s)
- Versão web de PSI-BLAST
- Abra a página web de explosão13 e clique na seção de explosão de proteína.
- No campo "Sequência de consulta de Enter", digite a sequência de aminoácidos da proteína (aqui VIT_05s0077g01970) que será usada como sonda para identificar outros membros da família.
Nota: Uma boa proteína representante deve ser usado (uma proteína mostrando todas as características importantes que caracterizam a família). - No campo "Choose conjunto de pesquisa", selecione o banco de dados "Proteína de referência" (refseq_protein) e o organismo de interesse (V. vinifera - taxid:29760).
- No campo "programa seleção", selecionar o algoritmo de PSI-BLAST e clique no botão para executar a análise BLAST.
Nota: Clicando em "parâmetros de algoritmo" é possível ajustar alguns parâmetros avançados (sequências de destino Max, matriz de Scoring, limiar de PSI-BLAST, etc.). - A primeira explosão redonda recupera todas as sequências exibindo partidas relevantes com a consulta (e-valor acima do limiar selecionado - por padrão 0.005; 0,001 neste experimento). Desmarque todas as entradas, que claramente não pertencem à família sob exame clicando sobre o carrapato na coluna "Selecione para PSI-BLAST" e executar a segunda iteração do PSI-BLAST clicando no botão de explosão como na etapa 1.1.4.
- Recentemente identificadas sequências são destacadas em amarelo. Desmarcar o hits obtido claramente errado e descobrir ainda mais iterações, conforme descrito na etapa 1.1.5.
- Continue com iterações até que o algoritmo não encontra qualquer entrada relevante ou atingir convergência (não há novas entradas são encontradas). Baixe a lista de membros da família gene putativo para mais análises. Inspecione visualmente os sucessos obtidos em cada iteração para evitar a presença de falsos positivos.
- Versão autônoma de PSI-BLAST
- Baixe a versão autônoma do explosão clicando no botão "baixar explosão" na página inicial explosão13.
Nota: O software de explosão independente é uma versão de linha de comando de interface web descrito antes. Permite executar a pesquisa PSI-BLAST contra um banco de dados local ou remoto personalizado. Além disso, ele permite pesquisar com um pré-definido posição específica Pontuação Matrix (PSSM).
- Baixe a versão autônoma do explosão clicando no botão "baixar explosão" na página inicial explosão13.
2. manual inspeção dos membros da família PSI-BLAST-identificado
- Alinhamento múltiplo
- Recolher as sequências de ácido aminoácidas anteriormente identificadas em um arquivo no formato FASTA e enviá-lo para o MEGA software14 para prosseguir com o alinhamento múltiplo.
- Abra o software MEGA, clique no botão "Alinhar", clique em "Editar/construir alinhamento", clique em "Criar um novo alinhamento", clique em "Proteína".
- Clique em "Editar" no menu de alinhamento e "Inserir sequência de arquivo". Procure pelo arquivo FASTA criado antes e confirme o upload de todas as sequências pesquisadas.
- Clique em "Alinhamento" no menu de alinhamento e "Alinhar pelo músculo". Usar parâmetros padrão, clique o botão "Calcular" e aguarde a conclusão do alinhamento múltiplo.
- Inspecione visualmente o alinhamento múltiplo para excluir incorretamente previu os membros da família. O canonical Pedagoga (13 x) PxCxHxxHxxCxxxW (7x) CxxCW motivo, (em especial a presença do resíduo prolina antes o terceiro cisteína), é a característica chave necessária para definir os membros da família ATL.
- Análise do logotipo específico
- Apresenta a lista definitiva dos membros da família (96 sequências de videira cumpram os requisitos para ser considerado ATL) o Em múltiplas por motivo Elicitação (MEME)15 para definir motivos conservados toda a família.
- MEME página inicial, clique no botão "MEME" e complete o "submissão formulário de dados" com informações específicas sobre a família de interesse.
- Use a análise MEME para confirmar a presença dos dois temas esperados dentro de videira ATL os membros da família, ou seja, o anel-H2 e os motivos GLD.
- Como alternativa, execute os passos 2.1 e 2.2 simultaneamente usando a suíte de softwares de Bioinformática (ver Tabela de materiais).
- Upload de arquivo FASTA (consulte a etapa 2.1.1) para a suíte. Selecione "Arquivo" no menu, em seguida, "Importar" e clique em "do arquivo". Procure o arquivo FASTA e clique em "Abrir".
- Selecione todas as sequências de importados na lista e clique no botão "Alinhar/montar" na barra de ferramentas, clique em "Pairwise alinhamento múltiplo". Selecione "Alinhamento muscular" e clique em "Okey" para iniciar o alinhamento usando parâmetros padrão.
- Para visualizar o logotipo do alinhamento, clique em "Gráficos" → "opções" e selecione "Logotipo da sequência".
3. análise dos parâmetros físicos de proteína e domínios
- Como a definição dos diferentes parâmetros físicos dos membros da família pesquisados é importante ter uma descrição abrangente da família, apresentar a lista de membros da família de ferramentas específicas da web.
- Ponto isoelétrico (pI) e do peso molecular (kDa), use a ferramenta de ProtParam16 no site Expasy com parâmetros padrão.
- Para localização Subcellular da proteína, usar ferramentas diferentes para obter uma previsão mais confiável como ngLOC v 1.017 com configurações padrão, targetP v 1.118 com configurações padrão e proteína gatuno Localização subcellular v 1.219 com um corte de probabilidade de 0,5. Para sites de fosforilação, use o MUsite v 1.0 web ferramenta20 com parâmetros padrão.
- Investiga os domínios da proteína adicional em membros da família.
- Abra o banco de dados de Pfam Web page21, selecione a ferramenta "Busca", enviar sequências de proteínas na caixa de consulta e clique em "Ir" para executar a análise.
Nota: Cada sequência da proteína é analisada individualmente. Um valor de 1.0, na configuração padrão e permite discriminar entre sucessos significativos e não-significativas. - Abra o servidor TMHMM22 do centro para análise de sequências biológicas investigar a presença de regiões transmembranares putativos.
- Abra o banco de dados de Pfam Web page21, selecione a ferramenta "Busca", enviar sequências de proteínas na caixa de consulta e clique em "Ir" para executar a análise.
4. cromossômica distribuição, duplicações e organização intron-Exon
- Mapear os membros da família sobre os cromossomos com base na informação obtida do site Centro de biotecnologia do Grapevine genoma CRIBI24ATL.
- Procure o PhenoGram Web site Home Page25. Gravar o "arquivo de entrada" como um arquivo de texto delimitado por tabulação com as características específicas dos genes a serem mapeados a cromossomos, de acordo com as diretrizes exaustivas e exemplos sobre a compilação do arquivo fornecido, seguindo o caminho "Phenogram" → " Documentação"→"Opções"→"Input file".
- Escreva o "título" do trabalho. Selecione o genoma a ser desenhada. Para genomas não implementadas no software, tais como o genoma da videira, selecione "outros" no menu drop-down. Gravar o arquivo de genoma de acordo com as orientações e exemplos fornecidos, seguindo o caminho "Phenogram" → "Documentação" → "Opções" → "Genoma" e enviá-lo.
- Usar parâmetros de padrão de "Espaçamento de fenótipo", "Fenótipo cor", "Formato de imagem" ou selecionadas alternativas nos respectivos menus e clique em "Plot" obter a visualização dos genes em cromossomos.
- Defina o estado de duplicação dos membros da família usando o software de MCScanX26.
- Baixe e descompacte uma cópia do MCscanX em uma máquina local, executando o comando linhas 1 (arquivo complementar 1). Entrar na pasta MCscanX e criar os executáveis necessários executando as linhas de comando 2 (arquivo complementar 1).
Nota: Instalação de MCscanX é conhecida por falhar em algumas máquinas de Linux de 64 bits devido a um problema em relação a função chdir. Se uma mensagem de erro é retornada relacionada a essa função após a fazer execução de comando, as linhas de comando 3 (arquivo complementar 1) deve ser executadas e o comando "make" deve ser tentado depois. - Baixe as proteínas V. vinifera e o arquivo de anotação, executando o comando linhas 4 (arquivo complementar 1).
Nota: A videira anotação arquivo precisa ser descompactado e o gato de informação único de cromossomos em um único arquivo executando as linhas de comando 5 (arquivo complementar 1). - Executar um "todos contra todos" blastp de busca usando o arquivo de proteína de V. vinifera como tanto a consulta e o assunto.
- Crie um banco de dados pesquisáveis explosão usando o arquivo proteína V. vinifera executando o comando linhas 6 (arquivo complementar 1). Executar a busca de blastp usando o arquivo V. vinifera proteínas como uma consulta no banco de dados criado anteriormente, executando o comando linhas 7 (arquivo complementar 1).
- Converta o arquivo de anotação em um formato adequado para MCScanX. Execute o comando linhas 8 (arquivo complementar 1) para baixar a parseMSCanXgff.pl de script perl personalizados. Realizar a análise de linhas de comando 9 (arquivo complementar 1) em execução.
Nota: Um vitis.gff de arquivo é gerado que contém as coordenadas do gene no seguinte formato:
SP # gene posição terminando a posição inicial
onde o "sp" é um código de duas letras para a espécie (Vv para videira) Considerando que o "#" é o nome do cadafalso. Note que o script perl personalizado fornecido é adequado para a maioria de conversão, embora algumas modificações de código podem ser necessária em alguns casos específicos, devido à diversidade das informações fornecidas no arquivo de anotação disponível. - Lançamento MCScanX executando o comando linhas 10 (arquivo complementar 1).
Nota: O "vitis" é o prefixo de anotação e o arquivo de saída de explosão. Isto representa um requisito obrigatório para o software executar. - Analise os resultados de MCScanX. MCScanX produz um arquivo de texto "vitis.collinearity", que contém blocos colineares. Esse arquivo pode ser inspecionado por qualquer editor de texto (ver exemplo 1 complementar 1 arquivode saída).
Nota: Um diretório "mcscaxOutput.html" é gerado, que contém os arquivos html com alinhamentos múltiplos de blocos colineares contra cada cromossomo de referência. Esses arquivos podem ser olhados através de um navegador da web. - Classifica os genes parólogo, baseados em suas posições relativas em cromossomos 11 (arquivo complementar 1) de linhas de comando em execução.
Nota: Classificação de gene parólogo é descrita no Quadro suplementar II. O arquivo de saída gerado "vitis.gene_type" contém todas as informações de origem com um formato delimitado por tabulação de simples. - Realizar análise de enriquecimento para avaliar se a família do gene originou-se predominantemente através de um mecanismo específico, executando o comando linhas 12 (arquivo complementar 1).
Nota: O arquivo "vitis.gene_type" é gerado no passo 4.2.8, Considerando que o arquivo "gene_family_file" representa um arquivo de texto uma linha em que o nome da família (por exemplo, ATL_genes) é seguido por locus nomes para todos os genes pertencentes à família separados por uma tabulação. O teste estatístico aplicado para o enriquecimento é um teste exato de Fisher e o p-valores de diferentes origens são armazenadas no arquivo "OutputFile".
- Baixe e descompacte uma cópia do MCscanX em uma máquina local, executando o comando linhas 1 (arquivo complementar 1). Entrar na pasta MCscanX e criar os executáveis necessários executando as linhas de comando 2 (arquivo complementar 1).
- Visualize a organização intron-exon dos genes usando Interactive árvore da vida (iTOL)27, uma ferramenta on-line para a visualização, anotação e gestão de árvores filogenéticas.
- Carregar uma árvore filogenética na seção "Upload" do site iTOL. A árvore é construída de acordo com a seção 5 abaixo. Para cada gene de membro da família, recupere predição da estrutura genética de V1 anotação do genoma da videira (site CRIBI citado acima). Calcule o comprimento (em PA) de exões putativos intrões e regiões untranslated (UTRs).
- Use o dataset "Domínios da proteína" para visualização gráfica do padrão intron-exon.
5. nomenclatura e análise filogenética
- Analise as relações entre os membros da família ATL através da construção de uma árvore filogenética de alta qualidade e a definição de uma nomenclatura de família.
- Para uma família de gene de videira, siga as regras estabelecidas pela videira Comité de nomenclatura de Super8.
- Recupere sequências da . thaliana ATL, necessárias como referência para videira gene nomenclatura8, a partir de banco de dados o UniProt28 .
- Gravar um arquivo FASTA, incluindo todas as sequências de nucleotídeos de videira e membros da família a. thaliana gene a ser incluídos na análise filogenética. As sequências de nucleótidos permitam o máximo de variabilidade entre membros da família (em comparação com sequências de proteínas).
- Árvore filogenética
Nota: O uso do pipeline 29 Phylogeny.fr é recomendado para obter uma árvore filogenética de alta qualidade, mas não obrigatório.- Procure o homepage de Phylogeny.fr29e selecione o pipeline "Análise da filogenia".
Nota: "One Click" é adequado na maioria dos casos, mas se for necessário, é possível selecionar específicas configurações avançadas ("Advanced") ou até mesmo uma análise totalmente personalizada ("à la Carte"; Veja passo 5.2.5). - Escrever o nome"da análise", o upload do arquivo FASTA criado anteriormente (passo 5.2.1 e clique em "Submit" para executar a análise.
- Alternativamente, se o procedimento descrito acima (passos 5.2.1, 5.2.2) resulta em uma mensagem de erro, concluir cada etapa do gasoduto filogenia suíte individualmente, da seguinte maneira.
- O músculo software homepage30, carregar o arquivo FASTA na "Etapa 1", selecione "Pearson/FASTA" como "Formato de saída" em "Passo 2" e clique em "Enviar" no "Passo 3" para alinhar sequências de consulta.
- Clique em "Carregar arquivo de alinhamento" e salvar como arquivo FASTA para novas etapas.
- Processo o arquivo FASTA de alinhamento para eliminar mal alinhados posições usando a ferramenta de servidor Gblocks31. Carregar o arquivo FASTA de alinhamento, selecione "DNA" como "Tipo de sequência" e escolheu a opção (ões) de rigor que melhor se encaixa com a análise (por exemplo, para videira ATL gene familiar selecione todas as três opções propostas para "menos rigorosa seleção" porque de divergência de alta sequência). Clique em "Obter blocos" para executar a análise.
- Clique em "Alinhamento resultante" na parte inferior da página de saída e salvar os resultados como um novo arquivo FASTA.
- O homepage de Phylogeny.fr29, selecione "A la Carte" como pipeline de "Análise de filogenia". Em seguida, desmarque a opção "Alinhamento múltiplo" e "Curadoria de alinhamento". Clique em "Criar fluxo de trabalho", carregar o arquivo Gblocks-curadoria FASTA (etapa 5.2.5.4), com parâmetros de padrão em "Configurações", selecione "Bootstrapping procedimento" e clique em "Enviar" para executar a análise.
- Ramos de colapso mal suportado (ou seja, valores de bootstrap < 70%), clicando em "Colapso ramos" na seção "Select e ação" e baixar os resultados finais no formato de Brasilia para novas análises.
- Procure o homepage de Phylogeny.fr29e selecione o pipeline "Análise da filogenia".
- Atribua um nome de gene baseado a filogenia.
- Examine a árvore filogenética para avaliar a confiabilidade da estrutura de árvore por meio de upload para a suite iTOL citada acima (seção 4.3).
- Atribua manualmente um nome de gene para cada membro da família. No caso de um para um orthologues, atribuir a Arabidopsis-como o nome (por exemplo, AtATL3 → VviATL3). Diferenciar os genes de videira (dois ou mais) derivando de um homólogo Arabidopsis único com a mesma distância filogenética usando números, ou letras se o gene de Arabidopsis termina com um número (por exemplo, AtATL23 → VviATL23a, VviATL23b).
- No caso de orthologues um-para-muitos ou muitos-para-muitos, atribuir um novo nome de gene composto a Arabidopsis-como nome (aqui, "ATL") combinado com um número maior que o maior número já usado para V. vinifera e de Arabidopsis (ex., VviATL83).
- Complete a nomenclatura da família recém-definido decrescente da parte superior à parte inferior da árvore filogenética.
6. grapevine órgão e fase de criação de perfil de expressão
- Gere os dados úteis dados matriz contendo expressão para os membros da família.
- Baixe o V. vinifera CV Corvina gene expressão Atlas datamatrix de link distribuído no ResearchGate plataforma32. Este arquivo contém os valores de expressão de RMA normalizado a serem utilizados em etapas.
- Extrair os valores de expressão de cada gene familiar do Atlas datamatrix e escrever um "datamatrix de trabalho", contendo a mesma linha de cabeçalho, como o Atlas datamatrix. Salve o datamatrix"trabalho" como um arquivo de texto delimitado por tabulação.
- Efectuar a análise hierárquica de bi-agrupado usando software Multi Visualizador de experimento (MeV).
- Baixe e instale o software de MeV33.
- Carregar o datamatrix"trabalho" (passo 6.1.2) seguindo o caminho do "Arquivo" → "Carregar dados" → "Browse" e selecione o arquivo de texto. Selecione "matriz de cor única" e remover o carrapato de "Anotação de carga", quando uma anotação automática não é fornecida. Selecione o valor da expressão superior mais à esquerda da visualização da tabela de expressão e clique no botão "Load".
- Ajuste os dados aplicando a transformação de Log2 ("Ajustar dados" → "Transformações de Log" → "Log2 transformar") e normalização de Gene/linha ("Ajustar dados" → "Gene/linha ajustes" → "Median Center Gene/Row"). Defina o limite de escala apropriada ("Exibir" → "definir cor escala limites").
- Calcular o Clustering hierárquico seguindo o caminho de "Análise" → "Clustering" → "HCL".
7. expressão de criação de perfil em resposta a estresses bióticos e abióticos
- Repita a etapa 6.1 com o ID de adesão GSE Obtida de estudos investigando o estresse biótico e abiótico em grapevine e respectivas publicações. Por exemplo, experimentos, fornecendo o perfil de transcriptoma de bagas de videira infectada com o patógeno fungo Botrytis cinerea , usando o microarray NimbleGen uva todo-genoma podem ser visitados com GSE ID de GSE52586. Repita as etapas 6.1.1 e 6.1.2.
- Procurar o NCBI sequência lê arquivo34 com a SRA/BioProject ID (por exemplo, SRP055458 ou PRJNA275778 para experiências "sombreamento de flor de videira") e baixar tudo associado sequência primas leituras. RNA-seq datasets de muitos estudos diferentes são processadas usando um único pipeline para consistência.
- Brevemente, aparar leituras FASTQ sequência primas (single - e par-final) e filtro de qualidade com Trimmomatic35. Uso que um AVGQUAL e MINLEN filtro de 20 e 40, respectivamente e todos os parâmetros padrão.
- Índice do 12 X videira referência genoma1 usando Bowtie236. Baixe 12 X videira referência genoma (por exemplo, construir bowtie2) antes de executar o comando bowtie2 .
- Obter contagem matriz tabelas com htseq-Conde37 usando o arquivo videira V1 gene modelo anotação (GFF/GTF).
- Realizar análise de expressão (re-) gene diferencial em R38 com se39 bibliotecas para matrizes de RMA-normalizado e DESeq240 bibliotecas para contagem matriz tabelas obtidas medidas 7.1.1 e 7.2.1, respectivamente.
- Realizar uma comparação de "dois-grupo" padrão (isto é, o "tratamento" / "controle"). Certifique-se de que o projeto matriz/agrupamentos de condições "controla" e "tratamento" são especificados corretamente.
Nota: Um design típico para a análise de expressão diferencial de microarray (GSE52586) para comparar EL-33 bagas infectadas com Botrytis cinerea contra bagas (saudável) de controle na mesma fase de desenvolvimento com se executando as linhas de comando 13 é mostrada no arquivo complementar 1. Um projeto típico para análise de expressão diferencial de RNA-seq (SRP055458 ou PRJNA275778) para comparar flor (em 7 dias após a queda do tampão) sob tratamento de sombra contra o controle com DESeq2 executando o comando linhas 14 é mostrado na complementar arquivo 1 . - Obter as listas de genes diferencialmente expressos (DEG) em cada contraste, para se, use as funções lmFit(), seguido por eBayes()e então por topTable() funções, enquanto que para DESeq2, use o DESeqDataSetFromMatrix(), DESeq()e results() funções. Abaixo, um fluxo de trabalho típico a ser seguido.
- Para a análise da expressão diferencial de microarray, ver linhas de comando 15 (arquivo complementar 1). Para análise de expressão diferencial de RNA-seq ver linhas de comando 16 (arquivo complementar 1). Repita os passos acima para todos os outros contrastes com esquema diferente design apropriado (ver exemplos na etapa 7.3.1)
- Realizar uma comparação de "dois-grupo" padrão (isto é, o "tratamento" / "controle"). Certifique-se de que o projeto matriz/agrupamentos de condições "controla" e "tratamento" são especificados corretamente.
- Nas listas de DEGs gerados, extrair todas as linhas que não correspondem à adesão de ATL V1, reter colunas contendo a mudança de dobrar log2 (tratamento/controle) > | 0.5 | e ajustado p-valores (FDR) < 0,05 e mesclagem-los adequadamente em uma tabela de matriz, se um estudo cai "abióticos" ou colectâneas de "interação biótica/patógeno".
- Construa o heatmaps clusterizado hierárquica (colectâneas abióticas e bióticas) em R, usando as bibliotecas gplots.
Nota: Chamar a função heatmap.2 constrói o heatmap juntamente com a linha dendrograms das tabelas respectivas matriz. Argumentos adicionais usando o cellnote função ajuda a distinguir diferencialmente expressos (log2FC > 0,5, FDR < 0.05) genes ATL em cada comparação entre uma grande variedade de condições experimentais por um * símbolo. Aplicar o fluxo de trabalho típico em R executando o comando linhas 17 (arquivo complementar 1) ou alternativamente, repita os passos 6.2.2 a 6.2.5 para construir o heatmaps usando software de MeV.
8. análise das relações entre a sequência parólogo divergência e expressão de Gene co
- Construa a matriz de similaridade emparelhada. Os elementos da matriz de similaridade são os valores de similaridade da sequência calculadas a partir os alinhamentos de proteína emparelhadas.
- Use o alto-relevo agulha web servidor41 com as configurações padrão para fazer alinhamentos de sequência emparelhadas e salvar como arquivo de texto. Abra o arquivo de texto de saída e remover todas as linhas de comentário, juntamente com nomes de coluna e linha para gerar um arquivo chamado "similarityTable.txt".
Nota: Uma mesa tão dispõe de uma linha para cada gene ATL relatando os valores de similaridade calculados em cada um do alinhamento emparelhado. A ordem dos loci em linhas e colunas é o mesmo modo que uma matriz simétrica é gerada com o respeito dos valores de diagonal.
- Use o alto-relevo agulha web servidor41 com as configurações padrão para fazer alinhamentos de sequência emparelhadas e salvar como arquivo de texto. Abra o arquivo de texto de saída e remover todas as linhas de comentário, juntamente com nomes de coluna e linha para gerar um arquivo chamado "similarityTable.txt".
- Construa a matriz com dados de expressão co calculando o coeficiente de correlação de Pearson. O procedimento a seguir exige R e o módulo perl PDL.
- Baixe os valores de expressão para os 96 genes ATL, executando o comando linhas 18 (arquivo complementar 1) dentro de um terminal. Realizar uma análise de expressão co usando um script perl personalizado que pode ser baixado através da execução de linhas de comando 19 (arquivo complementar 1). Esse script irá calcular o coeficiente de correlação de Pearson entre pares dos loci ATL como anteriormente relatado.
- Abra o script executando o comando linhas 20 (arquivo complementar 1) e siga as instruções de saída.
- Execute o teste de Mantel carregando as matrizes de dois dados e executando as estatísticas de execução de linhas de comando 21 (arquivo complementar 1), com "nrep" que representa o número de permutações. O teste consiste em calcular a correlação entre os elementos destas matrizes, permuting as matrizes e então calcular a estatística de teste a mesma novamente.
Nota: Todos os valores obtidos do teste de estatística são usados para construir uma distribuição de referência do teste estatístico, que será usado para calcular um p-valor para testar a significância. O número de permutações define a precisão com que o p-valor pode ser obtido.
Resultados
O gene VIT_05s0077g01970, identificado como o mais parecido com a. thaliana ATL2 (At3g16720), através de uma pesquisa BLASTp, foi usado como sonda para inspeccionar os membros da família ATL no genoma da videira (V. vinifera cv Pinot Noir PN40024). A análise de PSI-BLAST convergiu depois de alguns ciclos, revelando uma lista de putativos genes pertencentes à família de gene videira ATL (figura 1A). A presença do anel-H2 domínio canônico para cada candidato foi avaliada pelo inspeção visual do alinhamento muscular de todas as entradas identificados na análise (figura 1B). Apenas os genes contendo os aminoácidos conservados corretamente espaçados, os dois resíduos de histidina, bem como os resíduos de prolina antes o terceiro cisteína foram considerados como ATLs de acordo com a definição original de ATL em Arabidopsis5. Um total de 96 genes videira cumprido os requisitos e foram considerados para a caracterização mais. Cada membro da família ATL foi analisado para definir as características específicas do gene e a correspondente proteína codificada, ou seja, a presença de outros domínios conhecidos além do anel-H2, transmembranares ou hidrofóbicas regiões ricas, subcellular localização e sítios de fosforilação putativo (tabela 1 e tabela 2).

Figura 1: pesquisa PSI-BLAST e alinhamento de videira putativo ATLs. (A) Screenshot dos hits top 10 da primeira pesquisa PSI-BLAST iteração usando a sequência de proteínas VIT_05s0077g01970 como isca. (B) parte do alinhamento da videira selecionados 96 ATLs putativos, mostrando seu domínio de anel-H2 e o logotipo correspondente obtidos usando um conjunto de biologia molecular (ver Tabela de materiais). Reproduzido de Ariani et al . licenciado sob uma licença Creative Commons Attribution 4.0 internacional42.Por favor clique aqui para ver uma versão maior desta figura.
| Nome | Identificação do gene | Comprimento do gene (bp) | Número de intron | UniProt ID | Comprimento de proteínas (aa) | Motivo de anel-H2 | Número de domínio TM/H | Outros domínios | ||
| VviATL3 | VIT_09s0002g00220 | 1245 | 0 | F6HXK6 | 304 | PxC | 1 | |||
| VviATL4 [VviRHX1A] | VIT_15s0021g00890 | 1827 | 3 | D7SM36 | 203 | PxC | 0 | |||
| VviATL18 | VIT_11s0118g00780 | 1113 | 2 | F6HCI8 | 193 | PC | 0 | |||
| VviATL23a | VIT_18s0001g01060 | 935 | 0 | F6H0E4 | 114 | PxC | 0,5 | |||
| VviATL23b | VIT_18s0001g01050 | 399 | 0 | E0CQX3 | 132 | PxC | 1 | |||
| VviATL24 | VIT_17s0000g06460 | 4466 | 4 | D7SI89 | 217 | PxC | 1 | |||
| VviATL27 | VIT_00s0264g00020 | 2554 | 4 | D7T1R5 | 235 | PxC | 1 | |||
| VviATL43 | VIT_11s0052g00530 | 1576 | 2 | D7SQD9 | 457 | PxC | 3 | |||
| VviATL54a | VIT_18s0001g06640 | 3221 | 1 | F6H0Y5 | 405 | PxC | 1 | |||
| VviATL54b | VIT_03s0017g00670 | 2774 | 1 | F6HTI0 | 427 | PxC | 1 | |||
| VviATL55 [VviRING1] | VIT_07s0191g00230 | 1844 | 0 | F6HRP9 | 372 | PxC | 1 | |||
| VviATL63 | VIT_06s0004g06930 | 804 | 0 | D7SJU6 | 267 | PxC | 1 | |||
| VviATL65 | VIT_03s0063g01890 | 2068 | 0 | F6HQI8 | 396 | PxC | 1 | |||
| VviATL82 | VIT_01s0026g02540 | 820 | 0 | F6HPQ9 | 233 | PC | 0,5 | |||
| VviATL83 | VIT_17s0000g08400 | 1887 | 0 | F6GSQ4 | 143 | PC | 0 | |||
| VviATL84 | VIT_06s0004g00120 | 1853 | 0 | F6GUP5 | 368 | PC | 0,5 | ZF-RING_3 | ||
| VviATL85 | VIT_12s0034g01400 | 786 | 0 | F6H965 | 261 | PC | 0,5 | |||
| VviATL86 | VIT_12s0034g01390 | 1434 | 1 | D7T016 | 451 | PC | 0,5 | |||
| VviATL87 | VIT_18s0001g03270 | 1002 | 0 | F6H0T2 | 333 | PC | 0,5 | ZF-RING_3 | ||
| VviATL88 | VIT_08s0040g00590 | 1320 | 0 | F6HQR2 | 314 | PC | 0 | ZF-RING_3 | ||
Tabela 1: primeira 20 VviATL de genes e características da sequência das proteínas correspondentes. TM: transmembrana; H: hidrofóbicos; 0,5 indica a presença de uma ou mais regiões hidrofóbicas. Reproduzido de Ariani et al . licenciado sob uma licença Creative Commons Attribution 4.0 internacional42.

Tabela 2: detalhes sobre o primeiro 20 VviATL posição do gene no V. vinifera genoma, estado de duplicação e as características físico-químicas de proteínas ATL e localização. (um) número de sítios de fosforilação predito por Musite; (b) predições similares obtidas pelo menos dois software são destacadas em negrito; ngLOC foi usado com configurações padrão, Considerando que TargetP v 1.1 e proteína Prowler localização Subcellular foram usados com um corte de probabilidade de 0,5. NUC, núcleo; MIT, mitocôndrias; CHL, cloroplasto; PLA, a membrana plasmática; S, via secretora (presença de um peptídeo sinal); M, mitocôndria; C, cloroplasto; Ó ou -, outros locais; nd, não determinado (ou seja, valor abaixo do limiar). Reproduzido de Ariani et al . licenciado sob uma licença Creative Commons Attribution 4.0 internacional42. Clique aqui para baixar este arquivo.
Uma análise filogenética, incluindo as sequências de nucleotídeos dos genes de ATL-codificação de videira identificados em conjunto com as sequências de referência a. thaliana ATL gene da família foi usada para a nomenclatura ATL de videira, de acordo com as diretrizes do o sNCGGa8. Noventa e seis e 83 sequências nucleotídicas de V. vinifera e a. thaliana, respectivamente, foram submetidas ao pipeline de Phylogeny.fr para obter uma árvore filogenética confiável.As último sequências foram usadas mais tarde para anotar e nomear os genes de videira com base em relacionamentos sólidos (Figura 2). Seguindo esta abordagem, a 13 de 96 videira ATLs recebeu um identificador específico, considerando sua homologia-para-um com um a. thaliana ATL. Os nomes dos outros 83 genes foram atribuídos com base na árvore filogenética, com um progressivo numeração de cima para baixo, a partir de um número de gene ATL mais elevado do que o usado no número mais alto a. thaliana.
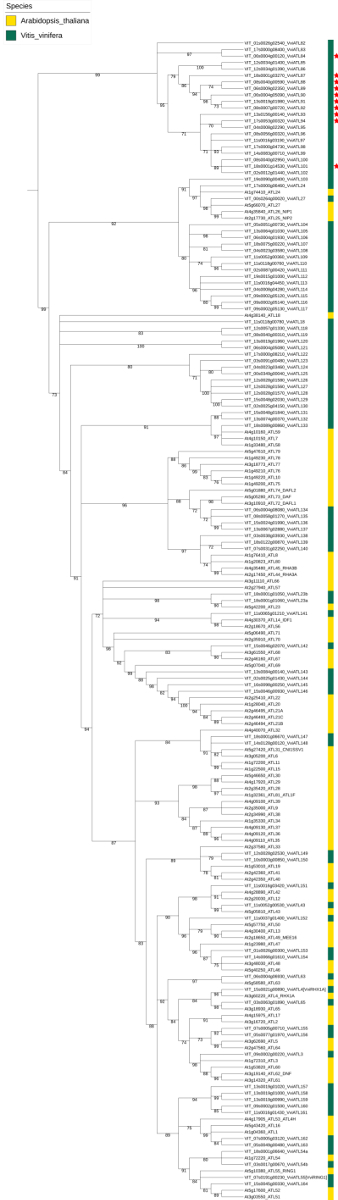
Figura 2: Árvore filogenética de V. vinifera a. thaliana ATL E3 ubiquitina ligase-codificação genes e. A árvore não enraizada foi gerada com a suite de Phylogeny.fr (V. vinifera (em verde) e 83 genes ATL de a. thaliana relatado no banco de dados UniProt (em amarelo). Foram obtidos valores de apoio de filial 100 repetições de inicialização. As estrelas vermelhas indicam a presença de um domínio de dedo (BZF) zinco BCA2 as proteínas correspondentes. Reproduzido de Ariani et al . licenciado sob uma licença Creative Commons Attribution 4.0 internacional42. Clique aqui para ver uma versão maior desta figura.
Mapeamento de genes ATL-codificação para os cromossomos de videira mostrou uma ampla distribuição em todo o genoma, sugerindo que a duplicação do inteiro-genoma como a principal força evolutiva na expansão da família do gene ATL em videira. Com efeito, 31 ATLs foram encontradas em regiões cromossômicas homólogas potencialmente provenientes de eventos de duplicação do genoma inteiro ou segmentar. Além disso, a mesma análise destacou 13 tandemly de duplicação de genes, uma duplicata proximal e 51 duplicatas dispersas (Figura 3). Considerando o grande número de genes duplicados da família ATL, realizamos um teste de enriquecimento (teste exato de Fisher) para verificar a retenção preferencial dos genes duplicados durante o fracionamento do genoma. Com um p-valor < 0.001, este teste confirma a hipótese de que a duplicação de genes ATL foram retidos aleatoriamente mais do que esperado, sugerindo um papel para a família do gene ATL durante videira adaptação e evolução.
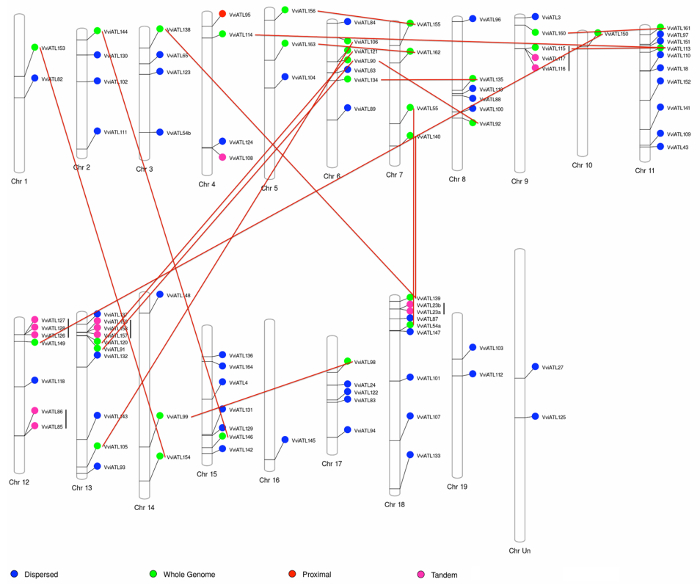
Figura 3: Distribuição de gene Grapevine ATL-codificação na V. vinifera cromossomos e a duplicação estado. Os genes ATL 96 videira com informação cromossômica exata disponível no banco de dados foram mapeados para os 19 cromossomos de V. vinifera . As cores indicam o evento original de duplicação. Linhas pretas verticais e linhas vermelhas identificam pares derivados de duplicações em tandem e duplicações de genoma inteiro, respectivamente. Reproduzido de Ariani et al . licenciado sob uma licença Creative Commons Attribution 4.0 internacional42. Clique aqui para ver uma versão maior desta figura.
Para investigar as funções biológicas putativos dos ATLs em videira, foi realizada uma meta-análise sobre o V. vinifera CV Corvina global expressao Atlas12. O conjunto de dados inclui valores de expressão de todo o genoma de 54 órgãos diferentes videira e estádios de desenvolvimento e foi usado para executar uma análise bi-cluster hierárquica. Resultados não só confirmaram que todos os 96 ATLs foram expressos em pelo menos um dos tecidos 54/estágios, mas também apontou a presença de cinco grupos principais de perfis de expressão (Figura 4A). Brevemente, grupos A e E mostraram-se em frente de comportamentos, em particular o primeiro é caracterizado por um general downregulation de genes ATL em amostras juvenis, incluindo estágios iniciais de baga, folha jovem, gavinhas, inflorescência e maioria das etapas do broto. Por outro lado, no mesmo cluster A, maduras amostras tais como bagas no amadurecimento e pós-colheita murchando estágios, tecidos lenhosas e estágios finais de sementes desenvolvimento ATL genes mostrou um upregulation predominante. Genes em Cluster C foram principalmente ativador na maioria das amostras, enquanto os genes ATL em cluster D eram frequentemente upregulated em estágios finais do desenvolvimento da baga. Finalmente, cluster B não apresentaram qualquer variação relevante nos perfis de expressão.
Uma abordagem similar foi aplicada para estudar a expressão da videira ATL familiares em resposta a estresses bióticos e abióticos, usando conjuntos de dados específicos, construídos para este fim. Uma enorme quantidade de dados de expressão derivando de microarray e RNA-seq experiências estão disponíveis a partir de bancos de dados de acesso público como Gene Expression Omnibus (GEO) e ArrayExpress. Uma vez coletados e convenientemente normalizada, a informação foi explorada durante mais insights sobre a função potencial do ATLs na resposta da planta ao estresse. Analisar os perfis de expressão de videira ATLs em resposta a estresses bióticos revelou que 62 de 96 transcrições mostraram uma significativa modulação (mudança de dobra log2 (FC) > | 0.5 |) pelo menos duas condições, com uma taxa falsa da descoberta (FDR) < (0.05 Figura 4B). O número aumenta para 81, considerando somente o limiar do FDR em uma única condição. Estes resultados sugeriram um envolvimento direto da família do gene ATL na resposta aos agentes patogénicos, também em videira. Em particular, um grupo de 12 genes (VviATL3-27-54b-55-90-97-123-144-148-149-156) foram fortemente upregulated em resposta a maioria dos patógenos, incluindo biotrófica e necrotrophic e outros herbívoros e assim, merece atenção para ainda mais funcional análises.
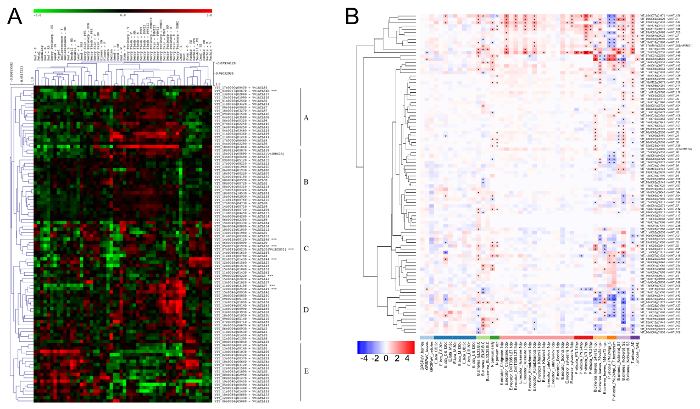
Figura 4: expressão de gene ATL clusteringof hierárquica em videira Atlas e no dataset videira de estresse biótico. (A) a expressão log transformada valores de genes ATL videira em videira Atlas12 foram usados para análise de cluster hierárquica com base na métrica de distância de Pearson. A cor escala representa a mais elevada (vermelho) ou inferior (verde) expressão níveis com respeito para a abundância de transcrição mediana de cada gene em todas as amostras. Letras de À E do lado direito indicam os diferentes clusters identificados.AB: depois da explosão; B: estouro; broto-w: broto de inverno; F: floração; FB: floração começa; FS: conjunto de frutas; G: verde; Senhor: meados-maturação; PFS: pós-fruta conjunto; PHWI-II-III: pós-colheita minguante 1, 2 e 3 meses; R: maturação; S: senescentes; haste-w: caule lenhoso; V: veraison; WD: bem desenvolvido; Y: jovens. (B) a cor escala representa aumento (vermelho) ou diminuído (azul) prega mudanças videira ATL da expressão do gene em amostras infectadas comparado com controles para cada condição. Asteriscos indicam a expressão diferencial significativa (FDR < 0,05) de cada ATL nas condições correspondentes. Reproduzido de Ariani et al . licenciado sob uma licença Creative Commons Attribution 4.0 internacional42. Clique aqui para ver uma versão maior desta figura.
Suplementares tabela 1: candidatos de genes ATL por splicing alternativo. (um) ATL gene ID de acordo com a previsão de uva gene V1 e anotação, identificação do gene (b), ATL de acordo com o V2 gene uva previsão e anotação43, (c) número de putativo ATL alternativas emenda variantes, (d) informações sobre a sequência de cada variante ATL putativo de código. Clique aqui para baixar este arquivo.
Suplementares tabela 2: Clique aqui para baixar este arquivo.
Arquivo complementar 1: Clique aqui para baixar este arquivo.
Discussão
Na era genômica, muitas famílias de gene foram caracterizadas profundamente em várias espécies de plantas. Esta informação é preliminar do estudos funcionais e fornecer um quadro para investigar mais profundamente o papel dos diferentes membros de uma família. Neste contexto, há também a necessidade de um sistema de nomenclatura que permite para identificar exclusivamente cada membro de uma família, evitando a redundância e confusões que podem surgir quando os nomes são atribuídos de maneira independente de diferentes genes por grupos de pesquisa diferentes.
Após a consideração pensativa, a comunidade científica de videira concordou com genes de videira nome em uma família com base em semelhanças com os genes de Arabidopsis e estabeleceu uma série de regras que devem ser aplicados para descrever novas famílias de genes em videira, Basicamente a partir de comparação filogenética das sequências nucleotídicas entre videira e membros da família de Arabidopsis8. Portanto, apenas os genes que já são anotados e nomeados corretamente em Arabidopsis podem ser usados na nomenclatura de videira. O procedimento aplicado para a identificação de videira ATL orthologues em Arabidopsis descrito aqui foi realizado, portanto, unicamente para cumprir a exigência de atribuição de nomenclatura correta videira gene familiar. No entanto, para outras espécies de plantas, abordagens alternativas poderiam ser uma opção. Por exemplo, homologia pode ser inferida utilizando um sucessos de explosão bidirecional (BBH), onde os orthologues são definidos como pares de genes em duas espécies que são mais semelhantes (ou seja, com maior pontuação de alinhamento) um ao outro do que para qualquer outro gene no outro espécie de44. No entanto, esse método poderia faltar muitos orthologues no caso de alta taxa de duplicação de genes, tais como em plantas e animais45. Além disso, no caso de genes codificação de ATL, BBH pode recuperar genes falta a estrutura exacta de ATL-tipo anel-H2 (incluindo o resíduo prolina) ou genes que não são anotados e nomeados como ATLs em Arabidopsis. Embora sob uma perspectiva evolucionista, essa busca pode ser relevante, a recuperação de orthologues que não são anotados não teria cumprido o escopo de videira ATL gene familiar anotação e nomenclatura e orthologues que não são anotadas como ATLs Não pode ser usado para os membros da família nome videira. Outra possibilidade é inferir homologia com base em aminoácidos em vez de sequências nucleotídicas usando InParanoid46, ou o mais recente Hieranoid 247, embora tais fluxos de trabalho não são expressamente recomendados pela comunidade científica.
Meta-análise de expressão, que pode ser definida como uma abordagem sistemática para estudar e combinar diferentes repositórios de dataset publicamente disponível de dados de expressão, permite destacar compartilhados e diferentes mecanismos moleculares em uma variedade de condições. Assim, a integração de informações de expressão do gene de múltiplas experiências transcriptomic em grande escala pode melhorar a caracterização de uma família de gene, definindo os perfis de expressão dos membros da família através de experimentos, minimizando assim o impacto de fatores específicos do experimento e apoiar uma suposição mais robusta da função do gene putativo em processos particulares. No entanto, a utilização de dados de microarray exige a integração de dados de expressão obtidos com diferentes plataformas, considerando suas próprias limitações. Por exemplo, na plataforma de microarray de Nimblegen de videira, uma proporção significativa de probesets para os genes correspondentes representados na matriz (~ 13.000 genes) têm problemas potencialmente Cruz-hibridação48. No caso da família ATL de videira, 15 genes podem ser afetados por tal fenômeno. No entanto, como discutido por Cramer et al 48, a Cruz-identificação dos membros da família gene altamente similar pela mesma sonda poderia fornecer informações interessantes sobre a expressão, em condições específicas, não apenas de um único gene, mas de dois genes mais compartilhamento sequência de alta semelhanças e, portanto, potencialmente compartilhamento de metas e funções. Outro problema potencial relacionado a conjuntos de dados microarray é o limite de detecção da expressão de plataformas microarray, que não são muito sensíveis. Para resolver os dois diz respeito, isto é., Cruz-hibridação e a sensibilidade do sinal, uma solução possível seria a considerar apenas RNAseq expressão de conjuntos de dados. No entanto, a meta-análise de dados RNAseq de muito grandes conjuntos de dados de muitos estudos diferentes pode tornar-se altamente demorada e pode exigir muitos recursos computacionais e alta especialização.
Embora a abordagem aqui apresentada pretende ser exaustiva, pode ser certamente mais complementado com outras análises. Em primeiro lugar, para alcançar ainda mais insights sobre a evolução molecular e a relação filogenética entre membros da família gene em plantas, a análise filogenética poderia ser estendida construindo uma árvore filogenética usando alinhamentos múltiplos da sequência dos membros da família de várias espécies de plantas. Também é possível calcular o tempo evolutivo de genes de família, uma estimativa das suas taxas de sinônimos e não-sinônimo de substituição durante a evolução, determinando os valores de Ks (número de substituições sinônimas por sinônimo local em um determinado período de tempo) e Ka (número de substituições nonsynonymous por site não-sinônimos no mesmo período). O rácio de Ka/Ks é usado para inferir os mecanismos de eventos de duplicação de gene após divergência dos seus antepassados. Um valor de Ka/Ks = 1 sugere seleção neutra, um valor de Ka/Ks de < 1 sugere purificando a seleção e um valor de Ka/Ks de > 1 sugere selecção positiva49. Além disso, se a análise da estrutura genética revela a presença de intrões, a caracterização de família do gene pode ser novamente prorrogada para a detecção de alternativa variantes de emenda. De fato, baseado em uma pesquisa profunda de RNA-seq dados de diferentes tecidos, condições de estresse e genótipos43, 21 (da 96) ATLs são fortes candidatos para eventos splicing alternativos, com potencial número de isoformas variando de 2 a 16 para estes ATLs (ver Complementar tabela 1). Transcrições alternativas frequentemente produzem isoformas de proteínas que variam em sequências de aminoácidos e essas mudanças podem alterar as propriedades de celulares das proteínas e podem causar alterações da modulação sutil à perda da função do produto do gene. Por esse motivo, eventos de splicing alternativos estiveram envolvidos em funções de vegetais importantes, incluindo a resposta ao estresse, resistência a doenças, fotossíntese e floração50,51.Integração de informações de promotor do gene ATL que contém putativo cis-elementos reguladores52 ou encontrar moléculas (por exemplo, microRNA e tempo não-codificantes do RNA) potencialmente alvo ATLs53 também pode ser complementado para revelam insights de sistema sobre o complexo molecular regulamento e interação de videira ATLs.
Em conclusão, a escolha das análises a serem executadas, bem como os procedimentos a aplicar para caracterizar uma nova família de gene em uma espécie de planta são impulsionados principalmente pelas regras da comunidade científica, bem como pelo escopo da identificação de família de gene. É importante ter em mente os passos de possível investigação subsequente, que irão explorar o conjunto de informações, entre que inclui a evolução de genes entre espécies de plantas, descrição de estrutura do genoma ou candidatos confiáveis para seleção em funcional estudos.
Divulgações
Os autores não têm nada para divulgar.
Agradecimentos
O trabalho foi apoiado pela Universidade de Verona, dentro do quadro de comum projeto 2014 (caracterização da família do gene ATL em videira e da sua participação na resistência à Plasmopara viticola).
Materiais
| Name | Company | Catalog Number | Comments |
| Personal computer | |||
| Basic Local Alignment Search Tool (BLAST) | https://blast.ncbi.nlm.nih.gov/Blast.cgi | ||
| Molecular Evolutionary Genetics Analysis (MEGA) | http://www.megasoftware.net/ | ||
| Motif-based sequence analysis tools (MEME) | http://meme-suite.org/ | ||
| Geneious | Biomatters Limited | http://www.geneious.com/ | |
| ProtParam Tool | http://web.expasy.org/protparam/ | ||
| ngLOC | http://genome.unmc.edu/ngLOC/index.html | ||
| TargetP v1.1 Server | http://www.cbs.dtu.dk/services/TargetP/ | ||
| Protein Prowler | http://bioinf.scmb.uq.edu.au:8080/pprowler_webapp_1-2/ | ||
| MUsite | http://musite.sourceforge.net/ | ||
| Pfam | http://pfam.xfam.org/ | ||
| TMHMM Server v. 2.0 | http://www.cbs.dtu.dk/services/TMHMM/ | ||
| ProtScale | http://web.expasy.org/protscale/ | ||
| Grape Genome Database (CRIBI) | http://genomes.cribi.unipd.it/grape/ | ||
| PhenoGram | http://visualization.ritchielab.psu.edu/phenograms/plot | ||
| MCScanX | http://chibba.pgml.uga.edu/mcscan2/ | ||
| Interactive Tree Of Life (iTOL) | http://itol.embl.de/ | ||
| UniProt | http://www.uniprot.org/ | ||
| Phylogeny.fr | http://www.phylogeny.fr/index.cgi | ||
| MUSCLE | http://www.ebi.ac.uk/Tools/msa/muscle/ | ||
| Gblocks Server | http://molevol.cmima.csic.es/castresana/Gblocks_server.html | ||
| Vitis vinifera cv. Corvina gene expression Atlas datamatrix | https://www.researchgate.net/publication/273383414_54sample_ datamatrix_geneIDs_Fasoli2012 | ||
| Multi Experiment Viewer (MeV) | http://mev.tm4.org/#/welcome | ||
| Sequence Read Archive (SRA) | https://www.ncbi.nlm.nih.gov/sra | ||
| R | https://www.r-project.org/ | ||
| EMBOSS Needle (EMBL-EBI) | http://www.ebi.ac.uk/Tools/psa/emboss_needle/ |
Referências
- Jaillon, O., et al. The grapevine genome sequence suggests ancestral hexaploidization in major angiosperm phyla. Nature. 449 (7161), 463-467 (2007).
- Adam-Blondon, A. -F., et al. Genetics, Genomics, and Breeding of Grapes. , Science Publishers. 211-234 (2011).
- Chen, L., Hellmann, H. Plant E3 Ligases: Flexible Enzymes in a Sessile World. Mol. Plant. 6 (5), 1388-1404 (2013).
- Vierstra, R. D. The ubiquitin-26S proteasome system at the nexus of plant biology. Nat. Rev. Mol. Cell Biol. 10 (6), 385-397 (2009).
- Serrano, M., Parra, S., Alcaraz, L. D., Guzmán, P. The ATL Gene Family from Arabidopsis thaliana and Oryza sativa Comprises a Large Number of Putative Ubiquitin Ligases of the RING-H2 Type. J. Mol. Evol. 62 (4), 434-445 (2006).
- Aguilar-Hernández, V., Aguilar-Henonin, L., Guzmán, P. Diversity in the Architecture of ATLs, a Family of Plant Ubiquitin-Ligases, Leads to Recognition and Targeting of Substrates in Different Cellular Environments. PLoS One. 6 (8), e23934(2011).
- Guzmán, P. The prolific ATL family of RING-H2 ubiquitin ligases. Plant Signal Behav. 7 (8), 1014-1021 (2012).
- Grimplet, J., et al. The grapevine gene nomenclature system. BMC Genomics. 15, 1077(2014).
- Prince, V. E., Pickett, F. B. Splitting pairs: the diverging fates of duplicated genes. Nat. Rev. Genet. 3 (11), 827-837 (2002).
- Magadum, S., Nerjee, U., Murugan, P., Gangapur, D., Ravikesavan, R. Gene duplication as a major force in evolution. J. Gen. 92 (1), 155-161 (2013).
- Wang, N. Patterns of Gene Duplication and Their Contribution to Expansion of Gene Families in Grapevine. Plant Mol. Biol. Rep. 31 (4), 852-861 (2013).
- Fasoli, M. The Grapevine Expression Atlas Reveals a Deep Transcriptome Shift Driving the Entire Plant into a Maturation Program. Plant Cell. 24 (9), 3489-3505 (2012).
- BLAST. BLAST2.6.0. , Available from: https://blast.ncbi.nlm.nih.gov/Blast.cgi (2016).
- MEGA. MEGA7.0.25 build 7170412. , Available from: http://www.megasoftware.net/ (2017).
- MEME. MEME Suite Version 4.11.4. , Available from: http://meme-suite.org/ (2017).
- ProtParam. ExPASy Server. , Available from: http://web.expasy.org/protparam/ (2005).
- ngLOC v1.0. , Available from: http://genome.unmc.edu/ngLOC/index.html (2007).
- TargetP v1.1 Server. , Available from: http://www.cbs.dtu.dk/services/TargetP/ (2000).
- Prowler v1.2. , Available from: http://bioinf.scmb.uq.edu.au:8080/pprowler_webapp_1-2/ (2005).
- MuSite v1.0. , Available from: http://musite.sourceforge.net/ (2010).
- Pfam. Pfam version 31.0. , Available from: http://pfam.xfam.org/ (2016).
- TMHMM v2.0c. , Available from: http://www.cbs.dtu.dk/services/TMHMM/ (2007).
- ExPASy. ProtScale. , Available from: http://web.expasy.org/protscale/ (2005).
- CRIBI. Grape genome database. , Available from: http://genomes.cribi.unipd.it/grape/ (2012).
- PhenoGram. , Available from: http://visualization.ritchielab.psu.edu/phenograms/plot (2012).
- ScanX v0.8. , Available from: http://chibba.pgml.uga.edu/mcscan2/ (2013).
- Interactive Tree Of Life (iTOL). Version3.5.3. , Available from: http://itol.embl.de/ (2016).
- UniProt. , Available from: http://www.uniprot.org/ (2016).
- Phylogeny.fr. , Available from: http://www.phylogeny.fr/index.cgi (2008).
- MUSCLE. , Available from: http://www.ebi.ac.uk/Tools/msa/muscle/ (2017).
- Gblocks Server. Version 0.91b. , Available from: http://molevol.cmima.csic.es/castresana/Gblocks_server.html (2002).
- Vitis vinifera cv. Corvina gene expression Atlas. , Available from: https://www.researchgate.net/publication/273383414_54sample_datamatrix_geneIDs_Fasoli2012 (2015).
- Multiple Experiment Viewer (MeV). Version 4.8.1. , Available from: http://mev.tm4.org/ (2017).
- Sequence Read Archive (SRA). , Available from: https://www.ncbi.nlm.nih.gov/sra (2017).
- Bolger, A. M., Lohse, M., Usadel, B. Trimmomatic: a flexible trimmer for Illumina sequence data. Bioinformatics. 30 (15), 2114-2120 (2014).
- Langmead, B., Salzberg, S. L. Fast gapped-read alignment with Bowtie 2. Nat Meth. 9 (4), 357-359 (2012).
- Anders, S., Pyl, P. T., Huber, W. HTSeq-a Python framework to work with high-throughput sequencing data. Bioinformatics. 31 (2), 166-169 (2015).
- R. Version 3.4.1. , Available from: https://www.r-project.org/ (2017).
- Ritchie, M. E. limma powers differential expression analyses for RNA-sequencing and microarray studies. Nucleic Acids Res. 43 (7), e47(2015).
- Love, M. I., Huber, W., Anders, S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2. Genome Biology. 15 (12), 550(2014).
- EMBL-EBI. EMBOSS Needle. , Available from: http://www.ebi.ac.uk/Tools/psa/emboss_needle/ (2017).
- Ariani, P. Genome-wide characterisation and expression profile of the grapevine ATL ubiquitin ligase family reveal biotic and abiotic stress-responsive and development-related members. Sci. Rep. 6, 38260(2016).
- Vitulo, N., et al. A deep survey of alternative splicing in grape reveals changes in the splicing machinery related to tissue, stress condition and genotype. BMC Plant Biol. 14 (1), 99(2014).
- Overbeek, R., Fonstein, M., D'Souza, M., Pusch, G. D., Maltsev, N. The use of gene clusters to infer functional coupling. Proc. Natl. Acad. Sci. USA. 96 (6), 2896-2901 (1999).
- Dalquen, D. A., Dessimoz, C. Bidirectional Best Hits Miss Many Orthologs in Duplication-Rich Clades such as Plants and Animals. Genome Biol. Evol. 5 (10), 1800-1806 (2013).
- Remm, M., Storm, C. E. V., Sonnhammer, E. L. L. Automatic clustering of orthologs and in-paralogs from pairwise species comparisons1. J. Mol. Biol. 314 (5), 1041-1052 (2001).
- Kaduk, M., Sonnhammer, E. Improved orthology inference with Hieranoid 2. Bioinformatics. 33 (8), (2017).
- Cramer, G. R., et al. Transcriptomic analysis of the late stages of grapevine (Vitis vinifera cv. Cabernet Sauvignon) berry ripening reveals significant induction of ethylene signaling and flavor pathways in the skin. BMC Plant Biol. 14, 370(2014).
- Juretic, N., Hoen, D. R., Huynh, M. L., Harrison, P. M., Bureau, T. E. The evolutionary fate of MULE-mediated duplications of host gene fragments in rice. Genome Res. 15 (9), 1292-1297 (2005).
- Filichkin, S. A. Genome-wide mapping of alternative splicing in Arabidopsis thaliana. Genome Res. 20 (1), 45-58 (2010).
- Quesada, V., Macknight, R., Dean, C., Simpson, G. G. Autoregulation of FCA pre-mRNA processing controls Arabidopsis flowering time. EMBO J. 22 (12), 3142-3152 (2003).
- Wong, D. C. J., Gutierrez, R. L., Gambetta, G. A., Castellarin, S. D. Genome-wide analysis of cis-regulatory element structure and discovery of motif-driven gene co-expression networks in grapevine. DNA Res. 24 (3), 311-326 (2017).
- Wong, D. C. J., Matus, J. T. Constructing Integrated Networks for Identifying New Secondary Metabolic Pathway Regulators in Grapevine: Recent Applications and Future Opportunities. Front. Plant Sci. 8, 505(2017).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados