Method Article
Генерация высших шаблона ДНК хроматина Качество Иммунопреципитация для высокопроизводительного секвенирования (чип-след)
В этой статье
Резюме
Сочетание иммунопреципитации хроматина и сверхвысокой пропускной последовательности (чип-далее) может определять и белок-ДНК взаимодействия в данной линии ткани или клетки. Изложенные в том, как генерировать высокое качество щепы шаблон для последующего секвенирования, используя опыт с TCF7L2 транскрипционный фактор в качестве примера.
Аннотация
Обломок-последовательности (чип-далее) методы непосредственно предложить целого генома охвата, где объединение иммунопреципитации хроматина (чип) и массивных параллельных последовательностей могут быть использованы для идентификации репертуара млекопитающих ДНК связаны факторов транскрипции в естественных условиях. "Следующего поколения" секвенирование генома технологии обеспечивают 1-2 порядков увеличение количества последовательностей, которые могут быть экономически эффективно генерируются на старые технологии что позволяет чип-далее методы для непосредственного предоставления целого генома покрытия для эффективного профилированию млекопитающего белок-ДНК взаимодействий.
Для успешного чип-последующие подходы, необходимо генерировать высокое качество чип ДНК-матрицы для получения наилучших результатов секвенирования. Описание основано вокруг опыт работы с белкового продукта гена наиболее сильно вовлечены в патогенез сахарного диабета 2 типа, а именно транскрипции фактора транскрипции фактора 7-подобный 2 (TCF7L2). Этот фактор также вовлечен в различные виды рака.
Изложенные в том, как генерировать высокое качество щепы ДНК-матрице, полученные из клеточной линии колоректального рака, HCT116, в целях создания высокого разрешения карты по виртуализации, чтобы определить гены связаны TCF7L2, давая дальнейшее понимание в свою ключевую роль в патогенезе сложных признаков.
Введение
На протяжении многих лет наблюдается неудовлетворенная потребность определить набор генов связаны и регулируются геном данного белка широко, в частности, в классе фактора транскрипции.
Одом и соавт. 1 использован иммунопреципитацию хроматина (чип) в сочетании с промоутером микрочипов в систематическом выявлении генов занимают заранее оговоренное транскрипционных регуляторов в человеческой печени и панкреатических островков. Впоследствии Джонсон и др.. 2 разработана крупномасштабная хроматин иммунопреципитации на основе прямых ультра высокой пропускной секвенирования ДНК (чип-след), чтобы всесторонне карту белок-ДНК взаимодействия по всей геномах млекопитающих. В качестве теста случае, они отображаются в естественных условиях связывания нейронов-ограничительные глушителем фактора (NRSF) до 1946 мест в геноме человека. Отображаемые данные высоким разрешением связывания позиции (+ 50 пар оснований), что способствовало как яsolation мотивов и выявления NRSF-связывающих мотивов. Эти чипсеты-SEQ данных также была высокой чувствительностью и специфичностью и статистической достоверности (p <10 -4), свойств, которые важны для выведения новых взаимодействий кандидата.
Робертсон и др.. 3 также используется чип-SEQ с целью картирования STAT1 цели в γ-интерферона (IFN-γ) стимулировали и нестимулированных человека HeLa S3 клетки в живом организме. Чип-SEQ, используя 15,1 и 12,9 млн. однозначно отображается последовательность читает, и, по оценкам, ложных открытие менее 0,001, они определили 41 582 и 11 004 предполагаемых STAT1-связывающие области в стимулированных и стимулированных клеток соответственно. Из 34 локусов как известно, содержат STAT1-интерферона реагировать сайты связывания 4-8, чип-далее найдены 24 (71%). Обломок-SEQ цели были обогащены последовательности похожи на известные STAT1 связывающие мотивы. Сравнения с двумя существующими чип-PCR наборы данных предложеночто чип-сл чувствительность была между 70% и 92%, а специфичность была по крайней мере 95%. Кроме того, было ясно, что чип-SEQ предлагает как низкий аналитической сложности и чувствительности, которая увеличивается с глубиной секвенирования.
Таким образом, геном "следующего поколения" секвенирования технологии обеспечивают 1-2 порядков увеличение количества последовательностей, которые могут быть экономически эффективно генерируются на старых технологий 9. Обломок-SEQ методами таким образом, напрямую обеспечит целого генома для эффективного охвата профилирование млекопитающих белок-ДНК взаимодействия 3.
В 2006 году в сильной ассоциации из вариантов фактора транскрипции 7-подобный 2 (TCF7L2) гена с диабетом 2 типа был обнаружен 10. Другие исследователи уже самостоятельно реплицировать этот вывод в разных национальностей и, что интересно, с первого генома широкие исследования ассоциации диабета 2 типа опубликованы в Nature 11,12 , науки и в других местах 13-15 16,17, самая сильная связь была действительно с TCF7L2; этого в настоящее время считается наиболее значительные генетические нахождения в сахарный диабет 2 типа на сегодняшний день 18-20. Кроме того, TCF7L2 была связана с риском рака 21,22, более того, эта связь стала более очевидной, когда локус 8q24 выявленные геномом широкого исследования ассоциативного ряда злокачественных опухолей, включая колоректальный рак, было показано, что в связи с крайним вверх TCF7L2-связывающим элементом, вызывающим транскрипции MYC 23,24. Таким образом, существует огромный интерес в определении нижестоящих генов регулируется настоящим ключевым фактором транскрипции.
На основе опыта TCF7L2 как пример методологии, в настоящем документе описывается, как генерировать высокое качество чип ДНК. Чип проводили в толстой клеточной линии карциномы, НСТ116, для последующего секвенирования с целью создания высокого резольногоution карту генов связаны TCF7L2 25 в попытке получить дальнейшее понимание в свою ключевую роль в патогенезе сложных признаков.
протокол
1. Перекрестные ссылки хроматина
- Рост клеток в 100x20mm блюда культуре клеток. Количество клеток может быть в диапазоне от 1 до 10 миллионов клеток на чашку в зависимости от типа клеток. Приблизительно 2 миллиона клеток является достаточным для одного иммунопреципитации.
- Перекрестная ссылка клеток в 1% формальдегида в течение 10 мин при комнатной температуре при периодическом качалки.
- Гасят сшивания путем добавления конечной концентрации 125 мМ глицина и инкубируют в течение 5 мин при комнатной температуре.
- Мытье клеток с 1X фосфатным буферным раствором (PBS), дважды декантируют PBS, а затем добавить 0,2 мл PBS.
- Урожай клетки с пластиковым скребком клетки в микроцентрифужных трубки.
- Спин вниз клеток при 2000 оборотах в минуту в течение 5 минут при 4 ° C.
- Аспирируйте супернатант. Ресуспендируют клеток в буфере SDS Лизиса (1% SDS, 10 мМ ЭДТА, 50 мМ Трис-HCl, рН 8,1) для целого клеточного лизата или держать их в качестве гранул для ядерных добычи.
- Клетки могут быть сохранены при -80 ° С или можно перейти ImmediatЭли с ультразвуком.
2. Подготовьте ядрами (перейдите к шагу 3.5 для клеточный лизат)
- Дополнение буфере лизиса клеток (5 мМ труб рН 8,0, 85 мМ KCl, 0,5% NP-40) с 1X ингибитор протеиназы каждого эксперимента.
- Ресуспендируйте талой осадок клеток в примерно в 10 раз превышает объем гранул с буфера для лизиса клеток.
- Dounce-гомогенизации в 10 раз с пестиком затем инкубируют на льду в течение 10 мин.
- Центрифуга образца при 4000 оборотах в минуту в течение 5 мин при 4 ° С, отбросить супернатант, и сохранить ядерное гранул.
3. Ультразвуковая обработка *
- Разминка SDS буфера для лизиса и дополнения количество буфера для использования с ингибитор протеиназ.
- Ресуспендируют осадок в ядерной SDS буфера для лизиса (приблизительно 0,5 мл буфера на 1-10 миллионов клеток)
- Инкубируют на льду в течение 10 мин.
- Добавить 0,5 мл аликвоты проб в микроцентрифужных пробирках.
- Разрушать ультразвуком на льду использованием ультразвукового Misonix 30 с и на 45 секс с амплитудой установку 2. Количество циклов для идеального размера фрагмента может быть определено первым опробовать различные номера цикла (например 2, 4, 8, 12, 16, 20 или более циклов). Другой марки ультразвуковой могут быть использованы, однако, условия будут меняться. Экспериментирование с количеством циклов и количество времени включения и выключения должны выполняться для того, чтобы определить идеальных условиях.
- Собирают 20 мкл каждого образца, чтобы проверить результаты обработки ультразвуком и сделать количественного. Остальные образцы можно хранить при -80 ° С.
- Развести 20 мкл образца, добавляя 30 мкл буфера 0.1X TE.
- Лечение образца с 1 мкл РНКазы А при 37 ° С в течение 1 часа, затем добавляют 1 мкл протеиназы К и инкубируют при 62 ° С в течение 2 часов.
- Выполнить 20 мкл образца на 2% агарозном геле.
- Очищают оставшееся количество образца с QIAquick набора для очистки ПЦР затем количественно NanoDrop использованием спектрофотометра.
* Для родных ChIP, microccocal нуклеазой качестве альтернативы может использоваться для сдвига ДНК.
4. Блок агарозы *
- Если бусы уже заблокированы, перейдите к шагу 5.1.
- Использование белка или белок G агарозы. За 5 иммуноосаждения (IPS), использовать 600 мкл 50% шарик суспензии (300 мкл из гранул)
- Для мытья бусы, спина их вниз на 800 оборотов в минуту в течение 1 мин при 4 ° С и отбросить супернатант. Добавить немного больше, чем 2 мл чип Буфер для разведения (0,01% SDS, 1,2 мМ ЭДТА, 167 мМ NaCl, 1,1% Тритон Х-100, 16,7 мМ Трис-HCl, рН 8,1) и смесь медленно обращения трубки 10 раз. Спин снова при 800 оборотах в минуту в течение 1 минуты при 4 ° С и отбросить супернатант. Повторите мыть 2 раза.
- Блок бусы, вращая при 4 ° С в течение ночи в блокирующем растворе. См. Таблицу 1 для рецепта блокирующий раствор.
5. Предварительно ясно хроматина
- Оттепель ультразвуком хроматина на льду.
- Спином вниз при 12000 оборотах в минуту Fили 10 мин при 4 ° С, затем помещают на лед сразу же, чтобы удалить SDS (белые гранулы).
- Сбор супернатант, отбросить гранул, и комбинировать образцы, если это необходимо.
- Выньте суммы, необходимые для эксперимента на основе расчетов (1-10 мкг хроматина в IP).
- Развести хроматина 10X чип буфером для разведения дополнен ингибитора протеиназ.
- Добавить 100 мкл заблокирован шариков на IP.
- Поворот при 4 ° С в течение 1 часа.
6. Иммунопреципитация
- Спин вниз образцов при 800 оборотах в минуту в течение 1 мин и супернатант перенос т в свежую пробирку.
- Спином вниз супернатанта при 800 оборотах в минуту в течение 1 мин и перевод на другую чистую пробирку.
- Скидка 20 мкл супернатанта, в качестве входной контроль, при температуре -20 ° C.
- Аликвотные хроматина количеству IP-адреса должно быть сделано в эксперименте.
- Добавить 2 мкг антитела на 1-10 мкг хроматина в каждом образце.
- Инкубируют в течение ночи 4 ° С с вращением.
- Добавить 100 мкл заблокирован бисера каждому IP образца.
- Инкубировать в течение 1 часа при 4 ° С с вращением.
- Пелле бус счет остановки при 800 оборотах в минуту в течение 1 мин и распоряжаться им, как большая часть супернатанта насколько это возможно.
- Вымойте бисером однажды с низким содержанием соли иммунных комплексов промывочного буфера. Добавить 1 мл буфера в каждую пробирку; вращаться при комнатной температуре в течение 5-8 мин; спином вниз при 800 оборотах в минуту в течение 1 мин, а затем отбросить супернатант. Повторите мыть один раз с высоким содержанием соли иммунных комплексов буфера и LiCl иммунных комплексов буфера и дважды ТЕ-буфером в общей сложности 5 промывок (табл. 2).
7. Элюирование
- Оттепель входные выборки от предыдущего дня, чтобы быть обработаны Элюанты.
- Сделать свежего буфера для элюции (табл. 3).
- Сделайте мастер-микс достаточного буфера для элюции необходимые для IP-адреса и образцы входной контроль плюс 1-2 дополнительных образцов.
- Добавить 100 мкл буфера для элюции каждый IP-образца и инкубировали при комнатной температуре в течение 15 мв с вращением.
- Спин вниз со скоростью 800 оборотов в минуту в течение 1 мин и добавляют супернатант в новую пробирку.
- Добавить еще 100 мкл буфера для элюции в каждую пробирку шариков и инкубируют при комнатной температуре в течение 15 мин с вращением.
- Vortex в течение 15 секунд после инкубации; замедления вращения при 5000 оборотах в минуту в течение 1 мин, затем соединить супернатанта с надосадочной жидкостью из первого элюирования. (Убедитесь, что в нем нет, оставшиеся шарики в супернатантах. Если вы не уверены, спин вниз супернатант снова на 5000 оборотов в минуту в течение 1 мин и собирают супернатант в новую пробирку.
- Добавить 180 мкл буфера для элюции 20 мкл образцов управления вводом.
8. Обратные сшивки
- К 200 мкл элюентами и входного контроля, добавить 8 мкл 5 М NaCl.
- Уплотнение труб с парафином и инкубируют на водяной бане при 65 ° С в течение ночи.
9. Очистки ДНК
- Лечение каждого образца с 1 мкл РНКазы А в течение 1 часа при 37 ° С.
- Добавить 4 μл 0,5 М ЭДТА, 8 мкл 1М Трис-HCl, перемешивают, затем добавляют 1 мкл протеиназы К к каждому образцу и инкубируют при 45 ° С в течение 2 часов.
- Очищают QIAquick образцов с использованием набора для очистки ПЦР. Образцы могут быть сохранены при -20 ° С и ПЦР проверки может быть сделано на более поздний срок.
* Кроме того, чип класса магнитных гранул может быть использован вместо агарозном для иммунопреципитации части.
10. ПЦР проверки
- Для контроля ПЦР, используйте праймеры для регионов, как известно, связан интерес белка. Кроме того, используйте праймеры для необязательного регионов в качестве отрицательного контроля.
- Перемешайте реагенты для реакции. Развести входного образца при 1:100 (табл. 3).
- Запустить реакцию. ПЦР программы:
Шаг 1: 94 ° C 3 мин
Шаг 2: 94 ° С 20 сек
59 ° C 30 сек
72 ° С 30 сек
(Повторите шаг 2 для по крайней мере 30 CYCLES)
Шаг 3: 72 ° С 2 мин
- Выполнить образцов на 1% агарозном геле.
- Обогащение также может быть определена количественно с ПЦР в реальном времени.
Результаты
После того, хроматин был ультразвуком и были обработаны РНКазой и протеиназы, образцы подвергали электрофорезу на 2% агарозном геле должна представлять мазка с основной массой ДНК в нужный размер. Если несколько различных циклов тестирования, постепенное снижение размера следует рассматривать как с увеличением числа циклов (рис. 2).
После завершения иммунопреципитации часть протокола обогащение может либо быть проверены с помощью ПЦР или ПЦР в реальном времени. Для ПЦР образцы работают на агарозном геле должно быть полос в Входные и чип (с использованием антител для белок, который TCF7L2 в данном случае) образца переулков и ничего или в крайнем случае, очень слабая полоса (фоновый шум) в IgG (отрицательный) контрольной полосе для положительного связывающей области. Для отрицательной области связывания должно быть очень слабым или вообще не группа для контроля IgG и чип переулков. Там должна быть полосы в полосу входного (рис. 3).
На рисунке 4 показаны же образцов рассмотрены ПЦР в реальном времени. Как и на предыдущем рисунке, не должно быть значительным кратное обогащение положительной области связывания для чипа образца по сравнению с контрольным IgG. Кроме того, должно быть очень мало обогащения, если таковые имеются, рассматривается в отрицательной области связывания.
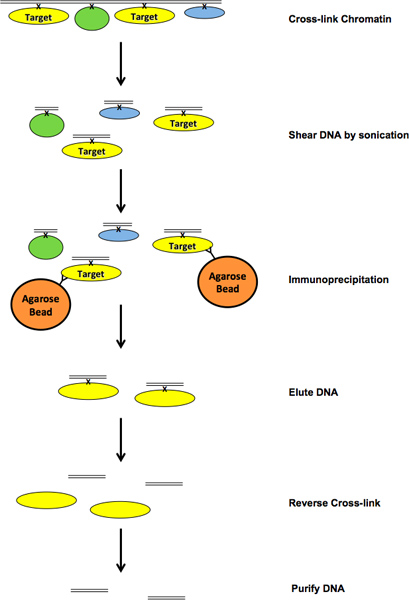
Рисунок 1. Технологическая схема чипа процесса. Нажмите здесь, чтобы увеличить рисунок .
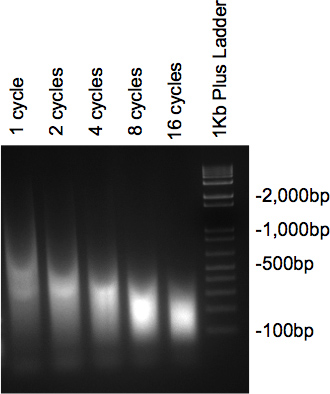
Рисунок 2. Гель проверить ДНК ультразвуком.
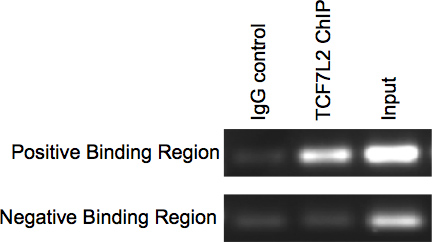
Рисунок 3. ПЦР Проверка чипа.

Рисунок 4. ПЦР в реальном времени из TCF7L2 чип.
| Реагент | Объем |
| Из гранул | 300 мкл |
| BSA (50 мг / мл) | 30 мкл |
| 100X ингибитор протеиназ | 10 мкл |
| ChIP буфером для разведения | 660 мкл |
| Общий | 1000 мкл |
Таблица 1. Рецепт для блокирования агарозы.
| Буфер | Компоненты |
| Низким содержанием соли иммунных комплексов промывочного буфера | 0,1% SDS 1% Тритон Х-100 2 мМ ЭДТА 20 мМ Трис-HCl, рН 8,1 150 мМ NaCl |
| Высоким содержанием соли иммунных комплексов промывочного буфера | 0,1% SDS 1% Тритон Х-100 2 мМ ЭДТА 20 мМ Трис-HCl, рН 8,1 500 мМ NaCl |
| LiCl иммунных комплексов промывочного буфера | 0,25 М LiCl 1% NP-40 1% Deoxycholate 1 мМ ЭДТА 10 мМ Трис-HCl, рН 8,1 |
| ТЕ-буфера | 10 мМ Трис-HCl, рН 8,1 1 мМ ЭДТА, рН 8,0 |
Таблица 2. Буферы ChIP стирки.
| Реагент | Объем |
| 20% SDS буфера | 10 мкл |
| 1 М NaHCO 3 | 20 мкл |
| H 2 O | 170 мкл |
Таблица 3. Элюирующий буфер для одного IP.
| Реагент | 50 мкл реакции | 20 мкл реакции |
| Воды | 27 мкл | 10,8 мкл |
| 5X реакции ПЦР-буфер | 10 мкл | 4 мкл |
| MgCl 2 | 4 мкл | 1,6 мкл |
| дНТФ (10 мМ) | 1 мкл | 0,4 мкл |
| Грунтовка смеси (5 мкм каждый) | 2 мкл | 0,8 мкл |
| Taq (Promega горячего старта) | 1 мкл | 0,4 мкл |
| ChIP ДНК | 5 мкл | 2 мкл |
Таблица 4. Объемы реакции ПЦР.
Обсуждение
Это теперь возможно проводить генома профиля белок-ДНК взаимодействия ассоциации с помощью чип-далее, как было недавно продемонстрировано с другими факторами транскрипции 2,3. Ключ к успешным результатам секвенирования поколения высокого качества шаблона хроматина ДНК иммунопреципитацией.
Как только ДНК-матрицы была сформирована и установлено быть должным образом обогащается, то можно взять его в библиотеке для подготовки к последующему секвенирования. Например, можно использовать протокол секвенирование библиотеки предоставляемых поставщиком, Illumina. Выбор размера этой библиотеки могут быть выполнены с помощью электрофореза в геле и последующее удаление и очистку ДНК в ~ 200 - 700 п.н. диапазона. Уменьшение размера и сужению диапазона размеров ДНК, собранных из очисткой на геле предназначен для улучшения позиционное разрешение Обломок-сл. По обогащения для небольших частей входных ДНК связан с коэффициентом десятичногоересть, можно было бы ожидать, что место расположения получит разрешение. Ужесточение выбор размера также улучшает равномерность размеров молекулярных колоний производится на платформе Illumina. Такая однородность размера колонии также увеличивает эффективное число чтение получены. Более короткие размер входного ДНК также производит более надежные колонии на платформе Illumina, и это может означать, что более короткие части ДНК в любом заданном распределении входной выборки будут представлены более эффективно в выходной последовательности, чем больше входной части из того же распределения.
Биоинформатики подходы к "следующего поколения" Анализ последовательности продолжают развиваться, и многие производители делает их программного обеспечения с открытым исходным кодом для дальнейшей доработки. Можно преобразовать говорится, что карта уникальных местах в геномной ДНК профиль фрагмента перекрытия. Значительные пики могут быть идентифицированы по threshholding профилей на высоте, эквивалентной примерно ложного обнаружения. Положение SPecific матрицы частот, полученных из этой работы могут быть использованы для идентификации и локализации ДНК-связывающих сайтов в человеческом геноме для данного фактора.
Но нужно быть осторожными в отношении того, какие факторы одна хочет учиться с чипом-след. Прежде чем приступать к такого исследования необходимо оценить, если антитела на рынке, который будет доступен в чипе установку в качестве бедных антитело может иметь весьма пагубные последствия для своих экспериментальных результатов. Кроме того, следует учитывать, если есть сращивание изоформы белка в исследовании, более того, TCF7L2 как известно, имеет множество изоформ поэтому мы особенно осторожным при выборе антитела, которые связаны с аминокислотами постоянно присутствует во всех основных изоформ этого фактора транскрипции 25.
Таким образом, сочетание иммунопреципитации хроматина и сверхвысокой пропускной последовательности (чип-далее) может определять и белок-ДНК взаимодействия в данной ткани или СELL линии. Мы наметили, как генерировать высокие качества щепы шаблон для последующего секвенирования.
Раскрытие информации
Авторы заявляют, что у них нет конкурирующих финансовых интересов.
Благодарности
Работа выполнена при поддержке Института развития премию из Детской больницы Филадельфии.
Материалы
| Name | Company | Catalog Number | Comments |
| QIAquick PCR Purification Kit | Qiagen | 28104 | |
| EZ-ChIP Kit | Millipore | 17-371 | |
| GoTaq Hot Start Polymerase | Promega | M5001 | |
| Misonix Sonicator | Qsonica | XL-2000 | |
| NanoDrop 1000 Spectrophotometer | Thermo-Scientific | ||
| Positive control primer sequences (TCF7L2-1) Forward- 5'-TCGCCCTGTCAATAATCTCC-3' Reverse- 5'-GCTCACCTCCTGTATCTTCG-3' Negative control primer sequences (CTRL-1) Forward-5'-ATGTGGTGTGGCTGTGATGGGAAC-3' Reverse- 5'-CGAGCAATCGGTAAATAGGTCTGG-3' | |||
Ссылки
- Odom, D. T., et al. Control of pancreas and liver gene expression by HNF transcription factors. Science. 303, 1378-1381 (2004).
- Johnson, D. S., Mortazavi, A., Myers, R. M., Wold, B. Genome-wide mapping of in vivo protein-DNA interactions. Science. 316, 1497-1502 (2007).
- Robertson, G., et al. Genome-wide profiles of STAT1 DNA association using chromatin immunoprecipitation and massively parallel sequencing. Nature Methods. 4, 651-657 (2007).
- Reich, N. C., Liu, L. Tracking STAT nuclear traffic. Nat. Rev. Immunol. 6, 602-612 (2006).
- Lodige, I., et al. Nuclear export determines the cytokine sensitivity of STAT transcription factors. The Journal of Biological Chemistry. 280, 43087-43099 (2005).
- Schroder, K., Sweet, M. J., Hume, D. A. Signal integration between IFNgamma and TLR signalling pathways in macrophages. Immunobiology. 211, 511-524 (2006).
- Vinkemeier, U. Getting the message across, STAT! Design principles of a molecular signaling circuit. The Journal of Cell Biology. 167, 197-201 (2004).
- Brierley, M. M., Fish, E. N. Stats: multifaceted regulators of transcription. J. Interferon Cytokine Res. 25, 733-744 (2005).
- Bentley, D. R. Whole-genome re-sequencing. Current Opinion in Genetics & Development. 16, 545-552 (2006).
- Grant, S. F., et al. Variant of transcription factor 7-like 2 (TCF7L2) gene confers risk of type 2 diabetes. Nature Genetics. 38, 320-323 (2006).
- Sladek, R., et al. A genome-wide association study identifies novel risk loci for type 2 diabetes. Nature. 445, 881-885 (2007).
- Wellcome Trust Case Control Consortium. Genome-wide association study of 14,000 cases of seven common diseases and 3,000 shared controls. Nature. 447, 661-678 (2007).
- Saxena, R., et al. Genome-wide association analysis identifies loci for type 2 diabetes and triglyceride levels. Science. 316, 1331-1336 (2007).
- Zeggini, E., et al. Replication of genome-wide association signals in UK samples reveals risk loci for type 2 diabetes. Science. 316, 1336-1341 (2007).
- Scott, L. J., et al. A genome-wide association study of type 2 diabetes in Finns detects multiple susceptibility variants. Science. 316, 1341-1345 (2007).
- Steinthorsdottir, V., et al. A variant in CDKAL1 influences insulin response and risk of type 2 diabetes. Nature Genetics. 39, 770-775 (2007).
- Salonen, J. T., et al. Type 2 Diabetes Whole-Genome Association Study in Four Populations: The DiaGen Consortium. American Journal of Human Genetics. 81, 338-345 (2007).
- Zeggini, E., McCarthy, M. I. TCF7L2: the biggest story in diabetes genetics since HLA. Diabetologia. 50, 1-4 (2007).
- Weedon, M. N. The importance of TCF7L2. Diabet. Med. 24, 1062-1066 (2007).
- Hattersley, A. T. Prime suspect: the TCF7L2 gene and type 2 diabetes risk. The Journal of Clinical Investigation. 117, 2077-2079 (2007).
- Yochum, G. S., et al. Serial analysis of chromatin occupancy identifies beta-catenin target genes in colorectal carcinoma cells. Proceedings of the National Academy of Sciences of the United States of America. 104, 3324-3329 (2007).
- Duval, A., Busson-Leconiat, M., Berger, R., Hamelin, R. Assignment of the TCF-4 gene (TCF7L2) to human chromosome band 10q25.3. Cytogenet. Cell Genet. 88, 264-265 (2000).
- Pomerantz, M. M., et al. The 8q24 cancer risk variant rs6983267 shows long-range interaction with MYC in colorectal cancer. Nature Genetics. 41, 882-884 (2009).
- Tuupanen, S., et al. The common colorectal cancer predisposition SNP rs6983267 at chromosome 8q24 confers potential to enhanced Wnt signaling. Nature Genetics. 41, 885-890 (2009).
- Zhao, J., Schug, J., Li, M., Kaestner, K. H., Grant, S. F. Disease-associated loci are significantly over-represented among genes bound by transcription factor 7-like 2 (TCF7L2) in vivo. Diabetologia. 53, 2340-2346 (2010).
- Benjamini, Y., Yekutieli, D. Quantitative trait Loci analysis using the false discovery rate. Genetics. 171, 783-790 (2005).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены