Method Article
높은 처리량 시퀀싱을위한 고품질 염색질 면역 침전의 DNA 템플릿의 생성 (칩 SEQ)
요약
염색질의 immunoprecipitation과 매우 높은 처리량 시퀀싱 (칩 SEQ)의 조합은 특정 조직이나 세포주에서 단백질 DNA 상호 작용을 파악하고 매핑 할 수 있습니다. 설명은 예를 들어 전사 인자 TCF7L2 경험을 사용하여 다음 순서를위한 고품질 칩 템플릿을 생성하는 방법이다.
초록
염색질의 immunoprecipitation (칩)을 결합하고 대규모 병렬 염기 서열은 생체 내 전사 인자에 의해 구속 포유류 DNA 서열의 레퍼토리를 식별하기 위해 활용 될 수있는 칩 시퀀싱 (칩 SEQ) 방법은 직접 전체 게놈 범위를 제공합니다. "다음 세대"게놈 시퀀싱 기술은 할 수 순서의 양에 크기가 증가 1-2 순서를 제공하는 비용 효율적 따라서 직접적으로 포유류의 효과적인 프로파일에 대한 전체 게놈의 범위를 제공하는 칩 SEQ 방법을 허용하는 오래된 기술을 통해 생성 단백질 DNA 상호 작용.
성공적인 칩 서열 방식의 경우, 하나는 고품질의 칩 DNA 템플릿 최적의 시퀀싱 결과를 얻기 위해 생성해야합니다. 설명이 가장 강하게 제 2 형 당뇨병, 즉 전사 인자의 전사 인자 2 7처럼 (TCF7L2의 병인에 관여하는 유전자의 단백질 제품과 경험을 기반으로합니다). 이 요소는 각종 암에 연루되어있다.
설명은 병인의 핵심 역할에서 더 통찰력을 제공, TCF7L2 구속 유전자를 확인하는 순서를 통해 고해상도지도를 구축하기 위해 대장 암 세포주 HCT116에서 파생 된 고품질의 칩 DNA 템플릿을 생성하는 방법입니다 복잡한 특성의.
서문
몇 년 동안, 특히 특정 단백질 게놈 넓은 전사 인자 클래스의 사람들에 의해 바인딩 조절 유전자의 집합을 식별하는 unmet 필요가있다.
오돔 등. 1 체계적으로 인간의 간 및 췌장에서 미리 지정된 전사 규제에 의해 점령 유전자를 식별하는 발기인 마이크로 어레이와 함께 염색질의 immunoprecipitation을 (칩)를 사용. 그 후, 존슨 등 2. 종합적으로 전체 포유류의 게놈에 걸쳐 단백질 DNA 상호 작용을 매핑하기 위해 직접 매우 높은 처리량 DNA 시퀀싱 (칩 SEQ)를 기반으로 대규모 염색질 immunoprecipitation의 분석을 개발했습니다. 테스트 케이스로, 그들은 생체 내에서 인간 게놈 1946 년 위치에 신경 제한적인 소음 계수 (NRSF)의 바인딩을 매핑됩니다. 데이터는 내가 모두를 촉진 바인딩 위치 선명한 해상도 (+ 50 염기쌍)를 표시주제 및 NRSF 결합 모티프의 식별 solation. 이 칩 SEQ 데이터는 높은 민감도와 특이도 및 통계적 신뢰도 (P <10 -4), 새로운 후보 상호 작용을 추론에 중요한 특성을 가지고 있었다.
로버트슨 등. 3도 STAT1 대상에 매핑하기 위해 칩 SEQ를 사용하여 인터페론-γ (IFN-γ) 자극 및 생체 내에서 인간의 헬라 S3 세포를 자극받지. 칩 서열, 15.1 및 12,900,000 고유 매핑 순서 읽고, 0.001의 추정 거짓 발견 속도를 사용하여, 그들은 각각 자극과 자극받지 않은 세포에서 41,582 및 11,004 추정 STAT1 결합 영역을 확인했다. STAT1 인터페론 반응 결합 부위 4-8를 포함하는 것으로 알려진 34 유전자 좌의, 칩 SEQ 24 (71 %)를 발견했다. 칩 SEQ 대상은 알려진 STAT1 바인딩 모티프와 유사한 서열에 충실 하였다. 기존의 두 칩 PCR 데이터와 비교 제안 설정이 칩 SEQ 민감도는 70 %와 92 %, 특이도했다 적어도 95 % 사이였다. 또한, 그것은 칩 SEQ 깊이를 연속으로 증가 낮은 분석의 복잡성과 민감성을 모두 제공한다는 것은 분명했습니다.
등 "차세대"게놈 시퀀싱 기술은 9 이전 기술을 통해 생성 된 비용 효율적으로 할 수 순서의 양에 크기가 증가 1-2 주문을 제공하기 때문에. 칩 SEQ 방법은 따라서 직접 포유류 단백질 DNA 상호 작용 3 효과적인 프로파일에 대한 전체 게놈의 범위를 제공합니다.
2006 년 전사 인자 2 7처럼 (TCF7L2) 제 2 형 당뇨병 유전자 변형의 강한 협회는 10 발견되었다. 다른 연구자들은 이미 독립적으로 자연 11,12에 발표 된 제 2 형 당뇨병의 첫 번째 게놈 넓은 협회 연구에서, 흥미롭게도 다른 인종이 발견을 복제하고있다 과학 13 ~ 15 및 16, 17 위로, 강한 협회는 TCF7L2와 참이었다;이 지금은 18 ~ 20 연대를 제 2 형 당뇨병에서 가장 중요한 유전 발견으로 간주됩니다. 또한, TCF7L2은 암 위험 21,22에 연결되었습니다, 대장 암 등 암의 수의 게놈 넓은 협회 연구에 의해 밝혀 8q24 현장은 극도의 상류에 의한 것으로 표시되었을 때 실제로,이 연결은 더 분명되었다 TCF7L2 바인딩 요소는 MYC 23,24의 전사를 운전. 등이 주요 전사 인자에 의해 조절 하류 유전자를 결정에 큰 관심이있다.
방법의 예로 TCF7L2 경험을 바탕으로, 본 논문은 높은 품질의 칩 DNA 템플릿을 생성하는 방법을 설명합니다. 칩은 높은 레솔을 빌드 순서에서 다음 순서를 위해, 대장 암 세포주 HCT116에서 실시되었다복잡한 특성의 병인에있는 그것의 중요한 역할에 추가 통찰력을 얻을 수있는 노력에 TCF7L2 (25)에 의해 구속 유전자의 ution지도.
프로토콜
1. 상호 링크 염색질
- 100x20mm 세포 배양 접시에 세포를 성장. 세포의 양이 1 세포의 종류에 따라 접시 당 1,000 만 셀에 이르기까지 다양하실 수 있습니다. 약 2 백만 세포는 하나의 면역 침전 충분합니다.
- 가끔 흔들 실온에서 10 분 동안 1 %의 포름 알데히드의 가교 세포.
- 상온에서 5 분 동안 125 mM의 글리신과 부화의 최종 농도를 추가하여 상호 연결을 끄다.
- 1X 인산염과 세포를 씻으은 가만히 따르다 PBS, 두 번 식염수 (PBS)를 버퍼, 그리고 PBS의 0.2 ML를 추가합니다.
- microcentrifuge 관에 플라스틱 셀 스크레이퍼 수확 세포.
- 4에서 5 분간 2,000 rpm으로 세포를 스핀 다운 ° C.
- 뜨는을 대기음. 전체 셀에 대한 SDS 용해 버퍼에있는 세포를 resuspend (1 % SDS, 10MM EDTA의 50mm 트리스 - 염산 산도 8.1) 해물이나 핵 추출 펠렛으로 보관하십시오.
- 세포 ° C 또는 하나의 immediat를 진행할 수 -80 저장할 수 있습니다초음파와 엘리.
2. (전체 세포 파쇄물을 위해 3.5 단계로 진행) 핵을 준비
- 1X 단백질 분해 효소 억제제 각 실험로 (5 개 mm 파이프 산도 8.0, 85 MM의 KCl을, 0.5 % NP-40) 세포 용해 버퍼를 보충합니다.
- 약 10 배 세포 용해 버퍼 펠릿 볼륨 해동 세포 펠렛을 resuspend을.
- 유 봉 다운스 - 균질화 10 배는 10 분 동안 얼음에 품어.
- 4에서 5 분 ° C에 대한 4,000 rpm에서 샘플을 원심 분리기, 상층 액을 버리고, 핵 펠렛을 저장합니다.
3. 초음파 *
- SDS의 용해 버퍼 및 단백질 분해 효소 억제제로 사용되는 버퍼의 보조 금액을 따뜻하게.
- SDS 용해 버퍼를 Resuspend 핵 펠렛 (1-10000000 세포 당 버퍼의 0.5 ML)
- 10 분 동안 얼음에 품어.
- 의 microcentrifuge 튜브에 샘플 0.5 ML의 aliquots를 추가합니다.
- 초와 45 초 30로 Misonix의 sonicator를 사용하여 젖은 얼음에 초음파 처리오프 2의 진폭 설정에서. 이상적인 조각의 크기 사이클 수를 먼저 다양한 사이클 번호 (예 : 2, 4, 8, 12, 16, 20 이상주기)을 시도하여 확인할 수 있습니다. sonicator의 다른 브랜드는, 그러나, 조건이 달라집니다 사용할 수 있습니다. 주기에 시간의 금액의 번호와 실험 및 해제는 이상적인 조건을 결정하기 위해 수행해야합니다.
- 초음파 결과를 확인하고, 정량화를 수행하기 위해 각 시료 20 μl를 수집합니다. 샘플의 나머지는 -80 ° C.에 저장할 수 있습니다
- 0.1X TE 버퍼 30 μl를 추가하여 샘플의 20 μl를 희석.
- RNase가 1 μL로 샘플을 치료 37 1 시간 동안 ° C는 2 시간 동안 62 ° C에서 단백질 분해 효소 K와 부화의 1 μl를 추가합니다.
- 2 % 아가로 오스 겔에 시료 20 μl를 실행합니다.
- 다음 Nanodrop 분광 광도계를 사용하여 정량화 QIAquick PCR 정화 키트와 샘플의 남은 양을 정화.
* 네이티브 치에 대한P, microccocal 핵산의 소화 전단 DNA를에 선택적으로 사용할 수 있습니다.
4. 차단 아가로 오스 비즈 *
- 구슬이 이미 차단 된 경우, 5.1 단계로 진행합니다.
- 단백질 또는 단백질 G 아가로 오스를 사용합니다. 5 immunoprecipitations를 들어 IPS (), 50 %의 비드 슬러리 (300 μL 비드 펠렛) 600 μl를 사용
- 구슬을 씻어, 4 ° C에서 1 분간 800 rpm으로 그들을 회전하고 상층 액을 버린다. 추가 약간 더 2보다 ML 칩 희석 버퍼 (0.01 % SDS, 1.2 mM의 EDTA, 167 mM의 NaCl을 1.1 % 트리톤 X-100, 16.7 MM 트리스 - 염산 산도 8.1) 천천히 반전 튜브 10X로 섞는다. 4 ° C에서 1 분간 800 rpm으로 다시 스핀 다운 및 뜨는을 폐기하십시오. 세척 2 번 이상 반복합니다.
- 솔루션을 차단에 4 ° C에서 하룻밤 회전 구슬을 차단합니다. 차단 솔루션의 조리법은 표 1을 참조하십시오.
5. 사전 맑은 염색질
- 해동 얼음에 염색질을 초음파.
- 12,000 RPM의 F에서 스핀 다운또는 4시 10 분 ° C 다음 SDS (흰색 펠렛)를 제거하는 즉시 얼음에 넣어.
- 상층 액을 수집, 펠렛을 폐기하고, 필요한 경우 샘플을 결합합니다.
- 계산 (IP 당 염색질의 1-10 UG)에 따라 실험에 필요한 금액을 꺼냅니다.
- 단백질 분해 효소 억제제와 보완 칩 희석 버퍼에 배 염색질을 희석.
- IP 당 차단 구슬의 100 μl를 추가합니다.
- 1 시간 동안 4 ° C에서 회전합니다.
6. 면역 침전
- 1 분간 800 rpm으로 샘플을 스핀 다운과 신선한 튜브에 뜨는을 전송합니다.
- 1 분 다른 깨끗한 튜브로 전송을 위해 800 rpm으로 상등액 스핀 다운.
- -20 ° C.에서 입력 컨트롤 역할을, 상층 액 20 μl를 저장
- 실험에서 수행 할 IP의 개수로 나누어지는 염색질합니다.
- 각 샘플에 염색질의 1-10 UG 당 항체의 2 UG를 추가합니다.
- 회전 하룻밤 4 ° C를 품어.
- 각 IP 샘플 차단 비즈 100 μl를 추가합니다.
- ° C 회전 4에서 1 시간 동안 배양한다.
- 펠릿 1 분 동안 800 rpm으로 스핀 다운 가능한 한 상층 액만큼을 취소하여 구슬.
- 낮은 소금 면역 복합체 세척 버퍼를 한 번 구슬을 씻으십시오. 각 튜브에 버퍼 1 ML을 추가, 5-8 분 동안 상온에서 회전, 1 분 동안 800 rpm으로 스핀 다운 후 상층 액을 버린다. 높은 소금 면역 복합체 세척 버퍼 LiCl을 면역 복합체 세척 버퍼를 한 번 씻어 반복 회 5 세척 (표 2)의 총 TE 버퍼와 함께.
7. 용출
- 전날 해동 입력 샘플 eluants으로 처리 할 수 있습니다.
- 용출 버퍼 신선한 (표 3)을 확인합니다.
- IP를 및 입력 컨트롤 샘플 플러스 1-2 추가 샘플에 필요한 충분한 용출 버퍼의 마스터 믹스를 확인하십시오.
- 각 IP 샘플 100 μL 용출 버퍼를 추가, 15 m에 대하여 상온에서 배양회전합니다.
- 1 분간 800 rpm으로 스핀 다운과 신선한 튜브에 뜨는을 추가합니다.
- 구슬의 각 튜브에 용출 버퍼의 또 다른 100 μl를 추가하고 회전 15 분간 실온에서 알을 품다.
- 부화 후 15 초 동안 소용돌이, 1 분 동안 5,000 rpm으로 스핀 다운, 다음 첫 번째 용출의 상층 액과 상층 액을 결합합니다. (무 상층 액에있는 구슬을 통해이 남아 있는지 확인합니다. 확실하지 않으면 1 분간 5,000 rpm에서 다시 상층 액을 아래로 회전하고 새로운 튜브에 뜨는를 수집합니다.
- 입력 제어 샘플 20 μL로 용출 버퍼 180 μl를 추가합니다.
8. 상호 링크를 역방향
- eluants 및 입력 컨트롤의 200 μL에, 5 M NaCl을 8 μl를 추가합니다.
- 65 ° C 하룻밤 물에 목욕 파라 필름 (parafilm)와 부화와 인감 튜브.
9. DNA 정제
- 37 1 시간 동안의 RNase의 1 μL 각 샘플을 치료 ° C.
- 4 μ 추가0.5 M EDTA (에틸렌 다이아 민 테트라 초산), 13 μL 1M 트리스 - 염산, 혼합,의 L는 2 시간 동안 45 ° C에서 각각의 샘플과 부화에 단백질 분해 효소 K 1 μl를 추가합니다.
- QIAquick PCR의 정제 키트를 사용하여 샘플을 정화. 샘플은 추후에 수행 할 수 있습니다 -20 ° C와 PCR 검사에 저장할 수 있습니다.
* 또한, 칩 고급 자석 구슬은 면역 침전 부분에 대한 아가의 장소에서 사용할 수 있습니다.
10. PCR 검사
- PCR 검사에 대한 관심의 단백질에 결합하는 것으로 알려져 지역을 위해 프라이머를 사용합니다. 또한, 음성 대조군으로 구속력이 지역의 프라이머를 사용합니다.
- 반응 시약을 혼합합니다. 1:100 (표 3)에서 입력 샘플을 희석.
- 반응을 실행합니다. PCR 프로그램 :
1 단계 : 94 ° C 3 분
2 단계 : 94 ° C 20 초
59 ° C 30 초
72 ° C 30 초
(적어도 30 CYCL에 대해 2 단계를 반복ES)
3 단계 : 72 ° C 2 분
- 1 % 아가 로스 겔에서 샘플을 실행합니다.
- 농축은 실시간 PCR을 정량적으로 측정 할 수 있습니다.
결과
염색질은 초음파되어 RNase의와 단백질 분해 효소로 처리되고 나면, 2 % 아가로 오스 겔에서 실행되는 샘플은 원하는 크기의 DNA 대량으로 얼룩을 제시해야한다. 여러 가지 사이클을 테스트하는 경우 크기가 점차 감소 사이클 수가 증가 (그림 2)로 볼 수 있어야합니다.
프로토콜의 면역 침전 부분을 완료 한 후 농축은 어느 PCR 또는 실시간 PCR 확인할 수 있습니다. 아가로 오스 겔에서 실행 PCR 샘플 입력 및 칩 대역 (이 경우 TCF7L2입니다 관심 단백질에 대한 항체를 사용하여) 샘플 차선 아무것도 또는 대부분에서,에서 매우 희미한 밴드 (잡음)가 있어야합니다 긍정적 인 바인딩 영역에 대한 IgG의 (음수) 제어 레인입니다. 음 바인딩 지역의 매우 희미한 또는 IgG의 제어 및 칩 차선 없음 대역이 있어야합니다. 입력 차선 (그림 3)의 대역이 있어야합니다.
그림 4는 실시간 PCR에 의해 검사 같은 샘플을 보여줍니다. 앞의 그림에서와 같이 IgG의 제어를 통해 칩 샘플에 대한 긍정적 인 바인딩 지역의 중요한 배 농축이 있어야합니다. 또한, 부정적인 바인딩 지역에서 본 아주 작은 농축있는 경우,이 있어야합니다.
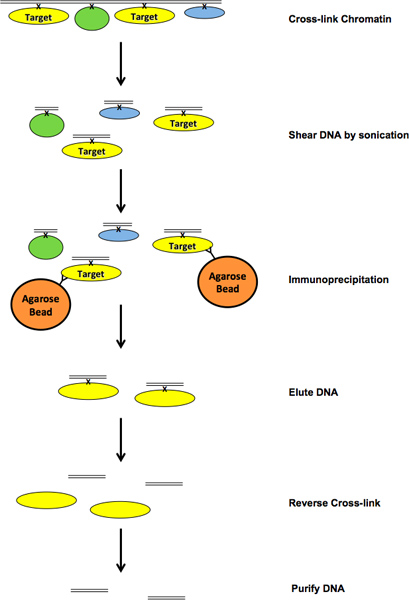
그림 1. 칩 공정의 흐름도. 큰 그림을 보려면 여기를 클릭하십시오 .
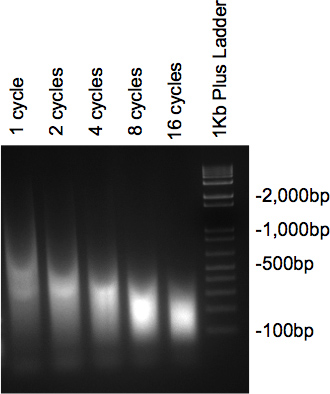
그림 2. 젤 DNA의 초음파의 점검.
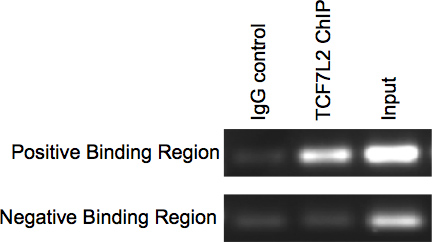
그림 3. PCR 칩의 점검.

4 그림. TCF7L2 칩의 실시간 PCR.
| 시약 | 음량 |
| 비드 펠렛 | 300 μL |
| BSA (50 MG / ML) | 30 μL |
| 100X 단백질 분해 효소 억제제 | 10 μL |
| 칩 희석 버퍼 | 660 μL |
| 합계 | 1,000 μL |
표 1. 아가를 차단위한 레시피.
| 버퍼 | 구성 요소 |
| 낮은 소금 면역 복합체 워시 버퍼 | 0.1 % SDS 1 % 트리톤 X-100 2 밀리미터 EDTA (에틸렌 다이아 민 테트라 초산) 20 MM 트리스 - 염산 pH를 8.1 150 mM의 NaCl을 |
| 높은 소금 면역 복합체 워시 버퍼 | 0.1 % SDS 1 % 트리톤 X-100 2 밀리미터 EDTA (에틸렌 다이아 민 테트라 초산) 20 MM 트리스 - 염산 pH를 8.1 500 mM의 NaCl을 |
| LiCl을 면역 복합체 워시 버퍼 | 0.25 M LiCl을 1 % NP-40 1 % Deoxycholate 1 ㎜ EDTA (에틸렌 다이아 민 테트라 초산) 10 MM 트리스 - 염산 pH를 8.1 |
| TE 버퍼 | 10 MM 트리스 - 염산 pH를 8.1 1 ㎜ EDTA (에틸렌 다이아 민 테트라 초산) 산도 8.0 |
표 2. 칩 세척 버퍼.
| 시약 | 음량 |
| 20 % SDS 버퍼 | 10 μL |
| 1 M NaHCO3를 | 20 μL |
| H 2 O | 170 μL |
표 3. 하나의 IP에 대한 용출 버퍼입니다.
| 시약 | 50 ㎕의 반응 | 20 ㎕의 반응 |
| 물 | 27 μL | 10.8 μL |
| 배 PCR 반응 버퍼 | 10 μL | 4 μL |
| MgCl 2 | 4 μL | 1.6 μL |
| dNTP (10 mM의) | 1 μL | 0.4 μL |
| 프라이머 믹스 (5 음 각) | 2 μL | 0.8 μL |
| DNA 형성 촉매 (Promega 사 Hotstart) | 1 μL | 0.4 μL |
| 칩 DNA | 5 μL | 2 μL |
표 4. PCR 반응 볼륨.
토론
아주 최근에 다른 전사 인자 2,3로 입증되었습니다로서, 지금은 칩 SEQ를 사용하여 단백질 DNA 상호 작용 협회의 게놈 전체 프로필을 수행 할 가능합니다. 성공적으로 시퀀싱 결과의 핵심은 높은 품질의 염색질의 immunoprecipitation의 DNA 템플릿의 세대입니다.
DNA 템플릿을 생성하고 적절하게 풍성하게 할 ascertained되면, 하나는 후속 시퀀싱 라이브러리 준비에 걸릴 수 있습니다. 예를 들어, 하나의 공급 업체 일루미나에서 제공하는 시퀀싱 라이브러리 프로토콜을 사용할 수 있습니다. 이 라이브러리의 크기 선택은 ~ 200 겔 전기 영동과 DNA의 연속 절단 및 정화하여 수행 할 수 있습니다 - 700 BP의 범위. 크기를 줄이고 젤 정화에서 수집 된 DNA의 크기 범위는 칩 서열의 위치 해상도를 개선하기위한 것입니다 축소. INT의 요소에 바인딩 입력 DNA의 작은 조각에 대한 풍부하여erest, 하나는 해당 사이트의 위치가 해상도를 얻을 것으로 기대된다. 타이트한 사이즈 선택은 Illumina의 플랫폼에서 생산 된 분자 식민지의 크기의 균일 성을 향상시킵니다. 이러한 식민지 크기의 균일도도 획득 효과적인 읽기 수를 증가합니다. 짧은 입력 DNA의 크기는 Illumina의 플랫폼에 더 강력한 식민지를 생성하고, 이것은 어떤 주어진 입력 샘플 분포에서 짧은 DNA 조각이 동일한 분포에서 이상 입력 조각보다 최종 시퀀스 출력에보다 효율적으로 표현 될 것입니다 의미합니다.
"차세대"서열 분석에 생물 정보학 접근 방식은 더 세련에 대한 자신의 소프트웨어를 오픈 소스 만드는 많은 공급 업체와 진화를 계속하고 있습니다. 하나는 DNA 단편 중복 프로파일에 고유 한 게놈 위치에 그지도를 읽고 변환 할 수 있습니다. 중요한 피크가 예상 거짓 발견 속도에 해당하는 높이에 threshholding 프로필에서 확인할 수 있습니다. 위치 SP이 작품에서 파생 된 ecific 주파수 행렬은 주어진 요소에 대한 인간 게놈에 걸쳐 DNA 결합 부위를 확인하고 지역화하는 데 사용할 수 있습니다.
그러나 한 존경과 함께 어떤 요인이 칩 서열에서 공부하는 한 가지 소원을.주의해야합니다 가난한 항체 하나의 실험 결과에 매우 해로운 영향을 미칠 수있는 항체, 칩 설정에서 사용할 수있는 시장에서 사용할 수있는 경우 이러한 연구에 착수하기 전에 하나를 평가해야한다. 연구중인 단백질의 결합 이소가있는 경우뿐만 아니라, 하나 고려해야한다, 우리가이 전사 인자의 주요 아형에 지속적으로 존재하는 아미노산에 결합하는 항체를 선택에 특히주의했다 그래서 실제로 TCF7L2 많은 아형이 알려져있다 25.
요약하면, 염색질의 immunoprecipitation과 매우 높은 처리량 시퀀싱 (칩 SEQ)의 조합은 특정 조직 또는 C 단백질 DNA 상호 작용을 파악하고 매핑 할 수 있습니다ELL 라인. 우리는 다음 순서를위한 고품질 칩 템플릿을 생성하는 방법을 설명했다.
공개
제작자들은 더 경쟁 금융 이익이 없다는 점 선언합니다.
감사의 말
작품은 필라델피아의 아동 병원에서 연구원 개발 포상에 의해 지원됩니다.
자료
| Name | Company | Catalog Number | Comments |
| QIAquick PCR Purification Kit | Qiagen | 28104 | |
| EZ-ChIP Kit | Millipore | 17-371 | |
| GoTaq Hot Start Polymerase | Promega | M5001 | |
| Misonix Sonicator | Qsonica | XL-2000 | |
| NanoDrop 1000 Spectrophotometer | Thermo-Scientific | ||
| Positive control primer sequences (TCF7L2-1) Forward- 5'-TCGCCCTGTCAATAATCTCC-3' Reverse- 5'-GCTCACCTCCTGTATCTTCG-3' Negative control primer sequences (CTRL-1) Forward-5'-ATGTGGTGTGGCTGTGATGGGAAC-3' Reverse- 5'-CGAGCAATCGGTAAATAGGTCTGG-3' | |||
참고문헌
- Odom, D. T., et al. Control of pancreas and liver gene expression by HNF transcription factors. Science. 303, 1378-1381 (2004).
- Johnson, D. S., Mortazavi, A., Myers, R. M., Wold, B. Genome-wide mapping of in vivo protein-DNA interactions. Science. 316, 1497-1502 (2007).
- Robertson, G., et al. Genome-wide profiles of STAT1 DNA association using chromatin immunoprecipitation and massively parallel sequencing. Nature Methods. 4, 651-657 (2007).
- Reich, N. C., Liu, L. Tracking STAT nuclear traffic. Nat. Rev. Immunol. 6, 602-612 (2006).
- Lodige, I., et al. Nuclear export determines the cytokine sensitivity of STAT transcription factors. The Journal of Biological Chemistry. 280, 43087-43099 (2005).
- Schroder, K., Sweet, M. J., Hume, D. A. Signal integration between IFNgamma and TLR signalling pathways in macrophages. Immunobiology. 211, 511-524 (2006).
- Vinkemeier, U. Getting the message across, STAT! Design principles of a molecular signaling circuit. The Journal of Cell Biology. 167, 197-201 (2004).
- Brierley, M. M., Fish, E. N. Stats: multifaceted regulators of transcription. J. Interferon Cytokine Res. 25, 733-744 (2005).
- Bentley, D. R. Whole-genome re-sequencing. Current Opinion in Genetics & Development. 16, 545-552 (2006).
- Grant, S. F., et al. Variant of transcription factor 7-like 2 (TCF7L2) gene confers risk of type 2 diabetes. Nature Genetics. 38, 320-323 (2006).
- Sladek, R., et al. A genome-wide association study identifies novel risk loci for type 2 diabetes. Nature. 445, 881-885 (2007).
- . Wellcome Trust Case Control Consortium. Genome-wide association study of 14,000 cases of seven common diseases and 3,000 shared controls. Nature. 447, 661-678 (2007).
- Saxena, R., et al. Genome-wide association analysis identifies loci for type 2 diabetes and triglyceride levels. Science. 316, 1331-1336 (2007).
- Zeggini, E., et al. Replication of genome-wide association signals in UK samples reveals risk loci for type 2 diabetes. Science. 316, 1336-1341 (2007).
- Scott, L. J., et al. A genome-wide association study of type 2 diabetes in Finns detects multiple susceptibility variants. Science. 316, 1341-1345 (2007).
- Steinthorsdottir, V., et al. A variant in CDKAL1 influences insulin response and risk of type 2 diabetes. Nature Genetics. 39, 770-775 (2007).
- Salonen, J. T., et al. Type 2 Diabetes Whole-Genome Association Study in Four Populations: The DiaGen Consortium. American Journal of Human Genetics. 81, 338-345 (2007).
- Zeggini, E., McCarthy, M. I. TCF7L2: the biggest story in diabetes genetics since HLA. Diabetologia. 50, 1-4 (2007).
- Weedon, M. N. The importance of TCF7L2. Diabet. Med. 24, 1062-1066 (2007).
- Hattersley, A. T. Prime suspect: the TCF7L2 gene and type 2 diabetes risk. The Journal of Clinical Investigation. 117, 2077-2079 (2007).
- Yochum, G. S., et al. Serial analysis of chromatin occupancy identifies beta-catenin target genes in colorectal carcinoma cells. Proceedings of the National Academy of Sciences of the United States of America. 104, 3324-3329 (2007).
- Duval, A., Busson-Leconiat, M., Berger, R., Hamelin, R. Assignment of the TCF-4 gene (TCF7L2) to human chromosome band 10q25.3. Cytogenet. Cell Genet. 88, 264-265 (2000).
- Pomerantz, M. M., et al. The 8q24 cancer risk variant rs6983267 shows long-range interaction with MYC in colorectal cancer. Nature Genetics. 41, 882-884 (2009).
- Tuupanen, S., et al. The common colorectal cancer predisposition SNP rs6983267 at chromosome 8q24 confers potential to enhanced Wnt signaling. Nature Genetics. 41, 885-890 (2009).
- Zhao, J., Schug, J., Li, M., Kaestner, K. H., Grant, S. F. Disease-associated loci are significantly over-represented among genes bound by transcription factor 7-like 2 (TCF7L2) in vivo. Diabetologia. 53, 2340-2346 (2010).
- Benjamini, Y., Yekutieli, D. Quantitative trait Loci analysis using the false discovery rate. Genetics. 171, 783-790 (2005).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유