Method Article
Generierung von High Quality Chromatin Immunpräzipitation DNA Vorlage für Hochdurchsatz-Sequenzierung (ChIP-seq)
In diesem Artikel
Zusammenfassung
Die Kombination von Chromatinimmunpräzipitation und ultra-Sequenzierung mit hohem Durchsatz (Chip-seq) zu identifizieren und abzubilden Protein-DNA-Wechselwirkungen in einem bestimmten Gewebe oder Zelllinie. Skizziert ist, wie man eine hohe Qualität ChIP Vorlage für anschließende Sequenzierung mit Erfahrung mit dem Transkriptionsfaktor TCF7L2 als Beispiel zu generieren.
Zusammenfassung
ChIP-Sequenzierung (ChIP-seq) Methoden direkt anbieten Ganzgenom Deckung, wo die Kombination Chromatinimmunpräzipitation (Chip) und massiv parallele Sequenzierung genutzt werden, um das Repertoire der Säuger-DNA-Sequenzen, die von Transkriptionsfaktoren in vivo gebunden zu identifizieren. "Next-Generation" Genom-Sequenzierung Technologien bieten 1-2 Größenordnungen Anstieg in Höhe von Sequenz, die sein können kostengünstig im Vergleich zu älteren Technologien erzeugt so dass für ChIP-seq Methoden, um direkt zur Verfügung Ganzgenom Abdeckung für wirksame Profilierung von Säugetieren Protein-DNA-Wechselwirkungen.
Für eine erfolgreiche ChIP-seq Ansätze, muss man erzeugen hochwertige ChIP DNA-Vorlage, um die besten Ergebnisse zu erhalten Sequenzierung. Die Beschreibung ist um Erfahrungen mit dem Proteinprodukt des Gens am stärksten in der Pathogenese von Diabetes Typ 2, nämlich der Transkriptionsfaktor Transkriptionsfaktor 7-like 2 (TCF7L2 gebracht basierend). Dieser Faktor ist auch in verschiedenen Tumoren in Verbindung gebracht.
Skizziert ist wie man qualitativ hochwertige ChIP DNA-Vorlage aus dem kolorektalen Karzinom Zelllinie HCT116 abgeleitet zu erzeugen, um eine hochauflösende Kartendarstellung durch Sequenzierung der Gene von TCF7L2 gebunden bestimmen zu bauen, geben weitere Einblicke in seine wichtige Rolle in der Pathogenese von komplexen Merkmalen.
Einleitung
Seit vielen Jahren hat es einen unerfüllten Bedarf, um den Satz von Genen, gebunden und durch ein bestimmtes Protein genomweiter, insbesondere in den Transkriptionsfaktor reguliert Klasse zu identifizieren.
Odom et al. 1 verwendet Chromatinimmunpräzipitation (Chip) mit Promoter Microarrays systematisch zu identifizieren, die Gene von vorgegebenen Transkriptionsfaktoren in der menschlichen Leber und Pankreas-Inseln besetzt kombiniert. Anschließend entwickelte Johnson et al. 2 eine groß angelegte Chromatinimmunpräzipitation Assay auf direkte Ultra Hochdurchsatz-DNA-Sequenzierung (ChIP-seq), um umfassend abzubilden Protein-DNA-Wechselwirkungen über die gesamte Säugergenomen basiert. Als Testfall sie in vivo der Bindung des Neuron-einschränkende Faktor Schalldämpfer (NRSF) bis 1946 Stellen im menschlichen Genom abgebildet. Die angezeigten Daten scharfen Auflösung von verbindlichen Position (+ 50 Basenpaare), die sowohl die i erleichtertsolation der Motive und der Identifizierung von NRSF-bindende Motive. Diese ChIP-seq Daten hatten auch hohe Sensitivität und Spezifität und statistische Sicherheit (P <10 -4), Eigenschaften, die wichtig für die Ableitung neuer Kandidat Wechselwirkungen sind.
Robertson et al. 3 auch ChIP-seq verwendet werden, um Ziele in der Karte STAT1 Interferon-γ (IFN-γ)-stimulierten und unstimulierten humanen HeLa S3-Zellen in vivo. Durch ChIP-seq, mit 15,1 und 12,9 Mio. eindeutig zugeordnet Sequenz liest, und eine geschätzte False Discovery Rate von weniger als 0.001, identifizierten sie 41.582 und 11.004 mutmaßliche STAT1-bindenden Regionen in stimulierten und unstimulierten Zellen. Von den 34 Loci bekannt STAT1 auf Interferon ansprechenden Bindungsstellen 4-8 enthalten, gefunden ChIP-seq 24 (71%). ChIP-seq Ziele wurden in Sequenzen ähnlich bekannt STAT1 Bindungsmotive bereichert. Vergleiche mit zwei bestehenden ChIP-PCR-Datensätze vorgeschlagendass ChIP-seq Sensitivität lag zwischen 70% und 92% und eine Spezifität von mindestens 95%. Darüber hinaus war es klar, dass ChIP-seq sowohl niedrige analytische Komplexität und Sensibilität, die mit zunehmender Tiefe Sequenzierung bietet.
Als solche "nächsten Generation" Genom-Sequenzierung Technologien bieten 1-2 Größenordnungen Anstieg in Höhe von Sequenz, die kostengünstig gegenüber älteren Technologien 9 erzeugt werden können. ChIP-seq Methoden daher direkt liefern Ganzgenom Abdeckung für wirksame Profilierung der Säuger Protein-DNA-Wechselwirkungen 3.
Im Jahr 2006 wurde eine starke Assoziation von Varianten in den Transkriptionsfaktor 7-like 2 (TCF7L2) Gen mit Typ-2-Diabetes 10 entdeckt. Andere Forscher haben bereits unabhängig diese Feststellung in verschiedenen Ethnien repliziert und interessanterweise von den ersten genomweiten Assoziationsstudien von Typ-2-Diabetes in Nature veröffentlicht 11,12 , Wissenschaft und anderswo 13-15 16,17 war die stärkste Assoziation mit der Tat TCF7L2, dies ist nun die wichtigste genetische Befund bei Typ-2-Diabetes als 18-20 Laufenden. Darüber hinaus hat TCF7L2 das Krebsrisiko 21,22 in Verbindung gebracht worden, ja, wurde diese Verbindung noch deutlicher, wenn der Locus 8q24 durch genomweite Assoziationsstudien von einer Reihe von Krebsarten, einschließlich kolorektalen Karzinomen ergab, wurde gezeigt, dass durch eine extreme stromaufwärts TCF7L2 bindende Element der Fahrt die Transkription von MYC 23,24. Daher besteht ein großes Interesse an der Feststellung der nachgeschalteten Gene durch diese Taste Transkriptionsfaktor reguliert.
Basierend auf den Erfahrungen mit TCF7L2 als Beispiel der Methodik, umreißt dieses Papier wie man qualitativ hochwertige ChIP-DNA-Template zu erzeugen. Chip wurde im kolorektalen Karzinom-Zelllinie HCT116, z. anschließende Sequenzierung, um Aufbau eines High-Resolution Karte der Gene durch TCF7L2 25 in dem Bestreben, weitere Einblicke in seine wichtige Rolle in der Pathogenese von komplexen Merkmalen ergeben gebunden.
Protokoll
Ein. Querverbindung Chromatin
- Wachsen Zellen in 100x20mm Zellkulturschalen. Die Menge der Zellen können von 1 bis 10 Millionen Zellen pro Schale je nach Zelltyp reichen. Etwa 2 Millionen Zellen ist ausreichend für eine Immunpräzipitation.
- Querverbindung Zellen in 1% Formaldehyd für 10 min bei Raumtemperatur unter gelegentlichem Schaukelstuhl.
- Quench Vernetzung durch Zugabe einer Endkonzentration von 125 mM Glycin und Inkubation für 5 min bei Raumtemperatur gerührt.
- Waschen der Zellen mit 1X Phosphate Buffered Saline (PBS) zweimal, dekantieren PBS, und fügen Sie 0,2 ml PBS.
- Ernten Sie die Zellen mit einem Kunststoff Zellenabstreifer in ein Mikrozentrifugenröhrchen.
- Spin down Zellen bei 2.000 rpm für 5 min bei 4 ° C.
- Saugen Sie den Überstand. Die Zellen in SDS-Lyse-Puffer (1% SDS, 10 mM EDTA, 50 mM Tris-HCl pH 8,1) für Ganzzelllysat oder halten sie als Pellet für nukleare Extraktion.
- Die Zellen können bei -80 ° C gespeichert werden, oder man kann immediat gehenEly mit Beschallung.
2. Bereiten Kerne (Fahren Sie mit 3.5 für Ganzzelllysat Schritt)
- Nachtrag Cell Lysis Buffer (5 mM PIPES pH 8,0, 85 mM KCl, 0,5% NP-40) mit 1X Proteinase-Inhibitor jedes Experiment.
- Resuspendieren aufgetaut Zellpellet in etwa das 10-fache Volumen Pellet mit Cell Lysis Buffer.
- Dounce nachhomogenisieren 10 mal mit Stößel dann auf Eis inkubieren 10 min.
- Zentrifuge Probe bei 4.000 rpm für 5 min bei 4 ° C, Überstand verwerfen und speichern Kernpellet.
3. Beschallung *
- Warm up SDS Lysis Buffer und Ergänzung Menge Puffer mit Proteinase-Inhibitor verwendet werden.
- Resuspendieren Kernpellet in SDS Lysis Buffer (ca. 0,5 ml Puffer pro 1-10.000.000 Zellen)
- Inkubation auf Eis für 10 min.
- 0,5 ml Aliquots der Proben zu Mikrozentrifugenröhrchen.
- Mit Ultraschall auf nassem Eis mit Misonix Ultraschallgerät mit 30 Sek. auf 45 Sek. undoff bei einer Amplitude von 2 Einstellung. Anzahl der Zyklen für ideal Fragmentgröße werden, indem zuerst versucht verschiedene Taktzahlen (zB 2, 4, 8, 12, 16 und 20 oder mehr Zyklen) ermittelt werden. Eine andere Marke von Ultraschallgerät verwendet, jedoch die Bedingungen variiert werden. Versuche mit der Anzahl der Zyklen und Zeit-und Ausschalten muß durchgeführt werden, um die optimalen Bedingungen zu bestimmen.
- Sammle 20 ul jeder Probe Beschallung Ergebnisse zu überprüfen und zu quantifizieren tun. Der Rest der Probe bei -80 ° C gelagert werden
- Verdünnen Sie die 20 ul Probe durch Zugabe von 30 ul 0,1 × TE-Puffer.
- Gönnen Probe mit 1 ul RNase A bei 37 ° C für 1 Stunde dann 1 ul Proteinase K und Inkubation bei 62 ° C für 2 Stunden.
- Führen Sie 20 ul der Probe auf einem 2% Agarosegel.
- Reinigen Sie die verbleibende Probe mit QIAquick PCR Purification Kit dann quantifizieren mit NanoDrop Spektralphotometer.
* Für nativen ChIP kann microccocal Nucleaseverdau alternativ zum Abscheren des DNA verwendet werden.
4. Blockieren Agarosekügelchen *
- Wenn Perlen bereits blockiert sind, gehen Sie zu Schritt 5.1.
- Verwenden Protein A oder Protein G-Agarose. Für 5 Immunpräzipitationen (IPs), verwenden Sie 600 ul 50% Kügelchenaufschlämmung (300 ul Wulst Pellet)
- Um die Perlen waschen, schleudern sie unten bei 800 rpm für 1 min bei 4 ° C und Überstand verwerfen. In etwas mehr als 2 ml ChIP Dilution Buffer (0,01% SDS, 1,2 mM EDTA, 167 mM NaCl, 1,1% Triton X-100, 16,7 mM Tris-HCl pH 8,1) zugeben und langsam umdrehen, die Röhre 10X. Spin down wieder bei 800 rpm für 1 min bei 4 ° C und Überstand verwerfen. Wiederholen Sie die Wäsche 2 weitere Male.
- Blockieren Perlen durch Drehen bei 4 ° C über Nacht in Blocking-Lösung. Siehe Tabelle 1 für das Rezept des Blocking-Lösung.
5. Pre-clear Chromatin
- Thaw beschallt Chromatin auf Eis.
- Spin down bei 12.000 rpm foder 10 min bei 4 ° C auf Eis sofort, SDS (weiß Pellet) entfernen setzen.
- Sammle Überstand verwerfen Pellet, und kombinieren Proben, wenn nötig.
- Nehmen Sie die Beträge für das Experiment auf Berechnungen (1-10 ug von Chromatin pro IP) benötigt.
- Verdünnen Chromatin 10X in ChIP Dilution Buffer mit Proteinase-Inhibitor ergänzt.
- In 100 ul blockiert Perlen pro IP.
- Drehen bei 4 ° C für 1 Stunde.
6. Immunpräzipitation
- Spin down Proben bei 800 rpm für 1 min und den Überstand in ein frisches Röhrchen.
- Spin down Überstand bei 800 rpm für 1 min und Übertragung auf ein anderes sauberes Röhrchen.
- Sparen Sie 20 ul der Überstand, die als Input-Steuerung dienen, bei -20 ° C.
- Aliquot des Chromatins der Anzahl von IPs in den Test erfolgen.
- In 2 ug Antikörper pro 1-10 ug von Chromatin zu jeder Probe.
- Inkubieren über Nacht bei 4 ° C Drehung.
- In 100 ul blockiert Perlen an jede IP-Probe.
- Inkubation für 1 h bei 4 ° C unter Rotation.
- Pellet die Perlen indem sie zum Stillstand bei 800 rpm für 1 min und entsorgen Sie so viel von der Überstand wie möglich.
- Waschen Sie Perlen einmal mit wenig Salz Immune Complex Waschpuffer. 1 ml Puffer in jedes Röhrchen, drehen bei Raumtemperatur 5-8 min; Spin-Down bei 800 rpm für 1 min, dann Überstand verwerfen. Wiederholen waschen einmal mit hoher Salz Immune Complex Waschpuffer und LiCl Immune Complex Waschpuffer und zweimal mit TE-Puffer für insgesamt 5 Wäschen (Tabelle 2).
7. Elution
- Thaw Eingang Proben aus vorherigen Tag, um mit den Fliessmitteln verarbeitet werden.
- Als Elutionspuffer frisch (Tabelle 3).
- Machen Sie einen Master-Mix von genug Elutionspuffer für IPs und Eingang Kontrollproben sowie 1-2 zusätzliche Proben benötigt.
- 100 l Elutionspuffer auf jedem IP Probe und Inkubation bei Raumtemperatur für 15 mMit Drehung.
- Spin down bei 800 rpm für 1 min und fügen Sie den Überstand in ein frisches Röhrchen.
- Einen weiteren hinzufügen 100 ul Elutionspuffer zu jedem Röhrchen von Perlen und Inkubation bei Raumtemperatur für 15 min mit der Drehung.
- Vortex für 15 sec nach der Inkubation; Abzentrifugieren bei 5000 Upm für 1 min, dann verbinden sich mit dem Überstand aus der ersten Elution Überstand. (Stellen Sie sicher, dass keine über Perlen in den Überständen links. Falls Sie unsicher sind, Spin-down Überstand erneut bei 5.000 rpm für 1 min und den Überstand in ein neues Röhrchen.
- In 180 ul Elutionspuffer auf die 20 ul Eingang Kontrollproben.
8. Reverse-Kreuz-link
- Zum 200 ul Elutionsmitteln und Eingabe-Steuerelemente, Add 8 ul 5 M NaCl.
- Seal-Röhrchen mit Parafilm und Inkubation im Wasserbad bei 65 ° C über Nacht.
9. DNA Purification
- Behandle jeden Probe mit 1 ul RNase A für 1 h bei 37 ° C.
- In 4 μl von 0,5 M EDTA, 8 ul 1 M Tris-HCl, mischen, dann fügen Sie 1 ul Proteinase K zu jeder Probe und Inkubation bei 45 ° C für 2 Stunden.
- Reinigen Sie die Proben mit dem QIAquick PCR Purification Kit. Die Proben können bei -20 ° C und PCR Check gespeichert werden können, zu einem späteren Zeitpunkt durchgeführt werden.
* Alternativ können ChIP-grade magnetischen Kügelchen anstelle Agarose für die Immunpräzipitation Teil verwendet werden.
10. PCR Check
- Für die PCR Überprüfung einfach Primer für Regionen bekannt, die durch das Protein von Interesse gebunden werden. Verwenden Sie außerdem Primer für nicht-bindenden Regionen als negative Kontrollen.
- Mischen Sie die Reagenzien für die Reaktion. Verdünnen Eingang Probe bei 1:100 (Tabelle 3).
- Führen Reaktion. PCR-Programm:
Schritt 1: 94 ° C 3 min
Schritt 2: 94 ° C 20 sec
59 ° C 30 sec
72 ° C 30 sec
(Wiederholen Sie Schritt 2 für mindestens 30 cycln)
Schritt 3: 72 ° C 2 min
- Führen Proben auf 1% Agarosegel.
- Die Anreicherung kann auch quantitativ mit Echtzeit-PCR bestimmt.
Ergebnisse
Sobald das Chromatin wurde mit Ultraschall behandelt wurde, und wurden mit RNase und Proteinase behandelt, sollten die Proben auf der 2% igen Agarosegel aufgetrennt ein Abstrich mit der Masse der DNA, die in der gewünschten Größe. Wenn mehrere Zyklen geprüft, ist eine allmähliche Abnahme in der Größe der Anzahl von Zyklen erhöht (Abbildung 2) zu sehen.
Nach Abschluss der Immunopräzipitation des Protokolls ist die Anreicherung kann entweder durch PCR oder Echtzeit-PCR überprüft. Für die PCR-Proben auf einem Agarosegel laufen sollte Bänder in der Eingangs-und Chip (unter Verwendung des Antikörpers für das interessierende Protein, das TCF7L2 in diesem Fall) Probenbahnen und nichts oder allenfalls eine sehr schwache Bande (Hintergrundrauschen) im BE IgG (negative) Kontrolle Fahrspur für die positive Bindung Region. Für die negative bindende Region sollte es sehr schwache oder gar keine Band für die IgG-Steuerung und ChIP Gassen. Es sollte eine Band im Input Spur (Abbildung 3).
Abbildung 4 zeigt die gleichen Proben mittels real-time PCR untersucht. Wie bei der vorherigen Figur, sollte es eine signifikante fache Anreicherung der positiven Bindungsregion für den Chip Probe über die IgG-Kontrolle. Auch sollte es sehr wenig Anreicherung, wenn überhaupt, in der negativen Bindungsregion gesehen werden.
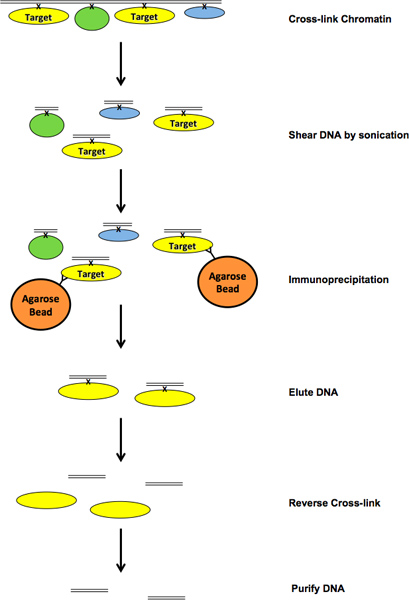
Abbildung 1. Flussdiagramm des Chip-Verfahren. Klicke hier, um eine größere Abbildung anzuzeigen .
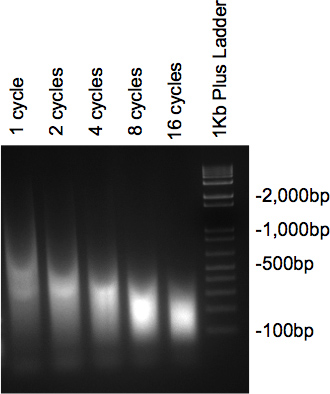
Abbildung 2. Gel von DNA Beschallung zu überprüfen.
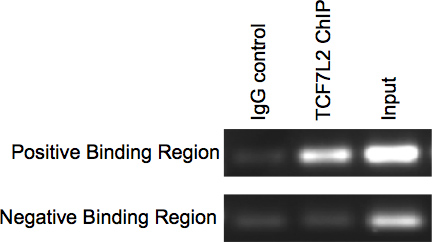
Abbildung 3. PCR von ChIP prüfen.

Abbildung 4. Echtzeit-PCR von TCF7L2 ChIP.
| Reagenz | Volume |
| Bead Pellet | 300 ul |
| BSA (50 mg / ml) | 30 ul |
| 100X Proteinase-Inhibitor | 10 ul |
| ChIP Dilution Buffer | 660 ul |
| Insgesamt | 1.000 ul |
Tabelle 1. Rezept für die Sperrung Agarose.
| Puffer | Components |
| Low Salz Immune Complex Wash Buffer | 0,1% SDS 1% Triton X-100 2 mM EDTA 20 mM Tris-HCl pH 8,1 150 mM NaCl |
| Hohe Salz Immune Complex Wash Buffer | 0,1% SDS 1% Triton X-100 2 mM EDTA 20 mM Tris-HCl pH 8,1 500 mM NaCl |
| LiCl Immune Complex Wash Buffer | 0,25 M LiCl 1% NP-40 1% Deoxycholate 1 mM EDTA 10 mM Tris-HCl pH 8,1 |
| TE Buffer | 10 mM Tris-HCl pH 8,1 1 mM EDTA pH 8,0 |
Tabelle 2. ChIP Waschpuffer.
| Reagenz | Volume |
| 20% SDS-Puffer | 10 ul |
| 1 M NaHCO 3 | 20 ul |
| H 2 O | 170 ul |
Tabelle 3. Elutionspuffer für eine IP.
| Reagenz | 50 ul Reaktion | 20 ul Reaktion |
| Wasser | 27 ul | 10,8 ul |
| 5X PCR Reaktionspuffer | 10 ul | 4 ul |
| MgCl 2 | 4 ul | 1,6 ul |
| dNTP (10 mM) | 1 ul | 0,4 ul |
| Primer-Mix (5 uM jeweils) | 2 ul | 0,8 ul |
| Taq (Promega Warmstart) | 1 ul | 0,4 ul |
| ChIP DNA | 5 ul | 2 ul |
Tabelle 4. PCR-Reaktion Bände.
Diskussion
Es ist nun möglich, die Durchführung einer genomweiten Profil von Protein-DNA-Wechselwirkungen Assoziation mit ChIP-seq, wie bereits vor kurzem mit anderen Transkriptionsfaktoren 2,3 demonstriert. Der Schlüssel zu einer erfolgreichen Sequenzierung Ergebnis ist die Erzeugung eines hochwertigen Chromatinimmunpräzipitation DNA-Vorlage.
Sobald die DNA-Matrize erzeugt wurde und festgestellt, angemessen angereichert werden, kann man dann nehmen Sie es in der Bibliothek Vorbereitung für die anschließende Sequenzierung. Zum Beispiel kann man die Sequenzierung Bibliothek Protokoll vom Verkäufer, Illumina vorgesehen. Die Größe Auswahl dieser Bibliothek kann durch Gelelektrophorese und anschließende Entfernung und Reinigung von DNA in die ~ 200 durchgeführt werden - bis 700-bp-Bereich. Die Reduzierung der Größe und Verengung der Größenbereich von DNA aus Gelreinigung gesammelt beabsichtigt ist, Ortsauflösung von ChIP-seq verbessern. Durch die Anreicherung für kleinere Stücke von Input-DNA gebunden an den Faktor intERest, würde man erwarten, dass vor Ort Standort wird Auflösung gewinnen. Engere Auswahl Größe verbessert auch die einheitliche Größe der molekularen Kolonien auf der Illumina-Plattform produziert. Solche Kolonie einheitliche Größe erhöht sich auch die effektive Lese Zahl erhalten. Kürzere Eingang DNA Größe produziert auch robuster Kolonien auf der Illumina-Plattform und kann dies bedeuten, dass kürzere DNA-Stücke innerhalb einer gegebenen Probe Eingang Verteilung wird effizienter in der Schluss-Sequenz Ausgabe vertreten sind länger als Eingang Stücke aus derselben Verteilung.
Die bioinformatische Ansätze zur "nächsten Generation" Sequenzanalyse sind weiterhin zu entwickeln, mit vielen Anbietern, die ihre Software Open-Source für die weitere Verfeinerung. Man kann die Transformation liest diese Karte einzigartig genomische Standorten in einem DNA-Fragment Überlappung Profil. Signifikante Spitzen können durch threshholding Profile in einer Höhe entspricht einem geschätzten False Discovery Rate identifiziert werden. Die Position specific Frequenz Matrizen aus dieser Arbeit abgeleitet werden zur Identifizierung und Lokalisierung von DNA-Bindungsstellen für das menschliche Genom für einen gegebenen Faktor werden.
Aber man muss vorsichtig sein, in Bezug auf welche Faktoren man wünscht, mit ChIP-seq studieren. Bevor über eine solche Studie sollte man beurteilen, ob ein Antikörper ist auf dem Markt erhältlich, die den nutzbaren in der Chip-Einstellung ist, als eine schlechte Antikörper sehr nachteilige Auswirkungen auf eine der experimentellen Ergebnisse haben können. Darüber hinaus sollte man sich überlegen, ob es splice Isoformen des Proteins unter Studie sind, ja, TCF7L2 ist bekannt, dass viele Isoformen haben, so dass wir besonders vorsichtig bei der Auswahl eines Antikörpers, der an Aminosäuren konsequent in allen Haupt-Isoformen dieses Transkriptionsfaktors gebunden waren 25.
Zusammenfassend kann die Kombination von Chromatinimmunpräzipitation und ultra-Hochdurchsatz-Sequenzierung (ChIP-seq) Identifizierung und Kartierung von Protein-DNA-Wechselwirkungen in einem bestimmten Gewebe oder cell Linie. Wir haben dargelegt, wie eine hohe Qualität ChIP Vorlage für anschließende Sequenzierung zu generieren.
Offenlegungen
Die Autoren erklären, dass sie keine finanziellen Interessen haben.
Danksagungen
Die Arbeit wird von einem Institut Development Award aus dem Kinderkrankenhaus von Philadelphia unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| QIAquick PCR Purification Kit | Qiagen | 28104 | |
| EZ-ChIP Kit | Millipore | 17-371 | |
| GoTaq Hot Start Polymerase | Promega | M5001 | |
| Misonix Sonicator | Qsonica | XL-2000 | |
| NanoDrop 1000 Spectrophotometer | Thermo-Scientific | ||
| Positive control primer sequences (TCF7L2-1) Forward- 5'-TCGCCCTGTCAATAATCTCC-3' Reverse- 5'-GCTCACCTCCTGTATCTTCG-3' Negative control primer sequences (CTRL-1) Forward-5'-ATGTGGTGTGGCTGTGATGGGAAC-3' Reverse- 5'-CGAGCAATCGGTAAATAGGTCTGG-3' | |||
Referenzen
- Odom, D. T., et al. Control of pancreas and liver gene expression by HNF transcription factors. Science. 303, 1378-1381 (2004).
- Johnson, D. S., Mortazavi, A., Myers, R. M., Wold, B. Genome-wide mapping of in vivo protein-DNA interactions. Science. 316, 1497-1502 (2007).
- Robertson, G., et al. Genome-wide profiles of STAT1 DNA association using chromatin immunoprecipitation and massively parallel sequencing. Nature Methods. 4, 651-657 (2007).
- Reich, N. C., Liu, L. Tracking STAT nuclear traffic. Nat. Rev. Immunol. 6, 602-612 (2006).
- Lodige, I., et al. Nuclear export determines the cytokine sensitivity of STAT transcription factors. The Journal of Biological Chemistry. 280, 43087-43099 (2005).
- Schroder, K., Sweet, M. J., Hume, D. A. Signal integration between IFNgamma and TLR signalling pathways in macrophages. Immunobiology. 211, 511-524 (2006).
- Vinkemeier, U. Getting the message across, STAT! Design principles of a molecular signaling circuit. The Journal of Cell Biology. 167, 197-201 (2004).
- Brierley, M. M., Fish, E. N. Stats: multifaceted regulators of transcription. J. Interferon Cytokine Res. 25, 733-744 (2005).
- Bentley, D. R. Whole-genome re-sequencing. Current Opinion in Genetics & Development. 16, 545-552 (2006).
- Grant, S. F., et al. Variant of transcription factor 7-like 2 (TCF7L2) gene confers risk of type 2 diabetes. Nature Genetics. 38, 320-323 (2006).
- Sladek, R., et al. A genome-wide association study identifies novel risk loci for type 2 diabetes. Nature. 445, 881-885 (2007).
- . Wellcome Trust Case Control Consortium. Genome-wide association study of 14,000 cases of seven common diseases and 3,000 shared controls. Nature. 447, 661-678 (2007).
- Saxena, R., et al. Genome-wide association analysis identifies loci for type 2 diabetes and triglyceride levels. Science. 316, 1331-1336 (2007).
- Zeggini, E., et al. Replication of genome-wide association signals in UK samples reveals risk loci for type 2 diabetes. Science. 316, 1336-1341 (2007).
- Scott, L. J., et al. A genome-wide association study of type 2 diabetes in Finns detects multiple susceptibility variants. Science. 316, 1341-1345 (2007).
- Steinthorsdottir, V., et al. A variant in CDKAL1 influences insulin response and risk of type 2 diabetes. Nature Genetics. 39, 770-775 (2007).
- Salonen, J. T., et al. Type 2 Diabetes Whole-Genome Association Study in Four Populations: The DiaGen Consortium. American Journal of Human Genetics. 81, 338-345 (2007).
- Zeggini, E., McCarthy, M. I. TCF7L2: the biggest story in diabetes genetics since HLA. Diabetologia. 50, 1-4 (2007).
- Weedon, M. N. The importance of TCF7L2. Diabet. Med. 24, 1062-1066 (2007).
- Hattersley, A. T. Prime suspect: the TCF7L2 gene and type 2 diabetes risk. The Journal of Clinical Investigation. 117, 2077-2079 (2007).
- Yochum, G. S., et al. Serial analysis of chromatin occupancy identifies beta-catenin target genes in colorectal carcinoma cells. Proceedings of the National Academy of Sciences of the United States of America. 104, 3324-3329 (2007).
- Duval, A., Busson-Leconiat, M., Berger, R., Hamelin, R. Assignment of the TCF-4 gene (TCF7L2) to human chromosome band 10q25.3. Cytogenet. Cell Genet. 88, 264-265 (2000).
- Pomerantz, M. M., et al. The 8q24 cancer risk variant rs6983267 shows long-range interaction with MYC in colorectal cancer. Nature Genetics. 41, 882-884 (2009).
- Tuupanen, S., et al. The common colorectal cancer predisposition SNP rs6983267 at chromosome 8q24 confers potential to enhanced Wnt signaling. Nature Genetics. 41, 885-890 (2009).
- Zhao, J., Schug, J., Li, M., Kaestner, K. H., Grant, S. F. Disease-associated loci are significantly over-represented among genes bound by transcription factor 7-like 2 (TCF7L2) in vivo. Diabetologia. 53, 2340-2346 (2010).
- Benjamini, Y., Yekutieli, D. Quantitative trait Loci analysis using the false discovery rate. Genetics. 171, 783-790 (2005).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten