Method Article
Reconstituição do canal receptor de glutamato bacteriano por encapsulamento de um sistema de expressão livre de células
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
Este protocolo descreve o método de emulsão invertida usado para encapsular um sistema de expressão livre de células (CFE) dentro de uma vesícula unilamelar gigante (GUV) para a investigação da síntese e incorporação de uma proteína de membrana modelo na bicamada lipídica.
Resumo
Os sistemas de expressão livre de células (CFE) são ferramentas poderosas em biologia sintética que permitem a biomimética de funções celulares como biossensoriamento e regeneração de energia em células sintéticas. A reconstrução de uma ampla gama de processos celulares, no entanto, requer a reconstituição bem-sucedida das proteínas da membrana na membrana das células sintéticas. Embora a expressão de proteínas solúveis seja geralmente bem-sucedida em sistemas CFE comuns, a reconstituição de proteínas de membrana em bicamadas lipídicas de células sintéticas provou ser um desafio. Aqui, é demonstrado um método para reconstituição de uma proteína de membrana modelo, receptor bacteriano de glutamato (GluR0), em vesículas unilamelares gigantes (GUVs) como células sintéticas modelo baseado na encapsulação e incubação da reação CFE dentro de células sintéticas. Utilizando esta plataforma, é demonstrado o efeito da substituição do peptídeo sinal N-terminal de GluR0 por peptídeo sinal de proteorodopsina na translocação cotranslacional bem-sucedida de GluR0 em membranas de GUVs híbridos. Este método fornece um procedimento robusto que permitirá a reconstituição livre de células de várias proteínas de membrana em células sintéticas.
Introdução
A biologia sintética de baixo para cima ganhou interesse crescente na última década como um campo emergente com inúmeras aplicações potenciais em bioengenharia, administração de medicamentos e medicina regenerativa 1,2. O desenvolvimento de células sintéticas como pedra angular da biologia sintética de baixo para cima, em particular, atraiu uma ampla gama de comunidades científicas devido às aplicações promissoras de células sintéticas, bem como suas propriedades físicas e bioquímicas semelhantes a células que facilitam estudos biofísicos in vitro 3,4,5,6 . As células sintéticas são frequentemente projetadas em vesículas unilamelares gigantes do tamanho de células (GUVs) nas quais diferentes processos biológicos são recriados. A reconstituição do citoesqueleto celular 7,8, a regeneração de energia dependente de luz9, a comunicação celular10,11 e o biossensoriamento12 são exemplos de esforços feitos para reconstruir comportamentos semelhantes a células em células sintéticas.
Embora alguns processos celulares dependam de proteínas solúveis, muitas características das células naturais, como detecção e comunicação, geralmente utilizam proteínas de membrana, incluindo canais iônicos, receptores e transportadores. Um grande desafio no desenvolvimento de células sintéticas é a reconstituição de proteínas de membrana. Embora os métodos tradicionais de reconstituição de proteínas de membrana em bicamadas lipídicas dependam da purificação mediada por detergente, tais métodos são trabalhosos, ineficazes para proteínas tóxicas para o hospedeiro de expressão ou muitas vezes não são adequados para a reconstituição de proteínas de membrana em GUVs13.
Um método alternativo para a expressão de proteínas são os sistemas de expressão livre de células (CFE). Os sistemas CFE têm sido uma ferramenta poderosa na biologia sintética que permite a expressão in vitro de várias proteínas usando lisado celular ou maquinário de transcrição-tradução purificado14. Os sistemas CFE também podem ser encapsulados em GUVs, permitindo assim reações de síntese de proteínas compartimentadas que podem ser programadas para várias aplicações, como a criação de células sintéticas coletoras de luz9 ou biossensores mecanossensíveis15,16. Análoga aos métodos de expressão de proteínas recombinantes, a expressão de proteínas de membrana é desafiadora em sistemas CFE17. Agregação, dobramento incorreto e falta de modificação pós-traducional em sistemas CFE são os principais gargalos que impedem a síntese bem-sucedida de proteínas de membrana usando sistemas CFE. A dificuldade de reconstituição de proteínas de membrana de baixo para cima usando sistemas CFE se deve em parte à ausência de uma via complexa de biogênese de proteínas de membrana que dependa de peptídeos de sinal, partículas de reconhecimento de sinal, translocões e moléculas de acompanhamento. No entanto, recentemente, vários estudos sugeriram que a presença de estruturas membranosas, como microssomas ou lipossomas durante a tradução, promove a expressão bem-sucedida de proteínas de membrana 18,19,20,21. Além disso, Eaglesfield et al. e Steinküher et al. descobriram que a inclusão de domínios hidrofóbicos específicos conhecidos como peptídeos sinal no terminal N da proteína de membrana pode melhorar significativamente sua expressão22,23. Em conjunto, esses estudos sugerem que o desafio da reconstituição da proteína de membrana em células sintéticas pode ser superado se a tradução da proteína ocorrer na presença da membrana GUV e se o peptídeo sinal N-terminal adequado for utilizado.
Aqui, é apresentado um protocolo para encapsulamento da síntese proteica usando reações CFE de elementos recombinantes (PURE) para reconstituição de proteínas de membrana em GUVs. O receptor bacteriano de glutamato24 (GluR0) é selecionado como a proteína modelo de membrana, e o efeito de seu peptídeo sinal N-terminal em sua reconstituição de membrana é estudado. O efeito do peptídeo sinal de proteorodopsina, que demonstrou melhorar a eficiência de reconstituição da proteína de membrana por Eaglesfield et al.22, é investigado pela construção de uma variante mutada de GluR0 denotada como PRSP-GluR0 e sua expressão e localização de membrana com GluR0 de tipo selvagem (referido como WT-GluR0 a seguir) que abriga seu peptídeo sinal nativo é comparado. Este protocolo é baseado no método de emulsão invertida25 com modificações que o tornam robusto para encapsulamento CFE. No método apresentado, as reações de CFE são primeiro emulsionadas usando uma solução lipídica em óleo que gera gotículas de tamanho mícron que contêm o sistema CFE e são estabilizadas pela monocamada lipídica. As gotículas de emulsão são então colocadas em camadas sobre uma interface óleo-água saturada com outra monocamada lipídica. As gotículas de emulsão são então forçadas a viajar através da interface óleo-água por meio de força centrífuga. Por meio desse processo, as gotículas obtêm outra monocamada, gerando assim uma vesícula lipídica de bicamada. Os GUVs contendo a reação CFE são então incubados, durante os quais a proteína de membrana é expressa e incorporada à membrana GUV. Embora este protocolo seja especificado para expressão livre de células de GluR0, ele pode ser usado para síntese livre de células de outras proteínas de membrana ou diferentes aplicações de células sintéticas, como reconstituição de citoesqueleto ou estudos de fusão de membrana26.
Protocolo
Os reagentes e equipamentos utilizados para este estudo são fornecidos na Tabela de Materiais.
1. Reações de CFE em massa na presença de pequenas vesículas unilamelares (SUVs)
- Preparação para SUV
NOTA: Esta etapa deve ser realizada em uma hotte seguindo as instruções de segurança para trabalhar com clorofórmio.- Prepare 5 mM de SUVs de 1-palmitoil-2-oleoil-glicero-3-fosfocolina (POPC) em um frasco de vidro, transferindo 76 μL de solução estoque de POPC de 25 mg / mL dissolvida em clorofórmio.
- Enquanto gira suavemente o frasco de vidro, sopre um fluxo suave de argônio no frasco para formar uma película de lipídios secos no fundo. Em seguida, transfira o frasco de vidro para um dessecador com a tampa frouxamente aparafusada para evaporar o excesso de clorofórmio.
- Mantenha o frasco de vidro no exsicador por 1 h. Em seguida, adicione 0,5 mL de água deionizada ultrapura para dissolver o filme lipídico e o vórtice por aproximadamente 2 min.
- Configure um aparelho de miniextrusão embebendo dois suportes de filtro em água deionizada e colocando-os em cada um dos suportes internos da membrana. Em seguida, embeba um filtro de policarbonato de 100 nm e coloque-o entre os dois suportes internos da membrana mantidos juntos pelo invólucro externo da extrusora e pela porca de retenção. Coloque esta configuração no suporte da extrusora.
- Lave duas seringas estanques a gás de 1 mL 3 vezes com água deionizada ultrapura.
- Carregue a amostra de mistura lipídica-água em uma das seringas estanques a gás de 1 mL e coloque-a em uma extremidade da mini extrusora usando os clipes do braço oscilante para manter a seringa no lugar. Insira a segunda seringa na outra extremidade da miniextrusora e certifique-se de que esteja totalmente pressionada.
NOTA: Ao carregar a seringa com a mistura lipídica-água, certifique-se de que não há ar na seringa antes de passá-la pela miniextrusora. - Passe suavemente a mistura lipídica-água da seringa original para a seringa vazia através do aparelho miniextrusora. Repita esta etapa 11 vezes para formar SUVs. Transfira os SUVs para um tubo de microcentrífuga de 1,5 mL.
NOTA: A solução SUV pode ser armazenada a 4 °C por até 2 semanas.
- Conjunto de reação CFE
- Monte a reação CFE seguindo o protocolo de expressão livre de células fornecido pelo fabricante com pequenas modificações detalhadas a seguir.
- Misture 10 μL da Solução 1 (contendo aminoácidos, NTPs, tRNAs e substratos para enzimas e tampão necessário), 1 μL de Solução 2 (proteínas em glicerol a 20%), 2 μL de Solução 3 (ribossomo (20 μM)), quantidade apropriada do DNA que codifica para sfGFP-sfCherry solúvel (1-10) 27 (doravante referido como sfGFP solúvel), WT-GluR0-sfGFP ou PRSP-GluR0-sfGFP (10-60 ng / 1000 pares de bases), 1 μL de inibidor de RNAse murina e 4 μL de solução SUV 5 mM para proteínas de membrana ou 4 μL de água para proteínas solúveis.
- Traga o volume final da reação para 20 μL adicionando água deionizada ultrapura.
NOTA: Ao montar o sistema CFE, todos os componentes devem ser mantidos no gelo. Todos os materiais são sensíveis à temperatura e podem se degradar se atingirem a temperatura ambiente.
- Incubação e monitoramento da reação CFE
- Transfira a solução CFE para uma placa cônica de fundo em V de 96 poços. Para evitar a evaporação durante o curso da reação, cubra a placa com um filme de vedação.
- Incubar a placa a 37 °C num leitor de placas durante 4-5 h enquanto monitoriza a reação CFE medindo o sinal GFP com um ganho de 100 a comprimentos de onda de excitação/emissão de 488 nm/528 nm a cada 2 min.
2. Reações CFE encapsuladas em GUVs
- Preparação da solução tampão externa GUV
- Em um tubo de microcentrífuga de 1,5 mL, misture 1,5 μL de espermidina 1 M, 37,5 μL de ATP 100 mM, 25 μL de GTP 100 mM, 12,5 μL de CTP 100 mM, 12,5 μL de UTP 100 mM, 25 μL de fosfato de creatina 1 M, 18 μL de acetato de magnésio 1 M, 93,33 μL de glutamato de potássio 3 M, 50 μL de 1 M HEPES KOH (pH 7,4), 1,15 μL de ácido folínico 332 mM, 100 μL de glicose 2 M e 50 μL de solução estoque de mistura 6 mM de cada um dos 20 aminoácidos (preparados seguindo o protocolo descrito por Sun et al.28).
- Traga o volume final da solução para 1 mL adicionando água deionizada ultrapurificada.
NOTA: Todos os componentes devem ser mantidos no gelo. Adicione a mistura de aminoácidos no final para evitar a depleção de aminoácidos28. A solução pode ser alíquota em alíquotas de 330 μL e conservada a -20 °C até à utilização.
- Preparação da mistura lipídica em óleo
NOTA: esta etapa deve ser realizada em uma hotte seguindo as instruções de segurança para trabalhar com clorofórmio.- Em uma capela de exaustão, misture 17,3 μL de solução estoque de POPC 25 mg / mL e 1,08 μL de copolímero de poli (butadieno) -b-poli (óxido de etileno) (PEO-b-PBD) de 50 mg / mL em um frasco de vidro de 15 mL.
NOTA: A solução final de lipídio em óleo contém 0,5 mM de lipídio com 95% e 5% de POPC e PEO-b-PBD, respectivamente. PEO-b-PBD foi usado para aumentar a estabilidade da membrana durante a expressão da proteína, mas foi mantido em uma proporção molar baixa para reduzir a tendência do copolímero de se agregar em micelas separadas das moléculas lipídicas29,30. - Sopre cuidadosamente um fluxo suave de gás argônio no frasco de vidro enquanto gira o frasco para evaporar o clorofórmio.
- Pipete 1,2 mL de óleo mineral leve no frasco de vidro de 15 mL contendo os lipídios secos.
- Misture os lipídios e o óleo por vórtice na velocidade máxima por 10-20 s. A mistura de lipídio-copolímero dissolvida parecerá turva.
- Para garantir que todos os agregados lipídicos possíveis no óleo sejam totalmente dissolvidos e dispersos por todo o óleo, coloque o frasco de vidro em um forno a cerca de 50 ° C por 20 min antes do vórtice por mais 10-20 s na velocidade máxima.
- Em uma capela de exaustão, misture 17,3 μL de solução estoque de POPC 25 mg / mL e 1,08 μL de copolímero de poli (butadieno) -b-poli (óxido de etileno) (PEO-b-PBD) de 50 mg / mL em um frasco de vidro de 15 mL.
- Montagem e encapsulamento da reação CFE (a Figura 1 resume as etapas a seguir).
- Prepare 300 μL da solução externa GUV em um tubo de microcentrífuga de 1,5 mL misturando 270 μL de solução tampão externa CFE preparada na etapa 2.1, 15 μL de NaCl 5 M e 15 μL de KCl 4,5 M.
NOTA: Nesta etapa, adicione 0.45 μL de 1 M 1,4-ditiotreitol (DTT) à solução externa de GUV. O objetivo da adição de NaCl e KCl é ajustar a osmolalidade da solução externa para combiná-la com a solução interna de CFE. O volume exato de NaCl e KCl depende do ajuste de osmolalidade desejado. Pode-se adicionar NaCl ou KCl ou ambos para ajustar a osmolalidade. - Pipete suavemente 300 μL de mistura lipídica em óleo sobre a solução externa GUV.
NOTA: Ao adicionar a mistura lipídica-óleo, é importante que o óleo não se misture com a solução externa aquosa. Após a adição, deve haver uma interface visível entre a mistura lipídica em óleo e a solução externa de GUV. - Incubar a interface óleo-água à temperatura ambiente durante 2 h para permitir que a monocamada lipídica se forme e se estabilize na interface.
- Enquanto isso, siga a seção 1.2.1 para montar uma reação CFE contendo o DNA do plasmídeo que codifica as variantes da proteína de membrana ou GFP solúvel. Substitua os 4 μL de solução SUV 5 mM ou água por 4 μL de sacarose 1 M. Essa reação será a solução interna do GUV.
NOTA: Um osmômetro foi usado para medir a osmolalidade das soluções interna e externa. A osmolalidade da solução externa foi então ajustada de acordo com a adição de NaCl ou água. A osmolalidade da reação CFE é tipicamente em torno de 1600 mOsm/Kg. A adição de sacarose à reação aumenta a densidade da solução interna, permitindo assim que as vesículas viajem pela interface óleo-água durante a etapa de centrifugação. Uma alternativa à sacarose é a solução de gradiente de densidade Opti-Prep. - Adicione 600 μL de mistura lipídica-óleo ao tubo de microcentrífuga contendo a reação CFE e pipete para cima e para baixo vigorosamente por ~ 1 min para emulsionar a reação em solução lipídica em óleo e formar a monocamada lipídica ao redor das células sintéticas.
NOTA: A solução final não deve ter bolhas e deve parecer opaca. - Pipete suavemente a emulsão da solução interna sobre a camada de óleo no tubo de microcentrífuga de 1,5 mL onde a interface óleo-água foi configurada.
NOTA: Tenha cuidado para não perturbar ou desestabilizar a interface. - Centrifugue por 10 min a 2.000 x g a 4 °C.
NOTA: A velocidade de centrifugação foi otimizada para este protocolo. Adir et al.31 relataram uma velocidade de centrifugação diferente. - Terminada a centrifugação, remova cuidadosamente o excesso de óleo e a solução externa do tubo da microcentrífuga usando um pipetador. Remover a solução externa até que o volume restante esteja em torno de 100 μL.
NOTA: Um pellet de GUVs geralmente é visível na parte inferior do tubo da microcentrífuga. No entanto, a falta de um pellet visível não significa necessariamente nenhum rendimento GUV. Em vez de usar um pipetador, o excesso de óleo em cima da solução externa pode ser removido por aspiração. É fundamental garantir que a solução lipídica em óleo seja completamente removida. A contaminação por óleo na solução GUV pode causar imagens de baixa qualidade. - Ressuspenda o grânulo GUV na solução de 100 μL restante, pipetando suavemente para cima e para baixo. Em seguida, transfira a solução GUV para uma placa de fundo plano limpa de 96 poços para incubar.
- Prepare 300 μL da solução externa GUV em um tubo de microcentrífuga de 1,5 mL misturando 270 μL de solução tampão externa CFE preparada na etapa 2.1, 15 μL de NaCl 5 M e 15 μL de KCl 4,5 M.
3. Incubação e imagem de reação CFE encapsulada
- Cubra a placa usando um filme de vedação para evitar a evaporação. Incubar a placa a 37 °C durante 5-6 h. Pode-se usar um leitor de placas e seguir a etapa 1.3.2 para preparar o leitor de placas para a etapa de incubação.
- Quando a incubação terminar, coloque a placa de 96 poços no estágio de imagem de um microscópio invertido equipado com uma câmera EMCCD (ou uma câmera sCMOS), laser controlado por DAQ-MX (ou um sistema combinador de laser integrado) e um confocal de disco giratório CSU-X1 (ou um confocal de varredura a laser). Concentre-se em qualquer ROI contendo GUVs e capture imagens em um comprimento de onda de excitação de 488 nm usando uma objetiva Plan-Apochromat 60 x/1.4 NA.
- Salve imagens de GUVs no formato .tiff.
- Abra as imagens em um software de processamento de imagem (por exemplo, ImageJ ou Fiji). Abra o painel de configuração Brilho/Contraste . Ajuste o brilho e o contraste para as configurações apropriadas que tornam as proteínas fluorescentes visíveis.
- Se o objetivo for comparar a intensidade do sinal de diferentes proteínas expressas, primeiro empilhe imagens individuais de GUVs contendo diferentes proteínas usando o painel Imagens para empilhar localizado no submenu Pilhas de > de imagens . Em seguida, ajuste o brilho e o contraste de todas as imagens usando o painel Brilho/Contraste .
Resultados
Antes do encapsulamento das reações CFE, duas variantes de GluR0-sfGFP contendo peptídeos sinais nativos e proteorodopsina (as sequências de peptídeos sinal são apresentadas na Tabela Suplementar 1) e o sfGFP solúvel foram expressos individualmente em reações em massa, e sua expressão foi monitorada pela detecção do sinal sfGFP usando um leitor de placas (Figura 2A). As proteínas de membrana foram expressas na ausência ou presença de SUVs de 100 nm. Além disso, usando uma curva de calibração que correlaciona o sinal sfGFP com sua concentração (Figura Suplementar 1), foram estimadas as concentrações de proteínas sintetizadas (Tabela Suplementar 2). Claramente, o sfGFP solúvel teve a maior expressão entre as três proteínas, o que sugere que a expressão de proteínas de membrana impõe uma carga ao sistema CFE, retardando assim a reação e diminuindo seu rendimento. Além disso, em média, as reações que expressam proteínas de membrana na presença de SUVs mostraram maior sinal sfGFP em comparação com reações sem SUVs. Essa observação se alinha com os achados de Steinküher et al., que mostraram que a expressão de proteínas de membrana reduz a capacidade dos sistemas CFE de produzir proteínas23. No entanto, dada a demonstração bem-sucedida da expressão de proteínas na reação CFE em massa, pode-se raciocinar que o CFE encapsulado também sintetizará proteínas dentro de GUVs.
Em seguida, reações individuais de CFE foram encapsuladas em GUVs usando o método de emulsão invertida para expressar variantes de GluR0, ou seja, WT-GluR0, PRSP-GluR0 e sfGFP solúvel. Enquanto WT-GluR0, abrigando o peptídeo sinal nativo GluR0, demonstrou excelente expressão e localização da membrana (Figura 2B, painel esquerdo), sua contraparte, PRSP-GluR0, que possui peptídeo sinal N-terminal de proteorodopsina, não mostrou forte localização de membrana semelhante. O PRSP-GluR0 mostrou-se mais propenso à agregação e formação de pontos (Figura 2B, painel do meio). Como esperado, a sfGFP solúvel foi expressa em GUVs e permaneceu no lúmen GUV (Figura 2B, painel direito; consulte a Figura Suplementar 2 para imagens de coortes de GUVs).
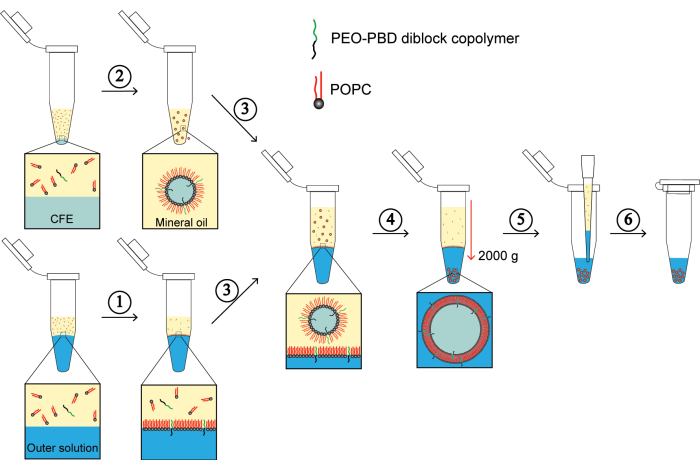
Figura 1: Etapas experimentais da emulsão invertida. (1) As etapas 2.3.1 a 2.3.3 do protocolo são visualizadas para demonstrar a montagem da monocamada lipídica na interface da mistura lipídico-óleo e da solução tampão externa. (2) A visualização da etapa 2.3.5 do protocolo é mostrada aqui para representar a formação da monocamada lipídica em torno de gotículas emulsionadas encapsulando a solução interna de CFE. (3) A etapa 2.3.6 do protocolo mostra a adição dos GUVs de monocamada ao tubo de microcentrífuga com a monocamada lipídica na interface de uma mistura lipídico-óleo e solução tampão externa. (4) A etapa 2.3.7 é descrita aqui, na qual a centrifugação leva à formação de um pellet GUV na solução externa. (5) A etapa 2.3.8 é mostrada aqui, indicando o processo de remoção do excesso de mistura de lipídios em óleo e solução externa. (6) Finalmente, a etapa 2.3.9 é descrita aqui, onde o pellet GUV é ressuspenso na solução externa e os GUVs estão prontos para incubação, seguido de imagem. Clique aqui para ver uma versão maior desta figura.
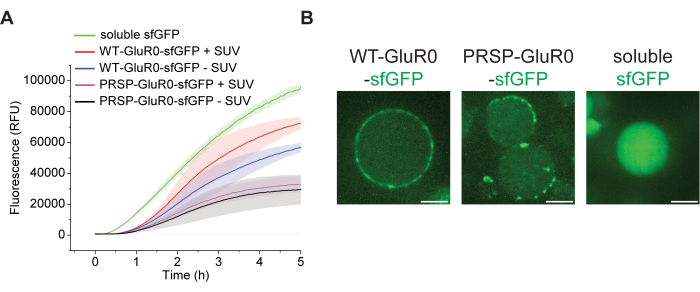
Figura 2: Expressão de proteínas em reações CFE em massa e em GUVs encapsulando reações CFE. (A) Leituras de fluorescência de reações CFE individuais em massa expressando WT-GluR0-sfGFP, PRSP-GluR0-sfGFP e sfGFP solúvel. O gráfico sfGFP solúvel representa o sinal de uma reação de 2,5 μL (o volume de reação padrão é de 20 μL) para evitar a supersaturação das medições do leitor de placas. Os dados são apresentados como média ± DP, n = 3. (B) Esquerda: Uma imagem confocal representativa de uma reação de CFE encapsulada por GUV expressando WT-GluR0-sfGFP. Meio: Uma imagem confocal representativa de GUVs encapsulando a reação CFE expressando PRSP-GluR0-sfGFP. Direita: Uma imagem confocal representativa de uma reação CFE encapsulada por GUV expressando sfGFP solúvel. Barras de escala: 10 μm. Clique aqui para ver uma versão maior desta figura.
Figura suplementar 1: A curva de calibração do sinal sfGFP e sua análise de regressão linear correspondente. Clique aqui para baixar este arquivo.
Figura suplementar 2: Imagem confocal representativa de coortes de GUVs expressando (esquerda) WT-GluR0-sfGFP, (meio) PRSP-GluR0-sfGFP e (direita) sfGFP solúvel. Barra de escala: 10 μm. Clique aqui para baixar este arquivo.
Tabela Suplementar 1: A sequência de aminoácidos dos peptídeos sinais de proteorodopsina e do tipo selvagem. Clique aqui para baixar este arquivo.
Tabela suplementar 2: A concentração de proteínas sintetizadas em reações CFE em massa. Clique aqui para baixar este arquivo.
Discussão
Praticamente qualquer processo celular que dependa da transferência de moléculas ou informações através da membrana celular, como sinalização celular ou excitação celular, requer proteínas de membrana. Assim, a reconstituição de proteínas de membrana tornou-se o principal gargalo na realização de vários projetos de células sintéticas para diferentes aplicações. A reconstituição tradicional mediada por detergente de proteínas de membrana em membranas biológicas requer métodos de geração de GUV, como inchaço suave ou eletroformação. As abordagens de inchaço geralmente produzem vesículas de tamanho pequeno, e o rendimento da eletroformação cai significativamente quando soluções complicadas, o que geralmente é o caso na geração de células sintéticas, são encapsuladas32. Além disso, os detergentes solubilizam a proteína da membrana, e sua remoção durante o processo de reconstituição pode causar desdobramento da proteína33,34. Por outro lado, a abordagem aqui apresentada baseia-se na incorporação cotraducional da proteína de membrana na bicamada lipídica, que se assemelha mais à via natural de biogênese da proteína nas células22.
Do ponto de vista técnico, o protocolo apresentado é vantajoso para outros métodos comuns de encapsulamento, como eletroformação e encapsulamento contínuo de cruzamento de interface de gotículas 7,8,35,36 (cDICE), para facilitar a implementação, pois o único equipamento de laboratório necessário para a geração de GUV é uma centrífuga. Ao contrário da eletroformação, o método de emulsão invertida permite o encapsulamento de diferentes combinações de moléculas com várias concentrações. Além disso, em comparação com a técnica original de emulsão invertida25, essa abordagem gera GUVs mais estáveis que são adequados para encapsulamento de lisados CFE ou sistemas PURE. A maior estabilidade do GUV deve-se à presença de copolímero dibloco na composição da membrana GUV37, bem como à longa incubação da interface óleo-água que permite que a interface seja saturada com moléculas lipídicas. Por fim, ao contrário das abordagens microfluídicas, o protocolo apresentado aqui não requer pequenos canais e tubulações. Portanto, a reação CFE pode ser encapsulada assim que é montada, e o menor tempo de montagem do GUV devido à falta de fluxo e possível entupimento evita o início prematuro da reação CFE. Embora a demonstração da expressão de proteínas de membrana neste protocolo seja exclusiva para reações PUREfrex, pode-se estender este método para sintetizar proteínas usando diferentes sistemas CFE disponíveis, como sistemas CFE bacterianos ou mamíferos baseados em lisados.
A abordagem apresentada aqui tem limitações que são causadas pela natureza dependente do óleo do processo de formação de GUV e pela intenção de ter GUVs estáveis. Essa abordagem é normalmente mais longa em comparação com outros métodos, como cDICE ou microfluídica, devido ao longo tempo de incubação da interface óleo-água que é necessário para a estabilização da interface e alto rendimento de GUV. Além disso, a composição lipídica é limitada principalmente ao POPC com pequenas doses de outros lipídios ou copolímeros em bloco, enquanto outros métodos, como a eletroformação, são mais adequados para a incorporação de lipídios com diferentes propriedades físicas e químicas. Embora a composição da membrana GUV neste método seja uma mistura de POPC e PBD-PEO para maximizar o rendimento do CFE, possíveis variações na composição da membrana GUV podem ser testadas. No entanto, uma otimização adicional dos parâmetros pode ser necessária para outras proteínas de membrana. Como a emulsificação de gotículas ocorre por pipetagem manual, os GUVs gerados por esse método são polidispersos e bastante heterogêneos em tamanho. Além disso, o fato de os lipídios serem dissolvidos na fase orgânica pode ocasionalmente causar uma camada de óleo entre os dois folhetos da membrana GUV ou contaminar a câmara de imagem com óleo que pode ser prejudicial à qualidade da imagem. Uma possível solução alternativa para o desafio do óleo residual é substituir o óleo mineral por um solvente orgânico volátil, como o éter dietílico, como mostrado por Tsumoto et al.38, para contar com a evaporação do solvente junto com a centrifugação durante a formação do GUV.
Embora não haja demonstração da função do canal neste trabalho, inspirado em ensaios anteriores usados para sondar a funcionalidade do canal mecanosensível ou sensível à luz reconstituída, um ensaio baseado em microscopia de fluorescência é descrito. A abertura do canal GluR0 é relatada para aumentar a condutividade da membrana para íons K + 24. Como as reações CFE já contêm uma alta concentração de K+, os indicadores típicos de potássio não serão adequados para avaliar a funcionalidade do canal. No entanto, como o influxo de potássio altera o potencial de membrana, indicadores de potencial de membrana sensíveis, como DiBAC4 (3) 22 ou BeRST 139 , podem relatar a atividade de GluR0 na presença de glutamato.
A reconstituição bem-sucedida de proteínas de membrana em células sintéticas abre inúmeras possibilidades para a criação de células sintéticas com habilidades sem precedentes que imitam mais de perto as células naturais. Uma grande desvantagem atual das células sintéticas é sua incapacidade de reproduzir e reciclar energia. No entanto, com esquemas de regeneração de energia dependentes de luz e produtos químicos que dependem fortemente de proteínas de membrana, pode-se imaginar células sintéticas de longa duração40. A utilização de sistemas CFE permite a reconstituição de múltiplas proteínas de membrana que podem executar coletivamente determinadas tarefas. Por exemplo, a reconstituição de um canal iônico dependente de ligante semelhante ao GluR0 descrito aqui, juntamente com diferentes canais iônicos dependentes de voltagem, pode levar à construção de uma célula sintética excitável semelhante a um neurônio.
Divulgações
Os autores declaram não haver conflitos de interesse.
Agradecimentos
A APL reconhece o apoio da National Science Foundation (EF1935265), dos Institutos Nacionais de Saúde (R01-EB030031 e R21-AR080363) e do Escritório de Pesquisa do Exército (80523-BB)
Materiais
| Name | Company | Catalog Number | Comments |
| 100 nm polycarbonate filter | STERLITECH | 1270193 | |
| 96 Well Clear Bottom Plate | ThermoFisher Scientific | 165305 | |
| BioTek Synergy H1M Hybrid Multi-Mode Reader | Agilent | 11-120-533 | |
| Creatine phosphate | Millipore Sigma | 10621714001 | |
| CSU-X1 Confocal Scanner Unit | Yokogawa | CSU-X1 | |
| Density gradient medium (Optiprep) | Millipore Sigma | D1556 | Optional to switch with sucrose in inner solution |
| Filter supports | Avanti | 610014 | |
| Fisherbrand microtubes (1.5 mL) | Fisher Scientific | 05-408-129 | |
| Folinic acid calcium salt hydrate | Millipore Sigma | F7878 | |
| Glucose | Millipore Sigma | 158968 | |
| HEPES | Millipore Sigma | H3375 | |
| iXon X3 camera | Andor | DU-897E-CS0 | |
| L-Glutamic acid potassium salt monohydrate | Millipore Sigma | G1501 | |
| Light mineral oil | Millipore Sigma | M5904 | |
| Magnesium acetate tetrahydrate | Millipore Sigma | M5661 | |
| Mini-extruder kit (including syringe holder and extruder stand) | Avanti | 610020 | |
| Olympus IX81 Inverted Microscope | Olympus | IX21 | |
| Olympus PlanApo N 60x Oil Microscope Objective | Olympus | 1-U2B933 | |
| PEO-b-PBD | Polymer Source | P41745-BdEO | |
| pET28b-PRSP-GluR0-sfGFP plasmid DNA | Homemade | N/A | |
| pET28b-sfGFP-sfCherry(1-10) plasmid DNA | Homemade | N/A | |
| pET28b-WT-GluR0-sfGFP plasmid DNA | Homemade | N/A | |
| POPC lipid in chloroform | Avanti | 850457C | |
| Potassium chloride | Millipore Sigma | P9541 | |
| PUREfrex 2.0 | Cosmo Bio USA | GFK-PF201 | |
| Ribonucleotide Solution Set | New England BioLabs | N0450 | |
| RNase Inhibitor, Murine | New England BioLabs | M0314S | |
| RTS Amino Acid Sampler | Biotechrabbit | BR1401801 | |
| Sodium chloride | Millipore Sigma | S9888 | |
| Spermidine | Millipore Sigma | S2626 | |
| Sucrose | Millipore Sigma | S0389 | |
| VAPRO Vapor Pressure Osmometer Model 5600 | ELITechGroup | VAPRO 5600 |
Referências
- Liu, A. P., Fletcher, D. A. Biology under construction: In vitro reconstitution of cellular function. Nat Rev Mol Cell Biol. 10 (9), 644-650 (2009).
- Lin, A. J., Sihorwala, A. Z., Belardi, B. Engineering tissue-scale properties with synthetic cells: Forging one from many. ACS Synth Biol. 12 (7), 1889-1907 (2023).
- Powers, J., Jang, Y. Advancing biomimetic functions of synthetic cells through compartmentalized cell-free protein synthesis. Biomacromolecules. 24 (12), 5539-5550 (2023).
- Jiang, W., et al. Artificial cells: Past, present and future. ACS Nano. 16 (10), 15705-15733 (2022).
- Groaz, A., et al. Engineering spatiotemporal organization and dynamics in synthetic cells. WIREs Nanomed Nanobiotech. 13 (3), 1685 (2021).
- Sharma, B., Moghimianavval, H., Hwang, S. W., Liu, A. P. Synthetic cell as a platform for understanding membrane-membrane interactions. Membranes. 11 (12), 912 (2021).
- Bashirzadeh, Y., et al. Actin crosslinker competition and sorting drive emergent GUV size-dependent actin network architecture. Commun Biol. 4 (1), 1-11 (2021).
- Bashirzadeh, Y., Moghimianavval, H., Liu, A. P. Encapsulated actomyosin patterns drive cell-like membrane shape changes. iScience. 25 (5), 104236 (2021).
- Berhanu, S., Ueda, T., Kuruma, Y. Artificial photosynthetic cell producing energy for protein synthesis. Nat Commun. 10 (1), 1325 (2019).
- Ji, Y., Chakraborty, T., Wegner, S. V. Self-regulated and bidirectional communication in synthetic cell communities. ACS Nano. 17 (10), 8992-9002 (2023).
- Moghimianavval, H., Loi, K. J., Hwang, S. W., Bashirzadeh, Y., Liu, A. P. Light-based juxtacrine signaling between synthetic cells. bioRxiv. , (2024).
- Boyd, M. A., Thavarajah, W., Lucks, J. B., Kamat, N. P. Robust and tunable performance of a cell-free biosensor encapsulated in lipid vesicles. Science Advances. 9 (1), 6605 (2023).
- Schneider, B., et al. Membrane Protein expression in cell-free systems. Methods Mol Biol. 601, 165-186 (2010).
- Noireaux, V., Liu, A. P. The new age of cell-free biology. Annu Rev Biomed Eng. 22 (1), 51-77 (2020).
- Majumder, S., et al. Cell-sized mechanosensitive and biosensing compartment programmed with DNA. Chem. Commun. 53 (53), 7349-7352 (2017).
- Poddar, A., et al. Membrane stretching activates calcium permeability of a putative channel Pkd2 during fission yeast cytokinesis. MBoC. 33 (14), (2022).
- Sachse, R., Dondapati, S. K., Fenz, S. F., Schmidt, T., Kubick, S. Membrane protein synthesis in cell-free systems: From bio-mimetic systems to bio-membranes. FEBS Letters. 588 (17), 2774-2781 (2014).
- Dondapati, S. K., et al. Functional reconstitution of membrane proteins derived from eukaryotic cell-free systems. Front Pharmacol. 10, 917 (2019).
- Majumder, S., et al. In vitro synthesis and reconstitution using mammalian cell-free lysates enables the systematic study of the regulation of LINC complex assembly. Biochemistry. 61 (14), 1495-1507 (2022).
- Niwa, T., et al. Comprehensive study of liposome-assisted synthesis of membrane proteins using a reconstituted cell-free translation system. Sci Rep. 5 (1), 18025 (2015).
- Moghimianavval, H., Hsu, Y. Y., Groaz, A., Liu, A. P. In vitro reconstitution platforms of mammalian cell-free expressed membrane proteinsmembrane proteins. Methods Mol Biol. 2433, 105-120 (2022).
- Eaglesfield, R., Madsen, M. A., Sanyal, S., Reboud, J., Amtmann, A. Cotranslational recruitment of ribosomes in protocells recreates a translocon-independent mechanism of proteorhodopsin biogenesis. iScience. 24 (5), 102429 (2021).
- Steinküher, J., et al. Improving cell-free expression of membrane proteins by tuning ribosome cotranslational membrane association and nascent chain aggregation. bioRxiv. , (2023).
- Chen, G. Q., Cui, C., Mayer, M. L., Gouaux, E. Functional characterization of a potassium-selective prokaryotic glutamate receptor. Nature. 402 (6763), 817-821 (1999).
- Pautot, S., Frisken, B. J., Weitz, D. A. Production of unilamellar vesicles using an inverted emulsion. Langmuir. 19 (7), 2870-2879 (2003).
- Hsu, Y. Y., et al. Calcium-triggered DNA-mediated membrane fusion in synthetic cells. Chemical Communications. 59 (57), 8806-8809 (2023).
- Moghimianavval, H., et al. Engineering functional membrane-membrane interfaces by interspy. Small. 19 (13), 2202104 (2023).
- Sun, Z. Z., et al. Protocols for implementing an Escherichia coli based TX-TL cell-free expression system for synthetic biology. J Vis Exp. (79), e50762 (2013).
- Jacobs, M. L., Boyd, M. A., Kamat, N. P. Diblock copolymers enhance folding of a mechanosensitive membrane protein during cell-free expression. PNAS. 116 (10), 4031-4036 (2019).
- Kostarelos, K., Tadros, T. F., Luckham, P. F. Physical conjugation of (tri-) block copolymers to liposomes toward the construction of sterically stabilized vesicle systems. Langmuir. 15 (2), 369-376 (1999).
- Adir, O., et al. Preparing protein producing synthetic cells using cell free bacterial extracts, liposomes and emulsion transfer. J Vis Exp. (158), e60829 (2020).
- van de Cauter, L., van Buren, L., Koenderink, G. H., Ganzinger, K. A. Exploring giant unilamellar vesicle production for artificial cells - current challenges and future directions. Small Methods. 7 (12), 2300416 (2023).
- Seddon, A. M., Curnow, P., Booth, P. J. Membrane proteins, lipids and detergents: Not just a soap opera. Biochim Biophys Acta Biomembr. 1666 (1), 105-117 (2004).
- Guo, Y. Detergent-free systems for structural studies of membrane proteins. Biochem Soc Trans. 49 (3), 1361-1374 (2021).
- Bashirzadeh, Y., Wubshet, N., Litschel, T., Schwille, P., Liu, A. P. Rapid encapsulation of reconstituted cytoskeleton inside giant unilamellar vesicles. J Vis Exp. (177), e63332 (2021).
- Hwang, S. W., et al. Hybrid vesicles enable mechano-responsive hydrogel degradation. Angew Chemie Int Ed. 62 (41), e202308509 (2023).
- Rideau, E., Dimova, R., Schwille, P., Wurm, F. R., Landfester, K. Liposomes and polymersomes: a comparative review towards cell mimicking. Chem Soc Rev. 47 (23), 8572-8610 (2018).
- Tsumoto, K., Hayashi, Y., Tabata, J., Tomita, M. A reverse-phase method revisited: Rapid high-yield preparation of giant unilamellar vesicles (GUVs) using emulsification followed by centrifugation. Colloids Surf A: Physicochem Eng Asp. 546, 74-82 (2018).
- Huang, Y. L., Walker, A. S., Miller, E. W. A photostable silicon rhodamine platform for optical voltage sensing. J Am Chem Soc. 137 (33), 10767-10776 (2015).
- Bailoni, E., et al. Minimal out-of-equilibrium metabolism for synthetic cells: A membrane perspective. ACS Synth Biol. 12 (4), 922-946 (2023).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados