Method Article
Rekonstitution des bakteriellen Glutamatrezeptorkanals durch Verkapselung eines zellfreien Expressionssystems
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Dieses Protokoll beschreibt das invertierte Emulsionsverfahren, das zur Verkapselung eines zellfreien Expressionssystems (CFE) in einem riesigen unilamellären Vesikel (GUV) zur Untersuchung der Synthese und des Einbaus eines Modellmembranproteins in die Lipiddoppelschicht verwendet wird.
Zusammenfassung
Zellfreie Expressionssysteme (CFE) sind leistungsfähige Werkzeuge in der synthetischen Biologie, die die Biomimikry zellulärer Funktionen wie Biosensorik und Energieregeneration in synthetischen Zellen ermöglichen. Die Rekonstruktion einer Vielzahl zellulärer Prozesse erfordert jedoch die erfolgreiche Rekonstitution von Membranproteinen in die Membran synthetischer Zellen. Während die Expression löslicher Proteine in gängigen CFE-Systemen in der Regel erfolgreich ist, hat sich die Rekonstitution von Membranproteinen in Lipiddoppelschichten synthetischer Zellen als schwierig erwiesen. In dieser Arbeit wird ein Verfahren zur Rekonstitution eines Modellmembranproteins, des bakteriellen Glutamatrezeptors (GluR0), in riesigen unilamellären Vesikeln (GUVs) als synthetische Modellzellen auf der Grundlage der Verkapselung und Inkubation der CFE-Reaktion in synthetischen Zellen demonstriert. Unter Verwendung dieser Plattform wird der Effekt der Substitution des N-terminalen Signalpeptids von GluR0 durch das Proteorhodopsin-Signalpeptid auf die erfolgreiche cotranslationale Translokation von GluR0 in Membranen von hybriden GUVs demonstriert. Mit dieser Methode steht ein robustes Verfahren zur Verfügung, das die zellfreie Rekonstitution verschiedener Membranproteine in synthetischen Zellen ermöglicht.
Einleitung
Die synthetische Bottom-up-Biologie hat in den letzten zehn Jahren als aufstrebendes Gebiet mit zahlreichen Anwendungsmöglichkeiten in der Biotechnologie, der Wirkstoffverabreichung und der regenerativen Medizin zunehmend an Interesse gewonnen 1,2. Insbesondere die Entwicklung synthetischer Zellen als Eckpfeiler der synthetischen Bottom-up-Biologie hat aufgrund der vielversprechenden Anwendungen synthetischer Zellen sowie ihrer zellähnlichen physikalischen und biochemischen Eigenschaften, die biophysikalische In-vitro-Studien erleichtern, ein breites Spektrum an wissenschaftlichen Gemeinschaften angezogen 3,4,5,6 . Synthetische Zellen werden oft in zellgroßen riesigen unilamellären Vesikeln (GUVs) hergestellt, in denen verschiedene biologische Prozesse nachgebildet werden. Die Rekonstitution des Zellzytoskeletts 7,8, die lichtabhängige Energieregeneration9, die zelluläre Kommunikation10,11 und die Biosensorik12 sind Beispiele für Bemühungen, zellähnliche Verhaltensweisen in synthetischen Zellen zu rekonstruieren.
Während einige zelluläre Prozesse auf löslichen Proteinen beruhen, verwenden viele Eigenschaften natürlicher Zellen, wie z. B. Sensorik und Kommunikation, häufig Membranproteine, einschließlich Ionenkanäle, Rezeptoren und Transporter. Eine große Herausforderung in der synthetischen Zellentwicklung ist die Rekonstitution von Membranproteinen. Obwohl traditionelle Methoden der Membranproteinrekonstitution in Lipiddoppelschichten auf einer Detergenz-vermittelten Reinigung beruhen, sind solche Methoden mühsam, unwirksam für Proteine, die für den Expressionswirt toxisch sind, oder oft nicht für die Membranproteinrekonstitution in GUVsgeeignet 13.
Eine alternative Methode zur Proteinexpression sind zellfreie Expressionssysteme (CFE). CFE-Systeme sind ein leistungsfähiges Werkzeug in der synthetischen Biologie, das die In-vitro-Expression verschiedener Proteine entweder mit Zelllysat oder gereinigter Transkriptions-Translations-Maschinerie ermöglicht14. CFE-Systeme können auch in GUVs verkapselt werden, was kompartimentierte Proteinsynthesereaktionen ermöglicht, die für verschiedene Anwendungen programmiert werden können, wie z. B. die Erzeugung von lichtsammelnden synthetischen Zellen9 oder mechanosensitiven Biosensoren15,16. Analog zu rekombinanten Proteinexpressionsmethoden stellt die Expression von Membranproteinen in CFE-Systemen eine Herausforderungdar 17. Aggregation, Fehlfaltung und fehlende posttranslationale Modifikation in CFE-Systemen sind große Engpässe, die eine erfolgreiche Membranproteinsynthese mit CFE-Systemen behindern. Die Schwierigkeit der Rekonstitution von Bottom-up-Membranproteinen mit Hilfe von CFE-Systemen ist zum Teil auf das Fehlen eines komplexen Biogenesewegs für Membranproteine zurückzuführen, der auf Signalpeptiden, Signalerkennungspartikeln, Translokons und Chaperoning-Molekülen beruht. In jüngster Zeit haben jedoch mehrere Studien darauf hingewiesen, dass das Vorhandensein von membranösen Strukturen wie Mikrosomen oder Liposomen während der Translation eine erfolgreiche Expression von Membranproteinen fördert 18,19,20,21. Darüber hinaus haben Eaglesfield et al. und Steinküher et al. herausgefunden, dass der Einschluss spezifischer hydrophober Domänen, die als Signalpeptide bekannt sind, in den N-Terminus des Membranproteins dessen Expression signifikant verbessern kann 22,23. Insgesamt deuten diese Studien darauf hin, dass die Herausforderung der Rekonstitution von Membranproteinen in synthetischen Zellen überwunden werden kann, wenn die Proteintranslation in Gegenwart der GUV-Membran erfolgt und wenn das richtige N-terminale Signalpeptid verwendet wird.
In dieser Arbeit wird ein Protokoll zur Verkapselung der Proteinsynthese unter Verwendung von rekombinanten Elementen (PURE) CFE-Reaktionen zur Rekonstitution von Membranproteinen in GUVs vorgestellt. Der bakterielle Glutamatrezeptor24 (GluR0) wird als Modellmembranprotein ausgewählt und die Wirkung seines N-terminalen Signalpeptids auf seine Membranrekonstitution untersucht. Die Wirkung des Proteorhodopsin-Signalpeptids, das von Eaglesfield et al.22 gezeigt wurde, dass es die Rekonstitutionseffizienz von Membranproteinen verbessert, wird untersucht, indem eine mutierte Variante von GluR0 mit der Bezeichnung PRSP-GluR0 konstruiert und seine Expression und Membranlokalisation mit Wildtyp-GluR0 (im Folgenden als WT-GluR0 bezeichnet), das sein natives Signalpeptid beherbergt, verglichen wird. Dieses Protokoll basiert auf dem invertierten Emulsionsverfahren25 mit Modifikationen, die es für die CFE-Verkapselung robust machen. Bei der vorgestellten Methode werden die CFE-Reaktionen zunächst mit einer Lipid-in-Öl-Lösung emulgiert, die mikrometergroße Tröpfchen erzeugt, die das CFE-System enthalten und durch die Lipidmonoschicht stabilisiert werden. Die Emulsionströpfchen werden dann auf eine Öl-Wasser-Grenzfläche geschichtet, die mit einer weiteren Lipid-Monoschicht gesättigt ist. Die Emulsionströpfchen werden dann durch die Zentrifugalkraft gezwungen, über die Öl-Wasser-Grenzfläche zu wandern. Durch diesen Prozess erhalten die Tröpfchen eine weitere Monoschicht, wodurch ein zweischichtiges Lipidvesikel entsteht. Die GUVs, die die CFE-Reaktion enthalten, werden dann inkubiert, wobei das Membranprotein exprimiert und in die GUV-Membran eingebaut wird. Obwohl dieses Protokoll für die zellfreie Expression von GluR0 spezifiziert ist, kann es für die zellfreie Synthese anderer Membranproteine oder verschiedene synthetische Zellanwendungen wie die Rekonstitution des Zytoskeletts oder Membranfusionsstudien verwendet werden26.
Protokoll
Die für diese Studie verwendeten Reagenzien und Geräte sind in der Materialtabelle aufgeführt.
1. Bulk-CFE-Reaktionen in Gegenwart von kleinen unilamellären Vesikeln (SUVs)
- SUV-Vorbereitung
HINWEIS: Dieser Schritt muss in einem Abzug gemäß den Sicherheitshinweisen für die Arbeit mit Chloroform durchgeführt werden.- Bereiten Sie 5 mM 1-Palmitoyl-2-oleoyl-glycero-3-phosphocholin (POPC) SUVs in einem Glasfläschchen vor, indem Sie 76 μl 25 mg/ml POPC-Stammlösung, gelöst in Chloroform, umfüllen.
- Während Sie das Glasfläschchen vorsichtig drehen, blasen Sie einen sanften Argonstrahl in das Fläschchen, um am Boden einen Film aus getrockneten Lipiden zu bilden. Übertragen Sie anschließend das Glasfläschchen in einen Exsikkator, wobei die Kappe locker verschraubt ist, um überschüssiges Chloroform zu verdampfen.
- Bewahren Sie das Glasfläschchen 1 h lang im Exsikkator auf. Fügen Sie dann 0,5 ml ultrareines deionisiertes Wasser hinzu, um den Lipidfilm aufzulösen und etwa 2 Minuten lang zu wirbeln.
- Richten Sie eine Mini-Extrusionsvorrichtung ein, indem Sie zwei Filterträger in entionisiertem Wasser einweichen und auf jeden der internen Membranträger legen. Tränken Sie dann einen 100-nm-Polycarbonatfilter und platzieren Sie ihn zwischen den beiden internen Membranstützen, die durch das Außengehäuse des Extruders und die Haltemutter zusammengehalten werden. Platzieren Sie dieses Setup in den Extruderständer.
- Spülen Sie zwei gasdichte 1-mL-Spritzen 3 Mal mit ultrareinem entionisiertem Wasser.
- Laden Sie die Probe des Lipid-Wasser-Gemisches in eine der gasdichten 1-ml-Spritzen und platzieren Sie sie mit den Schwenkarmclips an einem Ende des Mini-Extruders, um die Spritze an Ort und Stelle zu halten. Führen Sie die zweite Spritze in das andere Ende des Mini-Extruders ein und stellen Sie sicher, dass sie vollständig eingedrückt ist.
HINWEIS: Wenn Sie die Spritze mit dem Lipid-Wasser-Gemisch befüllen, stellen Sie sicher, dass sich keine Luft in der Spritze befindet, bevor Sie sie durch den Mini-Extruder leiten. - Das Lipid-Wasser-Gemisch wird vorsichtig von der Originalspritze in die leere Spritze durch den Mini-Extruder geleitet. Wiederhole diesen Schritt 11 Mal, um SUVs zu formen. Übertragen Sie die SUVs in ein 1,5-ml-Mikrozentrifugenröhrchen.
HINWEIS: Die SUV-Lösung kann bis zu 2 Wochen bei 4 °C gelagert werden.
- CFE-Reaktionsanordnung
- Assemblieren Sie die CFE-Reaktion gemäß dem vom Hersteller bereitgestellten zellfreien Expressionsprotokoll mit geringfügigen Änderungen, die im Folgenden beschrieben werden.
- Mischen Sie 10 μl Lösung 1 (enthält Aminosäuren, NTPs, tRNAs und Substrate für Enzyme sowie den erforderlichen Puffer), 1 μl Lösung 2 (Proteine in 20 % Glycerin), 2 μl Lösung 3 (Ribosom (20 μM)), eine geeignete Menge der DNA, die für lösliches sfGFP-sfCherry(1-10)27 (im Folgenden als lösliches sfGFP bezeichnet), WT-GluR0-sfGFP oder PRSP-GluR0-sfGFP (10-60 ng/1000 Basenpaare) kodiert, 1 μl muriner RNAse-Inhibitor und 4 μl 5 mM SUV-Lösung für Membranproteine oder 4 μl Wasser für lösliche Proteine.
- Bringen Sie das Endvolumen der Reaktion auf 20 μl, indem Sie ultrareines entionisiertes Wasser hinzufügen.
HINWEIS: Bei der Montage des CFE-Systems müssen alle Komponenten auf Eis gehalten werden. Alle Materialien sind temperaturempfindlich und können sich zersetzen, wenn sie Raumtemperatur erreichen.
- Inkubation und Überwachung der CFE-Reaktion
- Übertragen Sie die CFE-Lösung auf eine konische 96-Well-Platte mit V-Boden. Um eine Verdunstung während der Reaktion zu verhindern, decken Sie die Platte mit einer Siegelfolie ab.
- Inkubieren Sie die Platte bei 37 °C in einem Plattenlesegerät für 4-5 Stunden und überwachen Sie dabei die CFE-Reaktion, indem Sie das GFP-Signal mit einer Verstärkung von 100 bei 488 nm/528 nm Anregungs-/Emissionswellenlängen alle 2 Minuten messen.
2. In GUVs eingekapselte CFE-Reaktionen
- Herstellung der äußeren GUV-Pufferlösung
- Mischen Sie in einem 1,5-ml-Mikrozentrifugenröhrchen 1,5 μl 1 M Spermidin, 37,5 μl 100 mM ATP, 25 μl 100 mM GTP, 12,5 μl 100 mM CTP, 12,5 μl 100 mM UTP, 25 μl 1 M Kreatinphosphat, 18 μl 1 M Magnesiumacetat, 93,33 μl 3 M Kaliumglutamat, 50 μl 1 M HEPES KOH (PH 7,4), 1,15 μl 332 mM Folinsäure, 100 μl 2 M Glukose und 50 μl aus Stammlösung von 6 mM Mischung jeder der 20 Aminosäuren (hergestellt nach dem von Sun et al.28 beschriebenen Protokoll).
- Bringen Sie das endgültige Volumen der Lösung auf 1 ml, indem Sie hochreines deionisiertes Wasser hinzufügen.
HINWEIS: Alle Komponenten müssen auf Eis aufbewahrt werden. Fügen Sie die Aminosäuremischung am Ende hinzu, um einen Aminosäuremangelzu vermeiden 28. Die Lösung kann in 330 μl Aliquoten aliquotiert und bis zur Verwendung bei -20 °C gelagert werden.
- Zubereitung des Lipid-in-Öl-Gemisches
HINWEIS: Dieser Schritt muss in einem Abzug gemäß den Sicherheitshinweisen für die Arbeit mit Chloroform durchgeführt werden.- Mischen Sie in einem Abzug 17,3 μl 25 mg/ml POPC-Stammlösung und 1,08 μl 50 mg/ml Poly(butadien)-b-poly(ethylenoxid) (PEO-b-PBD)-Copolymer in einem 15-ml-Glasfläschchen.
HINWEIS: Die endgültige Lipid-in-Öl-Lösung enthält 0,5 mM Lipid mit 95 % bzw. 5 % POPC bzw. PEO-b-PBD. PEO-b-PBD wurde verwendet, um die Membranstabilität während der Proteinexpression zu verbessern, wurde jedoch in einem niedrigen molaren Verhältnis gehalten, um die Tendenz des Copolymers zu reduzieren, zu Mizellen zu aggregieren, die von den Lipidmolekülen getrennt sind29,30. - Blasen Sie vorsichtig einen sanften Strahl Argongas in das Glasfläschchen, während Sie das Fläschchen drehen, um das Chloroform zu verdampfen.
- Pipettieren Sie 1,2 mL helles Mineralöl in das 15 mL Glasfläschchen mit den getrockneten Lipiden.
- Mischen Sie die Lipide und das Öl, indem Sie sie 10-20 s lang bei maximaler Geschwindigkeit vortexen. Das gelöste Lipid-Copolymer-Gemisch sieht trüb aus.
- Um sicherzustellen, dass alle möglichen Lipidaggregate im Öl vollständig aufgelöst und im Öl verteilt sind, stellen Sie das Glasfläschchen 20 Minuten lang bei ca. 50 °C in einen Ofen, bevor Sie es weitere 10-20 s bei maximaler Geschwindigkeit vortexen.
- Mischen Sie in einem Abzug 17,3 μl 25 mg/ml POPC-Stammlösung und 1,08 μl 50 mg/ml Poly(butadien)-b-poly(ethylenoxid) (PEO-b-PBD)-Copolymer in einem 15-ml-Glasfläschchen.
- Assemblierung und Verkapselung der CFE-Reaktion (Abbildung 1 fasst die folgenden Schritte zusammen).
- Bereiten Sie 300 μl der GUV-Außenlösung in einem 1,5-ml-Mikrozentrifugenröhrchen vor, indem Sie 270 μl der in Schritt 2.1 hergestellten CFE-Außenpufferlösung, 15 μl 5 M NaCl und 15 μl 4,5 M KCl mischen.
HINWEIS: In diesem Schritt werden 0,45 μl 1 M 1,4-Dithiothreitol (DTT) in die äußere GUV-Lösung gegeben. Der Zweck der Zugabe von NaCl und KCl besteht darin, die Osmolalität der äußeren Lösung so anzupassen, dass sie mit der inneren CFE-Lösung übereinstimmt. Das genaue Volumen von NaCl und KCl hängt von der gewünschten Osmolalitätseinstellung ab. Man kann entweder NaCl oder KCl oder beides hinzufügen, um die Osmolalität einzustellen. - Pipettieren Sie vorsichtig 300 μl des Lipid-in-Öl-Gemisches auf die äußere GUV-Lösung.
HINWEIS: Bei der Zugabe der Lipid-Öl-Mischung ist es wichtig, dass sich das Öl nicht mit der wässrigen äußeren Lösung vermischt. Nach der Zugabe sollte eine sichtbare Grenzfläche zwischen dem Lipid-in-Öl-Gemisch und der GUV-Außenlösung vorhanden sein. - Die Öl-Wasser-Grenzfläche wird 2 Stunden lang bei Raumtemperatur inkubiert, damit sich die Lipid-Monoschicht an der Grenzfläche bilden und stabilisieren kann.
- In der Zwischenzeit ist Abschnitt 1.2.1 zu befolgen, um eine CFE-Reaktion zu assemblieren, die die Plasmid-DNA enthält, die für die Membranproteinvarianten oder lösliches GFP kodiert. Ersetzen Sie die 4 μl 5 mM SUV-Lösung oder Wasser durch 4 μl 1 M Saccharose. Diese Reaktion wird die innere Lösung des GUV sein.
HINWEIS: Zur Messung der Osmolalität der inneren und äußeren Lösungen wurde ein Osmometer verwendet. Die Osmolalität der äußeren Lösung wurde dann durch Zugabe von NaCl oder Wasser entsprechend eingestellt. Die Osmolalität der CFE-Reaktion liegt typischerweise bei etwa 1600 mOsm/kg. Die Zugabe von Saccharose zur Reaktion erhöht die innere Lösungsdichte, so dass die Vesikel während des Zentrifugationsschritts über die Öl-Wasser-Grenzfläche wandern können. Eine Alternative zu Saccharose ist die Opti-Prep Dichtegradientenlösung. - Geben Sie 600 μl Lipid-Öl-Gemisch in das Mikrozentrifugenröhrchen, das die CFE-Reaktion enthält, und pipettieren Sie ~1 Minute lang kräftig auf und ab, um die Reaktion in der Lipid-in-Öl-Lösung zu emulgieren und die Lipid-Monoschicht um die synthetischen Zellen zu bilden.
HINWEIS: Die endgültige Lösung sollte keine Blasen aufweisen und undurchsichtig aussehen. - Pipettieren Sie die innere Lösungsemulsion vorsichtig auf die Ölschicht in das 1,5-ml-Mikrozentrifugenröhrchen, in dem die Öl-Wasser-Grenzfläche eingerichtet wurde.
HINWEIS: Achten Sie darauf, die Benutzeroberfläche nicht zu stören oder zu destabilisieren. - 10 min bei 2.000 x g bei 4 °C zentrifugieren.
HINWEIS: Die Zentrifugationsgeschwindigkeit wurde für dieses Protokoll optimiert. Adir et al.31 berichteten über eine andere Zentrifugationsgeschwindigkeit. - Entfernen Sie nach Beendigung der Zentrifugation vorsichtig das überschüssige Öl und die äußere Lösung mit einer Pipette aus dem Mikrozentrifugenröhrchen. Entfernen Sie die äußere Lösung, bis das verbleibende Volumen etwa 100 μl beträgt.
HINWEIS: Ein Pellet aus GUVs ist normalerweise am Boden des Mikrozentrifugenröhrchens sichtbar. Das Fehlen eines sichtbaren Pellets bedeutet jedoch nicht zwangsläufig, dass keine GUV-Ausbeute erzielt wird. Anstatt eine Pipette zu verwenden, kann das überschüssige Öl auf der äußeren Lösung durch Aspiration entfernt werden. Es ist wichtig sicherzustellen, dass die Lipid-in-Öl-Lösung vollständig entfernt wird. Ölverunreinigungen in der GUV-Lösung können zu Bildern mit geringer Qualität führen. - Resuspendieren Sie das GUV-Pellet in der restlichen 100-μl-Lösung, indem Sie es vorsichtig auf und ab pipettieren. Übertragen Sie anschließend die GUV-Lösung zur Inkubation auf eine saubere 96-Well-Platte mit klarem flachem Boden.
- Bereiten Sie 300 μl der GUV-Außenlösung in einem 1,5-ml-Mikrozentrifugenröhrchen vor, indem Sie 270 μl der in Schritt 2.1 hergestellten CFE-Außenpufferlösung, 15 μl 5 M NaCl und 15 μl 4,5 M KCl mischen.
3. Inkubation und Bildgebung von verkapselten CFE-Reaktionen
- Decken Sie die Platte mit einer Siegelfolie ab, um eine Verdunstung zu verhindern. Inkubieren Sie die Platte bei 37 °C für 5-6 h. Man kann einen Plattenleser verwenden und Schritt 1.3.2 befolgen, um den Plattenleser für den Inkubationsschritt vorzubereiten.
- Sobald die Inkubation abgeschlossen ist, stellen Sie die 96-Well-Platte auf den Bildgebungstisch eines inversen Mikroskops, das mit einer EMCCD-Kamera (oder einer sCMOS-Kamera), einem DAQ-MX-gesteuerten Laser (oder einem integrierten Laserkombiniersystem) und einer konfokalen CSU-X1-Spinning-Disk (oder einem konfokalen Laserscanning) ausgestattet ist. Fokussieren Sie sich auf beliebige ROI-Aufnahmen mit GUVs und nehmen Sie Bilder bei einer Anregungswellenlänge von 488 nm mit einem Plan-Apochromat 60 x/1,4 NA-Objektiv auf.
- Speichern Sie Bilder von GUVs in .tiff Format.
- Öffnen Sie die Bilder in einer Bildbearbeitungssoftware (z.B. ImageJ oder Fiji). Öffnen Sie das Einstellungsfeld für Helligkeit/Kontrast . Passen Sie die Helligkeit und den Kontrast an die entsprechenden Einstellungen an, um fluoreszierende Proteine sichtbar zu machen.
- Wenn das Ziel darin besteht, die Signalintensität verschiedener exprimierter Proteine zu vergleichen, stapeln Sie zunächst einzelne Bilder von GUVs, die unterschiedliche Proteine enthalten, mithilfe des Bedienfelds "Bilder zum Stapeln ", das sich im Untermenü "Bild > Stapel" befindet. Passen Sie dann die Helligkeit und den Kontrast aller Bilder mit dem Bedienfeld Helligkeit/Kontrast an.
Ergebnisse
Vor der Verkapselung der CFE-Reaktionen wurden zwei Varianten von GluR0-sfGFP, die native und Proteorhodopsin-Signalpeptide beherbergen (die Signalpeptidsequenzen sind in der ergänzenden Tabelle 1 dargestellt), und das lösliche sfGFP einzeln in Massenreaktionen exprimiert, und ihre Expression wurde durch Detektion des sfGFP-Signals mit einem Plattenlesegerät überwacht (Abbildung 2A). Membranproteine wurden in Abwesenheit oder Anwesenheit von 100 nm SUVs exprimiert. Zusätzlich wurden unter Verwendung einer Kalibrierungskurve, die das sfGFP-Signal mit seiner Konzentration korreliert (Ergänzende Abbildung 1), die Konzentrationen der synthetisierten Proteine geschätzt (Ergänzende Tabelle 2). Offensichtlich hatte lösliches sfGFP die höchste Expression unter allen drei Proteinen, was darauf hindeutet, dass die Expression von Membranproteinen eine Belastung für das CFE-System darstellt, wodurch die Reaktion verlangsamt und ihre Ausbeute verringert wird. Darüber hinaus zeigten Reaktionen, die Membranproteine in Gegenwart von SUVs exprimierten, im Durchschnitt ein höheres sfGFP-Signal im Vergleich zu Reaktionen, bei denen SUVs fehlten. Diese Beobachtung stimmt mit den Ergebnissen von Steinküher et al. überein, die zeigten, dass die Expression von Membranproteinen die Fähigkeit der CFE-Systeme zur Produktion von Proteinen verringert23. Angesichts der erfolgreichen Demonstration der Proteinexpression in der CFE-Massenreaktion kann man jedoch davon ausgehen, dass verkapseltes CFE auch Proteine in GUVs synthetisiert.
Als nächstes wurden einzelne CFE-Reaktionen in GUVs unter Verwendung der invertierten Emulsionsmethode verkapselt, um Varianten von GluR0, nämlich WT-GluR0, PRSP-GluR0 und lösliches sfGFP, zu exprimieren. Während WT-GluR0, das das native GluR0-Signalpeptid beherbergt, eine ausgezeichnete Expression und Membranlokalisation zeigte (Abbildung 2B, linkes Bild), zeigte sein Gegenstück, PRSP-GluR0, das über ein Proteorhodopsin-N-terminales Signalpeptid verfügt, keine ähnlich starke Membranlokalisation. Es wurde festgestellt, dass PRSP-GluR0 anfälliger für Aggregation und Punktbildung ist (Abbildung 2B, mittleres Bild). Wie erwartet wurde lösliches sfGFP in GUVs exprimiert und blieb im GUV-Lumen (Abbildung 2B, rechtes Bild; siehe Ergänzende Abbildung 2 für Bilder von Kohorten von GUVs).
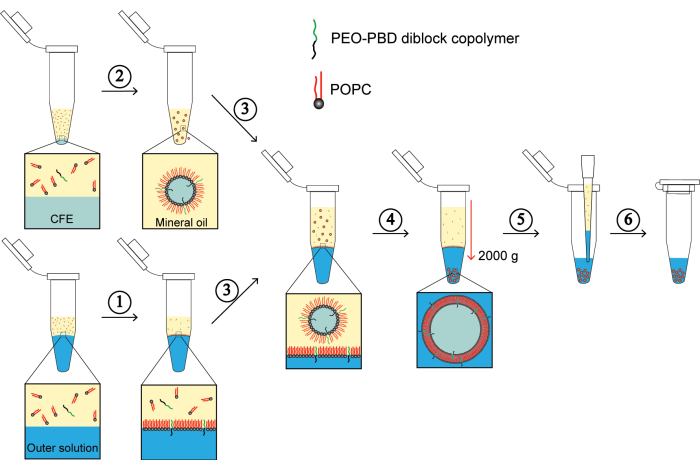
Abbildung 1: Experimentelle Schritte der invertierten Emulsion. (1) Die Schritte 2.3.1 bis 2.3.3 des Protokolls werden visualisiert, um den Aufbau der Lipidmonoschicht an der Grenzfläche zwischen dem Lipid-Öl-Gemisch und der äußeren Pufferlösung zu demonstrieren. (2) Die Visualisierung von Schritt 2.3.5 des Protokolls ist hier dargestellt, um die Bildung der Lipidmonoschicht um emulgierte Tröpfchen darzustellen, die die innere CFE-Lösung einkapseln. (3) Schritt 2.3.6 des Protokolls zeigt die Zugabe der Monolagen-GUVs zum Mikrozentrifugenröhrchen mit der Lipidmonoschicht an der Grenzfläche eines Lipid-Öl-Gemisches und einer äußeren Pufferlösung. (4) Hier ist der Schritt 2.3.7 dargestellt, in dem die Zentrifugation zur Bildung eines GUV-Pellets in der äußeren Lösung führt. (5) Hier ist Schritt 2.3.8 dargestellt, der den Prozess der Entfernung des überschüssigen Lipid-in-Öl-Gemisches und der äußeren Lösung angibt. (6) Schließlich ist hier Schritt 2.3.9 dargestellt, in dem das GUV-Pellet in der äußeren Lösung resuspendiert wird und die GUVs zur Inkubation bereit sind, gefolgt von der Bildgebung. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
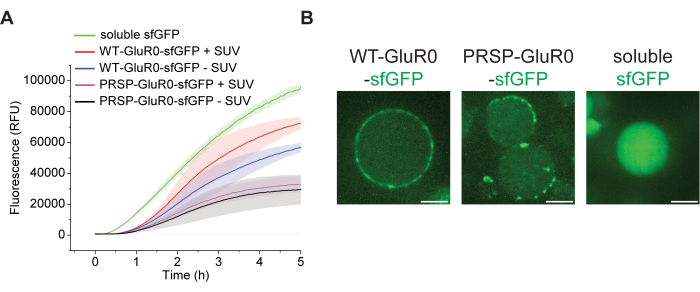
Abbildung 2: Proteinexpression in CFE-Massenreaktionen und in GUVs, die CFE-Reaktionen verkapseln. (A) Fluoreszenzmessungen einzelner CFE-Massenreaktionen, die WT-GluR0-sfGFP, PRSP-GluR0-sfGFP und lösliches sfGFP exprimieren. Das lösliche sfGFP-Diagramm stellt das Signal einer 2,5-μl-Reaktion dar (das Standardreaktionsvolumen beträgt 20 μl), um eine Übersättigung der Plattenlesermessungen zu vermeiden. Die Daten werden als Mittelwert ± S.D, n = 3 dargestellt. (B) Links: Ein repräsentatives konfokales Bild einer GUV-verkapselnden CFE-Reaktion, die WT-GluR0-sfGFP exprimiert. Mitte: Ein repräsentatives konfokales Bild von GUVs, die die CFE-Reaktion verkapseln, die PRSP-GluR0-sfGFP exprimiert. Rechts: Ein repräsentatives konfokales Bild einer GUV-verkapselnden CFE-Reaktion, die lösliches sfGFP exprimiert. Maßstabsbalken: 10 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Ergänzende Abbildung 1: Die sfGFP-Signalkalibrierungskurve und die entsprechende lineare Regressionsanalyse. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Abbildung 2: Repräsentatives konfokales Bild von Kohorten von GUVs, die (links) WT-GluR0-sfGFP, (Mitte) PRSP-GluR0-sfGFP und (rechts) lösliches sfGFP exprimieren. Maßstabsleiste: 10 μm. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Tabelle 1: Die Aminosäuresequenz von Wildtyp- und Proteorhodopsin-Signalpeptiden. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Tabelle 2: Die Konzentration synthetisierter Proteine in CFE-Massenreaktionen. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Diskussion
Praktisch jeder zelluläre Prozess, der von der Übertragung von Molekülen oder Informationen über die Zellmembran abhängt, wie z. B. die Zellsignalisierung oder die Zellanregung, erfordert Membranproteine. Daher ist die Rekonstitution von Membranproteinen zum Hauptengpass bei der Realisierung verschiedener synthetischer Zelldesigns für unterschiedliche Anwendungen geworden. Die traditionelle Detergenzien-vermittelte Rekonstitution von Membranproteinen in biologischen Membranen erfordert GUV-Erzeugungsmethoden wie sanftes Quellen oder Elektroformation. Quellungsansätze erzeugen in der Regel kleine Vesikel, und die Elektroformation führt zu einem signifikanten Abfall, wenn komplizierte Lösungen, was häufig bei der Erzeugung synthetischer Zellen der Fall ist, eingekapselt werden32. Darüber hinaus lösen Detergenzien das Membranprotein auf, und ihre Entfernung während des Rekonstitutionsprozesses kann zu einer Fehlfaltung des Proteins führen33,34. Andererseits beruht der hier vorgestellte Ansatz auf dem kotranslationalen Einbau des Membranproteins in die Lipiddoppelschicht, der eher dem natürlichen Proteinbiogeneseweg in Zellen ähnelt22.
Aus technischer Sicht ist das vorgestellte Protokoll vorteilhaft gegenüber anderen gängigen Verkapselungsmethoden, wie z. B. der Elektroformation und der kontinuierlichen Tröpfchengrenzflächenverkapselung 7,8,35,36 (cDICE), da die einzige für die GUV-Erzeugung erforderliche Laborausrüstung eine Zentrifuge ist. Im Gegensatz zur Elektroformung ermöglicht das Verfahren der invertierten Emulsion die Verkapselung verschiedener Kombinationen von Molekülen mit unterschiedlichen Konzentrationen. Darüber hinaus erzeugt dieser Ansatz im Vergleich zur ursprünglichen invertierten Emulsionstechnik25 stabilere GUVs, die für die Verkapselung von CFE-Lysaten oder PURE-Systemen geeignet sind. Die höhere GUV-Stabilität ist auf das Vorhandensein von Diblock-Copolymer in der Zusammensetzung der GUV-Membran37 sowie auf die lange Inkubation der Öl-Wasser-Grenzfläche zurückzuführen, die es ermöglicht, die Grenzfläche mit Lipidmolekülen zu sättigen. Im Gegensatz zu mikrofluidischen Ansätzen benötigt das hier vorgestellte Protokoll keine kleinen Kanäle und Schläuche. Daher kann die CFE-Reaktion sofort nach dem Zusammenbau eingekapselt werden, und die kürzere Zeit der GUV-Assemblierung aufgrund von mangelnder Strömung und möglicher Verstopfung verhindert einen vorzeitigen Start der CFE-Reaktion. Während die Demonstration der Membranproteinexpression in diesem Protokoll ausschließlich für PUREfrex-Reaktionen gilt, kann diese Methode auf die Synthese von Proteinen mit verschiedenen verfügbaren CFE-Systemen erweitert werden, wie z. B. lysatbasierten bakteriellen oder Säugetier-CFE-Systemen.
Der hier vorgestellte Ansatz weist Einschränkungen auf, die durch die ölabhängige Natur des GUV-Entstehungsprozesses und die Absicht, stabile GUVs zu haben, verursacht werden. Dieser Ansatz ist im Vergleich zu anderen Methoden, wie cDICE oder Mikrofluidik, in der Regel länger, da die lange Inkubationszeit der Öl-Wasser-Grenzfläche, die für die Grenzflächenstabilisierung und eine hohe GUV-Ausbeute erforderlich ist, lange Zeit hat. Darüber hinaus ist die Lipidzusammensetzung in erster Linie auf POPC mit geringen Dosen anderer Lipide oder Blockcopolymere beschränkt, während andere Methoden, wie z. B. die Elektroformation, eher für den Einbau von Lipiden mit unterschiedlichen physikalischen und chemischen Eigenschaften geeignet sind. Während die Zusammensetzung der GUV-Membran bei dieser Methode eine Mischung aus POPC und PBD-PEO ist, um die CFE-Ausbeute zu maximieren, können mögliche Variationen in der Zusammensetzung der GUV-Membran getestet werden. Für andere Membranproteine könnte jedoch eine weitere Optimierung der Parameter erforderlich sein. Da die Tröpfchenemulgierung durch manuelles Pipettieren erfolgt, sind die mit dieser Methode erzeugten GUVs polydispers und recht heterogen groß. Darüber hinaus kann die Tatsache, dass Lipide in der organischen Phase gelöst sind, gelegentlich eine Ölschicht zwischen den beiden Segeln der GUV-Membran verursachen oder die Bildgebungskammer mit Öl verunreinigen, was sich nachteilig auf die Bildqualität auswirken kann. Eine mögliche Lösung für die Herausforderung des Restöls besteht darin, Mineralöl durch ein flüchtiges organisches Lösungsmittel wie Diethylether zu ersetzen, wie von Tsumoto et al.38 gezeigt, um sich auf die Lösungsmittelverdampfung zusammen mit der Zentrifugation während der GUV-Bildung zu verlassen.
Obwohl es in dieser Arbeit keine Demonstration der Kanalfunktion gibt, wird in Anlehnung an frühere Assays, die zur Untersuchung der rekonstituierten mechano- oder lichtempfindlichen Kanalfunktionalität verwendet wurden, ein auf Fluoreszenzmikroskopie basierender Assay skizziert. Es wird berichtet, dass die Öffnung des GluR0-Kanals die Membranleitfähigkeit für K+ -Ionenerhöht 24. Da CFE-Reaktionen bereits eine hohe Konzentration an K+ enthalten, sind typische Kaliumindikatoren nicht geeignet, um die Funktionalität des Kanals zu beurteilen. Da jedoch der Kaliumeinstrom das Membranpotential verändert, könnten empfindliche Membranpotentialindikatoren wie DiBAC4(3)22 oder BeRST 139 die GluR0-Aktivität in Gegenwart von Glutamat anzeigen.
Die erfolgreiche Rekonstitution von Membranproteinen in synthetischen Zellen eröffnet zahlreiche Möglichkeiten, synthetische Zellen mit noch nie dagewesenen Fähigkeiten zu schaffen, die natürlichen Zellen näher kommen. Ein großer Nachteil synthetischer Zellen ist derzeit ihre Unfähigkeit, Energie zu reproduzieren und zu recyceln. Mit licht- und chemikalienabhängigen Energieregenerationsschemata, die stark auf Membranproteinen beruhen, kann man sich jedoch langlebige synthetische Zellen vorstellen40. Die Verwendung von CFE-Systemen ermöglicht die Rekonstitution mehrerer Membranproteine, die gemeinsam bestimmte Aufgaben erfüllen können. Zum Beispiel kann die hier beschriebene Rekonstitution eines ligandengesteuerten Ionenkanals ähnlich wie GluR0 zusammen mit verschiedenen spannungsgesteuerten Ionenkanälen zur Konstruktion einer erregbaren neuronenähnlichen synthetischen Zelle führen.
Offenlegungen
Die Autoren erklären, dass keine Interessenkonflikte bestehen.
Danksagungen
APL bedankt sich für die Unterstützung durch die National Science Foundation (EF1935265), die National Institutes of Health (R01-EB030031 und R21-AR080363) und das Army Research Office (80523-BB)
Materialien
| Name | Company | Catalog Number | Comments |
| 100 nm polycarbonate filter | STERLITECH | 1270193 | |
| 96 Well Clear Bottom Plate | ThermoFisher Scientific | 165305 | |
| BioTek Synergy H1M Hybrid Multi-Mode Reader | Agilent | 11-120-533 | |
| Creatine phosphate | Millipore Sigma | 10621714001 | |
| CSU-X1 Confocal Scanner Unit | Yokogawa | CSU-X1 | |
| Density gradient medium (Optiprep) | Millipore Sigma | D1556 | Optional to switch with sucrose in inner solution |
| Filter supports | Avanti | 610014 | |
| Fisherbrand microtubes (1.5 mL) | Fisher Scientific | 05-408-129 | |
| Folinic acid calcium salt hydrate | Millipore Sigma | F7878 | |
| Glucose | Millipore Sigma | 158968 | |
| HEPES | Millipore Sigma | H3375 | |
| iXon X3 camera | Andor | DU-897E-CS0 | |
| L-Glutamic acid potassium salt monohydrate | Millipore Sigma | G1501 | |
| Light mineral oil | Millipore Sigma | M5904 | |
| Magnesium acetate tetrahydrate | Millipore Sigma | M5661 | |
| Mini-extruder kit (including syringe holder and extruder stand) | Avanti | 610020 | |
| Olympus IX81 Inverted Microscope | Olympus | IX21 | |
| Olympus PlanApo N 60x Oil Microscope Objective | Olympus | 1-U2B933 | |
| PEO-b-PBD | Polymer Source | P41745-BdEO | |
| pET28b-PRSP-GluR0-sfGFP plasmid DNA | Homemade | N/A | |
| pET28b-sfGFP-sfCherry(1-10) plasmid DNA | Homemade | N/A | |
| pET28b-WT-GluR0-sfGFP plasmid DNA | Homemade | N/A | |
| POPC lipid in chloroform | Avanti | 850457C | |
| Potassium chloride | Millipore Sigma | P9541 | |
| PUREfrex 2.0 | Cosmo Bio USA | GFK-PF201 | |
| Ribonucleotide Solution Set | New England BioLabs | N0450 | |
| RNase Inhibitor, Murine | New England BioLabs | M0314S | |
| RTS Amino Acid Sampler | Biotechrabbit | BR1401801 | |
| Sodium chloride | Millipore Sigma | S9888 | |
| Spermidine | Millipore Sigma | S2626 | |
| Sucrose | Millipore Sigma | S0389 | |
| VAPRO Vapor Pressure Osmometer Model 5600 | ELITechGroup | VAPRO 5600 |
Referenzen
- Liu, A. P., Fletcher, D. A. Biology under construction: In vitro reconstitution of cellular function. Nat Rev Mol Cell Biol. 10 (9), 644-650 (2009).
- Lin, A. J., Sihorwala, A. Z., Belardi, B. Engineering tissue-scale properties with synthetic cells: Forging one from many. ACS Synth Biol. 12 (7), 1889-1907 (2023).
- Powers, J., Jang, Y. Advancing biomimetic functions of synthetic cells through compartmentalized cell-free protein synthesis. Biomacromolecules. 24 (12), 5539-5550 (2023).
- Jiang, W., et al. Artificial cells: Past, present and future. ACS Nano. 16 (10), 15705-15733 (2022).
- Groaz, A., et al. Engineering spatiotemporal organization and dynamics in synthetic cells. WIREs Nanomed Nanobiotech. 13 (3), 1685 (2021).
- Sharma, B., Moghimianavval, H., Hwang, S. W., Liu, A. P. Synthetic cell as a platform for understanding membrane-membrane interactions. Membranes. 11 (12), 912 (2021).
- Bashirzadeh, Y., et al. Actin crosslinker competition and sorting drive emergent GUV size-dependent actin network architecture. Commun Biol. 4 (1), 1-11 (2021).
- Bashirzadeh, Y., Moghimianavval, H., Liu, A. P. Encapsulated actomyosin patterns drive cell-like membrane shape changes. iScience. 25 (5), 104236 (2021).
- Berhanu, S., Ueda, T., Kuruma, Y. Artificial photosynthetic cell producing energy for protein synthesis. Nat Commun. 10 (1), 1325 (2019).
- Ji, Y., Chakraborty, T., Wegner, S. V. Self-regulated and bidirectional communication in synthetic cell communities. ACS Nano. 17 (10), 8992-9002 (2023).
- Moghimianavval, H., Loi, K. J., Hwang, S. W., Bashirzadeh, Y., Liu, A. P. Light-based juxtacrine signaling between synthetic cells. bioRxiv. , (2024).
- Boyd, M. A., Thavarajah, W., Lucks, J. B., Kamat, N. P. Robust and tunable performance of a cell-free biosensor encapsulated in lipid vesicles. Science Advances. 9 (1), 6605 (2023).
- Schneider, B., et al. Membrane Protein expression in cell-free systems. Methods Mol Biol. 601, 165-186 (2010).
- Noireaux, V., Liu, A. P. The new age of cell-free biology. Annu Rev Biomed Eng. 22 (1), 51-77 (2020).
- Majumder, S., et al. Cell-sized mechanosensitive and biosensing compartment programmed with DNA. Chem. Commun. 53 (53), 7349-7352 (2017).
- Poddar, A., et al. Membrane stretching activates calcium permeability of a putative channel Pkd2 during fission yeast cytokinesis. MBoC. 33 (14), (2022).
- Sachse, R., Dondapati, S. K., Fenz, S. F., Schmidt, T., Kubick, S. Membrane protein synthesis in cell-free systems: From bio-mimetic systems to bio-membranes. FEBS Letters. 588 (17), 2774-2781 (2014).
- Dondapati, S. K., et al. Functional reconstitution of membrane proteins derived from eukaryotic cell-free systems. Front Pharmacol. 10, 917 (2019).
- Majumder, S., et al. In vitro synthesis and reconstitution using mammalian cell-free lysates enables the systematic study of the regulation of LINC complex assembly. Biochemistry. 61 (14), 1495-1507 (2022).
- Niwa, T., et al. Comprehensive study of liposome-assisted synthesis of membrane proteins using a reconstituted cell-free translation system. Sci Rep. 5 (1), 18025 (2015).
- Moghimianavval, H., Hsu, Y. Y., Groaz, A., Liu, A. P. In vitro reconstitution platforms of mammalian cell-free expressed membrane proteinsmembrane proteins. Methods Mol Biol. 2433, 105-120 (2022).
- Eaglesfield, R., Madsen, M. A., Sanyal, S., Reboud, J., Amtmann, A. Cotranslational recruitment of ribosomes in protocells recreates a translocon-independent mechanism of proteorhodopsin biogenesis. iScience. 24 (5), 102429 (2021).
- Steinküher, J., et al. Improving cell-free expression of membrane proteins by tuning ribosome cotranslational membrane association and nascent chain aggregation. bioRxiv. , (2023).
- Chen, G. Q., Cui, C., Mayer, M. L., Gouaux, E. Functional characterization of a potassium-selective prokaryotic glutamate receptor. Nature. 402 (6763), 817-821 (1999).
- Pautot, S., Frisken, B. J., Weitz, D. A. Production of unilamellar vesicles using an inverted emulsion. Langmuir. 19 (7), 2870-2879 (2003).
- Hsu, Y. Y., et al. Calcium-triggered DNA-mediated membrane fusion in synthetic cells. Chemical Communications. 59 (57), 8806-8809 (2023).
- Moghimianavval, H., et al. Engineering functional membrane-membrane interfaces by interspy. Small. 19 (13), 2202104 (2023).
- Sun, Z. Z., et al. Protocols for implementing an Escherichia coli based TX-TL cell-free expression system for synthetic biology. J Vis Exp. (79), e50762 (2013).
- Jacobs, M. L., Boyd, M. A., Kamat, N. P. Diblock copolymers enhance folding of a mechanosensitive membrane protein during cell-free expression. PNAS. 116 (10), 4031-4036 (2019).
- Kostarelos, K., Tadros, T. F., Luckham, P. F. Physical conjugation of (tri-) block copolymers to liposomes toward the construction of sterically stabilized vesicle systems. Langmuir. 15 (2), 369-376 (1999).
- Adir, O., et al. Preparing protein producing synthetic cells using cell free bacterial extracts, liposomes and emulsion transfer. J Vis Exp. (158), e60829 (2020).
- van de Cauter, L., van Buren, L., Koenderink, G. H., Ganzinger, K. A. Exploring giant unilamellar vesicle production for artificial cells - current challenges and future directions. Small Methods. 7 (12), 2300416 (2023).
- Seddon, A. M., Curnow, P., Booth, P. J. Membrane proteins, lipids and detergents: Not just a soap opera. Biochim Biophys Acta Biomembr. 1666 (1), 105-117 (2004).
- Guo, Y. Detergent-free systems for structural studies of membrane proteins. Biochem Soc Trans. 49 (3), 1361-1374 (2021).
- Bashirzadeh, Y., Wubshet, N., Litschel, T., Schwille, P., Liu, A. P. Rapid encapsulation of reconstituted cytoskeleton inside giant unilamellar vesicles. J Vis Exp. (177), e63332 (2021).
- Hwang, S. W., et al. Hybrid vesicles enable mechano-responsive hydrogel degradation. Angew Chemie Int Ed. 62 (41), e202308509 (2023).
- Rideau, E., Dimova, R., Schwille, P., Wurm, F. R., Landfester, K. Liposomes and polymersomes: a comparative review towards cell mimicking. Chem Soc Rev. 47 (23), 8572-8610 (2018).
- Tsumoto, K., Hayashi, Y., Tabata, J., Tomita, M. A reverse-phase method revisited: Rapid high-yield preparation of giant unilamellar vesicles (GUVs) using emulsification followed by centrifugation. Colloids Surf A: Physicochem Eng Asp. 546, 74-82 (2018).
- Huang, Y. L., Walker, A. S., Miller, E. W. A photostable silicon rhodamine platform for optical voltage sensing. J Am Chem Soc. 137 (33), 10767-10776 (2015).
- Bailoni, E., et al. Minimal out-of-equilibrium metabolism for synthetic cells: A membrane perspective. ACS Synth Biol. 12 (4), 922-946 (2023).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten