Method Article
Hibridização da Reação em Cadeia de RNA Fluorescência de Monte Inteiro Hibridização in situ de Genes Quimiossensoriais em Apêndices Olfativos de Mosquitos
Neste Artigo
Resumo
O artigo descreve os métodos e reagentes necessários para realizar a hibridização em cadeia de reação em cadeia de hibridização por fluorescência completa de RNA (HCR, RNA, WM-FISH) para revelar informações sobre a resolução espacial e celular de genes de receptores quimiossensoriais na antena do mosquito e palpo maxilar.
Resumo
Os mosquitos são vetores eficazes de doenças mortais e podem navegar em seu ambiente químico usando receptores quimiossensoriais expressos em seus apêndices olfativos. Compreender como os receptores quimiossensoriais estão organizados espacialmente nos apêndices olfativos periféricos pode oferecer insights sobre como o odor é codificado no sistema olfatório do mosquito e informar novas maneiras de combater a disseminação de doenças transmitidas por mosquitos. O surgimento da hibridização de terceira geração de reação em cadeia de RNA, hibridação in situ de montagem total de RNA (HCR, RNA, WM-FISH) permite o mapeamento espacial e o perfil de expressão simultânea de múltiplos genes quimiossensoriais. Aqui, descrevemos uma abordagem passo a passo para a realização do HCR RNA WM-FISH na antena do mosquito Anopheles e palpo maxilar. Investigamos a sensibilidade desta técnica examinando o perfil de expressão dos receptores olfatórios ionotrópicos. Perguntamos se a técnica de HCR WM-FISH descrita era adequada para estudos multiplexados por meio da ligação de sondas de RNA a três fluoróforos espectralmente distintos. Os resultados forneceram evidências de que o HCR RNA WM-FISH é robustamente sensível para detectar simultaneamente múltiplos genes quimiossensoriais na antena e apêndices olfativos palpos maxilares. Investigações posteriores atestam a adequação do HCR WM-FISH para o perfil de co-expressão de alvos de RNA duplo e triplo. Esta técnica, quando aplicada com modificações, pode ser adaptável para localizar genes de interesse nos tecidos olfativos de outras espécies de insetos ou em outros apêndices.
Introdução
Mosquitos vetores como Anopheles gambiae dependem de um rico repertório de genes quimiossensoriais expressos em seus apêndices olfativos periféricos para prosperar em um mundo químico complexo e identificar odores comportamentalmente relevantes emanados de hospedeiros humanos, detectar fontes de néctar e localizar sítios de oviposição1. A antena do mosquito e o palpo maxilar são enriquecidos com genes quimiossensoriais que conduzem a detecção de odor nesses apêndices olfativos. Três classes principais de canais iônicos ligados a ligantes conduzem a detecção de odor nos apêndices olfativos de mosquitos: os receptores odorantes (ORs), que funcionam com um co-receptor odorante obrigatório (Orco); os receptores ionotrópicos (RIs), que interagem com um ou mais correceptores de RI (IR8a, IR25a e IR76b); os receptores gustativos quimiossensoriais (RGs), que funcionam como um complexo de três proteínas para detectar dióxido de carbono (CO2)1,2.
A hibridização in situ por fluorescência de RNA é uma ferramenta poderosa para detectar a expressão deRNAm 3 endógeno. Em geral, este método utiliza uma sonda de ácido nucleico de fita simples marcada com fluoróforo com sequência complementar a um RNAm alvo. A ligação da sonda de RNA fluorescente ao RNA alvo permite a identificação de células que expressam um transcrito de interesse. Avanços recentes agora permitem a detecção de transcritos em tecidos de mosquitos de montagem total 4,5. A primeira geração da tecnologia de reação em cadeia de hibridização (HCR) usou um amplificador HCR baseado em RNA; isso foi melhorado em um método de segunda geração que, em vez disso, usou DNA projetado para o amplificador HCR 6,7. Essa atualização resultou em um aumento de 10x no sinal, uma redução drástica no custo de produção e uma melhoria significativa na durabilidade dos reagentes 6,7.
No protocolo, descrevemos a utilização de um método de hibridização in situ por fluorescência de RNA de terceira geração (HCR RNA WM-FISH) desenvolvido para detectar a localização espacial e expressão de qualquer gene 8,9. Este método de duas etapas utiliza primeiro sondas de ácido nucleico específicas para o mRNA de interesse, mas que também contêm uma sequência de reconhecimento do iniciador; a segunda etapa utiliza grampos marcados com fluoróforos que se ligam à sequência do iniciador para amplificar o sinal fluorescente (Figura 1). Este método também permite a multiplexação de duas ou mais sondas de RNA e amplificação de sinais de sonda para facilitar a detecção e quantificação de RNA8. A visualização da abundância de transcritos e dos padrões de localização de RNA de genes quimiossensoriais expressos nos apêndices olfativos oferece a primeira linha de conhecimento sobre as funções gênicas quimiossensoriais e a codificação de odores.
Protocolo
1. Considerações e preparação dos materiais
- Decida se a montagem completa ou a criossecção do tecido serão apropriadas. Este protocolo é otimizado para imagens in situ de RNA na antena do mosquito Anopheles e palpo maxilar sem criossecção. Se as amostras tiverem espessura superior a 5 mm, recomenda-se a criossecção para permitir a penetração da sonda.
- Identifique os genes de interesse e copie as sequências, incluindo íntrons e éxons de um banco de dados adequado. Transcreva a sequência gênica em RNA para síntese.
- Determine se as sondas devem ser compradas de fornecedores comerciais ou se as sondas serão sintetizadas em laboratório.
NOTA: Neste estudo, as sondas in situ foram adquiridas de um fornecedor comercial (ver Tabela de Materiais). Alternativamente, pode ser sintetizado como relatado anteriormente10. Os reagentes utilizados no estudo estão descritos na Tabela 1. Os materiais necessários para preparar os reagentes estão listados na Tabela de Materiais.
2. Pré-fixação tecidual
- Anestesiar 10-15 mosquitos Anopheles coluzzii adultos fêmeas ou machos (cepa N'Gousso), com idade entre 5 e 10 dias após a emergência, coletando-os com um aspirador bucal em um copo de papel ou tubo plástico e colocando-os em um balde contendo gelo. Uma anestesia induzida pelo frio bem-sucedida pode ser confirmada quando os mosquitos estão imóveis.
- Decapitá-los removendo as cabeças da região do pescoço com um par de pinças. Com a mão não dominante, segure o tórax com uma pinça e separe o pescoço do corpo com uma pinça segurada pela mão dominante. Colocar as cabeças num tubo de 1,5 ml contendo 500 μL de tampão quitinase-quimotripsina dimetilsulfóxido (tampão CCD) sobre gelo.
OBS: Animais congelantes podem deformar as antenas; Recomenda-se uma rápida derrubada no gelo. A idade do mosquito durante o teste pode variar dependendo do projeto. É importante confirmar e coordenar seu estado fisiológico, como se eles são alimentados com sangue, famintos ou acasalados. Neste estudo, apêndices olfativos foram amostrados de mosquitos que foram alimentados com sangue e acasalados. - Pré-aqueça as cabeças do mosquito em tampão CCD a 37 ° C em um bloco de calor por 5 min e, em seguida, transfira o tubo contendo as cabeças do mosquito para um forno de hibridização e gire a 37 ° C. O tempo de incubação no tampão CCD depende do tipo de tecido. Para antenas Anopheles fêmeas, use 20 min, antenas masculinas requerem 15 min, enquanto maior tempo de incubação (1 h) é necessário para os palpos maxilares.
- Transfira todo o conteúdo do tubo para um vidro de relógio dissecante. Despeje suavemente a amostra na depressão de um vidro de relógio (dissecação). Se as cabeças do mosquito estiverem presas dentro do tubo, use uma pipeta para adicionar tampão CCD no tubo e enxaguar a cabeça.
- Use pinça para transferir cuidadosamente as cabeças ou quaisquer antenas/palpos destacados durante a incubação e fixe em 1 mL do pré-fixador.
NOTA: O buffer CCD restante pode ser preservado a -20 ° C. O buffer pode ser reutilizado até 3x. Se o pré-fixador ficar levemente acastanhado por causa do transporte do tampão CCD pelos tecidos, substitua-o por mais 1 mL de fixador. - Gire as cabeças em pré-fixador por 24 h a 4° C usando um nutador.
OBS: A velocidade de balanço do nutador utilizado neste estudo não foi ajustável; Foi utilizada a velocidade padrão definida (12 rpm) pelo fabricante.
3. Dissecção tecidual
- Enxaguar as cabeças 4x (5 min por lavagem) com 1 mL de PBS-Tween a 0,1% no gelo. Para evitar a perda de amostra, use uma pipeta acoplada à ponta de carregamento de gel para remover o líquido.
NOTA: Duas lavagens rápidas seguidas de uma lavagem de 10 minutos e uma lavagem rápida final poderia ser realizada em vez da etapa 3.1. - Transfira as cabeças no tubo para um vidro de relógio dissecante. Despeje suavemente a amostra na depressão de um vidro de relógio. Enxágue as cabeças dos mosquitos que estão presas dentro do tubo com 0,1% de PBS-Tween.
- Sob um microscópio dissecante, remova os tecidos de interesse (antenas/palpos) da cabeça com pinças afiadas. Segure a parte posterior da cabeça com pinça e pegue uma antena com outra pinça da base. Pinça limpa com papel umedecido com solvente sem RNAse. Remova os palpos usando o mesmo processo.
- Transfira as antenas e palpos com pinças para tubos vazios sem DNA/RNase colocados no gelo. Separe as diferentes partes do tecido em tubos diferentes pré-marcados.
- Desidratar o tecido em 400 μL de solvente contendo uma mistura de metanol (MeOH 80%) e dimetilsulfóxido (DMSO 20%) por 1 h à temperatura ambiente.
NOTA: Não há necessidade de colocar tecido em um nutador; Deixe-o descansar em um rack de tubo à temperatura ambiente na bancada do laboratório. Recomenda-se o uso de mistura de solventes frescos. Para uma solução-mãe de 500 μL, misturar 400 μL de MeOH com 100 μL de DMSO. - Substitua o reagente desidratante por 400 μL de metanol absoluto (100%) e desidrate os tecidos durante a noite a -20 °C. Deixe os tecidos se acomodarem por gravidade e use uma pipeta para remover o líquido.
NOTA: As amostras são viáveis para um período prolongado de desidratação, até 4 noites foram testadas sem perda de sinal.
4. Pós-fixação tecidual
- Reidratar os tecidos em uma série de quatro etapas de 400 μL de MeOH/PBS-Tween por 10 min em gelo. Comece com 75% MeOH/25% PBS-Tween seguido por 50% MeOH/ 50% PBS-Tween, depois 25% MeOH/ 75% PBS-Tween e, finalmente, 100% PBS-Tween.
NOTA: Uma ponta de carregamento de gel com uma pequena abertura pode ser usada para remover o tampão do tubo para evitar a perda dos tecidos. A remoção do tampão pode ser feita sob um microscópio dissecante. - Lavar com 400 μL de solução salina tamponada com fosfato contendo Tween-20 a 0,1% (PBS-T) por 10 min à temperatura ambiente. Lave colocando a amostra num nutador.
- Fazer uma solução de 20 μg/mL de proteinase-K e incubar em 400 μL de solução de proteinase-K por 30 min à temperatura ambiente.
NOTA: Diluir 20 mg/mL de proteinase-K (solução estoque de 1000x), ou seja, 2 μL em 2 mL de PBS-Tween a 0,1%. As sobras podem ser armazenadas em um freezer de -20°C. - Pare a digestão enzimática do tecido lavando 2x por 10 min com 400 μL de PBS-tween a 0,1%.
- Adicionar 400 μL do pós-fixador e incubar durante 20 min à temperatura ambiente. Lavar 3x, 15 min por lavagem, com 400 μL de PBS-Tween a 0,1%.
5. Hibridização da sonda
- Incubar o tecido em 400 μL de tampão de hibridização por 5 min. Certifique-se de que o tecido está completamente submerso no tampão pipetando suavemente.
- Prepare-se para a próxima etapa aquecendo uma alíquota de tampão de hibridização de sonda a 37°C por 30 min.
- Retire o tampão e pré-hibridize com 400 μL de tampão de hibridização pré-aquecido por 30 min a 37°C.
- Fazer solução de sonda para os receptores quimiossensoriais alvo (IR8a, IR76b, IR25a, IR41t.1, IR75d, IR7t, IR64a ou Orco) adicionando 8 pM de sonda. Adicionar 8 μL de estoque de sonda de 1 μM a 500 μL de tampão de hibridização pré-aquecido da sonda.
- Remova o tampão de hibridização da sonda pré-aquecido e substitua-o por 400 μL de solução de sonda aquecida.
- Incubar o tecido a 37 ° C por duas noites em um nutador colocado dentro de uma incubadora e coberto sob uma caixa.
6. Amplificação da sonda
- Aqueça o tampão de lavagem da sonda a 37 ° C. Retire o excesso de solução da sonda enxaguando o tecido 5x, 10 min por lavagem, com 400 μL de tampão de lavagem da sonda a 37 ° C, nutando na incubadora.
NOTA: A preparação para o passo 6.4 pode começar. Veja a nota na etapa 6.4. - Lavar amostras 2x, 5 min por lavagem, com 400 μL de 5x citrato salino-sódico contendo 10% de Tween-20 (SSCT) à temperatura ambiente. Descongelar o tampão de amplificação à temperatura ambiente em uma bancada.
- Preparar o tecido para amplificação incubando com 400 μL de tampão de amplificação por 10 min à temperatura ambiente. Devido à viscosidade do tampão de amplificação, os tecidos podem não estar completamente submersos. Misture pipetando suavemente o tampão de amplificação abaixo do tecido e expelindo o tampão em cima do tecido até que eles fiquem submersos.
- Preparar separadamente 18 pM de hairpin h1 e 18 pM de hairpin h2 aquecendo 6 μL de estoque de 3 μM a 95 °C por 90 s e resfriar à temperatura ambiente em uma gaveta escura por 30 min. Certifique-se de que os tubos de PCR estejam bem tampados para evitar a evaporação dos grampos de cabelo durante o aquecimento em um termociclador.
NOTA: Para economizar tempo, a etapa 6.4 pode ser iniciada durante a4ª etapa de lavagem na etapa 6.1. Os grampos de cabelo devem ser aquecidos separadamente para evitar reação cruzada. Os grampos de cabelo não devem ser diluídos com qualquer tampão nesta etapa. Os grampos de cabelo podem ser comprados do fabricante da sonda junto com as sondas. - Substitua o tampão de amplificação adicionado no passo 6.3 por uma mistura contendo os grampos aquecidos (do passo 6.4) juntamente com 100 μL de tampão de amplificação. Uma reação típica conterá 6 μL de h1, 6 μL de h2 e 100 μL de tampão de amplificação.
NOTA: Os grampos h1 e h2 podem ser combinados após o resfriamento à temperatura ambiente e, em seguida, adicionados a 100 μL de tampão de amplificação. Para evitar a transferência das amostras de tecido de um tubo para outro, o tampão de amplificação na etapa 6.3 pode ser removido e substituído pela mistura de grampos diluídos em tampão de amplificação. - Incubar o tecido durante a noite e nutar no escuro à temperatura ambiente usando a velocidade padrão do nutador.
7. Montagem da amostra de tecido
- Tampão de amplificação diluído no tecido incubado com 300 μL de 5x SSCT (cloreto de sódio-citrato de sódio diluído em Tween-20).
NOTA: A diluição é útil para reduzir a viscosidade e facilita a remoção do tampão de amplificação do tecido. Utilizamos Triton-X 100 para o passo pré-fixativo e Tween-20 para o passo pós-fixativo devido às diferenças em suas ações como agentes de permeabilização da membrana celular. - Lavar o tecido 5x com 400 μL de 5x SSCT à temperatura ambiente.
NOTA: Conservar temporariamente o tecido a 4 °C até estar pronto para ser montado, se necessário. Não ultrapassamos 2 dias antes da aquisição de imagens. - Faça 5 gotículas de solução de montagem em uma lâmina de vidro. Corte a ponta de uma pipeta de 200 μL para torná-la mais larga e transfira o tecido para uma nova lâmina de vidro.
- Pegue as amostras de tecido por sua base com pinças e mergulhe e enxágue suavemente em uma série de gotículas de solução de montagem. Tenha cuidado para não quebrar os tecidos nesta etapa.
- Monte com solução de montagem, coloque a tampa e sele com esmalte. Imagem in situ de amostras de tecido usando microscópio confocal.
Resultados
Detecção robusta de genes quimiossensoriais em antena de anofelinos
Investigamos a sensibilidade do método HCR FISH (Figura 1) para detectar a expressão de receptores quimiossensoriais em tecidos olfativos de mosquitos. Guiados pelos dados de transcrição de RNA relatados anteriormente na antena fêmea do mosquito Anopheles, geramos sondas para atingir uma variedade de IRs. Os valores médios de transcrição de quatro estudos independentes de transcriptoma antenal revelaram que Ir41t.1 (11 RPKM), Ir75d (12 RPKM) e Ir7t (13 RPKM) foram menos abundantes na antena em comparação com os co-receptores Ir25a (197 RPKM) e Ir76b (193 RPKM)5,11,12,13,14. Geramos sondas para atingir Ir41t.1, Ir75d e Ir7t. IR64a (31 RPKM) também foi alvo dado que seu nível de transcrição é aproximadamente 3x mais abundante do que o menor valor de transcrição entre os genes candidatos de interesse. Deve-se notar aqui que os valores de RPKM de estudos transcriptômicos representam uma medida em massa de transcritos de toda a antena. Como tal, pode ser que apenas alguns neurônios expressem altamente um transcrito do gene IR, ou pode ser que um gene IR seja pouco expresso em muitas células. Os valores de RPKM podem, portanto, não necessariamente corresponder ao nível de abundância de um IR dentro de um neurônio. Além disso, a amplificação do sinal proporcionada pelo método HCR também pode dificultar o uso desse método para medir com precisão os níveis de transcrição neuronal com base em sinais fluorescentes. Nossa recente publicação discutiu esses conceitos mais detalhadamente5. Os dados sugerem que o HCR WM-FISH é altamente sensível à detecção de transcritos de RNAm de tecidos antenais, como mostrado na Figura 2.
Co-marcação multiplexada de diferentes alvos de RNA em apêndices quimiossensoriais
Para examinar a co-localização de alvos de RNA, geramos sondas conjugadas a diferentes fluoróforos. Hibridização dupla in situ direcionada a transcritos do gene co-receptor do receptor odorante (Orco) e do gene co-receptor do receptor ionotrópico mais amplamente expresso (Ir25a) revelou a colocalização dessas distintas famílias de receptores quimiossensoriais em um subconjunto de populações celulares na antena (Figura 3A) e palpo maxilar (Figura 3B). Também investigamos a colocalização de transcritos de três genes co-receptores de RI (Ir8a, Ir25a e Ir76b). Os padrões de colocalização sugerem que as células Ir76b positivas expressam Ir25a, enquanto as células Ir8a positivas co-localizam parcialmente com Ir76b e Ir25a (Figura 4). A análise da co-expressão dos correceptores de RI demonstra a robustez do uso do HCR WM-FISH para estudos multiplexados.
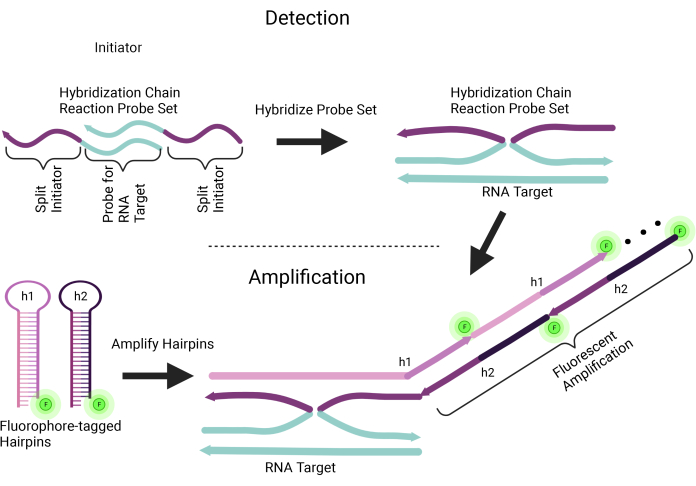
Figura 1: Esquema da reação em cadeia de hibridização in situ . Este método funciona em duas etapas: detecção e amplificação. Uma sonda definida para reação em cadeia de hibridização in situ compreende um iniciador dividido e uma sequência de ácidos nucleicos específica para o alvo de RNA. Múltiplos pares de conjuntos de sondas podem ser projetados para hibridizar várias regiões no alvo do RNA; Isso define a etapa de detecção. A etapa de amplificação requer que os pinos de cabelo marcados com fluoróforos (h1 e h2) se liguem especificamente ao iniciador conjugado a um conjunto de sondas. Após a ligação, o sinal de RNA marcado pelo fluoróforo é amplificado pela ligação repetida de pinos marcados com fluoróforo. Clique aqui para ver uma versão maior desta figura.

Figura 2: Localização do RNA dos genes quimiossensoriais de IR. HCR WM-FISH de antenas fêmeas de Anopheles coluzzii para atingir IRs. As sondas de RNA incluem (A) IR41t.1, (B) IR75d, (C) IR7t e (D) IR64a. As inserções em caixas tracejadas brancas são imagens ampliadas do tecido. Clique aqui para ver uma versão maior desta figura.

Figura 3: Dupla co-localização in situ de genes quimiossensoriais na antena e palpo maxilar. Imagens confocais Z-stack de células expressando Ir25a (verde) e Orco (magenta) na antena (A) e (B) palpo maxilar de fêmeas de mosquitos Anopheles coluzzii . As inserções em caixas tracejadas brancas são imagens ampliadas do tecido. A escala é de 10 μm. A Figura 3B foi modificada de15. Clique aqui para ver uma versão maior desta figura.

Figura 4: Mapeamento da colocalização espacial de múltiplos alvos de RNA. Imagens in situ mostrando padrões de colocalização de três co-receptores IR, IR25a (magenta), IR8a (verde) e IR76b (azul) na antena do mosquito. As setas brancas apontam para as células que expressam os três correceptores de RI (IR25a, IR8a e IR76b). A escala é de 20 μm. Este valor foi modificado de5. Clique aqui para ver uma versão maior desta figura.
Tabela 1: Reagentes para reação em cadeia de hibridização in situ . Reagentes necessários para realizar a hibridização em cadeia de hibridização fluorescente in situ . Clique aqui para baixar esta tabela.
Discussão
A terceira geração da reação em cadeia de hibridização (HCR) é notável por sua sensibilidade e robustez para visualizar vários alvos deRNA8. O HCR WM-FISH tem sido utilizado com sucesso em embriões de Drosophila, galinhas, camundongos e zebrafish, bem como em larvas de nematoides e zebrafish 10,16,17. Antenas de mosquito e palpos maxilares são tipicamente propensos a alta autofluorescência e fraca penetração de sonda, que são particularmente desafiadores ao conduzir métodos tradicionais de montagem completa in situ. Essas desvantagens foram diminuídas pela etapa de amplificação do sinal integrada ao protocolo HCR, que melhora a relação sinal-fundo e permite a detecção de transcritos de RNAm quimiorreceptores olfatórios (Figura 2). O design do protocolo HCR garante que as sondas de iniciação estejam presas aos polímeros de amplificação, o que permite a obtenção de imagens de alvos de RNA com baixo sinal. Na configuração multiplexada, diferentes amplificadores ortogonais de HCR foram amarrados a fluoróforos espectralmente únicos. Essa abordagem foi fundamental para evitar o sangramento espectral de um canal de imagem vizinho.
Para otimizar o método de RCE para uso com apêndices de mosquitos, fizemos várias observações. Os amplificadores HCR são sensíveis à luz e devem ser sempre armazenados em uma caixa em um freezer de -20°C. Em nossa experiência, a incubação extensa dos tecidos em tampão CCD, proteinase K ou curto tempo de fixação em paraformaldeído poderia resultar na quebra de antenas ou palpos durante as etapas de lavagem. Antenas e palpos maxilares também devem ser sempre agarrados pela base para evitar quebra. Na fase inicial de adaptação deste protocolo, perdemos tecidos consistentemente durante a série de trocas de tampão (passos 5 e 6). Para minimizar tais perdas, o número de tecidos foi duplicado no início do experimento, as etapas de lavagem e trocas de tampão foram realizadas ao microscópio, e pontas de carregamento de gel não maiores que 0,5 mm foram usadas para remover os tampões dos tecidos.
O HCR WM-FISH tem sido bem sucedido em apêndices olfativos periféricos de mosquitos vetores Anopheles gambiae 5,15 e Aedes aegypti 4,18, e o protocolo pode ainda ser otimizado e adaptado a outras partes do tecido do inseto ou diferentes animais. O protocolo original projetado pelo fabricante não incorpora incubação em tampão CCD. Esta etapa foi integrada ao protocolo de digestão e permeabilização da cutícula quitinosa. Estendemos o tempo de incubação nos conjuntos de sonda por duas noites para dar mais tempo para a penetração da sonda, uma modificação do protocolo do fabricante que recomenda um tempo de incubação de 16 h que funciona bem para amostras genéricas em uma lâmina. A otimização deste protocolo para outras partes do tecido do inseto exigiria a variação do tempo de incubação no tampão CCD; Partes de tecidos periféricos com cutículas espessas provavelmente exigirão tempo prolongado de incubação. A concentração de proteinase K também deve ser ajustada experimentalmente durante a adaptação deste protocolo para diferentes tecidos ou estágios de desenvolvimento dos animais. Quando se trabalha com tecidos com espessura superior a 5 mm, a penetração da sonda torna-se um desafio. Nesta situação, esforços adicionais são necessários para realizar a criossecção tecidual ou alterar ainda mais o protocolo descrito neste estudo.
O HCR WM-FISH limita-se a visualizar simultaneamente apenas alguns alvos gênicos ao mesmo tempo em comparação com a transcriptômica espacial, o que poderia potencialmente permitir a obtenção simultânea de imagens simultâneas de milhares de genes19. A síntese comercial de sondas de RNA é cara; A alternativa seria produzir as sondas e amplificadores em laboratório, mas isso pode ser desafiador para a maioria dos grupos e é trabalhoso e demorado. Esse protocolo também é limitado pelas dificuldades encontradas pela penetração da sonda de RNA através de amostras espessas de montagem total, o que poderia exigir alterações no protocolo (por exemplo, preparação do tecido passo 2) ou secção do tecido intacto. Se o método HCR WM-FISH descrito aqui não conseguir detectar um transcrito de RNA em um apêndice olfatório, o gene pode ser expresso em apenas algumas células e exigir um extenso levantamento do tecido, possivelmente não é transcrito ou pode ser transcrito em um nível abaixo do limite de detecção desse método. RT-PCR quantitativo ou RNA seq poderiam ser realizados para validar tais achados.
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
Agradecemos a Margo Herre e ao laboratório Leslie Vosshall por compartilharem seu protocolo de hibridização in situ para apêndices olfativos do Aedes aegypti . Este trabalho foi apoiado por subsídios dos Institutos Nacionais de Saúde para C.J.P. (NIAID R01Al137078), uma bolsa HHMI Hanna Gray para J.I.R, um Johns Hopkins Postdoctoral Accelerator Award para J.I.R, e uma bolsa de pós-doutorado do Johns Hopkins Malaria Research Institute para J.I.R. Agradecemos ao Johns Hopkins Malaria Research Institute e à Bloomberg Philanthropies pelo apoio.
Materiais
| Name | Company | Catalog Number | Comments |
| Amplification buffer | Molecular Instruments | Molecular Instruments, Inc. | In Situ Hybridization + Immunofluorescence | 50 mL |
| Calcium Chloride (CaCl2) 1M | Sigma-Aldrich | 21115-100ML | |
| Chitinase | Sigma-Aldrich | C6137-50UN | |
| Chymotrypsin | Sigma-Aldrich | CHY5S-10VL | |
| Dimethyl sulfoxide (DMSO) | Sigma-Aldrich | 472301 | |
| Eppendorf tube | VWR | 20901-551 | 1.5 mL |
| Forceps | Dumont | 11251 | Number 5 |
| Gel loading tip | Costar | 4853 | 1-200 µL tip |
| Hairpins | Molecular Instruments | Molecular Instruments, Inc. | In Situ Hybridization + Immunofluorescence | h1 and h2 initiator splits |
| HEPES (1M) | Sigma-Aldrich | H0887 | |
| IR25a probe | Molecular Instruments | Probe Set ID: PRK149 | AGAP010272 |
| IR41t.1 probe | Molecular Instruments | Probe Set ID: PRK978 | AGAP004432 |
| IR64a probe | Molecular Instruments | Probe Set ID: PRK700 | AGAP004923 |
| IR75d probe | Molecular Instruments | Probe Set ID: PRK976 | AGAP004969 |
| IR76b probe | Molecular Instruments | Probe Set ID: PRI998 | AGAP011968 |
| IR7t probe | Molecular Instruments | Probe Set ID: PRL355 | AGAP002763 |
| IR8a probe | Molecular Instruments | Probe Set ID: PRK150 | AGAP010411 |
| LoBind Tubes | VWR | 80077-236 | 0.5 mL DNA/RNA LoBind Tubes |
| Magnessium Chloride (MgCl2) 1M | Thermo Fisher | AM9530G | |
| Methanol | Fisher | A412-500 | |
| Nuclease-free water | Thermo Fisher | 43-879-36 | |
| Nutator | Denville Scientific | Model 135 | 3-D Mini rocker |
| Orco probe | Molecular Instruments | Probe set ID PRD954 | AGAP002560 |
| Paraformaldehyde (20% ) | Electron Microscopy Services | 15713-S | |
| Phosphate Buffered Saline (10X PBS) | Thermo Fisher | AM9625 | |
| Probe hybridization buffer | Molecular Instruments | https://www.molecularinstruments.com/ | 50 mL |
| Probe wash buffer | Molecular Instruments | Molecular Instruments, Inc. | In Situ Hybridization + Immunofluorescence | 100 mL |
| Proteinase-K | Thermo Fisher | AM2548 | |
| Saline-Sodium Citrate (SSC) 20x | Thermo Fisher | 15-557-044 | |
| SlowFade Diamond | Thermo Fisher | S36972 | mounting solution |
| Sodium Chloride (NaCl) 5M | Invitrogen | AM9760G | |
| Triton X-100 (10%) | Sigma-Aldrich | 93443 | |
| Tween-20 (10% ) | Teknova | T0027 | |
| Watch glass | Carolina | 742300 | 1 5/8" square; transparent |
Referências
- Konopka, J. K., et al. Olfaction in Anopheles mosquitoes. Chem Senses. 46, (2021).
- Raji, J. I., Potter, C. J. Chemosensory ionotropic receptors in human host-seeking mosquitoes. Curr Opin Insect Sci. 54, 100967 (2022).
- Young, A. P., Jackson, D. J., Wyeth, R. C. A technical review and guide to RNA fluorescence in situ hybridization. PeerJ. 8, e8806 (2020).
- Herre, M., et al. Non-canonical odor coding in the mosquito. Cell. 185 (17), 3104-3123.e28 (2022).
- Raji, J. I., Konopka, J. K., Potter, C. J. A spatial map of antennal-expressed ionotropic receptors in the malaria mosquito. Cell Rep. 42 (2), 112101 (2023).
- Choi, H. M. T., et al. Programmable in situ amplification for multiplexed imaging of mRNA expression. Nat Biotechnol. 28 (11), 1208-1212 (2010).
- Choi, H. M. T., Beck, V. A., Pierce, N. A. Next-generation in situ hybridization chain reaction: higher gain, lower cost, greater durability. ACS Nano. 8 (5), 4284-4294 (2014).
- Choi, H. M. T., et al. Third-generation in situ hybridization chain reaction: multiplexed, quantitative, sensitive, versatile, robust. Development. 145 (12), dev165753 (2018).
- Schwarzkopf, M., et al. Hybridization chain reaction enables a unified approach to multiplexed, quantitative, high-resolution immunohistochemistry and in situ hybridization. Development. 148 (22), dev199847 (2021).
- Choi, H. M. T., et al. Mapping a multiplexed zoo of mRNA expression. Development. 143 (19), 3632-3637 (2016).
- Pitts, R. J., Derryberry, S. L., Zhang, Z., Zwiebel, L. J. Variant ionotropic receptors in the malaria vector mosquito Anopheles gambiae tuned to amines and carboxylic acids. Sci Rep. 7, 40297 (2017).
- Rinker, D. C., Zhou, X., Pitts, R. J., Rokas, A., Zwiebel, L. J. Antennal transcriptome profiles of anopheline mosquitoes reveal human host olfactory specialization in Anopheles gambiae. BMC Genomics. 14, 749 (2013).
- Maguire, S. E., Afify, A., Goff, L. A., Potter, C. J. Odorant-receptor-mediated regulation of chemosensory gene expression in the malaria mosquito Anopheles gambiae. Cell Rep. 38 (10), 110494 (2022).
- Athrey, G., et al. Chemosensory gene expression in olfactory organs of the anthropophilic Anopheles coluzzii and zoophilic Anopheles quadriannulatus. BMC Genomics. 18 (1), 751 (2017).
- Task, D., et al. Chemoreceptor co-expression in Drosophila melanogaster olfactory neurons. eLife. 11, e72599 (2022).
- Shah, S., et al. Single-molecule RNA detection at depth by hybridization chain reaction and tissue hydrogel embedding and clearing. Development. 143 (15), 2862-2867 (2016).
- Trivedi, V., Choi, H. M. T., Fraser, S. E., Pierce, N. A. Multidimensional quantitative analysis of mRNA expression within intact vertebrate embryos. Development. 145 (1), dev156869 (2018).
- Herre, M., Greppi, C. RNA in situ hybridization and immunohistochemistry to visualize gene expression in peripheral chemosensory tissues of mosquitoes. Cold Spring Harb Protoc. 2023 (1), 48-54 (2023).
- Marx, V. Method of the Year: spatially resolved transcriptomics. Nat Methods. 18 (1), 9-14 (2021).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados