Method Article
Mapeando interações do genoma 3D de mamíferos com Micro-C-XL
Neste Artigo
Resumo
Um protocolo para mapeamento da organização tridimensional do genoma com resolução de nucleossomos usando o método de captura de conformação cromossômica Micro-C-XL é apresentado aqui.
Resumo
A organização cromossômica tridimensional (3D) é um fator importante na regulação do genoma e especificação do tipo celular. Por exemplo, acredita-se que elementos cis-regulatórios, conhecidos como potenciadores, regulem a atividade de promotores distais via interação no espaço 3D. Tecnologias de captura de conformação cromossômica (3C) em todo o genoma, como Hi-C, transformaram nossa compreensão de como os genomas são organizados nas células. A compreensão atual da organização do genoma 3D é limitada pela resolução com a qual a organização topológica dos cromossomos no espaço 3D pode ser resolvida. Micro-C-XL mede o enovelamento cromossômico com resolução no nível do nucleossomo, a unidade básica da cromatina, utilizando nuclease microcócica (MNase) para fragmentar genomas durante o protocolo de captura da conformação cromossômica. Isso resulta em uma melhor relação sinal-ruído nas medições, facilitando assim a melhor detecção de locais de isolamento e loops cromossômicos em comparação com outras tecnologias 3D de todo o genoma. Um protocolo passo-a-passo detalhado e visualmente suportado para preparar amostras Micro-C-XL de alta qualidade a partir de células de mamíferos é apresentado neste artigo.
Introdução
Micro-C-XL é uma técnica genômica para medir a conformação do genoma 3D com resolução de nucleossomos. O Micro-C-XL baseia-se na tecnologia Hi-C baseada em ligadura de proximidade amplamente utilizada, que transformou nossa compreensão de como os genomas 3D são organizados1. O Micro-C-XL e sua primeira iteração, Micro-C, foram inicialmente desenvolvidos em Saccharomyces cerevisiae2,3 e posteriormente adaptados a sistemas celulares de mamíferos, para os quais o protocolo demonstrou todo o seu potencial na detecção de características de curto alcance do genoma 3D, como alças cromossômicas e sítios de isolamento. Esta versão é baseada em publicações recentes de mamíferos Micro-C-XL 4,5. Como o Micro-C-XL substitui o Micro-C, o Micro-C-XL é doravante referido como Micro-C no manuscrito.
As principais diferenças entre Micro-C e Hi-C6 são as seguintes: 1) fragmentação do genoma com nuclease microcócica (MNase) em comparação com enzimas de restrição e 2) reticulantes adicionais com maior espaçamento atômico entre os grupos reativos em comparação com apenas formaldeído. Ambas as etapas contribuem significativamente para a melhoria da relação sinal-ruído do Micro-C em comparação com o Hi-C convencional. O tamanho da fragmentação limita a resolução para a qual a organização do genoma 3D pode ser resolvida durante o protocolo de ligadura de proximidade. A MNase é uma nuclease que digere preferencialmente o DNA acessível e deixa intacto o DNA nucleossomal protegido. O footprinting nucleossomal usando o sequenciamento de MNase mostrou que os nucleossomos cobrem totalmente a maioria dos genomas eucarióticos7. Como os nucleossomos estão distribuídos ao longo do genoma com um espaçamento médio de 160-220 pb, dependendo da espécie e do tipo celular, a MNase é a enzima ideal para o mapeamento de alta resolução da arquitetura do genoma.
O uso de um reticulador adicional em combinação com formaldeído (FA) no método Micro-C melhora adicionalmente a relação sinal-ruído 2,8. Reticulantes específicos de amina com espaçadores atômicos mais longos entre os grupos reativos facilitam ligações cruzadas proteína-proteína. Estes são tipicamente disuccinimidil glutarato (DSG) ou etilenoglicol bis-succinimidil succinato (EGS) com espaçadores de 7,7 Å e 16,1 Å, respectivamente. A redução do ruído através do EGS ou DSG é particularmente aparente em experimentos com altas taxas de fragmentação, como o Micro-C, e presumivelmente ocorre devido a uma redução na taxa de eventos de ligaduraaleatória8.
Um protocolo Hi-C 3.0 recentemente desenvolvido que utiliza ligações cruzadas ESG/DSG e múltiplas combinações de enzimas de restrição reduz o ruído em experimentos Hi-C e melhora significativamente a detecção de alças cromossômicas e locais de isolamento 8,9. Ainda assim, uma comparação site a site de vários recursos de dados de interação descobriu que o Micro-C tinha detecção superior de recursos de curto alcance, como loops cromossômicos e locais de isolamento, em comparação com o Hi-C 3.0 e o Hi-C8 convencional. No entanto, o Hi-C 3.0 melhora a detecção de características de curto alcance e mantém uma forte detecção de compartimentalização do genoma em comparação com o Hi-C convencional. Em resumo, a escolha de um método de captura da conformação cromossômica deve ser determinada pela questão objetiva e biológica.
Aqui, fornecemos um protocolo passo-a-passo para experimentos Micro-C bem-sucedidos que podem desvendar a organização do genoma 3D.
Protocolo
1. Cultura celular e reticulação
- Cultivar células de acordo com as necessidades experimentais para obter um mínimo de 1 x 107 células. Aqui, as células foram cultivadas a 37 °C com 5% de CO2 em meio E14 (DMEM com piruvato e L-glutamina, 15% FBS, 1x LIF, 1x NEAA, 1% pen-strep, 0,1 mM β-mercaptoetanol [ver Tabela de Materiais]) e passadas a cada dois dias.
NOTA: Este protocolo foi aplicado com sucesso a vários tipos celulares de várias espécies, tais como Homo sapiens e Mus musculus. Neste exemplo, foram usadas células-tronco embrionárias (ES) de camundongos. - Colher as células na confluência de 70%-80% aspirando o meio. Lavar uma vez com 5 mL de DPBS e incubar as células com 3 mL de tripsina 0,25% pré-aquecida por prato de 10 cm por 2-3 min a 37 °C.
- Temperar a tripsina com 7 mL de meio E14 pré-aquecido e transferir as células destacadas para um tubo de 50 mL.
- Pellet as células por centrifugação a 300 x g por 5 min à temperatura ambiente (TR). Descarte o sobrenadante e ressuspenda o pellet de células no meio. Conte as células com um contador de células.
NOTA O protocolo é robusto a alguma variação nas concentrações celulares, e vários contadores de células têm sido usados para quantificar os números de células; no entanto, esteja ciente do intervalo dinâmico do contador de células usado. Isso é tipicamente entre 1 x 105-1 x 107 células/mL. Embora a velocidade e a duração da centrifugação tenham sido testadas para este tipo de célula, células menores e diferentes podem exigir uma velocidade de centrifugação maior ou tempos de rotação mais longos, e a centrifugação deve ser ajustada de acordo. - Coletar as células por centrifugação a 300 x g por 5 min na RT e ressuspender as células em DPBS em uma concentração final de 1 x 106 células/mL. Por exemplo, se o rendimento for de 1 x 107 células, ressuspenda as células em 10 mL de DPBS.
- Para a primeira etapa de reticulação, adicionar formaldeído (AG) a 37% para uma concentração final de 1% à suspensão celular e incubar a suspensão celular por 10 min em TR com rotação (15-20 rpm); como exemplo, se o rendimento for de 1 x 107 células, adicione 270 μL de AG à suspensão celular de 10 mL.
NOTA: A solução FA é normalmente estável em RT por até 3 meses após a abertura. - Atenuar a reação adicionando glicina 2,5 M a uma concentração final de 0,25 M e incubar por 5 min em TR com rotação (15-20 rpm). Por exemplo, se o rendimento for de 1 x 107 células, adicione 1,027 mL de glicina 2,5 M à suspensão celular.
- Coletar as células por centrifugação a 300 x g por 5 min na RT. Descarte o sobrenadante e ressuspenda as células em 5 mL de DPBS. Repetir a etapa de centrifugação uma vez e ressuspender as células peletizadas em DPBS para 4 x 106 células/mL. Por exemplo, se o rendimento for de 1 x 107 células, ressuspenda as células em 2,5 mL de DPBS.
- Para a segunda etapa de reticulação, preparar uma solução-estoque 0,3 M de bis-succinimidil succinato de etilenoglicol (EGS) em DMSO (13,6 mg de EGS em 100 μL de DMSO). Adicionar EGS a uma concentração final de 3 mM à suspensão celular e incubar em TR por 40 min com rotação (15-20 rpm). Por exemplo, se o rendimento for de 1 x 107 células, adicione 25 μL da solução-mãe EGS 0,3 M a 2,5 mL da suspensão celular.
NOTA: Equilibrar o EGS para RT durante pelo menos 20 minutos antes da pesagem para preparar a solução-mãe. - Atenuar a reação adicionando glicina 2,5 M a uma concentração final de 0,4 M e incubar por 5 min em TR com rotação (15-20 rpm). Por exemplo, se o rendimento for de 1 x 107 células, adicione 400 μL de glicina 2,5 M à suspensão celular.
- Coletar as células por centrifugação a 1.000 x g por 5 min no TR e ressuspender as células peletizadas em DPBS para 5 x 106 células/mL. Distribuir 5 x 10 6 células/tubo para bibliotecas preparativas e 1 x 106 células/tubo para titulação para digestão de MNase.
NOTA: Sugere-se titular o grau de digestão ideal para cada lote de células reticuladas. Idealmente, coletar de duas a três alíquotas de 1 x 10 6 células para experimentos de titulação de MNase (etapa 2) e duas a quatro alíquotas de 5 x 106 células para experimentos preparativos (etapa 3). - Coletar as células por centrifugação a 1.000 x g por 5 min no TR e remover o sobrenadante. Congele rapidamente os pellets de células em nitrogênio líquido e armazene-os a -80 °C.
NOTA: Bibliotecas Micro-C bem-sucedidas podem ser preparadas a partir de amostras armazenadas por até 3 meses.
2. Titulação da MNase
NOTA: A realização de uma titulação de MNase é necessária para determinar a concentração ideal de MNase antes de processar a biblioteca preparativa das células duplamente reticuladas.
- Para realizar a titulação da MNase, descongelar uma pastilha de 1 x 106 células no gelo por 10 min e ressuspender as células em 500 μL de DPBS (adicionar 1x BSA se as células aderirem à parede). Incubar a suspensão celular no gelo por 20 min.
- Coletar as células por centrifugação a 10.000 x g por 5 min no TR e remover o sobrenadante. Ressuspender as células peletizadas em 500 μL de tampão MB#1 (50 mM NaCl, 10 mM Tris-HCl, 5 mM MgCl 2, 1 mM CaCl 2,0,2% NP-40, 1x inibidor de protease [ver Tabela de Materiais], pH 7,4).
- Recolher as células por centrifugação a 10.000 x g durante 5 minutos e remover o sobrenadante. Ressuspender as células em 200 μL de tampão MB#1 e dividir a amostra em quatro tubos.
- Descongelar um frasco de MNase (20 U/μL) e diluir com Tris 10 mM, pH 7,4, em séries de 1:2, 1:4, 1:4 e 1:4 para atingir concentrações de 10 U/μL, 2,5 U/μL, 0,625 U/μL e 0,1256 U/μL, respectivamente (um para cada condição de digestão). Com intervalos de tempo apropriados (10-20 s), adicionar 1 μL de solução de MNase a uma das quatro amostras, vórtice, e incubar em um termomisturador a 37 °C por 10 min (agitação de 800 rpm). Continue adicionando 1 μL das diluições restantes de MNase às alíquotas celulares restantes.
- Interromper a digestão do Mnase adicionando 200 μL de tampão STOP recém-preparado (150 μL de Tris 10 mM, pH 7,4, 25 μL de SDS a 10 %, 25 μL de 20 mg/mL de proteinase K, 2 μL de 0,5 M EGTA) a cada tubo na mesma ordem e com o mesmo intervalo de tempo que a MNase foi adicionada. Incubar a 65 °C durante 2 h.
- Adicionar 500 μL de álcool fenol-clorofórmio-isoamílico (PCI) a cada amostra e misturar cuidadosamente por vórtice. Centrifugar a 19.800 x g por 5 min em TR para separar as fases e transferir a fase aquosa para novos tubos (aproximadamente 200 μL/amostra).
CUIDADO: O PCI contém inúmeros componentes tóxicos e só deve ser manuseado em uma capa de segurança química. Consulte o fabricante para obter mais detalhes. - Purificar o ADN utilizando um kit comercial de purificação do ADN (ver Tabela de Materiais) de acordo com as instruções do fabricante e eluir as amostras em 12 μL de tampão de eluição .
NOTA: A concentração de SDS da etapa de desproteinização (etapa 2.5) é inibitória para alguns kits de purificação de DNA. O kit de purificação de DNA usado aqui tem um desempenho comparável à precipitação de etanol. - Adicionar 2-5 μL de corante de carga e passar as amostras em gel de agarose a 1,5% a 120 V por 30-50 min (até separar adequadamente; Figura 1A).
- Escolha o melhor grau de digestão para o experimento e continue com a digestão preparativa de MNase. Um grau de digestão ideal exibe pouco ou nenhum fragmento subnucleossomal e uma proporção de 70%-90% mono para dinucleossomo.
NOTA: Neste experimento, o grau de digestão da pista 4 na Figura 1A foi determinado como sendo a digestão ótima obtida com 0,625-1,25 U de Mnase para 2,5 x 10 5 células (= 2,5-5 U de Mnase para 1 x 106 células). Para uma discussão detalhada, consulte os resultados representativos.
3. Digestão preparativa da MNase
- Para fragmentar a cromatina aos mononucleossomos via digestão com MNase, descongelar alíquotas de células 5 x 106 previamente duplamente reticuladas e ressuspender em 1 mL de DPBS. Incubar no gelo durante 20 minutos (adicionar 1x BSA se as células aderirem à parede do tubo).
- Coletar as células por centrifugação a 10.000 x g por 5 min no TR e descartar o sobrenadante. Ressuspender o pellet em 500 μL de tampão MB#1. Repita a etapa de centrifugação uma vez. Ressuspender o pellet em 1 mL de tampão MB#1 e fazer alíquotas de 200 μL (1 x 106 células por alíquota).
- Com base na titulação da MNase (descrita na etapa 2), digerir a cromatina adicionando a quantidade apropriada de MNase (geralmente 2,5-10 U/μL por 1 x 106 células) a cada alíquota. Misture bem (vórtice e gire rapidamente) e incube em um termomisturador a 37 °C por 10 min com agitação de 800 rpm.
- Interromper a digestão da MNase adicionando 1,6 μL de EGTA 0,5 M (4 mM final) a cada alíquota e incubar num termomisturador a 65 °C durante 10 minutos com agitação a 800 rpm.
- Coletar a amostra por centrifugação a 10.000 x g por 5 min no TR e descartar o sobrenadante. Ressuspender o pellet celular em 500 μL de 1x NEBuffer 2.1.
- Agrupar amostras equivalentes a uma entrada de 5 x 106 células ou menos para processamento posterior.
NOTA: Se mais de 5 x 10 6 células forem processadas, processe essas amostras em paralelo, pois as condições enzimáticas são otimizadas para 5 x 106 células. - Antes de prosseguir para as etapas de ligadura de proximidade, transfira 10% da amostra como controle de entrada para controlar o nível de digestão da MNase. Adicionar 150 μL de Tris 10 mM, pH 7,4, 25 μL de SDS a 10% e 25 μL de 20 mg/mL de proteinase K a esta amostra e incubar durante a noite a 65 °C.
4. Processamento final de DNA e ligadura de proximidade
- Recolher a amostra restante por centrifugação a 10.000 x g durante 5 min a 4 °C e eliminar o sobrenadante. Ressuspender o pellet em 90 μL da mistura mestre Micro-C 1 recém-preparada (Tabela 1) e incubar em um termomisturador por 15 min a 37 °C com agitação de 800 rpm.
- Adicionar 10 μL de 5 U/μL de Fragmento de Klenow e incubar num termomisturador durante 15 minutos a 37 °C com agitação a 800 rpm.
- Adicionar 100 μL da mistura principal Micro-C 2 recém-preparada (Tabela 2) e incubar em um termomisturador por 45 min a 25 °C com agitação de 800 rpm. Após a incubação, extinguir a reação enzimática adicionando EDTA a uma concentração final de 30 mM. Incubar num misturador de termólogos durante 20 minutos a 65 °C com agitação a 800 rpm.
- Recolher a amostra por centrifugação a 10.000 x g durante 5 min a 4 °C e eliminar o sobrenadante. Ressuspender a amostra em 500 μL da mistura mestre Micro-C 3 recém-preparada (Tabela 3) e incubar por 2,5 h em TR com rotação (15-20 rpm).
- Recolher a amostra por centrifugação a 10.000 x g durante 5 min a 4 °C e eliminar o sobrenadante. Ressuspender a amostra em 200 μL da mistura mestra Micro-C 4 recém-preparada (Tabela 4) e incubar em um termomisturador por 15 min a 37 °C com agitação de 800 rpm.
- Para reticulação reversa e desproteinação, adicionar 25 μL de 20 mg/mL de proteinase K e 25 μL de SDS a 10% à amostra e incubar a 65 °C durante a noite com mistura intermitente.
5. Purificação do DNA dinucleossomal e seleção de tamanho
- Adicionar 500 μL de PCI às amostras e ao controle de entrada, e misturar por vórtice. Separe as fases por centrifugação a 19.800 x g por 5 min e transfira a fase aquosa superior para um tubo fresco.
- Concentrar o DNA usando um kit de purificação de DNA ou por precipitação de etanol. Eluir as amostras em 30 μL (passo 5.3) e os controlos de entrada (passo 3.11) em 15 μL e executar um gel de agarose a 1,5 % para separar os mononucleossomas e os dinucleossomas (Figura 1B).
NOTA: A concentração de SDS da etapa de desproteinização é inibitória para alguns kits de purificação de DNA. O kit de purificação de DNA usado aqui (veja Tabela de Materiais) tem um desempenho comparável à precipitação de etanol. Dependendo do número de células utilizadas, a entrada pode variar de 100 ng a 10 μg. Normalmente, 1-5 μg de DNA é extraído de 5 x 106 células. - Excise os fragmentos de DNA que têm um tamanho di-nucleossomal (aproximadamente 300 pb). Use um kit de eluição de gel de DNA disponível comercialmente (consulte Tabela de Materiais) para extrair o DNA do gel de agarose e eluir em 150 μL.
6. Preparação das esferas de estreptavidina
- Transferir 10 μL de esferas de estreptavidina (ver Tabela de Materiais) por amostra para um tubo de reação.
- Coloque-o em um ímã apropriado para tubos de 1,5 mL (consulte a Tabela de Materiais). Após a liberação da solução (1-2 min), remova o sobrenadante e ressuspenda as esferas em 300 μL de 1x TBW (5 mM Tris-HCl, pH 7,5, 0,5 mM EDTA, 1 M NaCl, 0,05% Tween 20) por amostra. Repita esta etapa uma vez.
- Ressuspender as esferas em 150 μL de tampão 2x P&B (10 mM Tris-HCl, pH 7,5, 1 mM EDTA, 2 M NaCl) por amostra processada na etapa 7.
7. Preparação da biblioteca pull-down e on-bead de estreptavidina
- Adicionar 150 μL de contas pré-preparadas (passo 6.3) a 150 μL da amostra (passo 5.3). Incubar por 20 min em TR com rotação (15-20 rpm).
- Coloque os tubos em um ímã apropriado e aguarde até que a solução se limpe (1-2 min). Retire o sobrenadante e ressuspenda as esferas em 300 μL de 1x TBW. Repita esta etapa.
- Coloque os tubos em um ímã apropriado e aguarde até que a solução se limpe (1-2 min). Retirar o sobrenadante e ressuspender as esferas em 100 μL de 0,1x TE (1 mM Tris, 0,1 mM EDTA, pH 8,0).
- Coloque os tubos em um ímã apropriado e aguarde até que a solução se limpe (1-2 min). Retirar o sobrenadante, ressuspender as esferas em 50 μL de 0,1x TE e transferi-las para tubos de PCR.
NOTA: O volume de 0,1x TE utilizado (50 μL) corresponde ao volume de entrada do kit de preparação da biblioteca de sequenciamento de ADN (ver Tabela de Materiais) utilizado neste protocolo. Se um kit ou estratégia diferente for usado, ajuste o volume de acordo. - Executar as etapas de manipulação de DNA do kit de preparação da biblioteca de sequenciamento de acordo com o protocolo do fabricante. Essas etapas geralmente incluem embotamento do DNA, cauda A, ligadura do adaptador e excisão em U. A última etapa (excisão em U) é específica para o kit utilizado neste estudo. Se utilizar esse kit, siga as instruções do fabricante do passo 1 ao passo 2.6 do protocolo do kit utilizando adaptadores não diluídos e não considere o armazenamento de -20 °C após o passo 2.6.
NOTA: As alterações de tampão exigidas pelos kits de sequenciamento devem ser trocadas pela ligação das contas a um ímã e lavagens (passos 7.3 a 7.4), uma vez que o ADN ainda está ligado às esferas de estreptavidina. Além disso, como o DNA está ligado às esferas magnéticas, ignore todas as etapas de purificação e seleção de tamanho após a ligadura do adaptador do protocolo do kit. Continue para a etapa 7.6 (este protocolo). - Após a conclusão da ligadura do adaptador, lave a amostra conforme descrito na etapa 7.2. Descarte o sobrenadante e ressuspenda as esferas em 20 μL de 0,1x TE.
8. Estimativa dos ciclos de PCR necessários
NOTA: Estimar os ciclos de PCR necessários para amplificação de bibliotecas é aconselhável. Normalmente, uma biblioteca Micro-C requer 8-15 ciclos de PCR. Embora a etapa não seja essencial, ajuda a evitar a amplificação excessiva e reduz o risco de duplicações de PCR.
- Para definir o número mínimo de ciclos de PCR necessários, realizar a PCR com 1 μL de amostra de estreptavidina-biotina-DNA (passo 7.6). Para fazer isso, adicione a mistura mestre de PCR (3,2 μL de H2O, 0,4 μL de primer i5, 0,4 μL de primer i7 e 5 μL de DNA polimerase de alta fidelidade Q5) à amostra. Realizar PCR com 16 ciclos de acordo com as instruções do fabricante para o kit de preparação da biblioteca de DNA utilizado.
NOTA: Os primers usados aqui são comprados em um kit separado (consulte Tabela de Materiais) que precisa ser compatível com o kit de preparação da biblioteca usado. - Após a PCR, colete as esferas com um ímã apropriado e meça a concentração de DNA de 1 μL do sobrenadante usando um instrumento de quantificação de DNA de alta sensibilidade (ver Tabela de Materiais). Para obter a concentração total de DNA, multiplique esse valor por 10 (volume total da reação de PCR). Estimar o número necessário de ciclos de PCR conforme discutido nos resultados representativos.
- Adicionar 2 μL de tampão de carga 6x aos 9 μL restantes da mistura de PCR. Resolver a biblioteca amplificada da PCR em gel de agarose a 1% para determinar o sucesso da ligadura do adaptador, resultando em aproximadamente 450 pb de tamanho (Figura 1C).
NOTA: O gel deve mostrar uma banda distinta de aproximadamente 420 pb (biblioteca Micro-C mais adaptadores). Bandas de 120 pb representam dímeros adaptadores e são indicativas de bibliotecas de baixa complexidade. Bandas de menor peso molecular podem aparecer (menos de 100 pb), e estes são primers não utilizados da reação de PCR.
9. Amplificação da biblioteca de sequenciamento
- Adicionar a mistura mestre de PCR (65 μL de H2O, 100 μL de DNA polimerase de alta fidelidade Q5, 8 μL de primer i5, 8 μL de primer i7) aos 19 μL restantes da amostra de estreptavidina-biotina-DNA. Divida a mistura de reação em alíquotas de 50-100 μL.
NOTA: O volume ideal para o PCR depende da máquina de PCR; tipicamente, 50 μL oferece a amplificação mais reprodutível em máquinas comuns de PCR. - Realizar a PCR de acordo com as instruções do fabricante do kit de preparação da biblioteca de DNA com o número de ciclos determinado na etapa 8. Se o passo 8 foi omitido, recomenda-se 14 ciclos para PCR.
- Purificar o ADN com esferas paramagnéticas (ver Tabela de Materiais) na proporção de 1:0,9 de acordo com o protocolo do fabricante. Eluir em 20 μL de TA 0,1%.
Observação : se os dímeros do adaptador forem detectados na etapa 8.5, execute a purificação duas vezes usando a mesma proporção. - Determine a concentração de DNA e execute as amostras em um sistema de controle de qualidade (consulte Tabela de Materiais). Garantir a boa qualidade da biblioteca Micro-C pela presença de uma única banda precisa (Figura 1D).
NOTA: Os dímeros adaptadores são incomuns em preparações de biblioteca de contas devido às lavagens da amostra antes da PCR. Assim, o aparecimento de dímeros adaptadores indica baixa complexidade da biblioteca. Se dímeros adaptadores forem observados, é altamente recomendável controlar a qualidade da amostra com baixo sequenciamento de entrada.
10. Sequenciamento de DNA e processamento de dados
- Sequencie a biblioteca Micro-C com sequenciamento de extremidade emparelhada de acordo com os requisitos do provedor de sequenciamento.
NOTA: Idealmente, as amostras são sequenciadas em uma plataforma no modo de extremidade emparelhada com 50 pb por leitura. Plataformas mais antigas que oferecem comprimentos de leitura mais curtos, como 2 x 35 pb, também foram usadas com sucesso. É importante ressaltar que, se regiões genômicas repetitivas forem estudadas, pode ser aconselhável sequenciar com uma extensão de leitura mais longa. - Para avaliar a qualidade da biblioteca Micro-C, execute um sequenciamento de entrada baixo com 5 x 106 a 1 x 107 leituras por amostra.
- Processe os arquivos de sequenciamento (arquivos fastq) com o Distiller10. Mapeie as leituras contra o genoma de referência apropriado, aqui mm10.
Observação : os arquivos de sequenciamento podem ser processados usando vários pipelines em computadores locais ou clusters de computação. Para amostras com baixas profundidades de sequenciamento, tamanhos de compartimento maiores, como 10.000 pb, 50.000 pb, 100.000 pb e 500.000 pb, podem reduzir as demandas de computação e os tamanhos de arquivos. O Distiller (utilizado neste estudo) gera todos os tipos de arquivos necessários para avaliar a qualidade da biblioteca Micro-C. O arquivo *.stats gerado contém as informações sobre a taxa do mapa, razões cis-trans e orientação de leitura estratificada pela distância entre os pares de leitura. Esses parâmetros são visualizados na Figura 2, e a avaliação da qualidade da biblioteca Micro-C é discutida nos resultados representativos. O software de processamento também gera arquivos mcool que podem ser carregados diretamente no HiGlass (https://docs.higlass.io/) para gerar matrizes de interação9.
Resultados
A preparação bem-sucedida de bibliotecas Micro-C pode ser avaliada em várias etapas do protocolo. O passo mais importante é a escolha de um grau de digestão MNase adequado. Portanto, a concentração de MNase deve ser titulada para produzir consistentemente 70%-90% de mononucleossomos sobre dinucleossomos para cada amostra. É importante notar que a digestão da cromatina é diferente para eu- e heterocromatina, com a MNase digerindo a heterocromatina de forma menos eficiente. Assim, o grau ótimo de digestão depende da região de interesse da cromatina e do tipo celular estudado, uma vez que a proporção relativa de eu- e heterocromatina é específica do tipo celular. Portanto, é aconselhável titular cuidadosamente a concentração de MNase necessária e primeiro avaliar o sucesso do experimento Micro-C por meio de sequenciamento de baixa entrada.
Um padrão típico de titulação de MNase da cromatina tratada com quantidades decrescentes de MNase é mostrado na Figura 1A. Aqui, a cromatina de 250.000 células por reação é digerida com uma diluição de quatro vezes a MNase. A maior concentração (10 U de MNase, Lane 2) mostra cromatina superdigerida quase exclusivamente consistindo de DNA mononucleossomal (~150 pb). Notavelmente, o centro da banda mononucleossomal é menor no gel de agarose em comparação com as bandas correspondentes nas amostras com concentrações reduzidas de MNase, indicando uma superdigestão do DNA nucleossomal. Nucleossomos superdigeridos são ligados ineficientemente na reação de ligadura de proximidade; portanto, a amostra na faixa 2 é subótima para experimentos Micro-C. A faixa 3 (2,5 U de MNase) exibe um grau de digestão quase apropriado para experimentos de Micro-C. Aqui, a banda mononucleossomal é a espécie dominante, e o esfregaço subnucleossomal, indicativo de nucleossomos superdigeridos, é reduzido; no entanto, ainda está presente. O grau de digestão na Lane 4 (0,635 U de MNase) é uma condição ideal para um experimento Micro-C neste exemplo de titulação. Uma banda mononucleossomal clara sem DNA subnucleossomal está presente. A intensidade da banda para o DNA mononucleossomo e dinucleossomo é quase igual, indicando um rendimento de mononucleossomo de 66% ou mais. Vale a pena notar que o DNA dinucleossomal é aproximadamente duas vezes o tamanho do DNA mononucleossomal (~320 pb vs. ~150 pb), de modo que sua intensidade de banda por mol de DNA é duas vezes maior em comparação com sua contraparte mononucleossomal. O grau de digestão em Lane 5 (0,156 U de MNase) mostra cromatina subdigerida com quase nenhum DNA nucleossomal, e esta, portanto, representa uma amostra subótima.
Em conclusão, neste exemplo, a digestão de 2,5 x 10 5 células ES de camundongo com 0,625 U de MNase (correspondendo a2,5 U de MNase para 1 x 106 células em 200 μL) oferece o ponto de partida mais promissor para digestãos preparativas em experimentos de Micro-C. No entanto, uma concentração intermediária de MNase entre as condições utilizadas para as amostras na faixa 3 e na faixa 4 (correspondendo a 5 U de MNase para 1 x 106 células em 200 μL) também deve ser considerada. É importante ressaltar que a digestão da cromatina com MNase não pode ser dimensionada linearmente, e não é recomendado aumentar a digestão preparativa mais de 4x. Para preparar bibliotecas Micro-C de mais de 1 x 10 6 células, é aconselhável digerir a cromatina em alíquotas de 1 x 106 células e agrupá-las após a inativação da MNase.
Para avaliar o sucesso do protocolo de ligadura de proximidade, o controle de entrada, que é digerido por MNase e não ligado por proximidade (passo 3.8), deve ser comparado com a amostra ligada à proximidade (passo 5.3) por eletroforese em gel de agarose a 1,5% (Figura 1B). A banda mononucleossomal ligada à proximidade tem um tamanho aproximado de 300 pb, semelhante ao dos dinucleossomos. Portanto, a razão de sinal da banda mono para di- nucleossomal deve mudar de predominantemente mononucleossomos (Lane 1) para di-nucleossomos (Lane 3 e Lane 4). Como o gel de agarose nesta etapa é o DNA dinucleossomal que é excisado e purificado, dividir as amostras em várias faixas é aconselhável para evitar a sobrecarga de carga.
Recomenda-se avaliar a qualidade e a quantidade da biblioteca de sequenciamento preparada por PCR mínimo. Aqui, o DNA de 1 μL de contas (1/20 da amostra total) é amplificado por 16 ciclos em 10 μL de reação de PCR. A concentração total da biblioteca mínima de PCR normalmente varia de 50-500 ng após 16 ciclos de PCR. Em teoria, isso corresponde a uma biblioteca de 1-10 μg da amostra restante de 19 μL se ela também fosse amplificada por 16 ciclos. Recomenda-se usar o número mínimo de ciclos de PCR necessários para gerar uma biblioteca de aproximadamente 100 ng a partir do DNA total. Assumindo amplificação logarítmica na PCR, a concentração teórica do DNA obtido a partir da entrada de 19 μL em 16 ciclos pode ser dividida sucessivamente por dois para calcular o número de ciclos de PCR necessários para gerar uma biblioteca de 100 ng. Por exemplo, um rendimento de 100 ng de 1 μL após 16 ciclos corresponde a um rendimento de 1.900 ng amplificado a partir de 19 μL. Nesse cenário, 12 ciclos devem idealmente gerar uma biblioteca de sequenciamento de 118 ng a partir do DNA total (1.900 ng/[2 × 2 × 2 × 2] = 118 ng). A amostra restante de 9 μL da PCR mínima pode então ser usada para avaliar a qualidade da biblioteca por eletroforese em gel de agarose (Figura 1C). A visualização deve mostrar uma banda distinta a 420 pb e nenhuma banda para dímeros adaptadores (120 pb). Fragmentos menores também podem aparecer, e estes correspondem a iniciadores de PCR não utilizados.
Em seguida, recomenda-se analisar e confirmar a preparação bem-sucedida de amostras Micro-C por meio de sequenciamento de baixa entrada antes de se comprometer com o sequenciamento profundo com uso intensivo de recursos. Normalmente, as bibliotecas são sequenciadas a uma profundidade de leitura de 5 x 106 a 1 x 107 e avaliadas com base nos seguintes critérios: a taxa de duplicação de leitura de sequenciamento, a taxa de interação cis- versus transcromossômica e a frequência de orientação de leitura de sequenciamento. As bibliotecas Micro-C são processadas com o Distiller, um pipeline de serviço completo que processa os dados de sequenciamento de arquivos de leitura (formato Fastq) para arquivos de pares de leitura (formato Bedpe) e matrizes de interação escaláveis (formatos Cool e Mcool) usando cooler, pairtools e cooltools10,11,12. O pipeline também gera um arquivo de resumo que é ideal para avaliar a qualidade das bibliotecas Micro-C10 (https://github.com/open2c/distiller-nf). A taxa de duplicação de PCR fornece informações sobre a complexidade da biblioteca de sequenciamento e pode ser extraída do arquivo *.stats gerado. Bibliotecas Micro-C de alta qualidade têm taxas de duplicação de PCR inferiores a 5%-10% quando geradas a partir de 5 milhões ou mais células. Notavelmente, algumas plataformas de sequenciamento geram duplicatas de PCR durante a formação do cluster, independentemente da complexidade da biblioteca de sequenciamento. A Figura 2A mostra as taxas de duplicação relativa de dois experimentos: um que consideramos uma amostra boa e outra amostra ruim. Neste exemplo, ambos os exemplos exibiram taxas de mapa aceitáveis. Os próximos critérios para avaliar a qualidade das bibliotecas Micro-C são a razão cis versus trans e as frequências de orientação de leitura. Dentro do núcleo, os cromossomos habitam territórios cromossômicos individuais e, portanto, raramente interagem com outros cromossomos. Uma alta taxa de interações transcromossômicas detectadas indica uma alta taxa de ligações aleatórias. Ressalta-se que, nesse nível de análise, a amostra ruim apresentou alta taxa de interações transcromossômicas em relação à amostra boa (Figura 2B). Para Micro-C, uma taxa de interação cis-cromossômica de 70% ou mais é desejável.
Uma biblioteca Micro-C tem um tamanho de fragmento semelhante à banda de DNA dinucleossomal, que pode co-purificar com a amostra ligada de proximidade e contaminar o experimento. Esses contaminantes são sempre interações cromossômicas cis. Portanto, é importante avaliar também as taxas de orientação de leitura. A taxa de contaminação dinucleossômica pode ser estimada por sequenciamento de baixa entrada. O DNA dinucleossomal provém de dois nucleossomos vizinhos que não foram clivados pela MNase. Assim, as leituras de sequenciamento resultantes sempre exibirão uma orientação de leitura para frente e para trás (F e R), e a distância entre os pares de leitura será de cerca de 320 pb. Fragmentos ligados por proximidade, em comparação, podem ser ligados em quatro orientações, produzindo pares de leitura com F-R, R-R, R-F e F-F, idealmente com igual abundância (Figura 2C). Além disso, eles exibem várias distâncias entre os dois pares de leitura. Para estimar a quantidade de contaminantes dinucleossomais, a frequência das orientações de leitura pode ser calculada a partir dos arquivos *stats gerados pelo destilador (Figura 2D). Notadamente, neste trabalho, a fração de leitura de F-R (vermelho) foi maior na amostra ruim em relação à amostra boa, e isso se tornou mais evidente quando as orientações de leitura foram estratificadas por distância (Figura 2E). A fração F-R é dominada por fragmentos dinucleossomais em comparação com bibliotecas Micro-C quando os pares de leitura são estratificados em leituras com distâncias <562 pb ou ≥562 pb. Aqui, a fração de leituras com distância <562 pb é dominada por leituras F-R, enquanto a fração com distâncias ≥562 pb exibe uma distribuição uniforme entre as quatro orientações possíveis, indicando que a super-representação global de leituras F-R deriva de contaminantes dinucleossomais. A escolha de 562 bp como limite para subconfiguração é definida pelo binning no arquivo *stats gerado. Embora não seja necessário para esse controle de qualidade, uma subconfiguração mais definida pode ser obtida extraindo as distâncias do arquivo *pairs, que também é gerado pelo destilador. É importante notar que as leituras dinucleossômicas não diminuem a qualidade da amostra de Micro-C, pois podem ser identificadas e ignoradas durante o processamento de dados. No entanto, eles não contêm informações valiosas sobre interações 3D e diluem as leituras informativas.
Assim, a titulação cuidadosa da MNase e o controle de qualidade minucioso com sequenciamento de baixa entrada são as melhores ferramentas para otimizar a qualidade dos experimentos Micro-C.
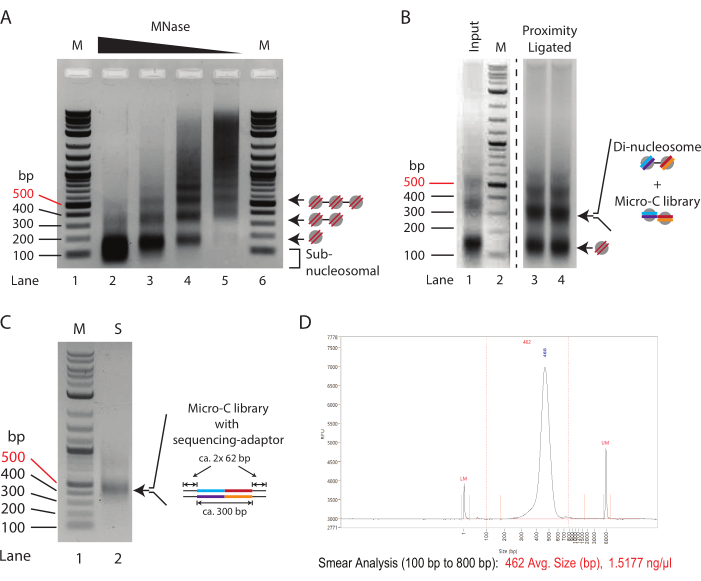
Figura 1: Estágios intermediários do protocolo Micro-C . (A) Eletroforese em gel de agarose da cromatina de 2,5 x 105 células ES de camundongo digeridas com concentrações variáveis de MNase. As bandas mono, di e trinucleossômicas são indicadas por setas. M: Escada de DNA (Faixa 1/6); 10 U de MNase por 250.000 células (Faixa 2); 2,5 U de MNase por 250.000 células (Faixa 3); 0,625 U de MNase por 250.000 células (Faixa 4); 0,156 U de MNase por 250.000 células (Faixa 2). (B) A eletroforese em gel de agarose a 1,0% das amostras preparadas com Micro-C (Faixa 3 e Faixa 4) e o controle de entrada digerido pela MNase (Faixa 1). A faixa 1 e a faixa 2 (M: escada de DNA) são aprimoradas para enfatizar a mudança relativa na intensidade do fragmento mono a dinucleossomal. As bandas mono e dinucleossomal são indicadas por setas. A banda dinucleossomal na amostra ligada à proximidade combina DNA dinucleossomal e biblioteca Micro-C. (C) A eletroforese em gel de agarose a 1,0% das bibliotecas de sequenciamento Micro-C amplificada a partir de amostra de 1 μL para avaliar a qualidade. Faixa 1 (M): Escada de DNA; Faixa 2 (S): Biblioteca Mirco-C. (D) Rastreio do Analisador de Fragmentos da biblioteca Micro-C final. Clique aqui para ver uma versão maior desta figura.

Figura 2: Estatísticas amostrais para o sequenciamento de baixa entrada de uma amostra boa e uma amostra ruim . (A) Gráfico de barras da porcentagem mapeada (verde) e não mapeada (vermelho) lida. (B) Fração normalizada de leituras mapeando interações cis e transcromossômicas. Os conjuntos de dados foram normalizados para a leitura do mapeamento cis. As leituras do mapeamento cis foram estratificadas pela distância entre a primeira e a segunda leituras das amostras sequenciadas pareadas: ≤1 kbp (amarelo), >1 kbp e ≤10 kbp (laranja) e >10 kbp (vermelho). (C) Esquema das espécies moleculares potenciais com os tamanhos dinucleossomais. (D) Porcentagens de orientações de pares de leitura de todas as leituras da amostra boa e da amostra ruim. (E) O mesmo que o painel (D), mas estratificado por distâncias (esquerda, <562 pb e direita, ≥562 pb). Clique aqui para ver uma versão maior desta figura.
| Componentes | 1x | 4,4x |
| 10x NEBuffer 2.1, | 10 μL | 44 μL |
| 2 μL 100 mM ATP | 2 μL | 8,8 μL |
| TDT de 100 mM | 5 μL | 22 μL |
| H2O | 68 μL | 299,2 μL |
| 10 U/μL T4 PNK | 5 μL | 22 μL |
| Total | 90 μL | 396 μL |
Tabela 1: Mistura mestre Micro-C 1. Composição da mistura mestre para a reação mastigatória final.
| Componentes | 1x | 4,4x |
| 1 mM Biotina-dATP | 10 μL | 44 μL |
| 1 mM Biotina-dCTP | 10 μL | 44 μL |
| Mistura de 10 mM de dTTP e dGTP | 1 μL | 4,4 μL |
| 10x T4 DNA Ligase Tampão | 5 μL | 22 μL |
| 200x BSA | 0,25 μL | 1,1 μL |
| H2O | 23,75 μL | 104,5 μL |
Tabela 2: Micro-C master mix 2. Composição da mistura mestre para a reação de marcação final.
| Componentes | 1x | 4,4x |
| 10x tampão de reação NEB T4 Ligase | 50 μL | 220 μL |
| H2O | 422,5 μL | 1859 μL |
| T4 DNA Ligase | 25 μL | 110μL |
Tabela 3: Mistura mestre Micro-C 3. Composição da mistura mestre para a reação de ligadura de proximidade.
| Componentes | 1x | 4,4x |
| 10x NEBuffer 1,1 | 20 μL | 88 μL |
| H2O | 180 μL | 792 μL |
| Nuclease ExoIII | 10 μL | 44 μL |
Tabela 4: Mistura mestre Micro-C 4. Composição da mistura mestre para a reação de remoção de biotina.
Discussão
O sucesso de um experimento Micro-C depende de algumas etapas críticas no protocolo que precisam ser cuidadosamente executadas. Primeiro, a ligação cruzada com o reticulador adicional DSG ou EGS pode levar à agregação de células, dependendo do tipo de célula. A adição de 0,1%-0,5% de BSA à reação de reticulação reduz significativamente a agregação sem afetar a eficiência da reticulação. Ligações cruzadas ineficientes podem resultar em aumento das taxas de interações transcromossômicas indicativas de ligaduras aleatórias. O segundo, mas mais crucial, passo neste protocolo é a digestão da cromatina com MNase. A digestão subótima da cromatina leva à ligadura de proximidade ineficiente (excesso de digestão) ou ao aumento das taxas de dinucleossomos não ligados à proximidade (subdigestão). A eficiência da reação de ligadura pode ser avaliada por eletroforese em gel de agarose (Figura 1B) e é adicionalmente melhor estimada por sequenciamento de baixa entrada. Se o sequenciamento de baixa entrada revelar uma alta taxa de duplicação (ligadura ineficiente) ou taxas aumentadas de dinucleossomos, o grau de digestão da MNase deve ser reavaliado. Notavelmente, a perda de amostra ao executar o protocolo pode levar à redução da complexidade da biblioteca. A concentração de uma amostra é melhor avaliada após a purificação do DNA (etapa 5.3) ou por PCR mínimo (etapa 8). O rendimento total de DNA de 5 x 106 células de mamíferos após a purificação do DNA é tipicamente de >2 μg. A concentração de DNA deve ser controlada após a digestão da MNase, digestão ExoIII e purificação do DNA. Nucleases endógenas, cuja abundância é específica do tipo celular e espécie-específica, podem ser uma fonte de degradação do DNA. Além disso, a purificação de DNA baseada em coluna pode levar à perda de amostra devido à incompatibilidade com o SDS de reações de desproteinação. A precipitação de etanol pode ser considerada se a concentração de DNA for baixa nesta etapa.
Como o Micro-C requer titulação de MNase específica da amostra, é um desafio aplicar o Micro-C a populações de pequenas células, como com pequenos órgãos de vários organismos modelo, embriões e células únicas, organoides ou biópsias de pacientes. Aqui, o Hi-C 3.0 oferece uma alternativa bem estabelecida usando uma reação de desfecho por endonucleases de restrição seqüenciais-específicas 8,9.
O Micro-C é uma tecnologia de conformação cromossômica de alta resolução amplamente aplicável, com alta faixa dinâmica e baixa relação sinal-ruído, o que a torna particularmente adequada para investigar características cromossômicas de curto alcance 4,5,8, como alças cromossômicas. A resolução do Micro-C permite a captura de alças promotora-potencializadora, que estão além do limite de detecção de Hi-C, possibilitando eficientemente uma análise mais detalhada da relação entre organização e regulação do genoma13,14,15. Além disso, estratégias de captura de DNA foram recentemente combinadas com Micro-C para aumentar a resolução locus-específica dos locus genômicos alvo para níveis sem precedentes, revelando novos insights sobre a ultraestrutura do genoma 3D16,17,18. Em resumo, vislumbramos que o Micro-C e seus derivados serão uma tecnologia chave para dissecar o papel do genoma 3D na regulação transcricional e, consequentemente, na diferenciação e manutenção do tipo celular.
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
Agradecemos a Christl Gaubitz e Kathleen Stewart-Morgan pela leitura crítica do manuscrito. Agradecemos a Anja Groth e ao laboratório Groth por seu apoio na criação de nosso laboratório. Agradecemos à equipe da Plataforma de Genômica CPR/reNEW pelo apoio: H. Wollmann, M. Michaut e A. Kalvisa. O Centro de Medicina de Células-Tronco da Fundação Novo Nordisk (reNEW) é apoiado pelo número de concessão da Fundação Novo Nordisk NNF21CC0073729. O Centro de Pesquisa de Proteínas (CPR) da Fundação Novo Nordisk é apoiado pelo número NNF14CC0001 de concessão da Fundação Novo Nordisk. Agradecemos ao laboratório Brickman do Novo Nordisk Center for Stem Cell Medicine, reNEW Copenhagen, pelas células ES de camundongos.
Materiais
| Name | Company | Catalog Number | Comments |
| 1 mM Biotin dATP | Jenna Bioscience | NU-835-Bio14-S | |
| 1 mM Biotin dCTP | Jenna Bioscience | NU-809-BioX-S | |
| 10 mM dGTP | NEB | N0442S | |
| 10 mM dTTP | NEB | N0443S | |
| 10 U/ml T4 PNK | NEB | M0201L | |
| 100 U/L Exonuclease III | NEB | M0206L | |
| 10x NEBuffer 1.1 | NEB | B7001S | |
| 10x NEBuffer 2.1 | NEB | B7202S | |
| 10x T4 DNA Ligase buffer | NEB | B0202A | |
| 1x DPBS w/o Mg2+ and Ca2+ | ThermoFisher | 14190144 | |
| 1x LIF | |||
| 2_Mercaptoethanol 50 mM | Gibco | 31350010 | 0.1 mM b-mercaptoethanol |
| 37% Formaldehyde | Sigma Aldrich | 252549-500ML | Caution. See manufactures MSDS |
| 400 U/ml T4 DNA Ligase | NEB | M0202L | |
| 5 U/ml Klenow Fragment | NEB | M0210L | |
| Agarose | BIO-RAD | 1613102 | Caution. See manufactures MSDS |
| BSA 20mg/ml | NEB | B9000S | |
| CaCl2 | |||
| cell counter | |||
| Dimethyl Sulfoxide (DMSO) | Sigma Aldrich | D8418-100ML | Caution. See manufactures MSDS |
| Dynabeads MyOne Streptavidin C1 | Invitrogen | 65001 | |
| DynaMag-2 Magnet | Invitrogen | 12321D | refered to as: magnet magnet for 1.5 ml tubes |
| DynaMag-PCR Magnet | Invitrogen | 492025 | refered to as: magnet magnet for PCR tubes |
| EDTA Ultrapure 0.5M pH 8.0 | Invitrogen | 15575-038 | |
| EGTA Ultrapure 0.5M pH 8.0 | BioWorld | 40121266-1 | |
| Ethanol 96% | VWR Chemicals | 20824365 | quality control system |
| Ethidium Bromide | Invitrogen | 15585-011 | |
| Ethylene glycol bis(succinimidyl succinate) (EGS) | ThermoFisher | 21565 | |
| Fetl Bovin Serum | Sigma Aldrich | F7524 | 15% FBS |
| Gel Loading dye purple (6X) | NEB | B7024S | |
| Glycine | PanReac AppliChem | A1067.0500 | |
| Halt Proteinase inhibitor (100x) | ThermoFisher | 78430 | Caution. See manufactures MSDS |
| IGEPAL CA-630 (NP-40) | Sigma Aldrich | 18896-50ML | |
| MgCl 1 M | Invitrogen | AM9530G | |
| Micrococcal Nuclease (MNase) | Worthington | LS004798 | |
| mouse embryonic stem cells | |||
| NaCl | Sigma Aldrich | S9888-1KG | |
| NEBNext Multiplex Oligos for Illumina (Dual Index primers) | NEB | E7600S | amplification primers for sequencing libraries |
| NEBNext Ultra II DNA library prep kit for Illumina | NEB | E7645L | sequencing library preparation kit |
| NEBNext Ultra II Q5 Master mix | NEB | M0544S | Caution. See manufactures MSDS |
| Non-Essential Amino Acids Solution | Gibco | 11140050 | 1x NEAA |
| Penicillin-Streptomycin (10,000 U/mL) | Gibco | 15140148 | 1% Pen-Strep |
| Proteinase K (40 mg/ml) | GoldBio | P-480-1 | Caution. See manufactures MSDS |
| QIAquick Gel extraction kit | QIAgen | 28706 | refered to as: DNA gel elution kit |
| QIAquick PCR purification kit | QIAgen | 28106 | refered to as: commercial DNA purification kit |
| Qubit dsDNA HS Assay kit | Invitrogen | Q32854 | high sensitivity DNA quantification instrument |
| Quick load purple 1kb plus DNA Ladder | NEB | N0550S | |
| SPRIselect size selection beads | Beckman Coulter | B23319 | paramagnetic beads |
| ThermoMixer C | Eppendorf | 5382000015 | refered to as: thermomixer |
| Tris | Merck | 10708976001 | |
| Trypsin | |||
| Tween20 | Sigma Aldrich | P7949-100ML | |
| Ultrapure 10% SDS | Invitrogen | 15553-035 | |
| Ultrapure Phenol Chloroform Isoamyl Alcohol (PCI) | Invitrogen | 15593-031 | |
| Fragment Analyzer |
Referências
- Lieberman-Aiden, E., et al. Comprehensive mapping of long-range interactions reveals folding principles of the human genome. Science. 326 (5950), 289-293 (2009).
- Hsieh, T. -H. S., Fudenberg, G., Goloborodko, A., Rando, O. J. Micro-C XL: Assaying chromosome conformation from the nucleosome to the entire genome. Nature Methods. 13 (12), 1009-1011 (2016).
- Hsieh, T. -H. S., et al. Mapping nucleosome resolution chromosome folding in yeast by Micro-C. Cell. 162 (1), 108-119 (2015).
- Krietenstein, N., et al. Ultrastructural details of mammalian chromosome architecture. Molecular Cell. 78 (3), 554-565 (2020).
- Hsieh, T. -H. S., et al. Resolving the 3D landscape of transcription-linked mammalian chromatin folding. Molecular Cell. 78 (3), 539-553 (2020).
- Belaghzal, H., Dekker, J., Gibcus, J. H. Hi-C 2.0: An optimized Hi-C procedure for high-resolution genome-wide mapping of chromosome conformation. Methods. 123, 56-65 (2017).
- van Holde, K. E. Chromatin. , Springer. New York, NY. (1989).
- Oksuz, B. A. Systematic evaluation of chromosome conformation capture assays. Nature Methods. 18, 1046-1055 (2021).
- Lafontaine, D. L., Yang, L., Dekker, J., Gibcus, J. H. Hi-C 3.0: Improved protocol for genome-wide chromosome conformation capture. Current Protocols. 1 (7), 198(2021).
- Goloborodko, A., Venev, S., Abdennur, N., Di Tommaso, P. Mirnylab/distiller-nf; v033. Zenodo. , (2019).
- Venev, S., et al. Open2c/cooltools: v0.4.1. Zenodo. , (2021).
- Abdennur, N., Mirny, L. A. Cooler: Scalable storage for Hi-C data and other genomically labeled arrays. Bioinformatics. 36 (1), 311-316 (2019).
- Zhang, S., Übelmesser, N., Barbieri, M., Papantonis, A. Enhancer-promoter contact formation requires RNAPII and antagonizes loop extrusion. BioRxiv. , (2022).
- Barshad, G., et al. RNA polymerase II and PARP1 shape enhancer-promoter contacts. BioRxiv. , (2022).
- Hansen, A. S., et al. Distinct classes of chromatin loops revealed by deletion of an RNA-binding region in CTCF. Molecular Cell. 76 (3), 395-411 (2019).
- Hua, P., et al. Defining genome architecture at base-pair resolution. Nature. 595 (7865), 125-129 (2021).
- Downes, D. J. High-resolution targeted 3C interrogation of cis-regulatory element organization at genome-wide scale. Nature Communications. 12, 531(2021).
- Goel, V. Y., Huseyin, M. K., Hansen, A. S. Region capture Micro-C reveals coalescence of enhancers and promoters into nested microcompartments. BioRxiv. , (2022).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados