Method Article
Micro-C-XL을 사용한 포유류 3D 게놈 상호 작용 매핑
요약
게놈 전체 염색체 형태 캡처 방법인 Micro-C-XL을 사용하여 뉴클레오솜 분해능으로 3차원 게놈 조직을 매핑하기 위한 프로토콜이 여기에 제시되어 있습니다.
초록
3차원(3D) 염색체 조직은 게놈 조절 및 세포 유형 사양의 주요 요소입니다. 예를 들어, 인핸서(enhancer)로 알려진 시스 조절 요소는 3D 공간에서의 상호작용을 통해 원위 프로모터의 활성을 조절하는 것으로 생각됩니다. Hi-C와 같은 게놈 전체 염색체 형태 캡처(3C) 기술은 세포에서 게놈이 어떻게 구성되는지에 대한 우리의 이해를 변화시켰습니다. 3D 게놈 조직에 대한 현재의 이해는 3D 공간에서 염색체의 위상 조직을 분해할 수 있는 해상도에 의해 제한됩니다. Micro-C-XL은 염색체 형태 캡처 프로토콜 중에 미세구균 뉴클레아제(MNase)를 활용하여 게놈을 단편화함으로써 염색질의 기본 단위인 뉴클레오솜 수준에서 분해능으로 염색체 접힘을 측정합니다. 그 결과 측정에서 신호 대 잡음비가 향상되어 다른 게놈 전체 3D 기술에 비해 절연 부위 및 염색체 루프를 더 잘 감지할 수 있습니다. 이 기사에서는 포유류 세포에서 고품질 Micro-C-XL 샘플을 준비하기 위한 시각적으로 지원되는 상세한 단계별 프로토콜을 제공합니다.
서문
Micro-C-XL은 뉴클레오솜 분해능으로 3D 게놈 형태를 측정하는 게놈 전체 기술입니다. Micro-C-XL은 널리 사용되는 근접 결찰 기반 Hi-C 기술을 기반으로 하며, 이는 3D 게놈이 구성되는 방식에 대한 우리의 이해를 변화시켰습니다1. Micro-C-XL과 그 첫 번째 반복인 Micro-C는 처음에 맥주효모균 2,3에서 개발되었으며 이후 포유류 세포 시스템에 적용되었으며, 이 프로토콜은 염색체 루프 및 절연 부위와 같은 3D 게놈의 단거리 특징을 검출하는 데 있어 완전한 잠재력을 입증했습니다. 이 버전은 최근 포유류 Micro-C-XL 간행물 4,5를 기반으로 합니다. Micro-C-XL이 Micro-C를 대체하기 때문에 Micro-C-XL은 이후 원고에서 Micro-C로 지칭됩니다.
Micro-C와 Hi-C6 의 주요 차이점은 다음과 같습니다: 1) 제한 효소와 비교하여 미세구균 뉴클레아제(MNase)를 사용한 게놈 단편화 및 2) 포름알데히드에 비해 반응성 그룹 사이에 더 큰 원자 간격을 갖는 추가 교차결합제. 두 단계 모두 기존 Hi-C에 비해 Micro-C의 향상된 신호 대 잡음비에 크게 기여합니다. 단편화 크기는 근접 결찰 프로토콜 중에 3D 게놈 조직을 분해할 수 있는 분해능을 제한합니다. MNase는 접근 가능한 DNA를 우선적으로 소화하고 뉴클레오솜 보호 DNA를 그대로 유지하는 뉴클레아제입니다. MNase 염기서열분석을 이용한 뉴클레오솜 발자국 측정은 뉴클레오솜이 대부분의 진핵생물 게놈을 완전히 덮고 있음을 보여주었다7. 뉴클레오솜은 종과 세포 유형에 따라 평균 160-220bp의 간격으로 게놈 전체에 분포되어 있기 때문에 MNase는 게놈 아키텍처의 고해상도 매핑에 이상적인 효소입니다.
Micro-C 방법에서 포름알데히드(FA)와 함께 추가 가교제를 사용하면 신호 대 잡음비 2,8이 추가로 향상됩니다. 반응성 그룹 사이에 더 긴 원자 스페이서가 있는 아민 특이적 교차결합제는 단백질-단백질 교차결합을 촉진합니다. 이들은 전형적으로 각각 7.7 Å 및 16.1 Å 스페이서를 갖는 디숙시니미딜 글루타레이트(DSG) 또는 에틸렌 글리콜 비스-숙시니미딜 숙시네이트(EGS)이다. EGS 또는 DSG를 통한 노이즈 감소는 Micro-C와 같이 단편화 비율이 높은 실험에서 특히 두드러지며, 무작위 연결 사건8의 속도 감소로 인해 발생하는 것으로 추정된다.
ESG/DSG 가교 및 제한 효소의 다중 조합을 활용하는 최근 개발된 Hi-C 3.0 프로토콜은 Hi-C 실험에서 노이즈를 줄이고 염색체 루프 및 절연 부위의 검출을 크게 향상시킵니다 8,9. 그럼에도 불구하고 다양한 상호 작용 데이터 기능을 사이트별로 비교한 결과, Micro-C는 Hi-C 3.0 및 기존 Hi-C8에 비해 염색체 루프 및 절연 부위와 같은 단거리 기능을 더 잘 감지하는 것으로 나타났습니다. 그러나 Hi-C 3.0은 기존 Hi-C에 비해 단거리 특징의 검출을 개선하고 게놈 구획화의 강력한 검출을 유지합니다. 요약하면, 염색체 형태 포획 방법의 선택은 객관적이고 생물학적인 질문에 의해 결정되어야 합니다.
여기에서는 3D 게놈 조직을 밝힐 수 있는 성공적인 Micro-C 실험을 위한 단계별 프로토콜을 제공합니다.
프로토콜
1. 세포 배양 및 가교
- 실험에 따른 배양 세포는 최소 1 x 107 세포를 얻을 필요가 있다. 여기에서, 세포는 E14 배지 (피루브산 및 L- 글루타민을 함유 한 DMEM, 15 % FBS, 1x LIF, 1x NEAA, 1 % 펜 - 스트렙, 0.1 mM β- 메르캅토 에탄올 [재료 표 참조])에서 5 % CO 2로 37 °C에서 성장시키고2 일마다 통과시켰다.
참고: 이 프로토콜은 호모 사피엔스(Homo sapien) 및 무스 무스큘러스(Mus musculus)와 같은 여러 종의 다양한 세포 유형에 성공적으로 적용되었습니다. 이 예에서는 마우스 배아 줄기(ES) 세포를 사용했습니다. - 배지를 흡인하여 70%-80% 밀도로 세포를 수확합니다. 5mL의 DPBS로 한 번 세척하고 37°C에서 2-3분 동안 10cm 접시당 3mL의 예열된 0.25% 트립신으로 세포를 배양합니다.
- 예열된 E14 배지 7mL로 트립신을 퀀칭하고 분리된 세포를 50mL 튜브로 옮깁니다.
- 실온(RT)에서 5분 동안 300 x g 에서 원심분리하여 세포를 펠렛화합니다. 상층액을 버리고 세포 펠릿을 배지에 재현탁시킵니다. 셀 카운터로 셀을 계산합니다.
참고 프로토콜은 세포 농도의 일부 변화에 강하며 세포 수를 정량화하기 위해 다양한 세포 카운터가 사용되었습니다. 그러나 사용되는 셀 카운터의 동적 범위를 알고 있어야 합니다. 이것은 일반적으로 1 x 105-1 x 107 cells/mL 사이입니다. 이 세포 유형에 대해 원심분리 속도와 지속 시간을 테스트했지만 다른 작은 세포는 더 높은 원심분리 속도 또는 더 긴 탈수 시간이 필요할 수 있으며 그에 따라 원심분리를 조정해야 합니다. - RT에서 5분 동안 300 x g 에서 원심분리하여 세포를 수집하고 1 x 106 cells/mL의 최종 농도에서 DPBS에 세포를 재현탁시킵니다. 예를 들어, 수율이 1 x 107 세포인 경우 세포를 10mL의 DPBS에 재현탁시킵니다.
- 첫 번째 가교 단계의 경우, 37 % 포름 알데히드 (FA)를 세포 현탁액에 1 %의 최종 농도로 첨가하고, 회전 (15-20 rpm)으로 RT에서 10 분 동안 세포 현탁액을 배양합니다. 예를 들어, 수율이 1 x 107 세포인 경우 10mL 세포 현탁액에 270μL의 FA를 추가합니다.
알림: FA 용액은 일반적으로 개봉 후 최대 3개월 동안 RT에서 안정적입니다. - 최종 농도 0.25M에 2.5M 글리신을 첨가하여 반응을 소멸시키고 회전(15-20rpm)으로 RT에서 5분 동안 배양합니다. 예를 들어, 수율이 1 x 107 세포인 경우 세포 현탁액에 1.027mL의 2.5M 글리신을 추가합니다.
- RT에서 5분 동안 300 x g 에서 원심분리하여 세포를 수집합니다. 상층액을 버리고 5mL의 DPBS에 세포를 재현탁시킵니다. 원심분리 단계를 한 번 반복하고 펠릿화된 세포를 DPBS에 4 x 106 cells/mL로 재현탁시킵니다. 예를 들어, 수율이 1 x 107 세포인 경우 세포를 2.5mL의 DPBS에 재현탁시킵니다.
- 두 번째 가교 단계의 경우, DMSO에서 에틸렌 글리콜 비스-숙시니미딜 숙시네이트(EGS) 0.3M 원액을 준비합니다(DMSO 100μL에 EGS 13.6mg). 최종 농도 3mM의 EGS를 세포 현탁액에 첨가하고, 회전(15-20 rpm)으로 40분 동안 실온에서 배양합니다. 예를 들어, 수율이 1 x 107 세포인 경우 25μL의 0.3M EGS 원액을 2.5mL의 세포 현탁액에 추가합니다.
알림: 원액을 준비하기 위해 계량하기 전에 최소 20분 동안 EGS를 RT로 평형화합니다. - 최종 농도 0.4M에 2.5M 글리신을 첨가하여 반응을 소멸시키고 회전(15-20rpm)으로 RT에서 5분 동안 배양합니다. 예를 들어, 수율이 1 x 107 세포인 경우 세포 현탁액에 400μL의 2.5M 글리신을 추가합니다.
- RT에서 5분 동안 1,000 x g에서 원심분리하여 세포를 수집하고, 펠릿화된 세포를 DPBS에서 5 x 106 cells/mL로 재현탁시킵니다. 분취용 라이브러리용 5 x 10 6 cells/tube와 MNase 분해를 위한 적정용 1 x 106 cells/tube를 분배합니다.
참고: 각 가교 세포 배치에 대해 최적의 분해도를 적정하는 것이 좋습니다. 이상적으로는 MNase 적정 실험(2단계)을 위해 1 x 106 세포의 2-3개 부분 표본을 수집하고, 분취 실험(3단계)을 위해 5 x 106 세포의 부분 표본 2-4개를 수집합니다. - RT에서 5분 동안 1,000 x g 에서 원심분리하여 세포를 수집하고 상층액을 제거합니다. 세포 펠릿을 액체 질소에 스냅 얼린 후 -80°C에서 보관합니다.
참고: 성공적인 Micro-C 라이브러리는 최대 3개월 동안 보관된 샘플에서 준비할 수 있습니다.
2. MNase 적정
참고: 이중 가교 세포의 분취 라이브러리를 처리하기 전에 MNase의 최적 농도를 결정하기 위해 MNase 적정을 수행해야 합니다.
- MNase 적정을 수행하려면 얼음 위에서 1 x 106 세포의 펠릿 1개를 10분 동안 해동하고 세포를 500μL의 DPBS에 재현탁시킵니다(세포가 벽에 달라붙으면 1x BSA 추가). 세포 현탁액을 얼음 위에서 20분 동안 배양합니다.
- RT에서 5분 동안 10,000 x g에서 원심분리하여 세포를 수집하고 상층액을 제거합니다. 펠릿화된 세포를 500μL의 MB#1 완충액(50mM NaCl, 10mM Tris-HCl, 5mM MgCl2, 1mMCaCl2, 0.2% NP-40, 1x 프로테아제 억제제[재료 표 참조], pH 7.4)에 재현탁시킵니다.
- 10,000 x g 에서 5분 동안 원심분리하여 세포를 수집하고 상층액을 제거합니다. 200μL의 MB#1 완충액에 세포를 재현탁시키고 샘플을 4개의 튜브로 분할합니다.
- MNase (20 U / μL)의 한 바이알을 해동하고 10 : 2, 1 : 4, 1 : 4 및 1 : 4의 연속으로 10 mM Tris, pH 7.4로 희석하여 각각 10 U / μL, 2.5 U / μL, 0.625 U / μL 및 0.1256 U / μL (각 분해 조건에 대해 하나씩). 적절한 시간 간격(10-20초)으로 1μL의 MNase 용액을 4개의 샘플 중 하나인 와류에 추가하고 37°C의 열혼합기에서 10분(800rpm 진탕) 동안 배양합니다. 나머지 MNase 희석액에서 1μL를 나머지 세포 부분 표본에 계속 추가합니다.
- 200 μL의 갓 준비된 STOP 완충액 (10 mM Tris의 150 μL, pH 7.4, 10 % SDS의 25 μL, 20 mg / mL proteinase K의 25 μL, 0.5 M EGTA의 2 μL)을 MNase가 첨가 된 동일한 순서와 동일한 시간 간격으로 각 튜브에 첨가하여 Mnase 분해를 중지하십시오. 65 °C에서 2 시간 동안 배양합니다.
- 각 샘플에 500μL의 페놀-클로로포름-이소아밀 알코올(PCI)을 추가하고 볼텍싱으로 완전히 혼합합니다. 상온에서 5분 동안 19,800 x g 에서 원심분리하여 상을 분리하고 수성상을 새 튜브(약 200μL/샘플)로 옮깁니다.
주의: PCI에는 수많은 독성 성분이 포함되어 있으므로 화학 안전 후드를 착용한 상태에서만 취급해야 합니다. 자세한 내용은 제조업체에 문의하십시오. - 제조업체의 지침에 따라 상용 DNA 정제 키트( 재료 표 참조)를 사용하여 DNA를 정제하고 12μL의 용출 완충액에서 샘플을 용리합니다.
참고: 탈단백질화 단계(2.5단계)의 SDS 농도는 일부 DNA 정제 키트에 대해 억제됩니다. 여기에 사용된 DNA 정제 키트는 에탄올 침전과 유사한 성능을 발휘합니다. - 2-5 μL의 로딩 염료를 첨가하고 120 V의 1.5 % 아가로스 겔에서 30-50 분 동안 샘플을 실행합니다 (적절하게 분리 될 때까지; 그림 1A).
- 실험에 가장 적합한 분해 정도를 선택하고 분취용 MNase 분해를 계속합니다. 최적의 분해도는 핵하 분절이 거의 또는 전혀 없고 70%-90% 단핵체 대 디뉴클레오솜 비율을 나타냅니다.
참고: 이 실험에서, 도 1A에서 레인 4의 분해도는 2.5 x 10 5 세포에 대해 0.625-1.25 U의 Mnase로 얻어진 최적의 분해로 결정되었다(= 1 x 106 세포에 대해 2.5-5 U의 Mnase). 자세한 내용은 대표 결과를 참조하십시오.
3. 분취용 MNase 분해
- MNase 분해를 통해 염색질을 단뉴클레오솜으로 단편화하려면 이전에 이중 가교된 5 x 106 세포 부분 표본을 해동하고 1mL의 DPBS에 재현탁합니다. 얼음 위에서 20분 동안 배양합니다(세포가 튜브 벽에 달라붙으면 BSA 1개 추가).
- RT에서 5분 동안 10,000 x g 에서 원심분리하여 세포를 수집하고 상층액을 버린다. 펠릿을 500μL의 MB#1 완충액에 재현탁시킵니다. 원심분리 단계를 한 번 반복합니다. 펠릿을 1mL의 MB#1 완충액에 재현탁시키고 200μL 부분 표본(부분 표본당 1 x 106 세포)을 만듭니다.
- MNase 적정(2단계에서 설명)을 기반으로 각 부분 표본에 적절한 양의 MNase(일반적으로 1 x 106 세포당 2.5-10 U/μL)를 첨가하여 크로마틴을 분해합니다. 잘 섞고(와류 및 빠르게 회전) 37°C의 열혼합기에서 800rpm 흔들면서 10분 동안 배양합니다.
- 각 부분 표본에 1.6μL의 0.5M EGTA(최종 4mM)를 추가하여 MNase 분해를 중지하고 65°C의 열혼합기에서 800rpm 진탕으로 10분 동안 배양합니다.
- RT에서 5분 동안 10,000 x g 에서 원심분리하여 샘플을 수집하고 상층액을 버립니다. 셀 펠릿을 500μL의 1x NEBuffer 2.1에 재현탁시킵니다.
- 추가 처리를 위해 5 x 106 셀 이하의 입력에 해당하는 샘플을 풀링합니다.
참고: 5 x 106 세포 이상을 처리해야 하는 경우 효소 조건이 5 x 106 세포에 최적화되어 있으므로 이러한 샘플을 병렬로 처리하십시오. - 근접 연결 단계를 진행하기 전에 샘플의 10%를 MNase 분해 수준을 제어하기 위한 입력 대조군으로 전송합니다. 이 샘플에 150 μL의 10 mM Tris, pH 7.4, 25 μL의 10 % SDS 및 25 μL의 20 mg / mL proteinase K를 첨가하고 65 ° C에서 밤새 배양합니다.
4. DNA 말단 처리 및 근접 결찰
- 남은 시료는 4°C에서 5분 동안 10,000 x g 에서 원심분리하여 수집하고, 상층액은 버린다. 펠릿을 90μL의 갓 준비한 Micro-C 마스터 믹스 1(표 1)에 재현탁시키고 800rpm 진탕으로 37°C에서 15분 동안 열혼합기에서 배양합니다.
- 10μL의 5U/μL Klenow Fragment를 추가하고 800rpm 진탕으로 37°C에서 15분 동안 열혼합기에서 배양합니다.
- 갓 준비한 Micro-C 마스터 믹스 2 100μL(표 2)를 추가하고 25°C에서 800rpm 진탕으로 45분 동안 열혼합기에서 배양합니다. 배양 후 EDTA를 최종 농도 30mM로 첨가하여 효소 반응을 소멸시킵니다. 65°C에서 800rpm으로 진탕하여 20분 동안 열혼합기에서 배양합니다.
- 4°C에서 5분 동안 10,000 x g 에서 원심분리하여 샘플을 수집하고 상층액을 버립니다. 샘플을 500μL의 갓 준비한 Micro-C 마스터 믹스 3(표 3)에 재현탁시키고 회전(15-20rpm)과 함께 RT에서 2.5시간 동안 배양합니다.
- 4°C에서 5분 동안 10,000 x g 에서 원심분리하여 샘플을 수집하고 상층액을 버립니다. 샘플을 200μL의 갓 준비한 Micro-C 마스터 믹스 4(표 4)에 재현탁시키고 37°C에서 800rpm 진탕으로 15분 동안 열혼합기에서 배양합니다.
- 역교차결합 및 탈단백질화를 위해 25μL의 20mg/mL proteinase K와 25μL의 10% SDS를 샘플에 추가하고 간헐적 혼합으로 65°C에서 밤새 배양합니다.
5. 디뉴클레오솜 DNA 정제 및 크기 선택
- 500 μL의 PCI를 샘플 및 입력 컨트롤에 추가하고 볼텍싱으로 혼합합니다. 19,800 x g 에서 5분 동안 원심분리하여 상을 분리하고 상부 수성상을 새 튜브로 옮깁니다.
- DNA 정제 키트를 사용하거나 에탄올 침전을 통해 DNA를 농축합니다. 샘플을 30μL(5.3단계)로 용리하고 입력 대조군(3.11단계)을 15μL로 용리하고 1.5% 아가로스 겔을 실행하여 단핵소체와 디뉴클레오솜을 분리합니다(그림 1B).
참고: 탈단백질화 단계의 SDS 농도는 일부 DNA 정제 키트에 대해 억제됩니다. 여기에 사용된 DNA 정제 키트( 재료 표 참조)는 에탄올 침전과 유사한 성능을 발휘합니다. 사용된 셀 수에 따라 입력 범위는 100ng에서 10μg입니다. 일반적으로 1-5μg의 DNA가 5 x 106 세포에서 추출됩니다. - 디뉴클레오솜 크기(약 300bp)를 가진 DNA 단편을 절제합니다. 시중에서 판매되는 DNA 겔 용출 키트( 재료 표 참조)를 사용하여 아가로스 겔에서 DNA를 추출하고 150μL에서 용리합니다.
6. 스트렙타비딘 비드의 제조
- 샘플당 10μL의 streptavidin 비드( 재료 표 참조)를 반응 튜브로 옮깁니다.
- 1.5mL 튜브에 적합한 자석에 넣습니다( 재료 표 참조). 용액이 맑아진 후(1-2분) 상층액을 제거하고 샘플당 300μL의 1x TBW(5mM Tris-HCl, pH 7.5, 0.5mM EDTA, 1M NaCl, 0.05% 트윈 20)에 비드를 재현탁시킵니다. 이 단계를 한 번 반복합니다.
- 7 단계에서 처리 된 샘플 당 150 μL의 2x B &W 완충액 (10 mM Tris-HCl, pH 7.5, 1 mM EDTA, 2 M NaCl)에 비드를 재현탁시킵니다.
7. Streptavidin 풀다운 및 온비드 라이브러리 준비
- 150μL의 사전 준비된 비드(단계 6.3)를 샘플 150μL(단계 5.3)에 추가합니다. 회전(15-20 rpm)과 함께 RT에서 20분 동안 배양합니다.
- 튜브를 적절한 자석에 넣고 용액이 맑아질 때까지 기다립니다(1-2분). 상층액을 제거하고 비드를 300μL의 1x TBW에 재현탁시킵니다. 이 단계를 반복합니다.
- 튜브를 적절한 자석에 놓고 용액이 맑아질 때까지 기다립니다(1-2분). 상층액을 제거하고 비드를 0.1x TE (1mM Tris, 0.1mM EDTA, pH 8.0)의 100μL에 재현탁시킵니다.
- 튜브를 적절한 자석에 놓고 용액이 맑아질 때까지 기다립니다(1-2분). 상층액을 제거하고 비드를 50μL의 0.1x TE에 재현탁시킨 다음 PCR 튜브로 옮깁니다.
참고: 사용된 0.1x TE의 부피(50μL)는 이 프로토콜에 사용된 DNA 염기서열 분석 라이브러리 준비 키트( 재료 표 참조)의 입력 부피에 해당합니다. 다른 키트나 전략을 사용하는 경우 그에 따라 볼륨을 조정하십시오. - 제조업체의 프로토콜에 따라 염기서열분석 라이브러리 준비 키트의 DNA 조작 단계를 수행합니다. 이러한 단계에는 일반적으로 DNA 둔화, A-tailing, 어댑터 결찰 및 U 절제가 포함됩니다. 마지막 단계(U 절제)는 이 연구에 사용된 키트에 따라 다릅니다. 해당 키트를 사용하는 경우 희석되지 않은 어댑터를 사용하여 키트 프로토콜의 1단계에서 2.6단계까지 제조업체의 지침을 따르고 2.6단계 이후 -20°C 보관을 고려하지 마십시오.
참고: 염기서열분석 키트에 필요한 완충액 변화는 DNA가 여전히 스트렙타비딘 비드에 결합되어 있기 때문에 비드를 자석에 결합하고 세척하여 교환해야 합니다(7.3-7.4단계). 또한 DNA가 마그네틱 비드에 결합되어 있기 때문에 키트 프로토콜에서 어댑터 결찰 후 정제 및 크기 선택 단계를 무시하십시오. 7.6단계(이 프로토콜)를 계속 진행합니다. - 어댑터 결찰이 완료된 후 7.2단계에서 설명한 대로 샘플을 세척합니다. 상층액을 버리고 비드를 20μL의 0.1x TE에 재현탁시킵니다.
8. 필요한 PCR 주기의 추정
참고: 라이브러리 증폭에 필요한 PCR 주기를 추정하는 것이 좋습니다. 일반적으로 Micro-C 라이브러리에는 8-15 주기의 PCR이 필요합니다. 이 단계가 필수는 아니지만 과증폭을 방지하고 PCR 중복의 위험을 줄이는 데 도움이 됩니다.
- 필요한 최소 PCR 주기 수를 정의하려면 1μL의 스트렙타비딘-비오틴-DNA 샘플로 PCR을 수행합니다(단계 7.6). 이를 위해 PCR 마스터 믹스(3.2μL의 H2O, 0.4μL의 i5 프라이머, 0.4μL의 i7 프라이머 및 5μL의 Q5 고충실도 DNA 중합효소)를 샘플에 추가합니다. 사용된 DNA 라이브러리 준비 키트에 대한 제조업체의 지침에 따라 16주기로 PCR을 수행합니다.
참고: 여기에 사용된 프라이머는 사용된 라이브러리 준비 키트와 호환되어야 하는 별도의 키트( 재료 표 참조)로 구입한 것입니다. - PCR 후 적절한 자석으로 비드를 채취하고 고감도 DNA 정량화 기기를 사용하여 상층액 1μL의 DNA 농도를 측정합니다( 재료 표 참조). 총 DNA 농도를 얻으려면 이 값에 10(PCR 반응의 총 부피)을 곱합니다. 대표 결과에서 논의된 바와 같이 필요한 PCR 주기 수를 추정합니다.
- 나머지 9μL의 PCR 혼합물에 2μL의 6x 로딩 버퍼를 추가합니다. 1% 아가로스 겔에서 PCR 증폭 라이브러리를 분해하여 성공적인 어댑터 결찰을 결정하면 약 450bp 크기가 됩니다(그림 1C).
알림: 젤은 약 420bp(Micro-C 라이브러리 및 어댑터)의 뚜렷한 밴드를 보여야 합니다. 120bp의 대역은 어댑터 이량체를 나타내며 복잡성이 낮은 라이브러리를 나타냅니다. 더 낮은 분자량의 밴드가 나타날 수 있으며(100bp 미만), 이는 PCR 반응에서 사용되지 않은 프라이머입니다.
9. 염기서열분석 라이브러리 증폭
- PCR 마스터 믹스(H2O65μL, Q5 고충실도 DNA 중합효소 100μL, i5 프라이머 8μL, i7 프라이머 8μL)를 나머지 19μL의 스트렙타비딘-비오틴-DNA 샘플에 추가합니다. 반응 혼합물을 50-100 μL 부분 표본으로 분할합니다.
참고: PCR의 최적 부피는 PCR 기계에 따라 다릅니다. 일반적으로 50μL는 일반 PCR 기계에서 가장 재현성 있는 증폭을 제공합니다. - 8단계에서 결정된 사이클 수로 DNA 라이브러리 준비 키트의 제조업체 지침에 따라 PCR을 수행합니다. 단계 8을 생략한 경우, PCR에 대해 14 사이클을 권장합니다.
- 상자성 비드( 재료 표 참조)로 제조업체의 프로토콜에 따라 1:0.9의 비율로 DNA를 정제합니다. 0.1% TE의 20μL에서 용리합니다.
알림: 8.5단계에서 어댑터 이량체가 감지되면 동일한 비율을 사용하여 두 번 정제를 수행하십시오. - DNA 농도를 측정하고 품질 관리 시스템에서 샘플을 실행합니다( 재료 표 참조). 단일 정밀 대역의 존재로 Micro-C 라이브러리의 우수한 품질을 보장합니다(그림 1D).
참고: 어댑터 이량체는 PCR 전에 시료를 세척하기 때문에 on-bead 라이브러리 준비에서 일반적이지 않습니다. 따라서 어댑터 이량체의 모양은 라이브러리 복잡성이 낮다는 것을 나타냅니다. 어댑터 이량체가 관찰되면 낮은 입력 시퀀싱으로 샘플의 품질을 관리하는 것이 좋습니다.
10. DNA 염기서열 분석 및 데이터 처리
- Micro-C 라이브러리를 염기서열분석 공급자의 요구 사항에 따라 paired-end 염기서열분석으로 시퀀싱합니다.
참고: 이상적으로, 샘플은 읽기당 50bp의 paired-end 모드의 플랫폼에서 시퀀싱됩니다. 2 x 35bp와 같이 더 짧은 읽기 길이를 제공하는 이전 플랫폼도 성공적으로 사용되었습니다. 중요한 것은 반복적인 게놈 영역을 연구하는 경우 더 긴 판독 길이로 염기서열을 분석하는 것이 좋습니다. - Micro-C 라이브러리의 품질을 평가하려면 샘플당 5 x 106 - 1 x 107 read로 낮은 입력 시퀀싱을 수행합니다.
- Distiller10을 사용하여 시퀀싱 파일(fastq 파일)을 처리합니다. 적절한 참조 게놈(여기서는 mm10)에 대해 판독을 매핑합니다.
참고: 시퀀싱 파일은 로컬 컴퓨터 또는 컴퓨팅 클러스터에서 다양한 파이프라인을 사용하여 처리할 수 있습니다. 염기서열분석 깊이가 낮은 샘플의 경우 10,000bp, 50,000bp, 100,000bp 및 500,000bp와 같이 빈 크기가 크면 컴퓨팅 요구와 파일 크기를 줄일 수 있습니다. Distiller(이 연구에서 사용됨)는 Micro-C 라이브러리의 품질을 평가하는 데 필요한 모든 파일 형식을 생성합니다. 생성된 *.stats 파일에는 맵 속도, cis-trans 비율 및 읽기 쌍 사이의 거리로 계층화된 읽기 방향에 대한 정보가 포함되어 있습니다. 이러한 파라미터는 그림 2에 시각화되어 있으며, Micro-C 라이브러리 품질 평가는 대표적인 결과에서 논의됩니다. 프로세싱 소프트웨어는 또한 상호 작용 매트릭스(9)를 생성하기 위해 HiGlass(https://docs.higlass.io/)에 직접 로드할 수 있는 mcool 파일을 생성합니다.
결과
Micro-C 라이브러리의 성공적인 준비는 프로토콜의 여러 단계에서 평가할 수 있습니다. 가장 중요한 단계는 적절한 MNase 분해 정도를 선택하는 것입니다. 따라서 MNase 농도는 모든 샘플에 대해 디뉴클레오솜에 비해 70%-90% 모노뉴클레오솜을 일관되게 산출하도록 적정해야 합니다. 크로마틴 분해는 eu 크로마틴과 헤테로 크로마틴에 따라 다르며, MNase는 헤테로크로마틴을 덜 효율적으로 소화한다는 점에 유의해야 합니다. 따라서, 최적의 소화도는 관심 염색질 영역과 연구된 세포 유형에 따라 달라지는데, 이는 eu- 염색질 및 헤테로-염색질의 상대적 비율이 세포 유형에 따라 다르기 때문입니다. 따라서 필요한 MNase 농도를 신중하게 적정하고 먼저 저입력 시퀀싱을 통해 Micro-C 실험의 성공을 평가하는 것이 좋습니다.
감소하는 양의 MNase로 처리된 크로마틴의 일반적인 MNase 적정 패턴은 그림 1A에 나와 있습니다. 여기에서 반응당 250,000개 세포의 크로마틴을 MNase의 4배 희석으로 분해합니다. 가장 높은 농도(MNase 10U, 레인 2)는 거의 독점적으로 모노 뉴클레오솜 DNA(~150bp)로 구성된 과소화된 염색질을 보여줍니다. 특히, 모노 뉴클레오솜 밴드의 중심은 MNase 농도가 감소된 샘플의 해당 밴드에 비해 아가로스 겔에서 더 낮게 실행되며, 이는 뉴클레오솜 DNA의 과분해를 나타냅니다. 과잉 소화된 뉴클레오솜은 근접 결찰 반응에서 비효율적으로 결찰됩니다. 따라서 레인 2의 샘플은 Micro-C 실험에 최적이 아닙니다. 레인 3(MNase 2.5U)은 Micro-C 실험에 거의 적합한 분해도를 나타냅니다. 여기서, 단핵소체 띠가 우세한 종이며, 과잉 소화된 뉴클레오솜을 나타내는 아핵소체 도말이 감소한다. 그러나 여전히 존재합니다. 레인 4의 분해도(MNase 0.635U)는 이 적정 예에서 Micro-C 실험에 이상적인 조건입니다. 하위 뉴클레오솜 DNA가 없는 투명한 단핵솜 밴드가 존재합니다. 단핵체 및 이핵체 DNA의 띠 강도는 거의 동일하며, 이는 단핵체 수율이 66% 이상임을 나타냅니다. 이핵체 DNA는 단핵체 DNA의 약 두 배 크기(~320bp 대 ~150bp)이므로 DNA 몰당 밴드 강도가 단핵소체 DNA에 비해 두 배 높다는 점은 주목할 가치가 있습니다. 레인 5(MNase 0.156U)의 분해도는 뉴클레오솜 DNA가 거의 없는 소화 부족 염색질을 보여주며, 따라서 이는 차선의 샘플을 나타냅니다.
결론적으로, 이 예에서 0.625U의 MNase를 사용한 2.5 x 105 마우스 ES 세포의 분해(200μL의 1 x 106 세포에 대한 2.5U의 MNase에 해당)는 Micro-C 실험에서 분취 분해를 위한 가장 유망한 출발점을 제공합니다. 그러나 레인 3과 레인 4의 샘플에 사용된 조건(200μL의 1 x 106 셀에 대한 MNase 5U에 해당) 사이의 중간 MNase 농도도 고려해야 합니다. 중요한 것은 MNase를 사용한 크로마틴 분해는 선형적으로 스케일링할 수 없으며 분취 분해를 4배 이상 상향 스케일링하지 않는 것이 좋습니다. 1 x 10 6 세포 이상에서 Micro-C 라이브러리를 준비하려면 1 x 106 세포의 분취액에서 크로마틴을 분해하고 MNase 비활성화 후 통합하는 것이 좋습니다.
근접 결찰 프로토콜의 성공을 평가하려면 근접 결찰이 아닌 MNase 분해된 입력 대조군(단계 3.8)을 근접 결찰 샘플(단계 5.3)과 1.5% 아가로스 겔 전기영동으로 비교해야 합니다(그림 1B). 근접 결찰 단핵소체 밴드는 대략 300bp의 크기를 가지며 디뉴클레오솜과 유사합니다. 따라서 모노 대 디뉴클레오솜 대역 신호 비율은 주로 모노뉴클레오솜(1번 레인)에서 디뉴클레오솜(3번 레인 및 4번 레인)으로 이동해야 합니다. 이 단계에서 아가로스 겔은 절제 및 정제되는 디뉴클레오솜 DNA이므로 과부하를 피하기 위해 샘플을 여러 레인으로 분할하는 것이 좋습니다.
준비된 염기서열분석 라이브러리의 품질과 수량을 최소 PCR로 평가하는 것이 좋습니다. 여기서 1μL의 비드(전체 샘플의 1/20)의 DNA는 10μL의 PCR 반응에서 16주기 동안 증폭됩니다. 최소 PCR 라이브러리의 총 농도는 일반적으로 16회 PCR 주기 후 50-500ng 범위입니다. 이론적으로, 이는 16 사이클 동안 증폭된 경우 나머지 19 μL 샘플의 1-10 μg 라이브러리에 해당합니다. 총 DNA에서 약 100ng의 라이브러리를 생성하는 데 필요한 최소 PCR 주기 수를 사용하는 것이 좋습니다. PCR에서 로그 증폭을 가정하면 16 사이클에서 19 μL 입력에서 얻은 DNA의 이론적 농도를 연속적으로 2로 나누어 100 ng 라이브러리를 생성하는 데 필요한 PCR 사이클 수를 계산할 수 있습니다. 예를 들어, 16 사이클 후 1 μL에서 100 ng 수율은 19 μL에서 증폭된 1,900 ng 수율에 해당합니다. 이 시나리오에서 12 사이클은 총 DNA(1,900ng/[2 × 2 × 2 × 2] = 118ng)에서 118ng 염기서열분석 라이브러리를 이상적으로 생성해야 합니다. 그런 다음 최소 PCR에서 나머지 9μL 샘플을 사용하여 아가로스 겔 전기영동으로 라이브러리의 품질을 평가할 수 있습니다(그림 1C). 시각화는 420bp에서 하나의 고유한 밴드를 표시하고 어댑터 이량체(120bp)에 대한 밴드는 표시하지 않아야 합니다. 더 작은 파편은 또한 나타날지도 모르고, 이들은 사용되지 않는 PCR 뇌관에 대응한다.
다음으로, 리소스 집약적인 심층 염기서열분석을 시작하기 전에 저입력 염기서열분석을 통한 성공적인 Micro-C 시료 전처리를 분석하고 확인하는 것이 좋습니다. 일반적으로 라이브러리는 5 x 106 내지 1 x 107의 판독 깊이로 염기서열 분석되고 염기서열분석 판독 중복률, 시스염색체 대 트랜스염색체 상호작용 비율, 염기서열분석 판독 방향 빈도 기준에 따라 평가됩니다. Micro-C 라이브러리는 cooler, pairtools 및 cooltools10,11,12를 사용하여 읽기 파일(Fastq 형식)에서 읽기 쌍 파일(Bedpe 형식) 및 확장 가능한 상호 작용 매트릭스(Cool 및 Mcool 형식)에 이르는 시퀀싱에서 데이터를 처리하는 풀 서비스 파이프라인인 Distiller로 처리됩니다. 파이프라인은 또한 Micro-C 라이브러리(10)(https://github.com/open2c/distiller-nf)의 품질을 평가하는데 이상적인 요약 파일을 생성한다. PCR 복제율은 염기서열분석 라이브러리 복잡도에 대한 정보를 제공하며 생성된 *.stats 파일에서 추출할 수 있습니다. 고품질 Micro-C 라이브러리는 500만 개 이상의 세포에서 생성될 때 PCR 복제율이 5%-10% 미만입니다. 특히, 일부 염기서열분석 플랫폼은 염기서열분석 라이브러리 복잡성과 무관하게 클러스터 형성 중에 PCR 중복을 생성합니다. 그림 2A는 두 실험의 상대적 중복률을 보여줍니다: 하나는 좋은 샘플로 간주되고 다른 하나는 나쁜 샘플로 간주됩니다. 이 예에서는 두 샘플 모두 허용 가능한 맵 속도를 표시했습니다. Micro-C 라이브러리의 품질을 평가하는 다음 기준은 시스 대 트랜스 비율 및 읽기 방향 주파수입니다. 핵 내에서 염색체는 개별 염색체 영역에 서식하므로 다른 염색체와 거의 상호 작용하지 않습니다. 검출된 경염색체 상호작용의 높은 비율은 무작위 결찰의 높은 비율을 나타냅니다. 이 수준의 분석에서 불량 샘플은 양호한 샘플에 비해 높은 비율의 trans-chromosomal 상호 작용을 보였다는 점에 유의해야 합니다(그림 2B). Micro-C의 경우 70% 이상의 시스-염색체 상호작용 비율이 바람직합니다.
Micro-C 라이브러리는 디뉴클레오솜 DNA 밴드와 유사한 단편 크기를 가지고 있으며, 근접 결찰 샘플과 함께 정제되어 실험을 오염시킬 수 있습니다. 이러한 오염 물질은 항상 시스-염색체 상호 작용입니다. 따라서 읽기 방향 비율도 평가하는 것이 중요합니다. 디뉴클레오솜 오염 속도는 저입력 염기서열분석(low-input sequencing)으로 추정할 수 있습니다. 디뉴클레오솜 DNA는 MNase에 의해 절단되지 않은 두 개의 인접한 뉴클레오솜에서 비롯됩니다. 따라서 결과 시퀀싱 판독은 항상 정방향-역방향 판독 방향(F 및 R)을 표시하며 판독 쌍 사이의 거리는 약 320bp입니다. 이에 비해 근접 연결 단편은 4가지 방향으로 결찰할 수 있으며, F-R, R-R, R-F 및 F-F와 함께 판독 쌍을 생성할 수 있으며, 이상적으로는 동일한 함량을 제공합니다(그림 2C). 또한 두 판독 쌍 사이의 다양한 거리를 표시합니다. 디뉴클레오솜 오염물의 양을 추정하기 위해 증류기에서 생성된 *stats 파일에서 판독 방향의 빈도를 계산할 수 있습니다(그림 2D). 특히, 이 연구에서 F-R 판독의 비율(빨간색)은 양호한 샘플에 비해 불량 샘플에서 더 높았으며, 이는 판독 방향이 거리에 따라 계층화되었을 때 더욱 분명해졌습니다(그림 2E). F-R 분획은 판독 쌍이 <562 bp 또는 ≥562 bp 거리의 판독으로 계층화될 때 Micro-C 라이브러리와 비교하여 di-nucleosomal 단편에 의해 지배됩니다. 여기서, 거리가 <562 bp인 판독의 분율은 F-R 판독에 의해 지배되는 반면, 거리가 ≥562 bp인 분율은 4개의 가능한 방향 사이에 균일한 분포를 나타내며, 이는 F-R 판독의 전체 과잉 표현이 이뉴클레오솜 오염 물질에서 비롯됨을 나타냅니다. 하위 집합에 대한 임계값으로 562bp를 선택하는 것은 생성된 *stats 파일의 범주화에 의해 정의됩니다. 이 품질 관리에 반드시 필요한 것은 아니지만, 증류기에 의해 생성되는 *pairs 파일에서 거리를 추출하여 보다 정의된 하위 집합을 달성할 수 있습니다. 디뉴클레오솜 판독은 데이터 처리 중에 식별되고 무시될 수 있으므로 Micro-C 샘플의 품질을 저하시키지 않는다는 점에 유의하는 것이 중요합니다. 그러나 3D 상호 작용에 대한 중요한 정보가 포함되어 있지 않으며 정보 읽기를 희석시킵니다.
따라서 신중한 MNase 적정과 저입력 염기서열분석을 통한 철저한 품질 관리는 Micro-C 실험의 품질을 최적화하는 최고의 도구입니다.
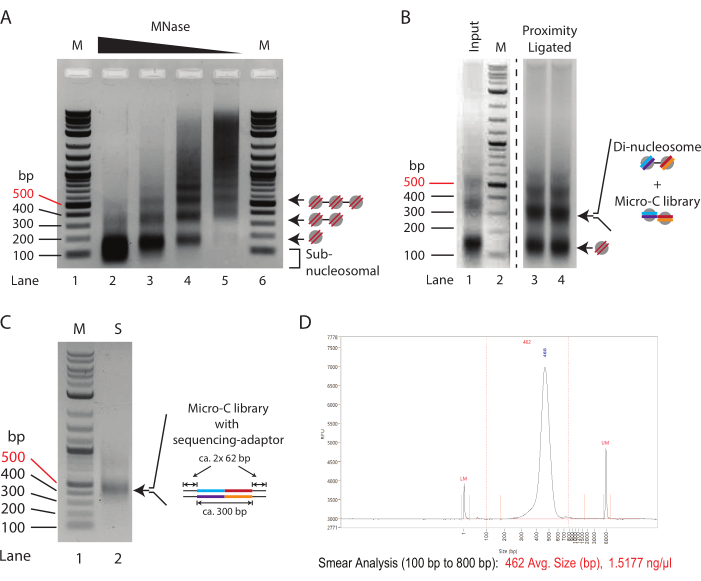
그림 1: Micro-C 프로토콜의 중간 단계 . (A) 다양한 MNase 농도로 분해된 2.5 x 105 개의 마우스 ES 세포에서 크로마틴의 아가로스 겔 전기영동. mono-, di- 및 tri-nucleosomal 밴드는 화살표로 표시됩니다. M: DNA 사다리(레인 1/6); 250,000개 셀당 10U의 MNase(레인 2); 250,000개 셀당 2.5U의 MNase(레인 3); 250,000개 셀당 0.625U의 MNase(레인 4); 250,000개 셀당 0.156U의 MNase(레인 2). (B) Micro-C 준비된 샘플(레인 3 및 레인 4) 및 MNase 분해 입력 컨트롤(레인 1)의 1.0% 아가로스 겔 전기영동. 레인 1 및 레인 2(M: DNA ladder)는 모노뉴클레오솜 및 디뉴클레오솜 단편 강도의 상대적 변화를 강조하기 위해 강화되었습니다. 단핵체 및 이핵체 띠는 화살표로 표시됩니다. 근접 결찰 샘플의 디뉴클레오솜 밴드는 디뉴클레오솜과 Micro-C 라이브러리 DNA를 결합합니다. (C) Micro-C 염기서열분석 라이브러리의 1.0% 아가로스 겔 전기영동을 1μL 샘플에서 증폭하여 품질을 평가했습니다. 레인 1 (M): DNA 사다리; 레인 2 (S): Mirco-C 라이브러리. (D) 최종 Micro-C 라이브러리의 단편 분석기 추적. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 2: 양호한 시료와 불량 시료의 저입력 시퀀싱에 대한 시료 통계량 . (A) 매핑된 비율(녹색)과 매핑되지 않은 읽기(빨간색)의 막대 그래프입니다. (B) cis 및 trans-chromosomal 상호 작용을 매핑하는 읽기의 정규화된 분율. 데이터 세트는 cis-mapping 읽기로 정규화되었습니다. 시스 매핑 판독은 쌍을 이루는 말단 서열 샘플의 첫 번째 판독과 두 번째 판독 사이의 거리(≤1kbp(노란색), >1kbp 및 ≤10kbp(주황색) 및 >10kbp(빨간색)로 계층화되었습니다. (C) 디뉴클레오솜 크기를 갖는 잠재적 분자 종의 개략도. (D) 양호한 샘플과 불량 샘플의 모든 판독에 대한 판독 쌍 방향의 백분율. (E) 패널 (D)와 동일하지만 거리로 계층화됩니다(왼쪽, <562bp 및 오른쪽, ≥562bp). 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
| 구성 요소 | 1배 | 4.4배 |
| NEBuffer 2.1 10개, | 10 μL | 44 μL |
| 2 μL 100 mM ATP | 2 μL | 8.8 μL |
| 100 mM DTT | 5 μL | 22 μL |
| H2O | 68 마이크로리터 | 299.2 마이크로리터 |
| 10 U/μL T4 PNK | 5 μL | 22 μL |
| 합계 | 90 μL | 396 마이크로리터 |
표 1: Micro-C 마스터 믹스 1. 최종 씹는 반응을 위한 마스터 믹스의 구성.
| 구성 요소 | 1배 | 4.4배 |
| 1mM 비오틴-dATP | 10 μL | 44 μL |
| 1mM 비오틴-dCTP | 10 μL | 44 μL |
| dTTP와 dGTP의 10mM 혼합 | 1 μL | 4.4 마이크로리터 |
| 10x T4 DNA 리가아제 완충액 | 5 μL | 22 μL |
| BSA 200배 | 0.25 마이크로리터 | 1.1 마이크로리터 |
| H2O | 23.75 마이크로리터 | 104.5 μL |
표 2: Micro-C 마스터 믹스 2. 최종 라벨링 반응을 위한 마스터 믹스의 조성.
| 구성 요소 | 1배 | 4.4배 |
| 10x NEB T4 리가아제 반응 완충액 | 50 μL | 220 마이크로리터 |
| H2O | 422,5 마이크로리터 | 1859 μL |
| T4 DNA 리가아제 | 25 μL | 110μL의 |
표 3: Micro-C 마스터 믹스 3. 근접 결찰 반응을 위한 마스터 믹스의 조성.
| 구성 요소 | 1배 | 4.4배 |
| NEBuffer 1.1 10개 | 20 μL | 88 μL |
| H2O | 180 μL | 792 μL |
| ExoIII 뉴클레아제 | 10 μL | 44 μL |
표 4: Micro-C 마스터 믹스 4. 비오틴 제거 반응을 위한 마스터 믹스의 조성물.
토론
Micro-C 실험의 성공 여부는 신중하게 실행해야 하는 프로토콜의 몇 가지 중요한 단계에 달려 있습니다. 첫째, 추가적인 가교결합제인 DSG 또는 EGS와의 가교결합은 세포 유형에 따라 세포의 응집을 유도할 수 있다. 가교 반응에 0.1%-0.5% BSA를 첨가하면 가교 효율에 영향을 주지 않고 응집이 크게 감소합니다. 비효율적인 교차결합은 무작위 결찰을 나타내는 trans-chromosomal 상호 작용의 비율을 증가시킬 수 있습니다. 이 프로토콜에서 두 번째이지만 가장 중요한 단계는 MNase를 사용한 염색질 분해입니다. 최적이 아닌 염색질 분해는 비효율적인 근접 결찰(과소화) 또는 비근접 결찰 디뉴클레오솜의 속도 증가(소화 부족)로 이어집니다. 결찰 반응의 효율은 아가로스 겔 전기영동(그림 1B)으로 평가할 수 있으며, 저투입 시퀀싱으로 가장 잘 추정할 수 있습니다. 낮은 입력 염기서열분석에서 높은 복제율(비효율적인 결찰) 또는 증가된 디뉴클레오솜 비율이 나타나는 경우 MNase 분해 정도를 재평가해야 합니다. 특히, 프로토콜을 실행할 때 샘플이 손실되면 라이브러리 복잡성이 줄어들 수 있습니다. 샘플의 농도는 DNA 정제 후(5.3단계) 또는 최소 PCR(8단계)로 평가하는 것이 가장 좋습니다. DNA 정제 후 5 x 106 포유류 세포의 총 DNA 수율은 일반적으로 >2μg입니다. DNA 농도는 MNase 분해, ExoIII 분해 및 DNA 정제 후에 제어되어야 합니다. 세포 유형 특이적 및 종 특이적 인 내인성 뉴클레아제는 DNA 분해의 원인이 될 수 있습니다. 또한 컬럼 기반 DNA 정제는 탈단백질화 반응에서 SDS와의 비호환성으로 인해 샘플 손실이 발생할 수 있습니다. 이 단계에서 DNA 농도가 낮으면 에탄올 침전을 고려할 수 있습니다.
Micro-C는 시료 특이적 MNase 적정이 필요하기 때문에 다양한 모델 유기체의 작은 장기, 배아 및 단일 세포, 오가노이드 또는 환자 생검과 같은 소세포 집단에 Micro-C를 적용하는 것은 어렵습니다. 여기에서 Hi-C 3.0은 염기서열 특이적 제한 엔도뉴클레아제 8,9에 의한 종말점 반응을 사용하여 잘 정립된 대안을 제공합니다.
Micro-C는 높은 동적 범위와 낮은 신호 대 잡음비를 가진 널리 적용 가능한 고분해능 염색체 구조 기술로, 염색체 루프와 같은 단거리 염색체 특징 4,5,8을 조사하는 데 특히 적합합니다. Micro-C의 분해능은 Hi-C의 검출 한계를 넘어서는 promoter-enhancer loop를 캡처할 수 있어 게놈 조직과 조절13,14,15 사이의 관계에 대한 보다 자세한 분석을 효율적으로 가능하게 합니다. 또한, DNA 포획 전략은 최근 Micro-C와 결합되어 표적 게놈 유전자좌의 유전자좌 특이적 분해능을 전례 없는 수준으로 증가시켜 3D 게놈16,17,18의 미세구조에 대한 새로운 통찰력을 드러냈습니다. 요약하면, Micro-C와 그 유도체는 전사 조절에서 3D 게놈의 역할을 해부하고 결과적으로 세포 유형 분화 및 유지를 위한 핵심 기술이 될 것으로 예상합니다.
공개
저자는 공개할 것이 없습니다.
감사의 말
원고를 비판적으로 읽어준 Christl Gaubitz와 Kathleen Stewart-Morgan에게 감사드립니다. 우리 연구실 설립에 도움을 주신 Anja Groth와 Groth 연구실에 감사드립니다. CPR/reNEW Genomics Platform의 지원해 주신 H. Wollmann, M. Michaut, A. Kalvisa에게 감사드립니다. 노보 노디스크 재단 줄기세포 의학 센터(reNEW)는 노보 노디스크 재단 보조금 번호 NNF21CC0073729의 지원을 받습니다. 노보 노디스크 재단 단백질 연구 센터(CPR)는 노보 노디스크 재단 보조금 번호 NNF14CC0001의 지원을 받습니다. 코펜하겐에 위치한 노보 노디스크 줄기세포 의학 센터(Novo Nordisk Center for Stem Cell Medicine)의 브릭맨(Brickman) 연구실에 마우스 ES 세포에 대해 감사의 뜻을 전합니다.
자료
| Name | Company | Catalog Number | Comments |
| 1 mM Biotin dATP | Jenna Bioscience | NU-835-Bio14-S | |
| 1 mM Biotin dCTP | Jenna Bioscience | NU-809-BioX-S | |
| 10 mM dGTP | NEB | N0442S | |
| 10 mM dTTP | NEB | N0443S | |
| 10 U/ml T4 PNK | NEB | M0201L | |
| 100 U/L Exonuclease III | NEB | M0206L | |
| 10x NEBuffer 1.1 | NEB | B7001S | |
| 10x NEBuffer 2.1 | NEB | B7202S | |
| 10x T4 DNA Ligase buffer | NEB | B0202A | |
| 1x DPBS w/o Mg2+ and Ca2+ | ThermoFisher | 14190144 | |
| 1x LIF | |||
| 2_Mercaptoethanol 50 mM | Gibco | 31350010 | 0.1 mM b-mercaptoethanol |
| 37% Formaldehyde | Sigma Aldrich | 252549-500ML | Caution. See manufactures MSDS |
| 400 U/ml T4 DNA Ligase | NEB | M0202L | |
| 5 U/ml Klenow Fragment | NEB | M0210L | |
| Agarose | BIO-RAD | 1613102 | Caution. See manufactures MSDS |
| BSA 20mg/ml | NEB | B9000S | |
| CaCl2 | |||
| cell counter | |||
| Dimethyl Sulfoxide (DMSO) | Sigma Aldrich | D8418-100ML | Caution. See manufactures MSDS |
| Dynabeads MyOne Streptavidin C1 | Invitrogen | 65001 | |
| DynaMag-2 Magnet | Invitrogen | 12321D | refered to as: magnet magnet for 1.5 ml tubes |
| DynaMag-PCR Magnet | Invitrogen | 492025 | refered to as: magnet magnet for PCR tubes |
| EDTA Ultrapure 0.5M pH 8.0 | Invitrogen | 15575-038 | |
| EGTA Ultrapure 0.5M pH 8.0 | BioWorld | 40121266-1 | |
| Ethanol 96% | VWR Chemicals | 20824365 | quality control system |
| Ethidium Bromide | Invitrogen | 15585-011 | |
| Ethylene glycol bis(succinimidyl succinate) (EGS) | ThermoFisher | 21565 | |
| Fetl Bovin Serum | Sigma Aldrich | F7524 | 15% FBS |
| Gel Loading dye purple (6X) | NEB | B7024S | |
| Glycine | PanReac AppliChem | A1067.0500 | |
| Halt Proteinase inhibitor (100x) | ThermoFisher | 78430 | Caution. See manufactures MSDS |
| IGEPAL CA-630 (NP-40) | Sigma Aldrich | 18896-50ML | |
| MgCl 1 M | Invitrogen | AM9530G | |
| Micrococcal Nuclease (MNase) | Worthington | LS004798 | |
| mouse embryonic stem cells | |||
| NaCl | Sigma Aldrich | S9888-1KG | |
| NEBNext Multiplex Oligos for Illumina (Dual Index primers) | NEB | E7600S | amplification primers for sequencing libraries |
| NEBNext Ultra II DNA library prep kit for Illumina | NEB | E7645L | sequencing library preparation kit |
| NEBNext Ultra II Q5 Master mix | NEB | M0544S | Caution. See manufactures MSDS |
| Non-Essential Amino Acids Solution | Gibco | 11140050 | 1x NEAA |
| Penicillin-Streptomycin (10,000 U/mL) | Gibco | 15140148 | 1% Pen-Strep |
| Proteinase K (40 mg/ml) | GoldBio | P-480-1 | Caution. See manufactures MSDS |
| QIAquick Gel extraction kit | QIAgen | 28706 | refered to as: DNA gel elution kit |
| QIAquick PCR purification kit | QIAgen | 28106 | refered to as: commercial DNA purification kit |
| Qubit dsDNA HS Assay kit | Invitrogen | Q32854 | high sensitivity DNA quantification instrument |
| Quick load purple 1kb plus DNA Ladder | NEB | N0550S | |
| SPRIselect size selection beads | Beckman Coulter | B23319 | paramagnetic beads |
| ThermoMixer C | Eppendorf | 5382000015 | refered to as: thermomixer |
| Tris | Merck | 10708976001 | |
| Trypsin | |||
| Tween20 | Sigma Aldrich | P7949-100ML | |
| Ultrapure 10% SDS | Invitrogen | 15553-035 | |
| Ultrapure Phenol Chloroform Isoamyl Alcohol (PCI) | Invitrogen | 15593-031 | |
| Fragment Analyzer |
참고문헌
- Lieberman-Aiden, E., et al. Comprehensive mapping of long-range interactions reveals folding principles of the human genome. Science. 326 (5950), 289-293 (2009).
- Hsieh, T. -H. S., Fudenberg, G., Goloborodko, A., Rando, O. J. Micro-C XL: Assaying chromosome conformation from the nucleosome to the entire genome. Nature Methods. 13 (12), 1009-1011 (2016).
- Hsieh, T. -H. S., et al. Mapping nucleosome resolution chromosome folding in yeast by Micro-C. Cell. 162 (1), 108-119 (2015).
- Krietenstein, N., et al. Ultrastructural details of mammalian chromosome architecture. Molecular Cell. 78 (3), 554-565 (2020).
- Hsieh, T. -H. S., et al. Resolving the 3D landscape of transcription-linked mammalian chromatin folding. Molecular Cell. 78 (3), 539-553 (2020).
- Belaghzal, H., Dekker, J., Gibcus, J. H. Hi-C 2.0: An optimized Hi-C procedure for high-resolution genome-wide mapping of chromosome conformation. Methods. 123, 56-65 (2017).
- van Holde, K. E. Chromatin. , Springer. New York, NY. (1989).
- Oksuz, B. A. Systematic evaluation of chromosome conformation capture assays. Nature Methods. 18, 1046-1055 (2021).
- Lafontaine, D. L., Yang, L., Dekker, J., Gibcus, J. H. Hi-C 3.0: Improved protocol for genome-wide chromosome conformation capture. Current Protocols. 1 (7), 198(2021).
- Goloborodko, A., Venev, S., Abdennur, N., Di Tommaso, P. Mirnylab/distiller-nf; v033. Zenodo. , (2019).
- Venev, S., et al. Open2c/cooltools: v0.4.1. Zenodo. , (2021).
- Abdennur, N., Mirny, L. A. Cooler: Scalable storage for Hi-C data and other genomically labeled arrays. Bioinformatics. 36 (1), 311-316 (2019).
- Zhang, S., Übelmesser, N., Barbieri, M., Papantonis, A. Enhancer-promoter contact formation requires RNAPII and antagonizes loop extrusion. BioRxiv. , (2022).
- Barshad, G., et al. RNA polymerase II and PARP1 shape enhancer-promoter contacts. BioRxiv. , (2022).
- Hansen, A. S., et al. Distinct classes of chromatin loops revealed by deletion of an RNA-binding region in CTCF. Molecular Cell. 76 (3), 395-411 (2019).
- Hua, P., et al. Defining genome architecture at base-pair resolution. Nature. 595 (7865), 125-129 (2021).
- Downes, D. J. High-resolution targeted 3C interrogation of cis-regulatory element organization at genome-wide scale. Nature Communications. 12, 531(2021).
- Goel, V. Y., Huseyin, M. K., Hansen, A. S. Region capture Micro-C reveals coalescence of enhancers and promoters into nested microcompartments. BioRxiv. , (2022).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유