Method Article
Mappatura delle interazioni del genoma 3D dei mammiferi con Micro-C-XL
In questo articolo
Riepilogo
Viene presentato un protocollo per mappare l'organizzazione tridimensionale del genoma con risoluzione nucleosomica utilizzando il metodo di cattura della conformazione cromosomica a livello di genoma Micro-C-XL.
Abstract
L'organizzazione tridimensionale (3D) dei cromosomi è un fattore importante nella regolazione del genoma e nella specificazione del tipo di cellula. Ad esempio, si ritiene che gli elementi cis-regolatori, noti come enhancer, regolano l'attività dei promotori distali attraverso l'interazione nello spazio 3D. Le tecnologie di cattura della conformazione cromosomica a livello di genoma (3C), come Hi-C, hanno trasformato la nostra comprensione di come i genomi sono organizzati nelle cellule. L'attuale comprensione dell'organizzazione del genoma 3D è limitata dalla risoluzione con cui l'organizzazione topologica dei cromosomi nello spazio 3D può essere risolta. Micro-C-XL misura il ripiegamento cromosomico con risoluzione a livello del nucleosoma, l'unità di base della cromatina, utilizzando la nucleasi micrococcica (MNasi) per frammentare i genomi durante il protocollo di cattura della conformazione cromosomica. Ciò si traduce in un migliore rapporto segnale/rumore nelle misurazioni, facilitando così il rilevamento migliore dei siti di isolamento e dei loop cromosomici rispetto ad altre tecnologie 3D a livello di genoma. In questo articolo viene presentato un protocollo dettagliato, visivamente supportato e passo-passo per la preparazione di campioni Micro-C-XL di alta qualità da cellule di mammifero.
Introduzione
Micro-C-XL è una tecnica a livello di genoma per misurare la conformazione del genoma 3D con risoluzione nucleosomica. Micro-C-XL si basa sulla tecnologia Hi-C basata sulla legatura di prossimità, ampiamente utilizzata, che ha trasformato la nostra comprensione di come sono organizzati i genomi 3D1. Micro-C-XL e la sua prima iterazione, Micro-C, sono stati inizialmente sviluppati in Saccharomyces cerevisiae2,3 e successivamente adattati ai sistemi cellulari di mammifero, per i quali il protocollo ha dimostrato il suo pieno potenziale nel rilevare le caratteristiche a corto raggio del genoma 3D, come le anse cromosomiche e i siti di isolamento. Questa versione si basa su recenti pubblicazioni sui mammiferi Micro-C-XL 4,5. Poiché Micro-C-XL sostituisce Micro-C, Micro-C-XL è d'ora in poi indicato come Micro-C nel manoscritto.
Le principali differenze tra Micro-C e Hi-C6 sono le seguenti: 1) frammentazione del genoma con nucleasi micrococcica (MNasi) rispetto agli enzimi di restrizione e 2) reticolanti aggiuntivi con maggiore spaziatura atomica tra i gruppi reattivi rispetto alla sola formaldeide. Entrambi i passaggi contribuiscono in modo significativo al miglioramento del rapporto segnale/rumore del Micro-C rispetto all'Hi-C convenzionale. La dimensione della frammentazione limita la risoluzione a cui l'organizzazione del genoma 3D può essere risolta durante il protocollo di legatura di prossimità. La MNasi è una nucleasi che digerisce preferenzialmente il DNA accessibile e lascia intatto il DNA protetto dai nucleosomi. L'impronta nucleosomica mediante sequenziamento della MNasi ha dimostrato che i nucleosomi coprono completamente la maggior parte dei genomi eucariotici7. Poiché i nucleosomi sono distribuiti in tutto il genoma con una spaziatura media di 160-220 bp, a seconda della specie e del tipo di cellula, la MNasi è l'enzima ideale per la mappatura ad alta risoluzione dell'architettura del genoma.
L'uso di un reticolante aggiuntivo in combinazione con la formaldeide (FA) nel metodo Micro-C migliora ulteriormente il rapporto segnale/rumoredi 2,8. I reticolanti specifici per le ammine con spaziatori atomici più lunghi tra i gruppi reattivi facilitano i legami tra proteina e proteina. Si tratta in genere di disuccinimidil glutarato (DSG) o glicole etilenico bis-succinimidil succinato (EGS) con distanziatori rispettivamente da 7,7 Å e 16,1 Å. La riduzione del rumore attraverso EGS o DSG è particolarmente evidente negli esperimenti con alti tassi di frammentazione, come Micro-C, e presumibilmente si verifica a causa di una riduzione del tasso di eventi di legatura casuale8.
Un protocollo Hi-C 3.0 sviluppato di recente che utilizza la reticolazione ESG/DSG e molteplici combinazioni di enzimi di restrizione riduce il rumore negli esperimenti Hi-C e migliora significativamente il rilevamento di loop cromosomici e siti di isolamento 8,9. Tuttavia, un confronto sito per sito di varie caratteristiche dei dati di interazione ha rilevato che Micro-C aveva una rilevazione superiore di caratteristiche a corto raggio, come i loop cromosomici e i siti di isolamento, rispetto sia a Hi-C 3.0 che a Hi-C8 convenzionale. Tuttavia, Hi-C 3.0 migliora il rilevamento delle caratteristiche a corto raggio e mantiene un forte rilevamento della compartimentazione del genoma rispetto all'Hi-C convenzionale. In sintesi, la scelta di un metodo di cattura della conformazione cromosomica dovrebbe essere determinata dalla questione oggettiva e biologica.
Qui, forniamo un protocollo passo-passo per esperimenti Micro-C di successo che possono svelare l'organizzazione del genoma 3D.
Protocollo
1. Coltura cellulare e reticolazione
- Colture cellulari secondo le esigenze sperimentali per ottenere un minimo di 1 x 107 cellule. Qui, le cellule sono state coltivate a 37 °C con il 5% di CO2 in terreno E14 (DMEM con piruvato e L-glutammina, 15% FBS, 1x LIF, 1x NEAA, 1% pen-streptococco, 0,1 mM di β-mercaptoetanolo [vedi tabella dei materiali]) e passate ogni due giorni.
NOTA: Questo protocollo è stato applicato con successo a vari tipi di cellule di più specie, come Homo sapienss e Mus musculus. In questo esempio, sono state utilizzate cellule staminali embrionali (ES) di topo. - Raccogliere le cellule al 70%-80% di confluenza aspirando il terreno. Lavare una volta con 5 mL di DPBS e incubare le cellule con 3 mL di tripsina allo 0,25% preriscaldata per piatto da 10 cm per 2-3 minuti a 37 °C.
- Spegnere la tripsina con 7 mL di terreno E14 preriscaldato e trasferire le cellule staccate in una provetta da 50 mL.
- Pellettare le cellule mediante centrifugazione a 300 x g per 5 minuti a temperatura ambiente (RT). Scartare il surnatante e risospendere il pellet cellulare nel terreno. Conta le celle con un contatore di celle.
NOTA Il protocollo è robusto per alcune variazioni nelle concentrazioni cellulari e sono stati utilizzati vari contatori di cellule per quantificare il numero di cellule; Tuttavia, prestare attenzione alla gamma dinamica del contatore di celle utilizzato. Questo è in genere compreso tra 1 x 105-1 x 107 cellule/mL. Sebbene la velocità e la durata della centrifugazione siano state testate per questo tipo di cellule, celle diverse e più piccole possono richiedere una velocità di centrifugazione più elevata o tempi di centrifugazione più lunghi e la centrifugazione deve essere regolata di conseguenza. - Raccogliere le cellule mediante centrifugazione a 300 x g per 5 minuti a RT e risospendere le cellule in DPBS a una concentrazione finale di 1 x 106 cellule/mL. Ad esempio, se la resa è di 1 x 107 cellule, risospendere le cellule in 10 mL di DPBS.
- Per la prima fase di reticolazione, aggiungere il 37% di formaldeide (FA) a una concentrazione finale dell'1% alla sospensione cellulare e incubare la sospensione cellulare per 10 minuti a RT con rotazione (15-20 rpm); ad esempio, se la resa è di 1 x 107 cellule, aggiungere 270 μL di FA alla sospensione cellulare da 10 mL.
NOTA: La soluzione FA è in genere stabile a RT fino a 3 mesi dopo l'apertura. - Estinguere la reazione aggiungendo 2,5 M di glicina ad una concentrazione finale di 0,25 M e incubare per 5 minuti a RT con rotazione (15-20 rpm). Ad esempio, se la resa è 1 x 107 cellule, aggiungere 1,027 mL di glicina 2,5 M alla sospensione cellulare.
- Raccogliere le cellule mediante centrifugazione a 300 x g per 5 minuti a RT. Scartare il surnatante e risospendere le cellule in 5 mL di DPBS. Ripetere la fase di centrifugazione una volta e risospendere le cellule pellettate in DPBS a 4 x 106 cellule/mL. Ad esempio, se la resa è di 1 x 107 cellule, risospendere le cellule in 2,5 mL di DPBS.
- Per la seconda fase di reticolazione, preparare una soluzione madre 0,3 M di glicole etilenico bis-succinimidil succinato (EGS) in DMSO (13,6 mg di EGS in 100 μL di DMSO). Aggiungere EGS ad una concentrazione finale di 3 mM alla sospensione cellulare e incubare a RT per 40 minuti con rotazione (15-20 rpm). Ad esempio, se la resa è di 1 x 107 cellule, aggiungere 25 μL di soluzione madre EGS 0,3 M a 2,5 mL di sospensione cellulare.
NOTA: Equilibrare l'EGS su RT per almeno 20 minuti prima di pesare per preparare la soluzione madre. - Estinguere la reazione aggiungendo 2,5 M di glicina ad una concentrazione finale di 0,4 M e incubare per 5 minuti a RT con rotazione (15-20 rpm). Ad esempio, se la resa è di 1 x 107 cellule, aggiungere 400 μL di glicina 2,5 M alla sospensione cellulare.
- Raccogliere le cellule mediante centrifugazione a 1.000 x g per 5 minuti a RT e risospendere le cellule pellettate in DPBS a 5 x 106 cellule/mL. Distribuire 5 x 106 cellule/provetta per le librerie preparative e 1 x 106 cellule/provetta per la titolazione per la digestione della MNasi.
NOTA: Si consiglia di titolare il grado di digestione ottimale per ogni lotto di cellule reticolate. Idealmente, raccogliere da due a tre aliquote di 1 x 10 6 cellule per gli esperimenti di titolazione MNasi (fase 2) e da due a quattro aliquote da 5 x 106 cellule per gli esperimenti preparativi (fase 3). - Raccogliere le cellule mediante centrifugazione a 1.000 x g per 5 minuti a RT e rimuovere il surnatante. Congelare i pellet cellulari in azoto liquido e conservarli a -80 °C.
NOTA: Le librerie Micro-C di successo possono essere preparate da campioni conservati per un massimo di 3 mesi.
2. Titolazione MNase
NOTA: L'esecuzione di una titolazione MNase è necessaria per determinare la concentrazione ottimale di MNasi prima di elaborare la libreria preparativa delle cellule a doppia reticolazione.
- Per eseguire la titolazione MNasi, scongelare un pellet di 1 x 106 cellule su ghiaccio per 10 minuti e risospendere le cellule in 500 μL di DPBS (aggiungere 1x BSA se le cellule si attaccano alla parete). Incubare la sospensione cellulare su ghiaccio per 20 min.
- Raccogliere le cellule mediante centrifugazione a 10.000 x g per 5 minuti a RT e rimuovere il surnatante. Risospendere le cellule pellettate in 500 μL di tampone MB#1 (50 mM NaCl, 10 mM Tris-HCl, 5 mM MgCl 2, 1 mM CaCl 2,0,2% NP-40, 1x inibitore della proteasi [vedi tabella dei materiali], pH 7,4).
- Raccogliere le cellule mediante centrifugazione a 10.000 x g per 5 minuti e rimuovere il surnatante. Risospendere le cellule in 200 μL di tampone MB#1 e dividere il campione in quattro provette.
- Scongelare un flaconcino di MNasi (20 U/μL) e diluire con 10 mM Tris, pH 7,4, in una serie di 1:2, 1:4, 1:4 e 1:4 per ottenere concentrazioni rispettivamente di 10 U/μL, 2,5 U/μL, 0,625 U/μL e 0,1256 U/μL (una per ogni condizione di digestione). Con opportuni intervalli di tempo (10-20 s), aggiungere 1 μL di soluzione di MNasi a uno dei quattro campioni, vortex, e incubare su un termomiscelatore a 37 °C per 10 minuti (agitazione a 800 giri/min). Continuare con l'aggiunta di 1 μL dalle diluizioni MNase rimanenti alle aliquote cellulari rimanenti.
- Arrestare la digestione della Mnase aggiungendo 200 μL di tampone STOP appena preparato (150 μL di 10 mM Tris, pH 7,4, 25 μL di SDS al 10%, 25 μL di proteinasi K da 20 mg/mL, 2 μL di EGTA 0,5 M) a ciascuna provetta nello stesso ordine e con lo stesso intervallo di tempo in cui è stata aggiunta la MNasi. Incubare a 65 °C per 2 ore.
- Aggiungere 500 μL di alcol fenolo-cloroformio-isoamilico (PCI) a ciascun campione e mescolare accuratamente mediante vortice. Centrifugare a 19.800 x g per 5 minuti a RT per separare le fasi e trasferire la fase acquosa in nuove provette (circa 200 μL/campione).
ATTENZIONE: Il PCI contiene numerosi componenti tossici e deve essere maneggiato solo in una cappa di sicurezza chimica. Si prega di consultare il produttore per maggiori dettagli. - Purificare il DNA utilizzando un kit di purificazione del DNA commerciale (vedere la tabella dei materiali) secondo le istruzioni del produttore ed eluire i campioni in 12 μL di tampone di eluizione.
NOTA: La concentrazione di SDS dalla fase di deproteinizzazione (fase 2.5) è inibitoria per alcuni kit di purificazione del DNA. Il kit di purificazione del DNA utilizzato qui funziona in modo paragonabile alla precipitazione dell'etanolo. - Aggiungere 2-5 μL di colorante di carico ed eseguire i campioni su un gel di agarosio all'1,5% a 120 V per 30-50 minuti (fino a quando non sono stati separati correttamente; Figura 1A).
- Scegli il miglior grado di digestione per l'esperimento e continua con la digestione preparativa della MNasi. Un grado di digestione ottimale mostra pochi o nessun frammento subnucleosomiale e un rapporto mono- e di-nucleosoma del 70%-90%.
NOTA: In questo esperimento, il grado di digestione della corsia 4 nella Figura 1A è stato determinato come la digestione ottimale ottenuta con 0,625-1,25 U di Mnase per 2,5 x 10 5 cellule (= 2,5-5 U di Mnase per 1 x 106 cellule). Per una discussione dettagliata, vedere i risultati rappresentativi.
3. Digestione preparativa della MNasi
- Per frammentare la cromatina nei mononucleosomi tramite digestione MNasi, scongelare aliquote cellulari 5 x 106 reticolate in precedenza e risospendere in 1 mL di DPBS. Incubare su ghiaccio per 20 minuti (aggiungere 1x BSA se le cellule si attaccano alla parete del tubo).
- Raccogliere le cellule mediante centrifugazione a 10.000 x g per 5 minuti a RT ed eliminare il surnatante. Risospendere il pellet in 500 μL di tampone MB#1. Ripetere una volta la fase di centrifugazione. Risospendere il pellet in 1 mL di tampone MB#1 e fare aliquote da 200 μL (1 x 106 cellule per aliquota).
- Sulla base della titolazione della MNasi (descritta nella fase 2), digerire la cromatina aggiungendo la quantità appropriata di MNasi (di solito 2,5-10 U/μL per 1 x 106 cellule) a ciascuna aliquota. Mescolare bene (vorticare e centrifugare velocemente) e incubare su un termomiscelatore a 37 °C per 10 minuti agitando a 800 giri/min.
- Arrestare la digestione della MNasi aggiungendo 1,6 μL di EGTA 0,5 M (4 mM finali) a ciascuna aliquota e incubare su un termomiscelatore a 65 °C per 10 minuti con agitazione a 800 giri/min.
- Raccogliere il campione mediante centrifugazione a 10.000 x g per 5 minuti a RT ed eliminare il surnatante. Risospendere il pellet cellulare in 500 μL di 1x NEBuffer 2.1.
- Raggruppare campioni equivalenti a un input di 5 x 106 celle o meno per l'ulteriore elaborazione.
NOTA: Se devono essere processate più di 5 x 10 6 cellule, trattare questi campioni in parallelo, poiché le condizioni enzimatiche sono ottimizzate per 5 x 106 cellule. - Prima di procedere alle fasi di legatura di prossimità, trasferire il 10% del campione come controllo di ingresso per controllare il livello di digestione della MNasi. Aggiungere 150 μL di Tris 10 mM, pH 7,4, 25 μL di SDS al 10% e 25 μL di proteinasi K da 20 mg/mL a questo campione e incubare per una notte a 65 °C.
4. Elaborazione finale del DNA e legatura di prossimità
- Raccogliere il campione rimanente mediante centrifugazione a 10.000 x g per 5 minuti a 4 °C ed eliminare il surnatante. Risospendere il pellet in 90 μL di Micro-C master mix 1 appena preparato (Tabella 1) e incubare su un termomiscelatore per 15 minuti a 37 °C con agitazione a 800 giri/min.
- Aggiungere 10 μL di frammento di Klenow da 5 U/μL e incubare su un termomiscelatore per 15 minuti a 37 °C con agitazione a 800 giri/min.
- Aggiungere 100 μL di Micro-C master mix 2 appena preparato (Tabella 2) e incubare su un termomiscelatore per 45 minuti a 25 °C con agitazione a 800 giri/min. Dopo l'incubazione, spegnere la reazione enzimatica aggiungendo EDTA a una concentrazione finale di 30 mM. Incubare su un termomiscelatore per 20 minuti a 65 °C con agitazione a 800 giri/min.
- Raccogliere il campione mediante centrifugazione a 10.000 x g per 5 minuti a 4 °C ed eliminare il surnatante. Risospendere il campione in 500 μL di miscela master Micro-C 3 appena preparata (Tabella 3) e incubare per 2,5 ore a RT con rotazione (15-20 rpm).
- Raccogliere il campione mediante centrifugazione a 10.000 x g per 5 minuti a 4 °C ed eliminare il surnatante. Risospendere il campione in 200 μL di Micro-C master mix 4 appena preparato (Tabella 4) e incubare su un termomiscelatore per 15 minuti a 37 °C con agitazione a 800 giri/min.
- Per la reticolazione inversa e la deproteinazione, aggiungere al campione 25 μL di proteinasi K da 20 mg/mL e 25 μL di SDS al 10% e incubare a 65 °C per una notte con miscelazione intermittente.
5. Purificazione del DNA di-nucleosomiale e selezione delle dimensioni
- Aggiungere 500 μL di PCI ai campioni e al controllo dell'ingresso e miscelare mediante vortex. Separare le fasi mediante centrifugazione a 19.800 x g per 5 minuti e trasferire la fase acquosa superiore in una provetta fresca.
- Concentrare il DNA utilizzando un kit di purificazione del DNA o mediante precipitazione con etanolo. Eluire i campioni in 30 μL (fase 5.3) e i controlli in ingresso (fase 3.11) in 15 μL ed eseguire un gel di agarosio all'1,5 % per separare i mononucleosomi e i dinucleosomi (Figura 1B).
NOTA: La concentrazione di SDS dalla fase di deproteinizzazione è inibitoria per alcuni kit di purificazione del DNA. Il kit di purificazione del DNA utilizzato qui (vedi Tabella dei materiali) ha prestazioni paragonabili alla precipitazione dell'etanolo. A seconda del numero di celle utilizzate, l'input può variare da 100 ng a 10 μg. Tipicamente, 1-5 μg di DNA vengono estratti da 5 x 106 cellule. - Asportare i frammenti di DNA che hanno una dimensione di-nucleosomiale (circa 300 bp). Utilizzare un kit di eluizione su gel di DNA disponibile in commercio (vedere Tabella dei materiali) per estrarre il DNA dal gel di agarosio ed eluire in 150 μL.
6. Preparazione delle perle di streptavidina
- Trasferire 10 μL di perle di streptavidina (vedere la tabella dei materiali) per campione in una provetta di reazione.
- Inserirlo in un magnete appropriato per provette da 1,5 mL (vedere la tabella dei materiali). Dopo che la soluzione si è chiarita (1-2 minuti), rimuovere il surnatante e risospendere le microsfere in 300 μL di 1x TBW (5 mM Tris-HCl, pH 7,5, 0,5 mM EDTA, 1 M NaCl, 0,05% Tween 20) per campione. Ripeti questo passaggio una volta.
- Risospendere le microsfere in 150 μL di tampone B&W 2x (10 mM Tris-HCl, pH 7,5, 1 mM EDTA, 2 M NaCl) per campione processato nella fase 7.
7. Pull-down della streptavidina e preparazione della libreria on-bead
- Aggiungere 150 μL di microsfere pre-preparate (passaggio 6.3) a 150 μL del campione (passaggio 5.3). Incubare per 20 minuti a RT con rotazione (15-20 rpm).
- Posizionare i tubi in un magnete appropriato e attendere che la soluzione si chiarisca (1-2 minuti). Rimuovere il surnatante e risospendere le perle in 300 μL di 1x TBW. Ripeti questo passaggio.
- Posizionare i tubi in un magnete appropriato e attendere che la soluzione si schiarisca (1-2 min). Rimuovere il surnatante e risospendere le perle in 100 μL di 0,1x TE (1 mM Tris, 0,1 mM EDTA, pH 8,0).
- Posizionare i tubi in un magnete appropriato e attendere che la soluzione si schiarisca (1-2 min). Rimuovere il surnatante, risospendere le microsfere in 50 μL di TE 0,1x e trasferirle in provette PCR.
NOTA: Il volume di 0,1x TE utilizzato (50 μL) corrisponde al volume di ingresso del kit di preparazione della libreria di sequenziamento del DNA (vedere la tabella dei materiali) utilizzato in questo protocollo. Se si utilizza un kit o una strategia diversa, regolare il volume di conseguenza. - Eseguire le fasi di manipolazione del DNA del kit di preparazione della libreria di sequenziamento secondo il protocollo del produttore. Questi passaggi in genere includono l'ottundimento del DNA, l'A-tailing, la legatura dell'adattatore e l'escissione dell'U. L'ultimo passaggio (escissione U) è specifico per il kit utilizzato in questo studio. Se si utilizza quel kit, seguire le istruzioni del produttore dal punto 1 al punto 2.6 del protocollo del kit utilizzando adattatori non diluiti e non considerare la conservazione a -20 °C dopo il passaggio 2.6.
NOTA: Le modifiche del tampone richieste dai kit di sequenziamento devono essere sostituite legando le perle a un magnete e lavaggi (passaggi da 7.3 a 7.4), poiché il DNA è ancora legato alle perle di streptavidina. Inoltre, poiché il DNA è legato alle microsfere magnetiche, ignorare qualsiasi fase di purificazione e selezione delle dimensioni dopo la legatura dell'adattatore dal protocollo del kit. Continuare con il passaggio 7.6 (questo protocollo). - Al termine della legatura dell'adattatore, lavare il campione come descritto al punto 7.2. Scartare il surnatante e risospendere le perle in 20 μL di 0,1x TE.
8. Stima dei cicli di PCR richiesti
NOTA: Si consiglia di stimare i cicli di PCR necessari per l'amplificazione della libreria. Tipicamente, una libreria Micro-C richiede 8-15 cicli di PCR. Sebbene il passaggio non sia essenziale, aiuta a evitare l'amplificazione eccessiva e riduce il rischio di duplicati PCR.
- Per definire il numero minimo di cicli di PCR richiesti, eseguire la PCR con 1 μL di campione di streptavidina-biotina-DNA (fase 7.6). A tale scopo, aggiungere al campione la miscela master PCR (3,2 μL di H2O, 0,4 μL di primer i5, 0,4 μL di primer i7 e 5 μL di DNA polimerasi ad alta fedeltà Q5). Eseguire la PCR con 16 cicli secondo le istruzioni del produttore per il kit di preparazione della libreria di DNA utilizzato.
NOTA: I primer utilizzati qui vengono acquistati in un kit separato (vedere la Tabella dei materiali) che deve essere compatibile con il kit di preparazione della libreria utilizzato. - Dopo la PCR, raccogliere le perle con un magnete appropriato e misurare la concentrazione di DNA di 1 μL del surnatante utilizzando uno strumento di quantificazione del DNA ad alta sensibilità (vedi Tabella dei materiali). Per ottenere la concentrazione totale di DNA, moltiplicare questo valore per 10 (volume totale della reazione PCR). Stimare il numero richiesto di cicli di PCR come discusso nei risultati rappresentativi.
- Aggiungere 2 μL di tampone di caricamento 6x ai restanti 9 μL di miscela PCR. Risolvere la libreria amplificata PCR su un gel di agarosio all'1% per determinare il successo della legatura dell'adattatore, con una dimensione di circa 450 bp (Figura 1C).
NOTA: Il gel deve mostrare una banda distinta di circa 420 bp (libreria Micro-C più adattatori). Le bande di 120 bp rappresentano i dimeri dell'adattatore e sono indicative di librerie a bassa complessità. Possono comparire bande di peso molecolare inferiore (meno di 100 bp) e si tratta di primer inutilizzati della reazione PCR.
9. Amplificazione della libreria di sequenziamento
- Aggiungere la miscela master PCR (65 μL di H2O, 100 μL di DNA polimerasi ad alta fedeltà Q5, 8 μL di primer i5, 8 μL di primer i7) ai restanti 19 μL del campione di streptavidina-biotina-DNA. Dividere la miscela di reazione in aliquote da 50-100 μL.
NOTA: Il volume ottimale per la PCR dipende dalla macchina PCR; tipicamente, 50 μL offrono l'amplificazione più riproducibile nelle comuni macchine PCR. - Eseguire la PCR secondo le istruzioni del produttore del kit di preparazione della libreria di DNA con il numero di cicli determinato al punto 8. Se il passaggio 8 è stato omesso, si consigliano 14 cicli per la PCR.
- Purificare il DNA con microsfere paramagnetiche (vedi Tabella dei Materiali) in un rapporto di 1:0,9 secondo il protocollo del produttore. Eluire in 20 μL di TE allo 0,1%.
NOTA: Se al punto 8.5 vengono rilevati dimeri adattatori, eseguire la purificazione due volte utilizzando lo stesso rapporto. - Determinare la concentrazione di DNA ed eseguire i campioni su un sistema di controllo qualità (vedi Tabella dei materiali). Garantire una buona qualità della libreria Micro-C grazie alla presenza di un'unica banda precisa (Figura 1D).
NOTA: I dimeri adattatori sono rari nelle preparazioni di librerie su microsfere a causa dei lavaggi del campione prima della PCR. Pertanto, l'aspetto dei dimeri dell'adattatore indica una bassa complessità della libreria. Se si osservano dimeri adattatori, si consiglia vivamente di controllare la qualità del campione con un basso sequenziamento di ingresso.
10. Sequenziamento del DNA ed elaborazione dei dati
- Sequenziare la libreria Micro-C con il sequenziamento paired-end in base ai requisiti del provider di sequenziamento.
NOTA: Idealmente, i campioni vengono sequenziati su una piattaforma in modalità paired-end con 50 bp per lettura. Anche le piattaforme più vecchie che offrono lunghezze di lettura più brevi, come 2 x 35 bp, sono state utilizzate con successo. È importante sottolineare che, se si studiano regioni genomiche ripetitive, potrebbe essere consigliabile eseguire il sequenziamento con una lunghezza di lettura maggiore. - Per valutare la qualità della libreria Micro-C, eseguire un sequenziamento a basso input con 5 x 10da 6 a 1 x 107 letture per campione.
- Elabora i file di sequenziamento (file fastq) con Distiller10. Mappare le letture rispetto al genoma di riferimento appropriato, qui mm10.
NOTA: i file di sequenziazione possono essere elaborati utilizzando varie pipeline su computer locali o cluster di calcolo. Per i campioni con basse profondità di sequenziamento, le dimensioni dei contenitori più grandi, ad esempio 10.000 bp, 50.000 bp, 100.000 bp e 500.000 bp, possono ridurre le richieste di elaborazione e le dimensioni dei file. Distiller (utilizzato in questo studio) genera tutti i tipi di file necessari per valutare la qualità della libreria Micro-C. Il file *.stats generato contiene le informazioni sulla velocità della mappa, i rapporti cis-trans e l'orientamento di lettura stratificati in base alla distanza tra le coppie di lettura. Questi parametri sono visualizzati nella Figura 2 e la valutazione della qualità della libreria Micro-C è discussa nei risultati rappresentativi. Il software di elaborazione genera anche file mcool che possono essere caricati direttamente in HiGlass (https://docs.higlass.io/) per generare matrici di interazione9.
Risultati
Il successo della preparazione delle librerie Micro-C può essere valutato in diverse fasi del protocollo. Il passo più importante è la scelta di un corretto grado di digestione della MNasi. Pertanto, la concentrazione di MNasi deve essere titolata per produrre costantemente il 70%-90% di mononucleosomi rispetto ai dinucleosomi per ogni campione. È importante notare che la digestione della cromatina è diversa per l'eu- e l'eterocromatina, con MNase che digerisce l'eterocromatina in modo meno efficiente. Pertanto, il grado di digestione ottimale dipende dalla regione della cromatina di interesse e dal tipo di cellula studiata, poiché la proporzione relativa di eu- ed eterocromatina è specifica del tipo di cellula. Pertanto, si consiglia di titolare attentamente la concentrazione di MNasi richiesta e di valutare prima il successo dell'esperimento Micro-C mediante sequenziamento a basso input.
Nella Figura 1A è mostrato un tipico pattern di titolazione della MNasi della cromatina trattata con quantità decrescenti di MNasi. Qui, la cromatina di 250.000 cellule per reazione viene digerita con una diluizione quadrupla di MNasi. La concentrazione più alta (10 U di MNasi, Lane 2) mostra una cromatina iperdigerita costituita quasi esclusivamente da DNA mononucleosomiale (~150 bp). In particolare, il centro della banda mononucleosomiale è più basso nel gel di agarosio rispetto alle bande corrispondenti nei campioni con concentrazioni ridotte di MNasi, indicando una digestione eccessiva del DNA nucleosomiale. I nucleosomi sovradigeriti sono legati in modo inefficiente nella reazione di legatura di prossimità; pertanto, il campione in Lane 2 non è ottimale per gli esperimenti Micro-C. La corsia 3 (2,5 U di MNase) mostra un grado di digestione quasi appropriato per gli esperimenti Micro-C. In questo caso, la banda mononucleosomiale è la specie dominante e lo striscio subnucleosomiale, indicativo di nucleosomi iperdigeriti, è ridotto; tuttavia, è ancora presente. Il grado di digestione in corsia 4 (0,635 U di MNasi) è una condizione ideale per un esperimento Micro-C in questo esempio di titolazione. È presente una chiara banda mononucleosomiale senza DNA sub-nucleosomiale. L'intensità della banda per il DNA mono- e di-nucleosoma è quasi uguale, indicando una resa mono-nucleosomica del 66% o superiore. Vale la pena notare che il DNA di-nucleosomiale è circa il doppio della dimensione del DNA mono-nucleosomiale (~320 bp vs. ~150 bp), quindi la sua intensità di banda per mole di DNA è due volte più alta rispetto alla sua controparte mono-nucleosomiale. Il grado di digestione in Lane 5 (0,156 U di MNasi) mostra una cromatina sottodigerita con quasi nessun DNA nucleosomiale, e questo, quindi, rappresenta un campione non ottimale.
In conclusione, in questo esempio, la digestione di 2,5 x 105 cellule ES di topo con 0,625 U di MNasi (corrispondenti a 2,5 U di MNasi per 1 x 106 cellule in 200 μL) offre il punto di partenza più promettente per le digestioni preparative negli esperimenti Micro-C. Tuttavia, deve essere considerata anche una concentrazione intermedia di MNasi tra le condizioni utilizzate per i campioni nella corsia 3 e nella corsia 4 (corrispondente a 5 U di MNasi per 1 x 106 cellule in 200 μL). È importante sottolineare che la digestione della cromatina con MNasi non può essere scalata linearmente e non è consigliabile aumentare la digestione preparativa più di 4 volte. Per preparare librerie Micro-C da più di 1 x 10 6 cellule, si consiglia di digerire la cromatina in aliquote di 1 x 106 cellule e raggrupparle dopo l'inattivazione della MNasi.
Per valutare il successo del protocollo di legatura di prossimità, il controllo in ingresso, che è digerito con MNasi e non legato per prossimità (fase 3.8), deve essere confrontato con il campione legato alla prossimità (fase 5.3) mediante elettroforesi su gel di agarosio all'1,5% (Figura 1B). La banda dei mononucleosomi legati alla prossimità ha una dimensione approssimativa di 300 bp, simile a quella dei di-nucleosomi. Pertanto, il rapporto del segnale della banda mono- e di-nucleosomiale dovrebbe spostarsi da prevalentemente mono-nucleosomi (Lane 1) a di-nucleosomi (Lane 3 e Lane 4). Poiché il gel di agarosio in questa fase è il DNA di-nucleosomiale che viene asportato e purificato, è consigliabile suddividere i campioni in più corsie per evitare il sovraccarico.
Si raccomanda di valutare la qualità e la quantità della libreria di sequenziamento preparata mediante PCR minima. Qui, il DNA di 1 μL di perline (1/20 del campione totale) viene amplificato per 16 cicli in 10 μL di reazione PCR. La concentrazione totale della libreria di PCR minima varia in genere da 50 a 500 ng dopo 16 cicli di PCR. In teoria, ciò corrisponde a una libreria di 1-10 μg dal restante campione da 19 μL se fosse anche amplificato per 16 cicli. Si raccomanda di utilizzare il numero minimo di cicli di PCR necessari per generare una libreria di circa 100 ng dal DNA totale. Assumendo l'amplificazione logaritmica nella PCR, la concentrazione teorica del DNA ottenuta dall'ingresso di 19 μL a 16 cicli può essere divisa successivamente per due per calcolare il numero di cicli di PCR necessari per generare una libreria di 100 ng. Ad esempio, una resa di 100 ng da 1 μL dopo 16 cicli corrisponde a una resa di 1.900 ng amplificata da 19 μL. In questo scenario, 12 cicli dovrebbero idealmente generare una libreria di sequenziamento di 118 ng dal DNA totale (1.900 ng/[2 × 2 × 2 × 2] = 118 ng). Il restante campione di 9 μL della PCR minima può quindi essere utilizzato per valutare la qualità della libreria mediante elettroforesi su gel di agarosio (Figura 1C). La visualizzazione dovrebbe mostrare una banda distinta a 420 bp e nessuna banda per i dimeri dell'adattatore (120 bp). Possono comparire anche frammenti più piccoli, che corrispondono a primer PCR inutilizzati.
Successivamente, si raccomanda di analizzare e confermare il successo della preparazione del campione Micro-C mediante sequenziamento a basso input prima di impegnarsi in un sequenziamento profondo ad alta intensità di risorse. In genere, le librerie vengono sequenziate a una profondità di lettura da 5 x 106 a 1 x 107 e valutate in base ai seguenti criteri: il tasso di duplicazione della lettura del sequenziamento, il tasso di interazione cis rispetto a quello transcromosomico e la frequenza di orientamento della lettura del sequenziamento. Le librerie Micro-C vengono elaborate con Distiller, una pipeline a servizio completo che elabora i dati dal sequenziamento dei file di lettura (formato Fastq) ai file di coppie di lettura (formato Bedpe) e alle matrici di interazione scalabili (formati Cool e Mcool) utilizzando cooler, pairtools e cooltools10,11,12. La pipeline genera anche un file di riepilogo, ideale per valutare la qualità delle librerie Micro-C10 (https://github.com/open2c/distiller-nf). Il tasso di duplicazione della PCR fornisce informazioni sulla complessità della libreria di sequenziamento e può essere estratto dal file *.stats generato. Le librerie Micro-C di alta qualità hanno tassi di duplicazione PCR inferiori al 5%-10% se generate da 5 milioni o più di cellule. In particolare, alcune piattaforme di sequenziamento generano duplicati PCR durante la formazione del cluster indipendentemente dalla complessità della libreria di sequenziamento. La Figura 2A mostra i tassi di duplicazione relativi di due esperimenti: uno che consideriamo un campione buono e un campione cattivo. In questo esempio, entrambi i campioni hanno visualizzato frequenze di mappa accettabili. I criteri successivi per valutare la qualità delle librerie Micro-C sono il rapporto cis rispetto a trans e le frequenze di orientamento di lettura. All'interno del nucleo, i cromosomi abitano i singoli territori cromosomici e, quindi, raramente interagiscono con altri cromosomi. Un alto tasso di interazioni transcromosomiche rilevate indica un alto tasso di legature casuali. Va notato che a questo livello di analisi, il campione difettoso ha mostrato un alto tasso di interazioni transcromosomiche rispetto al campione buono (Figura 2B). Per Micro-C, è auspicabile un tasso di interazione cis-cromosomico pari o superiore al 70%.
Una libreria Micro-C ha una dimensione del frammento simile alla banda del DNA di-nucleosomiale, che può co-purificarsi con il campione legato di prossimità e contaminare l'esperimento. Questi contaminanti sono sempre interazioni cis-cromosomiche. Pertanto, è importante valutare anche i tassi di orientamento alla lettura. Il tasso di contaminazione di-nucleosomiale può essere stimato mediante sequenziamento a basso input. Il DNA di-nucleosomiale deriva da due nucleosomi vicini che non sono stati scissi dalla MNasi. Pertanto, le letture di sequenziamento risultanti mostreranno sempre un orientamento di lettura avanti-indietro (F e R) e la distanza tra le coppie di lettura sarà di circa 320 bp. I frammenti legati alla prossimità, in confronto, possono essere legati in quattro orientamenti, producendo coppie di lettura con F-R, R-R, R-F e F-F, idealmente con uguale abbondanza (Figura 2C). Inoltre, visualizzano varie distanze tra le due coppie di lettura. Per stimare la quantità di contaminanti di-nucleosomiali, la frequenza degli orientamenti di lettura può essere calcolata dai file *stats generati dal distillatore (Figura 2D). In particolare, in questo lavoro, la frazione di letture F-R (rosso) era più alta nel campione difettoso rispetto al campione buono, e questo è diventato più evidente quando gli orientamenti di lettura sono stati stratificati in base alla distanza (Figura 2E). La frazione F-R è dominata da frammenti di-nucleosomiali rispetto alle librerie Micro-C quando le coppie di lettura sono stratificate in letture con distanze <562 bp o ≥562 bp. In questo caso, la frazione di letture con distanza <562 bp è dominata da letture F-R, mentre la frazione con distanze ≥562 bp mostra una distribuzione uniforme tra i quattro possibili orientamenti, indicando che la sovrarappresentazione globale delle letture F-R deriva da contaminanti di-nucleosomiali. La scelta di 562 bp come soglia per il sottoinsieme è definita dal binning nel file *stats generato. Sebbene non sia necessario per questo controllo di qualità, è possibile ottenere un sottoinsieme più definito estraendo le distanze dal file *pairs, anch'esso generato dal distillatore. È importante notare che le letture di-nucleosomiali non riducono la qualità del campione Micro-C in quanto possono essere identificate e ignorate durante l'elaborazione dei dati. Tuttavia, non contengono informazioni preziose sulle interazioni 3D e diluiscono le letture informative.
Pertanto, un'attenta titolazione della MNase e un accurato controllo di qualità con sequenziamento a basso input sono gli strumenti migliori per ottimizzare la qualità degli esperimenti Micro-C.
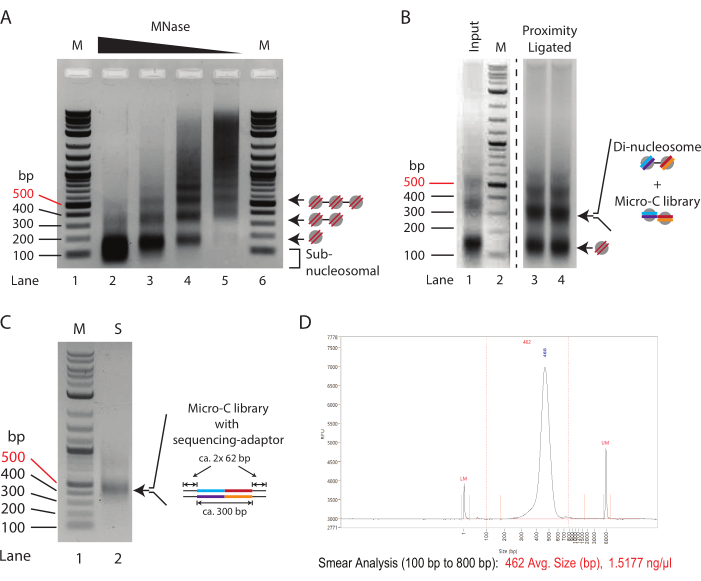
Figura 1: Fasi intermedie del protocollo Micro-C . (A) Elettroforesi su gel di agarosio della cromatina da 2,5 x 105 cellule ES di topo digerite con concentrazioni variabili di MNasi. Le bande mono-, di- e tri-nucleosomiali sono indicate da frecce. M: Scala del DNA (Corsia 1/6); 10 U di MNasi per 250.000 cellule (Corsia 2); 2,5 U di MNasi per 250.000 cellule (Corsia 3); 0,625 U di MNasi per 250.000 cellule (corsia 4); 0,156 U di MNasi per 250.000 cellule (corsia 2). (B) L'elettroforesi su gel di agarosio all'1,0% dei campioni preparati con Micro-C (Corsia 3 e Corsia 4) e il controllo dell'ingresso digerito MNase (Corsia 1). La corsia 1 e la corsia 2 (M: scala del DNA) sono migliorate per enfatizzare il cambiamento relativo nell'intensità del frammento da mono a dinucleosomiale. Le bande mono- e di-nucleosomiali sono indicate da frecce. La banda di-nucleosomiale nel campione legato alla prossimità combina il DNA di-nucleosomiale e il DNA della libreria Micro-C. (C) L'elettroforesi su gel di agarosio all'1,0% delle librerie di sequenziamento Micro-C amplificata da un campione da 1 μL per valutarne la qualità. Corsia 1 (M): scala del DNA; Corsia 2 (S): Biblioteca Mirco-C. (D) Traccia Fragment Analyzer della libreria Micro-C finale. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 2: Statistiche di esempio per il sequenziamento a basso input di un campione buono e di un campione cattivo. (A) Grafico a barre delle letture percentuali mappate (verde) e non mappate (rosso). (B) Frazione normalizzata delle letture che mappano le interazioni cis e trans-cromosomiche. I set di dati sono stati normalizzati alla lettura cis-mapping. Le letture di mappatura cis sono state stratificate in base alla distanza tra la prima e la seconda lettura dei campioni sequenziati all'estremità accoppiata: ≤1 kbp (giallo), >1 kbp e ≤10 kbp (arancione) e >10 kbp (rosso). (C) Schema delle potenziali specie molecolari con le dimensioni di-nucleosomiali. (D) Percentuali di orientamenti delle coppie di lettura di tutte le letture del campione valido e del campione cattivo. (E) Come il pannello (D) ma stratificato per distanze (sinistra, <562 bp e destra, ≥562 bp). Fare clic qui per visualizzare una versione più grande di questa figura.
| Componenti | 1x | 4,4 volte |
| 10x NEBuffer 2.1, | 10 μL | 44 μL |
| 2 μL 100 mM ATP | 2 μL | 8,8 μL |
| 100 mM DTT | 5 μL | 22 μL |
| H2O | 68 μL | 299,2 μL |
| 10 U/μL T4 PNK | 5 μL | 22 μL |
| Totale | 90 μL | 396 μL |
Tabella 1: Miscela master Micro-C 1. Composizione della miscela master per la reazione di fine masticazione.
| Componenti | 1x | 4,4 volte |
| 1 mM di Biotina-dATP | 10 μL | 44 μL |
| 1 mM di Biotina-dCTP | 10 μL | 44 μL |
| Miscela di 10 mM di dTTP e dGTP | 1 μL | 4,4 μL |
| 10x tampone DNA ligasi T4 | 5 μL | 22 μL |
| 200x BSA | 0,25 μL | 1,1 μL |
| H2O | 23,75 μL | 104,5 μL |
Tabella 2: Miscela master Micro-C 2. Composizione della miscela master per la reazione di etichettatura finale.
| Componenti | 1x | 4,4 volte |
| 10x Tampone di reazione della ligasi NEB T4 | 50 μL | 220 μL |
| H2O | 422,5 μL | 1859 μL |
| T4 DNA ligasi | 25 μL | 110μL |
Tabella 3: Miscela master Micro-C 3. Composizione della miscela master per la reazione di legatura di prossimità.
| Componenti | 1x | 4,4 volte |
| 10x NEBuffer 1.1 | 20 μL | 88 μL |
| H2O | 180 μL | 792 μL |
| Nucleasi ExoIII | 10 μL | 44 μL |
Tabella 4: Miscela master Micro-C 4. Composizione della miscela master per la reazione di rimozione della biotina.
Discussione
Il successo di un esperimento Micro-C dipende da alcuni passaggi critici del protocollo che devono essere eseguiti con attenzione. Innanzitutto, la reticolazione con il reticolante aggiuntivo DSG o EGS può portare all'aggregazione di cellule, a seconda del tipo di cellula. L'aggiunta dello 0,1%-0,5% di BSA alla reazione di reticolazione riduce significativamente l'aggregazione senza influire sull'efficienza della reticolazione. Una reticolazione inefficiente può comportare un aumento dei tassi di interazioni transcromosomiche che sono indicative di legature casuali. Il secondo, ma più cruciale, passo di questo protocollo è la digestione della cromatina con MNasi. Una digestione non ottimale della cromatina porta a un'inefficiente legatura di prossimità (sovradigestione) o a un aumento dei tassi di dinucleosomi non legati alla prossimità (sottodigestione). L'efficienza della reazione di legatura può essere valutata mediante elettroforesi su gel di agarosio (Figura 1B) ed è inoltre stimata al meglio mediante sequenziamento a basso input. Se il sequenziamento a basso input rivela un alto tasso di duplicazione (legatura inefficiente) o un aumento dei tassi di dinucleosomi, il grado di digestione della MNasi deve essere rivalutato. In particolare, la perdita di campione durante l'esecuzione del protocollo può portare a una riduzione della complessità della libreria. La concentrazione di un campione viene valutata al meglio dopo la purificazione del DNA (fase 5.3) o mediante PCR minima (fase 8). La resa totale di DNA da 5 x 106 cellule di mammifero dopo la purificazione del DNA è tipicamente di >2 μg. La concentrazione di DNA deve essere controllata dopo la digestione MNasi, la digestione ExoIII e la purificazione del DNA. Le nucleasi endogene, la cui abbondanza è specifica per il tipo di cellula e la specie, possono essere una fonte di degradazione del DNA. Inoltre, la purificazione del DNA basata su colonna può portare alla perdita di campione a causa dell'incompatibilità con la SDS dovuta alle reazioni di deproteinazione. La precipitazione dell'etanolo può essere presa in considerazione se la concentrazione di DNA è bassa in questa fase.
Poiché Micro-C richiede una titolazione MNase specifica per il campione, è difficile applicare Micro-C a piccole popolazioni cellulari, ad esempio con piccoli organi di vari organismi modello, embrioni e singole cellule, organoidi o biopsie di pazienti. In questo caso, Hi-C 3.0 offre un'alternativa consolidata utilizzando una reazione endpoint mediante endonucleasi di restrizione sequenza-specifica 8,9.
Micro-C è una tecnologia di conformazione cromosomica ad alta risoluzione ampiamente applicabile con un'elevata gamma dinamica e un basso rapporto segnale/rumore, che la rende particolarmente adatta per lo studio delle caratteristiche cromosomiche a corto raggio 4,5,8, come le anse cromosomiche. La risoluzione di Micro-C consente di catturare i loop promotore-enhancer, che sono oltre il limite di rilevamento di Hi-C, consentendo in modo efficiente un'analisi più dettagliata della relazione tra l'organizzazione del genoma e la regolazione13,14,15. Inoltre, le strategie di cattura del DNA sono state recentemente combinate con Micro-C per aumentare la risoluzione locus-specifica dei loci genomici mirati a livelli senza precedenti, rivelando nuove intuizioni sull'ultrastruttura del genoma 3D16,17,18. In sintesi, prevediamo che Micro-C e i suoi derivati saranno una tecnologia chiave per sezionare il ruolo del genoma 3D nella regolazione trascrizionale e, di conseguenza, nel differenziamento e nel mantenimento del tipo cellulare.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Ringraziamo Christl Gaubitz e Kathleen Stewart-Morgan per la lettura critica del manoscritto. Ringraziamo Anja Groth e il laboratorio Groth per il loro supporto nella creazione del nostro laboratorio. Ringraziamo per il supporto lo staff della piattaforma CPR/reNEW Genomics: H. Wollmann, M. Michaut e A. Kalvisa. Il Centro per la Medicina delle Cellule Staminali della Fondazione Novo Nordisk (reNEW) è supportato dalla sovvenzione della Fondazione Novo Nordisk numero NNF21CC0073729. Il Centro per la ricerca sulle proteine (CPR) della Fondazione Novo Nordisk è supportato dalla sovvenzione numero NNF14CC0001 della Fondazione Novo Nordisk. Ringraziamo il laboratorio Brickman del Novo Nordisk Center for Stem Cell Medicine, reNEW Copenhagen, per le cellule ES di topo.
Materiali
| Name | Company | Catalog Number | Comments |
| 1 mM Biotin dATP | Jenna Bioscience | NU-835-Bio14-S | |
| 1 mM Biotin dCTP | Jenna Bioscience | NU-809-BioX-S | |
| 10 mM dGTP | NEB | N0442S | |
| 10 mM dTTP | NEB | N0443S | |
| 10 U/ml T4 PNK | NEB | M0201L | |
| 100 U/L Exonuclease III | NEB | M0206L | |
| 10x NEBuffer 1.1 | NEB | B7001S | |
| 10x NEBuffer 2.1 | NEB | B7202S | |
| 10x T4 DNA Ligase buffer | NEB | B0202A | |
| 1x DPBS w/o Mg2+ and Ca2+ | ThermoFisher | 14190144 | |
| 1x LIF | |||
| 2_Mercaptoethanol 50 mM | Gibco | 31350010 | 0.1 mM b-mercaptoethanol |
| 37% Formaldehyde | Sigma Aldrich | 252549-500ML | Caution. See manufactures MSDS |
| 400 U/ml T4 DNA Ligase | NEB | M0202L | |
| 5 U/ml Klenow Fragment | NEB | M0210L | |
| Agarose | BIO-RAD | 1613102 | Caution. See manufactures MSDS |
| BSA 20mg/ml | NEB | B9000S | |
| CaCl2 | |||
| cell counter | |||
| Dimethyl Sulfoxide (DMSO) | Sigma Aldrich | D8418-100ML | Caution. See manufactures MSDS |
| Dynabeads MyOne Streptavidin C1 | Invitrogen | 65001 | |
| DynaMag-2 Magnet | Invitrogen | 12321D | refered to as: magnet magnet for 1.5 ml tubes |
| DynaMag-PCR Magnet | Invitrogen | 492025 | refered to as: magnet magnet for PCR tubes |
| EDTA Ultrapure 0.5M pH 8.0 | Invitrogen | 15575-038 | |
| EGTA Ultrapure 0.5M pH 8.0 | BioWorld | 40121266-1 | |
| Ethanol 96% | VWR Chemicals | 20824365 | quality control system |
| Ethidium Bromide | Invitrogen | 15585-011 | |
| Ethylene glycol bis(succinimidyl succinate) (EGS) | ThermoFisher | 21565 | |
| Fetl Bovin Serum | Sigma Aldrich | F7524 | 15% FBS |
| Gel Loading dye purple (6X) | NEB | B7024S | |
| Glycine | PanReac AppliChem | A1067.0500 | |
| Halt Proteinase inhibitor (100x) | ThermoFisher | 78430 | Caution. See manufactures MSDS |
| IGEPAL CA-630 (NP-40) | Sigma Aldrich | 18896-50ML | |
| MgCl 1 M | Invitrogen | AM9530G | |
| Micrococcal Nuclease (MNase) | Worthington | LS004798 | |
| mouse embryonic stem cells | |||
| NaCl | Sigma Aldrich | S9888-1KG | |
| NEBNext Multiplex Oligos for Illumina (Dual Index primers) | NEB | E7600S | amplification primers for sequencing libraries |
| NEBNext Ultra II DNA library prep kit for Illumina | NEB | E7645L | sequencing library preparation kit |
| NEBNext Ultra II Q5 Master mix | NEB | M0544S | Caution. See manufactures MSDS |
| Non-Essential Amino Acids Solution | Gibco | 11140050 | 1x NEAA |
| Penicillin-Streptomycin (10,000 U/mL) | Gibco | 15140148 | 1% Pen-Strep |
| Proteinase K (40 mg/ml) | GoldBio | P-480-1 | Caution. See manufactures MSDS |
| QIAquick Gel extraction kit | QIAgen | 28706 | refered to as: DNA gel elution kit |
| QIAquick PCR purification kit | QIAgen | 28106 | refered to as: commercial DNA purification kit |
| Qubit dsDNA HS Assay kit | Invitrogen | Q32854 | high sensitivity DNA quantification instrument |
| Quick load purple 1kb plus DNA Ladder | NEB | N0550S | |
| SPRIselect size selection beads | Beckman Coulter | B23319 | paramagnetic beads |
| ThermoMixer C | Eppendorf | 5382000015 | refered to as: thermomixer |
| Tris | Merck | 10708976001 | |
| Trypsin | |||
| Tween20 | Sigma Aldrich | P7949-100ML | |
| Ultrapure 10% SDS | Invitrogen | 15553-035 | |
| Ultrapure Phenol Chloroform Isoamyl Alcohol (PCI) | Invitrogen | 15593-031 | |
| Fragment Analyzer |
Riferimenti
- Lieberman-Aiden, E., et al. Comprehensive mapping of long-range interactions reveals folding principles of the human genome. Science. 326 (5950), 289-293 (2009).
- Hsieh, T. -H. S., Fudenberg, G., Goloborodko, A., Rando, O. J. Micro-C XL: Assaying chromosome conformation from the nucleosome to the entire genome. Nature Methods. 13 (12), 1009-1011 (2016).
- Hsieh, T. -H. S., et al. Mapping nucleosome resolution chromosome folding in yeast by Micro-C. Cell. 162 (1), 108-119 (2015).
- Krietenstein, N., et al. Ultrastructural details of mammalian chromosome architecture. Molecular Cell. 78 (3), 554-565 (2020).
- Hsieh, T. -H. S., et al. Resolving the 3D landscape of transcription-linked mammalian chromatin folding. Molecular Cell. 78 (3), 539-553 (2020).
- Belaghzal, H., Dekker, J., Gibcus, J. H. Hi-C 2.0: An optimized Hi-C procedure for high-resolution genome-wide mapping of chromosome conformation. Methods. 123, 56-65 (2017).
- van Holde, K. E. Chromatin. , Springer. New York, NY. (1989).
- Oksuz, B. A. Systematic evaluation of chromosome conformation capture assays. Nature Methods. 18, 1046-1055 (2021).
- Lafontaine, D. L., Yang, L., Dekker, J., Gibcus, J. H. Hi-C 3.0: Improved protocol for genome-wide chromosome conformation capture. Current Protocols. 1 (7), 198(2021).
- Goloborodko, A., Venev, S., Abdennur, N., Di Tommaso, P. Mirnylab/distiller-nf; v033. Zenodo. , (2019).
- Venev, S., et al. Open2c/cooltools: v0.4.1. Zenodo. , (2021).
- Abdennur, N., Mirny, L. A. Cooler: Scalable storage for Hi-C data and other genomically labeled arrays. Bioinformatics. 36 (1), 311-316 (2019).
- Zhang, S., Übelmesser, N., Barbieri, M., Papantonis, A. Enhancer-promoter contact formation requires RNAPII and antagonizes loop extrusion. BioRxiv. , (2022).
- Barshad, G., et al. RNA polymerase II and PARP1 shape enhancer-promoter contacts. BioRxiv. , (2022).
- Hansen, A. S., et al. Distinct classes of chromatin loops revealed by deletion of an RNA-binding region in CTCF. Molecular Cell. 76 (3), 395-411 (2019).
- Hua, P., et al. Defining genome architecture at base-pair resolution. Nature. 595 (7865), 125-129 (2021).
- Downes, D. J. High-resolution targeted 3C interrogation of cis-regulatory element organization at genome-wide scale. Nature Communications. 12, 531(2021).
- Goel, V. Y., Huseyin, M. K., Hansen, A. S. Region capture Micro-C reveals coalescence of enhancers and promoters into nested microcompartments. BioRxiv. , (2022).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon