Method Article
Medição direta do KDM1A Target Engagement usando imunoensaios baseados em Quimiosonda
Neste Artigo
Resumo
Aqui, apresentamos um protocolo para medir KDM1A engajamento alvo em uma célula humana ou animal, tecido ou amostras de sangue tratadas com inibidores da KDM1A. O protocolo emprega a marcação do chemoprobe da enzima livre de KDM1A e a quantificação direta da ocupação do alvo usando immunoensaios quimioprobe-baseados e pode ser usada em estudos pré-clínicos e clínicos.
Resumo
A avaliação do engajamento alvo, definida como a interação de um fármaco com a proteína para a qual foi projetada, é um requisito básico para a interpretação da atividade biológica de qualquer composto no desenvolvimento de fármacos ou em projetos de pesquisa básica. No Epigenetics, o acoplamento do alvo é avaliado o mais frequentemente pela análise de marcadores do proxy em vez de medir a União do composto ao alvo. Os leituras biológicos a jusante que foram analisados incluem a modulação da marca do histona ou as mudanças da expressão de Gene. KDM1A é uma desmetililase de lisina que remove grupos metilo de mono e H3K4 dimetilizados, uma modificação associada ao silenciamento da expressão gênica. A modulação dos marcadores do proxy é dependente do tipo e da função da pilha da make-up genética das pilhas investigadas, que pode fazer a interpretação e a comparação do Cruz-caso completamente difícil. Para contornar estes problemas, um protocolo versátil é apresentado para avaliar os efeitos da dose e a dinâmica do acoplamento direto do alvo de KDM1A. O ensaio descrito faz uso de uma quimiosonda KDM1A para capturar e quantificar a enzima desinibida, pode ser amplamente aplicada a células ou amostras de tecido sem a necessidade de modificação genética, tem uma excelente janela de detecção, e pode ser usado tanto para pesquisa básica e análise de amostras clínicas.
Introdução
O demetilase específico 1 de lysine (KDM1A)1 é um demetilase envolvido no controle da transcrição do gene. Esta proteína emergiu como um alvo farmacológico candidato2 em Oncologia; incluindo leucemia mielóide aguda3 (AML), síndrome de mielodisplasia (MDS)4, mielofibrose (MF)5,6, câncer de pulmão de pequenas células (SCLC)7; em doença falciforme (SCD)8,9, e em doenças do sistema nervoso central, incluindo a doença de Alzheimer (AD), esclerose múltipla (MS); e na agressão10.
A maioria dos compostos inibidores da KDM1A no desenvolvimento clínico são derivados da ciclopropilamina e inibem a proteína por ligação covalente ao seu cofator11de flavina-dinucleotídeo (FAD). A inibição da KDM1A induz alterações na expressão gênica, mas essas alterações variam enormemente nos tecidos, tipos de células ou casos de doença. A inibição da KDM1A também altera as marcas de histona12, mas essas mudanças são geralmente produzidas localmente em um local específico no genoma, e são novamente, altamente tecido e células específicas.
O protocolo foi desenvolvido para medir diretamente o engajamento alvo de KDM1A em amostras biológicas e foi otimizado para o uso com inibidores derivados da ciclopropilamina. O ensaio é baseado na tecnologia ELISA e analisa, em paralelo, total e livre (ou seja, não acoplado por inibidor) KDM1A em um extrato de proteína nativa de uma amostra biológica em um ensaio de fase sólida. Como primeiro passo, a amostra biológica é lisada na presença da quimiosonda seletiva biotinilada KDM1A og-88113,14, derivada do inibidor seletivo KDM1A Ory-1001 (iadademstat), um potente inibidor da KDM1A em clínica desenvolvimento para o tratamento da doença oncológica. O chemoprobe tem um IC50 para KDM1A de 120 nanômetro e inclui um metade obrigatório do FAD lig a um polietileno biotinylated glicol (PEG)-cauda. A quimiosonda liga-se exclusivamente à KDM1A livre, mas não à KDM1A associada ao inibidor na amostra. Após a ligação do chemoprobe, os KDM1A que contêm complexos na amostra são capturados em placas do de microtitulação com a superfície revestida estreptavidina para determinar o KDM1A livre, ou em placas revestidas com um anticorpo KDM1A monoclonal da captação para determinar o KDM1A total. Após a lavagem, ambas as placas são incubadas com um anticorpo de detecção KDM1A, lavadas novamente, e incubadas com um anticorpo de IgG anticoelho conjugado HRP secundário para detecção usando um substrato luminescente e quantificação por medição relativa de luz (RLU) num luminômetro (Figura 1).
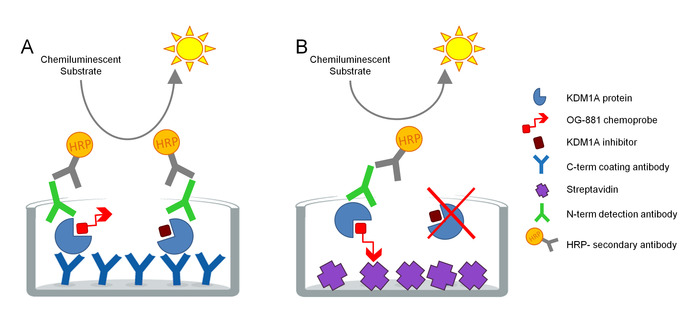
Figura 1. Esquema de ELISA enzima lig o ensaio immunoabsorvente do chemoprobe para o acoplamento do alvo KDM1A: A) determinação do total KDM1A usando o sanduíche ELISA e B) determinação do KDM1A livre usando o quimioprobe Elisa. Por favor clique aqui para ver uma versão maior desta figura.
Uma curva padrão é incluída em ambas as placas ELISA para verificar a linearidade de cada ensaio. A determinação do engajamento alvo de KDM1A em cada amostra é então calculada como um valor relativo para a amostra pré-dose ou tratada pelo veículo.
Protocolo
Amostras de sangue foram obtidas do Instituto de Investigación biomédica Sant Pau biobank de acordo com a legislação espanhola (Real Decreto de biobancos 1716/2011) e aprovação dos comitês de ética locais. Estudos com tecidos animais foram realizados de acordo com as diretrizes institucionais para o cuidado e uso de animais de laboratório (diretiva do Conselho das Comunidades Europeias 86/609/CEE), instituído pelo Comitê de ética para experimentação animal no PRAAL-PCB.
1. preparação de amostras biológicas para o ensaio.
PRECAUÇÃO: Este protocolo envolve a manipulação de amostras biológicas que podem ser submetidas à norma de patógenos de sangue (29 CFR 1910,1030), da administração de segurança e saúde ocupacional (OSHA), da Directiva 2000/54/CE do Parlamento Europeu e do Conselho de 18 de setembro de 2000 ou regulamentos equivalentes. Além disso, as amostras biológicas podem conter traços de compostos químicos experimentais biologicamente ativos e o protocolo pode envolver uma maior manipulação desses compostos. Reveja a ficha de dados de segurança (SDS) dos compostos utilizados antes do início do experimento e respeite rigorosamente todas as medidas de segurança aplicáveis estabelecidas no centro de investigação, incluindo a utilização de equipamento de protecção individual (EPI) adequado. Use vestuário de proteção adequado e usar blindagem adequada durante o curso do experimento. Descarte resíduos nos recipientes de resíduos apropriados (resíduos biológicos/citotóxicos).
Nota: Este protocolo começa com células ou amostras de indivíduos tratados com um inibidor da KDM1A e os seus controlos não tratados ou de veículo/placebo3.
- Células tratadas com o inibidor do veículo ou KDM1A in vitro
- Para as células cultivadas em suspensão, como culturas de 10 mL, transfira as suspensões em tubos cônicos limpos de 15 mL e prossiga para 1.1.3.
- Para as células aderentes (cultivadas em 75 cm2 frascos), retire o meio do balão e lave brevemente usando 4 ml de PBS. Retire as células de seus vasos usando 1,5 mL de 0,5% Trypsin-EDTA durante 2-5 min (as condições de trypsinization podem variar, siga as recomendações do provedor para a linha celular), adicione 4 mL de PBS e transfira as células em tubos cônicos limpos de 15 mL.
- Insira os tubos em uma centrífuga de bancada e colete as células por centrifugação por 5 min a 400 x g a 4 ° c. Retire o sobrenadante, ressuscitou a pelota em 1 mL de PBS dispensado usando uma micropipeta e transfira a suspensão em um tubo de microcentrífuga de 1,5 mL.
- Insira as amostras numa centrífuga de microtubos e Centrifugue-as durante 5 min a 400 x g a 4 ° c. Retire a PBS por aspiração com uma micropipeta e mantenha as pelotas no gelo e prossiga para a etapa 2; ou congelar as pelotas em gelo seco e armazená-los em-80 ° c até a etapa 3.
- Amostras de indivíduos ou animais tratados com um inibidor do veículo/placebo ou KDM1A
- Tecidos: corte o tecido em pequeno, ≈ 1 cm3 peças usando um bisturi. Congele as partes dos tecidos no líquido N2 em um recipiente do Dewar e armazene-os em-80 ° c até a etapa 3.
- Células mononucleares do sangue polymorphic (PBMCs): diluir 10 mL de sangue fresco (processo máximo 2 h após a retirada de sangue) coletado em tubos de K2-EDTA com 2 volumes de PBS em um tubo cônico de 50 ml. Isole os PBMCs do sangue usando tubos de separação de PBMC comercialmente obtidos de acordo com as instruções do fabricante. Mantenha as pelotas no gelo e prossiga para o passo 3,2; ou congelar as pelotas em gelo seco e armazená-los em-80 ° c até a etapa 3.
Nota: um pellet de células molhadas de 20 a 50 μL contém ≈ 1 x 107 células, dependendo do tamanho da célula. Um pellet de PBMC molhado obtido a partir de 10 mL de sangue humano saudável tem um volume de ≈ 20 μL e contém ≈ 1 x 107 PBMCs. os tecidos ou as pelotas da pilha podem ser armazenados em-80 º c por até 6 meses.
2. preparação da solução
- Prepare 2 μM OG-881 solução de trabalho: Tome uma alíquota de uso único de 10 μL da solução de estoque de sonda biotinilada de 20 mM OG-881 para fora do refrigerador de 4 ° c e deixe-a à temperatura ambiente (RT) por 10 min. Prepare a solução de trabalho de 2 μM por diluição serial do solut de estoque OG-881 íon em PBS, usando uma micropipeta com pontas de filtro e mudando a ponta entre as etapas diferentes da diluição.
- Prepare o inibidor da protease 10x: dissolva 1 pastilha em 1 mL de PBS em um tubo de microcentrífuga.
- Prepare o volume desejado de 1x tampão de lise celular com 25 nM OG-881 quimiosonda. Para cada mL, misturar 100 μL comercialmente obteve 10x tampão de lise celular, 150 μL de inibidor de protease 10x, 12,5 μL de 2 μM OG-881 e 737,5 μL de água destilada dupla tipo 1.
- Opcionalmente, prepare o volume desejado de 1x tampão de lise celular, mas com 25 nM ORY-1001 em vez de OG-881 como na etapa 2,3. Podem ser utilizados inibidores menos potentes, mas podem necessitar de concentrações mais elevadas, para utilização no controlo positivo com inibição de 100% (ver passo 3,5).
Nota: Tome as medidas apropriadas para evitar qualquer contaminação não intencional de soluções ou amostras com as soluções de ações do inibidor OG-881 ou KDM1A. Para calcular o volume desejado de 1x tampão de lise celular com 25 nM OG-881, suponha que 400 μL é exigido por 40 mg de tecido pulverizado, ou 200 μL por pellet molhado de 107 células.
3. extração de proteínas nativas
- Dos tecidos:
- Pulverize e Homogeneize um cubo de ≈ 1 cm3 do tecido congelado com um almofariz e um pilão refrigerados no gelo seco. Aliquot as amostras em frascos de uso único contendo ≈ 40 mg de pó de tecido, evite descongelar em todos os momentos. Prossiga para o passo 3.1.2. para processamento imediato ou armazenar a-80 ° c.
- Ressuscitem 40 mg de tecido pulverizado em 400 μL de tampão de lise celular 1x com 25 nM OG-881, vortex por 10 s, e force a amostra pelo menos cinco vezes através de uma agulha de seringa sem corte de 18 gauge até que a lise do tecido seja alcançada e uma luz turva amarela à suspensão laranja seja Obtido. Evite a formação de bolhas.
- Continue para a etapa 3,3
- A partir de pelotas de células (PBMCs e linhas celulares):
- Ressuscitem um pellet de ≈ 1 x 107 células em 200 μL de tampão de lise de células 1x contendo 25 nm OG-881. Vórtice as amostras brevemente e mantê-los no gelo por 5 min.
- SONICATE as amostras em um sonicador usando 3 pulsos de 20 s cada em 45 kHz; Coloque-os no gelo por 20 s entre pulsos.
Nota: assim que as amostras biológicas tiverem sido ressuscipended no tampão de lise celular 1x, mantenha-os no gelo durante o resto do processo.
- Mantenha as amostras no gelo por um adicional de 5 min, vórtice brevemente e Centrifugue as amostras por 10 min a 14 000 x g em uma centrífuga pré-resfriada a 4 ° c.
- Usando uma micropipeta de 1 mL, transfira os sobrenadantes em tubos de microcentrífuga 1,5 mL frescos e deixe-os no gelo durante 2 h. Avance para o passo 4.
- Opcionalmente, um controle positivo para simular 100% de engajamento de destino pode ser preparado da seguinte maneira:
- Ressuscitem a pelota da pilha ou o tecido pulverizado de um veículo ou uma amostra não tratada (da pré-dose) no volume exigido do amortecedor da lise da pilha 1x com 25 nanômetro ORY-1001 e processo como esboçado em etapa 3,1 a 3,3.
- Transfira os sobrenadantes do controle positivo em tubos de microcentrífuga de 1,5 mL frescos e deixe-os no gelo para 1 h. ORY-1001 inibem deliberadamente KDM1A e ligação do chemoprobe do bloco.
- Adicionar 5 μL de solução de trabalho de 2 μM OG-881 ao sobrenadante de controlo positivo (volume para controlo positivo gerado a partir de uma amostra de tecido de 40 mg) ou 2,5 μL de solução de trabalho de 2 μM OG-881 (volume para controlo positivo gerado a partir de uma amostra de 107células) para obter a mesma concentração OG-881 que as outras amostras e deixar no gelo durante 2 h. continue para o passo 4.
4. quantificação da proteína nativa utilizando o ensaio de Bradford
- Diluir o reagente de ensaio de proteína Bradford comercialmente adquirido 5 vezes com H2O tipo 1 água destilada dupla. Calcule o volume do reagente necessário para a quantidade total de amostras e normas (1 mL por amostra ou padrão + 5 mL de volume em excesso).
- Para a curva padrão da albumina de soro bovino (BSA), prepare um tubo do microcentrifugador com 1 mL diluída solução do ensaio da proteína de Bradford (em branco) e sete tubos do microcentrifugador com 995 μL da solução diluída do ensaio da proteína de Bradford. Adicione 5 μL de cada um dos padrões de BSA (concentração variando de 125 a 2.000 μg/mL) a cada um dos 7 tubos de microcentrífuga e misture-os suavemente invertendo os tubos várias vezes. Incubar por 5 min em RT.
- Transfira as normas diluídas para os cubos e leia o OD das amostras em branco e a albumina sérica bovina padrão em um espectrofotômetro a 280 nm.
- Para as amostras biológicas, prepare um tubo de microcentrífuga com 1 mL diluída solução de ensaio de proteína Bradford (em branco) e como muitos tubos de microcentrífuga com 999 μL do reagente de ensaio de proteína Bradford diluído como amostras que precisam ser quantificadas. Usando uma micropipeta P2 automática, adicione 1 μL de extrato de proteína nativa preparado na etapa 3 para cada tubo de microcentrífuga e misture suavemente invertendo os tubos várias vezes. Incubar as amostras 5 min em RT.
- Transfira os volumes para os cubos e leia o OD das amostras em um espectrofotômetro a 280 nm.
- Preferencialmente, prossiga imediatamente para a etapa 5. Alternativamente, armazene os extratos nativos da proteína em-80 ° c até a etapa 5. Evite congelar ciclos degelo.
5. luminescente ELISAs para total e livre KDM1A determinação
Nota: Mantenha a temperatura do laboratório constante a 23-24 ° c (RT).
- Revestimento de placas do de microtitulação com anticorpo da captação KDM1A ou streptavidin
- Total KDM1A ELISA: para cada placa, prepare 10 mL de anticorpo de captura KDM1A para uma concentração final de 2 μg/mL em PBS. Transfira 100 μL para cada poço da chapa.
- KDM1A ELISA livre: para cada placa, prepare 10 mL de estreptavidina a 10 μg/mL em PBS. Transfira 100 μL para cada poço da chapa.
- Top-sele as placas totais e livres de KDM1A ELISA com película adesiva e incubar as placas durante a noite em 4 ° c no refrigerador.
- Lavar e bloquear as placas
- Leve as placas para fora da geladeira e deixe-os equilibrados por cerca de 45 min em RT antes de usar.
- Prepare 1.000 mL tampão de lavagem (0,1% Tween em PBS) e 50 mL tampão de bloqueio (1% BSA em PBS) por placa.
- Lave as placas 3 vezes com tampão de lavagem. Neste e em etapas subseqüentes, bata a placa em toalhas de papel após cada etapa de lavagem para remover a solução residual.
- Adicionar 200 μL de tampão de bloqueio por poço a ambas as placas, vedação superior ambas as placas com uma película adesiva e incubar 2 h em RT.
- Preparação biológica da amostra
- Diluir os extratos de proteínas nativas obtidos no final da etapa 3 para a concentração apropriada usando PBS. A concentração recomendada variará em função do nível de expressão de KDM1A na amostra biológica. Os exemplos de escalas apropriadas são (1) pelotas da pilha: 0,5-10 μg por o poço. (2) PBMCs: 5-30 μg por poço. (3) tecido pulverizado (cérebro, pulmão, pele): 20-100 μg por poço. Mantenha as amostras no gelo durante a preparação. Quando possível, execute análises técnicas da amostra do triplicado.
- Prepare uma curva padrão usando o rKDM1A humano:
- Para preparar a solução de trabalho padrão KDM1A, Pipet o volume apropriado de rKDM1A para uma concentração final de 25 PG/μL, adicione 75 μL de 2 μM OG-881, e termine com 1 x PBS a um volume total de 6 mL em um tubo do falcão de 15 mL. Mantenha a solução de trabalho padrão KDM1A no gelo durante 1 h e misture a solução suavemente invertendo o tubo Falcon de 15 mL várias vezes a cada 20 min.
- Prepare a série de diluição padrão KDM1A em tubos de microcentrífuga de 1,5 ml de acordo com a tabela 1 (preparação padrão), em volume suficiente para a análise triplicada de 2 96 placas de microtitulação bem.
| SÉRIE PADRÃO | KDM1A solução de trabalho padrão (μL) | |
| (PG KDM1A/bem) | PBS (μL) | |
| 2500 para C-* | 800 | - |
| 2500 | 800 | - |
| 1750 | 560 | 240 |
| 1250 | 400 | 400 |
| 750 | 240 | 560 |
| 250 | 80 | 720 |
| 25 | 8 | 792 |
| 0 | 0 | 800 |
| Nota: | ||
| (1) o volume preparado de cada diluição é suficiente para ser executado em triplicado 2 placas de ensaio. | ||
| (2) a faixa recomendada é entre 2,5 e 5.000 PG/bem | ||
| * Para o controle negativo C-, sem anticorpo da deteção de KDM1A | ||
Tabela 1: preparação padrão. Para preparar a série padrão de proteína KDM1A, pipeta os volumes indicados de KDM1A solução de trabalho padrão e PBS em oito tubos de microcentrífuga de 1,5 mL devidamente rotulados.
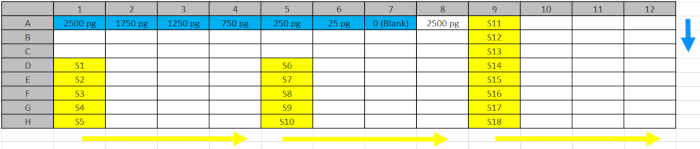
Tabela 2: projeto profundo da placa do poço. Padrões (azul) e amostras (amarelo) a partir do passo 5.4.2. foram pipetadas nas posições refletidas da placa de poço profundo para facilitar o carregamento nas placas ELISA seguindo a direção das setas azul (padrão) e amarela (amostras). Por favor, clique aqui para baixar este arquivo.

Tabela 3: projeto da placa de Elisa. A placa do ensaio inclui a curva padrão com quantidades de diminuição do alvo KDM1A de recombinação (no azul); as amostras biológicas (S) em amarelo; e os controles negativos correspondentes (contêm as amostras mas não o anticorpo preliminar da deteção) no branco, a ser carregados nas placas de ELISA da placa do poço profundo. O espaço em branco (0 na curva padrão) contém todos os reagentes de captura e detecção, mas nenhuma amostra. Por favor, clique aqui para baixar este arquivo.
- Elisa
- (Continuação do passo 5.2.4.) Após 2 h de incubação, descarte o tampão de bloqueio e lave as placas com tampão de lavagem.
- Transfira as amostras adequadamente diluídas (extratos de proteínas nativas e a curva padrão da etapa 5,3.) para um bloco de armazenamento refrigerado 96 profundo, seguindo a distribuição da placa mostrada na tabela 2 (design de placa de poço profundo).
- Mantenha este bloco no gelo até Pipetar 100 μL de amostra/poço nas placas ELISA total e livre, seguindo a distribuição da chapa mostrada na tabela 3 (ELISA Plate Design).
- Incubar por 1 h em RT, descarte as amostras e lave as placas 5 vezes com tampão de lavagem.
- Prepare 20 mL de anticorpo anti-KDM1A de detecção de coelhos a 0,125 μg/mL em tampão de bloqueio, adicione 100 μL por poço em cada placa do ensaio, excepto em poços correspondentes aos controlos negativos C-. Top-selar a placa e incubar 1 h em RT.
- Descarte a solução de anticorpos de detecção e lave as placas 6 vezes com tampão de lavagem.
- Prepare 25 ml de anticorpo secundário de cabra anti-coelho HRP para uma diluição 1:5000 em tampão de bloqueio, adicionar 100 μl por poço para as placas de de microtitulação; e incubar 1 h na RT.
-
Detecção quimioluminescente
- 30 min antes do final do Step 5.4.7. e condições de luz suave, misture partes iguais de luminol-Enhancer e solução de peróxido (10,5 mL: 10,5 mL, para 2 placas) em um frasco de âmbar e deixá-lo em RT.
Nota: Mantenha a solução de trabalho luminol numa garrafa de âmbar e evite a exposição prolongada a qualquer luz intensa. A exposição a curto prazo à iluminação laboratorial típica não prejudicará a solução de trabalho. - Pelo menos 20 minutos antes de medir a luminescência, ligue o leitor de microplacas a 25 ° c e configure leituras para 1.000 MS tempo de integração e 150 ms resolver tempo. As configurações de parâmetros podem exigir otimização em função do instrumento.
- Após 1 h de incubação em Step 5.4.7., descarte a solução de anticorpos secundários e lave as placas 6 vezes com tampão de lavagem.
- Pipetar 100 μL por poço da solução de trabalho luminol (substrato quimioluminescente) preparada na etapa 5.5.1. Pipeta muito lentamente e evite a formação de bolhas. Use um temporizador para controlar o tempo entre a adição da adição da solução e a medida da luminescência das placas e mantenha este tempo constante para conseguir uma boa reprodutibilidade inter do ensaio.
- Top-selar as placas e centrifugar a 500 x g em RT para 45 s em um centrifugador da placa para eliminar todas as bolhas restantes. Incubar as placas por 1 min em um agitador de chapa a 100 RPM.
- Insira a placa dentro do leitor e deixe-a por 3 min para estabilizar a temperatura a 25 ° c (sem película adesiva). Comece sempre com a placa de ELISA livre.
- Leia as unidades de luminescência relativa (RLU) de cada ensaio de Placa ELISA (livre e total de KDM1A).
- Salve e copie os valores brutos de RLU dos arquivos de Excel de dados brutos para análise posterior dos resultados.
- 30 min antes do final do Step 5.4.7. e condições de luz suave, misture partes iguais de luminol-Enhancer e solução de peróxido (10,5 mL: 10,5 mL, para 2 placas) em um frasco de âmbar e deixá-lo em RT.
6. cálculo do engajamento alvo
- Em um software de planilha, calcule os valores RLU Free e RLU total de amostras SX e amostras de referência ref (não tratada, veículo ou pré-dose amostra) de sua técnica replicar dados brutos conforme detalhado abaixo:
- Insira os dados RAW rlui total e RAW rlui individuais de espaços em branco, curva padrão, controles negativos C-e amostras biológicas (SX e ref) na folha de dados de análise (por exemplo, Excel). Também, incorpore os montantes (no PG) de KDM1A da curva padrão no datasheet.
- Calcule o RLU médio cru, os desvios padrão σRLU, e o coeficiente da variação CVRLU dos dados crus totais e crus livres individuais de rlui para cada DataPoint técnico da réplica.
- Aplique a eliminação de outlier (exemplo para triplicates): para cada rlui cru individual e cru RLU do ponto de dados livre de um ponto de DataPoint técnico do triplicado, aplique critérios de Grubbs quando o CV para o triplicado > 0,15, e rejeite o único valor cru do RLU do suspeito Quando
 ,
,
por meio de Z = 1,148 para n = 3 e 90% de intervalo de confiança (IC). - Se a eliminação de outlier foi aplicada, re-calcular o RAW média RLU, desvio padrão σ RLU e CVRLU do não-rejeitado (NR) RAW Rlui total e RAW rlui Free valores para cada DataPoint.
- Aplique a correção de plano de fundo: Calcule os valores médios RLU Free e RLU total para cada amostra padrão e cada amostra SX e referência de exemplo ref como:


- Represente graficamente os dados da seguinte forma:
- Plotar os valores RLUFree e RLUtotal (eixo Y) em relação à sua identificação de exemplo (eixo X) em um gráfico de barras.
- Também plotar os valores de RLU (eixo Y) dos padrões em um gráfico de dispersão em relação à sua quantidade de PG de proteína rKDM1A (eixo X) para medições gratuitas e totais, bem como as linhas de tendência lineares correspondentes e calcular o r2 (o quadrado do linear coeficiente de correlação).
- Calcule o engajamento de destino (TE); ou seja, a percentagem de KDM1A associada ao inibidor da KDM1A em cada amostra SX em relação a uma amostra de referência ref (não tratada, veículo ou pré-dose) da seguinte forma:
- Calcule a razão R da média RLU livre para total de valores para o SX e REF amostras como:

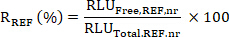
- Em seguida, calcule o engajamento de destino (TE) da amostra SX como:

Opcional: (1) se experimentos de repetição biológica N foram conduzidos, cada um com n repetições técnicas; primeiro Calcule o TESX para os conjuntos de replicação técnica. Posteriormente, calcule os valores médios de TE, SD e CV para o conjunto de replicantes biológicos.
- Calcule a razão R da média RLU livre para total de valores para o SX e REF amostras como:
- Rever se os critérios de aceitação do ensaio são cumpridos: Verifique se (1) o fundo do ensaio é aceitável e a média em branco < 0, 5 x 107 RLU; (2) a autoluminescência da amostra está ausente e os RLUs dos controles negativos C-estão abaixo do limite inferior de quantificação (LLOQ = média em branco + 10x DP); (3) a curva padrão rKDM1A é linear e R2 ≥ 0,98; (4) as amostras biológicas têm valores de RLU que caem na faixa dinâmica e linear do ensaio, ou seja, entre LLOQ e 2.500 PG/poço.
Nota: passos 6,1. a 6,4. pode ser prontamente automatizado em uma folha de dados de cálculo. - Exporte os dados de TE para uma fonte aberta ou software de estatística obtido comercialmente de escolha para a representação gráfica dos valores de TE e avaliações estatísticas adicionais.
Resultados
A linearidade da determinação KDM1A total e livre.
Uma série padrão foi preparada conforme descrito na etapa 5.3.2., usando 0 a 2500 PG de enzima recombinante humana KDM1A de comprimento total. Os valores de RLU de rKDM1A total e livre foram avaliados para verificar a linearidade (Figura 2a e 2B). Os dados são representados como média a partir de 3 experimentos com 3 repetições técnicas (n) ± DP. Os valores de RLU total e livre KDM1A detectados em PBMCs humanos do sangue de 3 voluntários independentes são sobrepostos na curva padrão na Figura 2C e 2D. Amostras de sangue foram obtidas do Instituto de Investigación biomédica Sant Pau biobank de acordo com a legislação espanhola (Real Decreto de biobancos 1716/2011) e aprovação dos comitês de ética locais.
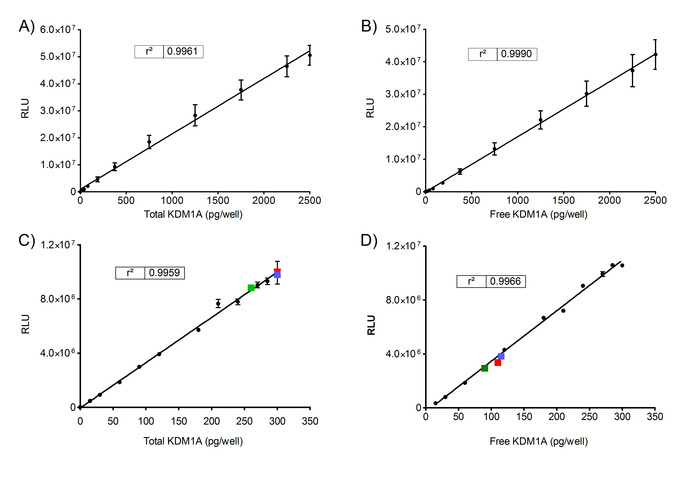
Figura 2. Determinação de rKDM1A total e livre em PBMCs de voluntários saudáveis. Valores de RLU do total rKDM1A avaliados por ELISA (A) e de RKDM1A livre avaliados pela captura de quimiosonda ELISA (B). Os dados foram obtidos a partir de 3 experimentos replicados, cada um analisado em triplicado (N = 3; n = 3). Valores de RLU do total KDM1A avaliados por ELISA (C) e de Free KDM1A (D) para os PBMCs de 3 voluntários independentes não tratados (quadrados vermelhos, azuis e verdes) sobrepostos na curva padrão. Os dados foram obtidos de um experimento analisado em triplicado (N = 1; n = 3). Os valores representados são as médias ± DP. por favor clique aqui para ver uma versão maior desta figura.
Análise de KDM1A engajamento alvo em células
As pilhas de AML foram cultivadas depois das recomendações do fornecedor. As células foram tratadas com veículo ou ORY-1001 em diferentes concentrações (0,25; 0,5; 1; 5 e 25 nM) (Figura 3). Os extratos proteicos nativos foram obtidos na presença de 25 nM OG-881 quimiosonda. 0,5 μg de proteína total foi utilizado para realizar a análise de engajamento-alvo, conforme descrito anteriormente. Foram determinados KDM1A total e livre, e o percentual de engajamento alvo de ORY-1001 para KDM1A foi calculado em relação ao veículo, conforme descrito.

Figura 3. Dose-resposta de KDM1A Target Engagement em uma linha celular de AML humano. As pilhas foram tratadas com o veículo ou o Ory-1001 em concentrações diferentes (0,25; 0,5; 1; 5 e 25 nanômetro) e usado para a determinação do acoplamento do alvo como descrito. Os dados foram obtidos a partir de 3 experimentos replicados, cada um analisado em triplicado (N = 3, n = 3). Os valores representados são as médias ± DP. por favor clique aqui para ver uma versão maior desta figura.
Análise do engajamento de alvos in vivo KDM1A
O objetivo deste experimento foi caracterizar o engajamento alvo de ORY-1001 em diferentes tecidos de ratos, em função do nível de dose. Para atingir esse objetivo, 15 ratos Sprague-Dawley (200-250 g) foram alojados em uma sala de segurança citostática para evitar a contaminação potencial pelo composto testado. Um máximo de 3 ratos/gaiola foram atribuídos aleatoriamente a 5 grupos de estudo. Os 5 grupos de estudo diferentes receberam, respectivamente, o veículo; 1 3 10 ou 30 μg/kg de ORY-1001 por administração oral durante 4 dias consecutivos. As soluções de ações compostas foram preparadas diariamente. Os animais foram pesados antes de cada administração para ajustar o volume exigido. Todos os animais foram alojados em temperatura ambiente constante (20-24 º C) e umidade relativa (45-65%) um ciclo claro-escuro de 12 h (luzes sobre em 6:00 AM). Alimentos e água estavam disponíveis ad libitum. Amostras de sangue foram coletadas 2 h após a última administração em tubos de K2EDTA e PBMCs foram isoladas de acordo com o procedimento descrito anteriormente na etapa 1.2.2. e preservado em-80 ° c até a extração nativa da proteína. Amostras pulmonares também foram coletadas 2 h após a última administração de fármacos, congeladas imediatamente em nitrogênio líquido e armazenadas a-80 ° c. Os estudos foram realizados de acordo com as diretrizes institucionais para o cuidado e uso de animais de laboratório (diretiva do Conselho das Comunidades Europeias 86/609/CEE), instituído pelo Comitê de ética para experimentação animal no PRAAL-PCB.
Após a pulverização, os extratos de proteínas nativas do pulmão foram obtidos conforme descrito e quantificado. 5 μg de proteína total de PBMCs agrupados ou 7,5 μg de proteína total do pulmão de 3 animais foram utilizados por grupo de dose para executar o ensaio de engajamento alvo KDM1A.
A dose-resposta de KDM1A de engajamento alvo em PBMCs e no tratamento pulmonar de ratos com ORY-1001 por gavagem oral, calculada em relação ao grupo de veículos, é mostrada na figura 4a e 4B. Como se pode ver na Figura 4C, a incubação ex vivo com 25 nm ory-1001 de extratos proteicos pulmonares dos animais tratados com o veículo produz te total ainda, mas não aumenta a te em amostras de ratos tratados por 4 dias com 30 μg/kg de ory-1001 , confirmando KDM1A já foi totalmente inibida in vivo.

Figura 4. Engajamento de alvos nativos in vivo e ex vivo KDM1A. Dose-resposta de KDM1A engajamento alvo em PBMCs (A) e amostras pulmonares (B) de ratos tratados com Ory-1001 por 4 dias consecutivos (p. o). Os dados foram obtidos de extratos agrupados de PBMCs de 3 animais por coorte, analisados em duplicata (N = 1, n = 2) ou dos pulmões de 3 animais individuais por coorte, analisados em triplicado (N = 3, n = 3). C. comparação da TE em extratos de proteínas pulmonares agrupadas de ratos tratados com veículo (à esquerda) ou 30 μg/kg ORY-1001; após 1 h de incubação ex vivo dos extratos sem (barras cinzentas) ou com 25 nM ORY-1001 (barras azuis) (N = 3, n = 3). Todos os dados são representados como médias ± DP. por favor clique aqui para ver uma versão maior desta figura.
Discussão
O protocolo apresentado aqui foi desenvolvido para medir diretamente o engajamento alvo de KDM1A usando um romance KDM1A com base na captura de quimiosonda ELISA. O método foi validado em linhagens de células humanas cultivadas e amostras ex vivo de humano, rato e rato e babuíno (incluindo PBMCs, pulmão, cérebro, pele, tumores), mas pode ser prontamente aplicada a outras espécies em que o anticorpo KDM1A resumos alvo e catalítico Centro são conservadas. Como OG-881 é um quimioprobe baseado na atividade, a qualidade da amostra é importante e a manipulação e a conservação apropriadas das amostras devem ser perseguidos especial durante as etapas iniciais do protocolo, para assegurar-se de que a atividade KDM1A esteja conservada.
O protocolo experimental atual foi otimizado para analisar KDM1A engajamento alvo por inibidores da FAD covalente. Também pode ser utilizado com inibidores reversíveis que bloqueiam o acesso ao cofator FAD de KDM1A. Inibidores potentes e reversíveis com tempos de permanência longos podem empregar o protocolo não modificado.
A quimiosonda OG-881 pode não ser adequada para inibidores reversíveis de baixa potência com altas taxas de desnível. A quimiosonda particular usada neste manuscrito não é penetrante celular e, portanto, as análises são realizadas ex vivo em amostras de lisadas.
O método pode ser executado em instrumentos que estão amplamente disponíveis em laboratórios de pesquisa e analíticos; não exige que as modificações genéticas sejam introduzidas nas pilhas, e pode facilmente ser aplicada aos tipos diferentes da amostra. Outra vantagem é que ele pode ser usado em amostras derivadas de diferentes espécies que são freqüentemente utilizadas na prova pré-clínica de estudos de conceito e em modelos de toxicologia e que foi traduzida com sucesso para analisar amostras clínicas.
Outros métodos têm sido utilizados para análise de KDM1A engajamento alvo. Muitos destes métodos usam marcadores do proxy como mudanças da marca do histona H3K4me2, usando alphalisa15; ou indução de marcadores de expressão utilizando a análise qRT-PCR ou FACS16. No entanto, em células ou tecidos, as marcas de histona são controladas por múltiplos fatores, e os ensaios que medem as alterações na marca da histona nem sempre proporcionam uma boa faixa dinâmica para análise. A inibição da KDM1A pode induzir mudanças potentes na expressão gênica e protéica, mas a resposta tende a depender muito heterogêneo e altamente dependente do contexto celular, o que pode complicar a análise da resposta da dose3,7.
A avaliação direta da ocupação do alvo é, portanto, a melhor opção para medir o engajamento alvo. Um ensaio que tem sido proposto para este é o teste de deslocamento térmico celular (CETSA), com base no aumento da estabilidade térmica das proteínas alvo após a ligação de inibidores. Este método pode, em princípio, ser aplicado a células não modificadas e diferentes tipos de tecidos e tem sido recentemente utilizado para avaliar a atividade celular dos inibidores da KDM1A em células cultivadas17. Entretanto, esta tecnologia foi usada raramente para estudos in vivo da farmacodinâmica18 e ao melhor de nosso conhecimento, seu uso não foi relatado em testes clínicos.
O protocolo fornecido aqui descreve um método com base em quimiosonda totalmente validado que tem sido usado para determinar KDM1A engajamento alvo em células e amostras de tecido. O método foi traduzido com sucesso para analisar amostras de indivíduos humanos tratados com um inibidor de KDM1A19 e será de grande uso para modelar respostas PK/PD em ensaios clínicos.
Divulgações
A autora Tamara Maes é diretora executiva e acionista, e os autores Cristina Mascaró e Raquel Ruiz Rodriguez são funcionários da ORYZON Genomics S.A. ORYZON Genomics S.A. desenvolve inibidores da KDM1A e detém patentes abrangendo compostos e métodos utilizados Neste artigo.
Agradecimentos
Este estudo foi financiado pela ORYZON Genomics. S.A., Hoffman-la Roche, e parcialmente apoiada pelo programa de colaboração CIIP-20152001 e RETOS RTC-2015-3332-1.
Materiais
| Name | Company | Catalog Number | Comments |
| 0,05% Trypsin-EDTA (1X) | Thermo Scientific | #25300-062 | |
| 10 X Protease Inhibitor Tablets | Roche | #11836153001 | |
| 96 deep well storage block | VWR | #734-1679 | |
| 96 well ELISA plates | Nunc | #436110 | |
| Adhesive black Film | Perkin Elmer | #6050173 | |
| Adhesive transparent Film | VWR | #60941-062 | |
| Biotinylated KDM1A probe OG-881 | Oryzon Genomics S.A. | NA | |
| Bovine Serum Albumin | Sigma | # 3117057001 | |
| Bovine Serum Albumin Standard | Thermo Scientific | #23208) | |
| Bradford Protein Assay | BioRad | #500-0001 | |
| Cell lysis buffer 10X | Cell Signaling | #9803 | |
| Centrifuge for 96- well plates | Hettich | Rotina 420R | |
| Flask | Thermo Scientific | #156499 | |
| Full length, enzymatically active human Recombinant LSD1 / KDM1A | Active Motif | #31426 | |
| Graphpad Prism 5 Project | GraphPad Software | NA | |
| Luminol-Enhacer and Peroxide Solution (Chemiluminescent Substrate) | Thermo Scientific | #37074 | |
| Micro Centrifuge | Eppendorf | 5415 R | |
| Microplate reader Infinite 200-Tecan | Tecan | Infinite 200 | |
| Mouse monoclonal capture antibody Anti-KDM1A (N-terminal epitope) | Abcam | #ab53269 | |
| Needle G18 gauge blunt | BD | #303129 | |
| ORY-1001 (iadademstat) | Oryzon Genomics S.A. | NA | |
| PBMC separation tubes 10 ml | Greiner bio-one | #163288 | |
| PBMC separation tubes 50 ml | Greiner bio-one | #227288 | |
| PBS 1x | Sigma | #D8537 | |
| Plate shaker | Heidolph Instruments | Rotamax 120 | |
| Polysorbate 20 | Sigma | #P7949 | |
| Rabbit monoclonal detection antibody Anti-KDM1A (C-terminal epitope) | Cell Signaling | #672184BF-100 | |
| Secondary antibody Peroxidase-conjugated Donkey Anti-rabbit IgG | Thermo Scientific | #31458 | |
| Spectrophotometer cuvette 1.5 | Deltalab | #302100 | |
| Spectrophotometer for cuvette | GE Healthcare | GeneQuant 1300 | |
| Streptavidin | Promega | #Z704A | |
| Syringe | BD | #303172 | |
| Type 1 ultrapure water | Millipore | Milli-Q Advantage A10 | |
| Ultrasonic cleaner | VWR | USC200T |
Referências
- Shi, Y. Histone demethylation mediated by the nuclear amine oxidase homolog LSD1. Cell. 119 (7), 941-953 (2004).
- Maiques-Diaz, A., Somervaille, T. C. LSD1: biologic roles and therapeutic targeting. Epigenomics. 8 (8), 1103-1116 (2016).
- Maes, T. ORY-1001, a Potent and Selective Covalent KDM1A Inhibitor, for the Treatment of Acute Leukemia. Cancer Cell. 33 (3), 495-511 (2018).
- Sugino, N. A novel LSD1 inhibitor NCD38 ameliorates MDS-related leukemia with complex karyotype by attenuating leukemia programs via activating super-enhancers. Leukemia. 31 (11), 2303-2314 (2017).
- Kleppe, M., Shank, K., Efthymia, P., Riehnhoff, H., Levine, R. L. Lysine-Specific Histone Demethylase, LSD1, (KDM1A) As a Novel Therapeutic Target in Myeloproliferative Neoplasms. Blood. 126, 601(2015).
- Jutzi, J. S., et al. LSD1 Inhibition Prolongs Survival in Mouse Models of MPN by Selectively Targeting the Disease Clone. HemaSphere. 2 (3), 54(2018).
- Mohammad, H. P. DNA Hypomethylation Signature Predicts Antitumor Activity of LSD1 Inhibitors in SCLC. Cancer Cell. 28 (1), 57-69 (2015).
- Rivers, A., et al. RN-1, a potent and selective lysine-specific demethylase 1 inhibitor, increases γ-globin expression, F reticulocytes, and F cells in a sickle cell disease mouse model. Experimental Hematology. 43 (7), 546-553 (2015).
- Rivers, A. Oral administration of the LSD1 inhibitor ORY-3001 increases fetal hemoglobin in sickle cell mice and baboons. Experimental Hematology. 67, 60-64 (2018).
- Buesa, C., et al. The dual LSD1/MAO-B inhibitor ORY-2001 prevents the development of the memory deficit in samp8 mice through induction of neuronal plasticity and reduction of neuroinflammation. Alzheimer’s & Dementia. 11 (7), P905(2015).
- Schmidt, D. M., McCafferty, D. G. Trans-2-Phenylcyclopropylamine is a mechanism-based inactivator of the histone demethylase LSD1. Biochemistry. 46 (14), 4408-4416 (2007).
- Forneris, F., Binda, C., Vanoni, M. A., Battaglioli, E., Mattevi, A. Human histone demethylase LSD1 reads the histone code. Journal of Biological Chemistry. 280 (50), 41360-41365 (2005).
- Oryzon Genomics, Methods to determine kdm1a target engagement and chemoprobes useful therefor. , WO2017158136 (2016).
- Mascaró, C., Ortega, A., Carceller, E., Rruiz Rodriguez, R., Cicero, F., Lunardi, S., Yu, L., Hilbert, M., Maes, T. Chemoprobe-based assays of histone lysine demethylase 1A target occupation enable in vivo pharmacokinetics and -dynamics studies of KDM1A inhibitors. Journal of Biological Chemistry. , In Press (2019).
- Rodriguez-Suarez, R. Development of Homogeneous Nonradioactive Methyltransferase and Demethylase Assays Targeting Histone H3 Lysine 4. Journal of Biomolecular Screening. 17 (1), 49-58 (2011).
- Lynch, J. T., Cockerill, M. J., Hitchin, J. R., Wiseman, D. H., Somervaille, T. C. CD86 expression as a surrogate cellular biomarker for pharmacological inhibition of the histone demethylase lysine-specific demethylase 1. Analytical Biochemistry. 442 (1), 104-106 (2013).
- Schulz-Fincke, J. Structure-activity studies on N-Substituted tranylcypromine derivatives lead to selective inhibitors of lysine specific demethylase 1 (LSD1) and potent inducers of leukemic cell differentiation. European Journal of Medicinal Chemistry. 144, 52-67 (2018).
- Ishii, T., et al. CETSA quantitatively verifies in vivo target engagement of novel RIPK1 inhibitors in various biospecimens. Scientific Report. 7, 13000(2017).
- Maes, T. ORY-2001: An Epigenetic drug for the treatment of cognition defects in Alzheimer’s disease and other neurodegenerative disorders. Alzheimer’s & Dementia. 12 (7), P1192(2017).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados