Method Article
Medición directa del compromiso objetivo de KDM1A mediante inmunoensayos basados en chemoprobe
En este artículo
Resumen
Aquí, presentamos un protocolo para medir el compromiso objetivo de KDM1A en una célula humana o animal, muestras de tejido o sangre tratadas con inhibidores de KDM1A. El protocolo emplea el etiquetado quimioprobe de la enzima KDM1A libre y la cuantificación directa de la ocupación objetivo mediante inmunoensayos basados en quimiosondas y se puede utilizar en estudios preclínicos y clínicos.
Resumen
La evaluación de la participación objetivo, definida como la interacción de un medicamento con la proteína para la que fue diseñado, es un requisito básico para la interpretación de la actividad biológica de cualquier compuesto en el desarrollo de fármacos o en proyectos de investigación básica. En la epigenética, la participación objetivo se evalúa con mayor frecuencia mediante el análisis de marcadores proxy en lugar de medir la unión del compuesto con el objetivo. Las lecturas biológicas aguas abajo que se han analizado incluyen la modulación de la marca de histona o los cambios en la expresión génica. KDM1A es una lisina demetilasa que elimina los grupos metilo de H3K4 mono y dimetilado, una modificación asociada con el silenciamiento de la expresión génica. La modulación de los marcadores proxy depende del tipo de célula y la función de la composición genética de las células investigadas, lo que puede dificultar la interpretación y la comparación entre mayúsculas y minúsculas. Para evitar estos problemas, se presenta un protocolo versátil para evaluar los efectos de la dosis y la dinámica de la participación directa de la meta de KDM1A. El ensayo descrito hace uso de una quimiosonda KDM1A para capturar y cuantificar enzimas desinhibidas, se puede aplicar ampliamente a células o muestras de tejido sin necesidad de modificación genética, tiene una excelente ventana de detección, y se puede utilizar tanto para la investigación básica y análisis de muestras clínicas.
Introducción
Lisina específica demetilasa 1 (KDM1A)1 es una demetilasa implicada en el control de la transcripción génica. Esta proteína ha surgido como un objetivo farmacológico candidato2 en oncología; incluyendo Leucemia mieloide aguda3 (LMA), Síndrome de mielodisplasia (MdS)4, Mielofibrosis (MF)5,6, Cáncer de pulmón de células pequeñas (SCLC)7; en la enfermedad de células falciformes (SCD)8,9, y en enfermedades del sistema nervioso central incluyendo la enfermedad de Alzheimer (AD), Esclerosis Múltiple (MS); y en agresión10.
La mayoría de los compuestos inhibidores de KDM1A en el desarrollo clínico son derivados de la ciclopropilina e inhiben la proteína mediante la unión covalente a su conucleótido de adenina de flavina (FAD) cofactor11. La inhibición de KDM1A induce cambios en la expresión génica, pero estos cambios varían enormemente entre tejidos, tipos de células o casos de enfermedad. La inhibición de KDM1A también cambia las marcas de histona12,sin embargo, estos cambios generalmente se producen localmente en un sitio específico en el genoma, y son de nuevo, altamente tejido y específico de las células.
El protocolo fue desarrollado para medir directamente la participación objetivo de KDM1A en muestras biológicas y ha sido optimizado para el uso con inhibidores derivados de la ciclopropilina. El ensayo se basa en la tecnología ELISA y analiza, en paralelo, Total y Libre (es decir, no unido por inhibidor) KDM1A en un extracto de proteína nativa de una muestra biológica en un ensayo de fase sólida. Como primer paso, la muestra biológica se lysed en presencia de la quimiosonda selectiva biotinilada KDM1A OG-88113,14, derivada del inhibidor selectivo KDM1A ORY-1001 (iadademstat), un potente inhibidor de KDM1A en clínica desarrollo para el tratamiento de enfermedades oncológicas. La quimiosonda tiene un IC50 para KDM1A de 120 nM e incluye una mitad de unión FAD vinculada a una cola de polietileno glicol biotinilado (PEG). La quimiosonda se une exclusivamente al KDM1A libre, pero no al KDM1A ligado a inhibidores en la muestra. Después de la unión de quimiosonda, el KDM1A que contiene complejos en la muestra se captura nado en placas microtíteres con superficie recubierta de estreptavidina para determinar KDM1A libre, o en placas recubiertas con un anticuerpo de captura monoclonal anti-KDM1A para determinar el Total de KDM1A. Después del lavado, ambas placas se incuban con un anticuerpo de detección anti-KDM1A, se lavan de nuevo, y se incuban con un anticuerpo secundario anticonejo anticonejo conjugado con conjugado con HRP para su detección utilizando un sustrato luminiscente y la cuantificación midiendo unidades de luz (RLU) en un luminómetro (Figura1).
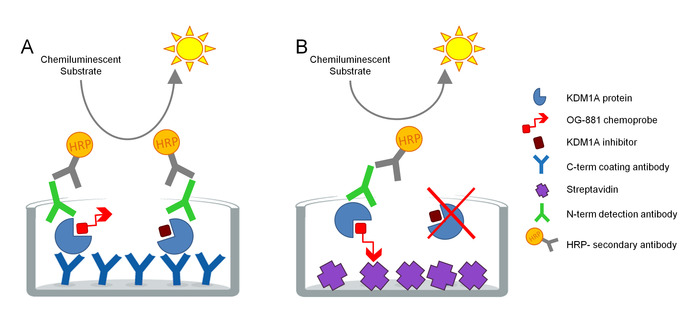
Figura 1. Esquema de elsay de inmunoabsorbente de quimiosonda ligada a enzimas ELISA para el compromiso objetivo KDM1A: A) Determinación del Total de KDM1A utilizando el sándwich ELISA y B) Determinación de KDM1A libre utilizando quimiosonda ELISA. Haga clic aquí para ver una versión más grande de esta figura.
Se incluye una curva estándar en ambas placas ELISA para verificar la linealidad de cada ensayo. La determinación de la participación objetivo de KDM1A en cada muestra se calcula entonces como un valor relativo a la muestra predosis o tratada por el vehículo.
Protocolo
Se obtuvieron muestras de sangre del Instituto de Investigación Biomédica Sant Pau Biobank de acuerdo con la legislación española (Real Decreto de Biobancos 1716/2011) y la aprobación de los comités de ética local. Los estudios con tejidos animales se realizaron de conformidad con las directrices institucionales para el cuidado y uso de animales de laboratorio (Directiva 86/609/CEE del Consejo de las Comunidades Europeas) establecidas por el Comité ético para la experimentación animal en el PRAAL-PCB.
1. Preparación de muestras biológicas para el ensayo.
ADVERTENCIA: Este protocolo implica la manipulación de muestras biológicas que pueden someterse a la norma de patógenos de la salud y la seguridad en el trabajo (OSHA) (29 CFR 1910.1030), la Directiva 2000/54/CE del Parlamento Europeo y de la Consejo de 18 de septiembre de 2000 o reglamentos equivalentes. Además, las muestras biológicas pueden contener trazas de compuestos químicos de investigación biológicamente activos y el protocolo puede implicar una mayor manipulación de dichos compuestos. Revisar la ficha de datos de seguridad (SDS) de los compuestos utilizados antes del inicio del experimento y observar estrictamente todas las medidas de seguridad aplicables establecidas en el centro de investigación, incluido el uso de equipos de protección personal (EPP) adecuados. Use ropa protectora adecuada y un blindaje adecuado durante el transcurso del experimento. Desechar los residuos en los contenedores de residuos apropiados (residuos biológicos/citotóxicos).
NOTA: Este protocolo comienza con células o muestras de sujetos tratados con un inhibidor de KDM1A y sus controles no tratados o tratados con vehículo/placebo3.
- Células tratadas con vehículo o inhibidor de KDM1A in vitro
- Para las células cultivadas en suspensión, como cultivos de 10 ml, transfiera las suspensiones a tubos cónicos limpios de 15 ml y proceda a 1.1.3.
- Para las células adherentes (cultivadas en frascos de 75 cm 2), retire el medio del matraz y lave brevemente con 4 ml de PBS. Separar las células de sus vasos usando 1.5 ml de 0.5% Trypsin-EDTA durante 2 - 5 min (condiciones de trippsinización pueden variar, seguir las recomendaciones del proveedor para la línea celular), agregar 4 mL PBS y transferir las células en tubos cónicos limpios de 15 ml.
- Inserte los tubos en una centrífuga de sobremesa y recoja las células por centrifugación durante 5 min a 400 x g a 4 oC. Retire el sobrenadante, resuspenda el pellet en 1 ml de PBS dispensado con un micropipeta y transfiera la suspensión a un tubo de microcentrífuga de 1,5 ml.
- Inserte las muestras en una centrífuga de microtubo y centrífuga durante 5 min a 400 x g a 4 oC. Retire el PBS por aspiración con un micropipeta y mantenga los pellets en el hielo y proceda al paso 2; o congelar los pellets en hielo seco y guardarlos a -80 oC hasta el paso 3.
- Muestras de sujetos o animales tratados con vehículo/placebo o inhibidor de KDM1A
- Tejidos: Cortar el tejido en trozos pequeños de 1 cm3 con un bisturí. Congele las piezas de tejido en líquido N2 en un recipiente de Dewar y guárdelas a -80 oC hasta el paso 3.
- Células mononucleares de sangre polimórfica (PPBCd): Diluir 10 ml de sangre fresca (máximo de proceso 2 h después de la extracción de sangre) recogida en tubos K2-EDTA con 2 volúmenes de PBS en un tubo cónico de 50 ml. Aísle los PBMC de la sangre utilizando tubos de separación PBMC obtenidos comercialmente de acuerdo con las instrucciones del fabricante. Mantener los pellets en el hielo y proceder al paso 3.2; o congelar los pellets en hielo seco y guardarlos a -80 oC hasta el paso 3.
NOTA: Un gránulo de células húmedas de 20 a 50 l contiene 1 x 107 células, dependiendo del tamaño de la célula. Un pellet pBMC húmedo obtenido a partir de 10 ml de sangre humana sana tiene un volumen de 20 s L y contiene 1 x 107 PbMCs. Los tejidos o pellets celulares se pueden almacenar a -80 oC durante un máximo de 6 meses.
2. Preparación de la solución
- Preparar una solución de trabajo de 2 M OG-881: Tomar una alícuota de un solo uso de 10 ml de la solución de 20 mM de sonda biotinada OG-881 fuera de la nevera de 4 oC y déjela a temperatura ambiente (RT) durante 10 min. Prepare la solución de trabajo de 2 mM mediante la serialización del solut de stock OG-881 ión en PBS, utilizando un micropipeta con puntas de filtro y cambiando la punta entre los diferentes pasos de dilución.
- Preparar 10x inhibidor de la proteasa: disolver 1 comprimido en 1 ml de PBS en un tubo de microcentrífuga.
- Prepare el volumen deseado de 1 tampón de lisis celular con 25 nM OG-881 quimiosondas. Para cada ml, mezcle 100 ml obtenido comercialmente 10x tampón de lisis celular, 150 ml de inhibidor de proteasa 10x, 12,5 ml de 2 oG-881 y 737,5 l de agua destilada doble de tipo 1.
- Opcionalmente prepare el volumen deseado del buffer de lisis de 1x Cell pero con 25 nM ORY-1001 en vez de OG-881 como en el paso 2.3. Se pueden utilizar inhibidores menos potentes, pero pueden requerir concentraciones más altas, para su uso en el control positivo con 100% de inhibición (ver Paso 3.5).
NOTA: Tome las medidas adecuadas para evitar cualquier contaminación involuntaria de soluciones o muestras con las soluciones de material inhibidor OG-881 o KDM1A. Para calcular el volumen deseado de 1 tampón de lisis celular con 25 nM OG-881, suponga mosquese que se requiere 400 l por cada 40 mg de tejido pulverizado, o 200 ml por pellet húmedo de 107 células.
3. Extracción de proteínas nativas
- De los tejidos:
- Pulverizar y homogeneizar un cubo de tejido congelado de 1 cmde peso con un mortero y un pestillo refrigerado sobre hielo seco. Alícuota las muestras en viales de un solo uso que contengan 40 mg de polvo tisular, evite la descongelación en todo momento. Continúe con el paso 3.1.2. para su procesamiento inmediato o para almacenar a -80 oC.
- Resuspender 40 mg de tejido en polvo en 400 l de tampón de lisis celular 1x con 25 nM OG-881, vórtice durante 10 s, y forzar la muestra al menos cinco veces a través de una aguja de jeringa contundente de calibre 18 hasta que se logre la lisis del tejido y se logre una suspensión de amarillo a naranja de color claro turbio y se produce una suspensión de amarillo ligero a naranja Obtenido. Evite la formación de burbujas.
- Continúe con el paso 3.3
- De pellets celulares (PBMC y líneas celulares):
- Resuspenda un gránulo de 1 x 107 células en 200 l de tampón de lisis de 1 x célula que contenga 25 nM OG-881. Vortex las muestras brevemente y mantenerlas en hielo durante 5 min.
- Sonicar las muestras en un sonicador usando 3 pulsos de 20 s cada uno a 45 kHz; colocarlos en hielo durante 20 s entre pulsos.
NOTA: Tan pronto como las muestras biológicas hayan sido resuspendidas en el tampón de lisis de células 1x, manténgalas en el hielo durante el resto del proceso.
- Mantenga las muestras en hielo durante 5 minutos adicionales, vórtice brevemente y centrifugar las muestras durante 10 min a 14 000 x g en una centrífuga preenfriada a 4 oC.
- Con un micropipeta de 1 ml, transfiera los sobrenadores a tubos de microcentrífuga frescos de 1,5 ml y déjelos en el hielo durante 2 h. Continúe en el paso 4.
- Opcionalmente, se puede preparar un control positivo para simular el 100% de la interacción objetivo de la siguiente manera:
- Resuspenda el pellet celular o el tejido en polvo de un vehículo o muestra no tratada (predosis) en el volumen requerido de 1tampón de lysis celular con 25 nM ORY-1001 y procese como se describe en los pasos 3.1 a 3.3.
- Transfiera los sobrenadores del control positivo en tubos de microcentrífuga frescos de 1,5 ml y déjelos en hielo durante 1 h. ORY-1001 inhibir deliberadamente KDM1A y bloquear la unión de quimiosonda.
- Añadir 5 ml de solución de trabajo de 2 oMg-881 al sobrenadante de control positivo (volumen para el control positivo generado a partir de una muestra de tejido de 40 mg) o 2,5 oL de solución de trabajo de 2 M OG-881 (volumen para control positivo generado a partir de una muestra de 107celdas) para obtener la misma concentración og-881 que las otras muestras y dejar en hielo durante 2 h. Continúe en el paso 4.
4. Cuantificación de proteínas nativas mediante el ensayo Bradford
- Diluir el reactivo de ensayo de proteína bradford de origen comercial 5 veces con agua destilada doble H2O Tipo 1. Calcular el volumen del reactivo necesario para la cantidad total de muestras y estándares (1 ml por muestra o volumen de exceso estándar + 5 ml).
- Para la curva estándar bovino Serum Albumin (BSA), prepare un tubo de microcentrífuga con una solución de ensayo de proteína bradford diluido de 1 ml (en blanco) y siete tubos de microcentrífuga con 995 ml de la solución diluida de ensayo de proteína sbradford. Añadir 5 l de cada una de las normas BSA (concentración que oscila entre 125 y 2.000 g/ml) a cada uno de los 7 tubos de microcentrífuga y mezclarlos invirtiendo suavemente los tubos varias veces. Incubar durante 5 min a RT.
- Transfiera los Estándares diluidos a las cubetas y lea la DO de las muestras estándar de albúmina de suero en blanco y bovino en un espectrofotómetro a 280 nm.
- Para las muestras biológicas, prepare un tubo de microcentrífuga con una solución de ensayo de proteína bradford diluido de 1 ml (en blanco) y tantos tubos de microcentrífuga con 999 ml del reactivo diluido de ensayo de proteína de Bradford como muestras que necesitan ser cuantificadas. Usando una micropipeta P2 automática, agregue 1 l de extracto de proteína nativo preparado en el paso 3 a cada tubo de microcentrífuga y mezcle invirtiendo suavemente los tubos varias veces. Incubar las muestras 5 min a RT.
- Transfiera los volúmenes a cubetas y lea la DO de las muestras en un espectrofotómetro a 280 nm.
- Preferentemente, proceda inmediatamente al paso 5. Alternativamente, almacene los extractos de proteínas nativas a -80 oC hasta el paso 5. Evite los ciclos de congelación de descongelación.
5. ELISA luminosas para la determinación Total y Gratuita de KDM1A
NOTA: Mantenga la temperatura de laboratorio constante a 23-24 oC (RT).
- Recubrimiento de placas microtíteas con anticuerpo KDM1A de captura o Streptavidin
- ElISA total de KDM1A: para cada placa, prepare 10 ml de anticuerpo de captura KDM1A a una concentración final de 2 g/ml en PBS. Transfiera 100 l a cada poceto de la placa.
- KDM1A ELISA libre: para cada placa, prepare 10 ml de estreptavidina a 10 g/ml en PBS. Transfiera 100 l a cada poceto de la placa.
- Selle las placas Total y Free KDM1A ELISA con película adhesiva e incubar las placas durante la noche a 4oC en el frigorífico.
- Lavado y bloqueo de las placas
- Saque las placas del refrigerador y déjelas equilibrar durante unos 45 minutos en RT antes de su uso.
- Prepare 1.000 ml de tampón de lavado (0,1% Tween en PBS) y búfer de bloqueo de 50 ml (1% BSA en PBS) por placa.
- Lave las placas 3 veces con tampón de lavado. En este y siguientes pasos, toque la placa en toallas de papel después de cada paso de lavado para eliminar la solución residual.
- Añadir 200 l de tampón de bloqueo por pocaa a ambas placas, sellar las dos placas con una película adhesiva e incubar 2 h en RT.
- Preparación biológica de muestras
- Diluir los extractos de proteína nativa obtenidos al final del paso 3 a la concentración adecuada utilizando PBS. La concentración recomendada variará en función del nivel de expresión de KDM1A en la muestra biológica. Ejemplos de rangos apropiados son (1) pellets celulares: 0.5 - 10 g por pozo. (2) PBMCs: 5 - 30 g por pozo. (3) Tejido pulverizado (cerebro, pulmón, piel): 20 - 100 g por pozo. Mantenga las muestras en el hielo durante la preparación. Cuando sea posible, ejecute análisis técnicos de muestras triplicados.
- Preparar una curva estándar utilizando rKDM1A humano:
- Para preparar la solución de trabajo estándar de KDM1A, pipetee el volumen adecuado de rKDM1A para una concentración final de 25 pg/L, agregue 75 ml de 2 oG-881 y complete con 1 x PBS a un volumen total de 6 ml en un tubo de halcón de 15 ml. Mantenga la solución de trabajo estándar KDM1A sobre hielo durante 1 h y mezcle la solución suavemente invirtiendo el tubo de halcón de 15 ml varias veces cada 20 minutos.
- Preparar la serie de dilución estándar KDM1A en tubos de microcentrífuga de 1,5 ml según la Tabla 1 (Preparación Estándar), en volumen suficiente para el análisis triplicado de dos placas de microtíter de 96 pocillos.
| SERIE STANDARD | KDM1A Solución de trabajo estándar (L) | |
| (pg KDM1A/pozo) | PBS (L) | |
| 2500 para C-* | 800 | - |
| 2500 | 800 | - |
| 1750 | 560 | 240 |
| 1250 | 400 | 400 |
| 750 | 240 | 560 |
| 250 | 80 | 720 |
| 25 | 8 | 792 |
| 0 | 0 | 800 |
| Nota: | ||
| (1) El volumen preparado de cada dilución es suficiente para correr en triplicado 2 placas de ensayo. | ||
| (2) El rango recomendado es entre 2.5 y 5,000 pg / bien | ||
| * Para el control negativo C-, sin anticuerpo de detección KDM1A | ||
Tabla 1: Preparación estándar. Para preparar la serie estándar de proteína KDM1A, pipetee los volúmenes indicados de solución de trabajo estándar KDM1A y PBS en ocho tubos de microcentrífuga de 1,5 ml debidamente etiquetados.
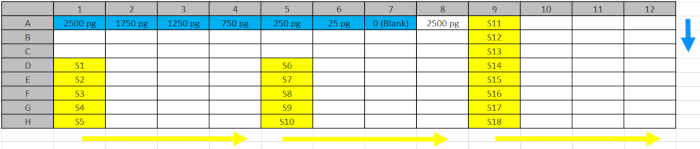
Tabla 2: Diseño de placas de pozoprofundo profundo. Estándares (azul) y muestras (amarillo) del paso 5.4.2. fueron canalizados en las posiciones reflejadas de la placa de pozo profundo para facilitar la carga en las placas ELISA siguiendo la dirección de las flechas azul (estándar) y amarilla (muestras). Haga clic aquí para descargar este archivo.

Tabla 3: Diseño de placas ELISA. La placa de ensayo incluye la curva estándar con cantidades decrecientes de objetivo KDM1A recombinante (en azul); las muestras biológicas (S) en amarillo; y los controles negativos correspondientes (contienen las muestras pero no el anticuerpo de detección primaria) en blanco, que se cargarán en las placas ELISA de la placa deep Well. El espacio en blanco (0 en la curva estándar) contiene todos los reactivos de captura y detección, pero ninguna muestra. Haga clic aquí para descargar este archivo.
- Elisa
- (Continuación del paso 5.2.4.) Después de 2 h de incubación, deseche el tampón de bloqueo y lave las placas con tampón de lavado.
- Transfiera las muestras adecuadamente diluidas (extractos de proteínas nativas y curva estándar del paso 5.3.) a un bloque de almacenamiento de pozos profundos refrigerados de 96 siguiendo la distribución de placas que se muestra en la Tabla 2 (Deep Well Plate Design).
- Mantenga este bloque en hielo hasta que se pipetee una muestra de 100 ol / bien en las placas ELISA total y libre siguiendo la distribución de placas que se muestra en la Tabla 3 (Diseño de placas ELISA).
- Incubar durante 1 h a RT, deseche las muestras y lave las placas 5 veces con tampón de lavado.
- Preparar 20 ml de anticuerpo de detección anti-KDM1A de conejo a 0,125 g/ml en tampón de bloqueo, añadir 100 ml por pocto en cada plato del ensayo, excepto en los pozos correspondientes a los controles negativos C-. Selle la placa e incubar 1 h en RT.
- Deseche la solución de anticuerpos de detección y lave las placas 6 veces con tampón de lavado.
- Preparar 25 ml de anticuerpo anticonejo de cabra secundario HRP a una dilución 1:5,000 en tampón de bloqueo, añadir 100 l por pocal a las placas de microtíter; e incubar 1 h a RT.
-
Detección quimioluminiscente
- 30 min antes del final del paso 5.4.7. y en condiciones de luz blanda, mezclar partes iguales de Luminol-Enhancer y Solución de peróxido (10,5 ml: 10,5 ml, para 2 placas) en una botella de ámbar y dejarla en RT.
NOTA: Mantenga la solución de trabajo de Luminol en un frasco de ámbar y evite la exposición prolongada a cualquier luz intensa. La exposición a corto plazo a la iluminación típica de laboratorio no dañará la solución de trabajo. - Al menos 20 minutos antes de medir la luminiscencia, encienda el lector de microplacas a 25 oC y configure lecturas a 1.000 ms de tiempo de integración y 150 ms de tiempo de asentado. La configuración de parámetros puede requerir una optimización en función del instrumento.
- Después de 1 h de incubación en el paso 5.4.7., deseche la solución secundaria de anticuerpos y lave las placas 6 veces con tampón de lavado.
- Pipeta de 100 ml por pozo de la solución de trabajo Deluminol (sustrato quimioluminiscente) preparada en el paso 5.5.1. Pipetear muy lentamente y evitar la formación de burbujas. Utilice un temporizador para controlar el tiempo entre la adición de la adición de la solución y la medición de luminiscencia de las placas y mantenga este tiempo constante para lograr una buena reproducibilidad entre ensayos.
- Selle la parte superior de las placas y centrífuga a 500 x g a RT durante 45 s en una centrífuga de placa para eliminar las burbujas restantes. Incubar las placas durante 1 min en una coctelera a 100 rpm.
- Inserte la placa dentro del lector y déjela durante 3 minutos para estabilizar la temperatura a 25 oC (sin película adhesiva). Comience siempre con la placa ELISA gratuita.
- Lea las unidades de luminiscencia relativa (RLU) de cada ensayo de placa ELISA (KDM1A libre y total).
- Guarde y copie los valores de RLU sin procesar de los archivos de Excel de datos sin procesar para un análisis posterior de los resultados.
- 30 min antes del final del paso 5.4.7. y en condiciones de luz blanda, mezclar partes iguales de Luminol-Enhancer y Solución de peróxido (10,5 ml: 10,5 ml, para 2 placas) en una botella de ámbar y dejarla en RT.
6. Cálculo de la participación objetivo
- En un software de hoja de cálculo, calcule los valores RLU Free y RLU Total de las muestras SX y las muestras de referencia REF (muestra sin tratar, vehículo o predosis) a partir de sus datos de réplica técnica Raw como se detalla a continuación:
- Introduzca los datos individuales Raw RLUi Total y Raw RLUi Free de espacios en blanco, curva estándar, controles negativos C- y muestras biológicas (SX y REF) en la hoja de datos de análisis (por ejemplo, Excel). Además, introduzca los importes (en pg) de KDM1A de la curva estándar en la hoja de datos.
- Calcule la RLU media sin procesar, las desviaciones estándar deRLUy el coeficiente de variación CVRLU a partir de los datos individuales de RLUi total y libre sin procesar individuales para cada punto de datos de réplica técnica.
- Aplique la eliminación de valores atípicos (ejemplo para triplicados): para cada punto de datos Raw RLU Total y Raw RLU Free RLUi individual desde un punto de datos de triplicado técnico, aplique los criterios de Grubbs cuando el CV para el triplicado > 0.15, y rechace el único sospechoso de valor Raw RLU valor Cuando
 ,
,
por el cual Z a 1.148 para n a 3 y intervalo de confianza del 90 % (CI). - Si se aplicó la eliminación de valores atípicos, vuelva a calcular la RLU media sin procesar, la desviación estándarrLU y CVRLU de los valores no rechazados (nr) Raw RLUi Total y Raw RLUi Free para cada punto de datos.
- Aplicar la corrección de fondo: Calcular los valores medios rLU libres y RLU Total para cada muestra estándar, y cada muestra SX y la muestra de referencia REF como:


- Representa gráficamente los datos de la siguiente manera:
- Trazar los valores RLUFree y RLUTotal (eje Y) en relación con su identificación de muestra (eje X) en un gráfico de barras.
- También trazar los valores RLU (eje Y) de los estándares en una gráfica de dispersión en relación con su cantidad de pg de proteína rKDM1A (eje X) para mediciones libres y totales, así como las líneas de tendencia lineales correspondientes y calcular el r2 (el cuadrado de la lineal valores de correlación).
- Calcular el compromiso objetivo (TE); es decir, el porcentaje de KDM1A enlazado por el inhibidor KDM1A en cada muestra SX en relación con una muestra de referencia REF (muestra no tratada, vehículo o predosis) de la siguiente manera:
- Calcule la relación R de los valores medios de RLU Free a Total para las muestras SX y REF como:

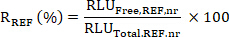
- A continuación, calcule el compromiso objetivo (TE) de la muestra SX como:

Opcional: (1) Si se llevaron a cabo N experimentos de réplica biológica, cada uno con n réplicas técnicas; primero calcule el TESX para los conjuntos de réplicas técnicas. Posteriormente, calcule los valores medios de TE, SD y CV para el conjunto de réplicas biológicas.
- Calcule la relación R de los valores medios de RLU Free a Total para las muestras SX y REF como:
- Revisar si se cumplen los criterios de aceptación del ensayo: Verifique que (1) el fondo del ensayo sea aceptable y la media en blanco < 0,05 x 107 RLU; (2) la autoluminiscencia de la muestra está ausente y las RPU de los controles negativos C- están por debajo del límite inferior de cuantificación (LLOQ - Espacio medio + 10x SD); (3) la curva estándar rKDM1A es lineal y r2 a 0,98; (4) las muestras biológicas tienen valores RLU que caen en el rango dinámico y lineal del ensayo, es decir, entre LLOQ y 2.500 pg/pozo.
NOTA: Pasos 6.1. a 6.4. puede automatizarse fácilmente en una hoja de datos de cálculo. - Exporte los datos de TE a un software de estadísticas de código abierto o obtenido comercialmente de su elección para la representación gráfica de los valores de TE y evaluaciones estadísticas adicionales.
Resultados
La linealidad de la determinación Total y Libre de KDM1A.
Se preparó una serie estándar como se describe en el paso 5.3.2., utilizando 0 a 2500 pg de enzima KDM1A recombinante humana de longitud completa. Los valores RLU de Total y Free rKDM1A se evaluaron para verificar la linealidad (Figura2A y 2B). Los datos se representan como media de 3 experimentos con 3 réplicas técnicas (n) a SD. Los valores RLU de KDM1A total y libre detectados en PMIHumana humana a partir de la sangre de 3 voluntarios independientes se superponen en la curva Estándar en las Figuras 2C y 2D. Se obtuvieron muestras de sangre del Instituto de Investigación Biomédica Sant Pau Biobank de acuerdo con la legislación española (Real Decreto de Biobancos 1716/2011) y la aprobación de los comités de ética local.
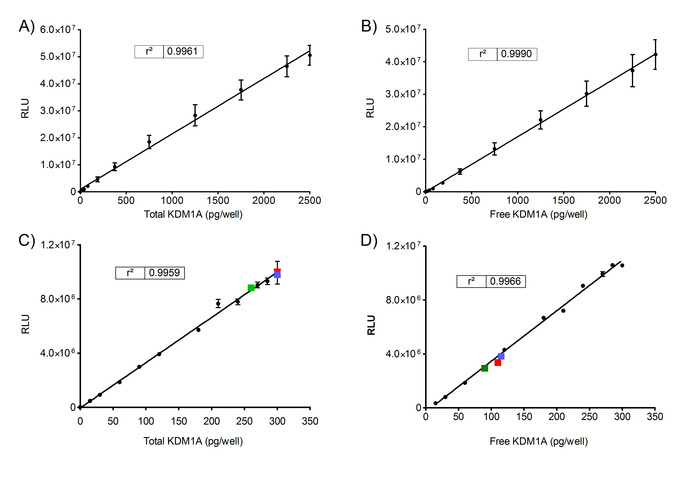
Figura 2. Determinación de rKDM1A Total y Gratis en PBMCs de voluntarios sanos. Valores RLU de RKDM1A total evaluados por ELISA (A) y de RKDM1A gratuito evaluados por captura quimiosonda ELISA (B). Los datos se obtuvieron de 3 experimentos de réplica, cada uno analizado en triplicado (N 3; n a 3). Valores RLU de KDM1A total evaluados por ELISA (C) y de KDM1A libre (D) para los PBB de 3 voluntarios independientes no tratados (cuadrados rojos, azules y verdes) superpuestos en la curva estándar. Los datos se obtuvieron de un experimento analizado en triplicado (N 1; n a 3). Los valores representados son los medios sD. Por favor, haga clic aquí para ver una versión más grande de esta figura.
Análisis de la participación objetivo de KDM1A en células
Las células AML se cultivaron siguiendo las recomendaciones del proveedor. Las células fueron tratadas con vehículo u ORY-1001 a diferentes concentraciones (0,25; 0,5; 1; 5 y 25 nM) (Figura3). Los extractos de proteína nativa se obtuvieron en presencia de 25 nM OG-881 quimiosonda. Se utilizaron 0,5 g de proteína total para realizar el análisis de compromiso objetivo como se describió anteriormente. Se determinaron KDM1A total y gratuito, y el porcentaje de compromiso objetivo de ORY-1001 a KDM1A se calculó en relación con el vehículo como se describe.

Figura 3. Dosis-respuesta de KDM1A Target Engagement en una línea de células AML humana. Las células fueron tratadas con vehículo u ORY-1001 a diferentes concentraciones (0,25; 0,5; 1; 5 y 25 nM) y se utilizaron para determinar el compromiso objetivo como se describe. Los datos se obtuvieron de 3 experimentos de réplica, cada uno analizado en triplicado (N 3, n a 3). Los valores representados son los medios sD. Por favor, haga clic aquí para ver una versión más grande de esta figura.
Análisis de la participación objetivo in vivo de KDM1A
El objetivo de este experimento era caracterizar el compromiso objetivo de ORY-1001 en diferentes tejidos de rata, en función del nivel de dosis. Para lograr este objetivo, 15 ratas Sprague-Dawley (200-250 g) fueron alojados en una sala de seguridad citostática para evitar la contaminación potencial por el compuesto probado. Un máximo de 3 ratas/jaula fueron asignadas aleatoriamente a 5 grupos de estudio. Los 5 grupos de estudio diferentes recibieron, respectivamente, vehículo; 1; 3; 10 o 30 g/kg de ORY-1001 por administración oral durante 4 días consecutivos. A diario se preparaban soluciones de stock compuesto. Los animales se pesaron antes de cada administración para ajustar el volumen requerido. Todos los animales fueron alojados a temperatura ambiente constante (20 - 24 oC) y humedad relativa (45 - 65 %) bajo un ciclo de 12 h de luz-oscuridad (se enciende a las 6:00 AM). La comida y el agua estaban disponibles ad libitum. Se recogieron muestras de sangre 2 h después de la última administración en tubos K2EDTA y los PBMC se aislaron de acuerdo con el procedimiento descrito anteriormente en el paso 1.2.2. y se conserva a -80 oC hasta la extracción de proteínas nativas. Las muestras pulmonares también se recogieron 2 h después de la última administración del medicamento, se congelaron inmediatamente en nitrógeno líquido y se almacenaron a -80 oC. Los estudios se realizaron de conformidad con las directrices institucionales para el cuidado y uso de animales de laboratorio (Directiva 86/609/CEE del Consejo de las Comunidades Europeas) establecidas por el Comité ético de experimentación animal en el PRAAL-PCB.
Después de la pulverización, los extractos de proteína nativa del pulmón se obtuvieron según lo descrito y cuantificado. Se utilizaron 5 g de proteína total a partir de PPM agrupados o 7,5 g de proteína total de pulmón de 3 animales por grupo de dosis para ejecutar el ensayo de compromiso objetivo KDM1A.
La dosis-respuesta de la participación objetivo de KDM1A en PBB y en el tratamiento pulmonar de ratas con ORY-1001 por vía oral, calculada en relación con el grupo de vehículos, se muestra en las figuras 4A y 4B. Como se puede ver en la Figura 4C, la incubación ex vivo con 25 nM ORY-1001 de extractos de proteína pulmonar de los animales tratados vehiculares produce TE completo todavía, pero no aumenta aún más te en muestras de ratas tratadas durante 4 días con 30 g/kg de ORY-1001 , confirmando que KDM1A ya estaba totalmente inhibido in vivo.

Figura 4. Compromiso objetivo de KDM1A nativo in vivo y ex vivo. Dosis-respuesta de la participación objetivo de KDM1A en PBMCs (A) y muestras pulmonares (B) de ratas tratadas con ORY-1001 durante 4 días consecutivos (p.o). Se obtuvieron datos de extractos de PPM agrupados de 3 animales por cohorte, analizados por duplicado (N 1, n 2) o de los pulmones de 3 animales individuales por cohorte, analizados en triplicado (N 3, n a 3). C. Comparación de TE en extractos de proteína pulmonar agrupada de ratas tratadas con vehículo (izquierda) o 30 g/kg ORY-1001; después de 1 h de incubación ex vivo de los extractos sin (barras grises) o con 25 nM ORY-1001 (barras azules) (N 3, n a 3). Todos los datos se representan como medios sD. Por favor, haga clic aquí para ver una versión más grande de esta figura.
Discusión
El protocolo presentado aquí fue desarrollado para medir directamente el compromiso objetivo de KDM1A utilizando una nueva captura de quimiosondas KDM1A basada en ELISA. El método ha sido validado en líneas celulares humanas cultivadas y muestras ex vivo de humanos, ratas y ratones y babuinos (incluyendo PPBCM, pulmón, cerebro, piel, tumores), pero se puede aplicar fácilmente a otras especies en las que los epítopos objetivo de anticuerpos KDM1A y catalíticos centro se conservan. Como OG-881 es una quimiosonda basada en la actividad, la calidad de la muestra es importante y se debe llevar a cabo una adecuada manipulación y conservación de las muestras especialmente durante los pasos iniciales del protocolo, para garantizar que se conserte la actividad de KDM1A.
El protocolo experimental actual se optimizó para analizar la participación objetivo de KDM1A mediante inhibidores de la orientación FAD covalentes. También se puede utilizar con inhibidores reversibles que bloquean el acceso al cofactor FAD de KDM1A. Los inhibidores reversibles potentes con largos tiempos de residencia pueden emplear el protocolo no modificado.
La quimiosonda OG-881 puede no ser adecuada para inhibidores reversibles de baja potencia con altas bajas tasas. La quimiosonda particular utilizada en este manuscrito no es penetrante celular y, por lo tanto, los análisis se realizan ex vivo en muestras lysed.
El método se puede ejecutar en instrumentos que están ampliamente disponibles en laboratorios de investigación y análisis; no requiere modificaciones genéticas para ser introducido en las células, y se puede aplicar fácilmente a diferentes tipos de muestras. Otra ventaja es que se puede utilizar en muestras derivadas de diferentes especies que se utilizan con frecuencia en la prueba preclínica de estudios conceptuales y en modelos toxicológicos y que se ha traducido con éxito para analizar muestras clínicas.
Se han utilizado otros métodos para el análisis de la participación objetivo de KDM1A. Muchos de estos métodos utilizan marcadores proxy como cambios de la marca histone H3K4me2, utilizando AlphaLisa15; o la inducción de marcadores de expresión mediante el análisis qRT-PCR o FACS16. Sin embargo, en las células o tejidos, las marcas de histona están controladas por múltiples factores, y los ensayos que miden los cambios en la marca de histona no siempre proporcionan un buen rango dinámico para el análisis. La inhibición de KDM1A puede inducir cambios potentes en la expresión genética y proteica, pero la respuesta tiende a un contexto muy heterogéneo y altamente dependiente de las células, lo que puede complicar los análisis de la respuesta a la dosis3,7.
Por lo tanto, la mejor opción para medir la participación objetivo es la evaluación directa de la ocupación del objetivo. Un ensayo que se ha propuesto para esto es el ensayo de cambio térmico celular (CETSA), basado en el aumento de la estabilidad térmica de las proteínas diana tras la unión de inhibidores. Este método puede, en principio, aplicarse a células no modificadas y diferentes tipos de tejido y recientemente se ha utilizado para evaluar la actividad celular de los inhibidores de KDM1A en células cultivadas17. Sin embargo, esta tecnología rara vez se ha utilizado para estudios de farmacodinámica in vivo18 y hasta el mejor de nuestro conocimiento, su uso no se ha divulgado en ensayos clínicos.
El protocolo proporcionado aquí describe un método totalmente validado basado en quimiosondas que se ha utilizado para determinar la participación objetivo de KDM1A en células y muestras de tejido. El método se ha traducido con éxito para analizar muestras de sujetos humanos tratados con un inhibidor de KDM1A19 y será de gran utilidad para modelar respuestas PK/PD en ensayos clínicos.
Divulgaciones
La autora Tamara Maes es directora ejecutiva y accionista, y las autoras Cristina Mascaró y Raquel Ruiz Rodríguez son empleados de Oryzon Genomics S.A. Oryzon Genomics S.A. desarrolla inhibidores de KDM1A y posee patentes que cubren compuestos y métodos utilizados en este artículo.
Agradecimientos
Este estudio fue financiado por Oryzon Genomics. S.A., Hoffman-La Roche, y parcialmente apoyado por el CIIP-20152001 y el programa de colaboración RETOS RTC-2015-3332-1.
Materiales
| Name | Company | Catalog Number | Comments |
| 0,05% Trypsin-EDTA (1X) | Thermo Scientific | #25300-062 | |
| 10 X Protease Inhibitor Tablets | Roche | #11836153001 | |
| 96 deep well storage block | VWR | #734-1679 | |
| 96 well ELISA plates | Nunc | #436110 | |
| Adhesive black Film | Perkin Elmer | #6050173 | |
| Adhesive transparent Film | VWR | #60941-062 | |
| Biotinylated KDM1A probe OG-881 | Oryzon Genomics S.A. | NA | |
| Bovine Serum Albumin | Sigma | # 3117057001 | |
| Bovine Serum Albumin Standard | Thermo Scientific | #23208) | |
| Bradford Protein Assay | BioRad | #500-0001 | |
| Cell lysis buffer 10X | Cell Signaling | #9803 | |
| Centrifuge for 96- well plates | Hettich | Rotina 420R | |
| Flask | Thermo Scientific | #156499 | |
| Full length, enzymatically active human Recombinant LSD1 / KDM1A | Active Motif | #31426 | |
| Graphpad Prism 5 Project | GraphPad Software | NA | |
| Luminol-Enhacer and Peroxide Solution (Chemiluminescent Substrate) | Thermo Scientific | #37074 | |
| Micro Centrifuge | Eppendorf | 5415 R | |
| Microplate reader Infinite 200-Tecan | Tecan | Infinite 200 | |
| Mouse monoclonal capture antibody Anti-KDM1A (N-terminal epitope) | Abcam | #ab53269 | |
| Needle G18 gauge blunt | BD | #303129 | |
| ORY-1001 (iadademstat) | Oryzon Genomics S.A. | NA | |
| PBMC separation tubes 10 ml | Greiner bio-one | #163288 | |
| PBMC separation tubes 50 ml | Greiner bio-one | #227288 | |
| PBS 1x | Sigma | #D8537 | |
| Plate shaker | Heidolph Instruments | Rotamax 120 | |
| Polysorbate 20 | Sigma | #P7949 | |
| Rabbit monoclonal detection antibody Anti-KDM1A (C-terminal epitope) | Cell Signaling | #672184BF-100 | |
| Secondary antibody Peroxidase-conjugated Donkey Anti-rabbit IgG | Thermo Scientific | #31458 | |
| Spectrophotometer cuvette 1.5 | Deltalab | #302100 | |
| Spectrophotometer for cuvette | GE Healthcare | GeneQuant 1300 | |
| Streptavidin | Promega | #Z704A | |
| Syringe | BD | #303172 | |
| Type 1 ultrapure water | Millipore | Milli-Q Advantage A10 | |
| Ultrasonic cleaner | VWR | USC200T |
Referencias
- Shi, Y. Histone demethylation mediated by the nuclear amine oxidase homolog LSD1. Cell. 119 (7), 941-953 (2004).
- Maiques-Diaz, A., Somervaille, T. C. LSD1: biologic roles and therapeutic targeting. Epigenomics. 8 (8), 1103-1116 (2016).
- Maes, T. ORY-1001, a Potent and Selective Covalent KDM1A Inhibitor, for the Treatment of Acute Leukemia. Cancer Cell. 33 (3), 495-511 (2018).
- Sugino, N. A novel LSD1 inhibitor NCD38 ameliorates MDS-related leukemia with complex karyotype by attenuating leukemia programs via activating super-enhancers. Leukemia. 31 (11), 2303-2314 (2017).
- Kleppe, M., Shank, K., Efthymia, P., Riehnhoff, H., Levine, R. L. Lysine-Specific Histone Demethylase, LSD1, (KDM1A) As a Novel Therapeutic Target in Myeloproliferative Neoplasms. Blood. 126, 601(2015).
- Jutzi, J. S., et al. LSD1 Inhibition Prolongs Survival in Mouse Models of MPN by Selectively Targeting the Disease Clone. HemaSphere. 2 (3), 54(2018).
- Mohammad, H. P. DNA Hypomethylation Signature Predicts Antitumor Activity of LSD1 Inhibitors in SCLC. Cancer Cell. 28 (1), 57-69 (2015).
- Rivers, A., et al. RN-1, a potent and selective lysine-specific demethylase 1 inhibitor, increases γ-globin expression, F reticulocytes, and F cells in a sickle cell disease mouse model. Experimental Hematology. 43 (7), 546-553 (2015).
- Rivers, A. Oral administration of the LSD1 inhibitor ORY-3001 increases fetal hemoglobin in sickle cell mice and baboons. Experimental Hematology. 67, 60-64 (2018).
- Buesa, C., et al. The dual LSD1/MAO-B inhibitor ORY-2001 prevents the development of the memory deficit in samp8 mice through induction of neuronal plasticity and reduction of neuroinflammation. Alzheimer’s & Dementia. 11 (7), P905(2015).
- Schmidt, D. M., McCafferty, D. G. Trans-2-Phenylcyclopropylamine is a mechanism-based inactivator of the histone demethylase LSD1. Biochemistry. 46 (14), 4408-4416 (2007).
- Forneris, F., Binda, C., Vanoni, M. A., Battaglioli, E., Mattevi, A. Human histone demethylase LSD1 reads the histone code. Journal of Biological Chemistry. 280 (50), 41360-41365 (2005).
- Oryzon Genomics, Methods to determine kdm1a target engagement and chemoprobes useful therefor. , WO2017158136 (2016).
- Mascaró, C., Ortega, A., Carceller, E., Rruiz Rodriguez, R., Cicero, F., Lunardi, S., Yu, L., Hilbert, M., Maes, T. Chemoprobe-based assays of histone lysine demethylase 1A target occupation enable in vivo pharmacokinetics and -dynamics studies of KDM1A inhibitors. Journal of Biological Chemistry. , In Press (2019).
- Rodriguez-Suarez, R. Development of Homogeneous Nonradioactive Methyltransferase and Demethylase Assays Targeting Histone H3 Lysine 4. Journal of Biomolecular Screening. 17 (1), 49-58 (2011).
- Lynch, J. T., Cockerill, M. J., Hitchin, J. R., Wiseman, D. H., Somervaille, T. C. CD86 expression as a surrogate cellular biomarker for pharmacological inhibition of the histone demethylase lysine-specific demethylase 1. Analytical Biochemistry. 442 (1), 104-106 (2013).
- Schulz-Fincke, J. Structure-activity studies on N-Substituted tranylcypromine derivatives lead to selective inhibitors of lysine specific demethylase 1 (LSD1) and potent inducers of leukemic cell differentiation. European Journal of Medicinal Chemistry. 144, 52-67 (2018).
- Ishii, T., et al. CETSA quantitatively verifies in vivo target engagement of novel RIPK1 inhibitors in various biospecimens. Scientific Report. 7, 13000(2017).
- Maes, T. ORY-2001: An Epigenetic drug for the treatment of cognition defects in Alzheimer’s disease and other neurodegenerative disorders. Alzheimer’s & Dementia. 12 (7), P1192(2017).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados