Method Article
Aplicação de Laser Microirradiação para exame de Single e Double Strand reparação em células de mamíferos
Neste Artigo
Resumo
Fluorescência confocal laser e microscopia microirradiação oferta ferramentas para indução de dano do ADN e monitoramento da resposta das proteínas de reparo de DNA em áreas selecionadas do sub nucleares. Esta técnica tem significativamente nosso conhecimento avançado de detecção de danos, sinalização e recrutamento. Este manuscrito demonstra essas tecnologias para examinar reparação simples e dupla vertente.
Resumo
Vias de reparo de DNA altamente coordenadas existem para detectar, impostos especiais de consumo e substituir as bases de DNA danificadas e coordenar a reparação de quebras da cadeia de DNA. Enquanto as técnicas de biologia molecular clarificaram a estrutura, funções enzimáticas e cinética de proteínas de reparo, ainda há uma necessidade de compreender como o reparo é coordenado dentro do núcleo. Microirradiação do laser oferece uma poderosa ferramenta para indução de dano do ADN e o recrutamento de proteínas de reparo de monitoramento. Indução de dano do ADN por microirradiação do laser pode ocorrer com uma gama de comprimentos de onda, e os usuários confiável podem induzir quebras da cadeia única, base lesões e quebras da cadeia dupla com uma gama de doses. Aqui, microirradiação do laser é usado para examinar o reparo de single e double quebras induzidas por dois comum do laser confocal comprimentos de onda, 355 nm e 405 nm. Além disso, a adequada caracterização da dose do laser aplicado por indução de misturas de dano específico é descrita, assim os usuários podem reproducibly executar análise e aquisição de dados de microirradiação do laser.
Introdução
Microscopia fluorescente tem emergido como uma técnica poderosa para visualizar a arquitetura celular, examinar a localização da proteína e monitorar as interações da proteína-proteína e proteína-ADN. Usando microscopia fluorescente para estudar respostas de dano de DNA após a aplicação do DNA global agentes nocivos, como luz ultravioleta (UV), radiação ionizante, oxidantes químicos ou agentes alquilantes, e/ou chemotherapeutics tem proporcionado a introspecção nova a iniciação, sinalização e recrutamento de DNA reparar proteínas para sites de DNA danos1,2. No entanto, estes global e assíncrono danificar eventos estão limitando, se informações detalhadas sobre a ordem de recrutamento, cinética da associação ou dissociação, e as relações entre as principais proteínas de reparo de DNA são procuradas. Felizmente, os avanços na exploração microscópios confocal do laser, mais ampla disponibilidade de comprimentos de onda do laser danos de indução e melhorias em proteínas fluorescentes nos últimos 25 anos forneceram pesquisadores com ferramentas melhoradas para examinar estas reparação de aspectos do DNA, através da indução de dano de DNA alvo.
Irradiação de células com laser microbeams fim de estudar as funções celulares e subcelulares é uma ferramenta bem estabelecida na célula e radiação de biologia3. Aplicação desta técnica para o estudo da reparação do DNA surgiu quando Cremer e colegas de trabalho usaram um UV altamente focada (257 nm) sistema de microbeam laser para induzir dano do ADN sobre um 0,5 µm em ovário de hamster chinês (CHO) local células4 e estabeleceu a indução de DNA photolesions por este sistema5. Enquanto oferecer melhorias significativas sobre os métodos de UV microbeam aquando da adopção da presente, danificando o sistema foi limitada devido à sua criação especializada e sua incapacidade de gerar dupla vertente quebra (DSBs)6. Investigações subsequentes de uma variedade de UV-B (290-320 nm) e UV-um comprimento de onda (320-400 nm) por um número de grupos revelou que photoproducts UV, oxidativos baseiam lesões, único filamento rompe (SSBs) e DSBs poderiam ser induzidas depende do comprimento de onda do laser e energia aplicada4,7,8,9,10 (revisto em 3). Além disso, combinações destes UV-B e UV-A comprimentos de onda com sensibilizante de agentes, como o psoraleno, bromodeoxyuridine (BrdU) e Hoechst corantes, também foram encontradas para induzir dano de DNA dependente de comprimento de onda, potência e duração da exposição, embora o poder necessário para induzir o dano é muitas vezes reduzido na presença destes agentes11,12,13,14,15. Esses avanços expandiu o uso de microirradiação, embora houvesse obstáculos técnicos ainda de ser abordada por uma maior adopção desses métodos.
Cremer e colegas de trabalho avançaram significativamente o campo de irradiação de micro centrando-se precisamente o microbeam UV para aplicar energia prejudicial significativas ao longo de uma área altamente localizada na célula. Como microscópios confocal e sistemas de laser microdissection avançaram, luz firmemente focalizada foi mais amplamente acessível; no entanto, fontes UV de acoplamento para escopos e lidar com as aberrações cromáticas, que eles induziram ainda apresentaram um desafio significativo para a maioria dos usuários3,6,16. Como tintas UV aumentaram em popularidade durante a década de 1990, óptica capaz de se concentrar e captura de fluorescência UV-animado tornou-se mais amplamente disponível16, e melhorias na exploração do laser ofereceram aos usuários a capacidade de criar altamente focada UV pontos de excitação dentro células6,17. No entanto, não foi até o início dos anos 2000 que o verdadeiro impacto desta combinação de feixes firmemente focalizadas com lasers de intensidade mais elevadas foi sentido, quando inúmeros relatos emergiram demonstrando que quebras da cadeia de DNA poderiam ser induzidas com e sem sensitizers na UV-A gama6,10,18,19,20, 405 nm21,22,23,24, 25,,26e até mesmo a mais visíveis comprimentos de onda como 488 nm27. Esses avanços permitidos para mais ampla adoção da técnica da microirradiação em um número de sistemas comerciais. Paralelamente a estes desenvolvimentos, técnicas de dois fotões também emergiram que permitiu precisa indução de dano do ADN; Embora esses avanços não serão discutidos aqui, há um número de artigos de revisão, discutindo essas metodologias9,28,29,30.
Com a acessibilidade atual de microscópios confocal capazes de proporcionar altamente focalizado de luz UV e a disponibilidade generalizada de proteínas fluorescentes para permitir o acompanhamento em tempo real das proteínas de reparo do DNA, técnicas de microirradiação têm evoluído para ferramentas poderosas para examinar caminhos de resposta e a reparação de danos de DNA. No entanto, os usuários precisam estar cientes de que a geração de dano do ADN é altamente dependente do comprimento de onda do laser e a energia aplicada à região sub nuclear. Uso de UV-C (~ 260 nm) comprimentos de onda permitem excitação direta de DNA e alta seletividade para a indução de UV photoproducts7,8. UV-B e UV-A comprimentos de onda produzem misturas de dano do ADN (lesão de base, SSBs e DSBs), dependente da energia aplicada e o fundo celular utilizada7. Fotossensibilizadores endógenas e níveis de antioxidante nas células alvo podem influenciar as misturas de dano de DNA produzidas por estes comprimentos de onda. Além disso, o uso de fotossensibilizadores exógenos (BrdU, etc.) pode ser úteis em reduzir a energia necessária para a indução de dano do ADN. No entanto, esses agentes podem induzir dano do ADN por si mesmos, e eles podem alterar o ciclo celular e a estrutura da cromatina, assim que seu uso pode produzir efeitos indesejados que precisam ser consideradas pelo usuário. Portanto, antes de usar o microirradiação para estudar reparação e resposta de dano do ADN, consideração cuidadosa da via de reparo de DNA de interesse, os comprimentos de onda disponíveis para uso e a mistura de dano de DNA criado é necessária.
Aqui, o microirradiação do laser é realizada em dois comprimentos de onda usados, 355 nm e 405 nm, sem sensitizers para demonstrar as misturas de danos no DNA induzidos por estes comprimentos de onda e as influências dessas misturas de danos têm em examinar a reparação deSSBs e DSBs. os usuários devem estar cientes de que estes comprimentos de onda não criar uma única espécie de quebras ou lesões de base. Para discriminar entre vias de reparo do DNA, usuários cuidadosamente deverá controlar a força aplicada sobre uma região específica do núcleo e caracterizar o dano induzido usando vários marcadores de quebra de fio e anticorpos de lesão do DNA. Se adequadamente aplicada e caracterizada, microirradiação do laser pode enriquecer algumas espécies de dano do ADN, que permite aos usuários avaliar o reparo de lesões de base e SSBs ou DSBs, com alguma especificidade. Portanto, nós fornecemos um método que permite aos usuários executar microirradiação do laser reproducibly, caracterizar as misturas de dano do DNA induzidas pela dose aplicada do laser e realizar a análise de dados.
Protocolo
1. cultura de células e a geração de células estáveis
- células CHO-K1 crescem em meio essencial mínimo suplementado com 10% de soro fetal bovino. Manter as células em uma incubadora umidificada com 5% CO 2 a 37 ° C.
- Células CHO-K1 transfect com 1 µ g de DNA de plasmídeo contendo XRCC1 humano com um C-terminal verde marca proteína fluorescente (GFP), usando um reagente de transfeccao comercial seguindo o fabricante ' instruções de s.
- CHO-K1 selecionar células estàvel expressando a fusão XRCC1-GFP humana usando 800 µ g/mL geneticin e enriquecer a população expressando GFP-XRCC1 usando fluorescência assistida célula classificação (FACS).
- Células de chapa a ser micro irradiados em vasos de cultura com fundos de lamela, assim que atingem aproximadamente 75% ou maior confluência no dia seguinte. Alguns espaços entre as células são desejáveis, especialmente se usando o registro para retornar para o mesmo campo de imagem (descrito na secção 3.2).
2. Set-up microscópio
- Selecione uma varredura a laser confocal microscópio equipado com laser adequado e óptica e software de controle para microirradiação / photostimulation. Apresentou experiências uso um laser confocal microscópio que foi modificado para incluir um 355 nm do laser, fibra-acoplado a um miniscanner de fotoativação do galvanômetro e controlado usando o fabricante ' software de controle e análise de s. Este sistema pode realizar microirradiação numa região de interesse (ROI) designado pelo usuário usando o photoactivation miniscanner (355 nm do laser) ou o galvanômetro confocal padrão (laser de 405 nm).
- Selecionar o comprimento de onda a ser usado para microirradiação, garantindo que todos os componentes ópticos no lightpath microirradiação são apropriados para o comprimento de onda escolhido.
Nota: O sistema apresentado usa um objectivo de imersão de óleo C-Apochromat (abertura numérica (NA) 1.2) 40 × juntamente com um cubo de filtro UV para 355 nm microirradiação e um objectivo seco de 20 × C-Apochromat (at 0.75) usando o padrão lightpath confocal para 405 nm microirradiação. O objectivo × 20, usado na instalação não é compatível com o comprimento de onda nm 355; Portanto, usamos o × 40 para 355 nm só. Com o objetivo de seco para danificar permite que protocolos de imunofluorescência ser aplicada a jusante sem limpar óleo de imersão, é por isso que nós utilizamos este objectivo quando possível. - Configurar o microscópio para o experimento de microirradiação. Para manter a consistência entre as experiências, designe uma configuração padronizada dos componentes do microscópio deve ser usado para cada tipo de microirradiação. Salvar essas configurações como uma predefinição dentro do microscópio ' s software, operacional, se possível.
Nota: No sistema apresentado, as células são microirradiadas e fotografada usando varredura unidirecional, resolução de 1024 x 1024 pixels com 1 x zoom de digitalização, digitalização em uma taxa de quadros de 8 quadros por segundo (fps) e com o pinhole definidas como aproximadamente 4 unidades arejado (AU), como determinado para o laser 488 nm (69 µm). Essa configuração de pinhole aberto foi escolhida para maximizar a quantidade de luz capturada em cada imagem, permitindo o uso da imagem latente do laser poderes inferiores e reduzindo o fotobranqueamento.
3. Microirradiação de laser
prato de cultura- lugar o preparado, contendo células de interesse, para a fase de microscópio e trava firmemente no lugar.
- , Se disponível, coloque slide septada em uma incubadora de palco-top e manter a 37 ° C com 5% CO 2 durante a indução de danos. Esta etapa é mais importante para viver-pilha timelapse de imagem ou imagem tempo sessões. Caso contrário lugar células no microscópio estágio e realizar irradiação à temperatura ambiente (~ 25 ˚ c) e em seguida retornar rapidamente as células para uma incubadora a 37 ° C com 5% de CO 2.
- Registrar campos de imagem para permitir que o pesquisador retornar ao mesmo local após a realização de fixação, coloração ou outros procedimentos. Executar registro de campos danificados usando um palco de microscópio codificado, automatizado ou vasos de lamela de cultura com uma grade gravado ou rotulado de locais XY de célula.
- Se usando o registro de imagem de software, selecione um recurso reconhecível do navio cultura (ou seja, o canto da lamela ou barreira entre poços), recolher uma imagem e registrar a localização de XY. Isto permitirá de alinhamento e registo dos locais dos campos selecionados após a preparação da amostra XY.
- Se usando o registro manual, gravar grade ou gravados locais para cada campo manualmente, tendo o cuidado de gravar a orientação e o posicionamento do prato no palco.
- Se não identificando características para os vasos de cultura de célula estão disponíveis, identificar campos danificados por inspecção visual da morfologia celular e densidade. Tome cuidado para selecionar campos com características suficientemente distintas para serem reconhecidos durante mais tarde imagem.
Nota: Isso pode ser demorado para executar a menos que a orientação e a área do navio cultura é fortemente restrito.
- Selecione um campo para microirradiação e concentrar a amostra. Para as células que expressam proteínas fluorescente etiquetadas, selecione o plano focal com secção transversal máximo nuclear no canal fluorescente de interesse. Para células não expressando rotulado de proteínas, ou para aqueles sem localização nuclear claro, contraste de fase ou interferência diferencial (DIC) de contraste imagens podem ser usada para encontrar o plano focal correto.
Nota: uma característica útil de contraste de fase e a imagem latente do DIC é o fato de que, como o plano focal se move através da amostra, a mesma característica pode aparecer clara ou escura, dependendo do plano focal relativo.- Selecione uma característica clara dentro do núcleo, tal como o nucléolo e mova o plano focal acima e para baixo enquanto observa esta mudança na aparência. O verdadeiro plano focal caberá dentro a transição da luz à obscuridade. Para concentrar a amostra, selecione o plano focal na qual o recurso selecionado tem o nítida contraste.
- Registrar a posição do campo de interesse. Criar um quadrado 3x3 pixel ROI dentro do software de microscópio, coloque este ROI sobre o núcleo de uma célula a ser danificada e definir este ROI o dano ROI.
- Recolher uma imagem pré-dano, incluindo a posição dos danos ROI.
- Para experimentos que não usam proteínas fluorescentes, adquirir um contraste de fase ou diferencial interferência contraste (DIC) brightfield imagem para identificar e registrar núcleos para danos.
- Para viver-pilha experimentos usando proteínas fluorescentes, adquirir uma imagem contendo brightfield e o canal de fluorescência para a proteína de interesse (ou seja, laser linha 488 nm para a excitação de GFP, laser de linha 561 nm para a excitação de RFP). Em experimentos usando o CHO-K1 XRCC1-GFP, animado pela linha 488 nm laser de fluorescência simultaneamente foi coletada com o canal de brightfield transmissível DIC.
- Iniciar danos de laser.
- No sistema apresentado, controle da dose do laser pelo tempo total gasto os danos ROI de digitalização. Porque o galvanômetro para a 355 nm do laser opera a uma taxa de varredura fixo, dose do laser é controlado pelo tempo gastado varredura repetidamente o dano selecionado ROI: os dados apresentados aqui, microirradiação em 355 nm é executada para 2 e 10 segundos.
- Por outro lado, controlar a dose de laser 405 nm, modulando a taxa de varredura e executar uma verificação dos danos selecionado ROI. No dados pressentiu-se aqui, use 8 e 0,5 fps para microirradiação em 405 nm. Uma taxa de varredura de fps 8 proporciona uma dose de laser menor do que 0,5 fps, porque o laser gasta menos tempo em cada pixel durante a verificação. Ambos os lasers são operados em 100% de energia. Ver secção 3.7 para obter instruções de medição da potência do laser diretamente.
Nota: Cada sistema de microscópio individuais pode diferir em como poder do laser é entregue a um ROI designado, similar a como a 355 nm e 405 nm diferem no sistema apresentado. Os usuários precisarão determinar este procedimento para o seu sistema de microscópio e relatar estes parâmetros dentro de suas seções métodos. - Para geração de imagens de células vivas, realizar a aquisição de imagem timelapse de canais brightfield e fluorescência. Ajuste a duração e a frequência de timelapse para otimizar a coleta de dados, idealmente, capturando o acúmulo da proteína fluorescente o dano ROI e sua dissociação ao longo do tempo do experimento. Experimentos utilizando XRCC1-GFP coletadas imagens a cada 30 segundos por 20 minutos.
Nota: A frequência de imagem o timelapse pode ser limitada por fotobranqueamento de fluoróforo, complicando a análise de acumulação e dissociação. Fotobranqueamento pode ser avaliado observando células defeituosas durante o timelapse e ajustando as condições de aquisição para minimizar a perda de sinal fluorescente nestas células defeituosas. Alguns fotobranqueamento pode ser inevitável, para que os usuários podem compensar a perda de sinal, normalizando as intensidades fluorescentes de células danificadas das células defeituosas em cada quadro do timelapse.- Após a conclusão do curso do tempo, selecione um novo campo de células por danos ou reparar células danificadas para posterior análise, conforme descrito na seção 4. Timelapse de imagem até que seja atingido o número desejado de células danificadas e
- continuar microirradiação.
Nota: Recomendamos que danificar um total de 10-25 células por condição selecionada a fim de avaliar a heterogeneidade de célula para célula em resposta aos danos induzidos. Embora mais confocal sistemas permitirá que os usuários danificar mais de uma célula durante cada microirradiação, isto apresenta tempos de iniciação de danos escalonada, que podem complicar a análise do tempo de recrutamento de pico ao longo de um grande número de células ou tempo de dissociação para eventos rápidos. Em alguns sistemas, indução de danos simultâneos sobre vários ROIs pode reduzir a dose de laser recebida por cada ROI independente. Portanto, a menos que estas questões de poder e sincronismo são abordadas diretamente pelo usuário, é recomendável que as células individualmente danificado.
- Para executar a análise por imunofluorescência (IF), danos e também corrigir imediatamente as células, conforme descrito na seção 4, ou permitir que as células reparar para selecionado incrementos de tempo (ou seja, 1, 5, 10 ou 20 min). Depois de decorrido o tempo desejado, corrigir e mancha as células, conforme detalhado na seção 4.
- Para aumentar o número total de células para análise se, danificar campos adicionais dentro do navio de cultura para gerar um curso multi campo tempo de pós-dano resposta. Registre a localização de XY de cada campo e o tempo que o dano ocorreu. Após o total desejado após decorrido o tempo de reparação, corrigir e manchar as células conforme detalhado abaixo.
- Após observar de recrutamento de proteína em doses selecionados, medida e relatório do laser poder níveis pós-fibra e pós-objetivo com precisão caracterizar e relatar a laser doses. Utilizamos um medidor de energia digital compacto e duas cabeças de diferentes fotodiodo sensor; um acoplado diretamente para a fibra de laser para medir a saída pós-fibra, e o outro colocado no palco microscópio para medir a saída post-objective.
- Para medir a potência do laser, coloque o sensor na configuração desejada (pós-fibra ou post-objective) e executar microirradiação conforme descrito acima, durante a gravação de medições efectuadas pelo medidor de energia. Lembre-se que a taxa de amostragem do medidor de energia pode não ser rápida o suficiente para capturar eventos de microirradiação muito rápidos, então pode ser necessário executar várias iterações do experimento para garantir que o medidor de energia detecta com precisão a dose aplicada.
Nota: Para a 355 nm do laser, medimos uma potência de pico médio de pós-fibra de cerca de 5 mW e aproximadamente 19 objetivo de pós µW. Para laser 405 nm, microirradiação foi realizada em loops de 30 iterações para superar a taxa de amostragem máxima de 0,01 s do medidor de energia e os poderes de média pico de 1,5 mW e 2.4 mW foram medidos com velocidades de varredura de 0,5 e 8 quadros por segundo , respectivamente. Cada medidor de energia é diferente. Os usuários precisarão determinar os comprimentos de onda operacionais e taxas de amostragem para o seu medidor de poder individual.
- Para medir a potência do laser, coloque o sensor na configuração desejada (pós-fibra ou post-objective) e executar microirradiação conforme descrito acima, durante a gravação de medições efectuadas pelo medidor de energia. Lembre-se que a taxa de amostragem do medidor de energia pode não ser rápida o suficiente para capturar eventos de microirradiação muito rápidos, então pode ser necessário executar várias iterações do experimento para garantir que o medidor de energia detecta com precisão a dose aplicada.
4. Mancha de procedimentos
- indução de pausa Analyze vertente se manchando.
- Células de correção com 3,7% de formaldeído (cuidado) em fosfato tampão salino (PBS) para solução de formaldeído 10 min. aspirar e lavar 3 vezes com PBS. O protocolo pode ser parado aqui colocando PBS volta sobre as células e as células podem ser armazenadas no 4 ˚ c por até 1 semana.
Atenção: O formol é tóxico e cancerígeno. Usar equipamento de proteção pessoal adequado e eliminar a substância tóxica como instruído por procedimentos institucionais de saúde e segurança ambientais. - Permeabilize pilhas usando 0,25% Triton X-100 em PBS por 10 minutos em temperatura ambiente (RT) e em seguida lavar 3 vezes com PBS.
- Ligação de anticorpo específico bloco incubando por 30 min em PBS contendo 1% albumina de soro bovino (BSA) em RT.
- Incubar com primário monoclonal antibody contra γH2AX e primário anticorpo policlonal contra 53BP-1, ambos diluído 1:750 em PBS contendo 1% de BSA para 1h no RT e em seguida lavagem 3 vezes com PBS.
- Incubar células com Alexa 488 cabra anti-mouse e Alexa 546 de cabra anticoelho tanto diluído em PBS contendo 1% de BSA para 1h no RT 1: 2000 e em seguida lavar 3 vezes com PBS.
- Mancha DNA nuclear com uma solução de 10 mg/mL de DAPI (4 ', 6-Diamidino-2-Phenylindole, cuidado) diluído para 1:5000 em PBS por 5 min, ou uso de corante nuclear comparáveis e em seguida lavar 3 vezes com PBS.
Cuidado: DAPI é tóxico e mutagênico. Usar equipamento de proteção pessoal apropriado e eliminar a substância tóxica como instruído por procedimentos institucionais de saúde e segurança ambientais. - Lugar PBS ou PBS + 0,1% de azida de sódio (cuidado) sobre as células manchadas. Protocolo pode ser parado aqui e células armazenadas no 4 ˚ c durante vários dias, ou proceder à aquisição de imagens na secção 5.
Atenção: azida de sódio é tóxica. Usar equipamento de proteção pessoal apropriado e eliminar as substâncias tóxicas como instruído por procedimentos institucionais de saúde e segurança ambientais.
- Células de correção com 3,7% de formaldeído (cuidado) em fosfato tampão salino (PBS) para solução de formaldeído 10 min. aspirar e lavar 3 vezes com PBS. O protocolo pode ser parado aqui colocando PBS volta sobre as células e as células podem ser armazenadas no 4 ˚ c por até 1 semana.
- Analisar indução de lesões de base ou DNA adutos se manchando.
- Fix e permeabilize células em metanol gelada (cuidado) por 20 min em -20 ° C.
Atenção: O metanol é tóxico e inflamável. Usar equipamento de proteção pessoal apropriado e eliminar a substância tóxica como instruído por procedimentos institucionais de saúde e segurança ambientais. - , Aspire a mimthanol e permitir que a amostra secar completamente durante 15 min. de protocolo pode ser parada aqui e células conservadas a-20 ˚ c secar por até 3 dias.
- Hidratar as células em PBS por 15 min.
- Denature DNA usando 2 N HCl (cuidado) por 45 min em RT e lavagem 3 vezes com PBS.
Atenção: O HCl é corrosivo. Usar equipamento de proteção pessoal apropriado e eliminar a substância corrosiva conforme instruído por procedimentos institucionais de saúde e segurança ambientais. - Neutralizar em pH 50 mM Tris-HCl 8,8 por 5 min, seguido de 3 lavagens com PBS.
- Células de incubar em tampão de bloqueio feito com soro de cabra normal de 5% e 0,1% Triton X-100 em PBS por 1h no RT.
- Incubar com anticorpos primários para 8-oxo-2´-Desoxiguanosina (oxodG-8, 1: 400) ou ciclobutano dímero de pirimidina (CPD, 1:1, 000) em tampão de bloqueio do soro a cabra por 1h no RT e em seguida lavar 3 vezes com PBS.
- Incubar células com Alexa 488 anti-rato cabra na proporção de 1: 2000 no buffer de bloqueio do soro de cabra por 1h no RT e lavar 3 vezes com PBS.
- Mancha DNA nuclear usando 1:5000 DAPI (10 mg/mL) em PBS por 5 min e lave 3 vezes com PBS.
- Se utilizar lamela septadas, coloque PBS ou PBS + 0,1% de azida de sódio sobre as células manchadas. Protocolo pode ser parado aqui e células armazenadas no 4 ˚ c durante vários dias, ou proceder à aquisição de imagens na secção 5.
- Alternativamente, montar células em meio de montagem preferencial, se uma lamela adicional pode ser colocada em cima do navio de cultura. Uma vez curados, montado as células podem ser armazenadas no 4 ˚ c por longos períodos de tempo.
- Fix e permeabilize células em metanol gelada (cuidado) por 20 min em -20 ° C.
5. Aquisição de experiências de imunofluorescência de imagem
- limpar o fundo da lamela da embarcação cultura com etanol e colocá-lo de volta ao palco microscópio confocal. Certifique-se que a orientação e a posição do navio no palco corresponde à orientação e posição gravada quando o dano foi induzido. Proteger o navio de cultura para garantir o registo ideal e imagem.
- Localizar previamente fotografada campos usando a técnica de registro selecionado (descrito em 3.2).
- De registo manual usando uma lamela com uma grade gravada, trazer a grade em foco e encontrar marcas de identificação. Então uso gravado coordenadas XY para localizar as células danificadas.
- Para registro automatizado baseado em imagem, localize a característica estrutural utilizada como referência na etapa 3.2.1 e em seguida alinhe a vista ao microscópio atual tão atentamente para a imagem gravada quanto possível. Uma vez alinhados, medir o local de estágio de microscópio XY atual e compará-lo com a localização de XY da imagem de referência. A distância linear entre os dois locais define o deslocamento X e Y. Aplica esse deslocamento para cada local de XY gravado para identificar o campo de imagem (s) que contém a célula irradiada. Esta função de deslocamento pode ser automatizada no software de controle do microscópio ou executada manualmente.
- Se não há pontos de registo adequado poderiam ser identificados, localizar manualmente as células analisando o slide e localizar as características identificadas anteriormente célula.
- Adquirir imagens usando configurações de microscópio apropriado para coletar tudo manchado de alvos, incluindo uma imagem de brightfield (configuração na seção 2.3 e centrando-se na seção 3.3). Nos experimentos apresentados, imagens multicanais foram coletadas usando as seguintes linhas de laser de excitação e fluorophores: 405 nm (DAPI), 488 nm (Alexa 488 e transmitida brightfield DIC) e 561 nm (Alexa 546). Todos os lasers passam através de um único filtro óptico-acústico sintonizável, controlando a transmissão de poder e de comprimento de onda do laser.
6. Análise de imagem de célula do recrutamento de proteínas fluorescentes para sites micro irradiados de viver
- Open adquiridas imagens em um aplicativo de análise de imagem (ou seja, elementos de NIS ou ImageJ). Se necessário, combine a imagem pré-irradiação com timelapse imagens para gerar uma sequência de imagem única da indução de danos pré e pós. Os usuários podem mostrar recrutamento medindo mudanças na intensidade fluorescente ao longo da área irradiada relativo à intensidade de fluorescência, medido através do núcleo de todo (ver resultados representante Figura 1 B. ).
- Para cada célula a ser medido, primeiro gerar uma referência ROI que representa o núcleo. Usar um algoritmo de limiarização do sinal fluorescente que contém os pixels que compõem o núcleo e depois converter esta área em um ROI. Se necessário, o ROI pode ser estabelecido manualmente usando o brightfield como referência. Ajustar o ROI ao longo do tempo para garantir que a área nuclear com precisão é coberta pelo ROI e relatar a intensidade média de fluorescência do sinal fluorescente dentro este ROI para cada frame.
- Para cada célula a ser medido criar um 6x6 pixel ROI e coloque sobre o ROI de dano para cada quadro do curso de tempo. Esta maior ROI é agora o dano ROI para análise. Relatório a média intensidade de fluorescência de ROI para cada frame.
Nota: O software de análise de imagem mais comercial contém módulos para rastreamento de objeto 2D, e há um número de macros desenvolvidos pelo usuário para ImageJ ou FIJI que facilitam o fluxo de trabalho mais rápido e maior rendimento (ver https://imagej.nih.gov/ij/plugins/). - Normalizar a intensidade de fluorescência de ROI médio dano àquele de uma correspondente referência ROI em cada quadro do timelapse. Aqui, a intensidade da fluorescência nuclear média é usada como referência o ROI (ver resultados representante Figura 1). normalização pode ser executada subtraindo-se a referência de média intensidade de fluorescência de ROI da média danificar a intensidade de fluorescência de ROI, ou dividindo-se a média danificar ROI intensidade de fluorescência pela referência de média intensidade de fluorescência ROI.
Nota: A normalização pela divisão pode produzir resultados imprevisíveis, se usado em situações com valores de referência de baixíssima intensidade. - Repetir para todas as células danificadas, bem como para controle de pelo menos dois, células defeituosas. Resultados de célula de controle podem ser usados para normalização, ainda mais, se necessário.
- Gráfico normalizado valores de intensidade ao longo do tempo para mostrar as mudanças na dinâmica de recrutamento em função do tratamento experimental.
7. Análise do recrutamento de proteína detectado por imunofluorescência de imagem
- abrir imagens pré e pós-danos em um aplicativo de análise de imagem. As imagens de pre-danos contenham os danos ROI(s) usado para microirradiação. O ROI(s) de copiar e colar para as imagens de pós-danos para identificar células alvo.
- Gerar um pixel de 6x6 ROI para análise de recrutamento de proteínas e coloque este ROI no local do dano ROI. Esta maior ROI é agora o dano ROI para análise. Relatar os danos de média intensidade de fluorescência ROI.
- Normalizar a intensidade de fluorescência de ROI médio dano àquele de uma ROI de referência correspondente. Aqui, a intensidade de fluorescência nuclear média da célula danificada é usada como referência o ROI (ver resultados representante Figura 1). Gerar a referência ROI usando um algoritmo de limiarização do sinal de DAPI para definir o núcleo e depois converter esta área para um ROI. Apresente-se a referência de média intensidade de fluorescência de ROI. Normalização pode ser executada subtraindo-se a referência de média intensidade de fluorescência de ROI do dano média intensidade de fluorescência de ROI, ou dividindo-se o dano de média intensidade de fluorescência de ROI pela referência de média intensidade de fluorescência ROI.
Nota: A normalização pela divisão pode produzir resultados imprevisíveis, se usado em situações com valores de referência de baixíssima intensidade. - Repita para todas as células danificadas e realizar a mesma análise em células de controle de pelo menos dois, conforme descrito acima.
- Gráfico normalizado valores de intensidade para cada evento microirradiação uma medida contra o tempo para mostrar as mudanças no recrutamento de proteínas ou tipo de dano do ADN em função do tratamento experimental.
Resultados
Caracterização de danos no DNA induzidos
Indução de lesões de base e quebras é dependente da dose do laser aplicada à área selecionada nuclear e o microambiente celular do modelo celular usado7. Proteínas fluorescentes fundidas para reparar as proteínas, como XRCC1, 53BP1, Ku70 ou Rad51, fornecer úteis single e double vertente marcadores de intervalo para estabelecer a energia mínima necessária para ver o acúmulo de uma proteína fluorescente dentro um dano ROI acima o fluorescência do fundo9,19,31. Uma vez que encontram-se as condições que induzem uma resposta, é fundamental para caracterizar a mistura de danos induzida por esse comprimento de onda específico e a dose. Atenuação da dose e duração no comprimento de onda usado pode permitir que o usuário minimizar a formação de misturas complexas de danos. Doses baixas do laser na faixa do UV-A tem demonstrados para produzir predominantemente SSBs e uma pequena quantidade de base lesões, apropriadas para estudar SSBR e BER vias10,28. Aumentando a dose cria lesões de base mais complexas, oxidativas e UV induzida e induz a um número mais significativo de DSBs7,10. Enquanto a indução de uma única espécie de dano do ADN é desejável para exame das vias de reparo de DNA específicas, é mais provável que os usuários estão induzindo uma mistura de lesões do DNA, com uma lesão específica como SSBs, sendo muito mais frequentes do que as lesões de base ou DSBs. Isso é semelhante às misturas de dano do DNA induzidas por agentes químicos, como o peróxido de hidrogênio (H2O2) ou metil Metanossulfonato (MMS)32. Os usuários precisam estar ciente de quando eles relatam resultados que danificam as misturas podem ocorrer e caracterização cuidadosa da dose e lesões no local do dano induzido são necessárias para assegurar a reprodutibilidade e a comparabilidade dos seus resultados.
Em estudos de microirradiação, XRCC1-GFP é muitas vezes usado como um marcador para a indução de lesões de base e SSBs9,28. XRCC1 é uma proteína de andaime que desempenha um papel importante na reparação SSB (SSBR) e excisão base reparar (BER) e também participa de outras vias de reparação, como nucleotídeos excisão reparação (NER)33,34,35 . Que desempenha um importante papel de coordenação na reparação do ADN, interagindo com um número de proteínas chaves, incluindo poly(ADP-ribose) polimerase 1 (PARP-1), DNA polimerase β (Pol β) e DNA ligase III. Utilizamos o XRCC1-GFP estàvel expressado em células CHO-K1 para determinar as doses do laser necessárias para gerar SSBs e DSBs. Primeiro identificamos a dose mínima necessária para induzir um recrutamento observável de XRCC1-GFP para cada comprimento de onda (Figura 1). Para a 355 nm comprimento de onda uma 2 tempo de interrupção de s sobre o dano definido ROI gerado um aumento sinal fluorescente dentro o ROI, indicando a indução de dano do ADN que era detectável sobre o fundo (Figura 1A). Para 405 nm, uma taxa de varredura de 8 fps era necessária para gerar um recrutamento observável ao dano ROI (Figura 1A). A dose foi então aumentada (10 s para 355 nm e 0,5 fps para 405 nm) para criar um mais intenso danificar ROI (Figura 1A).
Recrutamento e retenção de XRCC1-GFP no local do dano induzido então foi monitorada por imagens de timelapse. Retenção da proteína no local do dano ao DNA pode indicar reparo de ADN em curso, enquanto a dissociação das proteínas no site do dano induzido é muitas vezes considerada um marcador para conclusão de BER ou SSBR. No entanto, não houve nenhuma evidência clara vinculação a dissociação de XRCC1 de sites de dano de DNA induzida por laser, com a conclusão da reparação. Recrutamento da proteína para o local do dano é medido através do relato da intensidade média do sinal fluorescente dentro o ROI danificado sobre o sinal fluorescente médio medido para o núcleo inteiro (Figura 1B). Este tipo de normalização ajuda flutuações de intensidade endereço no sinal nuclear, embora outras técnicas de normalização podem ser empregadas dependendo da distribuição celular da proteína de interesse. Aqui, XRCC1 está localizada no compartimento nuclear, então medidas de normalização para a área nuclear a redistribuição do sinal para os danos ROI. A intensidade fluorescente média de ROI então é gravada para cada imagem em timelapse, incluindo a imagem pré-dano e graficamente em função do tempo (Figura 1C).
Então nós ainda mais caracterizado o dano induzido pelas duas doses de selecionados do laser para examinar a formação de lesões de base de DNA. Em primeiro lugar, a formação de CPD, uma volumosa lesão induzida por UV, foi sondada por imunofluorescência como um marcador para lesões do tipo NER(Figura 2). Em seguida, o lesion8-oxodG base oxidativamente induzida foi sondada como um marcador para lesões do tipo BER (Figura 2B). Nenhum aumento significativo nas lesões CPD foram observadas para a exposição de baixa dose para dois comprimentos de onda (2 s para 355 nm e fps 8 para 405 nm), enquanto a alta dose de tratamento em ambos os comprimentos de onda (10 s para 355 nm e 0,5 fps para 405 nm) mostraram um aumento significativo no fluorescente sinal observado dentro os danos ROI(Figura 2). Um gráfico de dispersão do CPD ROI significa intensidade para cada célula danificada mostra heterogeneidade na formação de danos e deteção em ambos os comprimentos de onda e doses, indicando que um baixo nível de lesões CPD pode estar presente nas doses mais baixas, mas a carga não pode ser significativamente detectadas até uma dose maior é aplicada. O gráfico de dispersão sugere também a ineficiência na pesquisa de anticorpos que possam limitar a quantificação exata das misturas danos.
Isto é ainda mais realçado na detecção de lesões de DNA oxidativamente induzidas pelo marcador 8-oxodG. Nenhum claro aumento sinal fluorescente dentro os danos ROI foi observado para 8-oxodG em comprimento de onda do laser ou dose utilizada (Figura 2B). O anticorpo usado para este trabalho é consistente com as anteriores Publicações9,10,36; no entanto, deve notar-se que pode haver limitações em observar a formação de 8-oxodG com anticorpos37,38. Também é recomendada para confirmação da ausência de lesões oxidativamente induzidas por um segundo marcador, como o recrutamento de 8-Oxoguanine DNA Glycosylase (OGG1), a enzima responsável pela remoção de 8-oxodG do DNA de10. Não observamos OGG1 recrutamento aos nossos sites de dano do ADN; no entanto, a formação de baixos níveis de dano do ADN oxidativamente induzido não pode ser descartada completamente.
Finalmente, examinamos a formação de DSBs usando dois marcadores, γH2AX e 53BP-1, no selectedoses de d do laser por imunofluorescência (Figura 3 & 4). ΓH2AX é comumente usado como um marcador de quebra de fio, mas sua especificidade para DSBs tem sido questionada em um número de relatórios39,40. Além disso, é um evento de fosforilação que propaga-se do local de descanso do vertente, para que localização do sinal de uma ruptura do fio possa ser limitada devido a essa propagação do sinal. Portanto, combinar γH2AX com 53BP-1 permite uma avaliação mais precisa da formação de ORL dentro os danos ROI.
A resposta do γH2AX e 53BP-1 a microirradiação é comprimento de onda e dose dependente. A dose baixa (2 s) estimulação em 355 nm não provoca nenhuma resposta às 5 e 20 min e uma irradiação post 10 min fraco e variável resposta(Figura 3)para ambos os marcadores. A dose elevada (10 s) 355 nm microirradiação induz um aumento sinal fluorescente dentro os danos ROI em 5, 10 e 20 min pós irradiação que é reduzida em 40 min (Figura 3B). Estes resultados indicam que cuidado a titulação da dose 355 nm é necessário para minimizar a Cruz-estimulação das vias de reparação, como demonstrado pela reduzida detecção da dupla vertente pausa marcador γH2AX nos pontos de tempo de início (< 20 min) com a dose baixa aplicado.
Experiências semelhantes foram realizadas usando baixa (8 fps) e alta dose (0,5 fps) 405 nm laser estimulação (Figura 4). Neste comprimento de onda observou-se significativa acumulação de intensidade fluorescente dentro os danos ROI para 53BP-1 e γH2AX, independentemente da dose aplicada, indicando que essas doses gerará uma mistura complexa de quebras simples e duplas quase imediata após a indução de dano do ADN (Figura 4). Além disso, as altas doses de show de 405 nm um aumento na coloração de γH2AX pan-nuclear dentro de 10 min de indução de danos (Figura 4B, inferior) que dificulta a deteção dos danos ROI ao relatório, enquanto o acúmulo de 53BP-1 é mais contidos os danos ROI.
Estes resultados demonstram claramente que a 405 nm microirradiação não é apropriado para monitoramento SSBR ou BER, e que vários marcadores de DNA adutos e vertente quebras deve ser empregado para caracterizar completamente as lesões induzidas e respostas de reparação do DNA.
Alteração de dose-dependente no recrutamento e retenção de XRCC1-GFP
Uma vez que o dano ao DNA induzido tem sido caracterizado, microirradiação do laser pode ser uma plataforma ideal para estudar a dinâmica das proteínas de reparo do DNA. A cinética de retenção e dissociação do XRCC1-GFP mostra uma dependência da dose (Figura 1), que não é inesperada dada a indução de misturas diferentes de dano por cada comprimento de onda. As maiores doses de irradiação (10 s e 0,5 fps) mostram um maior recrutamento de intensidade de XRCC1-GFP em relação as doses mais baixas e mais retenção do XRCC1-GFP no local do dano ao longo tempo de 20 min (Figura 1C). Isso indica que o dano de DNA criado em doses mais elevadas para 355 e 405 nm é provável não resolvido durante o curso da experiência, que é consistente com a aparência e a retenção de marcadores DSB, γH2AX e 53BP-1 (figuras 3 & 4 ).
Curiosamente, as doses mais baixas de dano (2 s e fps 8) mostram rápido recrutamento de XRCC1-GFP para os sites de danos e dissociação de XRCC1-GFP ao longo do tempo experimental para níveis pré-irradiação (Figura 1C). Sem a caracterização completa da mistura de danos, isto pode levar à conclusão de que SSBs e lesões de base completamente são resolvidas usando estas condições prejudiciais. No entanto, a presença de γH2AX e 53BP-1 a 40 min para 355 nm e a 5 min para a 405 nm pode levar a diferentes interpretações. Para a dose de s 355 nm 2, a mistura de danos pode ser predominantemente SSBs, então a aparência dos marcadores de ORL em 40 min pode indicar que algumas lesões unrepaired podem estar levando a DSBs ou que DSBs geradas por essa energia são reparados em uma escala de tempo mais longa. Diferenças de escala de tempo entre SSBR e DSB reparação tem sido relatado anteriormente28,41,42. Da mesma forma, a dissociação de baixa dose (8 fps) 405 nm pode indicar um baixo nível de SSBs ou um cluster de SSBs que rapidamente são convertidos em DSBs, que tem sido notado por alta danificar DNA microirradiação e outro agentes nocivos anteriormente43, 44 , 45.
Juntos, estes resultados destacam a importância de caracterizar as misturas de danos induzidos e utilizando DNA de várias proteínas de reparação e marcadores para interpretar o recrutamento e a retenção de DNA reparar proteínas aos sítios de dano induzido.
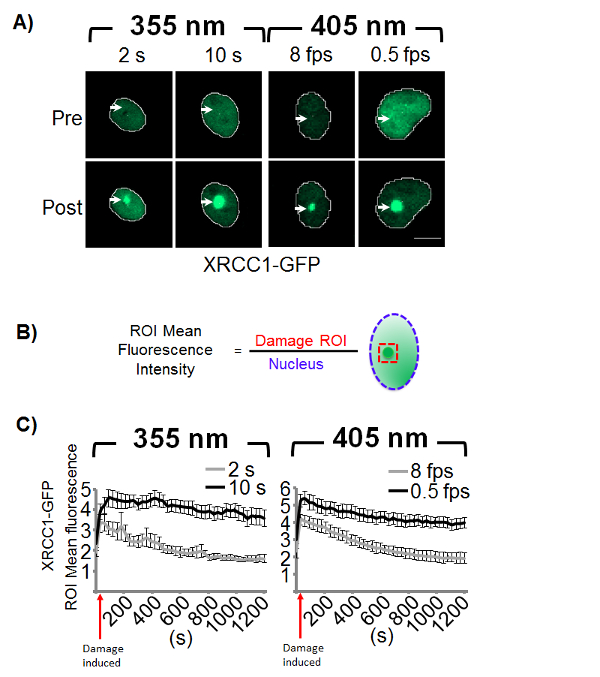
Figura 1 . Microirradiação do laser induz o recrutamento de XRCC1-GFP.
(A) CHO-K1 células expressando estàvel XRCC1-GFP foram irradiadas e fotografadas antes e imediatamente após a indução de danos. As setas indicam a localização da dose de irradiação de micro e barra de escala é 10 µm. (B) recrutamento é medido pela determinação da média intensidade fluorescente dentro os danos ROI e normalizando à média intensidade fluorescente do núcleo inteiro. (C) dinâmica de recrutamento podem ser medida para cada imagem de timelapse. Gráficos são representativas de dois experimentos independentes com barras de erro representando o SEM (n = 24). Clique aqui para ver uma versão maior desta figura.

Figura 2 . Microirradiação do laser induz dano de nucleotídeo.
Células CHO-K1 foram submetidas a microirradiação de laser e corrigidas imediatamente após a indução de danos. Realizou-se a imunofluorescência para detectar CPD e adutos de 8-oxodG. (A) top, gráfico de dispersão da fluorescência ROI média intensidades observadas em células danificadas depois da coloração da CPD. No fundo, imagens representativas de manchar o CPD. As setas indicam a localização de microirradiação e a barra de escala é de 10 µm (n = 12). (B) imagens representante para coloração de 8-oxodG. Setasindicar a localização de microirradiação e a barra de escala é 10 µm. clique aqui para ver uma versão maior desta figura.
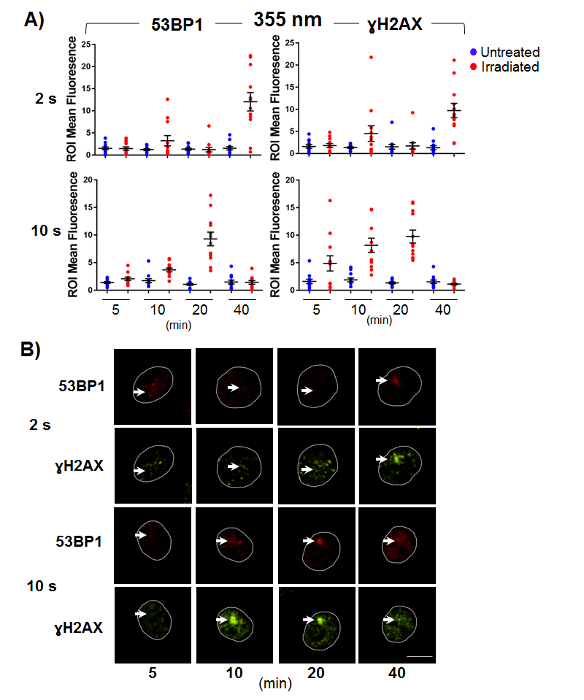
Figura 3 . Marcadores de DNA fita dupla ruptura respondem a 355 nm microirradiação de forma dose-dependente.
Células CHO-K1 foram submetidas a microirradiação e fixadas a tempo pontos indicado pós-estimulação. Realizou-se a imunofluorescência para o ORL marcadores γH2AX e 53BP-1. (A) dispersão de dano normalizado ROI dizer as intensidades de fluorescência medidas por células danificadas e sem danos. Barras de erro são representativos da SEM (n = 12). (B) representante imagens para γH2AX e 53BP-1 coloração. As setas indicam a localização de microirradiação e a barra de escala é 10 µm. clique aqui para ver uma versão maior desta figura.
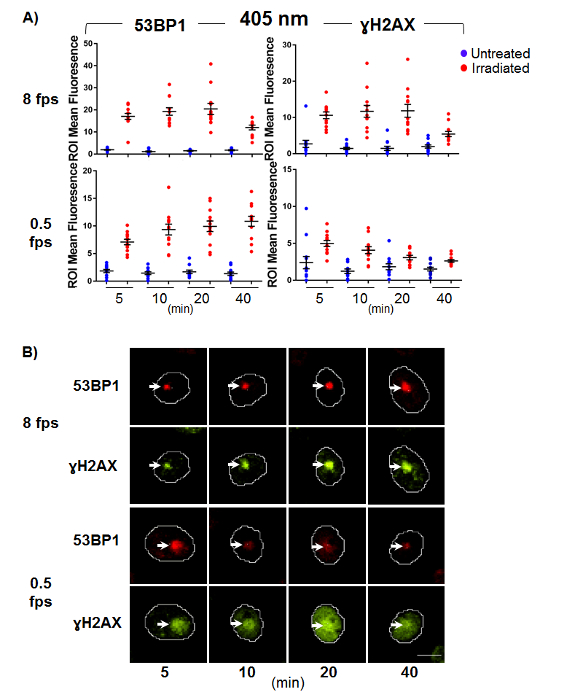
Figura 4. Marcadores de DNA fita dupla ruptura robustamente respondem a 405 nm microirradiação.
Células CHO-K1 foram submetidas a irradiação de micro e fixadas para a estimulação de post indicado de pontos de tempo. Imunofluorescência para o ORL marcadores γH2AX e 53BP-1. (A) dispersão de dano normalizado ROI dizer as intensidades de fluorescência medidas por células danificadas e sem danos. Barras de erro são representativos da SEM (n = 12). (B) representante imagens para γH2AX e 53BP-1 coloração. As setas indicam a localização de microirradiação e a barra de escala é 10 µm. clique aqui para ver uma versão maior desta figura.
Discussão
Usando as condições descritas aqui, qualquer microscópio confocal com uma UV-A ou 405 nm laser, integrado pelo fabricante ou adicionados pelo usuário final, pode induzir a danos no DNA dentro do núcleo de uma célula e permitir o recrutamento de proteínas de reparo do DNA para o site da induzida por dano do ADN a ser monitorado. No entanto, conforme indicado no protocolo e resultados representativos, seleção do comprimento de onda, aplicado o poder, e caracterização de danos são todos importantes questões que devem ser abordadas primeiro pelo usuário a fim de estudar um DNA específico reparar o caminho. Além disso, existem outras considerações no delineamento experimental que também deve ser considerado pelos usuários.
Para monitorar o comportamento de uma proteína em células vivas post microirradiação, uma proteína fluorescente etiquetada deve ser criada. A disponibilidade de uma vasta gama de proteínas fluorescentes que podem ser separadas espectralmente oferece ferramentas para criar fusões de proteína que podem ser usados para caracterizar respostas celulares para a indução de dano do ADN. Transfection transiente de proteínas fluorescentes de interesse permite a seleção rápida de condições prejudiciais, mutações ou até mesmo inibidores, enquanto células transfectadas estàvel oferecem mais controle sobre os níveis de expressão e permitir a outros bioquímicos caracterizações para ser executada, validação de resultados de microirradiação. Proteínas fluorescentes espectralmente distintas também podem ser utilizadas na mesma cela para monitorar as interações entre proteínas dentro da mesma via ou em caminhos diferentes. Proteínas fluorescentes também eliminam a necessidade de anticorpos específicos para cada proteína de interesse e permitir que a célula viva, monitoramento do comportamento de proteína por longos períodos de tempo após a indução de danos.
No entanto, o uso de proteínas fluorescentes também tem desvantagens. Sem origens genéticas deficientes nas somáticas de interesse, proteínas endógenas vão competir com as proteínas fluorescente etiquetadas. Conjugação da marca para o terminus N ou C da proteína fluorescente pode alterar o enrolamento de proteínas, impedir interações proteína-proteína-chave ou bloquear os sinais da translocação; alterar a função e potencialmente afetar a dinâmica de recrutamento. Estes efeitos foram demonstrados em uma série de relatórios onde coloração imunofluorescente demonstrou recrutamento dramaticamente diferente e tempos de retenção para proteínas endógenas no dano local28. Uso de transfecção transiente ou clones estáveis também pode alterar a resposta observada, normalmente através de variações nos níveis de expressão de proteínas. Além disso, o uso de proteínas fluorescentes múltiplas em uma única experiência pode também alterar a dinâmica de recrutamento, se os níveis de proteína não são incubados bem ou se o recrutamento da proteína fluorescente-com a tag maior bloqueia o recrutamento de outras proteínas . Finalmente, proteínas fluorescentes podem atuar como agentes de sensibilização fraca, aumentando a formação de espécies reactivas de oxigénio e potencialmente alterando o DNA danos misturas induzido46,47. Apesar destas deficiências, proteínas fluorescentes oferecem uma série de vantagens em estudar DNA reparar com microirradiação, e se os usuários incorporar controles apropriados, tais como a caracterização de dano aqui descritas, podem fornecer uma nova visão em danos resposta e da proteína-proteína interações3.
Se a imagem de célula viva não é necessária, imunofluorescência pode ser usada para monitorar a resposta aos danos induzidos. Fixação de células em alturas específicas pós indução de danos e coloração com anticorpos específicos para proteínas e lesões de interesse, instantâneos estáticos de indução de danos e o recrutamento e retenção de proteínas podem ser construídas de reparação. Anticorpos podem ser usados para monitorar o recrutamento de várias proteínas de interesse e/ou modificações borne-translational induzidas pela resposta de dano do ADN. Uso da imunofluorescência elimina a necessidade de proteínas fluorescentes e permite o comportamento de proteína endógena ser examinado. No entanto, esse método também tem suas desvantagens. Anticorpos altamente específicos são necessários, e permeabilização e bloqueio procedimentos precisam ser otimizados para permitir detecção de recrutamento com intensidade de sinal suficiente. Procedimentos de fixação não são instantâneos, e essa restrição física limita a resolução temporal desta abordagem. O site da indução de danos de mapeamento para que as células podem ser re-localizadas depois da coloração, também pode apresentar desafios significativos. O microscópio confocal usado neste trabalho permite registro baseado em imagem conforme descrito acima, para que as células danificadas podem ser realocadas com alta precisão. Se registro de palco ou lamela gravada não estiver disponível, o tempo de investimento envolvido em células realocando sem registro de estágio, juntamente com o atraso inerente entre a indução de danos e o recrutamento de imagem, podem fazer imunofluorescência atraente para alguns usuários. No entanto, os projetos experimentais microirradiação mais exacto e completos irão incorporar estes tipos de abordagens em paralelo com o uso de proteínas fluorescentes, conforme descrito no protocolo apresentado.
Aqui, tanto a imagem de célula viva e a imunofluorescência é usado para demonstrar a utilidade do microirradiação do laser. A coloração imunofluorescente nos permite analisar com precisão a mistura de dano do ADN criado e o recrutamento de proteínas induzidas por cada poder do laser, que nos permitem melhor interpretar as alterações observadas no recrutamento e retenção de XRCC1-GFP. Com base nestes resultados, o uso de 405 nm lasers deve ser limitado para exame de BER e SSBR proteínas. Além disso, o delineamento ideal incluiria as medições de energia após o objetivo, uma caracterização de mistura de danos completo para cada linha de celular usada e validação do recrutamento e retenção observada em fluorescente etiquetado proteínas com imunofluorescência. Obviamente, considerações de custo, tempo e equipamentos podem fazer estes desenhos experimentais ideais impossível para alguns usuários. No entanto, a importância de cada um desses elementos é demonstrada aqui, e os usuários devem ter estas considerações em mente ao iniciar experiências de microirradiação.
Divulgações
Os autores não têm nada para divulgar.
Agradecimentos
Os autores gostaria de agradecer ao Dr. Samuel H. Wilson no nacional Instituto de Ciências de saúde ambiental para a linha de celular utilizada neste trabalho.
Materiais
| Name | Company | Catalog Number | Comments |
| Nikon A1rsi laser scanning confocal microscope | Nikon | ||
| NIS Elements software | Nikon | ||
| 355 nm laser | PicoQuant VisUV | Radiation source | |
| Galvanometer photoactivation miniscanner | Bruker | ||
| Microscope slide photodiode power sensor | THORLabs | S170C | |
| Fiber photodiode power sensor | THORLabs | S150C | |
| Digital handheld optical power and energy meter | THORLabs | PM100D | |
| CHO-K1 | From Dr. Samuel H. Wilson, NIEHS | ||
| Minimal essential media | Hyclone | SH3026501 | |
| Fetal bovine serum | Atlanta biologicals | S11550 | |
| XRCC1-GFP | Origene | RG204952 | |
| Jetprime | Polyplus transfection | 11407 | |
| Geneticin | ThermoFisher | 10131035 | |
| 4 chambered coverglass | ThermoFisher | 155382 | |
| 8 chambered coverglass | ThermoFisher | 155409 | |
| Anti 53BP-1 | Novus | NB100304 | |
| Anti-phospho-histone H2AX | Millipore | 05-636-I | |
| Anti cyclobutane pyrimidine dimer | Cosmo Bio clone | CAC-NM-DND-001 | |
| Anti 8-oxo-2´-deoxyguanosine | Trevigen | 4354-MC-050 | |
| Alexa 488 goat anti-mouse | ThermoFisher | A11029 | |
| Alexa 546 goat anti-rabbit | ThermoFisher | A11010 | |
| 4’,6-Diamidino-2-Phenylindole (DAPI) | ThermoFisher | R37606 | Caution toxic! |
| Phosphate buffered saline | ThermoFisher | 0780 | |
| Normal goat serum | ThermoFisher | 31873 | |
| Bovine serum albumin (BSA) | Jackson Immuno Research | 001-000-162 | |
| 37% Formaldehyde | ThermoFisher | 9311 | Caution toxic! |
| Ethanol | Decon Labs | 2716 | |
| Methanol | VWR | BDH1135 | Caution toxic! |
| HCl | Fisher | SA49 | |
| Sodium azide | Sigma-Aldrich | S2002 | Caution toxic! |
| Tris Hydrochloride | Amresco | O234 | |
| Triton X-100 | Sigma-Aldrich | T8787 |
Referências
- Luijsterburg, M. S., et al. Stochastic and reversible assembly of a multiprotein DNA repair complex ensures accurate target site recognition and efficient repair. J Cell Biol. 189 (3), 445-463 (2010).
- Vermeulen, W. Dynamics of mammalian NER proteins. DNA Repair (Amst). 10 (7), 760-771 (2011).
- Berns, M. W. A history of laser scissors (microbeams). Methods Cell Biol. 82, 1-58 (2007).
- Cremer, C., Cremer, T., Zorn, C., Schoeller, L. Effects of laser uv-microirradiation (lambda = 2573 A) on proliferation of Chinese hamster cells. Radiat Res. 66 (1), 106-121 (1976).
- Cremer, C., Cremer, T., Fukuda, M., Nakanishi, K. Detection of laser--UV microirradiation-induced DNA photolesions by immunofluorescent staining. Hum Genet. 54 (1), 107-110 (1980).
- Walter, J., Cremer, T., Miyagawa, K., Tashiro, S. A new system for laser-UVA-microirradiation of living cells. J Microsc. 209, 71-75 (2003).
- Kielbassa, C., Roza, L., Epe, B. Wavelength dependence of oxidative DNA damage induced by UV and visible light. Carcinogenesis. 18 (4), 811-816 (1997).
- Kielbassa, C., Epe, B. DNA damage induced by ultraviolet and visible light and its wavelength dependence. Methods Enzymol. 319, 436-445 (2000).
- Kong, X., et al. Comparative analysis of different laser systems to study cellular responses to DNA damage in mammalian cells. Nucleic Acids Res. 37 (9), 68 (2009).
- Lan, L., et al. In situ analysis of repair processes for oxidative DNA damage in mammalian cells. Proc Natl Acad Sci U S A. 101 (38), 13738-13743 (2004).
- Krasin, F., Hutchinson, F. Strand breaks and alkali-labile bonds induced by ultraviolet light in DNA with 5-bromouracil in vivo. Biophys J. 24 (3), 657-664 (1978).
- Krasin, F., Hutchinson, F. Double-strand breaks from single photochemical events in DNA containing 5-bromouracil. Biophys J. 24 (3), 645-656 (1978).
- Rosenstein, B. S., Setlow, R. B., Ahmed, F. E. Use of the dye Hoechst 33258 in a modification of the bromodeoxyuridine photolysis technique for the analysis of DNA repair. Photochem Photobiol. 31 (3), 215-222 (1980).
- Cremer, T., Peterson, S. P., Cremer, C., Berns, M. W. Laser microirradiation of Chinese hamster cells at wavelength 365 nm: effects of psoralen and caffeine. Radiat Res. 85 (3), 529-543 (1981).
- Limoli, C. L., Ward, J. F. A new method for introducing double-strand breaks into cellular DNA. Radiat Res. 134 (2), 160-169 (1993).
- Carlsson, K., Mossberg, K., Helm, P. J., Philip, J. Use of UV excitation in confocal laser scanning fluorescence microscopy. Micron and Microscopica Acta. 23 (4), 413-428 (1992).
- Stelzer, E. H. K., Haar, F. -. M. Confocal Microscopy: Recent Developments. Advances in Imaging and Electron Physics. 106, 293-345 (1999).
- Rogakou, E. P., Boon, C., Redon, C., Bonner, W. M. Megabase chromatin domains involved in DNA double-strand breaks in vivo. J Cell Biol. 146 (5), 905-916 (1999).
- Tashiro, S., Walter, J., Shinohara, A., Kamada, N., Cremer, T. Rad51 accumulation at sites of DNA damage and in postreplicative chromatin. J Cell Biol. 150 (2), 283-291 (2000).
- Gassman, N. R., Stefanick, D. F., Kedar, P. S., Horton, J. K., Wilson, S. H. Hyperactivation of PARP triggers nonhomologous end-joining in repair-deficient mouse fibroblasts. PLoS One. 7 (11), 49301 (2012).
- Bolin, C., et al. The impact of cyclin-dependent kinase 5 depletion on poly(ADP-ribose) polymerase activity and responses to radiation. Cell Mol Life Sci. 69 (6), 951-962 (2012).
- Godon, C., et al. PARP inhibition versus PARP-1 silencing: different outcomes in terms of single-strand break repair and radiation susceptibility. Nucleic Acids Res. 36 (13), 4454-4464 (2008).
- Hanssen-Bauer, A., et al. XRCC1 coordinates disparate responses and multiprotein repair complexes depending on the nature and context of the DNA damage. Environ Mol Mutagen. 52 (8), 623-635 (2011).
- Mortusewicz, O., Amé, J. C., Schreiber, V., Leonhardt, H. Feedback-regulated poly(ADP-ribosyl)ation by PARP-1 is required for rapid response to DNA damage in living cells. Nucleic Acids Res. 35 (22), 7665-7675 (2007).
- Mortusewicz, O., Leonhardt, H., Cardoso, M. C. Spatiotemporal dynamics of regulatory protein recruitment at DNA damage sites. J Cell Biochem. 104 (5), 1562-1569 (2008).
- Mortusewicz, O., Rothbauer, U., Cardoso, M. C., Leonhardt, H. Differential recruitment of DNA Ligase I and III to DNA repair sites. Nucleic Acids Res. 34 (12), 3523-3532 (2006).
- Solarczyk, K. J., Zarębski, M., Dobrucki, J. W. Inducing local DNA damage by visible light to study chromatin repair. DNA Repair (Amst). 11 (12), 996-1002 (2012).
- Gassman, N. R., Wilson, S. H. Micro-irradiation tools to visualize base excision repair and single-strand break repair. DNA Repair (Amst). 31, 52-63 (2015).
- Botchway, S. W., Reynolds, P., Parker, A. W., O'Neill, P. Use of near infrared femtosecond lasers as sub-micron radiation microbeam for cell DNA damage and repair studies. Mutat Res. 704 (1-3), 38-44 (2010).
- Ferrando-May, E., et al. Highlighting the DNA damage response with ultrashort laser pulses in the near infrared and kinetic modeling. Front Genet. 4, 135 (2013).
- Lan, L., et al. Accumulation of Werner protein at DNA double-strand breaks in human cells. J Cell Sci. 118, 4153-4162 (2005).
- Salmon, T. B., Evert, B. A., Song, B., Doetsch, P. W. Biological consequences of oxidative stress-induced DNA damage in Saccharomyces cerevisiae. Nucleic Acids Res. 32 (12), 3712-3723 (2004).
- Caldecott, K. W. DNA single-strand break repair. Exp Cell Res. 329 (1), 2-8 (2014).
- London, R. E. The structural basis of XRCC1-mediated DNA repair. DNA Repair (Amst). 30, 90-103 (2015).
- Moser, J., et al. Sealing of chromosomal DNA nicks during nucleotide excision repair requires XRCC1 and DNA ligase III alpha in a cell-cycle-specific manner. Mol Cell. 27 (2), 311-323 (2007).
- Campalans, A., et al. Distinct spatiotemporal patterns and PARP dependence of XRCC1 recruitment to single-strand break and base excision repair. Nucleic Acids Res. 41 (5), 3115-3129 (2013).
- Cooke, M. S., Lunec, J. . Immunochemical detection of oxidative DNA damage. Vol. I. , 275-293 (2003).
- Rossner, P., Sram, R. J. Immunochemical detection of oxidatively damaged DNA. Free Radic Res. 46 (4), 492-522 (2012).
- Cleaver, J. E., Feeney, L., Revet, I. Phosphorylated H2Ax is not an unambiguous marker for DNA double-strand breaks. Cell Cycle. 10 (19), 3223-3224 (2011).
- Rybak, P., et al. Low level phosphorylation of histone H2AX on serine 139 (γH2AX) is not associated with DNA double-strand breaks. Oncotarget. 7 (31), 49574-49587 (2016).
- Haince, J. F., et al. PARP1-dependent kinetics of recruitment of MRE11 and NBS1 proteins to multiple DNA damage sites. J Biol Chem. 283 (2), 1197-1208 (2008).
- Caldecott, K. W. Single-strand break repair and genetic disease. Nat Rev Genet. 9 (8), 619-631 (2008).
- Lomax, M. E., Gulston, M. K., O'Neill, P. Chemical aspects of clustered DNA damage induction by ionising radiation. Radiat Prot Dosimetry. 99 (1-4), 63-68 (2002).
- Ma, W., Westmoreland, J. W., Gordenin, D. A., Resnick, M. A. Alkylation base damage is converted into repairable double-strand breaks and complex intermediates in G2 cells lacking AP endonuclease. PLoS Genet. 7 (4), 1002059 (2011).
- Siddiqi, M. A., Bothe, E. Single- and double-strand break formation in DNA irradiated in aqueous solution: dependence on dose and OH radical scavenger concentration. Radiat Res. 112 (3), 449-463 (1987).
- Sano, Y., Watanabe, W., Matsunaga, S. Chromophore-assisted laser inactivation--towards a spatiotemporal-functional analysis of proteins, and the ablation of chromatin, organelle and cell function. J Cell Sci. 127, 1621-1629 (2014).
- Wojtovich, A. P., Foster, T. H. Optogenetic control of ROS production. Redox Biol. 2, 368-376 (2014).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados