Method Article
Applicazione di Micro-irradiazione del Laser per l'esame di Single e Double Strand Break Repair in cellule di mammifero
In questo articolo
Riepilogo
Fluorescenza confocale microscopia e laser micro-irradiazione offrire strumenti per indurre il danno del DNA e monitoraggio della risposta delle proteine di riparazione del DNA in determinate aree sub-nucleare. Questa tecnica ha avanzato significativamente la nostra conoscenza di rilevamento dei danni, di segnalazione e di reclutamento. Questo manoscritto viene illustrato queste tecnologie per esaminare riparazione singola e doppia uscita.
Abstract
Vie di riparazione del DNA altamente Coordinate esistono per rilevare, asportare e sostituire le basi del DNA danneggiati e coordinare la riparazione delle rotture del filo del DNA. Mentre le tecniche di biologia molecolare hanno chiarito la struttura, le funzioni enzimatiche e cinetica delle proteine di riparazione, c'è ancora una necessità di capire come riparazione è coordinato all'interno del nucleo. Micro-irradiazione laser offre un potente strumento per indurre il danno del DNA e l'assunzione di proteine di riparazione di monitoraggio. Induzione di danno del DNA da micro-irradiazione laser può verificarsi con una gamma di lunghezze d'onda, e gli utenti in modo affidabile possono indurre rotture del singolo filo, lesioni della base e rotture a doppio filo con una gamma di dosi. Qui, micro-irradiazione del laser è utilizzato per esaminare riparazione di singolo e doppio filamento indotta da due comuni laser confocale lunghezze d'onda, 355 nm e 405 nm. Più ulteriormente, corretta caratterizzazione della dose applicata laser per indurre miscele danno specifico è descritto, così gli utenti possono eseguire riproducibile micro-irradiazione laser acquisizione e analisi.
Introduzione
La microscopia fluorescente è emerso come una tecnica potente per visualizzare architettura cellulare, esaminare la localizzazione della proteina e monitorare le interazioni proteina-proteina e proteina-DNA. Usando la microscopia fluorescente per studiare le risposte di danni del DNA dopo l'applicazione del global DNA agenti dannosi, come i raggi ultravioletti (UV), radiazioni ionizzanti, ossidanti chimici o agenti alchilanti, e/o chemioterapici ha fornito nuove informazioni l'iniziazione, la segnalazione e il reclutamento di DNA riparare le proteine ai siti di DNA danni1,2. Tuttavia, questi globale e asincrona di eventi dannosi sono limitanti, se l'ordine di reclutamento, la cinetica di associazione o dissociazione: informazioni dettagliate e le relazioni tra proteine chiave di riparazione del DNA sono ricercate. Fortunatamente, gli avanzamenti in microscopio confocale a scansione laser, disponibilità più ampia di lunghezze d'onda laser che inducono danni e miglioramenti in proteine fluorescenti negli ultimi 25 anni hanno fornito i ricercatori con strumenti migliorati per l'esame di questi aspetti del DNA ripristino, attraverso mirate induzione di danno del DNA.
L'irradiazione delle cellule con microfasci laser al fine di studiare le funzioni cellulare e subcellulare è uno strumento ben consolidato in biologia cellulare e radiazione3. Applicazione di questa tecnica per lo studio della riparazione del DNA è emerso quando Cremer e colleghi di lavoro utilizzato un UV altamente mirato (257 nm) laser microbeam sistema per indurre il danno del DNA sopra un 0,5 µm spot in ovaio di criceto cinese (CHO) cellule4 e stabilito l'induzione di DNA photolesions da questo sistema5. Mentre offrendo miglioramenti significativi rispetto ai metodi microbeam UV al tempo, adozione di questo danneggiare sistema era limitato a causa del suo allestimento specializzato e l'incapacità di generare interruzioni di doppio filo (DSBs)6. La ricerca successiva di una varietà di UV-B (290-320 nm) e UV-A lunghezze d'onda (320-400 nm) da un numero di gruppi ha rivelato che photoproducts UV, ossidativo base lesioni, singolo filamento si rompe (SSB) e DSBs potrebbe essere indotto dipende dalla lunghezza d'onda del laser e potenza applicata (rivisto in 3)4,7,8,9,10 . Inoltre, combinazioni di queste lunghezze d'onda UV-A con agenti, come psoralen, bromodeossiuridina (BrdU) e coloranti di Hoechst, sensibilizzanti e UV-B inoltre sono stati trovati per indurre il danno del DNA dipendono dalla lunghezza d'onda, potenza e durata dell'esposizione, anche se la potenza necessaria per indurre danni spesso si abbassa in presenza di questi agenti11,12,13,14,15. Questi avanzamenti esteso l'utilizzo di micro-irradiazione, anche se c'erano ancora caratteristiche tecnici ostacoli da affrontare per una più ampia adozione di questi metodi.
Cremer e colleghi di lavoro avanzato significativamente il campo di micro-irradiazione concentrandosi proprio il microbeam UV per applicare energia dannoso significativo sopra una zona altamente localizzata nella cella. Come microscopi confocali e sistemi di microdissezione laser avanzata, luce ben focalizzato era più largamente accessibile; Tuttavia, fonti UV di accoppiamento per gli ambiti e a che fare con le aberrazioni cromatiche hanno indotto ancora presentato sfide significative per la maggior parte degli utenti3,6,16. Come coloranti UV aumentato in popolarità per tutto il 1990, in grado di messa a fuoco ottica acquisizione fluorescenza UV-eccitato è diventato più ampiamente disponibili16e miglioramenti nella scansione laser offerto agli utenti la possibilità di creare altamente concentrato UV spot di eccitazione in cellule6,17. Tuttavia, non era fino agli inizi del 2000 che è stato sentito il vero impatto di questa combinazione di travi ben focalizzati con laser di intensità maggiore, quando numerosi rapporti emersero dimostrando che le rotture del filo del DNA potrebbero essere indotto con e senza sensibilizzatori nella Gamma di UV-A6,10,18,19,20, 405 nm21,22,23,24, 25,26e anche a più lunghezze d'onda visibili come 488 nm27. Questi progressi consentiti per adozione più diffusa della tecnica del micro-irradiazione su un numero di sistemi commerciali. Parallelamente a questi sviluppi, due-fotone tecniche emerse anche che ha permesso di preciso induzione di danno del DNA; anche se questi progressi non saranno discussi qui, ci sono una serie di articoli di revisione discutendo queste metodologie9,28,29,30.
Con l'attuale accessibilità dei microscopi confocali capace di fornire luce UV altamente concentrata e la diffusa disponibilità di proteine fluorescenti per consentire la registrazione in tempo reale delle proteine di riparazione del DNA, tecniche di micro-irradiazione sono evoluti in potenti strumenti per l'esame di vie di risposta e riparazione di danni del DNA. Tuttavia, gli utenti devono essere consapevoli del fatto che la generazione di danno del DNA dipende fortemente la lunghezza d'onda del laser e la potenza applicata alla regione sub-nucleare. Uso di UV-C (~ 260 nm) lunghezze d'onda consentono diretto del DNA eccitazione ed elevata selettività per l'induzione di UV photoproducts7,8. Lunghezze d'onda UV-A e UV-B produrre miscele di danno del DNA (lesione della base, SSB e DSBs), dipende la potenza applicata e dello sfondo del cellulare utilizzato7. Fotosensibilizzatori endogeni e anti-ossidanti livelli nelle cellule mirati possono influenzare le miscele di danni del DNA prodotte da queste lunghezze d'onda. Inoltre, l'uso di fotosensibilizzatori esogeni (BrdU, ecc.) può aiutare a ridurre l'energia necessaria per l'induzione di danno del DNA. Tuttavia, questi agenti possono indurre danni al DNA da loro stessi, e possono alterare il ciclo cellulare e la struttura della cromatina, così il loro uso può produrre effetti indesiderati che devono essere considerati dall'utente. Pertanto, prima di utilizzare micro-irradiazione per studiare la riparazione e la risposta al danno al DNA, un'attenta considerazione del pathway di riparazione del DNA di interesse, le lunghezze d'onda disponibili per l'uso e la miscela di danni del DNA creato è necessaria.
Qui, micro-irradiazione del laser viene eseguita a due lunghezze d'onda comunemente usate, 355 nm e 405 nm, senza sensibilizzatori per dimostrare le miscele di danno del DNA indotto da queste lunghezze d'onda e le influenze di queste miscele di danni hanno sull'esame la riparazione diSSB e DSBs. gli utenti devono essere consapevoli che queste lunghezze d'onda non si creano una singola specie di rotture del filo o lesioni della base. Al fine di discriminare tra le vie di riparazione del DNA, gli utenti devono attentamente controllare la potenza applicata su una specifica regione del nucleo e caratterizzare il danno indotto usando più strand break marcatori e gli anticorpi di lesione del DNA. Se correttamente applicato e caratterizzato, micro-irradiazione del laser è in grado di arricchire alcune specie di danno del DNA, consentendo agli utenti di valutare la riparazione delle lesioni della base e SSB o DSBs, con alcune specificità. Di conseguenza, abbiamo fornito un metodo che consente agli utenti di eseguire micro-irradiazione laser riproducibile, caratterizzano le miscele di danno del DNA indotte dalla dose applicata laser ed eseguire analisi di dati.
Protocollo
1. coltura delle cellule e la generazione di cellule stabili
- CHO-K1 crescere cellule nel minimo essenziale supplementato con 10% siero bovino fetale. Mantenere le cellule in un'incubatrice umidificata con 5% CO 2 a 37 ° C.
- Cellule di CHO-K1 transfect con 1 µ g di DNA del plasmide contenente XRCC1 umano con un C-terminale verde tag proteina fluorescente (GFP) utilizzando un reagente di transfezione commerciali seguendo il produttore ' istruzioni s.
- CHO-K1 selezionare le cellule che esprimono stabilmente la fusione XRCC1-GFP umana utilizzando 800 µ g/mL geneticin e arricchire la popolazione esprime XRCC1-GFP mediante fluorescenza assistita ordinamento delle cellule (FACS).
- Cellule di piastra da micro-irradiati in recipienti di coltura con fondelli coprioggetto, in modo che raggiungano circa 75% o maggiore confluenza il giorno seguente. Alcuni spazi tra le cellule sono desiderabili, particolarmente se si utilizza la registrazione per tornare al campo di immagine stesso (descritto nella sezione 3.2).
2. Set-up microscopio
- selezionare una scansione laser confocale microscopio dotato di idonei laser e ottica e software di controllo per micro-irradiazione/fotostimolazione. Presentato esperimenti uso un microscopio confocale che è stato modificato per includere un laser di 355 nm, accoppiati in fibra per un miniscanner di fotoattivazione di galvanometro di scansione laser e controllati tramite il produttore ' software di analisi e controllo di s. Questo sistema può eseguire micro-irradiazione a un'utente designato regione di interesse (ROI) utilizzando la fotoattivazione miniscanner (355 laser nm) o il galvanometro confocale standard (laser 405 nm).
- Selezionare la lunghezza d'onda da utilizzare per micro-irradiazione, assicurando che tutti i componenti ottici in lightpath il micro-irradiazione sono adatti per la lunghezza d'onda selezionata.
Nota: Il sistema presentato utilizza un obiettivo a immersione in olio 40 × C-Apochromat (apertura numerica (NA) 1.2) insieme a un cubo di filtro UV per 355 nm micro-irradiazione e un 20 × C-Apochromat (NA 0.75) asciutto obiettivo utilizzando il lightpath confocale standard per 405 nm micro-irradiazione. 20 × obiettivo utilizzato nel setup non è compatibile con la lunghezza d'onda di nanometro 355; di conseguenza abbiamo utilizzato il 40 × per 355 nm solo. Utilizzando l'obiettivo asciutto per danneggiare consente protocolli di immunofluorescenza da applicare a valle senza pulizia fuori olio di immersione, ed è per questo che utilizziamo questo obiettivo quando possibile. - Configurare il microscopio per l'esperimento di micro-irradiazione. Per mantenere la coerenza tra esperimenti, designare una configurazione standard dei componenti del microscopio per essere utilizzato per ogni tipo di micro-irradiazione. Salvare queste impostazioni come predefinito all'interno del microscopio ' s funzionamento del software, se possibile.
Nota: Nel sistema presentato, le cellule sono micro-irradiati e imaging utilizzando scansione unidirezionale, una risoluzione di scansione di 1024 x 1024 pixel con 1 x scanner zoom, ad un frame rate di 8 fotogrammi al secondo (fps) e con il foro stenopeico impostate su circa 4 arioso Units (AU), come determinato per il laser di 488 nm (69 µm). Questa impostazione aperta pinhole è stata scelta per massimizzare la quantità di luce catturata in ogni immagine, permettendo l'uso di imaging laser potenze inferiori e riducendo photobleaching.
3. Micro-irradiazione laser
- posto il preparato piastra di coltura, che contengono le cellule di interesse, sul tavolino del microscopio e bloccare saldamente in posizione.
- , Se disponibile, mettere il vetrino chambered in un'incubatrice di palcoscenico-top e mantenere a 37 ° C con 5% di CO 2 durante l'induzione di danno. Questo passaggio è più importante per timelapse di cellule vive di imaging o lunghe sessioni di imaging. Altrimenti le cellule posto sul microscopio fase ed eseguire irradiazione a temperatura ambiente (~ 25 ˚ c) e quindi tornare rapidamente le cellule di un incubatore a 37 ° C con 5% CO 2.
- Registro campi di immagine per permettere al ricercatore di tornare nella stessa posizione dopo aver eseguito il fissaggio, colorazione o altre procedure. Eseguire la registrazione dei campi danneggiati utilizzando una fase di microscopio codificato, automatizzati o recipienti di coltura coprioggetto con una griglia acidata o con etichetta delle posizioni XY di cella.
- Se utilizzando la registrazione dell'immagine software, selezionare una caratteristica riconoscibile della nave cultura (cioè, l'angolo del coprioggetto o barriera tra pozzi), raccogliere un'immagine e registrare la posizione XY. Questo vi permetterà di allineamento e di registrazione delle posizioni XY di campi selezionati seguendo la preparazione del campione.
- Se si utilizza la registrazione manuale, registrare griglia o acidati posizioni per ogni campo manualmente, facendo attenzione a registrare l'orientamento e il posizionamento del piatto sul palco.
- Se nessuna funzionalità di identificazione per i recipienti di coltura cellulare sono disponibili, individuare settori danneggiati mediante ispezione visiva della morfologia delle cellule e densità. Prendersi cura di selezionare i campi con le caratteristiche sufficientemente distinte per essere riconoscibili durante più successivamente imaging.
Nota: Questo può richiedere molto tempo per eseguire a meno che l'orientamento e la zona della nave cultura è strettamente limitata.
- Selezionare un campo per micro-irradiazione e mettere a fuoco il campione. Per le cellule che esprimono le proteine fluorescente contrassegnati, selezionare il piano focale con sezione trasversale massima nucleare nel canale fluorescente di interesse. Per le cellule che non esprimono etichettato proteine, o per quelli senza localizzazione nucleare chiaro, contrasto di fase o imaging (DIC) contrasto differenziale di interferenza può essere utilizzato per trovare il piano focale corretto.
Nota: una caratteristica utile di contrasto di fase e DIC imaging è il fatto che, come il piano focale si muove attraverso il campione, la stessa funzione può apparire chiaro o scuro a seconda del relativo piano focale.- Selezionare una caratteristica chiara all'interno del nucleo, come ad esempio un nucleolo e muoversi su e giù il piano focale osservando questo cambiamento di aspetto. Il vero piano focale si troverà all'interno la transizione dalla luce al buio. Per mettere a fuoco l'esempio, selezionare il piano focale in cui la feature selezionata ha il contrasto più nitido.
- Registrare la posizione del campo di interesse. Creare un quadrato di 3x3 pixel ROI all'interno del software di microscopio, posizionare questo ROI sopra il nucleo di una cellula di essere danneggiati e impostare questo ROI danno ROI.
- Raccogliere un'immagine pre-danni, compresa la posizione del danno ROI.
- Per esperimenti che non utilizzano proteine fluorescenti, acquisire un'immagine del campo chiaro di contrasto (DIC) interferenze contrasto di fase o differenziale di identificare e registrare i nuclei per danni.
- Per vivere-cella esperimenti utilizzando proteine fluorescenti, acquisire un'immagine contenente campo chiaro e il canale di fluorescenza per la proteina di interesse (cioè, laser linea 488 nm per l'eccitazione di GFP, laser linea 561 nm per l'eccitazione di RFP). Negli esperimenti utilizzando il CHO-K1 XRCC1-GFP, fluorescenza eccitata dalla linea laser 488 nm è stato raccolto simultaneamente con il canale di campo chiaro trasmesso DIC.
- Danni laser initiate.
- Nel sistema presentato, controllo la dose di laser con il tempo totale trascorso il danno ROI di scansione. Poiché il galvanometro per il laser 355 nm opera a una velocità di scansione fissa, dose di laser viene controllato mediante il tempo di scansione ripetutamente il danno selezionato ROI: nei dati presentati qui, micro-irradiazione a 355 nm viene eseguita per 2 e 10 secondi.
- Al contrario, controllare la dose di laser 405 nm modulando la frequenza di scansione ed esegue una scansione del danno selezionato ROI. In dati prisentito qui, utilizzare 8 e 0,5 fps per micro-irradiazione a 405 nm. Una velocità di scansione di 8 fps offre una dose di laser inferiore a 0,5 fps, perché il laser impiega meno tempo su ogni pixel durante la scansione. Entrambi i laser sono operati al 100% di potenza. Vedere la sezione 3.7 per istruzioni sulla misurazione potenza laser direttamente.
Nota: Ogni sistema di microscopio individuali può differire in come potenza del laser viene recapitato a un ROI designato, simile a come la 355 nm e 405 nm si differenziano per il sistema presentato. Gli utenti dovranno determinare questa procedura per il loro sistema di microscopio e riferire questi parametri all'interno delle loro sezioni metodi. - Per l'imaging di cellule vive, effettuare l'acquisizione immagine timelapse di canali campo chiaro e fluorescenza. Regolare la durata e la frequenza di timelapse per ottimizzare la raccolta di dati, catturando idealmente l'accumulo della proteina fluorescente a danni della ROI e sua dissociazione sopra il corso di tempo dell'esperimento. Gli esperimenti usando XRCC1-GFP raccolto immagini ogni 30 secondi per 20 minuti.
Nota: la frequenza dell'imaging timelapse può essere limitata da photobleaching del fluoroforo, analisi di accumulazione e di dissociazione di complicazione. Photobleaching può essere valutata osservando cellule intatte durante il timelapse e regolando le condizioni di acquisizione per minimizzare la perdita di segnale fluorescente in queste cellule non danneggiate. Alcuni photobleaching può essere inevitabile, così gli utenti possono compensare la perdita di segnale normalizzando le intensità di fluorescenza delle cellule danneggiate a quelle delle cellule intatte in ogni fotogramma del timelapse.- Una volta completato il corso di tempo, selezionare un nuovo campo di cellule per danni o riparare le cellule danneggiate per ulteriori analisi, come descritto nella sezione 4.
- Continua micro-irradiazione e timelapse imaging finché non viene raggiunto il numero desiderato di cellule danneggiate.
Nota: si consiglia di danneggiare un totale di 10-25 celle per condizione selezionata al fine di valutare l'eterogeneità della cellula--cellula in risposta al danno indotto. Anche se più sistemi di confocale permetterà agli utenti di danneggiare più di una cella durante ogni micro-irradiazione, questo introduce sfalsati danni iniziazione volte, che possono complicare l'analisi del tempo di reclutamento di picco sopra un gran numero di cellule o tempo di dissociazione per eventi veloci. In alcuni sistemi, induzione di danno simultanea sopra parecchi ROIs può ridurre la dose di laser ricevuta da ogni ROI indipendente. Di conseguenza, a meno che questi problemi di potere e di sincronizzazione vengono risolti direttamente dall'utente, è consigliabile che le celle vengano danneggiati singolarmente.
- Per eseguire analisi di immunofluorescenza (IF), danni e sia immediatamente correggere celle, come descritto nella sezione 4, o consentire alle cellule di riparazione per incrementi selezionati di tempo (cioè, 1, 5, 10 o 20 min). Una volta trascorso il tempo desiderato, difficoltà e macchia le celle, come dettagliato nella sezione 4.
- Per aumentare il numero complessivo delle cellule per analisi di, danni ulteriori campi all'interno del vaso di coltura per generare un corso a tempo multi-campo della risposta post-danno. Registrare la posizione XY di ogni campo e il tempo che si è verificato il danno. Dopo il totale desiderato passare del tempo di riparazione, difficoltà e macchia le celle come dettagliato qui sotto.
- Dopo l'osservazione del reclutamento di proteine alle dosi selezionate, misura e post-fibra rapporto laser Potenza livelli e post-obiettivo con precisione caratterizzano e segnalare laser dosi. Usiamo un misuratore di potenza digitale compatto e due teste del sensore fotodiodo differenti; uno accoppiato direttamente alla fibra laser per misurare la post-fibra produzione, e l'altro collocato sul palco microscopio per misurare la produzione post-objective.
- Per misurare la potenza del laser, sistemare il sensore nella configurazione desiderata (post-fibra o post-objective) ed eseguire micro-irradiazione come descritto in precedenza, durante la registrazione di misurazioni effettuate dal misuratore di potenza. Essere consapevoli del fatto che la frequenza di campionamento dello strumento potrebbe non essere abbastanza veloce per acquisire gli eventi di micro-irradiazione molto veloci, quindi potrebbe essere necessario eseguire più iterazioni dell'esperimento per garantire che il misuratore di potenza rileva con precisione la dose applicata.
Nota: Per il laser 355 nm, abbiamo misurato una potenza di picco medio di circa 19 µW post-obiettivo e post-fibra di circa 5 mW. Per il laser di 405 nm, micro-irradiazione è stato effettuato nei cicli di 30 iterazioni per superare la frequenza di campionamento massima di 0.01 s dello strumento e le potenze di picco medio di 1,5 mW e 2.4 mW sono stati misurati utilizzando velocità di scansione di 0,5 e 8 fotogrammi al secondo , rispettivamente. Ogni misuratore di potenza è diverso. Gli utenti dovranno determinare le lunghezze d'onda operative e frequenze di campionamento per loro misuratore di potenza individuale.
- Per misurare la potenza del laser, sistemare il sensore nella configurazione desiderata (post-fibra o post-objective) ed eseguire micro-irradiazione come descritto in precedenza, durante la registrazione di misurazioni effettuate dal misuratore di potenza. Essere consapevoli del fatto che la frequenza di campionamento dello strumento potrebbe non essere abbastanza veloce per acquisire gli eventi di micro-irradiazione molto veloci, quindi potrebbe essere necessario eseguire più iterazioni dell'esperimento per garantire che il misuratore di potenza rileva con precisione la dose applicata.
4. Le procedure di colorazione di immunofluorescenza
- induzione di rottura filo analizza se macchiando.
- Cellule fix con 3,7% formaldeide (attenzione) in fosfato tampone salino (PBS) per 10 min. aspirato soluzione di formaldeide e lavare 3 volte con PBS. Il protocollo può essere fermato qui inserendo PBS indietro sulle cellule e le cellule possono essere conservate a 4 ° c fino a 1 settimana.
Attenzione: La formaldeide è tossica e cancerogena. Dispositivi di protezione individuale adeguato usura e smaltire la sostanza tossica come indicato dalle procedure istituzionali di salute e sicurezza ambientale. - Permeabilize celle utilizzando 0,25% Triton X-100 in PBS per 10 min a temperatura ambiente (TA) e poi lavare 3 volte con PBS.
- Associazione di anticorpo specifico non di blocco mediante incubazione per 30 min in PBS contenente 1% di albumina di siero bovino (BSA) a TA.
- Incubare con primario monoclonal antibody contro γH2AX e primaria dell'anticorpo policlonale contro 53BP-1 sia diluito 1: 750 in PBS contenente BSA 1% per 1 h a RT e poi lavare 3 volte con PBS.
- Cellule Incubate con anti-topo di capra Alexa 488 e 546 Alexa Capra anti-coniglio sia diluito 1: 2000 in PBS contenente BSA 1% per 1 h a RT e poi lavare 3 volte con PBS.
- Macchia DNA nucleare con una soluzione di 10 mg/mL di DAPI (4 ', 6-Diamidino-2-Phenylindole, attenzione) diluito a 1: 5000 in PBS per 5 min, o uso paragonabile colorante nucleare e poi lavare 3 volte con PBS.
Attenzione: DAPI è tossico e mutageno. Indossare dispositivi di protezione personale ed eliminare la sostanza tossica come indicato dalle procedure istituzionali di salute e sicurezza ambientale. - Posto PBS o PBS + 0.1% sodio azide (attenzione) sulle cellule marcate. Protocollo può essere fermato qui e cellule conservati a 4 ° c per diversi giorni, o procedere all'acquisizione di immagini nella sezione 5.
Attenzione: sodio azide è tossica. Indossare dispositivi di protezione personale e smaltire le sostanze tossiche, come indicato dalle procedure istituzionali di salute e sicurezza ambientale.
- Cellule fix con 3,7% formaldeide (attenzione) in fosfato tampone salino (PBS) per 10 min. aspirato soluzione di formaldeide e lavare 3 volte con PBS. Il protocollo può essere fermato qui inserendo PBS indietro sulle cellule e le cellule possono essere conservate a 4 ° c fino a 1 settimana.
- Analyze induzione delle lesioni della base o del DNA addotti dalla macchiatura di se.
- Fix e permeabilize le cellule in metanolo ghiacciato (attenzione) per 20 min a -20 ° C.
Attenzione: Il metanolo è tossico ed infiammabile. Indossare dispositivi di protezione personale ed eliminare la sostanza tossica come indicato dalle procedure istituzionali di salute e sicurezza ambientale. - Aspirare il methanol e consentire l'esempio asciugare completamente per 15 min. protocollo può essere fermato qui e cellule conservate a-20 ˚ c a secco fino a 3 giorni.
- Reidratare le cellule in PBS per 15 min.
- Denaturare DNA usando 2 N HCl (attenzione) per 45 min a RT e lavare 3 volte con PBS.
Attenzione: HCl è corrosivo. Indossare dispositivi di protezione personale e smaltire la sostanza corrosiva, come indicato dalle procedure istituzionali di salute e sicurezza ambientale. - Neutralizzare in 50 mM Tris-HCl a pH 8.8 per 5 min seguita da 3 lavaggi con PBS.
- Incubare le cellule in tampone bloccante fatte con siero di capra normale 5% e 0,1% Triton X-100 in PBS per 1h a RT.
- Incubate con anticorpi primari per 8-oxo-2-deossiguanosina (8-oxodG, 1: 400) o cyclobutane pirimidina dimero (CPD, 1:1, 000) nel tampone bloccante del siero di capra per 1h a RT e poi lavare 3 volte con PBS.
- Incubare le cellule con Alexa 488 anti-topo di capra con un rapporto di 1: 2000 nel tampone bloccante del siero di capra per 1h a RT e lavare 3 volte con PBS.
- Macchia DNA nucleare utilizzando 1: 5000 DAPI (10 mg/mL) in PBS per 5 min e lavare 3 volte con PBS.
- Se utilizza chambered coprioggetto, posto PBS o PBS + 0.1% sodio azide indietro sulle cellule marcate. Protocollo può essere fermato qui e cellule conservati a 4 ° c per diversi giorni, o procedere all'acquisizione di immagini nella sezione 5.
- Montare in alternativa, le cellule nel mezzo di montaggio comodo, se coverglass aggiuntivo può essere posizionato sulla parte superiore della nave di cultura. Una volta indurito, celle montate possono essere conservate a 4 ° c per lunghi periodi di tempo.
- Fix e permeabilize le cellule in metanolo ghiacciato (attenzione) per 20 min a -20 ° C.
5. Acquisizione immagini per esperimenti di immunofluorescenza
- pulire il fondo del coprioggetto della nave cultura con etanolo e metterlo di nuovo sul palcoscenico al microscopio confocale. Assicurarsi che l'orientamento e la posizione dell'imbarcazione sulla fase corrisponde all'orientamento e posizione registrata quando il danno è stato indotto. Fissare il vaso di coltura per garantire la registrazione ottimale e imaging.
- Locate in precedenza imaged campi utilizzando la tecnica di registrazione selezionato (descritto in 3.2).
- Per la registrazione manuale usando un coprioggetto con una griglia acidata, portare la griglia messa a fuoco e trovare segni identificativi. Poi uso registrato coordinate XY per individuare le cellule danneggiate.
- Per registrazione automatizzata basata su immagine, individuare la caratteristica strutturale utilizzata come riferimento nel passaggio 3.2.1 e quindi allineare la vista corrente del microscopio dal vivo più vicino all'immagine registrata come possibile. Una volta allineati, misurare la posizione di fase di microscopio XY corrente e confrontarlo con la posizione di x-y dell'immagine. La distanza lineare tra le due posizioni definisce l'offset X e Y. Applicare questo offset per ogni posizione XY registrate per identificare i campi di immagine contenente la cella irradiata. Questa funzione di offset può essere automatizzata del software di controllo del microscopio o eseguita manualmente.
- Se non punti di registrazione appropriato potrebbero essere identificati, individuare manualmente cellule scansione la diapositiva e individuando le caratteristiche delle cellule precedentemente identificato.
- Acquisire immagini utilizzando impostazioni di microscopio appropriato per raccogliere tutto macchiati obiettivi, tra cui un'immagine di campo chiaro (impostazione nella sezione 2.3 e messa a fuoco nella sezione 3.3). Negli esperimenti presentati, immagini multicanale sono state raccolte utilizzando le seguenti linee del laser di eccitazione e fluorofori: 405 nm (DAPI), 488 nm (Alexa 488 e trasmessa DIC brightfield) e 561 nm (Alexa 546). Tutti i laser passano attraverso un filtro sintonizzabile acusto-ottico singolo controllo della trasmissione di lunghezza d'onda laser e di potere.
6. Analisi di immagine delle cellule del reclutamento di proteine fluorescenti ai siti di micro-irradiati di vivere
- Open acquisito immagini in un'applicazione di analisi di immagine (cioè, elementi di NIS o ImageJ). Se necessario, combinare l'immagine pre-irradiazione con le immagini della webcam per generare una sequenza di immagini singole di induzione di pre- e post-danno. Gli utenti possono mostrare il reclutamento misurando i cambiamenti nell'intensità fluorescente sopra la zona irradiata relativa all'intensità di fluorescenza misurata attraverso il nucleo intero (vedere Results rappresentante Figura 1 B. ).
- Per ogni cella deve essere misurata, in primo luogo generare un ROI che rappresenta il nucleo di riferimento. Utilizzare un algoritmo di soglia sul segnale fluorescente che contiene i pixel che compongono il nucleo e poi convertire questa zona in un ROI. Se necessario, il ROI può essere disegnato manualmente utilizzando il campo chiaro come riferimento. Regolare il ROI nel corso tempo affinché l'area nucleare rientra con precisione il ROI e segnalare l'intensità media di fluorescenza del segnale fluorescente all'interno di questo ROI per ogni frame.
- Per ogni cella deve essere misurata creare un 6x6 pixel ROI e metterlo sopra il danno ROI per ogni fotogramma del corso tempo. Questa maggiore ROI è ora il danno ROI per l'analisi. Segnalare l'intensità di fluorescenza ROI medio per ciascun frame.
Nota: Software di analisi di immagine commerciale contiene moduli per il rilevamento degli oggetti 2D, e ci sono una serie di macro sviluppati dall'utente ImageJ o Figi che facilitano il flusso di lavoro più veloce e un throughput più elevato (Vedi https://imagej.nih.gov/ij/plugins/). - Normalizzare l'intensità di fluorescenza del ROI di media danno a quello di riferimento corrispondente ROI in ogni fotogramma del timelapse. Qui, l'intensità media di fluorescenza nucleare viene utilizzato come riferimento di ROI (Vedi rappresentante Results Figura 1). la normalizzazione può essere eseguita sottraendo il riferimento di media intensità di fluorescenza di ROI dalla media danneggiare l'intensità di fluorescenza ROI, o dividendo la media danneggiare ROI intensità di fluorescenza il riferimento a media intensità di fluorescenza ROI.
Nota: Normalizzazione di divisione può portare a risultati imprevisti, se impiegati in situazioni con valori di intensità molto bassa riferimento. - Ripetere per tutte le cellule danneggiate, così come per il controllo di almeno due, cellule intatte. Risultati del controllo delle cellule possono essere utilizzati per ulteriore normalizzazione, se necessario.
- Graph normalizzati i valori di intensità nel corso del tempo per mostrare i cambiamenti nelle dinamiche di reclutamento in funzione del trattamento sperimentale.
7. Analisi di reclutamento di proteine rilevato dall'immunofluorescenza dell'immagine
- Apri immagini pre- e post-danni in un'applicazione di analisi di immagine. Le immagini pre-danno contengono il danno ROI(s) usato per micro-irradiazione. Copiare il ROI(s) e incollare le immagini post-danni per identificare le cellule mirate.
- Generare un 6x6 pixel ROI per l'analisi del reclutamento di proteine e posizionare questo ROI nella posizione del danno ROI. Questa maggiore ROI è ora il danno ROI per l'analisi. Segnalare i danni di media intensità di fluorescenza ROI.
- Normalizzare l'intensità di fluorescenza del ROI di media danno a quello di un corrispondente riferimento ROI. Qui, l'intensità media di fluorescenza nucleare della cellula danneggiata viene utilizzato come riferimento di ROI (Vedi rappresentante Results Figura 1). Generare il riferimento ROI utilizzando un algoritmo di soglia sul segnale DAPI per definire il nucleo e poi convertire questa zona in un ROI. Segnalare il riferimento di media intensità di fluorescenza di ROI. Normalizzazione può essere eseguita sottraendo il riferimento di media intensità di fluorescenza di ROI dal danno medio intensità di fluorescenza di ROI, o dividendo i danni di media intensità di fluorescenza di ROI il riferimento a media intensità di fluorescenza ROI.
Nota: Normalizzazione di divisione può portare a risultati imprevisti, se impiegati in situazioni con valori di intensità molto bassa riferimento. - Ripetere per tutte le cellule danneggiate ed eseguire la stessa analisi su controllo almeno due celle, come descritto in precedenza.
- Graph normalizzati i valori di intensità per ciascuna misurata micro-irradiazione evento contro il tempo per mostrare i cambiamenti nel tipo di danno del DNA o assunzione di proteine in funzione del trattamento sperimentale.
Risultati
Caratterizzazione del danno indotto del DNA
Induzione delle lesioni della base e le rotture del filo è dipendente dalla dose di laser applicato all'area selezionata nucleare e il microambiente cellulare del modello di cellulare utilizzato7. Le proteine fluorescenti fuse per riparare le proteine, come XRCC1, 53BP1, Ku70 o Rad51, fornire utile singolo e doppie filo pausa marcatori per stabilire l'energia minima necessaria per vedere l'accumulo di una proteina fluorescente all'interno di un danno ROI sopra il Priorità bassa fluorescenza9,19,31. Una volta individuate le condizioni che inducono una risposta, è fondamentale per caratterizzare la miscela di danno indotta da quella specifica lunghezza d'onda e la dose. Attenuazione della dose e durata alla lunghezza d'onda usato può consentire all'utente di ridurre al minimo la formazione di miscele complesse di danni. Dosi di laser a basso nella gamma UV-A sono state dimostrate per produrre prevalentemente SSB e una piccola quantità di lesioni della base, adatte per studiare SSBR e BER vie10,28. Aumentando la dose crea lesioni più complesse della base, ossidativa e UV indotta e induce un numero più significativo di DSBs7,10. Mentre l'induzione di una singola specie di danno del DNA è auspicabile per l'esame di specifiche vie di riparazione del DNA, è più probabile che gli utenti sono inducendo una miscela di lesioni del DNA, con una lesione specifica come essendo molto più frequenti di lesioni della base o DSBs SSB. Questo è simile a miscele di danno del DNA indotte da agenti chimici come il perossido di idrogeno (H2O2) o metile methanesulfonate (MMS)32. Gli utenti devono essere consapevoli quando si segnalano risultati che danno miscele possono verificarsi, e un'attenta caratterizzazione della dose e lesioni nel sito di danno indotto sono necessarie per garantire la riproducibilità e la comparabilità dei risultati.
Negli studi di micro-irradiazione, XRCC1-GFP è spesso usato come un indicatore per l'induzione delle lesioni della base e SSB9,28. XRCC1 è una proteina di impalcatura che svolge un ruolo importante nella riparazione SSB (SSBR) e bassa di asportazione (BER) di riparazione e partecipa anche ad altre vie di riparazione, come33,riparazione (NER) l'asportazione del nucleotide34,35 . Svolge un importante ruolo di coordinamento nella riparazione del DNA, interagendo con un numero di proteine chiave, tra cui poli polimerasi 1 (PARP-1), DNA polimerasi β (Pol β) e DNA ligasi III. Abbiamo utilizzato XRCC1-GFP stabilmente espressa in cellule CHO-K1 per determinare le dosi di laser per generare SSB e DSBs necessaria. Abbiamo identificato in primo luogo la dose minima necessaria per indurre un reclutamento osservabile di XRCC1-GFP per ogni lunghezza d'onda (Figura 1). Per la lunghezza d'onda di nanometro 355, un tempo di sosta s 2 sopra il danno definito ROI generato un segnale fluorescente aumentato entro quel ROI, che indica l'induzione di danno del DNA che era rilevabile sullo sfondo (Figura 1A). Per 405 nm, una velocità di scansione di 8 fps era necessaria per generare un reclutamento osservabile al danno ROI (Figura 1A). La dose è stata quindi aumentata (10 s per 355 nm e 0,5 fps per 405 nm) per creare un più intenso danneggiare ROI (Figura 1A).
Reclutamento e il mantenimento di XRCC1-GFP presso il sito del danno indotto è stato poi controllato da formazione immagine timelapse. Ritenzione della proteina nel sito di danno del DNA può indicare la riparazione del DNA in corso, mentre la dissociazione della proteina dal sito del danno indotto è spesso considerato un marker per il completamento del BER o SSBR. Tuttavia, c' non è stata alcuna chiara evidenza che collega la dissociazione di XRCC1 da siti di danno del DNA indotto da laser con il completamento della riparazione. Assunzione della proteina al sito di danno è misurata dalla segnalazione intensità media del segnale fluorescente all'interno il ROI danneggiato sopra il segnale fluorescente medio misurato per l'intero nucleo (Figura 1B). Questo tipo di normalizzazione aiuta le fluttuazioni di intensità di indirizzo nel segnale nucleare, anche se altre tecniche di normalizzazione possono essere impiegati a seconda della distribuzione cellulare della proteina di interesse. Qui, XRCC1 è localizzata nel compartimento nucleare, quindi misure di normalizzazione per l'area nucleare la ridistribuzione del segnale al danno ROI. L'intensità di fluorescenza media ROI viene poi registrato per ogni immagine in timelapse, inclusa l'immagine pre-danni e rappresentati graficamente in funzione del tempo (Figura 1C).
Quindi più ulteriormente abbiamo caratterizzato il danno indotto da due dosi di laser selezionato di esaminare la formazione di lesioni della base del DNA. In primo luogo, formazione di CPD, una lesione ingombrante indotta da UV, è stato sondato dall'immunofluorescenza come marcatore per le lesioni di tipo NER (Figura 2A). Quindi, la base oxidatively indotto lesion8-oxodG è stato sondato come marcatore per le lesioni di tipo BER (Figura 2B). Nessun aumento significativo nelle lesioni CPD sono stati osservati presso l'esposizione a basse dosi per due lunghezze d'onda (2 s per 355 nm a 8 fps per 405 nm), mentre l'alta dose di trattamento a due lunghezze d'onda (10 s per 355 nm e 0,5 fps per 405 nm) ha mostrato un aumento significativo in fluorescente segnale osservato all'interno il danno ROI (Figura 2A). Un grafico a dispersione del CPD ROI significa intensità per ogni cella danneggiata spettacoli eterogeneità nella formazione di danni e rilevamento a lunghezze d'onda sia dosi, che indica che un basso livello di lesioni CPD può essere presente alle dosi più basse, ma il carico non può essere significativamente rilevato fino a quando viene applicata una dose maggiore. Il scatterplot suggerisce anche tale inefficienza nella rilevazione dell'anticorpo che può limitare la quantificazione accurata delle miscele di danni.
Questo è ulteriormente evidenziato nella rilevazione delle lesioni del DNA oxidatively indotte dal marcatore 8-oxodG. Nessun chiaro aumento del segnale fluorescente all'interno del danno ROI è stata osservata per 8-oxodG a lunghezza d'onda laser o dose utilizzata (Figura 2B). L'anticorpo utilizzato per questo lavoro è coerenza con precedenti pubblicazioni9,10,36; Tuttavia, dovrebbe essere notato che ci possono essere limitazioni nell'osservare la formazione di 8-oxodG con anticorpi37,38. Conferma della mancanza di lesioni oxidatively indotte è consigliato anche da un secondo marker, come assunzione di 8-Oxoguanine DNA glicosilasi (OGG1), l'enzima responsabile della rimozione di 8-oxodG dal DNA10. Non abbiamo osservato OGG1 reclutamento ai nostri siti di danno del DNA; Tuttavia, la formazione di bassi livelli di danno oxidatively indotto del DNA non può essere esclusa completamente.
Infine, abbiamo esaminato la formazione di DSBs utilizzando due indicatori, γH2AX e 53BP-1, presso il selectele dosi d laser mediante immunofluorescenza (Figura 3 & 4). ΓH2AX è comunemente usato come un indicatore di rottura del filo, ma la sua specificità per DSBs è stato interrogato in un certo numero di rapporti39,40. Inoltre, è un evento di fosforilazione che si propaga dal sito di rottura del filo, quindi localizzazione del segnale di una rottura del filo può essere limitata a causa di questa propagazione del segnale. Pertanto, combinando γH2AX con 53BP-1 consente una verifica più accurata della formazione di DSB entro il danno ROI.
La risposta di γH2AX e 53BP-1 alla micro-irradiazione è lunghezza d'onda sia dose-dipendente. La dose bassa (2 s) stimolazione a 355 nm non suscita nessuna risposta alle 5 e 20 min e una risposta debole e variabile min 10 post irradiazione (Figura 3A) per entrambi gli indicatori. La dose elevata (10 s) di 355 nm micro-irradiazione induce un segnale fluorescente aumentato entro il danno ROI a 5, 10 e 20 min post irradiazione che viene ridotto a 40 min (Figura 3B). Questi risultati indicano che un'attenta titolazione della dose 355 nm è necessaria per ridurre al minimo la croce-stimolazione delle vie di riparazione, come dimostra la rilevazione ridotta del doppio filamento pausa marcatore γH2AX presso i primi punti di tempo (< 20 min) alla dose bassa applicato.
Esperimenti simili sono stati eseguiti utilizzando sia bassa (8 fps) e stimolazione del laser ad alte dosi (0,5 fps) 405 nm (Figura 4). A questa lunghezza d'onda accumulazione significativa di intensità fluorescente all'interno del danno ROI è stata osservata per 53BP-1 e γH2AX indipendentemente dalla dose applicata, che indica che queste dosi di generare quasi una miscela complessa di rotture del filo singolo e doppio immediato dopo l'induzione di danno del DNA (Figura 4). Inoltre, le dosi elevate di spettacolo di 405 nm un aumento nella macchiatura γH2AX pan-nucleare entro 10 min di induzione di danno (Figura 4B, in basso) che complica rilevamento del danno ROI di report, mentre l'accumulo di 53BP-1 è più contenuti all'interno del danno ROI.
Questi risultati dimostrano chiaramente che non è appropriato per il monitoraggio SSBR o BER 405 nm micro-irradiazione, e che più marcatori per DNA addotti e rotture del filo dovrebbero essere impiegato per caratterizzare le lesioni indotte e le risposte di riparazione del DNA.
Alterazione di dose-dipendente nel reclutamento e il mantenimento di XRCC1-GFP
Una volta che il danno al DNA indotto è stato caratterizzato, micro-irradiazione del laser può essere una piattaforma ideale per studiare la dinamica delle proteine di riparazione del DNA. La cinetica di ritenzione e la dissociazione della XRCC1-GFP Mostra una dipendenza di dose (Figura 1), che non è inaspettata dato l'induzione di miscele di diversi danni di ogni lunghezza d'onda. Le dosi elevate di irradiamento (10 s e 0,5 fps) mostrano una maggiore assunzione di intensità di XRCC1-GFP rispetto le dosi più basse e più lunga ritenzione della XRCC1-GFP presso il sito di danni nel corso del tempo 20 min (Figura 1C). Questo indica che il danno del DNA creato a dosi più elevate per 355 e a 405 nm è probabile non risolto durante il corso dell'esperimento, che è coerenza con l'aspetto e la conservazione di DSB marcatori, γH2AX e 53BP-1 (figure 3 & 4 ).
Interessante, le dosi più basse danno (2 s e 8 fps) mostrano rapido reclutamento di XRCC1-GFP ai siti danno e dissociazione di XRCC1-GFP nel corso sperimentale tempo ai livelli pre-irradiazione (Figura 1C). Senza la caratterizzazione completa della miscela danni, questo può portare alla conclusione che il SSB e lesioni della base sono completamente risolto utilizzando queste condizioni dannose. Tuttavia, la presenza di γH2AX e 53BP-1 a 40 min per 355 nm e a 5 min per la 405 nm può portare a diverse interpretazioni. Per la dose di s 355 nm 2, la miscela di danni può essere prevalentemente SSB, così l'aspetto degli indicatori DSB a 40 min può indicare che alcune lesioni unrepaired possono portare a DSBs o che DSBs generato da questa energia sono riparati su una scala di tempo più lungo. Le differenze di scala tempo tra riparazione SSBR e DSB sono state precedentemente segnalati28,41,42. Allo stesso modo, la dissociazione di dose bassa (8 fps) 405 nm può indicare un basso livello di SSB o un raggruppamento di SSB rapidamente convertiti in DSBs, che è stato notato per alta danno micro-irradiazione e altri agenti offensivi del DNA precedentemente43, 44 , 45.
Insieme questi risultati evidenziano l'importanza di caratterizzare le miscele di danno indotto e utilizzando più DNA riparare le proteine e marcatori per interpretare il reclutamento e il mantenimento del DNA ripristino le proteine nei siti di danno indotto.
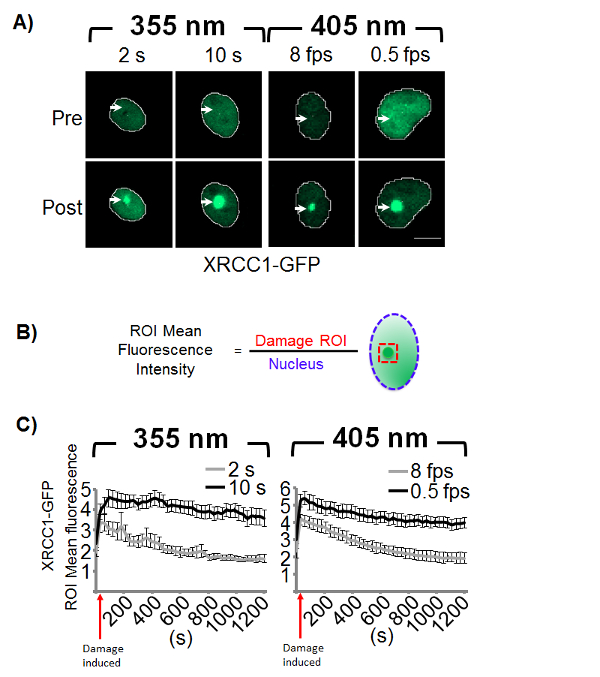
Figura 1 . Micro-irradiazione laser induce reclutamento di XRCC1-GFP.
(A) CHO-K1 le cellule che esprimono XRCC1-GFP sono state irradiate e imaged prima e immediatamente dopo induzione di danno. Le frecce indicano la posizione della dose di micro-irradiazione e la barra della scala è 10 µm. (B) reclutamento è misurata determinando l'intensità di fluorescenza media entro il danno ROI e normalizzante per la media intensità di fluorescenza del nucleo intero. (C) dinamica di reclutamento può essere misurato per ogni immagine della webcam. Grafici sono rappresentativi di due esperimenti indipendenti con barre di errore che rappresenta il SEM (n = 24). Clicca qui per visualizzare una versione più grande di questa figura.

Figura 2 . Micro-irradiazione laser induce il danno del nucleotide.
Cellule CHO-K1 è stata sottoposta per micro-irradiazione laser e risolti immediatamente dopo induzione di danno. L'immunofluorescenza è stata effettuata per rilevare CPD e 8-oxodG addotti. (A) top, dispersione della fluorescenza media ROI intensità osservata in cellule danneggiate dopo colorazione CPD. Fondo, immagini rappresentative di macchiatura CPD. Le frecce indicano la posizione di micro-irradiazione e la barra della scala è di 10 µm (n = 12). (B) immagini rappresentative per 8-oxodG macchiatura. Frecceindicare la posizione di micro-irradiazione e la barra della scala è 10 µm. Clicca qui per visualizzare una versione più grande di questa figura.
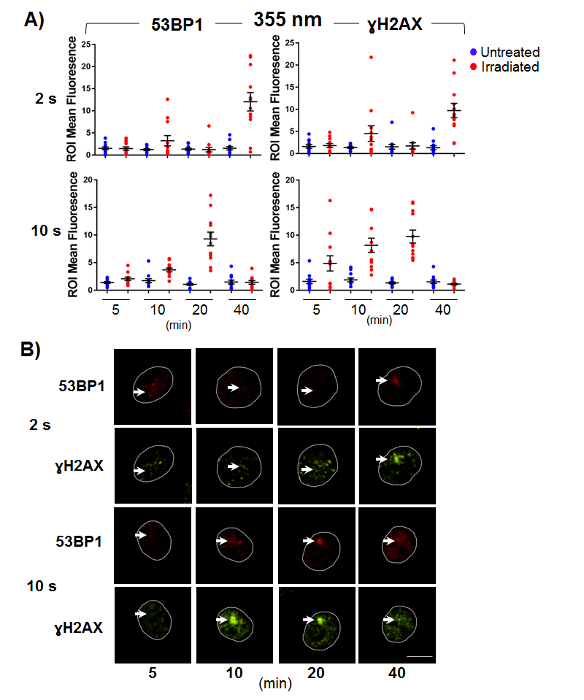
Figura 3 . Indicatori di rottura doppio filamento del DNA rispondono a 355 nm micro-irradiazione in un modo dipendente dalla dose.
Cellule CHO-K1 sono stati sottoposti a micro-irradiazione e fissate a tempo punti indicato post-stimolazione. Immunofluorescenza per il DSB marcatori γH2AX e 53BP-1 è stato effettuato. (A) tracciato di normalizzato danni ROI significa intensità di fluorescenza misurata per non danneggiate e danneggiate le cellule a dispersione. Barre di errore sono rappresentante del SEM (n = 12). (B) rappresentante immagini per γH2AX e 53BP-1 macchiatura. Le frecce indicano la posizione di micro-irradiazione e la barra della scala è 10 µm. Clicca qui per visualizzare una versione più grande di questa figura.
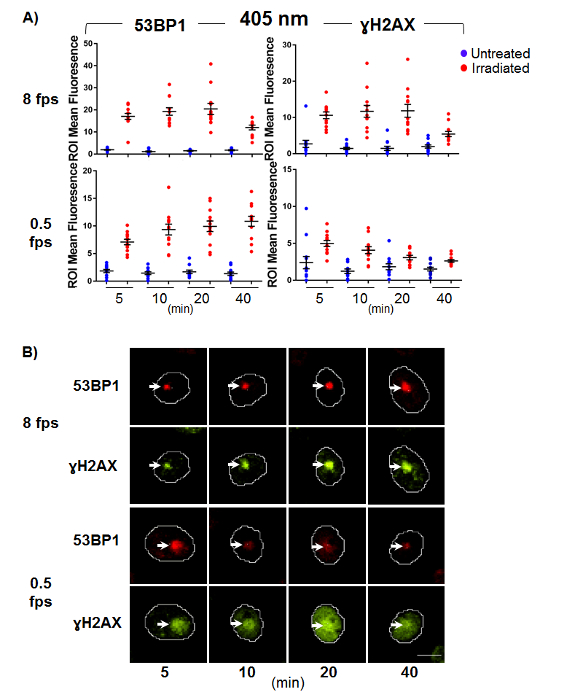
Nella figura 4. Indicatori di rottura doppio filamento del DNA robustamente rispondere a 405 nm micro-irradiazione.
Cellule CHO-K1 sono stati sottoposti a micro-irradiazione e fissate a tempo punti indicato post stimolazione. Immunofluorescenza per il DSB marcatori γH2AX e 53BP-1. (A) tracciato di normalizzato danni ROI significa intensità di fluorescenza misurata per non danneggiate e danneggiate le cellule a dispersione. Barre di errore sono rappresentante del SEM (n = 12). (B) rappresentante immagini per γH2AX e 53BP-1 macchiatura. Le frecce indicano la posizione di micro-irradiazione e la barra della scala è 10 µm. Clicca qui per visualizzare una versione più grande di questa figura.
Discussione
Utilizzando le condizioni descritte qui, qualsiasi microscopio confocale con un UV-A o laser a 405 nm, integrato dal produttore o aggiunti dall'utente finale, può indurre il danno del DNA all'interno del nucleo di una cellula e permettere l'assunzione di proteine di riparazione del DNA nel sito di ha indotto il danno del DNA da monitorare. Tuttavia, come indicato nel protocollo e risultati rappresentativi, selezione di lunghezza d'onda, applicato il potere, e caratterizzazione del danno sono tutti i più importanti problemi che devono essere affrontati per primi dall'utente al fine di studiare un DNA specifico percorso di riparazione. Inoltre, ci sono altre considerazioni nel disegno sperimentale che dovrà essere considerato anche dagli utenti.
Al fine di monitorare il comportamento di una proteina in cellule vive post micro-irradiazione, una proteina fluorescente identificata deve essere creata. La disponibilità di una vasta gamma di proteine fluorescenti che possono essere separati spettralmente offre strumenti per creare fusioni di proteina che possono essere utilizzate per la caratterizzazione delle risposte cellulari all'induzione di danno del DNA. Trasfezione transiente di proteine fluorescenti di interesse permette per lo screening rapido di condizioni dannose, mutazioni o anche inibitori, mentre cellule trasfettate stabilmente offrono un maggiore controllo sui livelli di espressione e permettono per altri biochimici caratterizzazioni da eseguire, convalida dei risultati micro-irradiazione. Spettralmente distinte proteine fluorescenti possono essere utilizzate anche nella stessa cella per monitorare le interazioni tra le proteine all'interno della stessa via o in vie differenti. Le proteine fluorescenti anche eliminano la necessità per gli anticorpi specifici per ogni proteina di interesse e consentono cellule vive monitoraggio del comportamento di proteina per lunghi periodi di tempo dopo l'induzione di danno.
Tuttavia, l'uso di proteine fluorescenti ha anche svantaggi. Senza background genetici carenti in proteine di interesse, proteine endogene saranno in concorrenza con le proteine fluorescente contrassegnate. Coniugazione del tag fluorescenti fino al capolinea N o C della proteina può alterare il protein folding, ostacolare interazioni proteina-proteina chiave o bloccare i segnali di traslocazione; alterando la funzione e potenzialmente influire sulle dinamiche di reclutamento. Questi effetti sono stati dimostrati in un certo numero di rapporti dove macchiatura immunofluorescente ha dimostrato drammaticamente diverso reclutamento e tempi di ritenzione per proteine endogene al sito danno28. Uso di trasfezione transiente o cloni stabili possa anche alterare la risposta osservata, in genere attraverso le variazioni nei livelli di espressione della proteina. Ulteriormente, l'uso di proteine fluorescenti multipli in un singolo esperimento cambiano anche le dinamiche di reclutamento, se i livelli della proteina non sono equilibrati bene o se l'assunzione della proteina fluorescente etichetta più grande blocca il reclutamento di altre proteine . Infine, le proteine fluorescenti possono agire come agenti di sensibilizzazione debole, aumentando la formazione di specie reattive dell'ossigeno e potenzialmente alterare il DNA danni miscele indotto46,47. Nonostante questi inconvenienti, proteine fluorescenti offrono una serie di vantaggi distinti nel DNA studio la riparazione con micro-irradiazione, e se gli utenti incorporare controlli appropriati, quali la caratterizzazione del danno qui descritto, può fornire la nuova comprensione in danno di interazioni proteina-proteina e risposta3.
Se live cell imaging non è necessaria, immunofluorescenza può essere utilizzato per monitorare la risposta al danno indotto. Fissando le cellule in momenti specifici post induzione di danno e macchiando con gli anticorpi specifici per proteine e lesioni di interesse, snapshot statici di induzione di danno e il reclutamento e il mantenimento delle riparazione proteine possono essere costruite. Anticorpi possono essere utilizzati per monitorare l'assunzione di più proteine di interesse e/o modificazioni post-traduzionali indotti dalla risposta al danno al DNA. Uso dell'immunofluorescenza Elimina la necessità di proteine fluorescenti e consente di comportamento di proteina endogena da esaminare. Tuttavia, questo metodo ha anche i suoi svantaggi. Gli anticorpi altamente specifici sono necessari, e permeabilizzazione e procedure di bloccare devono essere ottimizzate per consentire il rilevamento di reclutamento con sufficiente intensità del segnale. Procedure di fissaggio non sono istantanee, e questo vincolo fisico limita la risoluzione temporale di questo approccio. Mappatura del sito di induzione di danno così le cellule possono essere ri-si trova dopo la colorazione può anche presentare sfide significative. Il microscopio confocale utilizzato in questo lavoro permette registrazione basata su immagine come descritto in precedenza, quindi le cellule danneggiate possono essere spostate con alta precisione. Se tappa registrazione o acidato coprioggetto non è disponibile, il tempo investimenti coinvolti nella rilocazione cellule senza registrazione di fase, accoppiato con il ritardo inerente tra induzione di danno e il reclutamento imaged, possono rendere immunofluorescenza poco attraente per alcuni utenti. Tuttavia, i disegni sperimentali per il micro-irradiazione più accurati e completi incorporerà questi tipi di approcci in parallelo con l'uso di proteine fluorescenti, come descritto nel protocollo presentato.
Qui, sia live cell imaging e immunofluorescenza viene utilizzato per illustrare l'utilità del micro-irradiazione del laser. Macchiatura immunofluorescente permette di esaminare accuratamente la miscela di DNA danni creati e l'assunzione di proteine indotte da ogni potere del laser, che ci permettono di meglio interpretare le alterazioni osservate nel reclutamento e il mantenimento di XRCC1-GFP. Basato su questi risultati, l'uso di 405 nm laser deve essere limitato per l'esame delle proteine BER e SSBR. Ulteriormente, il disegno sperimentale ottimo dovrebbe includere misure di potenza dopo l'obiettivo, una caratterizzazione miscela danno completo per ogni linea cellulare utilizzato e la convalida del reclutamento e conservazione osservata in fluorescente contrassegnati proteine con immunofluorescenza. Ovviamente, attrezzature, tempo e considerazioni di costo possono rendere questi disegni sperimentali ottimali Impossibile per alcuni utenti. Tuttavia, l'importanza di ciascuno di questi elementi è dimostrata qui, e gli utenti dovrebbero tenere a mente queste considerazioni quando iniziano gli esperimenti micro-irradiazione.
Divulgazioni
Gli autori non hanno nulla a rivelare.
Riconoscimenti
Gli autori vorrei ringraziare il Dr. Samuel H. Wilson presso il National Institute of Environmental Health Sciences per le linee cellulari utilizzate in questo lavoro.
Materiali
| Name | Company | Catalog Number | Comments |
| Nikon A1rsi laser scanning confocal microscope | Nikon | ||
| NIS Elements software | Nikon | ||
| 355 nm laser | PicoQuant VisUV | Radiation source | |
| Galvanometer photoactivation miniscanner | Bruker | ||
| Microscope slide photodiode power sensor | THORLabs | S170C | |
| Fiber photodiode power sensor | THORLabs | S150C | |
| Digital handheld optical power and energy meter | THORLabs | PM100D | |
| CHO-K1 | From Dr. Samuel H. Wilson, NIEHS | ||
| Minimal essential media | Hyclone | SH3026501 | |
| Fetal bovine serum | Atlanta biologicals | S11550 | |
| XRCC1-GFP | Origene | RG204952 | |
| Jetprime | Polyplus transfection | 11407 | |
| Geneticin | ThermoFisher | 10131035 | |
| 4 chambered coverglass | ThermoFisher | 155382 | |
| 8 chambered coverglass | ThermoFisher | 155409 | |
| Anti 53BP-1 | Novus | NB100304 | |
| Anti-phospho-histone H2AX | Millipore | 05-636-I | |
| Anti cyclobutane pyrimidine dimer | Cosmo Bio clone | CAC-NM-DND-001 | |
| Anti 8-oxo-2´-deoxyguanosine | Trevigen | 4354-MC-050 | |
| Alexa 488 goat anti-mouse | ThermoFisher | A11029 | |
| Alexa 546 goat anti-rabbit | ThermoFisher | A11010 | |
| 4’,6-Diamidino-2-Phenylindole (DAPI) | ThermoFisher | R37606 | Caution toxic! |
| Phosphate buffered saline | ThermoFisher | 0780 | |
| Normal goat serum | ThermoFisher | 31873 | |
| Bovine serum albumin (BSA) | Jackson Immuno Research | 001-000-162 | |
| 37% Formaldehyde | ThermoFisher | 9311 | Caution toxic! |
| Ethanol | Decon Labs | 2716 | |
| Methanol | VWR | BDH1135 | Caution toxic! |
| HCl | Fisher | SA49 | |
| Sodium azide | Sigma-Aldrich | S2002 | Caution toxic! |
| Tris Hydrochloride | Amresco | O234 | |
| Triton X-100 | Sigma-Aldrich | T8787 |
Riferimenti
- Luijsterburg, M. S., et al. Stochastic and reversible assembly of a multiprotein DNA repair complex ensures accurate target site recognition and efficient repair. J Cell Biol. 189 (3), 445-463 (2010).
- Vermeulen, W. Dynamics of mammalian NER proteins. DNA Repair (Amst). 10 (7), 760-771 (2011).
- Berns, M. W. A history of laser scissors (microbeams). Methods Cell Biol. 82, 1-58 (2007).
- Cremer, C., Cremer, T., Zorn, C., Schoeller, L. Effects of laser uv-microirradiation (lambda = 2573 A) on proliferation of Chinese hamster cells. Radiat Res. 66 (1), 106-121 (1976).
- Cremer, C., Cremer, T., Fukuda, M., Nakanishi, K. Detection of laser--UV microirradiation-induced DNA photolesions by immunofluorescent staining. Hum Genet. 54 (1), 107-110 (1980).
- Walter, J., Cremer, T., Miyagawa, K., Tashiro, S. A new system for laser-UVA-microirradiation of living cells. J Microsc. 209, 71-75 (2003).
- Kielbassa, C., Roza, L., Epe, B. Wavelength dependence of oxidative DNA damage induced by UV and visible light. Carcinogenesis. 18 (4), 811-816 (1997).
- Kielbassa, C., Epe, B. DNA damage induced by ultraviolet and visible light and its wavelength dependence. Methods Enzymol. 319, 436-445 (2000).
- Kong, X., et al. Comparative analysis of different laser systems to study cellular responses to DNA damage in mammalian cells. Nucleic Acids Res. 37 (9), 68 (2009).
- Lan, L., et al. In situ analysis of repair processes for oxidative DNA damage in mammalian cells. Proc Natl Acad Sci U S A. 101 (38), 13738-13743 (2004).
- Krasin, F., Hutchinson, F. Strand breaks and alkali-labile bonds induced by ultraviolet light in DNA with 5-bromouracil in vivo. Biophys J. 24 (3), 657-664 (1978).
- Krasin, F., Hutchinson, F. Double-strand breaks from single photochemical events in DNA containing 5-bromouracil. Biophys J. 24 (3), 645-656 (1978).
- Rosenstein, B. S., Setlow, R. B., Ahmed, F. E. Use of the dye Hoechst 33258 in a modification of the bromodeoxyuridine photolysis technique for the analysis of DNA repair. Photochem Photobiol. 31 (3), 215-222 (1980).
- Cremer, T., Peterson, S. P., Cremer, C., Berns, M. W. Laser microirradiation of Chinese hamster cells at wavelength 365 nm: effects of psoralen and caffeine. Radiat Res. 85 (3), 529-543 (1981).
- Limoli, C. L., Ward, J. F. A new method for introducing double-strand breaks into cellular DNA. Radiat Res. 134 (2), 160-169 (1993).
- Carlsson, K., Mossberg, K., Helm, P. J., Philip, J. Use of UV excitation in confocal laser scanning fluorescence microscopy. Micron and Microscopica Acta. 23 (4), 413-428 (1992).
- Stelzer, E. H. K., Haar, F. -. M. Confocal Microscopy: Recent Developments. Advances in Imaging and Electron Physics. 106, 293-345 (1999).
- Rogakou, E. P., Boon, C., Redon, C., Bonner, W. M. Megabase chromatin domains involved in DNA double-strand breaks in vivo. J Cell Biol. 146 (5), 905-916 (1999).
- Tashiro, S., Walter, J., Shinohara, A., Kamada, N., Cremer, T. Rad51 accumulation at sites of DNA damage and in postreplicative chromatin. J Cell Biol. 150 (2), 283-291 (2000).
- Gassman, N. R., Stefanick, D. F., Kedar, P. S., Horton, J. K., Wilson, S. H. Hyperactivation of PARP triggers nonhomologous end-joining in repair-deficient mouse fibroblasts. PLoS One. 7 (11), 49301 (2012).
- Bolin, C., et al. The impact of cyclin-dependent kinase 5 depletion on poly(ADP-ribose) polymerase activity and responses to radiation. Cell Mol Life Sci. 69 (6), 951-962 (2012).
- Godon, C., et al. PARP inhibition versus PARP-1 silencing: different outcomes in terms of single-strand break repair and radiation susceptibility. Nucleic Acids Res. 36 (13), 4454-4464 (2008).
- Hanssen-Bauer, A., et al. XRCC1 coordinates disparate responses and multiprotein repair complexes depending on the nature and context of the DNA damage. Environ Mol Mutagen. 52 (8), 623-635 (2011).
- Mortusewicz, O., Amé, J. C., Schreiber, V., Leonhardt, H. Feedback-regulated poly(ADP-ribosyl)ation by PARP-1 is required for rapid response to DNA damage in living cells. Nucleic Acids Res. 35 (22), 7665-7675 (2007).
- Mortusewicz, O., Leonhardt, H., Cardoso, M. C. Spatiotemporal dynamics of regulatory protein recruitment at DNA damage sites. J Cell Biochem. 104 (5), 1562-1569 (2008).
- Mortusewicz, O., Rothbauer, U., Cardoso, M. C., Leonhardt, H. Differential recruitment of DNA Ligase I and III to DNA repair sites. Nucleic Acids Res. 34 (12), 3523-3532 (2006).
- Solarczyk, K. J., Zarębski, M., Dobrucki, J. W. Inducing local DNA damage by visible light to study chromatin repair. DNA Repair (Amst). 11 (12), 996-1002 (2012).
- Gassman, N. R., Wilson, S. H. Micro-irradiation tools to visualize base excision repair and single-strand break repair. DNA Repair (Amst). 31, 52-63 (2015).
- Botchway, S. W., Reynolds, P., Parker, A. W., O'Neill, P. Use of near infrared femtosecond lasers as sub-micron radiation microbeam for cell DNA damage and repair studies. Mutat Res. 704 (1-3), 38-44 (2010).
- Ferrando-May, E., et al. Highlighting the DNA damage response with ultrashort laser pulses in the near infrared and kinetic modeling. Front Genet. 4, 135 (2013).
- Lan, L., et al. Accumulation of Werner protein at DNA double-strand breaks in human cells. J Cell Sci. 118, 4153-4162 (2005).
- Salmon, T. B., Evert, B. A., Song, B., Doetsch, P. W. Biological consequences of oxidative stress-induced DNA damage in Saccharomyces cerevisiae. Nucleic Acids Res. 32 (12), 3712-3723 (2004).
- Caldecott, K. W. DNA single-strand break repair. Exp Cell Res. 329 (1), 2-8 (2014).
- London, R. E. The structural basis of XRCC1-mediated DNA repair. DNA Repair (Amst). 30, 90-103 (2015).
- Moser, J., et al. Sealing of chromosomal DNA nicks during nucleotide excision repair requires XRCC1 and DNA ligase III alpha in a cell-cycle-specific manner. Mol Cell. 27 (2), 311-323 (2007).
- Campalans, A., et al. Distinct spatiotemporal patterns and PARP dependence of XRCC1 recruitment to single-strand break and base excision repair. Nucleic Acids Res. 41 (5), 3115-3129 (2013).
- Cooke, M. S., Lunec, J. . Immunochemical detection of oxidative DNA damage. Vol. I. , 275-293 (2003).
- Rossner, P., Sram, R. J. Immunochemical detection of oxidatively damaged DNA. Free Radic Res. 46 (4), 492-522 (2012).
- Cleaver, J. E., Feeney, L., Revet, I. Phosphorylated H2Ax is not an unambiguous marker for DNA double-strand breaks. Cell Cycle. 10 (19), 3223-3224 (2011).
- Rybak, P., et al. Low level phosphorylation of histone H2AX on serine 139 (γH2AX) is not associated with DNA double-strand breaks. Oncotarget. 7 (31), 49574-49587 (2016).
- Haince, J. F., et al. PARP1-dependent kinetics of recruitment of MRE11 and NBS1 proteins to multiple DNA damage sites. J Biol Chem. 283 (2), 1197-1208 (2008).
- Caldecott, K. W. Single-strand break repair and genetic disease. Nat Rev Genet. 9 (8), 619-631 (2008).
- Lomax, M. E., Gulston, M. K., O'Neill, P. Chemical aspects of clustered DNA damage induction by ionising radiation. Radiat Prot Dosimetry. 99 (1-4), 63-68 (2002).
- Ma, W., Westmoreland, J. W., Gordenin, D. A., Resnick, M. A. Alkylation base damage is converted into repairable double-strand breaks and complex intermediates in G2 cells lacking AP endonuclease. PLoS Genet. 7 (4), 1002059 (2011).
- Siddiqi, M. A., Bothe, E. Single- and double-strand break formation in DNA irradiated in aqueous solution: dependence on dose and OH radical scavenger concentration. Radiat Res. 112 (3), 449-463 (1987).
- Sano, Y., Watanabe, W., Matsunaga, S. Chromophore-assisted laser inactivation--towards a spatiotemporal-functional analysis of proteins, and the ablation of chromatin, organelle and cell function. J Cell Sci. 127, 1621-1629 (2014).
- Wojtovich, A. P., Foster, T. H. Optogenetic control of ROS production. Redox Biol. 2, 368-376 (2014).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon