Method Article
Glicoproteômica da matriz extracelular: um método de análise GLICOP�TIDOS Intact Usando Espectrometria de Massa
Neste Artigo
Resumo
Este artigo descreve um método para preparar as amostras de tecido para análise MS cardiovasculares que permite (1) a análise da composição de proteínas de ECM, (2) a identificação de locais de glicosilação, e (3) a caracterização da composição de formas de glicano. Esta metodologia pode ser aplicada, com pequenas modificações, para o estudo da ECM em outros tecidos.
Resumo
A fibrose é uma característica de muitas doenças cardiovasculares e está associada com a secreção exacerbada e deposição da matriz extracelular (ECM). Usando proteómica, já anteriormente identificados mais de 150 ECM e proteínas ECM-associado em tecidos cardiovasculares. Notavelmente, muitas proteínas de ECM são glicosiladas. Esta modificação pós-translacional afecta proteína de dobragem, a solubilidade, a ligação e degradação. Nós desenvolvemos uma extracção sequencial e método para o enriquecimento de proteínas de ECM que é compatível com a espectrometria de massa em tandem com cromatograf ia líquida subsequente análise (LC-MS / MS) de glicopéptidos intactas. A estratégia baseia-se em incubações sequenciais com NaCl, SDS para descelularização de tecidos, e cloridrato de guanidina para a solubilização de proteínas de ECM. Os avanços recentes em LC-MS / MS de fragmentação incluem métodos, tais como combinações de dissociação colisão de maior energia (HCD) e transferência de electrões de dissociação (ETD), que permitema análise da composição de glicopéptidos directa de proteínas de ECM. No presente documento, descrevemos um método para preparar a ECM a partir de amostras de tecido. O método permite, não só para o perfil de proteína mas também a avaliação e caracterização de glicosilação por análise por MS.
Introdução
A fibrose é uma característica de muitas doenças. Fibroblastos proliferam e se diferenciam em direcção fenótipos altamente sintéticas, que estão associadas com a secreção exacerbada e deposição de matriz extracelular (ECM) 1. deposição de ECM excessivo pode continuar, mesmo após a lesão inicial diminuiu, levando a incapacidade funcional. Usando proteómica, já anteriormente identificados mais de 150 ECM e proteínas ECM-associado em tecido cardíaco, 2, 3. Eles não são apenas proteínas estruturais, mas também proteínas matricellular e proteases que contribuem para a remodelação contínua e adaptação dinâmica do coração. Notavelmente, muitas proteínas de ECM são glicosiladas 4. Esta modificação pós-translacional (PTM) envolve a adição de resíduos de açúcar a certas posies de aminoidos, e que afecta o dobramento de proteínas, a solubilidade, a ligação e degradação 5 .
Existem dois tipos principais de glicosilação que ocorrem em mamíferos. (1) N-glicosilação ocorre no azoto da carboxamida dos resíduos de asparagina (Asn) na sequcia de consenso Asn-Xaa-Tre / Ser, em que Xaa é qualquer aminoácido excepto prolina. (2) Em O-glicosilação, resíduos de açúcar anexar a resíduos de serina e treonina (Ser, Thr) ou, em muito menor extensão, a hidroxiprolina e hidroxilisina. Enquanto o O-glicosilação pode ocorrer em uma variedade de grupos de proteínas, N-glicosilação é restrito para proteínas secretadas ou domínios extracelulares de proteínas de membrana 5. Isso faz com que a N-glicosilação num alvo atractivo quando se estuda a ECM.
Proteomics estabelece um novo padrão para a análise das mudanças da proteína na doença. Até agora, a maioria dos estudos de proteômica têm sido focados na proteínas intracelulares 6. Isto é principalmente devido às seguintes razões. Em primeiro lugar, as proteínas intracelulares abundantes dificultar a identificaçãocação de componentes ECM escassos. Isto é particularmente importante no tecido cardíaco, em que representam proteínas mitocondriais e miofilamentos para uma grande proporção do teor de proteína 7. Em segundo lugar, as proteínas de ECM integrais estão fortemente reticulados e difíceis de solubilizar. Por fim, a presença de PTMs abundantes (isto é, glicosilação) altera a massa molecular, carga e propriedades electroforéticos de péptidos, que afectam tanto a separação e a identificação por espectrometria de massa em tandem cromatografia luida (LC-MS / MS). Nos últimos anos, temos desenvolvido e melhorado uma extracção sequencial e método para o enriquecimento de proteínas de ECM que é compatível com a análise subsequente espectrometria de massa (MS). A estratégia baseia-se incubações sequenciais.
A primeira etapa é realizada com NaCl, num tampão iónico que facilita a extracção de proteínas associadas a ECM e de ECM fracamente ligada, bem como proteínas de ECM recentemente sintetizadas. ele is detergente livre, não-desnaturação, não disruptiva de membranas celulares, e mais tratável através de ensaios bioquímicos 8. Em seguida, a descelularização é conseguida com dodecil sulfato de sódio (SDS). Neste passo, uma baixa concentração de SDS assegura a desestabilização da membrana e a libertação de proteínas intracelulares e a impedir a ruptura dos componentes da ECM não integrais mais solúveis. Finalmente, as proteínas de ECM são extraídos com um tampão de cloridrato de guanidina (GuHCl). GuHCl é eficaz na extracção de proteínas e proteoglicanos fortemente reticulados a partir de tecidos, tais como tendões, cartilagens 9 10, vasos 11, 12, 13 e o coração 2, 3. Aplicamos este fraccionamento bioquímico, em combinação com a LC-MS / MS, para explorar a remodelação de ECM na doença cardiovascular dois , 3, 11, 12, 13, 14. Os avanços recentes em MS incluem novos métodos de fragmentação, tais como combinações de dissociação de alta energia de colisão (HCD) e transferência de electrões de dissociação (ETD), que permitem a análise directa de glicopéptidos intactas 3, 15.
Aqui, descrevemos uma metodologia para preparar a ECM para análise MS que permite a análise da composição de proteína, a identificação dos sítios de glicosilação, e a caracterização das formas de glicano. Em comparação com as análises anteriores do ECM glicosilação 16, esta metodologia permite a avaliação directa da alteração de composição em perfis de glicosilao de uma maneira específica do local, utilizando o MS. Nós aplicamos esta metodologia para tecidos cardiovasculares. No entanto, ele pode alassim ser aplicada, com pequenas modificações, para o estudo da ECM em outras amostras de tecido e pode fornecer informações sem precedentes em biologia ECM.
Protocolo
O estudo foi aprovado pelo Comitê de Ética em Wandsworth local Research (número de referência: 06 / Q0803 / 37) e aprovação institucional recebido do escritório de pesquisa e desenvolvimento. Todos os pacientes deram consentimento informado por escrito.
1. Extração de Proteínas da Matriz Extracelular
NOTA: Os tecidos atriais humanos utilizados para estes experimentos foram obtidos a partir dos apêndices atriais durante a circulação extracorpórea, logo após a parada cardioplégica do coração. Todas as amostras foram coletadas em St. George Hospital, Londres, Reino Unido. Todas as amostras de tecido devem ser congelados a -80 ° C. Não utilizar amostras conservadas com fixadores, tais como paraformaldeído, que as proteínas de ligação cruzada.
- Preparar todos os tampões de extracção em avanço de experiências, conforme a Tabela 1, a fim de minimizar o tempo entre as etapas de extracção. Executar todas as incubações à temperatura ambiente (TA) num ambiente com temperatura controlada(Isto é, ~ 20 ° C) para assegurar a coerência entre as extracções.
- Pesar 20-50 mg de tecido. Se várias amostras são para ser extraída, cortar e pesá-los, um por um, para evitar o descongelamento completo dos tecidos. Utilizando um bisturi, dado o tecido em 3-4 pedaços mais pequenos (isto é, 2-3 mm) e colocá-los em conjunto em tubos de 1,5 mL.
- Adicionar 500 ul de solução salina tamponada com fosfato gelada (PBS; ver Tabela 1 e a Tabela de Materiais) e realizar cinco lavagens para minimizar a contaminação do sangue.
- Extracção Passo 1: A incubação com tampão de NaCl
- Após lavagem com PBS, colocar as amostras em tubos de 1,5 mL com tampas de rosca. Adicionar NaCl (ver Quadro 1) a 10 vezes (v: w) o peso do tecido. Vortex dos tubos à TA durante 1 h, a velocidade mínima (isto é, 600 rpm).
NOTA: A baixa velocidade de vórtice é crítico para evitar a disrupção mecânica do tecido durante este passo.Usar um adaptador de espuma para colocar todos os tubos durante a extracção. - Transferir os extractos para novos tubos e centrifugar a 16000 xg durante 10 min a 4 ° C. Armazenar os extractos à temperatura de -20 ° C até à sua utilização. Lava-se rapidamente as restantes pastilhas de tecido com tampão de NaCl fresco. Usar o mesmo tipo de tampão (isto é, 100 ul de tampão de NaCl) para lavar a impedir a extracção de outras proteínas com diferentes solubilidades (isto é, proteínas não extraíveis com NaCl).
- Após a lavagem, a assegurar a completa remoção do tampão para minimizar a sobreposição no teor de proteína com etapas de extracção subsequentes. Descartar o tampão NaCl a usada para a lavagem.
NOTA: A proporção entre o volume do tampão e o peso do tecido é importante para uma extracção reprodutível. A 10: 1 (v: w) para as extracções de NaCl e SDS e 5: 1 para o passo GuHCl fornecer quantidades suficientes de proteína sem saturar o tampão. As concentrações de proteína são aproximadamente 1-2 mg / mL após extraction.
- Após lavagem com PBS, colocar as amostras em tubos de 1,5 mL com tampas de rosca. Adicionar NaCl (ver Quadro 1) a 10 vezes (v: w) o peso do tecido. Vortex dos tubos à TA durante 1 h, a velocidade mínima (isto é, 600 rpm).
- Extracção Passo 2: Decelularização com tampão SDS
- Adicionar tampão de SDS (Tabela 1) em dez vezes (v: w) o peso do tecido; a utilização de baixas concentrações de SDS (ou seja, 0,1%) é crítico para evitar a perda de proteínas da MEC durante descelularização. Vortex dos tubos à temperatura ambiente durante 16 h a velocidade mínima (isto é, 600 rpm).
NOTA: A baixa velocidade de vórtice minimiza a ruptura mecânica da ECM. - Transferir os extractos para tubos novos. Centrifuga-se a 16000 xg durante 10 min a 4 ° C; armazenar a -20 ° C até à sua utilização. Lava-se rapidamente as pastilhas de tecido restantes com dd H2O para remover o SDS. Assegurar a remoção completa do líquido após a lavagem.
- Adicionar tampão de SDS (Tabela 1) em dez vezes (v: w) o peso do tecido; a utilização de baixas concentrações de SDS (ou seja, 0,1%) é crítico para evitar a perda de proteínas da MEC durante descelularização. Vortex dos tubos à temperatura ambiente durante 16 h a velocidade mínima (isto é, 600 rpm).
- Extracção Passo 3: A incubação com tampão de GuHCl
- Adicionar tampão de GuHCl (Tabela 1) em cinco vezes (v: p) o peso do tecido. Vortex dos tubos à temperatura ambiente durante 72 h a velocidade máxima (isto é, 3.200 rpm); vórtice vigoroso facilita a ruptura mecânica da ECM.
- Transferir os extractos para tubos novos. Centrifuga-se a 16000 × g durante 10 min a 4 ° C e armazenar a -20 ° C até à sua utilização.
2. Proteína Quantificação e Precipitation
NOTA: Devido à presença de detergentes, o tampão de SDS é incompatível com a quantificação directa da proteína com base em medições de absorvância a 280 nm. Para assegurar quantificação reprodutível, utilizar ensaios colorimétricos para todos os extractos de proteína 17.
- Quantificação.
- Preparar padrões para a curva de calibração, utilizando albumina de soro bovino (BSA) diluídas em série no tampão de extracção adequado (por exemplo, NaCl, SDS, ou GuHCl) 17. Durante este tempo, descongelar os extractos da amostra.
- Dilui-se as amostras em tampão de extracção para obter concentrações dentroa gama linear de absorvência; uma diluição 1:10 (v: v) produz resultados satisfatórios. Diluir as amostras de GuHCl com uma quantidade igual de ddH2O para quantificação, como o ensaio colorimétrico não é compatível com concentrações de> 4 M GuHCl.
- Usar um ácido bicinconínico (BCA) -based ensaio colorimétrico 17 (ver a Tabela de Materiais), seguindo as instruções do fabricante para os ensaios em placas de 96 poços; recomenda-se realizar medições pelo menos duplicados.
- Após incubação durante 30 minutos, fazer leituras de absorvância a um comprimento de onda de 570 nm, a fim de calcular a concentração de proteína usando a curva de calibração padrão de BSA 17.
- Protein Precipitation
- Thaw GuHCl extrai à TA. Aliquota de 10 ug de proteína para cada amostra para novos tubos. Para uma análise glicopeptídeo direta, alíquota de 50 mg. Adicionam-se 10 vezes o volume de etanol e incuBATE durante a noite a -20 ° C.
NOTA: GuHCl não é compatível com outras reações enzimáticas e a maioria das aplicações de eletroforese. A remoção de GuHCl é necessária antes deglycosylation e tripsina digestão. A solubilidade de GuHCl em etanol e, por outro lado, a baixa solubilidade de proteínas para permitir a remoção eficaz de GuHCl enquanto obtendo-se aproximadamente uma recuperação de 98% de proteínas de 18. - Centrifugar as amostras a 16000 × g durante 30 min a 4 ° C e aspirar o sobrenadante. Tome cuidado para não perturbar o pellet precipitada. Secam-se os peletes de 15 min, utilizando um concentrador de vácuo (ver a Tabela de Materiais) à TA.
NOTA: O protocolo pode ser interrompido aqui e as pastilhas secas armazenadas a -20 ° C até à sua utilização. - Opcionalmente, executar uma electroforese em gel de controlo de qualidade (QC, ver os métodos suplementares).
- Thaw GuHCl extrai à TA. Aliquota de 10 ug de proteína para cada amostra para novos tubos. Para uma análise glicopeptídeo direta, alíquota de 50 mg. Adicionam-se 10 vezes o volume de etanol e incuBATE durante a noite a -20 ° C.
3. Sequential desglicosilação para oAvaliação da N-glicosilação Ocupação do Site
- Durante a secagem da amostra (ver o passo 2.2.2), preparar o tampão de desglicosilação contendo desramificao enzimas de desglicosilação, conforme indicado na Tabela 1. Veja a Tabela de Materiais para detalhes do produto.
- Adicionar 10 ul de tampão de desglicosilação que contém enzimas para cada amostra. Assegurar a re-suspensão da pelota apropriado através da realização de um vórtice rápida e rotação para baixo de amostras.
NOTA: A remoção de monómeros de açúcar que utilizam enzimas desramificadas é essencial para a subsequente remoção completa e de sacáridos complexos ligados a O e facilita a clivagem posterior de açúcares ligados a N por PNGase-F. - Incubar durante 2 horas a 25 ° C para permitir a remoção de sulfato de heparano por heparinase II.Increase a temperatura para 37 ° C e incubar durante 36 horas com agitação suave.
NOTA: Tendo em conta os volumes de reacção baixas e tempos de incubação prolongados, agitadores utilização da incubadora e firmemente embalar múltiplos 1,5 mL banheiraes dentro de um tubo cónico de 50 ml que inclina-se no interior da incubadora a cerca de 45 °. - Após 36 h, centrifugar as amostras durante 1 min a 16.000 xg e evapora-se o H2O a partir das amostras usando um concentrador de vácuo à temperatura ambiente durante aproximadamente 45 min.
- Ressuspender as amostras secas com 10 mL de H 2 O contendo 18 a 50 U / mL com PNGase-F, que cliva todos os glicanos ligados a asparagina, numa reacção de desamidação.
NOTA: O ácido aspártico resultante transporta um excesso de massa de 2,98 Da, indicativo da presença de N-glicosilação durante a análise de MS. - Incubar durante 36 horas a 37 ° C sob agitação constante no incubador agitador.
4. Na solução de Tripsina-Digestão
NOTA: Este passo deve ser realizado para ambos os (isto é, usados para análise directa de glicopéptido,) e desglicosilados amostras não-desglicosilada (ou seja, utilizado para a avaliação de oc glicanocupancy).
- Utilizar 10 ug de proteína total para a avaliação de ocupação local de N-glicosilação (como indicado no passo anterior). Para amostras destinadas à análise directa glicopéptido, utilizar 50 ug de proteína como a quantidade de partida.
NOTA: As etapas seguintes são descritas para 10 de protea. Dimensionar-se th evolumes (ou seja, 5 vezes para 50 ug), conforme necessário. - Desnaturar as proteínas em cada amostra alíquota usando 9 M de ureia e tioureia 3 M, com concentrações finais de ureia 6 M e 2 M tioureia, respectivamente (por exemplo, para uma amostra de 10 ul, 20 ul de ureia / tioureia).
- Reduzir as proteínas por adição de ditiotreitol 100 mM (DTT, 3,33 mL, concentração final: 10 mM). Incubar a 37 ° C durante 1 h, com agitação a 240 rpm.
- Arrefecer as amostras até à TA antes de realizar a alquilação por adição de iodoacetamida 0,5 M (3,7 mL, concentração final: 50 mM). Incubar no escuro durante 1 h.
- Use pré-arrefecida (-20° C) de acetona (6 vezes o volume da amostra) a incubar as amostras durante a noite a -20 ° C. Precipitar por centrifugação a 14000 xg durante 25 min a 4 ° C.
- Aspirar o sobrenadante. Tome cuidado para não perturbar o pellet precipitada. Secam-se os peletes de proteína usando um concentrador de vácuo durante 30 minutos à temperatura ambiente.
- Ressuspender em 20 mL de bicarbonato de trietilamónio 0,1 M (TEAB), pH 8,2, contendo tripsina (0,01 ug / uL) e digerir durante a noite a 37 ° C e 240 rpm.
- Parar a digestão por acidificação das amostras com ido trifluoroacico a 10% (TFA, 2 mL para uma concentração final de 1% de TFA).
5. Limpeza de Peptídeo Usando colunas C18
NOTA: A remoção de contaminantes de interferência a partir da mistura de péptidos após digestão reduz ião supressão e melhora a relação sinal-para-ruído e cobertura sequência. Esta etapa deve ser realizada tanto para sa não-desglicosilada e desglicosiladamples.
- Activar a resina na placa C18 de rotação (ver a Tabela de Materiais) utilizando 200 uL de metanol por poço e centrifugar a 1000 x g durante 1 min.
- Lavar por adição de 200 ul por poço de 80% de acetonitrilo (ACN) e 0,1% de TFA em H 2 O. Centrifugar a 1.000 xg durante 1 min.
- Equilibra-se por adição de 200 ul por poço de 1% de ACN e 0,1% de TFA em H 2 O. Centrifugar a 1.000 xg durante 1 min. Repita este passo mais duas vezes.
- Carregar as amostras (a totalidade do volume) a partir do passo 4 para os poços que contêm a resina e centrifugar a 1500 x g durante 1 min. Recarregar o fluxo através de uma segunda vez e repetir a centrifugação.
- Lavar por adição de 200 ul por poço de 1% de ACN e 0,1% de TFA em H 2 O. Centrifugar a 1.500 xg durante 1 min. Repita este passo mais duas vezes.
- Elui-se as amostras com 170 uL por poço de 50% de ACN, 0,1% de TFA em H 2 O. Centrifugar a 1.500 xg durante 1 min. Repetir o passo anteriore combinar o eluato recolhido.
- Seca-se o eluído usando um concentrador de vácuo durante 2 h à TA. Se ele não está a ser utilizada imediatamente, manter as amostras secas a -80 ° C até à sua utilização.
NOTA: As amostras destinadas à identificação desglicosilada proteína única está pronto para usar para a LC-MS / MS ap este passo. Passos 6 e 7 não são necessárias para estas amostras. - Descongelar e ressuspender as amostras desglicosiladas em 2% de ACN e 0,05% de TFA em H 2 O para uma concentração de proteína final de 0,5 mg / mL. Prosseguir com o passo 6 para a análise glicopéptido directa de amostras não-desglicosilada.
- Opcionalmente, filtrar os péptidos antes da marcação com etiquetas de massa em tandem (TMT); ver Métodos Suplementares para fitration peptídeo.
6. A marcação com TMT (a análise directa GLICOP�TIDOS Only)
- Descongelar e ressuspender as pastilhas secas em 50 de TEAB 50 mM para se obter uma concentração de 1 ug / uL.
- Resusped o frasco 0,8 mg de TMT zero (0 TMT, ver a Tabela de Materiais) de reagente em 41 mL de ACN. Siga recommendantions do fabricante para a ressuspensão.
- Rotular as amostras de péptidos a uma razão de 50 ug a 0,4 mg péptidos TMT 0 (ou seja, 50 uL de péptidos de 20,5 mL de TMT 0). Incubar à temperatura ambiente durante 1 h.
- Extingue-se a reacção de marcação pela adição de 5% de hidroxilamina a uma razão de 6: 100 (ou seja, 4,23 mL de 5% de hidroxilamina). Incubar à temperatura ambiente durante 15 min.
- Secam-se as amostras de ptidos TMT 0 marcado com durante 1 h a RT utilizando um concentrador de vácuo. Ressuspender em 10? L de ddH 2 O.
NOTA: Devido aos resuos de glicano, glicopéptidos apresentar uma massa molecular maior do que os péptidos não glicosiladas. TMT 0 aumenta o estado de carga de glicopeptídeos. Isto reduz a sua massa em relação ao carregar proporções (m / z) e facilita a fragmentação DTE.
7. glicopeptídeo Enriquecimento
- Usar tampões de reacção fornecidos no kit (ver a Tabela de Materiais).
- Adicionar 50 uL de tampão de ligação a cada amostra 10 ul a partir do passo 6.5. Vortex a solução de resina glycocapture até se tornar homogénea. Usar uma interacção hidrófila cromatografia líquida zwitteriónico (ZIC-HILIC) com base na captura 15.
- Aliquota de 50 uL da suspensão de resina para novos tubos de 1,5 mL. Girar durante 1 min a 2500 x g e eliminar o sobrenadante. Adicionar 60 ul de amostra (isto é, a amostra com tampão de ligação) para os tubos que contêm os grânulos de resina. Misturar utilizando uma pipeta e incubar à temperatura ambiente durante 20 minutos em agitação a 1200 rpm.
- Centrifugar durante 2 minutos a 2000 xg, e transferir o sobrenadante para tubos novos. Mantenha os tubos. Adicionar 150 mL de tampão de lavagem para os tubos de resina. Misturar utilizando uma pipeta e incubar à temperatura ambiente durante 10 minutos em agitação a 1200 rpm.
- Centrifugação durante 2 min a 2500 x g. Transferiro sobrenadante aos mesmos tubos (do passo 7.4). Repetir os passos de lavagem duas vezes.
- Adicionar 75 ul de tampão de eluição e misturar com uma pipeta. Agita-se a 1200 rpm durante 5 min à temperatura ambiente e, em seguida, centrifugar os tubos durante 2 minutos a 2500 x g. Transfira os sobrenadantes para novos tubos de 1,5 mL. Repetir os passos de lavagem e, em seguida, transferir o sobrenadante eluído com o mesmo tubo.
- Centrifugar os tubos contendo o fluido (ou seja, glicopéptidos) durante 2 min a 2500 x g. Transferir o sobrenadante para novos tubos para assegurar a remoção de qualquer resina que sobraram das fases anteriores.
- Seca-se o total de 150 mL de eluído usando um concentrador de vácuo durante cerca de 2 h à TA. Ressuspender as glicopéptidos secou-baixo em 15 mL de 2% de ACN e 0,05% de TFA em ddH 2 O.
- Avance para executar LC-MS / MS utilizando HCD fragmentação para analisar a composio de protea de ECM, e de LC-MS / MS utilizando HCD e ETD fragmentação para caracterização glicopéptido. Consulte a seção 8.
Análise 8. Mass Spectrometry
- Executar LC-MS / MS utilizando HCD fragmentação para analisar a composio de protea de ECM; ver Métodos Suplementares para mais detalhes.
- Executar LC-MS / MS utilizando HCD e ETD fragmentação para caracterização glicoptido (ver os métodos suplementares para detalhes); a amostra enriquecida deve ser comparado com o material de entrada não enriquecido 15.
NOTA: As descrições detalhadas dos métodos de LC-MS / MS para análise indireta glicopeptídeo, análise glicopeptídeo direta e pesquisa de banco de dados são fornecidos nos Métodos Suplementares. Pesquisadores interessados na caracterização de proteínas ECM e composição glicanos utilizando espectrometria de massa são encorajados a referir-se a publicações anteriores 3, 11, 15.
Resultados
Um fluxo de trabalho esquemática do protocolo é fornecida na Figura 1.
protocolo de extracção de ECM
A eficiência da extracção pode ser monitorizada através da execução alíquotas formar cada extracto sobre geles de acrilamida-Tris e Bis usando coloração com prata para visualização. A Figura 2A mostra a complementaridade do NaCl, SDS e GuHCl extrai após extracção sequencial. Este QC permite a identificação de potenciais problemas com a qualidade da amostra, tais como a degradação de proteínas em excesso. Depois de extracção, as glicoproteínas de ECM são abundantes nos extractos GuHCl (Figura 2B).
desglicosila�o
Para avaliar a eficiência de deglycosylation, um controle não-desglicosilada deve ser executado no paParalela. Desglicosilação vezes têm de ser adequados para alcançar uma remoção completa e homogénea dos resíduos de açúcar, tal como exemplificado na Figura 3A. A Figura 3B mostra um exemplo representativo de amostras de forma eficiente desglicosilada pela adição de enzimas para enzimas de remoção de GAG e de desglicosilação que se destinam a menores N- e O-ligados oligossacáridos.
glicoproteômica
O protocolo para a avaliação da ocupação de sequons NXT / S melhora a cobertura da sequência da proteína para glicoproteínas de ECM após MS (Figura 3C) e permite uma triagem inicial da presença de glicoproteínas. Isso ajuda a reduzir o tempo de busca de glicopeptídeos, como bancos de dados pode ser personalizado para conter glicoproteínas previamente identificados. HCD-ETD fragmentação é utilizado para a identificação e caracterização da composição de oligossacáridos ligados a ECM g lycoproteins. A Figura 4A mostra um espectro representativo obtido para um péptido marcado com 18O após desglicosilação (análise glicopéptido indirecta). A Figura 4B é um exemplo representativo de um espectro obtido após análise de glicopéptidos intactas de extractos de ECM (análise glicopéptido directa).

Figura 1: Processo de Síntese. (A) após enriquecimento sequencial de proteínas de ECM, as análises de LC-MS / MS são realizados nos extractos desglicosiladas. (B) Alternativamente, extractos de ECM não são desglicosiladas ainda enriquecido para glicopéptidos. Por favor clique aqui para ver uma versão maior desta figura.
2" src = "/ files / ftp_upload / 55674 / 55674fig2.jpg" />
Figura 2: Extracção de proteínas de ECM. (A) Os extractos 3 diferentes do procedimento de extracção sequencial ( "Inglês Quickst") são complementares em seu teor de proteínas. Enquanto extractos de SDS são enriquecidos em proteínas intracelulares, extractos GuHCl conter a maioria das proteínas da MEC. fracionamento de sucesso é visualizado pelo padrão de coloração de prata diferente. Proteas (B) ECM, tais como a pequena rica em leucina proteoglicanos decorina, biglicano e mimecan são predominantemente detectada nos extractos GuHCl, com pouca presença nos extractos de SDS e NaCl. Por favor clique aqui para ver uma versão maior desta figura.
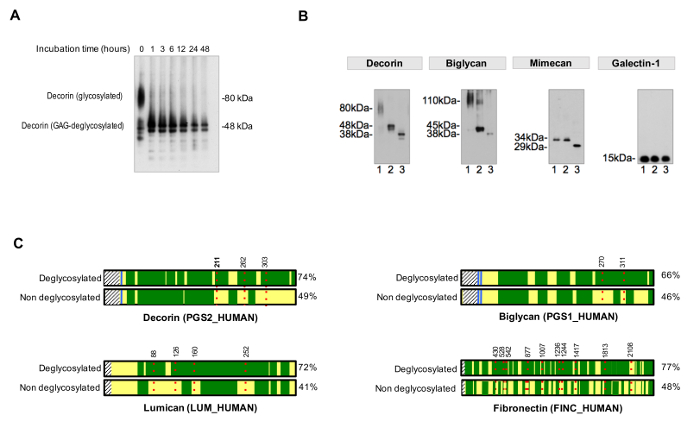
Figura 3. Análise de Glycosylation. Os tempos de incubação apropriados (A) são necessários para a desglicosilação completa. O exemplo mostra o efeito do tempo de incubação durante a remoção de cadeias de glicosaminoglicanos da decorina glicoproteína. Glicoproteínas (B) de ECM são decorados com correntes grandes e repetidas de glicosaminoglicano e curto e diversificada N- e oligossacidos ligados em O. A faixa 1 em cada um dos imunoblots representa extractos cardíacos não tratados. A pista 2 contém extractos tratados com enzimas que digerem os glicosaminoglicanos. Amostras em pista 3 contém, em adição, as enzimas para a remoção de oligossacarídeos N- e O-ligados. A galectina-1 não é glicosilada, por conseguinte, não há mudança no tamanho da proteína. Adaptado de Lynch M, et al. 4 (C) na análise de LC-MS / MS, as amostras tratadas com PNGase-F na presen de H 2 O 18 obter uma melhor cobertura da sequência (%, no lado direito) em comparação com amostras não-desglicosilada. Dáreas verdes representam arca cobertura sequência por LC-MS / MS. As linhas ponteadas representam vermelhas glycosites, com números que indicam a sua posição de aminoácido. Detecção de glicosilação de decorina na posição Asn 211 (N, destacadas em negrito) é mostrada em detalhe a título de exemplo na Figura 4. Por favor clique aqui para ver uma versão maior desta figura.
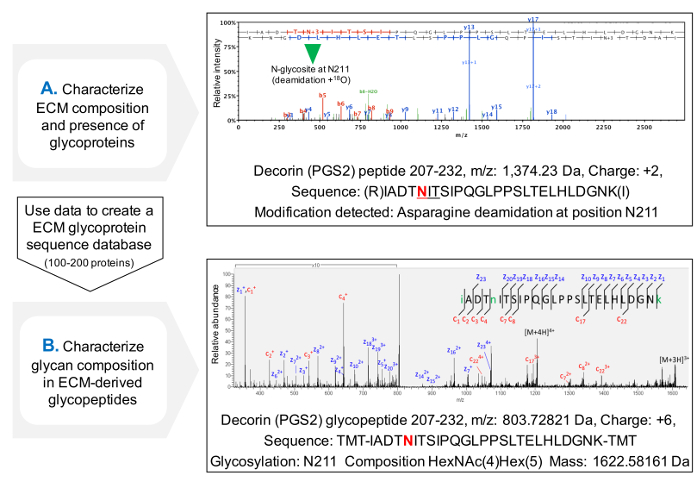
Figura 4. Análise por MS glicopéptido. (A) Utilizando uma abordagem proteómica espingarda em ECM extractos enriquecidos, glicopéptidos podem ser identificados pela presença de asparaginas desamidadas dentro sequons NXT / S e marcadas com 18 O. O exemplo mostra um espectro HCD MS / MS de um peptídeo contendo o de decorina anteriormente glicosilada Asn 211. Os dados obtidos podem ser utilizadospara criar um banco de dados personalizado de glicoproteínas ECM. (B) HCD-ETD fragmentação é utilizada para analisar o extracto enriquecido glicopéptido ECM. O espectro ETD MS / MS permite a caracterização da composição de glicano. Por favor clique aqui para ver uma versão maior desta figura.
| Soluções A. de Stock | |
| DTT (Ditiotreitol, C 4 H 10 O 2 S 2) | DTT 100 mM em ddH 2 O. 1 |
| EDTA (ácido etilenodiaminotetracético, C 10 H 16 N 2 O 8) | 250 mM de EDTA em ddH2O, pH 8,0. |
| GuHCl (cloridrato de guanidina, CH 6 ClN 3) | 8 M GuHCl emddH 2 O. |
| IAA (iodoacetamida, C 2 H 4 INO) | IAA 500 mM em ddH 2 O. 1,2 |
| Na acetato (acetato de sódio, C 2 H 3 NaO 2) | 1 M de acetato de Na em ddH2O, pH 5,8. |
| De NaCl (cloreto de sódio, NaCl) | NaCl 1 M em ddH 2 O. |
| Na fosfato dibásico (fosfato dissódico, Na 2 H 2 PO 4) | 1 M de Na fosfato dibásico em dd H2O, pH 6,8. |
| SDS (sulfato dodecilo de sódio, a NAC 12 H 25 SO 4) | 1% de SDS (35 mM) em ddH 2 O. 3 |
| TFA (ácido trifluoroacético, C 2 HF 3 O 2) | TFA a 10% (1,2 M) em ddH 2 O. |
| TEAB (bicarbonato de trietilamónio, C 7 H 17 NO 3) | 1M em TEAB em ddH 2 O, pH 8,5 |
| Tioureia (tioureia, CH 4 N 2 S) | 3 M tioureia em ddH 2 O. |
| Tris-HCl (Tris-cloridrato de (NH 11 C 4 O 3 [HCl]) | 100 mM de Tris-HCl em ddH2O, pH 7,5. |
| Ureia (ureia, CH 4 N 2 O) | 9 M de ureia em ddH 2 O. |
| Tampões B. Reacção | |
| C18 tampão de equilíbrio de limpeza | 1% de ACN, 0,1% de TFA em ddH2O |
| C18 limpa-se tampão de lavagem de coluna | 80% de ACN, 0,1% de TFA em H 2 O |
| C18 tampão de eluição de limpeza | 50% de acetonitrilo, 0,1% TFA em ddH2O |
| Tampão desglicosila�o (4x) | NaCl 600 mM e fosfato de sódio a 200 mM em ddH2O, pH 6,8. |
| Tampão de GuHCl 4 | 4 M de hidrocloreto de guanidina, 50 mM de acetato de Na e EDTA 25 mM em ddH2O, pH 5,8. Adicionar 1: 100 (v: v) de cocktail de inibidores de proteases antes da utilização. |
| NaCl 4 | NaCl 0,5 M, Tris-HCl a 10 e EDTA a 25 mM em ddH2O, pH 7,5. Adicionar 1: 100 (v: v) de cocktail de inibidores de proteases antes da utilização. |
| PBS (1x) | 1,7 mM de KH 2 PO 4, 5 mM de Na 2 HPO 4, NaCl 150 mM, pH 7,4. Adicionar EDTA 25 mM e 1: 100 (v: v) de cocktail de inibidores de proteases antes da utilização. |
| tampão de amostra (4x) | 100 mM de Tris, 2% de SDS, 40% glicerol, 0,02% de azul de bromofenol em ddH2O, pH 6,8. Adicionar 10% ß-mercaptoetanol antes do uso. |
| Tampão SDS a 4 | 0,1% de SDS e EDTA 25 mM em ddH 2 O. Adicionar 1: 100 (v: v) de mistura de inibidores de proteinase befuso de minério. |
| C. Enzimas | |
| Condroitinase ABC 5 | 0,5 U / mL em tampão de desglicosilação (1x) |
| queratanase 5 | 0,1 U / mL em tampão de desglicosilação (1x) |
| A heparinase II 5 | 0,1 U / mL em tampão de desglicosilação (1x) |
| α2-3,6,8,9-neuraminidase (sialidase) 5 | 0,025 U / mL em tampão de desglicosilação (1x) |
| β1,4-Galactosidase 5 | 0,015 U / mL em tampão de desglicosilação (1x) |
| p-N-acetilglucosaminidase 5 | 0,25 U / mL em tampão de desglicosilação (1x) |
| Endo-α-N-acetilgalactosaminidase (O-glicosidase) 5 | 0,013 U / mL em tampão de desglicosilação (1x) |
| Com PNGase-F (N-glicosidase-F) 6 | 50 U / ml em H 2 O 18 |
| tripsina | 0,01 mg / mL em tampão de TEAB |
| NOTAS Tabela. | |
| Manter uma solução estoque congelada a -20 ° C. | |
| 2 IAA deve ser mantido protegido da luz. | |
| 3 SDS prontamente cristaliza a <20 ° C. A fim de facilitar a solubilização de SDS a 1% (solução-mãe), aquecer o tampão sob água da torneira quente. | |
| 4 tampões de extracção pode ser armazenado à temperatura ambiente. Adicionar cocktail de largo espectro de inibidores de proteases, como indicado antes da utilização. | |
| 5 Estas enzimas devem ser adicionados durante o primeiro passo de desglicosilação. | |
| 6 com PNGase-F só deve ser adicionado durante o segundo passo de desglicosilação. | |
Tabela 1: Soluções Stock, buffers de reação e enzimas. Esta tabela indica a composição de cada tampão de solução de reserva e de reacção necessário para a extracção e processamento subsequente (incluindo digestões enzimáticas) de proteínas de ECM cardíacos antes da análise MS.
Discussão
Este protocolo proteômica foi optimizado ao longo dos últimos anos em nosso laboratório. Aqui, usamos tecido cardíaco, mas podem ser necessários apenas pequenos ajustes para a sua aplicação a outros tecidos. Por exemplo, o protocolo de extracção tem de ter a celularidade do tecido em consideração. tecido cardíaco é altamente celular em comparação com o tecido vascular. Quando se utiliza tecido vascular, a concentrao do SDS pode ser mais baixa (isto é, 0,08%) e o tempo de descelularização é mais curto (isto é, 4 h) 11, 12, 13. O uso de enzimas de desglicosilação é crucial para análise por LC-MS / MS de composição ECM. No entanto, os tempos de incubação ter de ser ajustada para diferentes tipos de tecidos. Por exemplo, heparinase II tempos de incubação prolongados necessários a 25 ° C quando se utiliza amostras, tais como a pele, os quais são ricos em proteínas da membrana basal (por exemplo, agrina, perlecano) (dados não mostrados). Análise glicopéptido directa pode ser realizada em meio condicionado a partir de células em cultura de 15. etapas de enriquecimento pode não ser necessária para a análise deste subproteome simplificado. Semelhante aos extractos de GuHCl, extractos de NaCl também são passíveis de análise glicoproteômica com pequenas modificações. Outros protocolos de extracção para o enriquecimento de proteínas de ECM pode ser adaptado para caracterizar glicopéptidos ECM 19, 20.
Glicosilação é a PTM mais complexa 5. A identificação indirecta de glicopéptidos é conseguida pela detecção de Asn desamidada com incorporado 18 O numa NXT / S sequon. Deamidated Asn em outras posições podem representar falsos positivos. Do mesmo modo, N-glicosilação deve ser considerado no contexto de ontologias proteína: proteínas intracelulares que contenham um / S sequon NXT não irá ser glicosilada mas pode dar origem a falsos positivos. Como searc atualh algoritmos não permitem o rastreio de PTMs em pré-determinadas sequências apenas (ou seja Asn na NXT / S), é necessária a filtragem manual dos dados. Identificação de presença / ausência de glicosilação nestas posições pode ser comparada entre as amostras e controle de doenças. Não há nenhuma enzima equivalente a PNGase F para S-desglicosilação (ou seja, a introdução de um deslocamento de massa de treonina ou serina). Portanto, a identificação de O-glicosilação está restringido à análise directa glicopéptido. análise directa glicopéptido é usado para obter informações sobre a composição de açúcares ligados a proteínas, mas não fornecem informação estrutural dos glicanos. Além disso, a composição de glicano é o resultado da síntese de glicano e processamento após a secreção.
O nosso método de extracção 3-passo para proteas ECM ( "Inglês Quickst") 6 permitiu a caracterização da ECM de uma variedade de tecidos cardiovasculares. Fraccionamento do tecidoem vários extractos é necessária para obter um proteoma ECM simplificado tal como discutido noutro local 6. proteínas intracelulares, de outro modo contribuem para uma gama dinâmica de abundâncias excessiva de proteínas, dentro dos extractos que iria dificultar a identificação de proteínas de ECM menos abundantes. Além disso, as proteínas intracelulares transportar O-glicosilaes 5 que iria complicar ECM enriquecimento glicopéptido e análise MS subsequente. Outros autores aplicado metodologias de extracção semelhantes a caracterizar por exemplo pulmão 21 e tecidos de cartilagem 10, no entanto eles não prosseguir a análise de glicosilação. Análise anterior de glicosilação focada na identificação de apenas glycosites, requerem a remoção do glicano do núcleo da proteína, e não pode avaliar a O-glicosilação 22, 23. matrizes de lectina e enriquecimento químico estão disponíveis para avaliação da typ glicanoes de amostras biológicas, com base na sua especificidade de ligação, mas estas técnicas não é possível atribuir tipos de glicanos de proteínas específicas 24 nem podem avaliar os locais de glicosilação.
Inicialmente, utilizou-se electroforese em gel antes de LC-MS / MS de proteínas de ECM. Embora a separação em gel é útil na obtenção de fracções de proteína simplificados propícios a análise por LC-MS / MS, os últimos instrumentos oferecem velocidades de varredura mais rápido. Assim, o passo de separação electroforética pode ser omitido. Isto proporciona uma vantagem adicional como grandes proteínas da MEC, os quais são retidos no topo do gel, são analisadas de forma mais eficiente. No entanto, a informação sobre o peso molecular das proteínas intactas é perdida. O passo de evaporação antes de PNGase F desglicosilação garante a remoção completa do normal H2O para minimizar os falsos negativos. Os resíduos de açúcar (isto é, massas de glicano variáveis) interferir com a separação por LC e comprometer a subsequente identificação do péptido por MS / MS. A pum-desglicosilação protocolo também é recomendado para amostras de proteínas totais de proteínas de ECM não focados na glicosilação.
Proteomics pode fornecer insights sem precedentes sobre a ECM. Além suporte estrutural, os glicanos ligados ao ECM são essenciais para a interacção hospedeiro-patogénio, comunicação célula-célula e a resposta imune 25, ou seja, a rejeição do enxerto após o transplante de órgãos. Glicoproteômica será uma ferramenta essencial na glycobiology.
Divulgações
Nenhum.
Agradecimentos
JBB é uma carreira Estabelecimento Fellow no Kings Hotel Center British Heart Foundation. MM é um membro sênior da Fundação Britânica do Coração (FS / 13/2/29892). O estudo foi apoiado por uma iniciativa de excelência (Centros de Competência para excelente Technologies - Comet) da Agência de Promoção de Investigação austríaco FFG: "Centro de Investigação de Excelência em Vascular Envelhecimento - Tyrol, VASCage" (número K-Project 843.536) eo Biomedical Research NIHR centro baseado no Serviço Nacional de Saúde Foundation Trust de Guy e St. Thomas' e faculdade Londres do rei, em parceria com o Hospital do rei College.
Materiais
| Name | Company | Catalog Number | Comments |
| A. Chemicals | |||
| Acetonitrile, MS-grade (ACN, C2H3N) | Thermo Scientific | 51101 | 5.2-5.8, 6.2, 7.11, Supp 2, 3, 4 |
| Cocktail of proteinase inhibitors | Sigma-Aldrich | P8340 | 1.3, 1.4.1, 1.5.1, 1.6.1 |
| Disodium phosphate (Na2H2PO4) | Sigma-Aldrich | S7907 | 3.1 |
| Dithiotreitol (DTT, C4H10O2S2) | Sigma-Aldrich | D0632 | 4.3 |
| Ethylenediaminetetraacetic acid (EDTA, C10H16N2O8) | Sigma-Aldrich | E9884 | 1.3, 1.4.1, 1.5.1, 1.6.1 |
| Ethanol (C2H6O) | VWR | 437433T | 2.2.1 |
| Guanidine hydrochloride (GuHCl, CH6ClN3) | Sigma-Aldrich | G3272 | 1.6.1 |
| Glycerol (C3H8O3) | Acros organics | 158920025 | Suppl 1.1 |
| H2O LC-MS Cromasolv | Sigma-Aldrich | 39253-1L-R | Throughout the protocol |
| H218O | Taiyo Nippon Sanso | FO3-0027 | 3.5 |
| Hydroxylamine (HA, H3NO) | Sigma-Aldrich | 467804 | 6.4 |
| Iodoacetamide (IAA, C2H4INO) | Sigma-Aldrich | A3221 | 4.4 |
| Phosphate-buffered Saline (PBS), 10X | Lonza | 51226 | 1.3 |
| Sodium acetate (C2H3NaO2) | Sigma-Aldrich | S7545 | 1.6.1 |
| Sodium chloride (NaCl) | Sigma-Aldrich | S9888 | 1.4.1, 3.2 |
| Sodium dodecyl sulfate (SDS, NaC12H25SO4) | Sigma-Aldrich | 466143 | 1.5.1 |
| Triethylammonium bicarbonate (TEAB, C7H17NO3) | Sigma-Aldrich | 11268 | 4.7, 6.1 |
| Trifluoroacetic acid (TFA, C2HF3O2) | Sigma-Aldrich | T62200 | 4.8, 5.2-5.8, 7.11, Supp 2, 3 |
| Thiourea (CH4N2S) | Sigma-Aldrich | T8656 | 4.2 |
| Tris-hydrochloride (Tris-HCl, NH11C4O3[HCl]) | Sigma-Aldrich | T3253 | 1.4.1, Suppl 1. |
| Urea (CH4N2O) | Sigma-Aldrich | U1250 | 4.2 |
| Name | Company | Catalog Number | Comments |
| B. Enzymes | |||
| α2-3,6,8,9-Neuraminidase (Sialidase) | EDM Millipore | 362280 (KP0012) | 3.1 |
| β1,4-Galactosidase | EDM Millipore | 362280 (KP0004) | 3.1 |
| β-N-Acetylglucosaminidase | EDM Millipore | 362280 (KP0013) | 3.1 |
| Chondroitinase ABC | Sigma-Aldrich | C3667 | 3.1 |
| Endo-α-N-acetylgalactosaminidase (O-glycosidase) | EDM Millipore | 362280 (KP0011) | 3.1 |
| Heparinase II | Sigma-Aldrich | H6512 | 3.1 |
| Keratanase | Sigma-Aldrich | G6920 | 3.1 |
| PNGase-F (N-Glycosidase F) | EDM Millipore | 362280 (KP0001) | 3.5 |
| Trypsin | Thermo Scientific | 90057 | 4.7 |
| Name | Company | Catalog Number | Comments |
| C. Reagent kits | |||
| 30 kDa MWCO spin filters | Amicon, Millipore | 10256744 | 5.9, Suppl 2 |
| Macro SpinColumn C-18, 96-Well Plate | Harvard Apparatus | 74-5657 | 5.1 |
| NuPAGE Novex BisTris Acrylamide Gels | Thermo-Scientific | NP0322PK2 | Suppl 1 |
| Pierce BCA Protein Assay Kit | Thermo Scientific | 23227 | 2.1.3 |
| ProteoExtract Glycopeptide Enrichment Kit | Merk Millipore | 72103 | 7 |
| Tandem mass tag 0 (TMT0) | Thermo Scientific | 900067 | 6.2, 6.3 |
| Name | Company | Catalog Number | Comments |
| D. Equipment and software | |||
| Acclaim PepMap100 C18 Trap, 5mm x 300µm, 5µm, 100Å | Thermo Scientific | 160454 | Suppl 3, 4 |
| Acclaim PepMap100 C18, 50cm x 75µm, 3µm, 100Å | Thermo Scientific | 164570 | Suppl 3 |
| Byonic Search Engine | Protein Metrics | Version 2.9.30 | Suppl 5 |
| Dionex UltiMate 3000 RSLCnano | Thermo Scientific | n/a | Suppl 3, 4 |
| EASY-Spray Ion Source | Thermo Scientific | ES081 | Suppl 4 |
| EASY-Spray PepMap RSLC C18, 50cm x 75µm, 2μm, 100Å | Thermo Scientific | ES803 | Suppl 4 |
| Mascot Search Engine | Matrix Science | Version 2.3.01 | Suppl 3 |
| Orbitrap Fusion Lumos Tribrid Mass Spectrometer | Thermo Scientific | IQLAAEGAAPFADBMBHQ | Suppl 4 |
| Proteome Discoverer Software | Thermo Scientific | Version 2.1.1.21 | Suppl 3, 5 |
| Picoview Nanospray Source | New Objective | 550 | Suppl 3 |
| Q Exactive HF Mass Spectrometer | Thermo Scientific | IQLAAEGAAPFALGMBFZ | Suppl 3 |
| Savant SpeedVac Concentrator | Thermo Scientific | SPD131DDA | 2.2.2, 3.4, 4.6, 5.7, 6.5, 7.11 |
| Scaffold Software | Proteome Software | Version 4.3.2 | Suppl 3 |
Referências
- Porter, K. E., Turner, N. A. Cardiac fibroblasts: at the heart of myocardial remodeling. Pharmacol. Ther. 123 (2), 255-278 (2009).
- Barallobre-Barreiro, J., et al. Proteomics analysis of cardiac extracellular matrix remodeling in a porcine model of ischemia/reperfusion injury. Circulation. 125 (6), 789-802 (2012).
- Barallobre-Barreiro, J., et al. Glycoproteomics reveals decorin peptides with anti-myostatin activity in human atrial fibrillation. Circulation. 134 (11), 817-832 (2016).
- Lynch, M., Barallobre-Barreiro, J., Jahangiri, M., Mayr, M. Vascular proteomics in metabolic and cardiovascular diseases. J. Intern. Med. 280 (4), 325-338 (2016).
- Varki, A., Lowe, J. B., Varki, A. Biological Roles of Glycans. Essentials of glycobiology. 2nd ed. , (2009).
- Barallobre-Barreiro, J., Lynch, M., Yin, X., Mayr, M. Systems biology - opportunities and challenges: The application of proteomics to study the cardiovascular extracellular matrix. Cardiovasc. Res. , (2016).
- Agnetti, G., Husberg, C., Van Eyk, J. E. Divide and conquer: the application of organelle proteomics to heart failure. Circ. Res. 108 (4), 512-526 (2011).
- Mason, R. M., Mayes, R. W. Extraction of cartilage protein-polysaccharides with inorganic salt solutions. Biochem. J. 131 (13), 535-540 (1973).
- Vogel, K. G., Peters, J. A. Isolation of proteoglycans from tendon. Methods. Mol. Biol. 171, 9-17 (2001).
- Wilson, R., et al. Comprehensive profiling of cartilage extracellular matrix formation and maturation using sequential extraction and label-free quantitative proteomics. Mol. Cell. Proteomics. 9 (6), 1296-1313 (2010).
- Barallobre-Barreiro, J., et al. Extracellular matrix remodeling in response to venous hypertension: proteomics of human varicose veins. Cardiovasc. Res. 110 (3), 419-430 (2016).
- Didangelos, A., Yin, X., Mandal, K., Baumert, M., Jahangiri, M., Mayr, M. Proteomics characterization of extracellular space components in the human aorta. Mol. Cell. Proteomics. 9 (9), 2048-2062 (2010).
- Didangelos, A., et al. Extracellular matrix composition and remodeling in human abdominal aortic aneurysms: a proteomics approach. Mol. Cell. Proteomics. 10 (8), (2011).
- Grandoch, M., et al. Loss of biglycan enhances thrombin generation in apolipoprotein E-deficient mice: Implications for inflammation and atherosclerosis. Arterioscler Thromb. Vasc. Biol. 36 (5), e41-e50 (2016).
- Yin, X., Bern, M., Xing, Q., Ho, J., Viner, R., Mayr, M. Glycoproteomic analysis of the secretome of human endothelial cells. Mol. Cell. Proteomics. 12 (4), 956-978 (2013).
- Parker, B. L., et al. Quantitative N-linked glycoproteomics of myocardial ischemia and reperfusion injury reveals early remodeling in the extracellular environment. Mol. Cell. Proteomics. 10 (8), (2011).
- Smith, P. K., et al. Measurement of protein using bicinchoninic acid. Anal. Biochem. 150 (1), 76-85 (1985).
- Pepinsky, R. B. Selective precipitation of proteins from guanidine hydrochloride-containing solutions with ethanol. Anal. Biochem. 195 (1), 177-181 (1991).
- de Castro-Brás, L. E., et al. Texas 3-step decellularization protocol: looking at the cardiac extracellular matrix. J. Proteomics. 86, 43-52 (2013).
- Naba, A., Clauser, K. R., Hynes, R. O. Enrichment of extracellular matrix proteins from tissues and digestion into peptides for mass spectrometry analysis. J Vis Exp. (101), e53057 (2015).
- Decaris, M. L., et al. Proteomic analysis of altered extracellular matrix turnover in bleomycin-induced pulmonary fibrosis. Mol. Cell. Proteomics. 13 (7), 1741-1752 (2014).
- Zhang, H., Li, X. J., Martin, D. B., Aebersold, R. Identification and quantification of N-linked glycoproteins using hydrazide chemistry, stable isotope labeling and mass spectrometry. Nat. Biotechnol. 21 (6), 660-666 (2003).
- Parker, B. L., et al. Site-specific glycan-peptide analysis for determination of N-glycoproteome heterogeneity. J. Proteome Res. 12 (12), 5791-5800 (2013).
- Li, Y., et al. Simultaneous analysis of glycosylated and sialylated prostate-specific antigen revealing differential distribution of glycosylated prostate-specific antigen isoforms in prostate cancer tissues. Anal. Chem. 83 (1), 240-245 (2011).
- Rienks, M., Papageorgiou, A. P., Frangogiannis, N. G., Heymans, S. Myocardial extracellular matrix: an ever-changing and diverse entity. Circ. Res. 114 (5), 872-888 (2014).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados