Method Article
Identificação de sequências de localização Plasmodesmal em proteínas em Planta
Neste Artigo
Resumo
Planta de conexões intercelulares, os dictiossomos (Pd), desempenham um papel central na planta de interações planta-vírus e fisiologia. Crítica para o transporte de Pd está classificando os sinais que dirigem proteínas à polícia. No entanto, nosso conhecimento sobre estas sequências é ainda em sua infância. Nós descrevemos uma estratégia para identificar sinais de localização de Pd em proteínas Pd-alvo.
Resumo
Dictiossomos (Pd) são ligações de celular para celular que funcionam como gateways através do qual moléculas pequenas e grandes são transportadas entre células vegetais. Considerando que o transporte de Pd de pequenas moléculas, como íons e água, presume-se ocorrer passivamente, célula a célula transporte de macromoléculas biológicas, tais proteínas, provavelmente ocorre através de um mecanismo ativo que envolve os sinais de direcionamentos específicos sobre o molécula transportada. A escassez de sinais de localização de dictiossomos identificados (Pd) (PLSs) restringiu severamente o entendimento de proteína-classificação vias envolvidas na planta transporte macromolecular de célula para célula e comunicação. De uma riqueza de planta endógenas e virais proteínas conhecidas para o tráfego por meio de Pd, PLSs apenas três foram relatados até à data, todos eles de proteínas vegetais endógenos. Assim, é importante desenvolver uma estratégia experimental sistemática e confiável para identificar uma sequência PLS funcional, que é necessário e suficiente para o direcionamento de Pd, diretamente no vivo células vegetais. Aqui, descrevemos uma tal estratégia usando como um paradigma da proteína de célula para célula de movimento (MP) do vírus do mosaico do tabaco (TMV). Estas experiências, que identificaram e caracterizam a primeira planta PLS viral, pode ser adaptadas para descoberta de PLS sequências em proteínas mais Pd-alvo.
Introdução
Dictiossomos (Pd) funcionam como condutos para transporte intercelular dos reguladores chaves do desenvolvimento da planta e morfogênese, variando de fatores de transcrição de mRNA e pequenas moléculas de RNA. Além disso, esta capacidade de transporte macromolecular da DP é utilizada pela maioria dos vírus de planta para sua propagação intercelular durante a infecção; para mover-se através de Pd, vírus de plantas evoluíram proteínas especializadas, denominadas proteínas de movimento (MPs), que visam especificamente a Pd1,2,3,4,5,6 , 7. vias moleculares de transporte Pd provavelmente estão intimamente interligadas com as sequências específicas que visam as proteínas transportadas para essas vias. Assim, a identificação destes sinais de localização do Pd (PLSs) pode ser diagnóstica da via de transporte correspondente do Pd. Isto é por analogia de Pd transporte8, por exemplo, a importação nuclear diferentes vias, que podem ser específicas para diferentes localização nuclear (NLS) de sinal sequências9,10. Conceitualmente, tanto NLSs e PLSs representam não-cleavable subcellular direcionamentos sequências que são necessárias e suficientes para o direcionamento. No entanto, ao contrário de NLSs11, as informações de sequência sobre PLSs são severamente limitadas. Especificamente, somente quatro sequências de proteínas envolvidas no direcionamento de Pd têm sido relatadas, com todos eles derivados de proteínas vegetais endógenos. O primeiro deles é representado por um domínio homeobox KN112 – um factor de transcrição que se move de camadas celulares interna a epiderme da planta folha13 – e sua KNOX homologs14. O segundo também é de um fator de transcrição, Dof, que contém um putativo PLS descrito como o tráfico intercelular (IT) motivo15. A terceira sequência é da proteína de membrana PDLP1 residente dictiossomos tipo 1, e é representado por um domínio transmembrana16. Finalmente, o quarto Pd segmentação sequência foi relatado recentemente para glicosilfostidilinosiol (GPI)-proteínas ancoradas e é representado por glicosilfostidilinosiol (GPI) modificação sinal17.
Curiosamente, até muito recentemente, nenhum PLSs têm sido relatados para MPs virais. Estudos anteriores indicaram a presença de sequências de PLS putativos em planta viral MPs18,19, mas não PLS verdadeiro, ou seja, uma sequência de aminoácidos mínima necessária e suficiente para o Pd direcionamento de uma carga independentes molécula ( ex., PCP) foi identificada em um MP viral. Ainda uma destas proteínas, MP do vírus do mosaico do tabaco (TMV), foi o primeiro para que a polícia de localização e transporte tem sido demonstrada20.
Para resolver esta lacuna, desenvolvemos uma estratégia experimental para identificar TMV MP PLS. Esta estratégia foi baseada em três conceitos. (i) definimos PLS como uma sequência de aminoácidos mínima necessária e suficiente para o direcionamento de proteína para Pd21. (ii) porque TMV MP visa primeiro Pd e translocates então através destes canais,22, que visa desatrelar essas duas atividades e identificando o PLS bona fide, que funciona apenas para o direcionamento de Pd e não para o transporte subsequente. (iii) foram analisados o PLS identificado por resíduos de aminoácidos importantes para a sua polícia visando a atividade, seja estrutural ou funcionalmente. Usando essa abordagem, nós delineado uma sequência de 50 amino ácido resíduos no amino-terminal do TMV MP que atua como PLS bona fide. Isto foi feito por produzir uma série de fragmentos de TMV MP que saturado de todo o comprimento da proteína, etiquetando seus carboxila-termini com PCP e transitoriamente expressá-los em tecidos vegetais. Localização de PD de cada um dos fragmentos testados foi determinada por coexpressing-los com uma proteína de marcador de Pd, PDCB1 (Pd callose proteína 1)23. O fragmento menor que ainda localizada, Pd, mas não atravessar Pd, considerou-se para representar o PLS. Finalmente, o PLS era alanina-digitalizadas para determinar os resíduos de aminoácidos chave necessários para sua estrutura e/ou função.
Considerando que aqui ilustramos esta abordagem descrevendo a identificação de TMV MP PLS, podem ser empregada para descobrir PLSs em qualquer outras proteínas Pd-alvo, se codificado por patógenos vegetais ou pelas plantas Isso ocorre porque nosso método não leva vantagem de quaisquer características únicas de MPs virais com relação à sua capacidade de alvo para Pd.
Protocolo
1. Material vegetal
- Escolha de espécies de plantas
- Use as espécies de plantas nativas à proteína de interesse, ou seja, um que codifica esta proteína para proteínas endógenas ou que representa o hospedeiro natural para o patógeno para proteínas virais. Além disso, as espécies de plantas selecionadas devem ser passíveis do método de escolha de transformação genética transitória.
Nota: Os estudos empregam rotineiramente Nicotiana benthamiana, que representa um bom anfitrião para TMV e é transformado de forma eficiente pela técnica de transformação genética mediada por Agrobacterium, i.e., agroinfiltration (ver passo 3). Plantas de s. benthamiana também são facilmente cultivadas e tem folhas grandes, que são facilmente inoculadas por Agrobacterium e facilmente analisadas por microscopia confocal. -
Crescimento de planta
- Plante sementes de s. benthamiana em solo molhado em alta densidade. Mantê-los em uma câmara de ambiente controlado em 20-25 ˚ c Membros abaixo dos 16 h de luz em ~ 75 µmol fótons m-2 s-1 e 8 h de escuridão. Depois que o diâmetro de euphyll atinge 0,5 cm, cuidadosamente transferir as mudas para vasos maiores e continuar o crescimento da mesma Câmara nas mesmas condições.
- Manter as plantas até que eles cresçam para estágio 4-8-folha dentro de 4 semanas, quando as folhas maiores são 4-6 cm de diâmetro; neste momento, eles podem ser usados para agroinfiltration (ver passo 3).
Nota: Importante, nunca use as plantas se começaram a flor porque, nesta fase do desenvolvimento, a arquitetura de folha varia frequentemente24,25, às vezes levando a resultados inconsistentes.
2. construção de vetor de expressão
-
Escolha do sistema de expressão
- Escolha a expressão sistema, i.e., vetores e vetor método de entrega, mais adequado para a expressão da proteína de interesse nas espécies de plantas estudadas.
Nota: Ocasionalmente, tais combinações específicas das espécies testadas proteína/planta podem exigir vetores específicos e vetor método de entrega para a expressão ideal e função da proteína de interesse. Para plantas dicotiledónias mais, no entanto, selecione binários plasmídeos como vetores de expressão e agroinfiltration como sistema de entrega.
- Escolha a expressão sistema, i.e., vetores e vetor método de entrega, mais adequado para a expressão da proteína de interesse nas espécies de plantas estudadas.
-
Marcação de proteínas fluorescentes
- Etiqueta fluorescente cada proteína expressa para análise de sua distribuição subcellular22, incluindo a localização do Pd.
Nota: Empregar autofluorescent proteínas, tais como o PCP e DsRed2, como etiquetas. Tag da proteína testada com PCP e coexpress-lo com um DsRed2 livre. Funções de PCP como marca e como uma molécula de carga não relacionado a vírus. DsRed2 funções para calibrar a eficiência global da expressão transiente e, porque é célula autónomos, para identificar a célula inicialmente transformada; Esta última função é importante, ao avaliar o movimento de célula para célula de proteínas testadas potencialmente não-celular-autónomos. Para coexpression com o marcador de Pd PDCB1, que também é celular autónomos, marca a proteína testada com PCP e PDCB1 com DsRed2. - Como regra geral, fundem-se a marca de autofluorescent para a carboxila-terminal da proteína expressa. No entanto, se as exposições de etiquetado proteína testado completos comprometida Pd direcionamento, transferi a tag para o amino-terminal da proteína.
- Clone as sequências de codificação das proteínas a ser testado em um vetor de expressão de instalações apropriadas.
Nota: Existem muitos vetores de expressão de diferentes plantas que permitem a marcação fluorescente. Usamos uma série de pSAT plasmídeos26 ou alguns dos seus derivados27,28, que são apropriados para a clonagem da sequência de interesse diretamente no frame com as tags sugeridas autofluorescent (consulte a etapa 2.2.1) em ambos os amino- e orientações de carboxila-terminal. - Transferi cada gaveta de expressão em um vetor binário de Agrobacterium.
Nota: Embora baseado no pSAT construções são apropriadas para entrega de peroxidação direta nos tecidos vegetais, agroinfiltration, que é o método de transformação aqui recomendado (ver passo 3), exige que a gaveta de expressão a ser localizada entre o T-DNA as fronteiras de um binário vetor29.
Nota: Todos os vetores de pSAT foram projetados para permitir essa transferência para o pPZP-RCS2 expressão multigene vetor binário26,30 clonando um passo a passo usando nucleases de corte raro ascos, PpoI-eu, eu-SceI, CeuI, PspI-PI e PI-TliI 26. pesquisadores que preferem utilizar a clonagem recombinatório, por exemplo, usando heteróloga salmão sites31, podem-se empregar pSAT vector variantes26,27. - Para coexpression, transferir as duas fitas de expressão, por exemplo, proteína-PCP testado e DsRed2 ou proteína-PCP testado e PDCB1-DsRed2 (ver passo 2.1), para o mesmo vetor binário de pPZP-RCS2.
Nota: Alternativamente, culturas de Agrobacterium abrigar construções binárias individuais podem ser misturadas juntos para infiltração de s. benthamiana (ver passo 3.1).
- Etiqueta fluorescente cada proteína expressa para análise de sua distribuição subcellular22, incluindo a localização do Pd.
3. Agroinfiltration
-
Adicionar 1 µ g de um plasmídeo baseados em pPZP-RCS2 de cultura de passo 2.2.5 para 100 µ l de células competentes da estirpe de Agrobacterium tumefaciens EHA105 ou GV3101 preparado como descrito32, incubar no gelo por 30 min, congelam em nitrogênio líquido por 1 min, e incube a 28 ° C por 15 min.
- Em seguida, adicionar 1 mL de meio LB (triptona 10 g/L, extrato de levedura 5 g/L e 10 g/L de NaCl) para a mistura de células competentes e incubar a 28 ° C, durante 2 h. Spin para baixo as células a 3.000 × g por 1 min, re-suspendê-las em 0,2 mL de LB e placa-los na LB ágar suplementado com antibióticos apropriados (por exemplo, espectinomicina 100 mg/L e rifampicin de 50 mg/L para pPZP RSC2-com base em vetores26,30). Cresce a 28 ° C, durante 48 h até colônias individuais são visíveis.
- Escolher e várias colónias individuais em uma placa de ágar LB fresca da placa (ver passo 3.1), crescer a 28 ° C, durante 48 h e tomar uma pequena quantidade de bactérias para análise por PCR de colônia33 para confirmar a presença das construções de binárias específicas.
-
Cresce cada colônia de Agrobacterium com o construto de interesse durante a noite a 28 ° C em 5 mL de LB suplementado com antibióticos apropriados (ver passo 3.1.1). Centrifugar as células a 3.000 × g e re-suspendê-las a OD600= 0,5 no buffer agroinfiltration (10 mM MgCl2, 10mm MES (pH 5,6), 150 µM acetosyringone). Incube durante 2 h à temperatura ambiente.
- Se usar uma mistura de plasmídeos binários em vez de um plasmídeo de expressão multigene único para coexpression de proteína, misture as culturas de célula correspondente na proporção de 1:1 v/v antes de infiltração. Então Incube as células a 28 ° C, durante 2 h.
-
Carrega o inóculo em uma seringa de plástico de 1 mL e pressione que o bico da seringa (sem agulha) contra a epiderme inferior (abaxial) da amadurecer N. benthamiana folhas. Segure a folha com um dedo protegido por luva a face adaxial34,35.
- Introduza o Agrobacterium médio de infiltração por injeção lenta. Para a significância estatística, se infiltre em diversas folhas em cada planta.
Nota: Observe que as folhas mais antigas não são utilizadas, como eles não produzem níveis de expressão consistente. - Vacinar todas as folhas em situ. Manter a planta infiltrada até observação conforme descrito no passo 1.2.
- Introduza o Agrobacterium médio de infiltração por injeção lenta. Para a significância estatística, se infiltre em diversas folhas em cada planta.
4. Confocal da microscopia
- Use uma lâmina para cortar cada folha em fragmentos entre as veias 24-48 h após a infiltração, (dependendo a eficiência da expressão transiente da proteína específica testada); o tamanho de cada fatia de tecido deve ser tal que a fatia é completamente coberta por vidro tampa (22 x 40 mm) usado para a microscopia.
- Lugar as fatias de folha no objeto deslize com a superfície abaxial da folha voltada para cima, coloque uma gota de água sobre a fatia de folha e cubra com o tampa de vidro. Imobilize o tampa de vidro com pequenos pedaços de fita em cada lado.
-
Observar os tecidos agroinfiltrated sob um microscópio confocal de varredura de laser e gravar as imagens resultantes.
- Primeiro, use uma lente de objetiva X 10 para identificar células com o sinal e use 63 x objectiva para observações mais detalhadas.
- Comprimento de onda do uso do 458 nm de um laser de íon argônio para excitar o PCP e 543 nm para excitar o DsRed2. Manter todas as configurações para aquisição de imagem, ou seja, a intensidade do laser e configurações de photomultiplier tubo (PMT), entre as experiências. Em média, examine células de 100-120 para cada condição experimental.
5. identificação dos PLS
- Diagnosticar a localização da proteína testada usando microscópio confocal (secção 4). Verifique se a proteína testada tem o padrão característico de punctate periférica25,36,37,38,39,40,41 e colocalizes com o marcador Pd PDCB123. Isto indica que a proteína testada provavelmente contém sequências PLS.
- Após confirmação da atividade da proteína testada, PLS progressivamente subdividir o quadro de leitura aberta (ORF) (número de adesão genbank BAF93925.1) em fragmentos menores, autofluorescently, (por exemplo, PCP,) marca cada um deles e analisar suas Localização subcellular conforme descrito na etapa 4. Inicialmente, recomenda-se o tamanho de 100 resíduos de aminoácidos para cada fragmento subdividido.
- Subdividir mais truncados fragmentos que têm a atividade PLS e verificar sua localização subcellular conforme descrito na etapa 4, até o menor fragmento que localiza ainda à polícia, ou seja, é suficiente para transportar a carga de marca autofluorescent para a polícia, é identificado. Este fragmento, presume-se para representar a sequência PLS.
- Excluir o putativo PLS da proteína testada completo, ou seja, fundir a parte restante da proteína teste sem putativo PLS a tag autofluorescent e determinar a Localização subcellular da proteína mutante resultante, conforme descrito na etapa 4. se esta proteína mutante que carece o putativo PLS falhar localizar à polícia, isso indicará que o identificado PLS não é apenas suficiente (ver passo 5.3), mas também é necessário para o transporte de Pd da proteína testado.
- Agroinfiltrate as folhas com uma mistura de culturas de Agrobacterium, abrigando a expressão constroem para o PCP-tag PLS e DsRed2 livre (ver passo 3.3). Observe o tecido infiltrado usando microscópio confocal com lente objetiva de 10x usando as mesmas configurações como no passo 4.3.
- Marca as células que contêm sinais tanto PCP e DsRed2 como células inicialmente infiltrados e marcar as células adjacentes que continha apenas o sinal de PCP como aqueles que mostra o movimento.
Nota: Este passo examina o PLS identificado por sua potencial capacidade de movimento de célula a célula; Isto é porque muitas proteínas que destino Pd (incluindo a maioria planta virais MPs) também se move Pd para as células vizinhas. A concentração de Agrobacterium usado para infiltração depende da eficiência de expressão de diferentes estruturas, respectivamente. Para agroinfiltration, certifique-se de usar a diluição de cultura de células que garante que (i) a transformação de células únicas, deixando as células adjacentes unlit, (ii) alcança eficiência semelhante de expressão. Por exemplo, nossos experimentos utilizam OD600 de 0,01 e 0,005 por expressão de PCP-tag PLS e livre DsRed2, respectivamente.
6. identificação da chave PLS resíduos usando alanina digitalização42
- Usar padrão protocolos PCR para amplificar o PLS sequência empregando um par de primers destinados a conter a mutação desejada, isto é, substituição do resíduo de aminoácido alvo com a alanina, em torno da região central de cada primer, conforme descrito de código 21. realizar o projeto da primeira demão e uso o kit mutagenesis local-dirigido para o mutagenesis local-dirigido de acordo com as instruções do fabricante.
- Purificar os produtos resultantes do PCR usando qualquer padrão kit de purificação de DNA e digeri-los a 37 ° C com DpnI para eliminar o parental (ou seja, não-mutante) misturado e hemimethylated o DNA. Isto esfrie por 5 min à temperatura ambiente (não gelo). Em seguida, transformar a mistura diretamente em células competentes de Escherichia coli 43e identificar colônias com as construções de mutante desejadas, conforme descrito no passo 3.2.
- Introduzir o mutante constrói o vetor binário, conforme descrito na etapa 2.2.4, execute agroinfiltration, conforme descrito na etapa 3 e avaliar Pd direcionamento conforme descrito nas etapas 4 e 5.1.
Resultados
Dados do representante, que fielmente ilustram os resultados esperados dos protocolos descritos e identificam o TMV MP PLS, adaptam-se de Yuan et al . 21. figura 1A primeiro resume grandes construções expressando o MP TMV completos (1-268), TMV MP PLS (compreendendo os primeiros 50 resíduos de aminoácidos da proteína, 1-50), e sua digitalização V4A derivados de alanina fundido ao CFP (gerado como descrito em etapas 2.2, 5.2 e 6) Considerando que a figura 1B resume e quantifica a Localização subcellular destas proteínas marcados. A Figura 1 ilustra os periférico punctate Pd localização padrões característicos observados para TMV MP-PCP e para TMV MP PLS-CFP, bem como para o marcador de Pd coexpressed, PDCB1-DsRed2 (veja etapa 5.1). Figura 2A ilustra Pd direcionamento de TMV MP-CFP e de distribuição TMV MP PLS-PCP e nucleocytoplasmic do coexpressed DeRed2 livre (ver passo 5.1). Finalmente, a Figura 2B ilustra a perda de Pd definião TMV MP e TMV MP PLS com uma substituição de alanina único V4A, indicando que este resíduo é importante para o PLS estrutura ou função; no mesmo células o marcador de Pd coexpressed PDCB1 localiza ainda aos Pd (consulte a etapa 6).
Estes dados mostram que esse procedimento conceitualmente e tecnicamente simples de produção sistemática de fragmentos de proteína, sua fusão para uma proteína de marcador-carga de autofluorescent e testes para a capacidade de localizar a polícia pode identificar uma sequência da proteína mínima necessária e suficiente para a polícia como alvo, ou seja, PLS. Para a correta interpretação destes dados de localização, é fundamental para realizar experiências colocalization com uma proteína conhecida de Pd, por exemplo, PDLP ou PDCB membros da família16,23 ou um da planta virais MPs esse tipo de Pd44 . Obviamente, o colocalization não tem que ser completa, nem todas as proteínas P-localização podem ser direcionadas para o Pd mesmo, mas um grau estatisticamente significativo de colocalization com pelo menos uma das proteínas de marcador de Pd é necessário para definir a atividade PLS.

Figura 1: identificação de TMV MP pls (A) Resumo das principais construções de PCP-fusão usado para identificar caixas de sequências, verde TMV MP pls TMV MP; PCP, caixas azuis; P, promotor; T, terminator. Os números à esquerda indicam os resíduos de aminoácidos incluídos em cada fragmento de TMV MP. (B) Resumo de Localização subcellular das proteínas de fusão mostrado em (A). Localização de PD ou nucleocytoplasmic localização foi determinada baseia a localização dos marcadores subcellular correspondentes, PDCB1-DsRed2 e DsRed2 livre, respectivamente. A percentagem de células exibindo cada padrão de localização é mostrada com base na pontuação de 100 células expressando por construir em três experimentos independentes. Dados são média ± erros-padrão (SE). (C) Pd direcionamento de TMV MP-PCP, TMV MP PLS-PCP e o marcador de Pd PDCB1-DsRed2. Sinal de PCP é em azul; Sinal de DsRed2 é em roxo; plastídeo autofluorescência foi filtrada. Imagens são seções confocal única. Barras de escala = 10 µm. DIC, Micrografia de contraste de interferência diferencial. Esta figura é uma adaptação de Yuan et al . 21. clique aqui para ver uma versão maior desta figura.
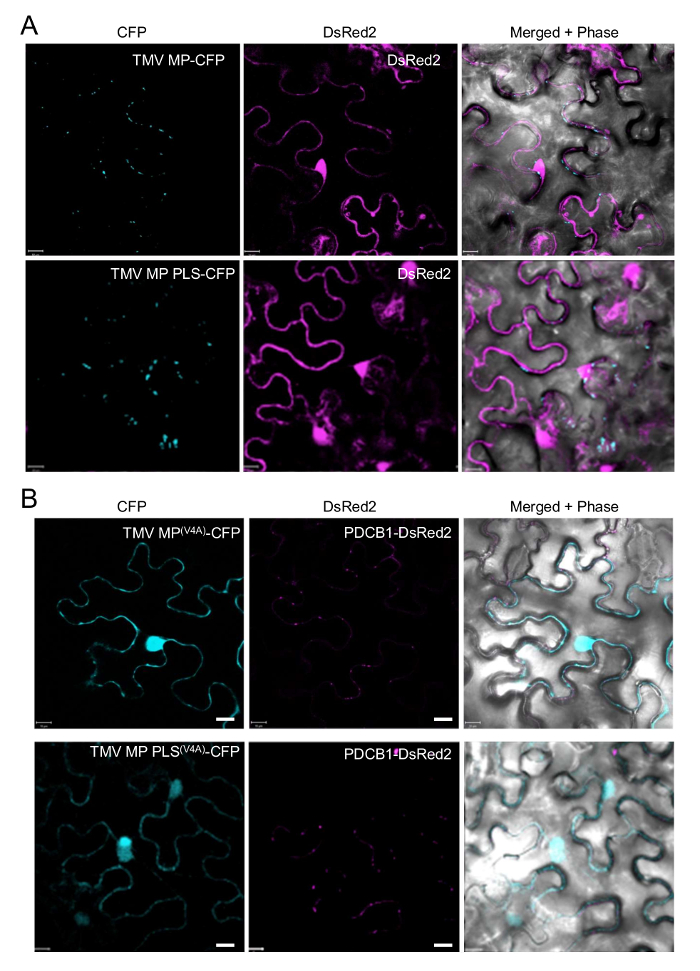
Figura 2: Pd direcionamento de TMV MP, TMV MP PLS e seus mutantes alanina-digitalização. (A) localização Subcellular de TMV MP-PCP, TMV MP PLS-PCP e o marcador de nucleocytoplasmic DsRed2. (B) perda de Pd direcionando a capacidade da alanina-digitalização V4A mutantes de TMV MP-PCP e TMV MP PLS-PCP. PD foram visualizadas por coexpression do marcador DP PDCB1-DsRed2. Sinal de PCP é em azul; Sinal de DsRed2 é em roxo; plastídeo autofluorescência foi filtrada. Imagens são seções confocal única. Barras de escala = 10 µm. DIC, Micrografia de contraste de interferência diferencial. Esta figura é uma adaptação de Yuan et al . 21. clique aqui para ver uma versão maior desta figura.
Discussão
Este protocolo tem quatro componentes principais: o conceito de identificar uma sequência que é necessário e suficiente para o direcionamento de Pd, divisão sistemática da proteína de interesse em fragmentos que são progressivamente reduzidos em comprimento, fundindo o testado fragmentos de uma proteína autofluorescent que serve como marca e como carga macromolecular e ensaio funcional para Pd direcionamento na vida da planta tecidos seguindo a expressão transiente das proteínas de fusão testado. Observe que a expressão transiente mediada por Agrobacterium gera dados dentro de um período relativamente curto de tempo, ou seja, vários dias, em comparação com os meses necessários para a produção de plantas transgênicas, frequentemente usado em semelhantes estudos15.
Coexpression da proteína de interesse com marcadores de Localização subcellular, por exemplo, PDCB1 ou DsRed2 livre, geralmente é realizado de um vetor de expressão multigene. No entanto, se a transferência de várias fitas de expressão em um único vetor é tecnicamente problemática, coexpression também pode ser realizada transferência de cada gaveta para uma construção binária separada e combinando volumes equivalentes de culturas líquidas do cepas de Agrobacterium resultantes para agroinfiltration. Porque este protocolo utiliza a expressão transiente, o vetor binário não precisa levar marcadores de seleção para transformação estável, embora sua presença não é prejudicial para a eficiência da expressão.
Este protocolo é otimizado para a expressão da proteína e direcionamento em s. benthamiana; no entanto, ele pode ser usado em outras espécies de plantas, incluindo Arabidopsis45,46, passível de transformação genética por Agrobacterium. Para outras espécies de plantas, ou eventualmente em caso contrário, este protocolo pode ser adaptado para utilizar peroxidação entrega41 para expressão transiente diretamente do pSAT-com base em vetores, evitando a necessidade de transferir as fitas de expressão para vetores binários.
Um ponto crítico neste protocolo é a condição fisiológica e desenvolvimento consistente do material vegetal. Especificamente, todas as plantas devem ser saudáveis ao longo de todo o experimento, e, para agroinfiltration, as plantas N. benthamiana devem ser menor que 4 semanas velho e na fase 4-8-folha com a folha maior, sendo 4-6 cm de diâmetro. Se as plantas são muito jovens, os efeitos da agroinfiltration será muito severos, interferindo na expressão e localização das proteínas marcadas. Por outro lado, as plantas mais velhas muitas vezes apresentam arquitetura variável de Pd24,25, afetando também a consistência dos padrões Pd-direcionamento.
Ainda assim, devido a diferentes condições fisiológicas da planta como um todo e de suas células transformadas em particular, a eficiência do Pd direcionamento em cada experimento um pouco pode variar. Assim, é importante analisar um número relativamente grande de células (> 100) por experiência e realizar pelo menos três réplicas biológicas de cada experimento. Geralmente, consideramos um fragmento testado para conter Pd segmentação de atividade, se exibe localização de Pd pelo menos 70% das células transformadas. Diferenças em Pd visando a eficiência de cada fragmento, por exemplo, % de células transformadas, mostrando o padrão de acumulação punctate Pd-específicos, devem ser analisados pelo teste t de Student e p-valores < 0,05-0,01, correspondente a probabilidade estatística de mais de 95-99%.
Outra questão importante é a posição da etiqueta fluorescente/carga na molécula de fusão. Algumas proteínas Pd-alvo também associam com o retículo endoplasmático (ER), e esta associação pode exigir a sequência amino-terminal desobstruída. Nestes casos, fusões de amino-terminal, na qual o carboxila-terminal do fragmento da proteína de interesse é fundido do amino-terminal da marca fluorescente, por exemplo, PCP ou DsRed2, deve ser usado. Este posicionamento padrão da marca pode ser alterado nos seguintes casos. (i) se as fusões de amino-terminal não são utilizáveis – ou seja, eles não apresentam padrões de Localização subcellular significativo, exprimem-se mal ou não geram sinal fluorescente suficiente para detecção confiável – e se a proteína de interesse não contêm sequências de amino-terminal sinal ou fusões carboxila-terminal no qual o amino-terminal do fragmento testado é fundido do carboxila-terminal da marca, devem ser tentadas. (ii) as fusões de carboxila-terminal também devem ser usadas se a proteína de interesse contém sequências de sinal em sua extremidade carboxila-terminal. (iii) se a proteína de interesse tem ambos os sinais de amino e carboxila-terminal, como os encontrados em proteínas GPI-ancorada, ou se ele contém um peptídeo sinal de amino-terminal, no entanto, sua fusão de amino-terminal não é utilizável, a marca deve ser colocada internamente ( ou a jusante do sinal do amino-terminal ou montante do sinal carboxila-terminal). Alguns procedimentos úteis para marcação fluorescente interna das proteínas são descritos em nossos anteriores Publicações47,48.
Neste momento, não há nenhuma sequência consenso conhecido para PLS. Como tal (na etapa 5.2) recomendamos, inicialmente, subdividindo a proteína em contíguo ~fragmentos de resíduos de comprimento de 100-200. Estes então podem ser feitas progressivamente mais curtos com base na atividade Pd-direcionamento observada ou a falta dela. Um deve estar atento de outros potenciais visando sinais presentes na proteína (por exemplo, NLS, peptídeos de sinal, etc.). No entanto, como a potencial sobreposição de tais sequências com sequências PLS é desconhecida, nestas regiões de proteína devem ser incluídas nesta análise mutacional.
Até agora, este protocolo foi usado para identificar um único PLS em uma molécula de proteína21. No entanto, potencialmente, uma proteína pode conter mais de uma sequência de sinal; na verdade vários NLSs têm sido descritas em muitas proteínas nucleares, incluindo a de fatores de transcrição GATA49. Assim, ao analisar diferentes fragmentos da proteína testado para a polícia como alvo, é possível que mais de um tal fragmento será identificado, indicando a presença de múltiplos PLSs que podem agir independentemente ou em sinergia.
A principal limitação do presente protocolo é que, transitoriamente a seguir agroinfiltration, visualização de Localização subcellular das proteínas expressas de interesse é melhor alcançado em células epidérmicas e na fase de crescimento relativamente estreita. Quando necessário, esta limitação pode ser superada através da produção de plantas transgênicas que estàvel expressam cada uma das proteínas testadas na maioria de seus tecidos e tipos de células. Além disso, este protocolo não seria útil para estudos de MPs virais ou outras proteínas Pd-alvo que funcionam apenas em espécies de plantas que não são passíveis de transformação genética transitória.
Enquanto desenvolvemos esta técnica para identificação de um PLS do TMV MP, pode ser usada para a descoberta da PLSs em qualquer outra planta MPs virais ou em proteínas endógenas de planta que localizar a Pd, transitoriamente ou permanentemente. Além disso, se combinado com adequada Localização subcellular marcadores47, este protocolo é apropriado para a identificação de muitos outros sinais de direcionamentos dentro de proteínas de interesse em plantas.
Divulgações
Não há conflitos de interesse declarados.
Agradecimentos
A falta de espaço, nós citados principalmente artigos de revisão, e pedimos desculpas aos nossos colegas, cuja obra original não foi citada. O trabalho no laboratório V.C. é suportado por concessões do NIH, NSF, USDA/NIFA, BARD e BSF para V.C., e o laboratório S.G.L. é suportado pelo NIH e fundos do departamentos de Fitopatologia e biologia vegetal-micróbio para S.G.L.
Materiais
| Name | Company | Catalog Number | Comments |
| Confocal laser scanning microscope (CLSM) | Zeiss | LSM5 | Any CLSM with similar capabilities is appropriate |
| Zen software for confocal microscope imaging | Zeiss | 2009 version | The software should be compatible with the CLSM used |
| Quickchange II site-directed mutagenesis kit | Agilent | 200523 | |
| Acetosyringone | Sigma-Aldrich | D134406 | |
| MES | Sigma-Aldrich | 69892 | |
| Syringes without needles | BD | 309659 | |
| MgCl2 | FisherScientific | M33-500 | |
| Spectinomycin | Sigma-Aldrich | S4014 | |
| Rifampicin | Sigma-Aldrich | R3501 | |
| Ampicillin | Sigma-Aldrich | A0166 |
Referências
- Lee, J. Y. Plasmodesmata: a signaling hub at the cellular boundary. Curr. Opin. Plant Biol. 27, 133-140 (2015).
- Kumar, D., Kumar, R., Hyun, T. K., Kim, J. Cell-to-cell movement of viruses via plasmodesmata. J. Plant Res. 128, 37-47 (2015).
- Kitagawa, M., Paultre, D., Rademaker, H. Intercellular communication via plasmodesmata. New Phytol. 205, 970-972 (2015).
- Jackson, D. Plasmodesmata spread their influence. F1000Prime Rep. 7, 25(2015).
- Brunkard, J. O., Runkel, A. M., Zambryski, P. C. The cytosol must flow: intercellular transport through plasmodesmata. Curr. Opin. Cell Biol. 35, 13-20 (2015).
- Yadav, S. R., Yan, D., Sevilem, I., Helariutta, Y. Plasmodesmata-mediated intercellular signaling during plant growth and development. Front. Plant Sci. 5, 44(2014).
- Sager, R., Lee, J. Y. Plasmodesmata in integrated cell signaling: insights from development and environmental signals and stresses. J. Exp. Bot. 65, 6337-6358 (2014).
- Jans, D. A., Xiao, C. Y., Lam, M. H. Nuclear targeting signal recognition: a key control point in nuclear transport? BioEssays. 22, 532-544 (2000).
- Miyamoto, Y., et al. Different modes of nuclear localization signal (NLS) recognition by three distinct classes of NLS receptors. J. Biol. Chem. 272, 26375-26381 (1997).
- Nair, R., Carter, P., Rost, B. NLSdb: database of nuclear localization signals. Nucleic Acids Res. 31, 397-399 (2003).
- Lee, J. Y., Yoo, B. C., Lucas, W. J. Parallels between nuclear-pore and plasmodesmal trafficking of information molecules. Planta. 210, 177-187 (2000).
- Kim, J. Y., Rim, Y., Wang, J., Jackson, D. A novel cell-to-cell trafficking assay indicates that the KNOX homeodomain is necessary and sufficient for intercellular protein and mRNA trafficking. Genes Dev. 19, 788-793 (2005).
- Lucas, W. J., et al. Selective trafficking of KNOTTED1 homeodomain protein and its mRNA through plasmodesmata. Science. 270, 1980-1983 (1995).
- Chen, H., Jackson, D., Kim, J. Y. Identification of evolutionarily conserved amino acid residues in homeodomain of KNOX proteins for intercellular trafficking. Plant Signal. Behav. 9, e28355(2014).
- Chen, H., Ahmad, M., Rim, Y., Lucas, W. J., Kim, J. Y. Evolutionary and molecular analysis of Dof transcription factors identified a conserved motif for intercellular protein trafficking. New Phytol. 198, 1250-1260 (2013).
- Thomas, C. L., Bayer, E. M., Ritzenthaler, C., Fernandez-Calvino, L., Maule, A. J. Specific targeting of a plasmodesmal protein affecting cell-to-cell communication. PLOS Biol. 6, e7(2008).
- Zavaliev, R., Dong, X., Epel, B. L. Glycosylphosphatidylinositol (GPI) modification serves as a primary plasmodesmal targeting signal. Plant Physiol. 172, 1061-1073 (2016).
- Sasaki, N., Park, J. W., Maule, A. J., Nelson, R. S. The cysteine-histidine-rich region of the movement protein of Cucumber mosaic virus contributes to plasmodesmal targeting, zinc binding and pathogenesis. Virology. 349, 396-408 (2006).
- Kaido, M., Funatsu, N., Tsuno, Y., Mise, K., Okuno, T. Viral cell-to-cell movement requires formation of cortical punctate structures containing Red clover necrotic mosaic virus movement protein. Virology. 413, 205-215 (2011).
- Creager, A. N. H., Scholthof, K. B. G., Citovsky, V., Scholthof, H. B. Tobacco mosaic virus: pioneering research for a century. Plant Cell. 11, 301-308 (1999).
- Yuan, C., Lazarowitz, S. G., Citovsky, V. Identification of a functional plasmodesmal localization signal in a plant viral cell-to-cell movement protein. mBio. 7, e02052-e02015 (2016).
- Ueki, S., Citovsky, V. To gate, or not to gate: regulatory mechanisms for intercellular protein transport and virus movement in plants. Mol. Plant. 4, 782-793 (2011).
- Simpson, C., Thomas, C. L., Findlay, K., Bayer, E., Maule, A. J. An Arabidopsis GPI-anchor plasmodesmal neck protein with callose binding activity and potential to regulate cell-to-cell trafficking. Plant Cell. 21, 581-594 (2009).
- Maule, A. J. Plasmodesmata: structure, function and biogenesis. Curr. Opin. Plant Biol. 11, 680-686 (2008).
- Roberts, I. M., et al. Dynamic changes in the frequency and architecture of plasmodesmata during the sink-source transition in tobacco leaves. Protoplasma. 218, 31-44 (2001).
- Tzfira, T., et al. pSAT vectors: a modular series of plasmids for fluorescent protein tagging and expression of multiple genes in plants. Plant Mol. Biol. 57, 503-516 (2005).
- Chakrabarty, R., et al. pSITE vectors for stable integration or transient expression of autofluorescent protein fusions in plants: probing Nicotiana benthamiana-virus interactions. Mol. Plant-Microbe Interact. 20, 740-750 (2007).
- Chung, S. M., Frankman, E. L., Tzfira, T. A versatile vector system for multiple gene expression in plants. Trends Plant Sci. 10, 357-361 (2005).
- Lee, L. Y., Gelvin, S. B. T-DNA binary vectors and systems. Plant Physiol. 146, 325-332 (2008).
- Goderis, I. J., et al. A set of modular plant transformation vectors allowing flexible insertion of up to six expression units. Plant Mol. Biol. 50, 17-27 (2002).
- Walhout, A. J., et al. GATEWAY recombinational cloning: application to the cloning of large numbers of open reading frames or ORFeomes. Methods Enzymol. 328, 575-592 (2000).
- Tzfira, T., et al. Transgenic Populus: a step-by-step protocol for its Agrobacterium-mediated transformation. Plant Mol. Biol. Rep. 15 (3), 219-235 (1997).
- Woodman, M. E., Savage, C. R., Arnold, W. K., Stevenson, B. Direct PCR of intact bacteria (colony PCR). Curr. Protoc. Microbiol. 42 (3D), 1-7 (2016).
- Kapila, J., De Rycke, R., Van Montagu, M., Angenon, G. An Agrobacterium-mediated transient gene expression system for intact leaves. Plant Sci. , 101-108 (1997).
- Wroblewski, T., Tomczak, A., Michelmore, R. Optimization of Agrobacterium-mediated transient assays of gene expression in lettuce, tomato and Arabidopsis. Plant Biotechnol. J. 3, 259-273 (2005).
- Boyko, V., Ferralli, J., Ashby, J., Schellenbaum, P., Heinlein, M. Function of microtubules in intercellular transport of plant virus RNA. Nat. Cell Biol. 2, 826-832 (2000).
- Heinlein, M., Epel, B. L., Padgett, H. S., Beachy, R. N. Interaction of tobamovirus movement proteins with the plant cytoskeleton. Science. 270, 1983-1985 (1995).
- Oparka, K. J., Prior, D. A. M., Santa-Cruz, S., Padgett, H. S., Beachy, R. N. Gating of epidermal plasmodesmata is restricted to the leading edge of expanding infection sites of tobacco mosaic virus (TMV). Plant J. 12, 781-789 (1997).
- Crawford, K. M., Zambryski, P. C. Non-targeted and targeted protein movement through plasmodesmata in leaves in different developmental and physiological states. Plant Physiol. 125, 1802-1812 (2001).
- Kotlizky, G., et al. A dysfunctional movement protein of Tobacco mosaic virus interferes with targeting of wild-type movement protein to microtubules. Mol. Plant-Microbe Interact. 14, 895-904 (2001).
- Ueki, S., Lacroix, B., Krichevsky, A., Lazarowitz, S. G., Citovsky, V. Functional transient genetic transformation of Arabidopsis leaves by biolistic bombardment. Nat. Protoc. 4, 71-77 (2009).
- Giesman-Cookmeyer, D., Lommel, S. A. Alanine scanning mutagenesis of a plant virus movement protein identifies three functional domains. Plant Cell. 5, 973-982 (1993).
- Ausubel, F. M., et al. Current Protocols in Molecular Biology. , Wiley-Interscience. New York. (1987).
- Waigmann, E., Ueki, S., Trutnyeva, K., Citovsky, V. The ins and outs of non-destructive cell-to-cell and systemic movement of plant viruses. Crit. Rev. Plant Sci. 23, 195-250 (2004).
- Lee, M. W., Yang, Y. Transient expression assay by agroinfiltration of leaves. Methods Mol. Biol. 323, 225-229 (2006).
- Burch-Smith, T. M., Schiff, M., Liu, Y., Dinesh-Kumar, S. P. Efficient virus-induced gene silencing in Arabidopsis. Plant Physiol. 142, 21-27 (2006).
- Tian, G. W., et al. High-throughput fluorescent tagging of full-length Arabidopsis gene products in planta. Plant Physiol. 135, 25-38 (2004).
- Tian, G. W., Chen, M. H., Zaltsman, A., Citovsky, V. A pollen-specific pectin methylesterase involved in pollen tube growth. Dev. Biol. 294, 83-91 (2006).
- Hunter, C. C., et al. Multiple nuclear localization signals mediate nuclear localization of the GATA transcription factor AreA. Eukaryot. Cell. 13, 527-538 (2014).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados