Method Article
Identificazione di sequenze di localizzazione Plasmodesmal in proteine In Planta
In questo articolo
Riepilogo
Connessioni intercellulari di pianta, la Plasmodesmi (Pd), giocano un ruolo centrale nella pianta interazioni pianta-virus e di fisiologia. Critica ai trasporti Pd sono smistamento segnali che indirizzano proteine al Pd. Tuttavia, la nostra conoscenza di queste sequenze è ancora nella sua infanzia. Descriviamo una strategia per identificare i segnali di localizzazione di Pd in proteine Pd-mirati.
Abstract
I Plasmodesmi (Pd) sono connessioni cellula--cellula che fungono da gateway attraverso cui piccole e grandi molecole sono trasportate tra cellule vegetali. Considerando che il trasporto del Pd di piccole molecole, quali gli ioni ed acqua, è presunta per accadere passivamente, cellula--cellula trasporto di macromolecole biologiche, tali proteine, si verifica molto probabilmente attraverso un meccanismo attivo che coinvolge specifici segnali di targeting sul trasportato della molecola. La scarsità di segnali di localizzazione identificati i Plasmodesmi (Pd) (PLSs) ha limitato severamente la comprensione della proteina-ordinamento pathways coinvolti nella pianta cellula--cellula macromolecolari trasporti e comunicazioni. Da una ricchezza di pianta proteine endogene e virale conosciute al traffico attraverso Pd, PLSs solo tre sono stati segnalati fin qui, tutti loro da proteine vegetali endogeni. Quindi, è importante sviluppare una strategia sperimentale sistematica e affidabile per identificare una sequenza PLS funzionale, che è necessario e sufficiente per il Pd targeting, direttamente nel soggiorno cellule vegetali. Qui, descriviamo una tale strategia utilizzando come paradigma della proteina della cellula--cellula movimento (MP) del virus del mosaico del tabacco (TMV). Questi esperimenti, che identificato e caratterizzato il primo impianto PLS virale, può essere adattati per l'individuazione di sequenze PLS in proteine più Pd-mirati.
Introduzione
I Plasmodesmi (Pd) funzionano come condotti per trasporto intercellulare di regolatori chiave di sviluppo di pianta e morfogenesi, che vanno da fattori di trascrizione di mRNA e piccole molecole di RNA. Inoltre, questa capacità di trasporto macromolecolare del Pd è utilizzata dalla maggior parte dei virus vegetali per la loro diffusione intercellulare durante l'infezione; per spostarsi tra Pd, virus vegetali si sono evolute proteine specializzate, chiamate proteine di movimento (MPs), che si rivolgono specificamente a Pd1,2,3,4,5,6 , 7. vie molecolari di trasporto Pd molto probabilmente sono intimamente interconnesse con le sequenze specifiche che mirano le proteine trasportate in queste vie. Così, identificazione di questi segnali di localizzazione di Pd (PLSs) può essere diagnostica del pathway di trasporto corrispondente di Pd. Questo è per analogia di Pd trasporto8, ad esempio, alle vie di importazione nucleare differenti, che possono essere specifiche per differenti localizzazione nucleare (NLS) di segnale sequenze9,10. Concettualmente, sia NLSs e PLSs rappresentare non liberabili sequenze di targeting subcellulare che sono necessarie e sufficienti per il targeting. Tuttavia, a differenza di NLSs11, le informazioni di sequenza PLSs sono severamente limitate. In particolare, sono state segnalate solo quattro sequenze di proteine coinvolte nel Pd targeting, con tutti i loro derivati da proteine vegetali endogeni. Il primo è rappresentato da un dominio homeobox di KN112 – un fattore di trascrizione che si muove da strati di cellule interne all'epidermide della pianta foglia13 – e i suoi omologhi di KNOX14. Quello secondo è anche da un fattore di trascrizione, Dof, che contiene un putativo PLS descritto come la tratta intercellulare (IT) motivo15. La terza sequenza è dalla proteina della membrana i plasmodesmi residente tipo 1 PDLP1, ed è rappresentato da un dominio transmembrana16. Infine, il quarto Pd sequenza di targeting recentemente è stato segnalato per glicosilfosfatidilinositolo (GPI)-proteine ancorate ed esso è rappresentato dal segnale di modificazione glicosilfosfatidilinositolo (GPI)17.
È interessante notare che, fino a poco tempo fa, nessun PLSs sono stati segnalati per MPs virale. Gli studi precedenti hanno indicato la presenza di presunti PLS sequenze in pianta virale MPs18,19, ma nessun vero PLS, vale a dire, una sequenza di amminoacido minimo necessaria e sufficiente per il Pd targeting di un carico indipendenti molecola ( ad es., PCP) è stata identificata in un MP virali. Ancora una di queste proteine, MP del virus del mosaico del tabacco (TMV), era il primo per cui Pd localizzazione e trasporti sono stati dimostrati20.
Per colmare questa lacuna, abbiamo sviluppato una strategia sperimentale per identificare TMV MP PLS. Questa strategia si basa su tre concetti. (i) abbiamo definito PLS come una sequenza di amminoacido minimo che è necessario e sufficiente per il targeting di proteina a Pd21. (ii) perché TMV MP in primo luogo destinato a Pd e poi trasloca attraverso questi canali22, abbiamo mirato a disgiungere queste due attività e identificare il PLS buona fede, che funziona solo per il targeting del Pd e non per il successivo trasporto. (iii) abbiamo analizzato il PLS identificato per residui dell'aminoacido importante per suo Pd targeting attività, sia strutturalmente che funzionalmente. Utilizzando questo approccio, abbiamo delineato una sequenza di 50-ammino acido residuo dell'estremità amminica di TMV MP che funge da PLS buona fede. Questo è stato fatto da producendo una serie di frammenti di TMV MP che satura l'intera lunghezza della proteina, tagging loro carbossilico-termini con PCP e transitoriamente esprimerli nei tessuti vegetali. Localizzazione del PD di ciascuno dei frammenti testati è stato determinato che li coexpressing con una proteina marker Pd, PDCB1 (Pd callosio proteina obbligatoria 1)23. Il frammento più piccolo che ancora localizzato al Pd, ma non ha fatto attraversare il Pd, è stato considerato per rappresentare PLS. Infine, il PLS era alanina-analizzati per determinare i residui dell'aminoacido chiave necessari per la sua struttura e/o funzione.
Considerando che qui illustriamo questo approccio descrivendo identificazione di TMV MP PLS, può essere impiegato per scoprire PLSs in qualsiasi altre proteine Pd-mirati, se codificato da patogeni vegetali o da impianti stessi; Questo è perché il nostro metodo non approfittare di eventuali caratteristiche uniche di MPs virale per quanto riguarda la loro capacità di destinazione al Pd.
Protocollo
1. il materiale vegetale
- Scelta delle specie vegetali
- Utilizzare le specie vegetali autoctone alla proteina di interesse, vale a dire, uno che codifica questa proteina per proteine endogene o che rappresenta l'ospite naturale per l'agente patogeno per proteine virali. Inoltre, le specie vegetali selezionati devono essere suscettibili del metodo scelto di trasformazione genetica transitoria.
Nota: Gli studi utilizzano abitualmente Nicotiana benthamiana, che rappresenta un buon padrone per TMV e si trasforma in modo efficiente con la tecnica di trasformazione genetica mediata da Agrobacterium, vale a dire, agroinfiltrazione (Vedi punto 3). N. benthamiana piante inoltre sono facilmente coltivate e hanno grandi foglie, che sono facilmente inoculati da Agrobacterium e facilmente analizzabile mediante microscopia confocale. -
Crescita delle piante
- Pianta N. benthamiana semi nel terreno bagnato ad alta densità. Tenerli in camera un ambiente controllato in 20-25 ˚ c sotto 16 h di luce a ~ 75 µmol fotoni m-2 s-1 e 8 ore di buio. Dopo che il diametro della euphyll raggiunge 0,5 cm, attentamente trasferire le piantine per pentole più grandi e continuare la crescita nella stessa camera alle stesse condizioni.
- Mantenere le piante fino a quando crescono alla fase 4-8-foglia entro 4 settimane, quando le foglie più grandi sono 4-6 cm di diametro; in questo momento, può essere utilizzati per agroinfiltrazione (Vedi punto 3).
Nota: Importante, mai utilizzare le piante se hanno cominciato a fiorire perché, in questa fase inerente allo sviluppo, l'architettura di foglia varia spesso24,25, portando talvolta a risultati incoerenti.
2. espressione vettoriale costruzione
-
Scelta del sistema di espressione
- Scegliere il sistema, cioè, vettori e vettore consegna metodo di espressione, più adatto per l'espressione della proteina di interesse per le specie vegetali studiate.
Nota: Occasionalmente, tali specifiche combinazioni di specie testate proteina/vegetali possono richiedono vettori specifici e vector metodo di consegna per l'ottimale espressione e funzione della proteina di interesse. Per le piante dicotiledoni più, tuttavia, selezionare plasmidi binari come vettori di espressione e agroinfiltrazione come sistema di consegna.
- Scegliere il sistema, cioè, vettori e vettore consegna metodo di espressione, più adatto per l'espressione della proteina di interesse per le specie vegetali studiate.
-
Proteina fluorescente tagging
- Fluorescente tag ogni proteina espressa per l'analisi della sua distribuzione subcellulare22, tra cui localizzazione Pd.
Nota: Impiegare autofluorescent proteine, come la PCP e DsRed2, come tag. Tag la proteina testata con PCP e coesprimere esso con un DsRed2 gratuito. Funzioni di PCP sia come tag e come una molecola di carico virus-indipendenti. DsRed2 funzioni per calibrare l'efficienza complessiva dell'espressione transitoria e, poiché esso è autonomo in cella, per identificare la cella inizialmente trasformata; Questa funzione di quest'ultima è importante nel valutare il movimento di cellula--cellula di proteine provate potenzialmente non-cella-autonomi. Per coexpression con il marcatore di Pd PDCB1, che è anche cella-autonoma, tag la proteina testata con PCP e PDCB1 con DsRed2. - Come regola generale, fusibile il tag autofluorescent carbossi-terminale della proteina espressa. Tuttavia, se le esposizioni di proteina testata full-length taggati compromessa Pd targeting, trasferire il tag per l'ammino-terminale della proteina.
- Clonare le sequenze di codificazione delle proteine per essere testato in un vettore di espressione pianta adatta.
Nota: Ci sono molti vettori di espressione di piante diverse che consentono di etichettare fluorescente. Utilizziamo una serie di pSAT plasmidi26 o alcuni dei loro derivati27,28, che sono adatti per la clonazione della sequenza di interesse direttamente nel telaio con i tag suggeriti autofluorescent (Vedi punto 2.2.1) in entrambi amminico- e orientamenti di carbossi-terminale. - Trasferire ogni cassetta di espressione in un vettore binario Agrobacterium.
Nota: Anche se basati su pSAT costrutti sono adatti per la consegna diretta Biobalistica nei tessuti vegetali, agroinfiltrazione, che è il metodo di trasformazione consigliato qui (Vedi punto 3), richiede che la cassetta di espressione per essere collocato tra il T-DNA bordi di un file binario vettore29.
Nota: Tutti i vettori di pSAT sono progettati per consentire tale trasferimento al pPZP-RCS2 espressione multigenica vettore binario26,30 da un singolo passaggio clonazione usando nucleasi raro taglio AscI, PpoI, -SceI-CeuI, PI-PspI e PI-TliI 26. i ricercatori che preferiscono utilizzare la clonazione recombinatorial, ad esempio, utilizzando eterologa lox siti31, possono impiegare pSAT vettore varianti26,27. - Per coexpression, trasferire entrambi cassette di espressione, ad esempio, testata proteina-CFP e DsRed2 o testata proteina-CFP e PDCB1-DsRed2 (Vedi punto 2.1), per lo stesso vettore binario pPZP-RCS2.
Nota: In alternativa, Agrobacterium culture che harboring costrutti binari individuali possono essere mescolate insieme per l'infiltrazione nel N. benthamiana (Vedi punto 3.1).
- Fluorescente tag ogni proteina espressa per l'analisi della sua distribuzione subcellulare22, tra cui localizzazione Pd.
3. agroinfiltrazione
-
Aggiungere 1 µ g di un plasmide basati su pPZP-RCS2 da cultura di 2.2.5 a 100 µ l di passaggio delle cellule competenti del ceppo di Agrobacterium tumefaciens EHA105 o GV3101 preparato come descritto32, Incubare in ghiaccio per 30 min, flash-congelamento in azoto liquido per 1 min, e Incubare a 28 ° C per 15 min.
- Quindi, aggiungere 1 mL di terreno LB (10 g/L triptone, 5g/L estratto di lievito e 10 g/L NaCl) alla miscela delle cellule competenti e incubare a 28 ° C per 2 h. Spin giù le cellule a 3.000 × g per 1 min, li risospendere in 0,2 mL di LB e impiattatelo su LB agar completati con gli antibiotici adatti (per esempio, spectinomicina 100 mg/L e 50 mg/L rifampicina per vettori basati su pPZP-RSC226,30). Crescere a 28 ° C per 48 h fino a quando le singole colonie sono visibili.
- Pick e piastra diverse singole colonie su una piastra di agar LB fresca (Vedi punto 3.1), crescere a 28 ° C per 48 h e prendere una piccola quantità di batteri per l'analisi di Colonia PCR33 per confermare la presenza dei costrutti specifici di binari.
-
Crescere ogni colonia di Agrobacterium con il costrutto di interesse durante la notte a 28 ° C in 5 mL di LB medium completate con gli antibiotici adatti (Vedi punto 3.1.1). Centrifugare le cellule a 3.000 × g e ri-li sospende a OD600= 0,5 nel buffer agroinfiltrazione (10mm MgCl2, 10mm MES (pH 5,6), 150 µM acetosyringone). Incubare per 2 ore a temperatura ambiente.
- Se usando una miscela di plasmidi binari piuttosto che un plasmide di espressione multigenica singolo per coexpression di proteina, mescolare le culture cella corrispondente al rapporto di 1:1 v/v prima di infiltrazione. Incubare le cellule a 28 ° C per 2 h.
-
Caricare l'inoculo in una siringa di plastica da 1 mL e premere che l'ugello della siringa (senza ago) contro dell'epidermide (abbagliai) più bassa della pienamente matura N. benthamiana lascia. Tenere la foglia con un dito guantato sul viso adassiale34,35.
- Introdurre l'agrobatterio nel mezzo di infiltrazione tramite iniezione lenta. Significatività statistica, infiltrarsi numerose foglie su ogni pianta.
Nota: Si noti che le foglie più vecchie non vengono utilizzate come non cedono i livelli di espressione coerente. - Inoculare tutte le foglie in situ. Mantenere la pianta infiltrata fino osservazione come indicato al punto 1.2.
- Introdurre l'agrobatterio nel mezzo di infiltrazione tramite iniezione lenta. Significatività statistica, infiltrarsi numerose foglie su ogni pianta.
4. microscopio confocale
- Utilizzare una lama per tagliare ogni foglia in frammenti tra le vene 24-48 h dopo l'infiltrazione, (a seconda del rendimento di espressione transitoria della proteina specifica testata); la dimensione di ogni fetta di tessuto deve essere tale che la fetta è completamente coperto da vetro di copertura (22 mm x 40 mm) utilizzato per la microscopia.
- Posto le fette foglia sull'oggetto diapositiva con la superficie del foglio abbagliai rivolto verso l'alto, mettere una goccia di acqua sulla fetta foglia e coprirlo con il vetro di copertura. Immobilizzare il vetro di copertura con piccoli pezzi di nastro su ogni lato.
-
Osservare i tessuti agroinfiltrate sotto un microscopio confocale di scansione laser e registrare le immagini risultanti.
- In primo luogo, utilizzare un obiettivo obiettivo 10x per identificare le celle con il segnale e quindi utilizzare obiettivo 63X per osservazioni più dettagliate.
- Uso la 458 nm lunghezza d'onda da un laser a ioni argon per eccitare la PCP e 543 nm per eccitare DsRed2. Mantenere tutte le impostazioni per l'acquisizione di immagini, cioè, intensità del laser e fotomoltiplicatore tubo (PMT) impostazioni, tra esperimenti. In media, esaminare le cellule di 100-120 per ogni condizione sperimentale.
5. identificazione di PLS
- Diagnosticare la localizzazione della proteina testata utilizzando il microscopio confocale (sezione 4). Verifica se la proteina testata ha il modello punctate periferico caratteristico25,36,37,38,39,40,41 e colocalizza con il marcatore di Pd PDCB123. Questo indica che la proteina testata probabilmente contiene sequenze di PLS.
- Dopo la conferma dell'attività della proteina testata, PLS progressivamente suddividere la sua cornice di lettura aperta (ORF) (numero di accessione genbank BAF93925.1) in frammenti più piccoli, autofluorescently, (ad es., PCP,) tag ciascuno di essi e analizzare loro localizzazione sottocellulare come descritto nel passaggio 4. Inizialmente, la dimensione di 100 residui dell'amminoacido per ogni frammento da è raccomandata.
- Suddividere ulteriormente i frammenti troncati che hanno l'attività PLS e controllare la loro localizzazione subcellulare come descritto nel passaggio 4 fino al più piccolo frammento che ancora si localizza al Pd, cioè è sufficiente per trasportare il carico di tag autofluorescent al Pd, è identificato. Questo frammento è presunto per rappresentare la sequenza PLS.
- Eliminare il PLS presunto dalla proteina testata full-length, cioè fusibile la parte restante della proteina della prova senza putativo PLS autofluorescent tag e determinare la localizzazione sottocellulare della proteina del mutante risultante come descritto nel passaggio 4. se questa proteina mutante che manca il presunto PLS non riesce a localizzare al Pd, questo indicherà che l'identificati PLS non è solo sufficiente (Vedi punto 5.3), ma anche necessario per il trasporto di Pd della proteina testato.
- Agroinfiltrate le foglie con una miscela di culture di Agrobacterium che harboring l'espressione costruire per la PCP-etichetta PLS e DsRed2 libero (Vedi punto 3.3). Osservare infiltrato tessuto usando il microscopio confocale con lente dell'obiettivo di 10 x utilizzando le stesse impostazioni passo 4.3.
- Punteggio di celle che contengono segnali PCP sia DsRed2 come cellule inizialmente infiltrate e segnare le celle adiacenti che conteneva solo il segnale di PCP come quelli quello movimento di esposizione.
Nota: Questo passaggio esamina il PLS identificato per la sua potenziale capacità di movimento della cellula--cellula; Questo è perché molte proteine destinate a Pd (tra cui la maggior parte pianta MPs virale) anche passare attraverso Pd alle cellule vicine. La concentrazione di Agrobacterium utilizzato per infiltrazione dipende dall'efficienza di espressione delle diverse strutture rispettivamente. Per agroinfiltrazione, assicurarsi di utilizzare la diluizione di coltura cellulare che garantisce (i) la trasformazione di singole cellule, lasciando le cellule adiacenti trasformate, (ii) raggiunge l'efficienza simile di espressione. Ad esempio, i nostri esperimenti utilizzano OD600 di 0,01 e 0,005 per espressione di PCP-etichetta PLS e libero DsRed2, rispettivamente.
6. identificazione di chiave PLS residui utilizzando alanina scansione42
- Utilizzare protocolli standard di PCR per amplificare il PLS codifica sequenza impiegando una coppia di primer disegnati per contenere la mutazione desiderata, cioè, sostituzione del residuo dell'amminoacido destinazione con alanina, intorno alla regione centrale di ogni primer come descritto 21. svolgere di disegno dell'iniettore e utilizzare il kit di mutagenesi sito-diretta per mutagenesi sito-diretta secondo le istruzioni del produttore.
- Purificare i prodotti PCR risultanti con qualsiasi kit di purificazione del DNA standard e digerire loro a 37 ° C con DpnI per eliminare il parentale (cioè, non-mutato) metilato e DNA hemimethylated. Questo raffreddare per 5 min a temperatura ambiente (non su ghiaccio). Poi trasformare la miscela direttamente in e. coli cellule competenti43e identificare le colonie con i costrutti mutanti desiderati come descritto nel passo 3.2.
- Introdurre i costrutti mutanti nel vettore binario come descritto al punto 2.2.4, eseguire agroinfiltrazione come descritto nel passaggio 3 e valutare Pd targeting come descritto nei passaggi 4 e 5.1.
Risultati
I dati rappresentativi, che fedelmente illustrano i risultati attesi dai protocolli descritti e identificano il TMV MP PLS, sono adattati da Yuan et al. 21. Figura 1A prima vengono riepilogate le principali costrutti esprimendo il full-length TMV MP (1-268), TMV MP PLS (comprendente i primi 50 residui dell'amminoacido della proteina, 1-50), e sua alanina scansione V4A derivati fusa per PCP (generata come descritte al punto 2.2 passaggi, 5.2 e 6) considerando che Figura 1B riassume e quantifica la localizzazione subcellulare di queste proteine etichettate. Figura 1 illustra i caratteristico periferico punctate Pd localizzazione modelli osservati per TMV MP-CFP e per TMV MP PLS-PCP anche per quanto riguarda il marcatore Pd coexpressed, PDCB1-DsRed2 (cfr. punto 5.1). Figura 2A illustra Pd targeting di TMV MP-CFP e della distribuzione TMV MP PLS-CFP e nucleocytoplasmic coexpressed gratuito DeRed2 (cfr. punto 5.1). Infine, Figura 2B illustra la perdita di Pd targeting da TMV MP e TMV MP PLS con una sostituzione di singola alanina V4A, che indica che questo residuo è importante per la struttura PLS o la funzione; nelle stesse cellule il marcatore Pd coexpressed PDCB1 ancora si localizza al Pd (Vedi punto 6).
Questi dati mostrano che questa procedura concettualmente e tecnicamente semplice di produzione sistematica di frammenti proteici, la loro fusione ad una proteina marker-cargo autofluorescent e test per la capacità di localizzare al Pd può identificare una sequenza della proteina minimal necessaria e sufficiente per Pd targeting, vale a dire, PLS. Per la corretta interpretazione di questi dati di localizzazione, è fondamentale per eseguire esperimenti di colocalizzazione con una proteina nota di Pd, ad esempio, PDLP o PDCB familiari16,23 o uno della pianta MPs virale che ordina al Pd44 . Ovviamente, la colocalizzazione non deve essere completa, in quanto non tutte le proteine P-localizzazione possono essere mirate per il Pd stesso, ma un grado statisticamente significativo di colocalizzazione con almeno una delle proteine marker Pd è necessario per la definizione dell'attività PLS.

Figura 1: identificazione di TMV MP pls (A) sintesi delle principali costrutti di PCP-fusione utilizzati per identificare TMV MP pls TMV MP caselle di sequenze, verde; PCP, scatole blu; P, promotore; T, terminator. I numeri a sinistra indicano i residui dell'amminoacido inclusi in ogni frammento di TMV MP. (B) sintesi della localizzazione subcellulare delle proteine di fusione mostrato in (A). Localizzazione di PD o nucleocytoplasmic localizzazione è stata determinata basato sulla localizzazione dei marcatori corrispondenti subcellulare, PDCB1-DsRed2 e DsRed2 gratis, rispettivamente. La percentuale delle cellule che esibiscono ogni modello di localizzazione è mostrata basata sul Punteggio 100 cellule esprimenti per costrutto in tre esperimenti indipendenti. Dati sono media ± errori standard (SE). (C) Pd targeting di TMV MP-CFP, TMV MP PLS-CFP e il marcatore di Pd PDCB1-DsRed2. Segnale di PCP è in blu; DsRed2 segnale è in viola; Plastidio autofluorescenza è stato filtrato. Le immagini sono singole sezioni confocale. Barre di scala = 10 µm. DIC, microfotografia contrasto differenziale di interferenza. Questa figura è adattata da Yuan et al. 21. Clicca qui per visualizzare una versione più grande di questa figura.
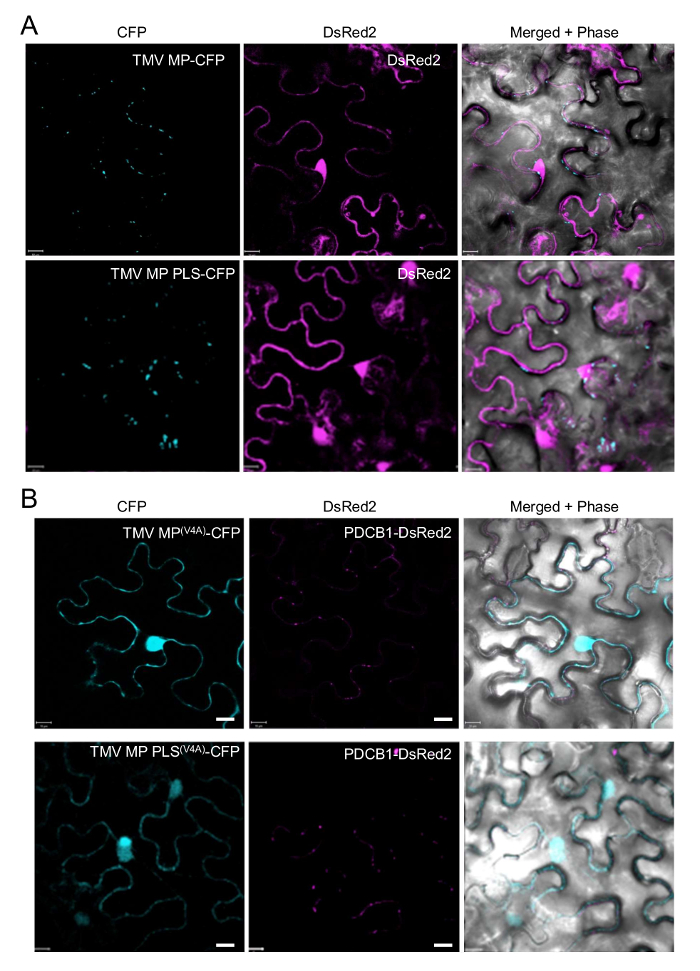
Figura 2: Pd targeting di TMV MP, TMV MP PLS e loro mutanti alanina-scansione. (A) localizzazione subcellulare di TMV MP-CFP, TMV MP PLS-CFP e il marcatore di nucleocytoplasmic DsRed2. (B) perdita di Pd targeting capacità dei mutanti V4A alanina-scansione di TMV MP-CFP e TMV MP PLS-CFP. PD sono state visualizzate da coexpression del marcatore Pd PDCB1-DsRed2. Segnale di PCP è in blu; DsRed2 segnale è in viola; Plastidio autofluorescenza è stato filtrato. Le immagini sono singole sezioni confocale. Barre di scala = 10 µm. DIC, microfotografia contrasto differenziale di interferenza. Questa figura è adattata da Yuan et al. 21. Clicca qui per visualizzare una versione più grande di questa figura.
Discussione
Questo protocollo ha quattro componenti principali: il concetto di identificazione di una sequenza che è necessario e sufficiente per il targeting al Pd, divisione sistematica della proteina di interesse in frammenti che sono progressivamente ridotti in lunghezza, fondendo il collaudato frammenti di una proteina di autofluorescent che serve sia come tag e come cargo macromolecolare e analisi funzionale per il Pd targeting in vita pianta tessuti dopo l'espressione transitoria delle proteine di fusione testata. Si noti che l'espressione transitoria mediata da Agrobacterium genera dati entro un periodo relativamente breve di tempo, vale a dire, diversi giorni, rispetto ai mesi necessari per la produzione di piante transgeniche, spesso utilizzato in simili studi15.
Coexpression della proteina di interesse con marcatori di localizzazione subcellulare, ad esempio, PDCB1 o DsRed2 gratis, di solito è eseguita da un vettore di espressione multigenica. Tuttavia, se il trasferimento di numerose cassette di espressione in un singolo vettore è tecnicamente problematico, coexpression può essere eseguita anche trasferendo ogni cassetta in una costruzione di binaria separata e combinando volumi equivalenti delle colture liquide della ceppi di Agrobacterium risultanti per agroinfiltrazione. Perché questo protocollo utilizza l'espressione transitoria, il vettore binario non necessità portare gli indicatori di selezione per trasformazione stabile, anche se la loro presenza non è dannosa per l'efficienza di espressione.
Questo protocollo è ottimizzato per l'espressione della proteina e targeting in N. benthamiana; Tuttavia, può essere utilizzato in altre specie di piante, tra cui Arabidopsis45,46, suscettibili di trasformazione genetica di Agrobacterium. Per altre specie di piante, o volendo in caso contrario, questo protocollo può essere adattato per utilizzare Biobalistica consegna41 per espressione transitoria direttamente da vettori basati su pSAT, evitando la necessità di trasferire le cassette di espressione al vettore binari.
Un punto critico in questo protocollo è la condizione fisiologica ed inerenti allo sviluppo coerenza del materiale vegetale. In particolare, tutte le piante devono essere sane in tutto l'intero esperimento e, per agroinfiltrazione, le piante N. benthamiana devono essere meno di 4 settimane vecchio e allo stadio di 4-8-foglie con la foglia più grande essendo 4-6 cm di diametro. Se le piante sono troppo giovane, gli effetti della agroinfiltrazione sarà troppo severi, interferendo con l'espressione e la localizzazione delle proteine con tag. D'altra parte, le più vecchie piante esibiscono spesso architettura variabile del Pd24,25, influenzando anche la coerenza dei modelli Pd-targeting.
Ancora, a causa di diverse condizioni fisiologiche dell'impianto nel suo complesso e delle sue cellule trasformate in particolare, l'efficienza di Pd targeting in ogni esperimento può variare un po'. Così, è importante analizzare un numero relativamente elevato di cellule (> 100) ogni esperimento ed eseguire almeno tre repliche biologiche di ogni esperimento. In generale, consideriamo un frammento testato per contenere Pd targeting per attività, se esibisce localizzazione Pd in almeno il 70% delle cellule trasformate. Differenze in Pd targeting per l'efficienza di ogni frammento, ad esempio, per cento delle cellule trasformate che mostra il motivo di accumulo punctate Pd-specifici, dovrebbero essere analizzate da dello studente t-test e i valori di p < 0,05-0,01, corrispondente alla probabilità statistica superiore a 95-99%.
Un'altra questione importante è la posizione del tag/carico fluorescente della molecola di fusione. Alcune proteine Pd-mirati anche associano il reticolo endoplasmico (ER), e questa associazione può richiedere la sequenza amino-terminale senza ostacoli. In questi casi, fusioni di amino-terminale, in cui il terminus carbossilico del frammento della proteina di interesse è fusa dell'estremità amminica del tag fluorescente, per esempio, PCP o DsRed2, deve essere utilizzato. Questa posizione di default del tag può essere modificata nei seguenti casi. (i) se le fusioni del amminico-terminale non sono utilizzabili – vale a dire, essi non presentano modelli di localizzazione subcellulare significative, sono scarsamente espressi o non generano segnale fluorescente sufficiente per rilevamento affidabile – e se non la proteina di interesse non contenere sequenze amino-terminale del segnale o fusioni di carbossi-terminale in cui l'estremità amminica del frammento testato è fusa a terminus carbossilico del tag, dovrebbe essere tentate. (ii) le fusioni di carbossi-terminale dovrebbero essere utilizzate anche se la proteina di interesse contiene sequenze di segnale al relativo terminus carbossilico. (iii) se la proteina di interesse ha entrambi i segnali amminico e carbossilico-terminale, come quelli trovati in proteine GPI ancorate, o se contiene un peptide di segnale del amminico-terminale ancora sua fusione del amminico-terminale non è utilizzabile, il tag deve essere posizionato internamente ( o a valle dell'ingresso del amminico-terminale o a monte dell'ingresso del carbossilico-terminale). Alcune procedure utili per l'etichettatura fluorescente interna di proteine sono descritti nelle nostre precedenti pubblicazioni47,48.
Allo stato attuale, non esiste nessuna sequenza consenso noti per PLS. Come tale (al punto 5.2) si consiglia, inizialmente suddividendo la proteina in contiguo ~frammenti residui-lungo 100-200. Questi possono poi essere fatti progressivamente più brevi sulla base l'attività osservata Pd-targeting o la loro mancanza. Uno dovrebbe essere consapevole di altri potenziali targeting segnali presenti nella proteina (ad es., NLS, peptidi di segnale, ecc.). Tuttavia, come la potenziale sovrapposizione di tali sequenze con sequenze di PLS è sconosciuta, queste regioni della proteina dovrebbero essere incluso in questa analisi mutational.
Finora, questo protocollo è stato usato per identificare un singolo PLS in una molecola di proteina21. Potenzialmente, tuttavia, una proteina può contenere più di una sequenza segnale; infatti più NLSs sono state descritte in molte proteine nucleari, comprese GATA trascrizione fattori49. Così, quando si analizzano diversi frammenti della proteina testata per il targeting del Pd, è possibile che più di un tale frammento sarà identificato, che indica la presenza di multiple PLSs che possono agire singolarmente o in sinergia.
Il limite principale di questo protocollo è che, seguendo transitoriamente agroinfiltrazione, visualizzazione della localizzazione subcellulare delle proteine espresse di interesse si ottiene in cellule epidermiche e in fase di crescita relativamente stretta. Quando necessario, questa limitazione può essere superata con la produzione di piante transgeniche che esprimono stabilmente ciascuna delle proteine provate nella maggior parte dei loro tessuti e tipi di cellule. Inoltre, questo protocollo non sarebbe utile per gli studi di MPs virali o altre proteine Pd-mirati che funzionano solo in specie di piante che non sono suscettibili di trasformazione genetica transitoria.
Mentre abbiamo sviluppato questa tecnica per l'identificazione di un PLS di TMV MP, può essere utilizzato per la scoperta di PLSs in qualsiasi altro impianto MPs virale o in proteine endogene di pianta che si localizzano Pd, transitoriamente o permanentemente. Inoltre, se combinato con localizzazione subcellulare appropriati marcatori47, questo protocollo è adatto per l'identificazione di molti altri segnali di destinazione all'interno di proteine di interesse in piante.
Divulgazioni
Conflitti di interesse non dichiarati.
Riconoscimenti
Per la mancanza di spazio, abbiamo citato principalmente articoli di recensione e ci scusiamo con i nostri colleghi cui lavoro originale non è stato citato. Il lavoro in laboratorio V.C. è supportato da sovvenzioni dal NIH, NSF, USDA/catino, BARD e BSF a V.C., e il laboratorio S.G.L è supportato da NIH e fondi dai dipartimenti di patologia vegetale e biologia vegetale-microbo a S.G.L
Materiali
| Name | Company | Catalog Number | Comments |
| Confocal laser scanning microscope (CLSM) | Zeiss | LSM5 | Any CLSM with similar capabilities is appropriate |
| Zen software for confocal microscope imaging | Zeiss | 2009 version | The software should be compatible with the CLSM used |
| Quickchange II site-directed mutagenesis kit | Agilent | 200523 | |
| Acetosyringone | Sigma-Aldrich | D134406 | |
| MES | Sigma-Aldrich | 69892 | |
| Syringes without needles | BD | 309659 | |
| MgCl2 | FisherScientific | M33-500 | |
| Spectinomycin | Sigma-Aldrich | S4014 | |
| Rifampicin | Sigma-Aldrich | R3501 | |
| Ampicillin | Sigma-Aldrich | A0166 |
Riferimenti
- Lee, J. Y. Plasmodesmata: a signaling hub at the cellular boundary. Curr. Opin. Plant Biol. 27, 133-140 (2015).
- Kumar, D., Kumar, R., Hyun, T. K., Kim, J. Cell-to-cell movement of viruses via plasmodesmata. J. Plant Res. 128, 37-47 (2015).
- Kitagawa, M., Paultre, D., Rademaker, H. Intercellular communication via plasmodesmata. New Phytol. 205, 970-972 (2015).
- Jackson, D. Plasmodesmata spread their influence. F1000Prime Rep. 7, 25(2015).
- Brunkard, J. O., Runkel, A. M., Zambryski, P. C. The cytosol must flow: intercellular transport through plasmodesmata. Curr. Opin. Cell Biol. 35, 13-20 (2015).
- Yadav, S. R., Yan, D., Sevilem, I., Helariutta, Y. Plasmodesmata-mediated intercellular signaling during plant growth and development. Front. Plant Sci. 5, 44(2014).
- Sager, R., Lee, J. Y. Plasmodesmata in integrated cell signaling: insights from development and environmental signals and stresses. J. Exp. Bot. 65, 6337-6358 (2014).
- Jans, D. A., Xiao, C. Y., Lam, M. H. Nuclear targeting signal recognition: a key control point in nuclear transport? BioEssays. 22, 532-544 (2000).
- Miyamoto, Y., et al. Different modes of nuclear localization signal (NLS) recognition by three distinct classes of NLS receptors. J. Biol. Chem. 272, 26375-26381 (1997).
- Nair, R., Carter, P., Rost, B. NLSdb: database of nuclear localization signals. Nucleic Acids Res. 31, 397-399 (2003).
- Lee, J. Y., Yoo, B. C., Lucas, W. J. Parallels between nuclear-pore and plasmodesmal trafficking of information molecules. Planta. 210, 177-187 (2000).
- Kim, J. Y., Rim, Y., Wang, J., Jackson, D. A novel cell-to-cell trafficking assay indicates that the KNOX homeodomain is necessary and sufficient for intercellular protein and mRNA trafficking. Genes Dev. 19, 788-793 (2005).
- Lucas, W. J., et al. Selective trafficking of KNOTTED1 homeodomain protein and its mRNA through plasmodesmata. Science. 270, 1980-1983 (1995).
- Chen, H., Jackson, D., Kim, J. Y. Identification of evolutionarily conserved amino acid residues in homeodomain of KNOX proteins for intercellular trafficking. Plant Signal. Behav. 9, e28355(2014).
- Chen, H., Ahmad, M., Rim, Y., Lucas, W. J., Kim, J. Y. Evolutionary and molecular analysis of Dof transcription factors identified a conserved motif for intercellular protein trafficking. New Phytol. 198, 1250-1260 (2013).
- Thomas, C. L., Bayer, E. M., Ritzenthaler, C., Fernandez-Calvino, L., Maule, A. J. Specific targeting of a plasmodesmal protein affecting cell-to-cell communication. PLOS Biol. 6, e7(2008).
- Zavaliev, R., Dong, X., Epel, B. L. Glycosylphosphatidylinositol (GPI) modification serves as a primary plasmodesmal targeting signal. Plant Physiol. 172, 1061-1073 (2016).
- Sasaki, N., Park, J. W., Maule, A. J., Nelson, R. S. The cysteine-histidine-rich region of the movement protein of Cucumber mosaic virus contributes to plasmodesmal targeting, zinc binding and pathogenesis. Virology. 349, 396-408 (2006).
- Kaido, M., Funatsu, N., Tsuno, Y., Mise, K., Okuno, T. Viral cell-to-cell movement requires formation of cortical punctate structures containing Red clover necrotic mosaic virus movement protein. Virology. 413, 205-215 (2011).
- Creager, A. N. H., Scholthof, K. B. G., Citovsky, V., Scholthof, H. B. Tobacco mosaic virus: pioneering research for a century. Plant Cell. 11, 301-308 (1999).
- Yuan, C., Lazarowitz, S. G., Citovsky, V. Identification of a functional plasmodesmal localization signal in a plant viral cell-to-cell movement protein. mBio. 7, e02052-e02015 (2016).
- Ueki, S., Citovsky, V. To gate, or not to gate: regulatory mechanisms for intercellular protein transport and virus movement in plants. Mol. Plant. 4, 782-793 (2011).
- Simpson, C., Thomas, C. L., Findlay, K., Bayer, E., Maule, A. J. An Arabidopsis GPI-anchor plasmodesmal neck protein with callose binding activity and potential to regulate cell-to-cell trafficking. Plant Cell. 21, 581-594 (2009).
- Maule, A. J. Plasmodesmata: structure, function and biogenesis. Curr. Opin. Plant Biol. 11, 680-686 (2008).
- Roberts, I. M., et al. Dynamic changes in the frequency and architecture of plasmodesmata during the sink-source transition in tobacco leaves. Protoplasma. 218, 31-44 (2001).
- Tzfira, T., et al. pSAT vectors: a modular series of plasmids for fluorescent protein tagging and expression of multiple genes in plants. Plant Mol. Biol. 57, 503-516 (2005).
- Chakrabarty, R., et al. pSITE vectors for stable integration or transient expression of autofluorescent protein fusions in plants: probing Nicotiana benthamiana-virus interactions. Mol. Plant-Microbe Interact. 20, 740-750 (2007).
- Chung, S. M., Frankman, E. L., Tzfira, T. A versatile vector system for multiple gene expression in plants. Trends Plant Sci. 10, 357-361 (2005).
- Lee, L. Y., Gelvin, S. B. T-DNA binary vectors and systems. Plant Physiol. 146, 325-332 (2008).
- Goderis, I. J., et al. A set of modular plant transformation vectors allowing flexible insertion of up to six expression units. Plant Mol. Biol. 50, 17-27 (2002).
- Walhout, A. J., et al. GATEWAY recombinational cloning: application to the cloning of large numbers of open reading frames or ORFeomes. Methods Enzymol. 328, 575-592 (2000).
- Tzfira, T., et al. Transgenic Populus: a step-by-step protocol for its Agrobacterium-mediated transformation. Plant Mol. Biol. Rep. 15 (3), 219-235 (1997).
- Woodman, M. E., Savage, C. R., Arnold, W. K., Stevenson, B. Direct PCR of intact bacteria (colony PCR). Curr. Protoc. Microbiol. 42 (3D), 1-7 (2016).
- Kapila, J., De Rycke, R., Van Montagu, M., Angenon, G. An Agrobacterium-mediated transient gene expression system for intact leaves. Plant Sci. , 101-108 (1997).
- Wroblewski, T., Tomczak, A., Michelmore, R. Optimization of Agrobacterium-mediated transient assays of gene expression in lettuce, tomato and Arabidopsis. Plant Biotechnol. J. 3, 259-273 (2005).
- Boyko, V., Ferralli, J., Ashby, J., Schellenbaum, P., Heinlein, M. Function of microtubules in intercellular transport of plant virus RNA. Nat. Cell Biol. 2, 826-832 (2000).
- Heinlein, M., Epel, B. L., Padgett, H. S., Beachy, R. N. Interaction of tobamovirus movement proteins with the plant cytoskeleton. Science. 270, 1983-1985 (1995).
- Oparka, K. J., Prior, D. A. M., Santa-Cruz, S., Padgett, H. S., Beachy, R. N. Gating of epidermal plasmodesmata is restricted to the leading edge of expanding infection sites of tobacco mosaic virus (TMV). Plant J. 12, 781-789 (1997).
- Crawford, K. M., Zambryski, P. C. Non-targeted and targeted protein movement through plasmodesmata in leaves in different developmental and physiological states. Plant Physiol. 125, 1802-1812 (2001).
- Kotlizky, G., et al. A dysfunctional movement protein of Tobacco mosaic virus interferes with targeting of wild-type movement protein to microtubules. Mol. Plant-Microbe Interact. 14, 895-904 (2001).
- Ueki, S., Lacroix, B., Krichevsky, A., Lazarowitz, S. G., Citovsky, V. Functional transient genetic transformation of Arabidopsis leaves by biolistic bombardment. Nat. Protoc. 4, 71-77 (2009).
- Giesman-Cookmeyer, D., Lommel, S. A. Alanine scanning mutagenesis of a plant virus movement protein identifies three functional domains. Plant Cell. 5, 973-982 (1993).
- Ausubel, F. M., et al. Current Protocols in Molecular Biology. , Wiley-Interscience. New York. (1987).
- Waigmann, E., Ueki, S., Trutnyeva, K., Citovsky, V. The ins and outs of non-destructive cell-to-cell and systemic movement of plant viruses. Crit. Rev. Plant Sci. 23, 195-250 (2004).
- Lee, M. W., Yang, Y. Transient expression assay by agroinfiltration of leaves. Methods Mol. Biol. 323, 225-229 (2006).
- Burch-Smith, T. M., Schiff, M., Liu, Y., Dinesh-Kumar, S. P. Efficient virus-induced gene silencing in Arabidopsis. Plant Physiol. 142, 21-27 (2006).
- Tian, G. W., et al. High-throughput fluorescent tagging of full-length Arabidopsis gene products in planta. Plant Physiol. 135, 25-38 (2004).
- Tian, G. W., Chen, M. H., Zaltsman, A., Citovsky, V. A pollen-specific pectin methylesterase involved in pollen tube growth. Dev. Biol. 294, 83-91 (2006).
- Hunter, C. C., et al. Multiple nuclear localization signals mediate nuclear localization of the GATA transcription factor AreA. Eukaryot. Cell. 13, 527-538 (2014).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon