Method Article
O isolamento de regiões específicas genômico e identificação de moléculas pela Associated enChIP
Neste Artigo
Resumo
The identification of molecules associated with specific genomic regions of interest is required to understand the mechanisms of regulation of the functions of these regions. This protocol describes procedures to perform engineered DNA-binding molecule-mediated chromatin imunoprecipitation (enChIP) for identification of proteins and RNAs associated with a specific genomic region.
Resumo
The identification of molecules associated with specific genomic regions of interest is required to understand the mechanisms of regulation of the functions of these regions. To enable the non-biased identification of molecules interacting with a specific genomic region of interest, we recently developed the engineered DNA-binding molecule-mediated chromatin immunoprecipitation (enChIP) technique. Here, we describe how to use enChIP to isolate specific genomic regions and identify the associated proteins and RNAs. First, a genomic region of interest is tagged with a transcription activator-like (TAL) protein or a clustered regularly interspaced short palindromic repeats (CRISPR) complex consisting of a catalytically inactive form of Cas9 and a guide RNA. Subsequently, the chromatin is crosslinked and fragmented by sonication. The tagged locus is then immunoprecipitated and the crosslinking is reversed. Finally, the proteins or RNAs that are associated with the isolated chromatin are subjected to mass spectrometric or RNA sequencing analyses, respectively. This approach allows the successful identification of proteins and RNAs associated with a genomic region of interest.
Introdução
A identificação de moléculas associadas com as regiões genómicas específicas de interesse é necessária para compreender os mecanismos de regulação das funções genómicas, tais como transcrição e regulação epigenética. Embora tenham sido desenvolvidas várias técnicas para análise bioquímica de regiões genômicas específicas 1-7, eles não são amplamente utilizados nesta fase por causa de seus problemas intrínsecos, tais como aplicação limitada (por exemplo, apenas para-elevado número de cópias ou loci loci com repetições) e muito tempo e esforço necessário.
Para realizar a análise bioquímica de regiões genômicas específicas facilmente, nós desenvolvemos duas tecnologias específicas do locus de cromatina imunoprecipitação (chip), nomeadamente insercional chip (Ichip) 8-13 e engenharia mediada por molécula de ligação a DNA ChIP (enChIP) 14-17 . Em Ichip, um lugar de interesse é marcado por sequências de reconhecimento de inserção de uma proteína de ligação de DNA exógeno, como LexA. O locus é então isolado através de purificação por afinidade, utilizando a proteína de ligação de ADN marcado. Em enChIP, modificadas moléculas de ligação a ADN, tais como proteínas de dedos de zinco, proteínas (Tal) de activador do tipo de transcrição, e agrupados regularmente interspaced repetições palindr�icas curtas (CRISPR) complexos, são utilizados para marcar um local de interesse (Figura 1). Subsequentemente, a região genómica que é isolado através de purificação por afinidade das moléculas de ligação de ADN marcados.
Uma das vantagens de enChIP sobre ichip é que a inserção de sequências de reconhecimento de uma proteína de ligação do ADN exógeno não é necessário. Segmentação de loci CRISPR usando complexos que consistem em uma forma cataliticamente inactivo de Cas9 (dCas9) e um guia de RNA (gRNA) é muito mais fácil do que a segmentação dessas regiões por iChip ou enChIP utilizando proteínas TAL e zinc-finger. Aqui, descrevemos um protocolo passo-a-passo para enChIP combinada com a espectrometria de massa e sequenciação de RNA (RNA-Seq) para identificar lócus associa-proteínas e RNAs ted, respectivamente.
Protocolo
1. Projeto de Engineered moléculas de DNA-binding Reconhecendo o alvo Locus
- Para enChIP usando complexos CRISPR, use a ferramenta web CRISPRdirect (http://crispr.dbcls.jp) para identificar sequências alvo gRNA candidato na região genômica de interesse descrito como anteriormente 18. Esta ferramenta da web retorna locais genómicos 23 pb da forma 5'-N 20 NGG-3 ', na região de alvo.
- Sintetizar um gBlock incluindo a sequência de promotor U6 e a sequência de 5'-N 20 em 5'-N 20 NGG-3 ', utilizando serviços comerciais (ver Figura 2) 19,20. Para fins de subclonagem, incluem sítios de enzimas de restrição adequados fora do gBlock.
- Inserir o gBlock num vector apropriado tal como previamente descrito 16. Vários vetores retrovirais para gBlocks estão disponíveis (ver Materiais).
- Para enChIP utilizando proteínas TAL, projetar proteínas TAL reconhecendo a target loci. Gerar plasmídeos que codificam proteínas TAL usando serviços comerciais. Gerar vectores de expressão que contêm as proteínas fundidas TAL com uma ou mais marcas, tais como 3 × marcar como previamente descrito 15.
2. Criação de Células para Análise enChIP
- Express 3 × FLAG-dCas9 e o gRNA (procedimento baseia-CRISPR) ou 3 × FLAG-Tal (à base de proteínas procedimento TAL) nas células a serem analisadas conforme descrito anteriormente 14-17.
- Use transfecção transiente, se a eficiência de transfecção é alta. Um vector de expressão contendo 3 × FLAG-dCas9 e o promotor CMV está disponível (ver Materiais).
- Considerar a criação de transformantes estáveis usando métodos convencionais, se a eficiência de transfecção transitória é baixa. Para além dos vectores retrovirais acima mencionados para gBlock, vectores retrovirais de 3 × FLAG-dCas9 estão disponíveis (ver Materiais).
- Confirmar a expressão da 3xFLAG-dCas9 ou proteína 3xFLAG-TAL nas células transfectadas por análise de imunotransferência utilizando um anticorpo anti-FLAG (Ab) tal como descrito anteriormente 14-17.
Nota: A expressão destas proteínas marcadas também podem ser confirmadas por coloração intracelular com anti-FLAG-FITC e uma análise de FACS subsequente. Um protocolo para a coloração intracelular pode ser baixado do nosso homepage (http://www.biken.osaka-u.ac.jp/lab/microimm/fujii/iChIP_protocols/english.html). - Confirmar a expressão do gRNA pela norma técnica de RT-PCR.
- Para a identificação quantitativa de proteínas que interagem com a região genômica de interesse, considere o uso de uma rotulagem isótopo estável por aminoácidos em cultura de células (SILAC) análise combinada com enChIP (enChIP-SILAC).
Nota: Os Meios para a análise SILAC podem ser adquiridos de fornecedores comerciais. SILAC é poderosa para detectar interacções específicas.- Células de cultura em SILAC meio pesado. Prepare células cultuvermelho em meio luz SILAC como controle negativo. Utilize pelo menos 5 × 10 7 células para cada meio SILAC (leve ou pesada). Para a rotulagem eficiente, adicione tanto L-Lisina-2HCl, 13 C 6 e L-Arginina-HCl, 13 C 6, 15 N 4 na mídia SILAC pesados. Nota: Pelo menos cinco divisões celulares são necessárias para proteínas de etiquetas.
- Por exemplo, células de cultura humanas fibrossarcoma HT1080 que expressam estavelmente 3xFLAG-dCas9 e um gRNA a 37 ° C e 5% de CO 2 em meio DMEM durante SILAC e soro fetal de bovino dialisado com Lisina-2HCl mais L-arginina-HCl (média de luz) ou 13 C 6 L-Lisina-2HCl mais 13 C 6 15 N 4 L-Arginina-HCl (meio-pesado) (veja Materiais) 16. Adicionam-se 50 mg de leve ou pesada de L-Lisina-2HCl e L-Arginina-HCl em 500 ml de meio.
- Células Trypsinize e replate antes de se tornarem confluentes, de modo que as células manter growt exponencialh.
- Células de cultura em SILAC meio pesado. Prepare células cultuvermelho em meio luz SILAC como controle negativo. Utilize pelo menos 5 × 10 7 células para cada meio SILAC (leve ou pesada). Para a rotulagem eficiente, adicione tanto L-Lisina-2HCl, 13 C 6 e L-Arginina-HCl, 13 C 6, 15 N 4 na mídia SILAC pesados. Nota: Pelo menos cinco divisões celulares são necessárias para proteínas de etiquetas.
3. A ligação cruzada de células com formaldeído
- Para as células em cultura em suspensão, de transferência de 2 x 10 7 células que expressam proteínas 3xFLAG-Tal ou 3xFLAG-dCas9 mais gRNA e suspender em 30 ml de meio de cultura regular em um tubo de centrífuga de 50 ml. Quando são usadas mais células, aumentar os volumes e as quantidades de reagentes proporcionalmente. Para as experiências de SILAC, misturar as células cultivadas em meio pesado ou médio da luz (5 x 10 7 células cada) e dividir o total de 1 x 10 células em 8 5 tubos contendo 2 x 10 7 células.
- Adicionar 810 ul de 37% de formaldeído a 30 ml de suspensão celular (concentração final 1%) e incuba-se a 37 ° C durante 5 min. Para as células aderentes, fixar directamente as células em placas de cultura pela adição de 810 ul de 37% de formaldeído a 30 ml de meio de cultura.
- Pare a reticulação por adição de 3,1 ml de 1,25 M de solução de glicina (127 mM de concentração final), eincubar à TA durante 10 min.
- Recolher as células por centrifugação (300 x g durante 5 min a 4 ° C). Cuidadosamente descarte o sobrenadante incluindo formaldeído e armazená-lo em um frasco adequado de lixo.
- Para as células aderentes, separar as células com um raspador de células e colheita em um tubo de 50 ml, e recolher as células, tal como descrito neste passo.
- Para as experiências de SILAC, misturar as células isoladas em cultura em meio pesado ou meio de luz (5 x 10 7 células cada), dividir o total de 1 x 10 8 células em 5 tubos contendo 2 x 10 7 células, e recolher as células tal como descrito no presente passo.
- Lava-se a pelete de células com 30 ml de PBS duas vezes por tubo. Cuidadosamente descarte o sobrenadante incluindo formaldeído e armazená-lo em um recipiente adequado de resíduos bottle.Handle o formaldeído de acordo com as diretrizes de segurança de produtos químicos. As células fixadas podem ser congeladas e armazenadas a -80 ° C.
4. Preparação da Cromatina (por 2 x 107 células)
- Suspender as células fixadas em 10 ml de tampão de lise celular (Tris 10 mM (pH 8,0), EDTA 1 mM, 0,5% de IGEPAL CA-630, e 1 × inibidores da protease) e incubar em gelo durante 10 min.
- Centrifugar a amostra (830 x g a 4 ° C durante 8 min) e o sobrenadante cuidadosamente descartar.
- Suspende-se o sedimento em 10 ml de tampão de lise nuclear (Tris 10 mM (pH 8,0), EDTA 1 mM, NaCl 0,5 M, 1% de Triton X-100, desoxicolato de sódio a 0,5%, 0,5% lauroilsarcosina, e inibidores da protease 1x). Incubar em gelo durante 10 min e agitar no vortex todos os 2-3 minutos.
- Centrifugar a amostra (830 × g a 4 ° C durante 8 min) e o sobrenadante cuidadosamente descartar.
- Lava-se a pelete em 10 ml de PBS. O sedimento (fracção cromatina) pode ser armazenada a -80 ° C, após o congelamento imediato em azoto líquido.
5. Sonication de cromatina (por 2 x 10 7 células)
- Suspender a fracção cromatina em 800 ul detampão de lise modificada 3 (Tris 10 mM (pH 8,0), EDTA 1 mM, NaCl 150 mM, desoxicolato de sódio a 0,1%, 0,1% SDS e inibidores de protease 1x) e transferir para um tubo de 1,5 ml.
- Sonicar a cromatina utilizando um sonicador (ver Materiais) e as seguintes condições: a saída: 3; dever: 100% (contínua); e tempo: livre. Realizar 10-18 ciclos de ultra-sons durante 10 segundos e arrefecimento em gelo durante 20 seg. Para evitar o aquecimento excessivo, incubar as amostras em gelo durante 2 minutos a cada 5-6 ciclos. Para evitar a formação de espuma, manter a posição da ponta da sonda de ultra-sons de 0,5 cm acima do fundo do tubo.
- Centrifugar a amostra a (16000 x g a 4 ° C durante 10 min) e transferir o sobrenadante para um tubo de 1,5 ml. A cromatina sonicado pode ser armazenado a -80 ° C, após o congelamento imediato em azoto líquido.
6. Avaliação da cromatina Fragmentação
- Misturar 10 ul da cromatina fragmentada com 85 ul de água destilada.
- Adicione 4 ulde NaCl 5 M e incuba-se a 65 ° CO / N.
- Adicionar 1 mL de 10 mg / ml de RNase A e incubar a 37 ° C durante 45 min.
- Adicionar 2 ul de EDTA 0,5 M (pH 8,0), 4 pi de 1 M de Tris (pH 6,8), e 1 uL de 20 mg / ml de Proteinase K, e, em seguida, incuba-se a 45 ° C durante 1,5 h.
- Separada de 10 ul da amostra por electroforese em um gel de agarose a 1% que não contém corantes de coloração tais como SYBR verde.
- Corar o gel para avaliar a distribuição dos comprimentos da cromatina fragmentada. Condições que geram um comprimento médio de 0,5-2 kpb (gama de 0,2-4 kpb) são recomendados.
- Purifica-se o ADN das amostras restantes por extracção com fenol-clorofórmio ou utilizando um kit de extracção de ADN (ver Materiais). O ADN purificado pode ser utilizado como o ADN de entrada para estimar os rendimentos de análises enChIP (ver 8.11).
7. Preparação de Dynabeads conjugado com anticorpos (por 2 x 10 7 células)
- Prépare duas 1,5 ml tubos, um para pré-limpeza utilizando IgG de rato normal e outra para incubação com o anti-FLAG Ab. Adicionar 150 ul de proteína G-conjugado esferas magnéticas (ver Materiais) para cada tubo.
- Colocar os tubos em um carrinho ímã e esperar por 3 min. Descartar o sobrenadante por pipetagem.
- Suspender as pérolas em 1 ml de PBS contendo 0,01% de Tween-20 (PBS-T). Colocar os tubos em um suporte magnético e esperar por 2 min. Desprezar o sobrenadante por pipetagem, em seguida, repita o passo.
- Suspender as pérolas em 1 ml de PBS-T contendo 0,1% de BSA.
- Adiciona-se 15 ug de IgG de rato normal ou anti-FLAG Ab e girar a 4 ° CO / N.
- Spin down brevemente, em seguida, colocar os tubos em um carrinho ímã e esperar por 3 min. Descartar o sobrenadante por pipetagem.
- Suspender as pérolas em 1 ml de PBS-T. Inverta as várias vezes de amostra e girar rapidamente. Colocar os tubos em um carrinho ímã e esperar por 3 min, em seguida, descartar o sobrenadante por pipetagem. Repita a lavagemmais duas vezes com PBS-T (um total de três passos de lavagem).
8. imunoprecipitação da cromatina (por 2 x 10 7 células)
- Misture a cromatina fragmentada preparado em 5.3) (aproximadamente 800 ul) com um quinto do volume (aproximadamente 200 ul) de tampão de lise modificada contendo 3 5% de Triton X-100 (concentração final de 1% de Triton X-100).
- Adicionar a solução da cromatina para o tubo em que contas magnéticas conjugadas com Proteína G conjugadas com IgG de ratinho normal foram preparados. Girar a 4 ° C durante 1 h.
- Colocar o tubo em um carrinho ímã e esperar por 3 min.
- Transferir o sobrenadante para dentro do tubo em que contas magnéticas conjugadas com Proteína G conjugadas com anti-FLAG Ab foram preparados. Girar a 4 ° CO / N.
- Colocar o tubo em um carrinho ímã e esperar por 3 min. Descartar o sobrenadante por pipetagem.
- Suspender as pérolas em 1 ml de tampão de baixa salinidade (20 mM de Tris (pH 8,0), EDTA a 2 mM, NaCl 150 mM, 1% de triton X-100, 0,1% de SDS, e 1x inibidores de protease) e rodar a 4 ° C durante 5 min. Colocar o tubo em um carrinho ímã e esperar por 3 min. Desprezar o sobrenadante por pipetagem e repita o passo de lavagem.
- Lavam-se as esferas com tampão de alto teor salino (Tris 20 mM (pH 8,0), EDTA a 2 mM, NaCl 500 mM, 1% de Triton X-100, 0,1% de SDS, e inibidores da protease 1x) duas vezes.
- Lavam-se as esferas com tampão de LiCl (10 mM de Tris (pH 8,0), EDTA 1 mM, 250 mM de LiCl, 0,5% de IGEPAL CA-630, desoxicolato de sódio a 0,5%, e inibidores de protease 1x) duas vezes.
- Lavam-se as esferas com TBS-IGEPAL CA-630 (Tris 50 mM (pH 7,5), NaCl 150 mM, IGEPAL CA-inibidores 630, e 1x de protease a 0,1%) uma vez.
- Suspender as pérolas em 200 ul de tampão de eluição (Tris a 50 mM (pH 7,5), NaCl 150 mM, 0,1% de IGEPAL CA-630, os inibidores da protease de 1x, e 500 ug / ml de 3 péptido × FLAG) e incubar a 37 ° C durante 20 min. Colocar o tubo em um carrinho ímã e esperar por 3 min. Transferir o sobrenadante para um novo tubo de 1,5 ml e repetir o Elutpasso ion.
- Purifica-se o ADN a partir de pequena porção (por exemplo, 5%) do eluato por extracção com fenol-clorofórmio ou utilizando um kit de extracção de ADN (ver Materiais). O ADN purificado pode ser utilizado para a PCR com conjuntos de iniciadores específicos para estimar os rendimentos de análises enChIP por comparação com o ADN de entrada, preparada na etapa 6.7, como descrito anteriormente 14,16.
9. SDS-PAGE, coloração e análise de espectrometria de massa
- Misturar o eluato (400 ul) com 1 ml de 2-propanol, 50 ul de acetato de sódio 3M (pH 5,2), e 5 ul de 20 mg / ml de glicogénio. Precipitar o cromatina a -20 ° CO / N.
- Centrifugar a amostra (16000 x g a 4 ° C durante 30 min) e desprezar o sobrenadante. Lavar o sedimento com 1 ml de etanol a 70% e centrifuga-se novamente (16000 x g a 4 ° C durante 10 min). Desprezar o sobrenadante completamente por pipetagem.
- Suspende-se o sedimento em 40 ul de tampão de amostra 2x (Tris 125 mM (pH 6,8), 10% de 2-mercaptoethanol, SDS a 4%, sacarose a 10%, e 0,004% de azul de bromofenol). Vortex durante 5 min para dissolver o sedimento completamente, e, em seguida, incuba-se a 100 ° C durante 30 minutos para desnaturar as proteínas e inverter a reticulação.
- Por SDS-PAGE, correr 40 ul da amostra no gel até o corante atinge 1 cm abaixo do poço. Nota: Normalmente, usamos 4-20% géis de gradiente, mas outros géis% pode ser usado.
- Manchar o gel com Coomassie Brilliant Blue ou prata.
- Corte o gel em cinco pedaços (2 mm de altura).
- Realiza-se em digestão em gel e análise espectrométrica de massa, tal como descrito anteriormente 13-16.
- Para as experiências de SILAC com 5 x 10 7 células cada (total de 1 x 10 8 células), suspender a pelete a partir dos cinco tubos iniciais em 40 ul de tampão de amostra 2x (não é necessário para intensificar).
10. A purificação de RNA e Análise de RNA-Seq
- Para purificar o ARN enChIP depois, adicionam-se 5 U / ml de ARNase Inibidor (SEe Materiais) para todas as soluções tampão, excepto tampão de lise modificada 3 e o tampão de eluição, para que adicionar 40 U / ml de RNase inibidor.
- Misturar o eluato (400 ul) com 16 ul de NaCl 5 M e incuba-se a 65 ° C durante 2 horas.
- Adicionar 1 ml de solução de ácido de guanidínio-fenol-base-(ver Materiais) para a amostra. Vortex durante 15 segundos e, em seguida incubar à temperatura ambiente durante 5-15 min. Centrifugar a amostra durante 15 min a 12.000 x g e RT.
- Transferir o sobrenadante para um novo tubo de 1,5 mL e adicionar 5 uL de p-bromoanisole. Vortex durante 15 segundos e, em seguida incubar à temperatura ambiente durante 3-5 minutos. Centrifugar a amostra durante 10 min a 12.000 x g e RT.
- Transferir o sobrenadante (aproximadamente 1 ml) para um novo tubo de 2 mL e adicionar 1 ml de 2-propanol. Inverter o tubo e incubar à TA durante 10 min. Carregar a mistura numa coluna de um kit de purificação de RNA (ver Materiais). Centrifugar a coluna durante 1 min a 12.000 × g e RT.
- lavagema coluna com 400 mL de RNA Wash Buffer em um kit de purificação de RNA (ver Materiais).
- Adicionar 80 ul de ADNase I cocktail (mistura de 5 ul de DNase I (1 U / ul), 8 uL de 10 × ADNase I Tampão de Reacção, 3 ul de DNase / RNase isenta de água, e 64 ul de ARN do Tampão de Lavagem em um kit de purificação de RNA (ver Materiais)) para a coluna. Incubar a amostra a 37 ° C durante 15 min e em seguida centrifugar durante 30 segundos a 12.000 x g e RT.
- Lavar a coluna com 400 mL de RNA pré-lavagem tampão em um kit de purificação de RNA (ver Materiais) duas vezes.
- Elui-se o ARN com 50 ul de ADNase / ARNase isenta de água. O RNA eluído pode ser utilizado para a sequenciação de ARN.
Resultados
Em geral, 1-30% de regiões genómicas alvo pode ser purificado utilizando enChIP. Figura 3 inclui dados representativos mostram os rendimentos em enChIP análises de telómeros direccionamento e o promotor do interferão (IFN) reguladora factor-1 (IRF-1) gene. Como exemplos de resultados típicos, Tabela 1 lista as proteínas associadas com o IRF-1 promotor de uma forma de IFNy específico identificado por enChIP-SILAC, Tabela 2 lista as proteínas se ligam a telómeros identificados por enChIP combinada com espectrometria de massa (enChIP-MS) e Tabela 3 lista os RNAs associadas com telômeros identificados por enChIP-RNA-Seq.
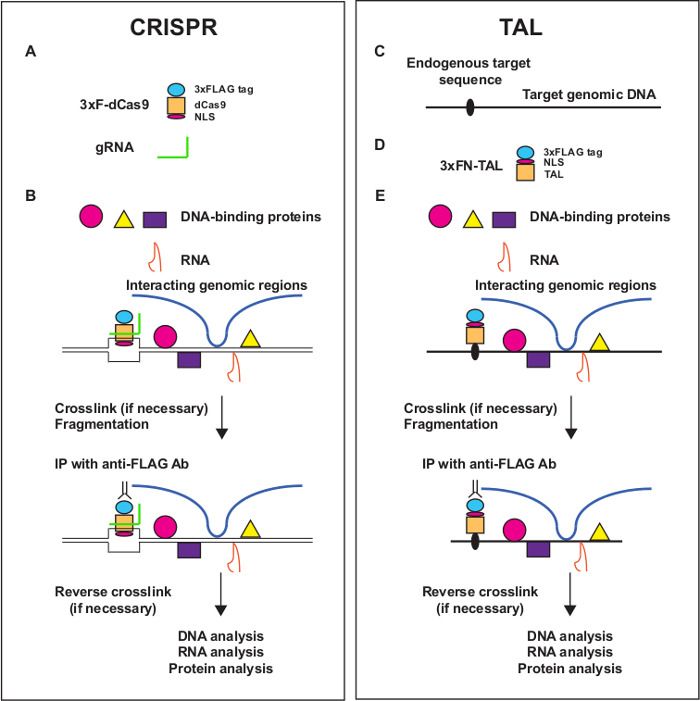
Figura 1. Visão geral do enChIP. enChIP usando CRISPR (A, B) e TAL (C, D, E ) é mostrado. Um sítio de interesse está marcado com moléculas de ligação de ADN modificadas, tais como uma proteína ou o sistema TAL CRISPR que consiste de uma forma cataliticamente inactiva de Cas9 (DCA) e a guia de ARN (gRNA). As interacções moleculares são fixadas com formaldeído ou outros agentes de reticulação, caso seja necessário. Subsequentemente, fixa cromatina é fragmentado por sonicação ou digestão enzimática. As regiões genómicas são marcados purificado por purificação de afinidade. Finalmente, a reticulação é invertida, e as moléculas interagem (regiões genômicas, RNAs e proteínas) são identificados utilizando sequenciamento próxima geração e espectrometria de massa. Por favor clique aqui para ver uma versão maior desta figura.

Figura 2. Seqüência de gBlock. O promotor U6 (em preto), o G nucleotídeo bntes a sequência de guia (em azul), a sequência de guia (gRNA espaçador) (em vermelho), a sequência de suporte (em verde), e uma sequência de terminador (em laranja) são mostrados.
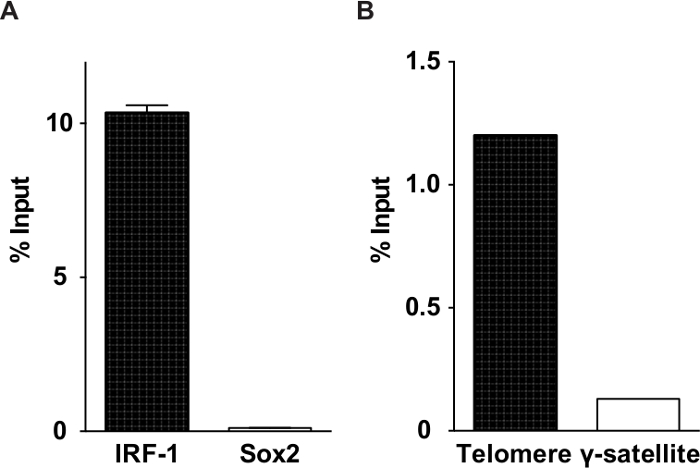
Figura 3. Rendimentos de análises enChIP representante. Insumos (A) Porcentagem para a meta de IRF-1 lócus ea Sox2 lócus não-alvo. Entradas (B) Porcentagem para os telômeros-alvo e não-alvo y-satélites. Os números foram adaptados e modificados a partir de publicações anteriores 15,16.
| Categorias | Proteínas |
| Transcrição | DDX1, PARP1, CKAP4, Pescadillo homólogo, PURβ, activado RNA polimerase II activador transcricional de p15, BTF3, Myb de ligação1A proteína |
| Desacetilação Histona, componentes corepressor | RBBP4, PA2G4, TBL3 |
| Acetiltransferase | Proteína arginina metiltransferase N-1 |
| DNA topoisomerase | DNA topoisomerase 2α |
| Histones | Histona H2A.Z, H3.2 histona |
Tabela 1:. Exemplos de proteínas associadas com o promotor de IRF-1 humano região de uma forma de IFNy específico identificado por enChIP-SILAC A tabela foi adaptada de uma publicação anterior 16.
| Categorias | Proteínas |
| Proteínas se ligam a telómeros de mamíferos | PML, RPA, CDK1, PARP1, PCBP1 |
| Telômero-binding proteínas em levedura ou outros organismos | IMP4 |
| As proteínas que interagem com o telómero de ligação proteínas [proteína de ligação dos telômeros associado] | DNA polimerase α (Pola1) [Cdc13p], ARMC6 [TRF2], CTBP1 [FoxP2-POT1], exportin-5 [terc], GNL3L [TRF1], exportin-1 [terc], 14-3-3 [terc] |
| Proteínas que localizam a heterochromatin | BEND3 |
| Proteínas reguladoras marcas epigenéticas | KDM5C |
| Proteínas cujas mutações afetam a função dos telômeros | Α DNA polimerase (Pola1), Hat1, Nup133, CDK7, DPOE1, PRDX1, TYSY, glutamato-cisteína-ligase, glutarredoxina, SMRC1 |
Tabela 2:. Exemplos de proteínas associadas com telômeros rato identificadas por enChIP-MS A tabela foi adapted de uma publicação anterior 15.
| Categorias | RNAs |
| Componentes da telomerase | Terc, rMRP |
| Telomeric RNAs | Terras |
| scaRNAs | Scarna6, Scarna10, Scarna13, Scarna2 |
| H / ACA snoRNAs | Snora23, Snora74a, Snora73b, Snora73a |
| C / D snoRNAs | Snord17, Snord15a, Snord118 |
| lncRNA | Neat1 |
Tabela 3: Exemplos de RNAs associadas com telômeros rato identificadas por enChIP-RNA-Seq A tabela foi adaptada de uma publicação anterior 17..
Discussão
Here, we describe the purification of specific genomic regions using engineered DNA-binding molecules such as the CRISPR system and TAL proteins, and the identification of proteins and RNAs bound to these genomic regions. Binding of engineered DNA-binding molecules to the genome may affect chromatin structure, including nucleosome positioning, and may abrogate genomic functions, as described in CRISPR interference experiments21. To avoid these potential aberrant effects, we propose specific guidelines for choosing target genomic regions. First, to avoid potential inhibition of the recruitment of RNA polymerases and transcription factors, as well as disruption of nucleosome positioning around the transcription start site, the target regions for analyses of promoter regions should be several hundred base pairs upstream of (5' to) the transcription start site. By contrast, when analyzing genomic regions with distinct boundaries, such as enhancers and silencers, genomic regions that are directly juxtaposed to these regions can be targeted because it is less likely that the binding of engineered DNA-binding molecules will affect their functions. Furthermore, it is best to avoid using target regions that are conserved among different species, because important DNA-binding molecules often bind to evolutionarily conserved regions and inhibition of their binding might disrupt the functions of the target genomic regions. In this regard, it is always necessary to check that the function of the target genomic region is maintained in the established cells used for enChIP analyses. Because multiple gRNAs can be tested easily and it is tedious and expensive to generate multiple versions of TAL or zinc-finger proteins recognizing different target genomic regions, enChIP using CRISPR is more advantageous than enChIP using other proteins.
It has been shown that dCas9 binds to off-target sites although affinity to those sites might be weaker than that to the target sites22-25. There are several ways to manage contamination of molecules bound to those off-target sites. First, the use of several, at least two, different gRNAs would be recommended. Those molecules commonly observed in enChIP using distinct gRNAs would be true positives. Second, comparison of different conditions for enChIP would be effective in cancelling contamination of non-specific molecules and molecules bound to off-target sites. Examples of those comparison sets would be (i) stimulation (-) and (+), or (ii) different cell types such as T cells vs. B cells. Finally, quantitative analysis of binding of candidate molecules should be performed to confirm their specific binding to the target sites. It is preferable to prepare cells expressing only dCas9 but not gRNA as a negative control.
Using enChIP analyses, we were able to successfully identify a number of known and novel molecules interacting with specific genomic regions (Tables 1-3)14-17. However, this technique failed to detect some other known proteins interacting with these regions. For example, STAT1 reportedly associates with the IRF-1 promoter upon IFNγ stimulation8, but our enChIP-SILAC analysis did not detect STAT1 as a protein induced to interact with this genomic region16. In addition, in the enChIP-MS analysis of telomeres, we did not detect shelterin proteins consisting of TRF-1 and TRF-215, which have been shown to interact with telomeres26. There are a few potential reasons for these discrepancies. First, the stoichiometry of binding of Stat1 to the IRF-1 promoter might be very low. It is reasonable that enChIP-MS, including enChIP-SILAC, detects proteins that are more abundantly associated with target genomic regions; hence, the analysis of more cells might be necessary to detect these proteins. Increases in the sensitivities of MS instruments would also contribute to the efficient detection of proteins with low stoichiometric binding. Second, some proteins, possibly including Stat1 and shelterins, might be difficult targets for MS analyses. Third, in our analysis of telomere-binding proteins15, the 3×FLAG-TAL proteins recognizing telomeres (3×FN-Tel-TAL) might have blocked the binding of shelterins to telomeres in a competitive fashion.
In contrast to the relative difficulty of detecting transcription factors binding to specific genomic regions using enChIP, we successfully identified epigenetic regulators such as histone modification enzymes using enChIP analyses. The success of this technique may be due to the fact that epigenetic regulators bind to a broad range of genomic regions; hence, more proteins per genomic region are available for MS. Because epigenetic regulators are increasingly recognized as important targets for drugs against intractable diseases such as cancer, enChIP would be a useful tool for the identification of epigenetic drug targets.
Divulgações
The authors have filed a patent on enChIP (Patent name: 'Method for isolating specific genomic regions using DNA-binding molecules recognizing endogenous DNA sequences'; Patent number: PCT/JP2013/74107). H.F. is a member of the Advisory Board of Addgene.
Agradecimentos
Este trabalho foi apoiado pela Fundação para a Ciência Takeda (TF); a Fundação Asahi Glass; o Memorial Foundation Uehara (HF); o Kurata Memorial Hitachi Ciência e Fundação de Tecnologia (FT e IC); Conceda-in-Aid para Jovens Cientistas (B) (# 25830131), Grant-in-Aid para a Investigação Científica (C) (# 15K06895) (TF); e um Grant-in-Aid para a Investigação Científica em 'Ciclo de transcrição' áreas inovadoras (# 25118512 & # 15H01354), Grant-in-Aid para a Investigação Científica (B) (# 15H04329), Grant-in-Aid para a Investigação exploratória ( # 26650059) e 'Genome Suporte' (# 221S0002) (HF) do Ministério da Educação, Cultura, Desporto, Ciência e Tecnologia do Japão.
Materiais
| Name | Company | Catalog Number | Comments |
| gBlock synthesis service | Life Technologies | Gene Synthesis by GeneArt | |
| gBlock synthesis service | IDT (Integrated DNA Technologies) | gBlocks Gene Fragments | |
| pSIR-neo | Addgene | 51128 | |
| pSIR-GFP | Addgene | 51134 | |
| pSIR-DsRed-Express2 | Addgene | 51135 | |
| pSIR-hCD2 | Addgene | 51143 | |
| TAL synthesis service | Life Technologies | GeneArt Precision TALs | |
| 3xFLAG-dCas9/pCMV-7.1 | Addgene | 47948 | |
| 3×FLAG-dCas9/pMXs-puro | Addgene | 51240 | |
| 3×FLAG-dCas9/pMXs-IG | Addgene | 51258 | |
| 3×FLAG-dCas9/pMXs-I2 | Addgene | 51259 | |
| 3×FLAG-dCas9/pMXs-neo | Addgene | 51260 | |
| anti-FLAG M2 Ab | Sigma-Aldrich | F1804 | |
| FITC-conjugated anti-FLAG M2 | Sigma-Aldrich | F4049 | |
| DMEM medium for SILAC | Life Technologies | 89985 | Other medium can be purchased from Life Technologies |
| Dialyzed FBS for SILAC | Life Technologies | 89986 | |
| L-Lysine-2HCl for SILAC | Life Technologies | 89987 | For Light medium |
| L-Arginine-HCl for SILAC | Life Technologies | 89989 | For Light medium |
| L-Lysine-2HCl, 13C6 for SILAC | Life Technologies | 89988 | For Heavy medium |
| L-Arginine-HCl, 13C6, 15N4 for SILAC | Life Technologies | 89990 | For Heavy medium |
| Complete, mini, EDTA-free | Roche Diagnostics | 4693159 | |
| Ultrasonic Disruptor UD-201 | Tomy Seiko | ||
| ChIP DNA Clean & Concentrator | Zymo Research | D5205 | |
| Dynabeads-Protein G | Life Technologies | DB10004 | |
| RNasin Plus RNase Inhibitor | Promega | N2611 | |
| Isogen II | Nippon Gene | 311-07361 | |
| Direct-zol RNA Miniprep kit | Zymo Research | R2050 | |
| LTQ Orbitrap Velos | Thermo Fisher Scientific | A component of a nanoLC-MS/MS system for MS analysis | |
| nanoLC | Advance, Michrom Bioresources | A component of a nanoLC-MS/MS system for MS analysis | |
| HTC-PAL autosampler | CTC Analytics | A component of a nanoLC-MS/MS system for MS analysis |
Referências
- Zhang, X. Y., Horz, W. Analysis of highly purified satellite DNA containing chromatin from the mouse. Nucleic Acids Res. 10, 1481-1494 (1982).
- Workman, J. L., Langmore, J. P. Nucleoprotein hybridization: a method for isolating specific genes as high molecular weight chromatin. Biochemstry. 24, 7486-7497 (1985).
- Boffa, L. C., Carpaneto, E. M., Allfrey, V. G. Isolation of active genes containing CAG repeats by DNA strand invasion by a peptide nucleic acid. Proc. Natl. Acad. Sci. USA. 92, 1901-1905 (1995).
- Jaskinskas, A., Hamkalo, B. A. Purification and initial characterization of primate satellite chromatin. Chromosome Res. 7, 341-354 (1999).
- Griesenbeck, J., Boeger, H., Strattan, J. S., Kornberg, R. D. Affinity purification of specific chromatin segments from chromosomal loci in yeast. Mol. Cell Biol. 23, 9275-9282 (2003).
- Ghirlando, R., Felsenfeld, G. Hydrodynamic studies on defined heterochromatin fragments support a 30-µm fiber having six nucleosomes per turn. J. Mol. Biol. 376, 1417-1425 (2008).
- Déjardin, J., Kingston, R. E. Purification of proteins associated with specific genomic loci. Cell. 136, 175-186 (2009).
- Hoshino, A., Fujii, H. Insertional chromatin immunoprecipitation: a method for isolating specific genomic regions. J. Biosci. Bioeng. 108, 446-449 (2009).
- Fujita, T., Fujii, H. Direct idenification of insulator components by insertional chromatin immunoprecipitation. PLoS One. 6, e26109(2011).
- Fujita, T., Fujii, H. Efficient isolation of specific genomic regions by insertional chromatin immunoprecipitation (iChIP) with a second-generation tagged LexA DNA-binding domain. Adv. Biosci. Biotechnol. 3, 626-629 (2012).
- Fujita, T., Fujii, H. Locus-specific biochemical epigenetics / chromatin biochemistry by insertional chromatin immunoprecipitation. ISRN Biochem. 2013, 913273(2013).
- Fujita, T., Fujii, H. Efficient isolation of specific genomic regions retaining molecular interactions by the iChIP system using recombinant exogenous DNA-binding proteins. BMC Mol. Biol. 15, 26(2014).
- Fujita, T., Kitaura, F., Fujii, H. A critical role of the Thy28-MYH9 axis in B cell-specific expression of the Pax5 gene in chicken B cells. PLoS One. 10, (2015).
- Fujita, T., Fujii, H. Efficient isolation of specific genomic regions and identification of associated proteins by engineered DNA-binding molecule-mediated chromatin immunoprecipitation (enChIP) using CRISPR. Biochem. Biophys. Res. Commun. 439, 132-136 (2013).
- Fujita, T., et al. Identification of telomere-associated molecules by engineered DNA-binding molecule-mediated chromatin immunoprecipitation (enChIP). Sci. Rep. 3, 3171(2013).
- Fujita, T., Fujii, H. Identification of proteins associated with an IFNγ-responsive promoter by a retroviral expression system for enChIP using CRISPR. PLoS One. 9, e103084(2014).
- Fujita, T., Yuno, M., Okuzaki, D., Ohki, R., Fujii, H. Identification of non-coding RNAs associated with telomeres using a combination of enChIP and RNA sequencing. PLoS One. 10, e0123387(2015).
- Naito, Y., Hino, K., Bono, H., Ui-Tei, K. CRISPRdirect: software for designing CRISPR/Cas guide RNA with reduced off-target sites. Bioinformatics. , (2014).
- Mali, P., et al. RNA-guided human genome engineering via Cas9. Science. 339, 823-826 (2013).
- Esvelt, K. M., et al. Orthogonal Cas9 proteins for RNA-guided gene regulation and editing. Nat. Methods. 10, 1116-1121 (2013).
- Qi, L. S., et al. Repurposing CRISPR as an RNA-guided platform for sequence-specific control of gene expression. Cell. 152, 1173-1183 (2013).
- Wu, X., et al. Genome-wide binding of the CRISPR endonuclease Cas9 in mammalian cells. Nat. Biotechnol. 32, 670-676 (2014).
- Kuscu, C., Arslan, S., Singh, R., Thorpe, J., Adli, M. Genome-wide analysis reveals characteristics of off-target sites bound by the Cas9 endonuclease. Nat Biotechnol. 32, 677-683 (2014).
- Cencic, R., et al. Protospacer adjacent motif (PAM)-distal sequences engage CRISPR Cas9 DNA target cleavage. PLoS One. 9, 109213(2014).
- O'Green, H., Henry, I. M., Bhakta, M. S., Meckler, J. F., Segal, D. J. A genome-wide analysis of Cas9 binding specificity using ChIP-seq and targeted sequence capture. Nucleic Acids Res. 43, 3389-3404 (2015).
- de Lange, T. Shelterin: the protein complex that shapes and safeguards human telomeres. Genes Dev. 19, 2100-2110 (2005).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados