Method Article
특정 게놈 지역과 enChIP으로 관련 분자의 식별의 분리
요약
The identification of molecules associated with specific genomic regions of interest is required to understand the mechanisms of regulation of the functions of these regions. This protocol describes procedures to perform engineered DNA-binding molecule-mediated chromatin imunoprecipitation (enChIP) for identification of proteins and RNAs associated with a specific genomic region.
초록
The identification of molecules associated with specific genomic regions of interest is required to understand the mechanisms of regulation of the functions of these regions. To enable the non-biased identification of molecules interacting with a specific genomic region of interest, we recently developed the engineered DNA-binding molecule-mediated chromatin immunoprecipitation (enChIP) technique. Here, we describe how to use enChIP to isolate specific genomic regions and identify the associated proteins and RNAs. First, a genomic region of interest is tagged with a transcription activator-like (TAL) protein or a clustered regularly interspaced short palindromic repeats (CRISPR) complex consisting of a catalytically inactive form of Cas9 and a guide RNA. Subsequently, the chromatin is crosslinked and fragmented by sonication. The tagged locus is then immunoprecipitated and the crosslinking is reversed. Finally, the proteins or RNAs that are associated with the isolated chromatin are subjected to mass spectrometric or RNA sequencing analyses, respectively. This approach allows the successful identification of proteins and RNAs associated with a genomic region of interest.
서문
관심의 특정 게놈 영역과 관련된 분자의 동정은 이러한 전사 및 후생 유전 학적 조절 게놈 기능 조절 메커니즘을 이해하기 위해 필요하다. 여러 기술들이 특정 게놈 영역 1-7의 생화학 적 분석을 위해 개발되었지만, 이들은 때문에 이러한 제한된 애플리케이션 (예에만 반복으로 높은 카피 수 궤적 또는 유전자좌에 대한)과 같은 그들의 본질적인 문제로,이 단계에서 널리 사용되지 너무 많은 시간과 노력이 필요합니다.
쉽게 특정 게놈 영역의 생화학 적 분석을 수행하기 위해, 우리는 14-17 개의 궤적 특정 염색질 면역 (칩) 기술, 즉 삽입 성 온칩 (iChIP) 8-13 및 설계 DNA 결합 분자 매개 칩 (enChIP)을 개발했다 . iChIP에서 관심의 궤적이 같은 렉스와 같은 외래 DNA 결합 단백질의 삽입 인식 서열에 의해 태그가A. 궤적은 다음 태그 DNA 결합 단백질을 이용하여 친 화성 정제에 의해 격리됩니다. enChIP에서, 아연 손가락 단백질, 전사 활성제와 같은 (TAL) 단백질과 같은 DNA 결합 분자를 설계하고, 클러스터 정기적으로 interspaced 짧은 회문 반복 (CRISPR) 단지는 (그림 1) 관심의 궤적을 태그하는 데 사용됩니다. 그 후, 게놈 영역은 태그 DNA 결합 분자의 친 화성 정제에 의해 격리됩니다.
iChIP 위에 enChIP의 장점 중 하나는 외래 DNA 결합 단백질의 인식 서열의 삽입이 필요하지 않다는 것이다. Cas9 (dCas9)의 촉매 비활성 형태로 구성된 CRISPR 건물 및 가이드 RNA (gRNA)를 사용하여 궤적의 타겟팅 TAL 아연 손가락 단백질을 사용하여 iChIP 또는 enChIP에 의해이 지역의 대상보다 훨씬 쉽다. 여기서, 우리는 질량 분석 및 RNA 서열과 결합 enChIP 단계별 프로토콜을 서술 (RNA-SEQ)는 궤적-소시아를 식별테드 단백질과 RNA를 각각.
프로토콜
대상 궤적을 인식 엔지니어링 DNA 결합 분자의 1. 디자인
- CRISPR 착체를 사용 enChIP 들어, 앞서 설명한 바와 같이 18 관심 게놈 영역 후보 gRNA 표적 서열을 확인하기 CRISPRdirect 웹 도구 (http://crispr.dbcls.jp)를 사용한다. 이 웹 도구는 형태의 23 bp의 게놈 사이트를 반환 5'-N (20) NGG-3 '대상 지역 내.
- U6 프로모터 서열 및 5'-N (20)의 시퀀스를 사용하여 상용 서비스 5'-N 20 NGG-3 '을 포함 gBlock 합성 19, 20 (도 2 참조). 목적을 서브 클로닝, gBlock 외부 적당한 제한 효소 부위를 포함한다.
- 이전 16 설명 된대로 적절한 벡터에 gBlock를 삽입합니다. gBlocks에 대한 몇 가지 레트로 바이러스 벡터는 (자료 참조)을 사용할 수 있습니다.
- TAL 단백질을 사용 enChIP 들어 TARG 인식 TAL 단백질 설계등의 궤적. 상용 서비스를 사용 TAL 단백질을 암호화하는 플라스미드를 생성합니다. 앞서 설명한 바와 같은 15 × 3의 FLAG와 같은 하나 또는 그 이상의 태그들과 융합 TAL 단백질을 포함하는 발현 벡터를 생성한다.
enChIP 분석을위한 세포의 2. 설립
- 3 × FLAG-dCas9 및 gRNA (CRISPR 기반 절차) 또는 익스프레스 3 이전에 14 ~ 17 설명 된대로 × FLAG-TAL 세포 (TAL 단백질 기반의 절차가) 분석합니다.
- 형질 전환 효율이 높으면 일시적인 형질 감염을 사용한다. 3 × FLAG-dCas9 및 CMV 프로모터를 포함하는 발현 벡터 (자료 참조)를 사용할 수 있습니다.
- 일시적 트랜 스펙 션 효율이 낮은 경우에는 통상적 인 방법을 사용하여 안정한 형질 전환을 확립 고려한다. gBlock에 대한 상기 레트로 바이러스 벡터에 추가하여, 3 × FLAG-dCas9의 레트로 바이러스 벡터는 (자료 참조)을 사용할 수 있습니다.
- 앞서 설명 된 바와 같이 14-17 항 FLAG 항체 (AB)를 사용하여 면역 블롯 분석에 의해 형질 감염된 세포에서 3xFLAG-dCas9 또는 3xFLAG-TAL 단백질의 발현을 확인한다.
주 :이 태그 된 단백질의 발현과 같은 항 - FLAG-FITC 및 후속 FACS 분석으로 세포 염색에 의해 확인 될 수있다. 세포 내 염색을위한 프로토콜은 홈페이지 (http://www.biken.osaka-u.ac.jp/lab/microimm/fujii/iChIP_protocols/english.html)에서 다운로드 할 수 있습니다. - 표준 RT-PCR 기술에 의해 gRNA의 발현을 확인합니다.
- 관심의 게놈 영역과 상호 작용하는 단백질의 정량 확인을 위해, 세포 배양 (SILAC) enChIP (enChIP-SILAC)와 결합 분석에 아미노산에 의해 안정 동위 원소 표지의 사용을 고려한다.
참고 : SILAC 분석을위한 미디어는 상용 공급 업체에서 구입하실 수 있습니다. SILAC 특정 상호 작용을 검출하는 강력한입니다.- SILAC 무거운 배지에서 배양 세포. 세포 cultu 준비음성 대조군으로 SILAC 광 매체에 빨간색. (무거운 또는 빛) 각 SILAC 매체 적어도 5 × 10 7 세포를 사용합니다. 효율적인 라벨은 모두 L- 라이신 - 2HCl, 13 C 6, L 아르기닌 - 염산, SILAC 중공업 미디어 13 C 6, 15 N 4를 추가합니다. 참고 : 적어도 5 세포 분열이 라벨 단백질이 필요하다.
- 예를 들어, 안정적 라이신 2HCl 플러스 L- 아르기닌 - 염산 (라이트 배지) SILAC위한 DMEM 배지 및 투석 된 소 태아 혈청에서 37 ° C, 5 % CO 2에서 3xFLAG-dCas9 및 gRNA 발현 배양 인간 섬유 육종 HT1080 세포 또는 13 C 6 L- 라이신 - 2HCl 플러스 13 C 6 15 N 4 L 아르기닌 염산 (헤비 매체) (16) (자료 참조). 매체의 500 ml의 빛 또는 무거운 L- 라이신 - 2HCl과 L- 아르기닌 염산 50 mg을 추가합니다.
- 세포가 지수 growt을 유지하도록를 Trypsinize 및 replate 세포는 그들이 합류되기 전에시간.
- SILAC 무거운 배지에서 배양 세포. 세포 cultu 준비음성 대조군으로 SILAC 광 매체에 빨간색. (무거운 또는 빛) 각 SILAC 매체 적어도 5 × 10 7 세포를 사용합니다. 효율적인 라벨은 모두 L- 라이신 - 2HCl, 13 C 6, L 아르기닌 - 염산, SILAC 중공업 미디어 13 C 6, 15 N 4를 추가합니다. 참고 : 적어도 5 세포 분열이 라벨 단백질이 필요하다.
포름 알데히드와 세포의 3. 가교
- 현탁 배양 세포에서, 전사 2 × 107 3xFLAG-TAL 단백질 또는 3xFLAG-dCas9 플러스 gRNA을 발현하는 세포와 50 mL의 원심 분리 튜브에 일정한 배지 30 ㎖에 서스펜드. 이상의 셀이 사용될 때, 비례 시약의 양 및 양을 증가시킨다. SILAC 실험에, 무거운 매체 또는 광 매체 (5 × 107 세포 당)에서 배양 세포를 혼합하여 2 × 107 세포를 함유하는 튜브 (5)에 1 × 108 세포의 총 나눈다.
- 세포 현탁액 (1 % 최종 농도)의 30 mL의 37 % 포름 알데히드의 810 μl를 첨가하고 5 분 동안 37 ℃에서 배양한다. 부착 세포에 직접 배지 30 ㎖에 37 % 포름 알데히드 810 μl를 첨가하여 배양 접시에 세포를 고정한다.
- 1.25 M 글리신 용액 (최종 농도 127 mM)을 3.1 mL를 첨가하여 가교를 중지실온에서 10 분 동안 품어.
- 원심 분리 (4 ℃에서 5 분 300 ×의 G)에 의해 세포를 수집합니다. 조심스럽게 상층 액을 포함하여 포름 알데히드를 무시하고, 적절한 폐기 용기에 보관하십시오.
- 부착 세포의 경우, 50 ㎖ 튜브에서 셀 스크레이퍼로 세포를 수확 분리하고,이 단계에 기재된 바와 같이 세포를 수집한다.
- SILAC 실험에, 무거운 매체 또는 광 매체 (5 × 107 세포 당)로 배양 분리 세포를 혼합 2 × 107 세포를 함유하는 5 튜브에 1 × 108 세포의 전체를 분할하고, 이것에 기재된 바와 같이 세포를 수집 단계.
- 튜브에 두 번 PBS의 30 mL를 세포 펠렛을 씻으십시오. 조심스럽게 포름 알데히드를 포함하여 상층 액을 제거하고 화학 물질 안전 지침에 따라 포름 알데히드 폐기물 bottle.Handle 적절한 폐기에 저장합니다. 고정 된 세포는 동결 및 -80 ℃에서 저장 될 수있다.
염색질 4. 준비 (당 2 × 107 세포)
- 세포 용해 완충 용액 10 ㎖ (10 mM 트리스 (PH 8.0), 1 mM의 EDTA, 0.5 % IGEPAL의 CA-630, 그리고 1 × 프로테아제 억제제)에 고정 된 세포를 중단하고 10 분 동안 얼음 위에서 배양한다.
- 원심 분리기 샘플 (8 분 4 ° C에서 830 ×의 g)을 조심스럽게 상층 액을 버린다.
- 핵 용해 완충액 10ml에 펠릿을 정지 (10 mM 트리스 (PH 8.0), 1 mM의 EDTA, 0.5 M의 NaCl, 1 % 트리톤 X-100, 0.5 % 나트륨 데 옥시 콜레이트, 0.5 % lauroylsarcosine 및 1X 프로테아제 억제제). 10 분 동안 얼음에 품어 모든 2 ~ 3 분 소용돌이.
- (830 ×을 샘플을 원심 분리기 8 분 4 ° C에서 g)을 조심스럽게 상층 액을 버린다.
- PBS 10ml에 펠렛을 씻으십시오. 펠렛 (염색질 분획)을 액체 질소에서 즉시 냉동 후 -80 ℃에서 저장 될 수있다.
염색질 5. 초음파 처리 (2 × 10 7 세포 당)
- 800 μL의 염색질 부분을 일시 중단변성 용해 완충액 3 (10 mM 트리스 (PH 8.0), 1 mM의 EDTA, 150 mM의 염화나트륨, 0.1 % 나트륨 데 옥시 콜레이트, 0.1 % SDS, 및 1X 프로테아제 억제제) 및 1.5 ㎖의 튜브에 옮긴다.
- 출력 : (원료 참조) 초음파기를 이용하여 염색질 및 다음의 조건 (3)을 초음파 처리; 의무 : 100 % (연속) 시간 : 무료. 10 초 동안 초음파의 10-18 사이클을 수행하고 20 초 동안 얼음에 냉각. 과도한 난방을 피하기 2 분마다 5 ~ 6주기 동안 얼음에 샘플을 품어. 발포 방지하기 위해, 0.5 cm 튜브 바닥 상기 초음파 프로브의 선단의 위치를 유지한다.
- 원심 분리기에서 샘플 (10 분 동안 4 ℃에서 16,000 ×의 g) 및은 1.5 ML 튜브에 뜨는을 전송합니다. 초음파 염색질은 액체 질소에서 즉시 냉동 후 -80 ℃에서 저장 될 수있다.
염색질 조각화 6. 평가
- 증류수 85 μL와 조각난 염색질의 10 μl를 섞는다.
- 4 μl를 추가5 M의 NaCl 65 ° CO / N에 품어.
- 10 ㎎ / ㎖의 RNase의 1 μl를 추가하고 45 분 동안 37 ° C에서 품어.
- 의 0.5 M EDTA (pH 8.0), 1 M 트리스 (PH 6.8)의 4 μL, 20 mg을 1 μL / ㎖ 테 K 2 μl를 추가하고 1.5 시간 동안 45 ℃에서 배양한다.
- 이러한 SYBR 그린과 같은 염색 염료를 포함하지 않는 1 % 아가 로스 겔에서 전기 영동에 의해 분리 된 샘플 10 μL.
- 단편화 된 염색질의 길이의 분포를 평가하는 겔을 염색. 0.5 KBP (0.2-4 KBP의 범위)의 평균 길이를 생성 조건이 좋습니다.
- 페놀 - 클로로포름 추출에 의해 또는 DNA 추출 키트 (자료를 참조)를 사용하여 나머지 샘플로부터 DNA를 정제 하였다. 정제 된 DNA는 enChIP 분석의 수율 (8.11 참조)를 추정하기 위해 입력 DNA로 사용할 수있다.
항체와 Dynabeads 공역 7. 준비 (2 × 10 7 세포 당)
- 사전두 개의 1.5 ml의 튜브, 정상 마우스의 IgG를 사용하여 미리 청소 하나는 안티 - 플래그 Ab의와 배양을위한 다른 깎다. 각각의 튜브에 단백질 G - 복합 자석 구슬 150 μl를 (자료 참조)를 추가합니다.
- 자석 스탠드에 튜브를 넣고 3 분을 기다립니다. 피펫으로 상층 액을 버린다.
- PBS는 0.01 % 트윈 20 (PBS-T)를 함유하는 1 ㎖의 비드를 중단. 자기 스탠드에 튜브를 넣고 2 분 동안 기다립니다. 피펫으로 상층 액을 버리고, 다음 단계를 반복합니다.
- 0.1 % BSA를 포함하는 PBS-T에 1 ㎖ 구슬을 정지.
- 정상 마우스의 IgG 또는 안티 - 플래그 Ab의 15 μg의 추가 4 ° CO / N로 회전.
- 다음, 간단히 스핀 다운 자석 스탠드에 튜브를 넣고 3 분을 기다립니다. 피펫으로 상층 액을 버린다.
- PBS-T에 1 ㎖ 구슬을 정지. 샘플을 여러 번 전환 간단히 스핀 다운. 다음 피펫으로 상층 액을 버리고, 자석 스탠드에 튜브를 넣고 3 분을 기다립니다. 세탁을 반복PBS-T (세 세척 단계의 총)을 두 번 더.
8. 염색질 면역 침전 (2 × 10 7 세포 당)
- 부피의 1/5 5 % 트리톤 X-100 (1 % 트리톤 X-100의 최종 농도)를 함유하는 개질 된 용해 완충액 (3)의 (약 200 μL)에 5.3에서 제조 단편화 염색질) (약 800 μL)을 혼합한다.
- 일반 마우스 IgG가 결합 단백질 G - 복합 자석 구슬을 준비했다있는 튜브로 염색질 솔루션을 추가합니다. 1 시간 동안 4 ° C에서 회전합니다.
- 자석 스탠드에 튜브를 넣고 3 분을 기다립니다.
- 안티 - 플래그 Ab의와 결합 단백질 G - 복합 자석 구슬을 준비했다있는 튜브에 뜨는을 전송합니다. 4 ° CO / N로 돌립니다.
- 자석 스탠드에 튜브를 넣고 3 분을 기다립니다. 피펫으로 상층 액을 버린다.
- 저염 완충액 (20 mM 트리스 (PH 8.0), 2 mM의 EDTA, 150 mM의 염화나트륨, 1 % 트라이 1 ㎖에 구슬을 정지톤 X-100, 0.1 % SDS, 및 1X 프로테아제 억제제)과 5 분 동안 4 ℃에서 회전한다. 자석 스탠드에 튜브를 넣고 3 분을 기다립니다. 피펫으로 상층 액을 제거하고 세척 단계를 반복합니다.
- 두 배 높은 소금 버퍼 (20 mM 트리스 (PH 8.0), 2 mM의 EDTA, 500 mM의 NaCl을, 1 % 트리톤 X-100, 0.1 % SDS, 및 1X 프로테아제 억제제)와 구슬을 씻으십시오.
- 두번의 LiCl 완충액 (10 mM 트리스 (PH 8.0), 1 mM의 EDTA, 250 mM의 LiCl을, 0.5 % IGEPAL CA-630을, 0.5 % 나트륨 데 옥시 콜레이트 및 1X 프로테아제 억제제)과 비드를 세척 하였다.
- 한 번 TBS-IGEPAL CA-630 (50 mM 트리스 (PH 7.5), 150 mM의 NaCl을, 0.1 % IGEPAL의 CA-630, 및 1X 프로테아제 억제제)와 구슬을 씻으십시오.
- 용출 완충액 200 μL의 구슬을 정지 (50 mM 트리스 (PH 7.5), 150 mM의 염화나트륨, 0.1 % IGEPAL의 CA-630, 1X 프로테아제 억제제, 및 500 μg의 / ㎖ × 3 FLAG 펩티드)과는 37 ° C에서 배양 20 분. 자석 스탠드에 튜브를 넣고 3 분을 기다립니다. 새로운 1.5 ML 튜브에 뜨는을 전송하고 엘 루트를 반복이온 공정.
- 페놀 - 클로로포름 추출에 의해 또는 DNA 추출 키트를 사용하여 용출의 DNA를 작은 부분에서 (예를 들면, 5 %)로 정제 (자료를 참조). 정제 된 DNA는 특정 프라이머 enChIP는 이전에 설명한 바와 같이 단계 (14, 16)에서 제조 한 6.7 입력 된 DNA와 비교하여 분석 수율을 추정하는 설정으로 PCR에 사용될 수있다.
9. SDS-PAGE, 염색 및 질량 분석
- 1 2- 프로판올 용액, 3M 아세트산 나트륨 (PH 5.2) 50 μL, 20 ㎎ / ㎖의 5 μL의 글리코겐과 용출 (400 μL)를 혼합한다. -20 ° CO / N에서 염색질을 침전.
- 원심 분리기 샘플 (30 분 4 ℃에서 16,000 ×의 g) 및 상층 액을 버린다. (10 분간 4 ℃에서 16,000 ×의 g)을 70 % 에탄올과 원심 분리기 다시 1 ㎖와 펠렛을 씻어. 피펫에 의해 완전히 상층 액을 버린다.
- 배의 샘플 버퍼 (125 mM 트리스 (PH 6.8), 10 % (2) - 용병의 40 μL에 펠렛을 일시 중단aptoethanol, 4 % SDS, 10 % 수 크로즈, 및 0.004 % 브로 모 페놀 블루). 5 분 동안 소용돌이를 완전히 펠렛을 용해하고 단백질을 변성하고, 가교를 반대로 30 분 동안 100 ° C에서 배양.
- 염료가 잘 1cm 이하에 도달 할 때까지에 대해 SDS-PAGE, 겔 시료 40 μL를 실행. 참고 : 보통 4~20% 구배 겔을 사용하지만, 다른 % 겔이 사용될 수있다.
- 쿠마 브릴리언트 블루 또는 실버 얼룩 젤 얼룩.
- 다섯 개 (2 mm 높이)에 젤을 잘라.
- 앞서 설명한 바와 같이 13-16 겔 소화 질량 분석에서 수행한다.
- 5 × 10 7 세포 각 (1 × 10 8 세포의 총)와 SILAC 실험의 경우, 2 배 샘플 버퍼의 40 μL의 초기 다섯 튜브 (그것을 확장 할 필요가 없습니다)에서 펠렛을 일시 중지합니다.
RNA와 RNA-서열 분석 (10) 정제
- enChIP 후 RNA를 정화하기 위해,의 RNase 억제제의 5 U / ㎖를 추가 (SE완충 용액의 모든 전자 물질), 변성 용해 버퍼 (3) 및 용출 완충액의 RNase 억제제를 제외하는 40 U / ㎖를 추가한다.
- 5 M NaCl을 16 μL로 용출액 (400 μL)를 혼합하고 2 시간 동안 65 ° C에서 품어.
- 샘플 산 - 구 아니 디늄 - 페놀 계 시약 1 ㎖를 (자료 참조)를 추가합니다. 다음 15 초 동안 소용돌이와는 5 ~ 15 분 동안 실온에서 품어. 12,000 × g에서 및 RT에서 15 분 동안 샘플을 원심 분리기.
- 새로운 1.5 ML 튜브에 뜨는을 전송하고 P-bromoanisole의 5 μl를 추가합니다. 다음 15 초 동안 소용돌이와는 3 ~ 5 분 동안 실온에서 품어. 12,000 × g에서 및 RT에서 10 분 동안 샘플을 원심 분리기.
- 새로운 2 ㎖의 튜브에 상청 (약 1ml)에 전송하고, 2- 프로판올 1 ㎖를 추가한다. 튜브를 전환하고 10 분 동안 RT에서 배양한다. (자료 참조) RNA 정제 키트의 컬럼에 혼합물을로드합니다. 12,000 ×에서 1 분 동안 열을 원심 분리기 g 및 RT.
- 빨래RNA 정제 키트에서 RNA 세척 버퍼 400 μL (자료 참조) 열입니다.
- DNase의 I (1 U / μL), 10 ×의 DNase I 반응 버퍼의 8 μL, DNase의 /의 RNase없는 물 3 μL,와 RNA 세척 버퍼의 64 μL의 5 μL의 DNase의 나는 칵테일의 80 μl를 추가 (혼합물 컬럼 RNA 정제 키트 (자재) 참조). 15 분 동안 37 ℃에서 샘플을 인큐베이션 한 후 12,000 × g 및 RT에서 30 초간 원심 분리기.
- 두 번 RNA 정제 키트에서 RNA 예비 세탁 버퍼 400 μL (자료 참조) 컬럼을 씻으십시오.
- DNase의 /의 RNA 분해 효소가없는 물을 50 μL와 RNA를 용출시킨다. 용출 된 RNA는 RNA 서열에 대해 사용될 수있다.
결과
전반적으로, 대상 게놈 영역 1-30 %의. enChIP을 사용하여 정제 3 enChIP의 수율을 보여주는 대표적인 데이터를 포함도 할 수는 텔로미어와 인터페론 (IFN) 규제 요인 1 (IRF-1) 유전자의 프로모터를 대상으로 분석한다. 전형적인 결과, 표 1의 예로서 enChIP-SILAC 표에 의해 식별 IFNγ 특정 방식 IRF-1 프로모터와 연관 단백질 2에는, 질량 스펙트럼과 결합 enChIP 의해 식별 텔로미어 - 결합 단백질 (enChIP-MS) 그리고 표 3 enChIP-RNA-SEQ 의해 식별과 연관된 텔로미어의 RNA.
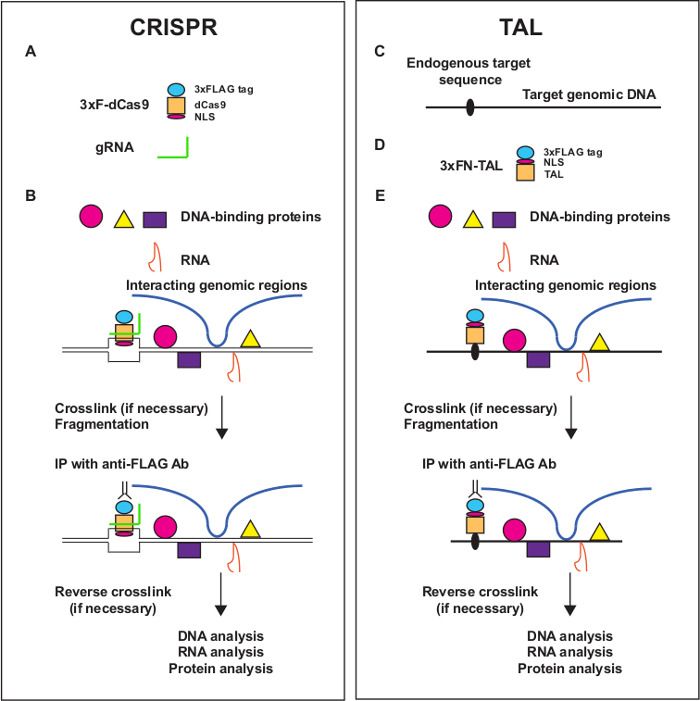
enChIP 1. 개요를 그림. enChIP 사용 CRISPR (A, B)와 TAL (C, D, E ) 표시됩니다. 관심의 궤적은 TAL 단백질 또는 Cas9 (DCAS)의 촉매 비활성 형태로 이루어진 CRISPR 시스템으로서 설계 DNA 결합 분자 태그 및 (gRNA)에 RNA를 안내한다. 분자의 상호 작용은 포름 알데히드 또는 다른 가교제, 필요하다면 고정되어 있습니다. 이어서, 고정 된 염색질은 초음파 처리 또는 효소 절단에 의해 파편화된다. 태그 게놈 영역은 친 화성 정제에 의해 정제된다. 마지막으로, 가교가 반전되고, 상호 작용하는 분자 (게놈 지역의 RNA, 단백질)이 차세대 시퀀싱 및 질량 분석기를 사용하여 식별됩니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

gBlock 그림 2. 순서. U6 프로모터 (검은 색), G 염기 B는 efore 가이드 시퀀스 (파란색), 가이드 순서 (빨간색) (gRNA 스페이서), (녹색) 비계 순서, 및 (오렌지) 터미네이터 서열이 표시됩니다.
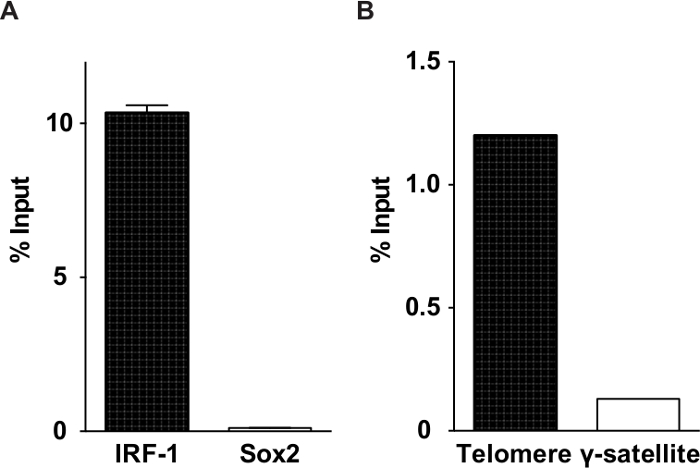
대표 enChIP 분석 3. 수익률 그림. 타겟 IRF-1 궤적과 비 표적 Sox2이 궤적에 대한 (A) 백분율 입력한다. 타겟 텔로미어 및 비 표적 γ-위성 대 (B)의 백분율 입력한다. 그림은 적응 이전 간행물 (15, 16)에서 수정되었습니다.
| 카테고리 | 단백질 |
| 전사 | DDX1, PARP1, CKAP4, Pescadillo의 동족체, PURβ, 활성화 된 RNA 중합 효소 II 전사 활성제 P15, BTF3, MYB가 결합단백질 1A |
| 히스톤 탈 아세틸 화, corepressor 구성 요소 | RBBP4, PA2G4, TBL3 |
| 아세틸 | 단백질 아르기닌 N-메틸화 된 1 |
| DNA 토포 아이소 머라 | DNA 토포 아이소 머라 제의 2α |
| 히스톤 | 히스톤 H2A.Z, 히스톤 H3.2 |
표 1. enChIP-SILAC 의해 식별 IFNγ 특정 방식으로 인간 IRF-1 프로모터 영역과 연관된 단백질의 예는 테이블 (16)로부터 이전에 발행하도록 구성되었다.
| 카테고리 | 단백질 |
| 포유류 텔로미어 결합 단백질 | PML, RPA, CDK1, PARP1, PCBP1 |
| 텔로미어-BI효모에서 찾아내는 단백질 또는 다른 생물 | IMP4 |
| 와 상호 작용하는 단백질 텔로미어가 결합 단백질 [관련 텔로미어 결합 단백질] | DNA 중합 효소 (α) (POLA1) Cdc13p] ARMC6 [(TRf2), CTBP1 [FOXP2-POT1] exportin-5 [3 급], GNL3L [TRF1] exportin -1- [TERT, 14-3-3 [TERT] |
| 이질에 현지화 단백질 | BEND3 |
| 단백질은 후생 유전 학적 마크를 조절 | KDM5C |
| 그 돌연변이 텔로미어의 기능에 영향을 미치는 단백질 | DNA 중합 효소 (α) (POLA1), HAT1, Nup133, CDK7, DPOE1, PRDX1, TYSY, 글루타민산 염 시스테인 리가, glutaredoxin, SMRC1 |
표 2. enChIP-MS에 의해 식별 마우스 텔로미어와 관련된 단백질의 예로는 표 adapte왔다이전 발행 15 D.
| 카테고리 | RNA를 |
| 텔로 머라 제의 구성 요소 | TERC, Rmrp |
| 텔로미어 RNA를 | TERRAs |
| scaRNAs | Scarna6, Scarna10, Scarna13, Scarna2 |
| H / ACA snoRNAs | Snora23, Snora74a, Snora73b, Snora73a |
| C / D snoRNAs | Snord17, Snord15a, Snord118 |
| lncRNA | Neat1 |
표 3 :. enChIP이-RNA-서열 표는 이전 발행 17에서 적응 된 식별 마우스 텔로미어와 관련된 RNA를 예.
토론
Here, we describe the purification of specific genomic regions using engineered DNA-binding molecules such as the CRISPR system and TAL proteins, and the identification of proteins and RNAs bound to these genomic regions. Binding of engineered DNA-binding molecules to the genome may affect chromatin structure, including nucleosome positioning, and may abrogate genomic functions, as described in CRISPR interference experiments21. To avoid these potential aberrant effects, we propose specific guidelines for choosing target genomic regions. First, to avoid potential inhibition of the recruitment of RNA polymerases and transcription factors, as well as disruption of nucleosome positioning around the transcription start site, the target regions for analyses of promoter regions should be several hundred base pairs upstream of (5' to) the transcription start site. By contrast, when analyzing genomic regions with distinct boundaries, such as enhancers and silencers, genomic regions that are directly juxtaposed to these regions can be targeted because it is less likely that the binding of engineered DNA-binding molecules will affect their functions. Furthermore, it is best to avoid using target regions that are conserved among different species, because important DNA-binding molecules often bind to evolutionarily conserved regions and inhibition of their binding might disrupt the functions of the target genomic regions. In this regard, it is always necessary to check that the function of the target genomic region is maintained in the established cells used for enChIP analyses. Because multiple gRNAs can be tested easily and it is tedious and expensive to generate multiple versions of TAL or zinc-finger proteins recognizing different target genomic regions, enChIP using CRISPR is more advantageous than enChIP using other proteins.
It has been shown that dCas9 binds to off-target sites although affinity to those sites might be weaker than that to the target sites22-25. There are several ways to manage contamination of molecules bound to those off-target sites. First, the use of several, at least two, different gRNAs would be recommended. Those molecules commonly observed in enChIP using distinct gRNAs would be true positives. Second, comparison of different conditions for enChIP would be effective in cancelling contamination of non-specific molecules and molecules bound to off-target sites. Examples of those comparison sets would be (i) stimulation (-) and (+), or (ii) different cell types such as T cells vs. B cells. Finally, quantitative analysis of binding of candidate molecules should be performed to confirm their specific binding to the target sites. It is preferable to prepare cells expressing only dCas9 but not gRNA as a negative control.
Using enChIP analyses, we were able to successfully identify a number of known and novel molecules interacting with specific genomic regions (Tables 1-3)14-17. However, this technique failed to detect some other known proteins interacting with these regions. For example, STAT1 reportedly associates with the IRF-1 promoter upon IFNγ stimulation8, but our enChIP-SILAC analysis did not detect STAT1 as a protein induced to interact with this genomic region16. In addition, in the enChIP-MS analysis of telomeres, we did not detect shelterin proteins consisting of TRF-1 and TRF-215, which have been shown to interact with telomeres26. There are a few potential reasons for these discrepancies. First, the stoichiometry of binding of Stat1 to the IRF-1 promoter might be very low. It is reasonable that enChIP-MS, including enChIP-SILAC, detects proteins that are more abundantly associated with target genomic regions; hence, the analysis of more cells might be necessary to detect these proteins. Increases in the sensitivities of MS instruments would also contribute to the efficient detection of proteins with low stoichiometric binding. Second, some proteins, possibly including Stat1 and shelterins, might be difficult targets for MS analyses. Third, in our analysis of telomere-binding proteins15, the 3×FLAG-TAL proteins recognizing telomeres (3×FN-Tel-TAL) might have blocked the binding of shelterins to telomeres in a competitive fashion.
In contrast to the relative difficulty of detecting transcription factors binding to specific genomic regions using enChIP, we successfully identified epigenetic regulators such as histone modification enzymes using enChIP analyses. The success of this technique may be due to the fact that epigenetic regulators bind to a broad range of genomic regions; hence, more proteins per genomic region are available for MS. Because epigenetic regulators are increasingly recognized as important targets for drugs against intractable diseases such as cancer, enChIP would be a useful tool for the identification of epigenetic drug targets.
공개
The authors have filed a patent on enChIP (Patent name: 'Method for isolating specific genomic regions using DNA-binding molecules recognizing endogenous DNA sequences'; Patent number: PCT/JP2013/74107). H.F. is a member of the Advisory Board of Addgene.
감사의 말
이 작품은 다케다 과학 재단 (TF)에 의해 지원되었다; 아사히 글라스 재단; 우에하라 기념 재단 (HF); 쿠라타 기념 히타치 과학 기술 재단 (TF 및 HF); 부여 - 에이드의 젊은 과학자를위한 (B) (# 25830131), 그랜트 - 에이드의 과학 연구 (C) (# 15K06895) (TF)에 대한; 및 보조금 에이드의 혁신 분야 '전사주기'에 대한 과학 연구를위한 (# 25118512 & # 15H01354), 그랜트 - 에이드의 과학 연구 (B) (# 15H04329)에 대한 그랜트 - 에이드의 탐색 적 연구 ( # 26650059)과 일본의 교육부, 문화, 스포츠, 과학 기술에서 '게놈 지원'(# 221S0002) (HF).
자료
| Name | Company | Catalog Number | Comments |
| gBlock synthesis service | Life Technologies | Gene Synthesis by GeneArt | |
| gBlock synthesis service | IDT (Integrated DNA Technologies) | gBlocks Gene Fragments | |
| pSIR-neo | Addgene | 51128 | |
| pSIR-GFP | Addgene | 51134 | |
| pSIR-DsRed-Express2 | Addgene | 51135 | |
| pSIR-hCD2 | Addgene | 51143 | |
| TAL synthesis service | Life Technologies | GeneArt Precision TALs | |
| 3xFLAG-dCas9/pCMV-7.1 | Addgene | 47948 | |
| 3×FLAG-dCas9/pMXs-puro | Addgene | 51240 | |
| 3×FLAG-dCas9/pMXs-IG | Addgene | 51258 | |
| 3×FLAG-dCas9/pMXs-I2 | Addgene | 51259 | |
| 3×FLAG-dCas9/pMXs-neo | Addgene | 51260 | |
| anti-FLAG M2 Ab | Sigma-Aldrich | F1804 | |
| FITC-conjugated anti-FLAG M2 | Sigma-Aldrich | F4049 | |
| DMEM medium for SILAC | Life Technologies | 89985 | Other medium can be purchased from Life Technologies |
| Dialyzed FBS for SILAC | Life Technologies | 89986 | |
| L-Lysine-2HCl for SILAC | Life Technologies | 89987 | For Light medium |
| L-Arginine-HCl for SILAC | Life Technologies | 89989 | For Light medium |
| L-Lysine-2HCl, 13C6 for SILAC | Life Technologies | 89988 | For Heavy medium |
| L-Arginine-HCl, 13C6, 15N4 for SILAC | Life Technologies | 89990 | For Heavy medium |
| Complete, mini, EDTA-free | Roche Diagnostics | 4693159 | |
| Ultrasonic Disruptor UD-201 | Tomy Seiko | ||
| ChIP DNA Clean & Concentrator | Zymo Research | D5205 | |
| Dynabeads-Protein G | Life Technologies | DB10004 | |
| RNasin Plus RNase Inhibitor | Promega | N2611 | |
| Isogen II | Nippon Gene | 311-07361 | |
| Direct-zol RNA Miniprep kit | Zymo Research | R2050 | |
| LTQ Orbitrap Velos | Thermo Fisher Scientific | A component of a nanoLC-MS/MS system for MS analysis | |
| nanoLC | Advance, Michrom Bioresources | A component of a nanoLC-MS/MS system for MS analysis | |
| HTC-PAL autosampler | CTC Analytics | A component of a nanoLC-MS/MS system for MS analysis |
참고문헌
- Zhang, X. Y., Horz, W. Analysis of highly purified satellite DNA containing chromatin from the mouse. Nucleic Acids Res. 10, 1481-1494 (1982).
- Workman, J. L., Langmore, J. P. Nucleoprotein hybridization: a method for isolating specific genes as high molecular weight chromatin. Biochemstry. 24, 7486-7497 (1985).
- Boffa, L. C., Carpaneto, E. M., Allfrey, V. G. Isolation of active genes containing CAG repeats by DNA strand invasion by a peptide nucleic acid. Proc. Natl. Acad. Sci. USA. 92, 1901-1905 (1995).
- Jaskinskas, A., Hamkalo, B. A. Purification and initial characterization of primate satellite chromatin. Chromosome Res. 7, 341-354 (1999).
- Griesenbeck, J., Boeger, H., Strattan, J. S., Kornberg, R. D. Affinity purification of specific chromatin segments from chromosomal loci in yeast. Mol. Cell Biol. 23, 9275-9282 (2003).
- Ghirlando, R., Felsenfeld, G. Hydrodynamic studies on defined heterochromatin fragments support a 30-µm fiber having six nucleosomes per turn. J. Mol. Biol. 376, 1417-1425 (2008).
- Déjardin, J., Kingston, R. E. Purification of proteins associated with specific genomic loci. Cell. 136, 175-186 (2009).
- Hoshino, A., Fujii, H. Insertional chromatin immunoprecipitation: a method for isolating specific genomic regions. J. Biosci. Bioeng. 108, 446-449 (2009).
- Fujita, T., Fujii, H. Direct idenification of insulator components by insertional chromatin immunoprecipitation. PLoS One. 6, e26109(2011).
- Fujita, T., Fujii, H. Efficient isolation of specific genomic regions by insertional chromatin immunoprecipitation (iChIP) with a second-generation tagged LexA DNA-binding domain. Adv. Biosci. Biotechnol. 3, 626-629 (2012).
- Fujita, T., Fujii, H. Locus-specific biochemical epigenetics / chromatin biochemistry by insertional chromatin immunoprecipitation. ISRN Biochem. 2013, 913273(2013).
- Fujita, T., Fujii, H. Efficient isolation of specific genomic regions retaining molecular interactions by the iChIP system using recombinant exogenous DNA-binding proteins. BMC Mol. Biol. 15, 26(2014).
- Fujita, T., Kitaura, F., Fujii, H. A critical role of the Thy28-MYH9 axis in B cell-specific expression of the Pax5 gene in chicken B cells. PLoS One. 10, (2015).
- Fujita, T., Fujii, H. Efficient isolation of specific genomic regions and identification of associated proteins by engineered DNA-binding molecule-mediated chromatin immunoprecipitation (enChIP) using CRISPR. Biochem. Biophys. Res. Commun. 439, 132-136 (2013).
- Fujita, T., et al. Identification of telomere-associated molecules by engineered DNA-binding molecule-mediated chromatin immunoprecipitation (enChIP). Sci. Rep. 3, 3171(2013).
- Fujita, T., Fujii, H. Identification of proteins associated with an IFNγ-responsive promoter by a retroviral expression system for enChIP using CRISPR. PLoS One. 9, e103084(2014).
- Fujita, T., Yuno, M., Okuzaki, D., Ohki, R., Fujii, H. Identification of non-coding RNAs associated with telomeres using a combination of enChIP and RNA sequencing. PLoS One. 10, e0123387(2015).
- Naito, Y., Hino, K., Bono, H., Ui-Tei, K. CRISPRdirect: software for designing CRISPR/Cas guide RNA with reduced off-target sites. Bioinformatics. , (2014).
- Mali, P., et al. RNA-guided human genome engineering via Cas9. Science. 339, 823-826 (2013).
- Esvelt, K. M., et al. Orthogonal Cas9 proteins for RNA-guided gene regulation and editing. Nat. Methods. 10, 1116-1121 (2013).
- Qi, L. S., et al. Repurposing CRISPR as an RNA-guided platform for sequence-specific control of gene expression. Cell. 152, 1173-1183 (2013).
- Wu, X., et al. Genome-wide binding of the CRISPR endonuclease Cas9 in mammalian cells. Nat. Biotechnol. 32, 670-676 (2014).
- Kuscu, C., Arslan, S., Singh, R., Thorpe, J., Adli, M. Genome-wide analysis reveals characteristics of off-target sites bound by the Cas9 endonuclease. Nat Biotechnol. 32, 677-683 (2014).
- Cencic, R., et al. Protospacer adjacent motif (PAM)-distal sequences engage CRISPR Cas9 DNA target cleavage. PLoS One. 9, 109213(2014).
- O'Green, H., Henry, I. M., Bhakta, M. S., Meckler, J. F., Segal, D. J. A genome-wide analysis of Cas9 binding specificity using ChIP-seq and targeted sequence capture. Nucleic Acids Res. 43, 3389-3404 (2015).
- de Lange, T. Shelterin: the protein complex that shapes and safeguards human telomeres. Genes Dev. 19, 2100-2110 (2005).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유