Method Article
enChIPによって特定のゲノム領域と関連する分子の同定の単離
要約
The identification of molecules associated with specific genomic regions of interest is required to understand the mechanisms of regulation of the functions of these regions. This protocol describes procedures to perform engineered DNA-binding molecule-mediated chromatin imunoprecipitation (enChIP) for identification of proteins and RNAs associated with a specific genomic region.
要約
The identification of molecules associated with specific genomic regions of interest is required to understand the mechanisms of regulation of the functions of these regions. To enable the non-biased identification of molecules interacting with a specific genomic region of interest, we recently developed the engineered DNA-binding molecule-mediated chromatin immunoprecipitation (enChIP) technique. Here, we describe how to use enChIP to isolate specific genomic regions and identify the associated proteins and RNAs. First, a genomic region of interest is tagged with a transcription activator-like (TAL) protein or a clustered regularly interspaced short palindromic repeats (CRISPR) complex consisting of a catalytically inactive form of Cas9 and a guide RNA. Subsequently, the chromatin is crosslinked and fragmented by sonication. The tagged locus is then immunoprecipitated and the crosslinking is reversed. Finally, the proteins or RNAs that are associated with the isolated chromatin are subjected to mass spectrometric or RNA sequencing analyses, respectively. This approach allows the successful identification of proteins and RNAs associated with a genomic region of interest.
概要
興味のある特定のゲノム領域に関連する分子の同定は、このような転写およびエピジェネティック制御などのゲノム機能の調節のメカニズムを理解するために必要です。いくつかの技術は、特定のゲノム領域1-7の生化学的分析のために開発されてきたが、それらは、ため、このような制限されたアプリケーション(例えば、唯一の反復を有する高コピー数の遺伝子座または遺伝子座など)と、それらの本質的な問題のため、この段階では広く使用されていませんあまりにも多くの時間と努力が必要です。
簡単に特定のゲノム領域の生化学的解析を実行するために、我々は2遺伝子座特異的クロマチン免疫沈降(チップ)技術、すなわち挿入チップ(iChIP)8-13と設計のDNA結合分子によって媒介されるチップ(enChIP)14-17を開発しました 。 iChIPでは、関心対象の遺伝子座は、レックスのような外因性のDNA結合タンパク質の認識配列を挿入することによってタグ付けされA.軌跡は、その後、タグ付けされたDNA結合タンパク質を用いたアフィニティー精製によって単離します。 enChIPでは、このようなジンクフィンガータンパク質、転写活性化因子のような(TAL)タンパク質などのDNA結合分子を、設計、およびクラスタ化された定期的interspaced短い回文繰り返し(CRISPR)錯体は、( 図1)は、対象の座をタグ付けするために使用されています。続いて、ゲノム領域は、タグ付けされたDNA結合分子の親和性精製によって単離されます。
iChIP enChIP上の利点の一つは、外来性DNA結合タンパク質の認識配列の挿入が必要ないことです。 Cas9の触媒的に不活性な形(dCas9)からなるCRISPR錯体とガイドRNA(gRNA)を使用して遺伝子座の標的化TAL、亜鉛フィンガータンパク質を使用してiChIPまたはenChIPにより、これらの領域の標的よりもはるかに簡単です。ここでは、遺伝子座アソシアを識別するために、(RNA-SEQ)質量分析およびRNAの配列決定と組み合わせenChIPのためのステップバイステップのプロトコルを記述テッドタンパク質およびRNAを、それぞれ。
プロトコル
ターゲット軌跡を認識エンジニアDNA結合分子の1デザイン
- CRISPR複合体を使用してenChIPについては、以前に18として記述関心のゲノム領域における候補gRNA標的配列を同定するためにCRISPRdirect Webツール(http://crispr.dbcls.jp)を使用します。このWebツールは、対象領域内に'形式5'-N 20 NGG-3の23 bpのゲノムのサイトを返します。
- U6プロモーター配列及び5'-N 20の配列商用サービスを使用して、5'-N 20 NGG-3 'を含むgBlock合成19,20( 図2参照)。目的をサブクローニングするために、gBlockの外に適切な制限酵素部位を含みます。
- 以前に16を説明したように適当なベクターにgBlockを挿入します。 gBlocksためのいくつかのレトロウイルスベクターは、(材料を参照)があります。
- TALタンパク質を使用してenChIPため、TARGを認識TALタンパク質を設計ら座。商用サービスを使用してTALタンパク質をコードするプラスミドを生成します。先に記載15のような3×FLAGのような1つ以上のタグと融合したTALタンパク質を含む発現ベクターを生成します。
enChIP解析のための細胞の樹立2。
- 以前に14〜17のように分析する細胞に3×FLAG-dCas9とgRNA(CRISPRベースの手順)または3×FLAG-TAL(TALのタンパク質ベースの手順)を発現します。
- トランスフェクション効率が高い場合、一過性トランスフェクションを使用してください。 3×FLAG-dCas9とCMVプロモーターを含む発現ベクターは、(材料を参照)が利用可能です。
- 一過性のトランスフェクション効率が低い場合、従来の方法を使用して安定した形質転換体を確立考えます。 gBlockする上述のレトロウイルスベクターに加えて、3×FLAG-dCas9のレトロウイルスベクターは、(材料を参照)があります。
- 以前に14〜17記載の抗FLAG抗体(AB)を用いたイムノブロット分析により、トランスフェクトされた細胞で3xFLAG-dCas9または3xFLAG-TALタンパク質の発現を確認してください。
注:これらのタグ化タンパク質の発現は、抗FLAG-FITCおよびその後のFACS分析と細胞内染色によって確認することができます。細胞内染色のためのプロトコルは、弊社ホームページ(http://www.biken.osaka-u.ac.jp/lab/microimm/fujii/iChIP_protocols/english.html)からダウンロードすることができます。 - 標準的RT-PCR法によりgRNAの発現を確認してください。
- 興味のあるゲノム領域と相互作用するタンパク質を定量的に同定するために、enChIP(enChIP-SILAC)と組み合わせた細胞培養(SILAC)分析におけるアミノ酸による安定同位体標識の使用を検討してください。
注:SILAC分析のためのメディアは、市販業者から購入することができます。 SILACは、特異的な相互作用を検出するのに強力です。- SILAC重い培地で細胞を培養します。細胞を準備cultu陰性対照としてSILAC光媒体中の赤。 (重鎖または軽)各SILAC媒体の少なくとも5×10 7個の細胞を使用してください。効率的な標識のために、両方のL-リジン2HCl、13 C 6およびL-アルギニン塩酸、SILAC重い培地で13 C 6、15 N 4を追加します 。注:少なくとも5の細胞分裂は、タンパク質を標識するために必要です。
- 例えば、培養ヒト線維肉腫HT1080細胞を安定的に37℃で3xFLAG-dCas9とgRNAを表現し、SILACのためのDMEM培地中で5%のCO 2およびリジン- 2HClプラスL-アルギニン塩酸(ライト中)とウシ胎児血清を透析または13 C 6 L-リジン-2HClプラス13 C 6〜15 N 4 L-アルギニン塩酸(ヘビーミディアム)16(材料を参照してください )。培地500mlに光や重いL-リジン-2HClおよびL-アルギニン塩酸50mgを追加します。
- 細胞が指数growtを維持するようにトリプシン処理およびreplate細胞は、それらがコンフルエントになる前にH。
- SILAC重い培地で細胞を培養します。細胞を準備cultu陰性対照としてSILAC光媒体中の赤。 (重鎖または軽)各SILAC媒体の少なくとも5×10 7個の細胞を使用してください。効率的な標識のために、両方のL-リジン2HCl、13 C 6およびL-アルギニン塩酸、SILAC重い培地で13 C 6、15 N 4を追加します 。注:少なくとも5の細胞分裂は、タンパク質を標識するために必要です。
ホルムアルデヒドで細胞を3架橋
- 懸濁培養細胞の場合、転送2×10 7 3xFLAG-TALタンパク質を発現する細胞または3xFLAG-dCas9プラスgRNA、50mlの遠心管中で、通常の培養液30mlに懸濁します。より多くの細胞が使用される場合、比例的な試薬の容積及び量を増加させます。 SILAC実験のために、重い媒体や光培地で培養した細胞(5×10 7細胞ずつ)混合し、2×10 7個の細胞を含む5チューブに1×10 8個の細胞の合計を分割します。
- 細胞懸濁液(最終濃度1%)の30 mlの37%ホルムアルデヒドの810マイクロリットルを加え、5分間37℃でインキュベートします。接着細胞については、直接培養培地の30 mlの37%ホルムアルデヒドの810マイクロリットルを添加することにより、培養皿で細胞を固定します。
- 1.25 Mグリシン溶液(最終濃度127 mM)の3.1mlのを追加することにより、架橋を停止し、室温で10分間インキュベートします。
- 遠心分離(4℃で5分間、300×g)と細胞を収集します。慎重にホルムアルデヒドを含む上清を破棄し、適切な廃棄ボトルに保管します。
- 接着細胞の場合は、50mlチューブ中の細胞スクレーパーと収穫して細胞を剥離し、この手順で説明したように細胞を収集します。
- SILAC実験のために、重い媒体や光培地で培養し分離した細胞(5×10 7細胞ずつ)混ぜ、2×10 7個の細胞を含む5チューブに1×10 8個の細胞の合計を分割し、これに記載されているように細胞を採取ステップ。
- チューブあたり二回のPBS 30mlで細胞ペレットを洗浄します。慎重にホルムアルデヒドを含む上清を破棄し、化学物質の安全性ガイドラインに従ってホルムアルデヒド廃棄物bottle.Handle適切な廃棄物に保管してください。固定した細胞を凍結し、-80℃で保存することができます。
クロマチンの4準備(あたり2×107細胞 )
- 細胞溶解緩衝液(10mMトリス(pH8.0)、1mMのEDTA、0.5%IGEPAL CA-630、および1×プロテアーゼ阻害剤)の10 mlの固定細胞を懸濁し、氷上で10分間インキュベートします。
- 遠心分離サンプル(8分間、4℃で830×gで)、慎重に上清を捨てます。
- 核溶解緩衝液(10mMトリス(pH8.0)、1mMのEDTA、0.5 MのNaCl、1%トリトンX-100、0.5%デオキシコール酸ナトリウム、0.5%ラウロイルサルコシン、および1×プロテアーゼ阻害剤)10mlにペレットを懸濁します。 10分間氷上でインキュベートし、2〜3分ごとにボルテックス。
- 遠心分離サンプル(830× 8分間4℃でグラム)と慎重に上清を捨てます。
- 10mlのPBSでペレットを洗浄します。ペレット(クロマチンの画分)を液体窒素中で即時凍結した後、-80℃で保存することができます。
クロマチン5.超音波処理(2×10 7個の細胞あたり)
- 800μlの中でクロマチン画分を一時停止修正された溶解緩衝液3(10mMトリス(pH8.0)、1mMのEDTA、150mMのNaCl、0.1%デオキシコール酸ナトリウム、0.1%SDS、及び1×プロテアーゼ阻害剤)、及び1.5 mlチューブに移します。
- 出力:超音波処理装置を用いて、クロマチン(材料を参照)、以下の条件超音波処理3。デューティ:100%(連続)。時刻:無料。 10秒間の超音波処理の10-18サイクルを実行し、20秒間、氷上で冷却します。過度の加熱を避けるために、2分ごとに5~6サイクルの間、氷上でサンプルをインキュベートします。発泡回避するために、0.5センチメートルチューブの底上記超音波処理プローブの先端の位置をキープ。
- 遠心分離(4℃で10分間16,000×g)でサンプルを1.5 mlチューブに上清を移します。超音波処理し、クロマチンを液体窒素中で即座に凍結した後、-80℃で保存することができます。
クロマチン断片化の6評価
- 蒸留水85μlの断片化クロマチンの10μLを混合します。
- 4μLを追加5 MのNaClと65°のCO / Nでインキュベートします。
- 10mg / mlのRNase Aの1μl添加し、45分間37℃でインキュベートします。
- 、0.5 M EDTA(pH8.0)を2μl加え、1Mトリス(pHは6.8)の4μL、および20 mg / mlのプロテイナーゼKを1μl、その後、1.5時間45℃でインキュベートします。
- 例えば、SYBRグリーンなどの染料を染色含まない1%アガロースゲルで電気泳動による試料の別々の10μlの。
- 断片化クロマチンの長さの分布を評価するために、ゲルを染色。 0.5〜2 kbpの(0.2-4 kbpの範囲)の平均長を生成する条件が推奨されています。
- フェノール-クロロホルム抽出により、またはDNA抽出キット(材料を参照)を使用して、残りの試料からDNAを精製します。精製されたDNAは、(8.11を参照)enChIP分析の収率を推定するために、入力DNAとして使用することができます。
抗体によるダイナビーズ共役の7.製造(2×10 7個の細胞あたり)
- 前抗FLAG抗体とのインキュベーションのために正常マウスIgGおよび他を使用して、2つの1.5ミリリットルチューブ、事前クリアのための1つを削減します。各チューブに(材料を参照)プロテインG結合磁性ビーズ150μlのを追加します。
- マグネットスタンドにチューブを置き、3分間待ちます。ピペットで上清を捨てます。
- 0.01%のTween-20(PBS-T)を含む1mlのPBS中のビーズを一時停止します。磁気スタンドにチューブを置き、2分間待ちます。ピペットで上清を捨て、ステップを繰り返します。
- 0.1%BSAを含むPBS-T 1ml中のビーズを一時停止します。
- 正常マウスIgGまたは抗FLAG抗体15μgのを追加し、4°CO / Nで回転します。
- その後、簡単にスピンダウンマグネットスタンドにチューブを置き、3分間待ちます。ピペットで上清を捨てます。
- PBS-T 1ml中のビーズを一時停止します。サンプル数回転倒し、簡単にスピンダウン。その後、ピペットで上清を捨て、マグネットスタンドにチューブを置き、3分間待ちます。洗濯を繰り返してPBS-T(3回の洗浄工程の合計)でさらに2回。
8.クロマチン免疫沈降(2×10 7個の細胞あたり)
- ボリュームの五分の一の5%トリトンX-100(1%トリトンX-100の最終濃度)を含む改変溶解バッファー3の(約200μL)と5.3で製造した断片化クロマチン)(約800μl)を混ぜます。
- 正常マウスIgGと結合させ、プロテインG結合磁性ビーズを準備されたチューブにクロマチン溶液を加えます。 1時間4℃で回転させます。
- マグネットスタンドにチューブを置き、3分間待ちます。
- 抗FLAG抗体とコンジュゲートプロテインG結合磁性ビーズを準備されたチューブに上清を移します。 4°CO / Nで回転します。
- マグネットスタンドにチューブを置き、3分間待ちます。ピペットで上清を捨てます。
- 低塩緩衝液(20mMトリス(pH8.0)を1mlのビーズをサスペンド、2mMのEDTA、150mMのNaCl、1%トリトンX-100、0.1%SDS、及び1×プロテアーゼ阻害剤)、5分間、4℃で回転します。マグネットスタンドにチューブを置き、3分間待ちます。ピペットで上清を捨て、洗浄ステップを繰り返します。
- 二倍の高塩緩衝液(20 mMトリス(pHは8.0)、2mMのEDTA、500mMのNaCl、1%トリトンX-100、0.1%SDS、及び1×プロテアーゼ阻害剤)でビーズを洗浄します。
- 二回(10mMトリス(pH8.0)、1mMのEDTA、250mMの塩化リチウム、0.5%IGEPAL CA-630、0.5%デオキシコール酸ナトリウム、および1×プロテアーゼ阻害剤)のLiCl緩衝液でビーズを洗浄します。
- 一度(50 mMトリス(pHは7.5)、150mMのNaCl、0.1%IGEPAL CA-630、および1×プロテアーゼ阻害剤)TBS-IGEPAL CA-630でビーズを洗浄します。
- 溶出緩衝液200μl(50 mMトリス(pHは7.5)、150mMのNaCl、0.1%IGEPAL CA-630、1×プロテアーゼ阻害剤、および500 / mlの3×FLAGペプチド)中のビーズをサスペンドし、37℃でインキュベート20分。マグネットスタンドにチューブを置き、3分間待ちます。新しい1.5 mlチューブに上清を移し、ELUTを繰り返しますイオンステップ。
- フェノール-クロロホルム抽出により、またはDNA抽出キット(材料を参照)を用いて溶出液の小部分(例えば、5%)からのDNAを精製します。精製されたDNAは、enChIPの収率を推定するために、特定のプライマーセットでPCRに使用することができ、入力DNA 14,16以前説明したように、ステップ6.7で調製と比較することにより分析します。
9. SDS-PAGE、染色、および質量分析
- 2-プロパノール、1mlの3M酢酸ナトリウム(pHは5.2)、および20 mg / mlのグリコーゲンの5μlと50μlの溶出液(400μl)を混ぜます。 -20°のCO / Nでクロマチンを沈殿させます。
- 遠心サンプル(4℃で30分間16,000×g)で、上清を捨てます。再び70%エタノールおよび遠心分離機の1ミリリットル(4℃で10分間16,000×g)でペレットを洗浄します。ピペットで完全に上清を捨てます。
- 2Xサンプルバッファー(125 mMトリス(pHは6.8)40μlの、10%の2-傭兵でペレットを中断aptoethanol、4%SDS、10%スクロース、および0.004%ブロモフェノールブルー)。完全にペレットを溶解するために5分間ボルテックスし、次いで、タンパク質を変性させ、架橋を逆に30分間、100℃でインキュベートします。
- 色素はよく以下1cmに達するまで、SDS-PAGE、ゲル上でサンプルを40μlを実行します。注:は、通常、4-20%勾配ゲルを使用し、それ以外%ゲルを使用することができます。
- クマシーブリリアントブルーまたは銀染色でゲルを染色します。
- 5個(2ミリメートルの高さ)にゲルをカットします。
- 先に説明したようにゲル13-16消化および質量分析で行います。
- 5×10 7細胞それぞれ(1×10 8細胞の合計)とSILAC実験のために、2倍の試料緩衝液40μlの中で最初の5チューブ(それがスケールアップする必要はありません)からのペレットを一時停止します。
RNAおよびRNA-のSeq解析の10精製
- enChIP後、RNAを精製するために、RNA分解酵素阻害剤の5 U / mlのを追加(SE緩衝液のすべてに電子材料)は、修正された溶解バッファー3と溶出バッファーを除いたRNアーゼ阻害剤の40 U / mlのを追加します。
- 5 M NaClを16μlの溶出液(400μl)を混合し、2時間65℃でインキュベートします。
- サンプルに(材料を参照)は、酸グアニジニウム - フェノール系試薬の1ミリリットルを追加します。 15秒間ボルテックスした後、5〜15分間室温でインキュベートします。 12,000×gで、室温で15分間サンプルを遠心します。
- 新しい1.5 mlチューブに上清を移し、P-ブロモアニソールの5μlを添加します。 15秒間ボルテックスした後、3〜5分間室温でインキュベートします。 12,000×gで、室温で10分間サンプルを遠心します。
- 新しい2 mlチューブに上清(約1ml)を転送し、2-プロパノールの1ミリリットルを追加します。チューブを反転し、室温で10分間インキュベートします。 (材料を参照)、RNA精製キットでカラムに混合物をロードします。 12,000×で1分間カラムを遠心 GおよびRT。
- ウォッシュRNA精製キット中のRNA洗浄緩衝液400μlの有するカラム(材料を参照してください)。
- DNアーゼIカクテルの80μlのDNアーゼの5μlに(混合物I(1 U /μl)を、10×DNアーゼI反応バッファー8μlの、3 DNアーゼ/μlのRNaseフリー水、および中のRNA洗浄バッファー64μLを追加カラムへのRNA精製キット(材料)を参照)。 15分間37℃でサンプルをインキュベートした後、12,000×gで、室温で30秒間遠心します。
- 二回のRNA精製キット中のRNA予洗バッファー400μlの(材料を参照)でカラムを洗浄します。
- DNアーゼ/ RNaseフリー水50μlのRNAを溶出。溶出したRNAは、RNAの配列決定のために使用することができます。
結果
全体的に、対象のゲノム領域1〜30%がenChIPを用いて精製することができる。 図3は、enChIPの収量を示す代表的なデータを含んでテロメアとインターフェロン(IFN)調節因子-1(IRF-1)遺伝子のプロモーターを標的と分析しています。典型的な結果としては、例えば、 表1 enChIP-SILACで識別されるIFNγ特異的にIRF-1プロモーターに関連するタンパク質、 表2質量分析(enChIP-MS)と組み合わせenChIPで識別テロメア結合タンパク質として、 、および表3 enChIP-RNA-、配列番号で識別されるテロメアに関連付けられたRNA。
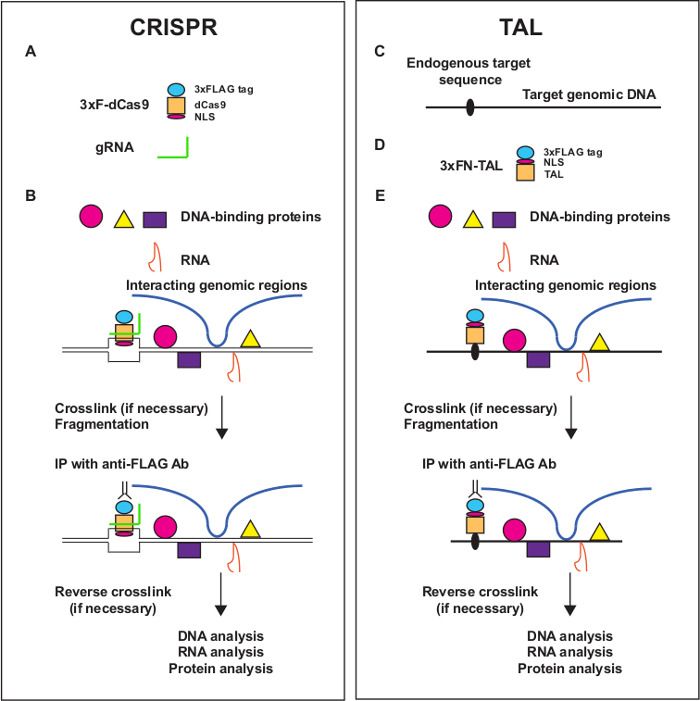
enChIPの1. 概要図 。 CRISPR(A、B)とTAL(C、D、Eを用い enChIP )が表示されます。興味の遺伝子座は、TALタンパク質またはCas9(DCAS)の触媒的に不活性な形で構成されるCRISPRシステムとして設計されたDNA結合分子でタグ付けし、(gRNA)のRNAを案内されます。必要に応じて、分子間相互作用は、ホルムアルデヒドまたは他の架橋剤で固定されています。その後、固定クロマチンは、超音波処理または酵素消化により断片化されています。タグ付けされたゲノム領域をアフィニティー精製によって精製されます。最後に、架橋を逆転させている、と相互作用する分子(ゲノム領域、RNAを、およびタンパク質が)、次世代シーケンシングおよび質量分析法を用いて識別されます。 この図の拡大版をご覧になるにはこちらをクリックしてください。

gBlockの図2のシーケンス。U6プロモーター(黒)、GヌクレオチドBガイド配列(青)、(赤)ガイド配列(gRNAスペーサー)、(緑色)足場配列、および(オレンジ色)ターミネーター配列EFORE示されています。
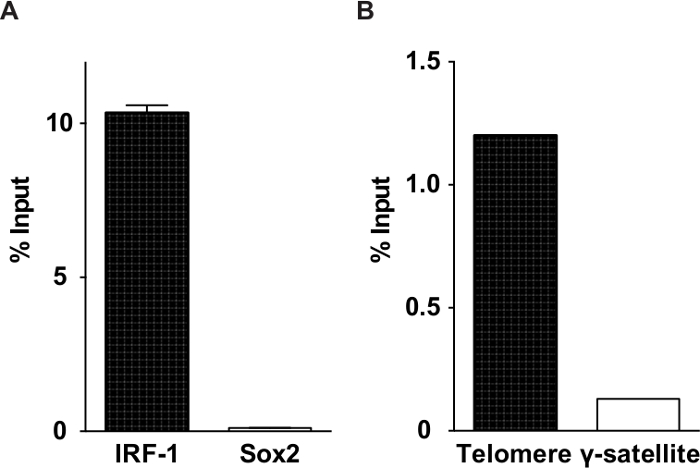
代表enChIP 3.収量を分析図 。 (A)対象IRF-1遺伝子座および非標的Sox2の遺伝子座のためのパーセントを入力。ターゲットテロメアと非標的γ-衛星の(B)の割合を入力。数値は適応し、前の出版物15,16から変更されています。
| カテゴリー | タンパク質 |
| 転写 | DDX1、PARP1、CKAP4、Pescadilloホモログ、PURβ、 活性化されたRNAポリメラーゼII転写活性化P15、 BTF3、のMyb結合タンパク質1A |
| ヒストン脱アセチル化、 コリプレッサーコンポーネント | RBBP4、PA2G4、TBL3 |
| アセチルトランスフェラーゼ | タンパク質アルギニンメチルトランスフェラーゼ1のN |
| DNAトポイソメラーゼ | DNAトポイソメラーゼ2α |
| ヒストン | ヒストンH2A.Z、ヒストンH3.2 |
表1:enChIP-SILAC で識別されるIFNγ特異的に人間のIRF-1プロモーター領域に関連するタンパク質の例の表は、以前の出版物16から適応されています。
| カテゴリー | タンパク質 |
| 哺乳類テロメア結合タンパク質 | PML、RPA、CDK1、PARP1、PCBP1 |
| テロメア-BI酵母におけるndingタンパク質 または他の生物 | IMP4 |
| テロメア結合と相互作用するタンパク質 タンパク質【関連テロメア結合タンパク質] | DNAポリメラーゼα(POLA1)[Cdc13p]、 ARMC6【TRF2]、CTBP1 [FOXP2-POT1]、 エクス-5 [TERT]、GNL3L [TRF1]、 エクス-1 [TERT]、14-3-3 [TERT] |
| ヘテロクロマチンに局在化するタンパク質 | BEND3 |
| エピジェネティックなマークを調節するタンパク質 | KDM5C |
| その変異テロメア機能に影響を及ぼすタンパク質 | DNAポリメラーゼα(POLA1)、HAT1、Nup133、 CDK7、DPOE1、PRDX1、TYSY、 グルタミン酸システインリガーゼ、グルタレドキシン、SMRC1 |
表2:enChIP-MS により同定したマウステロメアに関連するタンパク質の例には、表は、adapteされています以前の出版物15からD。
| カテゴリー | RNAを |
| テロメラーゼコンポーネント | TERC、Rmrp |
| テロメアのRNA | TERRAs |
| scaRNAs | Scarna6、Scarna10、Scarna13、Scarna2 |
| H / ACAのsnoRNA | Snora23、Snora74a、Snora73b、Snora73a |
| C / DのsnoRNA | Snord17、Snord15a、Snord118 |
| lncRNA | Neat1 |
表3:enChIP-RNA-、 配列番号で識別されるマウスのテロメアと関連するRNAの例の表は、以前の出版物17から適応されています。
ディスカッション
Here, we describe the purification of specific genomic regions using engineered DNA-binding molecules such as the CRISPR system and TAL proteins, and the identification of proteins and RNAs bound to these genomic regions. Binding of engineered DNA-binding molecules to the genome may affect chromatin structure, including nucleosome positioning, and may abrogate genomic functions, as described in CRISPR interference experiments21. To avoid these potential aberrant effects, we propose specific guidelines for choosing target genomic regions. First, to avoid potential inhibition of the recruitment of RNA polymerases and transcription factors, as well as disruption of nucleosome positioning around the transcription start site, the target regions for analyses of promoter regions should be several hundred base pairs upstream of (5' to) the transcription start site. By contrast, when analyzing genomic regions with distinct boundaries, such as enhancers and silencers, genomic regions that are directly juxtaposed to these regions can be targeted because it is less likely that the binding of engineered DNA-binding molecules will affect their functions. Furthermore, it is best to avoid using target regions that are conserved among different species, because important DNA-binding molecules often bind to evolutionarily conserved regions and inhibition of their binding might disrupt the functions of the target genomic regions. In this regard, it is always necessary to check that the function of the target genomic region is maintained in the established cells used for enChIP analyses. Because multiple gRNAs can be tested easily and it is tedious and expensive to generate multiple versions of TAL or zinc-finger proteins recognizing different target genomic regions, enChIP using CRISPR is more advantageous than enChIP using other proteins.
It has been shown that dCas9 binds to off-target sites although affinity to those sites might be weaker than that to the target sites22-25. There are several ways to manage contamination of molecules bound to those off-target sites. First, the use of several, at least two, different gRNAs would be recommended. Those molecules commonly observed in enChIP using distinct gRNAs would be true positives. Second, comparison of different conditions for enChIP would be effective in cancelling contamination of non-specific molecules and molecules bound to off-target sites. Examples of those comparison sets would be (i) stimulation (-) and (+), or (ii) different cell types such as T cells vs. B cells. Finally, quantitative analysis of binding of candidate molecules should be performed to confirm their specific binding to the target sites. It is preferable to prepare cells expressing only dCas9 but not gRNA as a negative control.
Using enChIP analyses, we were able to successfully identify a number of known and novel molecules interacting with specific genomic regions (Tables 1-3)14-17. However, this technique failed to detect some other known proteins interacting with these regions. For example, STAT1 reportedly associates with the IRF-1 promoter upon IFNγ stimulation8, but our enChIP-SILAC analysis did not detect STAT1 as a protein induced to interact with this genomic region16. In addition, in the enChIP-MS analysis of telomeres, we did not detect shelterin proteins consisting of TRF-1 and TRF-215, which have been shown to interact with telomeres26. There are a few potential reasons for these discrepancies. First, the stoichiometry of binding of Stat1 to the IRF-1 promoter might be very low. It is reasonable that enChIP-MS, including enChIP-SILAC, detects proteins that are more abundantly associated with target genomic regions; hence, the analysis of more cells might be necessary to detect these proteins. Increases in the sensitivities of MS instruments would also contribute to the efficient detection of proteins with low stoichiometric binding. Second, some proteins, possibly including Stat1 and shelterins, might be difficult targets for MS analyses. Third, in our analysis of telomere-binding proteins15, the 3×FLAG-TAL proteins recognizing telomeres (3×FN-Tel-TAL) might have blocked the binding of shelterins to telomeres in a competitive fashion.
In contrast to the relative difficulty of detecting transcription factors binding to specific genomic regions using enChIP, we successfully identified epigenetic regulators such as histone modification enzymes using enChIP analyses. The success of this technique may be due to the fact that epigenetic regulators bind to a broad range of genomic regions; hence, more proteins per genomic region are available for MS. Because epigenetic regulators are increasingly recognized as important targets for drugs against intractable diseases such as cancer, enChIP would be a useful tool for the identification of epigenetic drug targets.
開示事項
The authors have filed a patent on enChIP (Patent name: 'Method for isolating specific genomic regions using DNA-binding molecules recognizing endogenous DNA sequences'; Patent number: PCT/JP2013/74107). H.F. is a member of the Advisory Board of Addgene.
謝辞
この作品は、武田科学財団(TF)によってサポートされていました。旭硝子財団。上原記念財団(HF)。倉田記念日立科学技術財団(TFとHF)。費補助金若手(B)(#25830131)、費補助金基盤研究(C)(#15K06895)(TF)のため、そして費補助金革新的なエリア「転写サイクル」に関する科学研究費(#25118512&#15H01354)、費補助金基盤研究(B)(#15H04329)のため、萌芽研究( #26650059)、日本の文部科学省、文化、スポーツ、科学技術から「ゲノム支援」(#221S0002)(HF)。
資料
| Name | Company | Catalog Number | Comments |
| gBlock synthesis service | Life Technologies | Gene Synthesis by GeneArt | |
| gBlock synthesis service | IDT (Integrated DNA Technologies) | gBlocks Gene Fragments | |
| pSIR-neo | Addgene | 51128 | |
| pSIR-GFP | Addgene | 51134 | |
| pSIR-DsRed-Express2 | Addgene | 51135 | |
| pSIR-hCD2 | Addgene | 51143 | |
| TAL synthesis service | Life Technologies | GeneArt Precision TALs | |
| 3xFLAG-dCas9/pCMV-7.1 | Addgene | 47948 | |
| 3×FLAG-dCas9/pMXs-puro | Addgene | 51240 | |
| 3×FLAG-dCas9/pMXs-IG | Addgene | 51258 | |
| 3×FLAG-dCas9/pMXs-I2 | Addgene | 51259 | |
| 3×FLAG-dCas9/pMXs-neo | Addgene | 51260 | |
| anti-FLAG M2 Ab | Sigma-Aldrich | F1804 | |
| FITC-conjugated anti-FLAG M2 | Sigma-Aldrich | F4049 | |
| DMEM medium for SILAC | Life Technologies | 89985 | Other medium can be purchased from Life Technologies |
| Dialyzed FBS for SILAC | Life Technologies | 89986 | |
| L-Lysine-2HCl for SILAC | Life Technologies | 89987 | For Light medium |
| L-Arginine-HCl for SILAC | Life Technologies | 89989 | For Light medium |
| L-Lysine-2HCl, 13C6 for SILAC | Life Technologies | 89988 | For Heavy medium |
| L-Arginine-HCl, 13C6, 15N4 for SILAC | Life Technologies | 89990 | For Heavy medium |
| Complete, mini, EDTA-free | Roche Diagnostics | 4693159 | |
| Ultrasonic Disruptor UD-201 | Tomy Seiko | ||
| ChIP DNA Clean & Concentrator | Zymo Research | D5205 | |
| Dynabeads-Protein G | Life Technologies | DB10004 | |
| RNasin Plus RNase Inhibitor | Promega | N2611 | |
| Isogen II | Nippon Gene | 311-07361 | |
| Direct-zol RNA Miniprep kit | Zymo Research | R2050 | |
| LTQ Orbitrap Velos | Thermo Fisher Scientific | A component of a nanoLC-MS/MS system for MS analysis | |
| nanoLC | Advance, Michrom Bioresources | A component of a nanoLC-MS/MS system for MS analysis | |
| HTC-PAL autosampler | CTC Analytics | A component of a nanoLC-MS/MS system for MS analysis |
参考文献
- Zhang, X. Y., Horz, W. Analysis of highly purified satellite DNA containing chromatin from the mouse. Nucleic Acids Res. 10, 1481-1494 (1982).
- Workman, J. L., Langmore, J. P. Nucleoprotein hybridization: a method for isolating specific genes as high molecular weight chromatin. Biochemstry. 24, 7486-7497 (1985).
- Boffa, L. C., Carpaneto, E. M., Allfrey, V. G. Isolation of active genes containing CAG repeats by DNA strand invasion by a peptide nucleic acid. Proc. Natl. Acad. Sci. USA. 92, 1901-1905 (1995).
- Jaskinskas, A., Hamkalo, B. A. Purification and initial characterization of primate satellite chromatin. Chromosome Res. 7, 341-354 (1999).
- Griesenbeck, J., Boeger, H., Strattan, J. S., Kornberg, R. D. Affinity purification of specific chromatin segments from chromosomal loci in yeast. Mol. Cell Biol. 23, 9275-9282 (2003).
- Ghirlando, R., Felsenfeld, G. Hydrodynamic studies on defined heterochromatin fragments support a 30-µm fiber having six nucleosomes per turn. J. Mol. Biol. 376, 1417-1425 (2008).
- Déjardin, J., Kingston, R. E. Purification of proteins associated with specific genomic loci. Cell. 136, 175-186 (2009).
- Hoshino, A., Fujii, H. Insertional chromatin immunoprecipitation: a method for isolating specific genomic regions. J. Biosci. Bioeng. 108, 446-449 (2009).
- Fujita, T., Fujii, H. Direct idenification of insulator components by insertional chromatin immunoprecipitation. PLoS One. 6, e26109(2011).
- Fujita, T., Fujii, H. Efficient isolation of specific genomic regions by insertional chromatin immunoprecipitation (iChIP) with a second-generation tagged LexA DNA-binding domain. Adv. Biosci. Biotechnol. 3, 626-629 (2012).
- Fujita, T., Fujii, H. Locus-specific biochemical epigenetics / chromatin biochemistry by insertional chromatin immunoprecipitation. ISRN Biochem. 2013, 913273(2013).
- Fujita, T., Fujii, H. Efficient isolation of specific genomic regions retaining molecular interactions by the iChIP system using recombinant exogenous DNA-binding proteins. BMC Mol. Biol. 15, 26(2014).
- Fujita, T., Kitaura, F., Fujii, H. A critical role of the Thy28-MYH9 axis in B cell-specific expression of the Pax5 gene in chicken B cells. PLoS One. 10, (2015).
- Fujita, T., Fujii, H. Efficient isolation of specific genomic regions and identification of associated proteins by engineered DNA-binding molecule-mediated chromatin immunoprecipitation (enChIP) using CRISPR. Biochem. Biophys. Res. Commun. 439, 132-136 (2013).
- Fujita, T., et al. Identification of telomere-associated molecules by engineered DNA-binding molecule-mediated chromatin immunoprecipitation (enChIP). Sci. Rep. 3, 3171(2013).
- Fujita, T., Fujii, H. Identification of proteins associated with an IFNγ-responsive promoter by a retroviral expression system for enChIP using CRISPR. PLoS One. 9, e103084(2014).
- Fujita, T., Yuno, M., Okuzaki, D., Ohki, R., Fujii, H. Identification of non-coding RNAs associated with telomeres using a combination of enChIP and RNA sequencing. PLoS One. 10, e0123387(2015).
- Naito, Y., Hino, K., Bono, H., Ui-Tei, K. CRISPRdirect: software for designing CRISPR/Cas guide RNA with reduced off-target sites. Bioinformatics. , (2014).
- Mali, P., et al. RNA-guided human genome engineering via Cas9. Science. 339, 823-826 (2013).
- Esvelt, K. M., et al. Orthogonal Cas9 proteins for RNA-guided gene regulation and editing. Nat. Methods. 10, 1116-1121 (2013).
- Qi, L. S., et al. Repurposing CRISPR as an RNA-guided platform for sequence-specific control of gene expression. Cell. 152, 1173-1183 (2013).
- Wu, X., et al. Genome-wide binding of the CRISPR endonuclease Cas9 in mammalian cells. Nat. Biotechnol. 32, 670-676 (2014).
- Kuscu, C., Arslan, S., Singh, R., Thorpe, J., Adli, M. Genome-wide analysis reveals characteristics of off-target sites bound by the Cas9 endonuclease. Nat Biotechnol. 32, 677-683 (2014).
- Cencic, R., et al. Protospacer adjacent motif (PAM)-distal sequences engage CRISPR Cas9 DNA target cleavage. PLoS One. 9, 109213(2014).
- O'Green, H., Henry, I. M., Bhakta, M. S., Meckler, J. F., Segal, D. J. A genome-wide analysis of Cas9 binding specificity using ChIP-seq and targeted sequence capture. Nucleic Acids Res. 43, 3389-3404 (2015).
- de Lange, T. Shelterin: the protein complex that shapes and safeguards human telomeres. Genes Dev. 19, 2100-2110 (2005).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved