Method Article
Uso de Citometria de imagem para quantificação de fungos patogênicos em associação com células do hospedeiro
Neste Artigo
Resumo
Aqui, demonstramos como citometria de imagem pode ser usada para quantificação de fungos patogénicos em associação com as células hospedeiras em cultura. Esta técnica pode ser usado como uma alternativa para CFU enumeração.
Resumo
Estudos dos mecanismos de patogênese celulares de leveduras patogênicas como a Candida albicans, Histoplasma capsulatum e Cryptococcus neoformans comumente empregam infecção de hospedeiros mamíferos ou células hospedeiras (ou seja, macrófagos), seguido pela quantificação de leveduras utilizando análise de unidade formadora de colônia ou citometria de fluxo. Enquanto a unidade de formação de colónia enumeração tem sido o método mais comummente utilizado no campo, esta técnica tem as desvantagens e limitações, incluindo crescimento lento de algumas espécies de fungos em meios sólidos e eficiências de baixo e / ou variável de revestimento, que é de preocupação particular quando se compara o crescimento de estirpes de tipo selvagem e mutante. A citometria de fluxo podem proporcionar informação quantitativa rápida quanto viabilidade celular, no entanto, a adopção de citometria de fluxo para a detecção de leveduras patogénicas tem sido limitada por um número de razões práticas, incluindo o seu elevado custo e as considerações de biossegurança. Aqui, nós demonstramos uma baseada em imagemmetodologia de citometria usando a visão Cellometer (Nexcelom Bioscience, LLC), para a quantificação de leveduras patogênicos em co-cultura com macrófagos. Nossos estudos concentram-se na detecção de dois fungos patógenos humanos: Histoplasma capsulatum e Candida albicans H.. capsulatum coloniza macrófagos alveolares, replicando dentro do fagossoma do macrófago, e aqui, nós quantitativamente avaliar o crescimento de H. leveduras capsulatum em RAW 264,7 macrófagos usando laranja de acridina / coloração iodeto de propídio em combinação com a citometria de imagem. Nosso método fielmente recapitula as tendências de crescimento, medida pela unidade formadora de colônia tradicional enumeração, mas com um aumento significativo de sensibilidade. Além disso, avaliar diretamente a infecção de macrófagos ao vivo com uma cepa GFP-expressando de C. albicans. Nossa metodologia oferece um meio rápido, preciso e econômico para a detecção e quantificação de importantes fungos patógenos humanos em associação sagacidadeAs células hospedeiras h.
Introdução
Estudos de fungos patogénicos em associação com os seus hospedeiros e / ou células hospedeiras exigem frequentemente a quantificação de células de fungos viáveis ao longo de um curso de tempo ou sob diferentes condições de infecção. Enumeração das unidades formadoras de colónias (UFC) é o método padrão pelo qual o número de células de fungos viáveis foi medida, no entanto, esta técnica tem várias desvantagens e limitações. Em primeiro lugar, muitas espécies de fungos são de crescimento lento. O crescimento das colônias visíveis em meios sólidos pode levar 1-2 semanas, diminuindo significativamente o ritmo da investigação. Por outro lado, a manipulação de amostras durante CFU chapeamento é um processo laborioso, uma vez que várias diluições deve ser revestida para garantir um número de colónias contáveis. Em terceiro lugar, o número de UFC é tipicamente menor do que o número de organismos viáveis plaqueadas porque a eficiência de plaqueamento é bem abaixo de 100%. Por exemplo, a eficiência de revestimento para o dimórfico Histoplasma capsulatum patógeno fúngico pode ser tão alto como 90%, mas são habitualmente tão acessível quanto30% e são ainda mais baixos (10%) para o fungo relacionado Dimorfa Paracoccidioides brasiliensis 1, 2. Eficiência chapeamento de Candida albicans também está sujeito à variabilidade 3. Finalmente, a análise de CFU representa apenas as células vivas e dividindo activamente capazes de estabelecer o crescimento em meios sólidos, ao passo que, em muitas situações, pode ser útil para determinar a presença e concentração de células mortas inactivos e / ou metabolicamente.
Anteriormente, os métodos de citometria de fluxo para a quantificação de várias espécies de fungos patogénicos tem sido descrita 4-6. No entanto, devido a questões de biossegurança de contenção envolvidos com o uso de nível de biossegurança 2 (BSL2) ou patógenos de nível BSL3 em cytometers fluxo compartilhados, a adoção dessa técnica tem sido limitada. Como citometria de fluxo, citometria de imagem é um método sensível e rápido de quantificação celular. No entanto, a imagem de citometria pode ser realizada a uma fracção do custo com resultados comparáveis 7 -11. Aqui, descrevemos os métodos para a realização de citometria de imagem de fungos patogênicos em associação com células hospedeiras. Nós demonstramos nossos métodos usando dois fungos patógenos humanos: Histoplasma capsulatum e Candida albicans H.. capsulatum é um fungo dimórfico que causa doença respiratória, em seres humanos, que cresce levedura como brotamento e replica no interior de macrófagos alveolares Candida albicans é uma espécie comensais humanos que provoca ocasionalmente candidíase.. Nós mostramos que a citometria de imagem permite uma rápida quantificação dessas leveduras, juntamente com a capacidade de visualização.
Protocolo
1. Infecção de macrófagos com H. capsulatum, Candida albicans
- 16 horas antes da infecção, os macrófagos de sementes com uma densidade desejada em placas de 24 poços. Neste protocolo, foi utilizada uma densidade de 3,0 x 10 5 células / poço.
- Adicionar células fúngicas em crescimento de fase log a uma multiplicidade desejada de infecção (MOI). Este protocolo pode acomodar uma gama de MOI (0,2-5,0). Para a infecção de macrófagos RAW264.7 com H. capsulatum, foi utilizada uma MOI de 0,2.
- Após 1,5 horas para permitir a fagocitose, macrófagos lavar três vezes com PBS para remover fungos extracelulares.
- Incubar número desejado de horas antes da análise de amostras. Em baixa MOI (0,2 na nossa experiência), células de macrófagos infectados permanecem viáveis durante vários dias, e as amostras podem ser analisadas aproximadamente a cada 12-24 horas.
2. Lise de macrófagos
- Para libertar os fungos a partir de células de macrófagos, ao remover a mídia, lavar 3 vezes com PBS, eadicionar 0,5 ml de água estéril. Sob estas condições, os macrófagos e irá lisar células fúngicas permanecerá intacta.
- Incubar durante 5 min a temperatura ambiente.
- Transferir lysate para tubo estéril, manter em gelo.
- Transferir 20 uL de ligado a um tubo separado, em seguida, adicionar 20 ul AO / PI solução. Prosseguir diretamente para a etapa 5: "Preparação de Amostras para análise por citometria de imagem"
3. UFC chapeamento
- Realizar diluição de dez vezes de lisados (do passo 2.3) em meios.
- Placa de 100 ul de cada diluição em duplicado, em placas HMM-agarose. Incubar as placas numa câmara húmida a 37 ° C com 5% de CO 2 durante 7-8 dias.
- Contar manualmente colônias em placas indicam um mínimo de 100 e um máximo de 1.000 colônias distintas.
4. Visualização de fungos no interior dos macrófagos ao vivo
- Para a coleta de macrófagos ao vivo, lave as células três vezes com PBS. Adicionar 0,5 ml de PBS, e incubar durante 30 min a 4 ° C.
- Para remover os macrófagos dos poços de cultura de tecidos, pipeta suavemente para cima e para baixo várias vezes. Transferência líquida para tubo estéril, manter em gelo.
- Transferir 20 uL de amostra a um tubo separado, em seguida, adicionar 20 ul AO / PI solução. Prosseguir diretamente para a etapa 5: "Preparação de Amostras para análise por citometria de imagem".
5. Preparação de amostras para análise por citometria de imagem
- Pipetar a amostra-alvo e completamente em seguida, transferir 20 mL de amostra para a célula descartável câmara de contagem.
- Permitir que as células se instalar na câmara por 30 segundos.
- Insira câmara de contagem para o citômetro de imagem.
6. Configuração do Instrumento Cellometer
- Insira a ótica de fluorescência Módulos: VB-535-402 e VB-660-502 para o sistema e verifique se eles estão presos no local.
- VB-535-402 (excitação a 475 nm, emissão a 535 nm), é usada para a detecção de laranja de acridina e de GFP.
- VB-660-502 (excitatiem 540 nm, emissão a 660 nm) é utilizado para a detecção de iodeto de propídio.
- Ligue o citômetro de imagem e abrir o software que o acompanha.
7. Instalação de Software Cellometer
- Selecione o preset "Tipo de Ensaio" e "Tipo de célula" no menu suspenso de Ensaio.
- Para a viabilidade, o ensaio é optimizado para a detecção de laranja de acridina e iodeto de propídio.
- Para a detecção de infecção por Cândida albicans, o ensaio é optimizado para a detecção de GFP.
- Selecione "Opções" na parte superior e clique em "Take imagem de fundo", e permitir que a operação seja concluída.
- Clique em "Imagem de campo claro."
8. Imagem Procedimento de Aquisição
- Insira na câmara de amostra preparada para o citômetro de imagem.
- Use o botão de foco e ajustar o foco.
- De vez em foco, clique em "Count", e permitir que a operação de aquisição de imagem para ser concluído.
- Remove a câmara de contagem descartáveis e descartar adequadamente.
9. Imagem de Análise de Dados
- Concentração e de medição da viabilidade
- Uma vez que a contagem é concluída, a concentração e a viabilidade das células-alvo são apresentados na página de resultado.
- Clique em "Exportar" para exportar os dados para o FCS Express 4 para análise da população celular da GFP em Candida albicans experiência infecção.
- Análise FCS expresso de células infectadas com Cândida albicans
- Importe o arquivo ". NXDAT" em FCS Express 4 e enredo os resultados em um histograma de fluorescência.
- Aplicar portão população de células com o histograma para determinar a percentagem de população de células infectadas com Cândida albicans.
Resultados
Usamos o Cellometer Visão imagem citômetro para monitorar o crescimento de H. capsulatum do macrófago. Células RAW 264.7 foram infectados com H. células de levedura capsulatum e em vários pontos de tempo, as amostras foram submetidas a AO / PI coloração seguida por análise de citometria de imagem baseado. Em paralelo, as amostras foram analisadas por contagem de CFU tradicional. Em cada ponto de tempo, as amostras foram incubadas em água para lisar os macrófagos e as células de levedura libertadas foram identificados pelo programa de visão Cellometer (Figuras 1a e 1b). Observamos tendências altamente comparáveis na concentração de leveduras viáveis, como detectado por análise de citometria de-base de imagem e enumeração de CFU em cada ponto de tempo (Figura 1c). Como esperado, o número absoluto de leveduras vivas detectados por AO / PI coloração era consistentemente mais elevada do que o número de leveduras capazes de formação de colónias em meio sólido tal como avaliado por análise de CFU, destacando sinalificant números de células viáveis mas não cultiváveis.
A visualização das células de fungos vivos no interior de macrófagos pode ser conseguida utilizando-GFP expressando estirpes de levedura. Macrófagos da medula óssea derivados (BMDM) estavam infectados com C. células de levedura que expressam albicans proteína fluorescente verde (GFP) sob o controlo do promotor ADH1 12 (Figura 2A). Infecção de BMDM com GFP-C albicans leveduras em multiplicidades de infecção (MOI) variando de 0,1-10 correspondeu a aumentos incrementais na intensidade de GFP em macrófagos infectados (Figura 2a), bem como percentagem total de macrófagos infectados (Figura 2b).
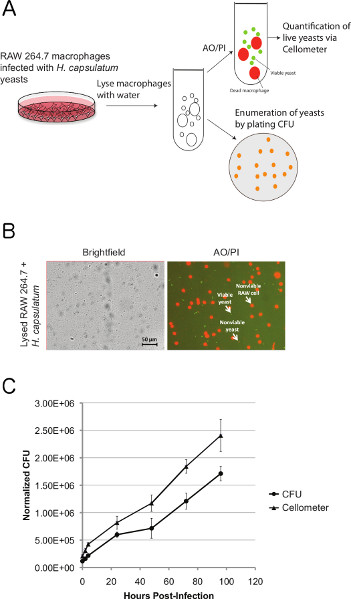
Figura 1. A comparação da análise de citometria de-imagem à base de CFU enumeração de H. capsulatum durante a infecção in vitro de macrófagos RAW 264.7. (a) RAW 264,7 macrófagos foram infectados com H. leveduras capsulatum, a um MOI de 0,2. Em intervalos de 24 h após a infecção, os macrófagos foram lisadas e leveduras viáveis avaliado pela AO / PI coloração em paralelo com CFU enumeração. (B) Representante brightfield (BR) e FL1/FL2 (verde / vermelho) imagens combinadas de AO / PI manchado H . capsulatum liberado lisadas RAW 264,7 macrófagos. Um algoritmo de segmentação contado apenas leveduras positivas AO e PI nas imagens fluorescentes, enquanto os grandes PI-manchadas RAW 264,7 macrófagos (vermelho) são excluídos. As imagens foram capturadas em aumento de 10x. (C) A comparação direta da proliferação de fungos, avaliada pela AO / PI coloração e contagem UFC. A análise de regressão linear das tendências de crescimento, medida pelo UFC e rendimento de análise Cellometer R 2 = 0,9927./ Files/ftp_upload/50599/50599fig1large.jpg "target =" _blank "> Clique aqui para ver a figura maior.

Figura 2. Quantificação de BMDM infecção com uma estirpe expressando GFP-de C. albicans. (a) Capturado brightfield (BR) (topo) e fluorescente imagens (meio) de GFP-C. albicans infectado BMDMs a aumentar MOI. Os histogramas de intensidade de fluorescência mostram um aumento na intensidade da fluorescência da GFP como os MOI aumenta, o que representa um aumento no número de C. albicans associada com células BMDM (baixo). O software identificou BMDMs infectados, com base na aplicação de uma marca linear com a intensidade de fluorescência da linha de base exibido pela população de controlo não infectadas (MOI = 0). (B) Percentagem infection em função do MOI, o que mostra um aumento na BMDMs infectados como MOI aumenta. Clique aqui para ver a figura maior .
Discussão
Citometria de imagem permite ao usuário capturar imagens de alta qualidade e, usando um software especializado, realizar rápida quantificação de células. Um desafio potencial para a adopção de citometria de imagem no domínio da patogênese microbiana é que os microrganismos sejam contados presente numa população mista de células, incluindo células hospedeiras de mamífero. Aqui, demonstramos que a citometria de imagem pode ser utilizada para a quantificação de leveduras patogénicas viáveis durante a infecção de macrófagos in vitro. Nossa metodologia não apenas fielmente recapitula a evolução do crescimento de fungos e a viabilidade observada durante a infecção dos macrófagos em ensaios in vitro, mas também exibe uma melhor sensibilidade em comparação com o ensaio de CFU tradicional 13.
Citometria de Imagem oferece diversas vantagens práticas quando comparado com qualquer um enumeração UFC ou de citometria de fluxo para a quantificação de células de levedura. Quando chapeamento UFC, os trabalhadores laboratório deve prato vários diluindoiões a fim de garantir um número de células contáveis em cada placa, o que pode ser muito trabalhoso. Citometria de Imagem elimina a necessidade de múltiplas diluições. Anteriormente, mostrámos que a formação de colónias em meios sólidos, é muitas vezes variável e a muito baixas concentrações de fungos pode falhar para formar colónias em todos, ao passo que a citometria de imagem permite a enumeração de células, independentemente da concentração de 13. A capacidade para detectar e contar fungos em concentrações muito baixas, pode ser muito útil durante os estágios iniciais de uma infecção de baixa dose, ou durante a recarga, quando o número e viabilidade pode estar na margem de detecção. Além disso, os dados gerados por citometria de imagem estão disponíveis instantaneamente, enquanto colônias pode demorar vários dias para se tornar visível. Enquanto muitas espécies de fungos pode ser detectada por citometria de fluxo, a possibilidade de contaminação cruzada é uma preocupação em instalações comuns. A câmara de contagem utilizado no presente estudo oferece uma vantagem significativa em termos de bio-segurança, como é disposable e auto-suficiente. Além disso, a imagem de citometria oferece as vantagens práticas de preço mais baixo e uma pegada menor quando comparada com a citometria de fluxo convencional, o que pode ser importante para laboratórios de investigação mais pequena 11.
Para além das suas vantagens práticas, citometria de imagem de células de levedura fornece informações que CFU enumeração não pode. Primeiro, CFU enumeração é apenas capaz de detectar fungos que são capazes de estabelecer o crescimento de colónias no meio escolhido, o que não pode representar com precisão o número de fungos viáveis presentes em uma experiência de infecção em tempo real. Além disso, a capacidade de estabelecer o crescimento de colónias em meios sólidos pode variar quando se comparam as estirpes do tipo selvagem e mutante de fungos, os quais podem ser uma fonte de polarização experimental 14, e estes preconceitos ocultos podem ser reveladas por comparação da citometria de imagem e quantificação de CFU estirpes de tipo selvagem e mutante. Uma vez que o comportamento de uma estirpe particular, tem sido caracterizada por both CFU enumeração e citometria de imagem, acreditamos que a citometria de imagem pode ser usada como um meio alternativo para a quantificação de fungos.
Uma limitação da citometria de imagem, em geral, é que ela não permite a recolha de tantos pontos de dados como a citometria de fluxo. Uma vez que o citómetro de imagem deriva dados quantitativos a partir de um número limitado de imagens capturadas, é difícil para o sistema de recolha de dados sobre centenas de milhares a milhões de células por amostra. No entanto, essa limitação pode ser contornada através do agrupamento de várias amostras em um conjunto de dados para a análise, a fim de alcançar os pontos de dados similares como um citómetro de fluxo. Gostaríamos também de salientar que o método de coloração AO / PI usado aqui representa uma forma de medir a viabilidade celular, e não necessariamente indica vitalidade celular e / ou a capacidade de estabelecer o crescimento em meio sólido, em qualquer amostra. Portanto, enquanto o nosso método proporciona um método fácil de quantificação vivo / morto de leveduras, eleTambém será interessante para investigar o uso de citometria de imagem em combinação com indicadores do metabolismo celular, como a FUN-1 15.
Software de citometria de imagem identifica e conta com células de interesse com base no tamanho e forma, e, portanto, é absolutamente crítico que estes parâmetros são definidos cuidadosamente durante cada experimento. Ao realizar a citometria de imagem com uma nova estirpe de levedura, sugere-se que uma primeira comparar os dados gerados a partir de uma cultura de levedura pura para os dados gerados a partir de uma mistura de contagem de células hospedeiras de leveduras, além de assegurar que os parâmetros são ajustados de tal forma que o software pode distinguir entre a dois tipos de células.
No futuro, este trabalho será a base para o desenvolvimento de métodos para detectar e quantificar os micróbios através de citometria de imagem. Por exemplo, o desenvolvimento de métodos de citometria de imagem para detectar fungos dentro de homogeneizados de órgãos será de grande utilização no campo. Outro desafio futuro será develop e refinar os métodos de citometria de imagem e software de tal forma que ele pode ser usado para a quantificação de células bacterianas. Com o avanço dos sistemas ópticos, câmaras digitais, e uma grande variedade de manchas fluorescentes no campo, a capacidade de analisar a imagem e população de bactérias pode ser implementado em citómetros da imagem, o que pode proporcionar um método mais rápido para a concentração e viabilidade ou vitalidade medição em comparação com os métodos de UFC ou de densidade óptica. Em resumo, o trabalho aqui apresentado é uma prova de conceito de que a imagem mostrando citometria é um método sensível para a quantificação de leveduras patogénicas. A sofisticação do programa de análise de citometria de imagem baseado oferece a oportunidade de analisar as interacções entre as células hospedeiras e os fungos patogénicos de uma maneira altamente quantitativa. Esta tecnologia pode ser adaptada para abordar uma variedade de questões de pesquisa no domínio da patogênese fúngica.
Divulgações
Os autores Leo Li-Ying Chan e Benjamin Paradis são funcionários da Nexcelom Bioscience LLC.
Materiais
| Name | Company | Catalog Number | Comments |
| REAGENTS | |||
| DMEM | Life Technologies | 11965-084 | |
| Fetal Bovine Serum | Life Technologies | 16000044 | |
| AO/PI Solution | Nexcelom Bioscience | CSK-0102 | |
| Disposable Counting Chamber | Nexcelom Bioscience | CHT4-SD100 | |
| EQUIPMENT | |||
| Cellometer Vision | Nexcelom Bioscience | ||
| Cellometer Vision Software | Nexcelom Bioscience | ||
Referências
- Worsham, P. L., Goldman, W. E. Quantitative plating of Histoplasma capsulatum without addition of conditioned medium or siderophores. J. Med. Vet. Mycol. 26, 137-143 (1988).
- Goihman-Yahr, M., Pine, L., Albornoz, M. C., Yarzabal, L., de Gomez, M. H., San Martin, B., Ocanto, A., Molina, T., Convit, J. Studies on plating efficiency and estimation of viability of suspensions of Paracoccidioides brasiliensis yeast cells. Mycopathologia. 71, 73-83 (1980).
- Bhatti, M. A., Hjertstedt, J., Hahn, B. L., Sohnle, P. G. Inefficient delivery of yeast cells as an explanation for reduced plating efficiency of Candida albicans. Med. Mycol. 40, 465-469 (2002).
- Chang, W. L., vander Heyde, H. C., Klein, B. S. Flow cytometric quantitation of yeast: a novel technique for use in animal model work and in vitro immunologic assays. Journal of Immunological Methods. 211, 51-63 (1998).
- Green, L., Petersen, B., Steimel, L., Haeber, P., Current, W. Rapid determination of antifungal activity by flow cytometry. Journal of Clinical Microbiology. 32, 1088-1091 (1994).
- Kirk, S. M., Callister, S. M., Lim, L. C., Schell, R. F. Rapid susceptibility testing of Candida albicans by flow cytometry. Journal of Clinical Microbiology. 35, 358-363 (1997).
- Chan, L. L. -. Y., Lai, N., Wang, E., Smith, T., Yang, X., Lin, B. A rapid detection method for apoptosis and necrosis measurement using the Cellometer imaging cytometry. Apoptosis. 16, 1295-1303 (2011).
- Chan, L. L. -. Y., Shen, D., Wilkinson, A. R., Patton, W., Lai, N., Chan, E., Kuksin, D., Lin, B., Qiu, J. A novel image-based cytometry method for autophagy detection in living cells. Autophagy. 8, 1371-1382 (2012).
- Chan, L. L., Wilkinson, A. R., Paradis, B. D., Lai, N. Rapid Image-based Cytometry for Comparison of Fluorescent Viability Staining Methods. Journal of Fluorescence. 22, 1301-1311 (2012).
- Chan, L. L., Zhong, X., Pirani, A., Lin, B. A novel method for kinetic measurements of rare cell proliferation using Cellometer image-based cytometry. J. Immunol. Methods. 377, 8-14 (2012).
- Chan, L. L., Zhong, X., Qiu, J., Li, P. Y., Lin, B. Cellometer Vision as an alternative to flow cytometry for cell cycle analysis, mitochondrial potential, and immunophenotyping. Cytom. Part A. 79, 507-517 (2011).
- Hull, C. M., Johnson, A. D. Identification of a mating type-like locus in the asexual pathogenic yeast Candida albicans. Science. 285, 1271-1275 (1999).
- Berkes, C. A., Chan, L. L., Wilkinson, A., Paradis, B. Rapid Quantification of Pathogenic Fungi by Cellometer Image-Based Cytometry. J. Micro. Meth. 91, 468-476 (2012).
- Nguyen, V. Q., Sil, A. Temperature-induced switch to the pathogenic yeast form of Histoplasma capsulatum requires Ryp1, a conserved transcriptional regulator. Proc. Natl. Acad. Sci. U.S.A. 105 (12), 4880-4885 (2008).
- Wenisch, C., Linnau, K. F., Parschalk, B., Zedtwitz-Liebenstein, K., Georgopoulos, A. Rapid susceptibility testing of fungi by flow cytometry using vital staining. Journal of Clinical Microbiology. 35, 5-10 (1997).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados