Method Article
El uso de citometría de imagen para la cuantificación de hongos patógenos en asociación con las células huésped
En este artículo
Resumen
Aquí, demostramos cómo citometría de imagen se puede utilizar para la cuantificación de hongos patógenos en asociación con células huésped en cultivo. Esta técnica se puede utilizar como una alternativa a la CFU enumeración.
Resumen
Los estudios sobre los mecanismos de la patogénesis celulares de levaduras patógenas tales como Candida albicans, Histoplasma capsulatum y Cryptococcus neoformans emplean comúnmente infección de huéspedes mamíferos o células huésped (es decir, los macrófagos), seguido por la cuantificación de levadura utilizando formadoras de colonias unidad de análisis o la citometría de flujo. Mientras unidad formadora de colonias enumeración ha sido el método más comúnmente utilizado en el campo, esta técnica tiene desventajas y limitaciones, incluyendo el crecimiento lento de algunas especies de hongos en medios sólidos y eficiencias bajas y / o variable de chapado, lo cual es de particular preocupación cuando se comparan el crecimiento de las cepas de tipo salvaje y mutante. La citometría de flujo puede proporcionar una rápida información cuantitativa con respecto a viabilidad de la levadura, sin embargo, la adopción de la detección de citometría de flujo para las levaduras patógenas se ha visto limitado por un número de razones prácticas, incluyendo su alto costo y las consideraciones de seguridad de la biotecnología. Este sentido, demuestran una imagen basada enmetodología de citometría de uso de la Visión Cellometer (Nexcelom Bioscience, LLC) para la cuantificación de levaduras patógenas viables en co-cultivo con macrófagos. Nuestros estudios se centran en la detección de dos hongos patógenos humanos: Histoplasma capsulatum y Candida albicans H.. capsulatum coloniza los macrófagos alveolares mediante la replicación dentro del fagosoma macrófagos, y aquí, nos cuantitativamente evaluar el crecimiento de H. levaduras capsulatum en RAW 264,7 macrófagos con naranja de acridina / tinción con yoduro de propidio en combinación con citometría de imagen. Nuestro método recapitula fielmente las tendencias de crecimiento, medido por unidad formadora de colonia tradicional enumeración, pero con un aumento significativo de sensibilidad. Además, se evalúa directamente la infección de los macrófagos en vivo con una cepa que expresa GFP de C. albicans. Nuestra metodología ofrece un medio rápido, preciso, y económico para la detección y cuantificación de hongos patógenos humanos importantes en asociación ingeniocélulas huésped h.
Introducción
Estudios de hongos patógenos en asociación con sus anfitriones y / o células huésped a menudo requieren la cuantificación de células fúngicas viables durante un transcurso de tiempo o bajo diferentes condiciones de infección. Enumeración de las unidades formadoras de colonias (UFC) es el método estándar por el cual se ha medido el número de células de hongos viables, sin embargo, esta técnica tiene varios inconvenientes y limitaciones. En primer lugar, muchas especies de hongos son de crecimiento lento. El crecimiento de colonias visibles en medios sólidos puede tardar 1-2 semanas, disminuyendo significativamente el ritmo de la investigación. En segundo lugar, la manipulación de las muestras durante chapado UFC es un proceso laborioso, ya que varias diluciones deben ser chapados para asegurar un número contable de colonias. En tercer lugar, el número de UFC es típicamente menor que el número de organismos viables sembradas debido a que la eficiencia de plaqueo es muy por debajo de 100%. Por ejemplo, la eficiencia de la galjanoplastia para el hongo dimórfico Histoplasma capsulatum patógeno puede ser tan alta como 90%, pero son habitualmente tan bajo como30% y son aún más baja (10%) para el relacionado con hongos dimorfo Paracoccidioides brasiliensis 1, 2. Eficiencia de plaqueo para Candida albicans también está sujeta a la variabilidad 3. Por último, el análisis de CFU representa sólo las células en vivo y dividiendo activamente capaces de establecer el crecimiento en medios sólidos, mientras que en muchas situaciones, sería útil para determinar la presencia y concentración de las células muertas inactivos y / o metabólicamente.
Anteriormente, los métodos de citometría de flujo para la cuantificación de varias especies de hongos patógenos ha sido descrito 4-6. Sin embargo, debido a problemas de contención de bioseguridad relacionados con el uso de bioseguridad de nivel 2 (BSL2) o patógenos BSL3 nivel de citómetros de flujo compartidas, la adopción de esta técnica ha sido limitada. Al igual que la citometría de flujo, citometría de imagen es un método sensible y rápido de cuantificación celular. Sin embargo, la citometría de imagen se puede realizar a una fracción del costo con resultados comparables 7 -11. Aquí, se describen los métodos para realizar citometría de imagen de hongos patógenos en asociación con las células huésped. Demostramos nuestros métodos utilizando dos hongos patógenos humanos: Histoplasma capsulatum y Candida albicans H.. capsulatum es un hongo patógeno dimórfico que causa la enfermedad de las vías respiratorias, en los seres humanos, que crece como levadura en ciernes y se replica dentro de los macrófagos alveolares Candida albicans es una especie comensal humanos, que en ocasiones causa la candidiasis.. Se demuestra que la citometría de imagen permite una rápida cuantificación de estas levaduras, junto con la capacidad de visualización.
Protocolo
1. La infección de macrófagos con H. capsulatum, C. albicans
- 16 horas antes de la infección, los macrófagos de semillas a la densidad deseada en placas de 24 pocillos. En este protocolo, se utilizó una densidad de células de 3,0 x 10 5 / pocillo.
- Añadir células de hongos en crecimiento de fase log a una multiplicidad de infección deseada (MOI). Este protocolo se puede acomodar una variedad de MOI (0,2-5,0). Para la infección de macrófagos RAW264.7 con H. capsulatum, se utilizó un MOI de 0,2.
- Después de 1,5 horas para permitir la fagocitosis, los macrófagos lavar tres veces con PBS para eliminar los hongos extracelulares.
- Incubar obtener el número deseado de horas antes del análisis de la muestra. En una MOI baja (0,2 en nuestro experimento), células de macrófagos infectados permanecen viables durante varios días, y las muestras pueden ser analizadas aproximadamente cada 12-24 horas.
2. Lisis del macrófago
- Para liberar a los hongos de los macrófagos, retire los medios, lavar 3 veces con PBS, yañadir 0,5 ml de agua estéril. En estas condiciones, los macrófagos y lisar células fúngicas permanecerán intactos.
- Incubar durante 5 min a temperatura ambiente.
- Traslado lisado de tubo estéril, mantener en hielo.
- Transferencia de 20 l de lisado a un tubo separado, a continuación, añadir 20 l AO / solución PI. Vaya directamente al paso 5: "Preparación de la muestra para el análisis de citometría de imagen"
3. CFU Plating
- Realizar dilución de diez veces de los lisados (de la etapa 2.3) en los medios de comunicación.
- Placa 100 l de cada dilución por duplicado, en placas HMM-agarosa. Incubar las placas en una cámara humidificada a 37 ° C con 5% de CO 2 durante 7-8 días.
- Recuento manual de colonias en las placas que muestran un mínimo de 100 y un máximo de 1.000 colonias distintas.
4. Visualización de hongos dentro de los macrófagos en Vivo
- Para recoger los macrófagos vivos, lavar las células 3 veces con PBS. Añadir 0,5 ml de PBS y se incuba durante 30 min a 4 ° C.
- Para eliminar los macrófagos de los pocillos de cultivo de tejidos, pipetear suavemente hacia arriba y hacia abajo varias veces. Transfiera el líquido en el tubo estéril, mantener en hielo.
- Transferencia de 20 l de muestra a un tubo separado, a continuación, añadir 20 l AO / solución PI. Vaya directamente al paso 5: "Preparación de muestras para análisis de citometría de imagen".
5. Preparación de muestras para análisis de citometría de imagen
- Pipetear la muestra objetivo a fondo y después transferir 20 l de muestra en la cámara de recuento celular desechable.
- Permita que las células se asienten en la cámara durante 30 segundos.
- Insertar cámara de recuento en el citómetro de imagen.
6. Configuración del instrumento Cellometer
- Inserte la óptica de fluorescencia Módulos: VB-535-402 y VB-660-502 en el sistema y asegurarse de que están bloqueados en su lugar.
- VB-535-402 (excitación a 475 nm, emisión a 535 nm) se utiliza para el naranja de acridina y detección de GFP.
- VB-660-502 (excitatien a 540 nm, emisión a 660 nm) se utiliza para la detección de yoduro de propidio.
- Encienda el citómetro de imagen y abrir el software que lo acompaña.
7. Configuración del software Cellometer
- Seleccione el preset "Tipo de ensayo" y "Tipo de la célula" en el menú desplegable de ensayo.
- Para la viabilidad, el ensayo se optimiza para el naranja de acridina y la detección de yoduro de propidio.
- Para la detección de la infección por Candida albicans, el ensayo se optimiza para la detección de GFP.
- Seleccione "Opciones" en la parte superior y haga clic en "Take imagen de fondo", y permitir que se complete la operación.
- Haga clic en "Vista previa de imagen de campo claro".
8. Procedimiento de adquisición de imágenes
- Inserte el módulo de la muestra preparada en el citómetro de imagen.
- Utilice el botón de enfoque y ajustar el enfoque.
- Una vez en el foco, haga clic en "Cuenta", y permitir que la operación de adquisición de imágenes para completar.
- RemoVe la cámara de recuento desechables y disponer de manera adecuada.
9. Análisis de los datos de imagen
- La concentración y la medición de la viabilidad
- Una vez que el recuento se ha completado, la concentración y la viabilidad de las células diana se muestran en la página de resultados.
- Haga clic en "Exportar" para exportar los datos en FCS Express 4 para el análisis de la población de células de las buenas prácticas agrarias en el Candida albicans experimento infección.
- FCS análisis expreso de las células infectadas por Candida albicans-
- Importe el archivo ". NXDAT" en FCS Express 4 y la trama de los resultados en un histograma de fluorescencia.
- Aplicar puerta población de células para el histograma para determinar los porcentajes de la población de células infectadas por Candida albicans.
Resultados
Se utilizó el Cellometer Vision imagen citómetro para controlar el crecimiento de H. capsulatum en células de macrófagos. Las células RAW 264.7 fueron infectados con H. células de levadura capsulatum y en varios puntos de tiempo, las muestras fueron sometidas a tinción AO / PI seguida de análisis de citometría basada en imágenes. En paralelo, las muestras se analizaron por enumeración tradicional UFC. En cada punto de tiempo, las muestras se incubaron en agua para lisar los macrófagos, y las células de levadura liberadas fueron identificados por el software de Visión Cellometer (Figuras 1a y 1b). Observamos tendencias altamente comparables en la concentración de las levaduras viables tal como se detectó por análisis de citometría de imagen basada en y enumeración UFC en cada punto de tiempo (Figura 1c). Como era de esperar, el número absoluto de levaduras vivas detectados por AO / PI tinción fue consistentemente mayor que el número de levaduras capaces de la formación de colonias en medio sólido según la evaluación de análisis de CFU, destacando signonúmeros ificant de células viables pero no cultivables.
Visualización de células fúngicas dentro de los macrófagos en vivo puede llevarse a cabo utilizando GFP-expresando cepas de levadura. Macrófagos derivados de médula ósea (BMDM) se infectaron con C. células de levadura albicans que expresan la proteína fluorescente verde (GFP) bajo el control del promotor ADH1 12 (Figura 2a). La infección de BMDM con GFP-C albicans levaduras a multiplicidades de infección (MOI) que van desde 0,1 hasta 10 correspondían a los aumentos incrementales en la intensidad de GFP dentro de los macrófagos infectados (Figura 2a), así como el porcentaje de total de los macrófagos infectados (Figura 2b).
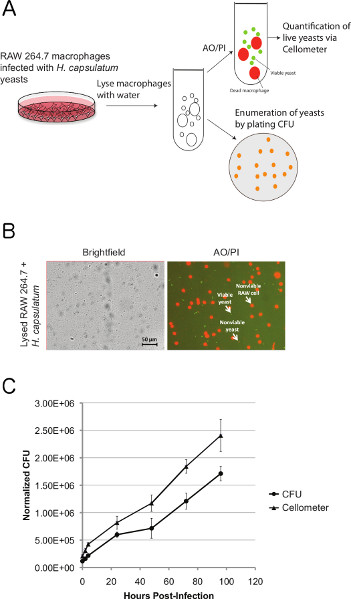
Figura 1. La comparación de análisis de citometría de imagen basado en UFC enumeración de H. capsulatum durante la infección in vitro de macrófagos RAW 264.7. (a) RAW 264,7 macrófagos fueron infectados con H. levaduras capsulatum en una MOI de 0,2. En intervalos de 24 h después de la infección, los macrófagos fueron zadas y levaduras viables evaluados por AO / PI tinción en paralelo con CFU enumeración. (B) Representante de campo claro (BR) y FL1/FL2 (verde / rojo) imágenes combinadas de AO / PI teñido H . capsulatum liberado de lisis macrófagos RAW 264.7. Un algoritmo de segmentación contaba sólo levaduras positivos AO y la PI en las imágenes fluorescentes, mientras que los grandes PI-manchados RAW 264,7 macrófagos (rojo) están excluidos. Las imágenes fueron capturadas con una lupa 10X. (C) La comparación directa de la proliferación de la levadura según la evaluación de AO / PI tinción y recuento CFU. El análisis de regresión lineal de las tendencias de crecimiento tal como se mide por análisis de CFU y rendimiento Cellometer R 2 = 0,9927./ Files/ftp_upload/50599/50599fig1large.jpg "target =" _blank "> Haga clic aquí para ver más grande la figura.

Figura 2. La cuantificación de la infección BMDM con una cepa que expresa GFP de C. albicans. (a) Capturado fluorescentes (media) imágenes de GFP-C campo claro (BR) (arriba) y albicans infectados BMDMs a aumentar la MOI. Los histogramas de la intensidad de fluorescencia muestran un aumento en la intensidad de fluorescencia de GFP medida que aumenta la MOI, lo que representa un aumento en el número C. albicans asociados con células BMDM (parte inferior). El software identifica BMDMs infectados en base a la aplicación de una marca lineal en la intensidad de la fluorescencia inicial mostrada por el control de la población no infectada (MOI = 0). (B) Porcentaje infection en función de MOI, que muestra un aumento en BMDMs infectados como MOI aumenta. Haz clic aquí para ver más grande la figura .
Discusión
Citometría de imagen permite al usuario capturar imágenes de alta calidad y el uso de software especializado, realizar una rápida cuantificación de células. Un reto potencial para la adopción de la citometría de imagen en el campo de la patogénesis microbiana es que los microbios a ser contadas están presentes en una población mixta de células, incluyendo las células huésped de mamífero. Aquí, demostramos que la citometría de imagen se puede utilizar para la cuantificación de levaduras patógenas viables durante la infección de macrófagos in vitro. Nuestra metodología no sólo recapitula fielmente las tendencias en el crecimiento de hongos y la viabilidad observado durante los ensayos de infección in vitro de macrófagos, pero también muestra una mejor sensibilidad en comparación con el tradicional ensayo de UFC 13.
Citometría de imagen ofrece varias ventajas prácticas en comparación con ya sea enumeración UFC o citometría de flujo para la cuantificación de células de levadura. Cuando enchapado UFC, personal de laboratorio debe plato varias DILUTiones con el fin de asegurar un número contable de las células en cada placa, que puede ser una labor muy intensa. Citometría de imagen elimina la necesidad de múltiples diluciones. Anteriormente, hemos demostrado que la formación de colonias en medios sólidos es a menudo variables, y en concentraciones muy bajas setas pueden dejar de formar colonias en absoluto, mientras que la citometría de imagen permite la enumeración de las células, independientemente de la concentración 13. La capacidad para detectar y contar los hongos a concentraciones muy bajas puede ser muy útil durante las primeras etapas de una infección de dosis baja, o durante el despacho, cuando el número y la viabilidad puede estar en el margen de detección. Además, los datos generados por citometría de imagen están disponibles al instante, mientras que las colonias pueden tardar varios días en estar visible. Mientras que muchas especies de hongos se pueden detectar mediante citometría de flujo, la posibilidad de contaminación cruzada es una preocupación en instalaciones compartidas. La cámara de recuento utilizado en este estudio ofrece una ventaja significativa en términos de seguridad de la biotecnología, ya que es disposable y autónomo. Además, la citometría de imagen ofrece las ventajas prácticas de precio más bajo y una huella más pequeña en comparación con la citometría de flujo convencional, que puede ser importante para la investigación más pequeños laboratorios 11.
Además de sus ventajas prácticas, citometría de imagen de células de levadura proporciona información que CFU enumeración no puede. En primer lugar, CFU enumeración es sólo capaz de detectar los hongos que son capaces de establecer el crecimiento de colonias en el medio elegido, que puede no representar con exactitud el número de hongos viables presentes en un experimento de infección en tiempo real. Además, la capacidad de establecer el crecimiento de colonias en medios sólidos puede diferir cuando se comparan las cepas de tipo salvaje y mutante de hongos, que pueden ser una fuente de sesgo experimental 14, y estos sesgos ocultos puede ser puesto de manifiesto por comparación de la citometría de imagen y cuantificación de UFC cepas de tipo salvaje y mutante. Una vez que el comportamiento de una cepa particular, se ha caracterizado por boª CFU enumeración y citometría de imagen, creemos que citometría de imagen puede ser utilizado como un medio alternativo para la cuantificación de hongos.
Una limitación de la citometría de imagen, en general, es que no permite colección de tantos puntos de datos como la citometría de flujo. Dado que el citómetro de imagen se deriva de datos cuantitativos a partir de un número limitado de imágenes capturadas, es difícil para el sistema para recoger datos sobre cientos de miles a millones de células por muestra. Sin embargo, esta limitación puede ser evitado mediante la agrupación de múltiples muestras en un conjunto de datos para el análisis con el fin de llegar a los puntos de datos similares a los de un citómetro de flujo. También nos gustaría hacer hincapié en que el método de tinción AO / PI utilizado aquí representa una manera de medir la viabilidad celular, y no indica necesariamente la vitalidad celular y / o la capacidad de establecer el crecimiento en medio sólido en cualquier muestra dada. Por lo tanto, mientras que nuestro método proporciona un método fácil de cuantificación en vivo / muerto de levaduras, seTambién será interesante investigar el uso de la citometría de imagen en combinación con indicadores del metabolismo celular tales como FUN-1 15.
Software de citometría de imagen identifica y cuenta células de interés basado en el tamaño y la forma, y por lo tanto, es absolutamente crítico que estos parámetros se ajustan cuidadosamente durante cada experimento. Al realizar citometría de imagen con una nueva cepa de levadura, se sugiere que una primera Comparación de los datos generados a partir de un cultivo de levadura pura a los datos generados a partir de contar una mezcla de levaduras, además de células huésped para asegurar que los parámetros se ajustan de tal manera que el software puede distinguir entre los dos tipos de células.
En el futuro, este trabajo será la base para un mayor desarrollo de los métodos para detectar y cuantificar microorganismos mediante citometría de imagen. Por ejemplo, el desarrollo de métodos de citometría de imagen para detectar hongos dentro de homogeneizados de órganos será de gran uso en el campo. Otro desafío futuro será desarrolle y refinar los métodos de imagen y de software de tal manera que puede ser utilizado para la cuantificación de las células bacterianas de citometría. Con el avance de los sistemas ópticos, cámaras digitales, y una amplia variedad de manchas fluorescentes en el campo, la capacidad de la imagen y analizar la población de bacterias puede ser implementado en citómetros de imagen, que puede proporcionar un método más rápido para la concentración y la viabilidad o la vitalidad de medición en comparación con el UFC o métodos de densidad óptica. En resumen, el trabajo presentado aquí es una prueba de concepto mostrando que la citometría de imagen es un método sensible para la cuantificación de levaduras patógenas. La sofisticación de software de análisis de citometría de imagen basado ofrece la oportunidad de analizar las interacciones entre las células huésped y hongos patógenos de una manera altamente cuantitativo. Esta tecnología se puede adaptar para hacer frente a una serie de preguntas de investigación en el campo de la patogénesis fúngica.
Divulgaciones
Los autores Leo Li-Ying Chan y Benjamin Paradis son empleados de Nexcelom Bioscience LLC.
Materiales
| Name | Company | Catalog Number | Comments |
| REAGENTS | |||
| DMEM | Life Technologies | 11965-084 | |
| Fetal Bovine Serum | Life Technologies | 16000044 | |
| AO/PI Solution | Nexcelom Bioscience | CSK-0102 | |
| Disposable Counting Chamber | Nexcelom Bioscience | CHT4-SD100 | |
| EQUIPMENT | |||
| Cellometer Vision | Nexcelom Bioscience | ||
| Cellometer Vision Software | Nexcelom Bioscience | ||
Referencias
- Worsham, P. L., Goldman, W. E. Quantitative plating of Histoplasma capsulatum without addition of conditioned medium or siderophores. J. Med. Vet. Mycol. 26, 137-143 (1988).
- Goihman-Yahr, M., Pine, L., Albornoz, M. C., Yarzabal, L., de Gomez, M. H., San Martin, B., Ocanto, A., Molina, T., Convit, J. Studies on plating efficiency and estimation of viability of suspensions of Paracoccidioides brasiliensis yeast cells. Mycopathologia. 71, 73-83 (1980).
- Bhatti, M. A., Hjertstedt, J., Hahn, B. L., Sohnle, P. G. Inefficient delivery of yeast cells as an explanation for reduced plating efficiency of Candida albicans. Med. Mycol. 40, 465-469 (2002).
- Chang, W. L., vander Heyde, H. C., Klein, B. S. Flow cytometric quantitation of yeast: a novel technique for use in animal model work and in vitro immunologic assays. Journal of Immunological Methods. 211, 51-63 (1998).
- Green, L., Petersen, B., Steimel, L., Haeber, P., Current, W. Rapid determination of antifungal activity by flow cytometry. Journal of Clinical Microbiology. 32, 1088-1091 (1994).
- Kirk, S. M., Callister, S. M., Lim, L. C., Schell, R. F. Rapid susceptibility testing of Candida albicans by flow cytometry. Journal of Clinical Microbiology. 35, 358-363 (1997).
- Chan, L. L. -. Y., Lai, N., Wang, E., Smith, T., Yang, X., Lin, B. A rapid detection method for apoptosis and necrosis measurement using the Cellometer imaging cytometry. Apoptosis. 16, 1295-1303 (2011).
- Chan, L. L. -. Y., Shen, D., Wilkinson, A. R., Patton, W., Lai, N., Chan, E., Kuksin, D., Lin, B., Qiu, J. A novel image-based cytometry method for autophagy detection in living cells. Autophagy. 8, 1371-1382 (2012).
- Chan, L. L., Wilkinson, A. R., Paradis, B. D., Lai, N. Rapid Image-based Cytometry for Comparison of Fluorescent Viability Staining Methods. Journal of Fluorescence. 22, 1301-1311 (2012).
- Chan, L. L., Zhong, X., Pirani, A., Lin, B. A novel method for kinetic measurements of rare cell proliferation using Cellometer image-based cytometry. J. Immunol. Methods. 377, 8-14 (2012).
- Chan, L. L., Zhong, X., Qiu, J., Li, P. Y., Lin, B. Cellometer Vision as an alternative to flow cytometry for cell cycle analysis, mitochondrial potential, and immunophenotyping. Cytom. Part A. 79, 507-517 (2011).
- Hull, C. M., Johnson, A. D. Identification of a mating type-like locus in the asexual pathogenic yeast Candida albicans. Science. 285, 1271-1275 (1999).
- Berkes, C. A., Chan, L. L., Wilkinson, A., Paradis, B. Rapid Quantification of Pathogenic Fungi by Cellometer Image-Based Cytometry. J. Micro. Meth. 91, 468-476 (2012).
- Nguyen, V. Q., Sil, A. Temperature-induced switch to the pathogenic yeast form of Histoplasma capsulatum requires Ryp1, a conserved transcriptional regulator. Proc. Natl. Acad. Sci. U.S.A. 105 (12), 4880-4885 (2008).
- Wenisch, C., Linnau, K. F., Parschalk, B., Zedtwitz-Liebenstein, K., Georgopoulos, A. Rapid susceptibility testing of fungi by flow cytometry using vital staining. Journal of Clinical Microbiology. 35, 5-10 (1997).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados