Method Article
Verwendung Bildzytometrie zur Quantifizierung von pathogenen Pilzen in Verbindung mit Wirtszellen
In diesem Artikel
Zusammenfassung
Hier zeigen wir, wie Bildzytometrie zur Quantifizierung von pathogenen Pilzen in Verbindung mit Wirtszellen in der Kultur verwendet werden. Diese Technik kann als Alternative zu CFU Aufzählung verwendet werden.
Zusammenfassung
Untersuchungen der zellulären Pathogenese pathogener Hefen wie Candida albicans, Histoplasma capsulatum und Cryptococcus neoformans verwenden üblicherweise Infektion von Säugetier-Hosts oder Wirtszellen (zB Makrophagen) durch Hefe Quantifizierung mittels koloniebildende Einheit Analyse oder Durchflusszytometrie gefolgt. Während koloniebildende Einheit Aufzählung ist das am häufigsten verwendete Verfahren auf dem Gebiet hat diese Technik Nachteile und Beschränkungen, einschließlich der langsamen Wachstum einiger Pilzarten auf festen Medien und nieder-und / oder variable Beschichtung Wirkungsgrade, die von besonderer Bedeutung ist bei einem Vergleich Wachstum von Wildtyp-und Mutanten. Durchflusszytometrie können schnelle quantitative Informationen über Hefelebensfähigkeit bieten hat jedoch Verabschiedung durchflusszytometrischen Nachweis für pathogene Hefen für eine Reihe von praktischen Gründen, einschließlich seiner hohen Kosten und die biologische Sicherheit Überlegungen beschränkt. Hier zeigen wir eine bildbasiertecytometric Methodik mit der Cellometer Vision (Nexcelom Bioscience, LLC) für die Quantifizierung von lebensfähigen pathogenen Hefen in Co-Kultur mit Makrophagen. Histoplasma capsulatum und Candida albicans H.:. Unsere Studien auf dem Nachweis von zwei menschlichen Pilzpathogene konzentrieren capsulatum kolonisiert Alveolarmakrophagen durch die Replikation innerhalb der Makrophagen phagosome, und hier haben wir quantitativ das Wachstum von H. capsulatum Hefen in RAW 264.7 Makrophagen mit Acridinorange / Propidiumiodidfärbung in Kombination mit Bildzytometrie. Unsere Methode treu rekapituliert Wachstumstrends wie durch traditionelle Kolonie bildende Einheit Aufzählung gemessen, aber mit deutlich erhöhter Empfindlichkeit. Zusätzlich haben wir direkt beurteilen Infektion von lebenden Makrophagen mit einem GFP-exprimierenden Stamm von C. albicans. Unsere Methodik bietet eine schnelle, genaue und kostengünstige Mittel zur Detektion und Quantifizierung von wichtigen menschlichen pilzliche Erreger in Verbindung with Wirtszellen.
Einleitung
Studien von pathogenen Pilzen in Verbindung mit ihren Gastgebern und / oder Wirtszellen erfordern oft Quantifizierung der lebensfähigen Pilzzellen über einen Zeitverlauf oder unter verschiedenen Bedingungen Infektion. Zählung der koloniebildenden Einheiten (CFU) der Standard-Methode, durch die die Zahl der lebensfähigen Pilzzellen gemessen wurde, jedoch hat dieses Verfahren einige Nachteile und Einschränkungen. Erstens sind viele Pilzarten langsam wachsende. Das Wachstum der sichtbaren Kolonien auf festen Medien kann 1-2 Wochen, deutlich verlangsamt das Tempo der Forschung. Zweitens ist Manipulation von Proben während der CFU Beschichtung ein langwieriger Prozess, da mehrere Verdünnungen müssen beschichtet, um eine zählbare Anzahl der Kolonien zu gewährleisten. Drittens, ist die Anzahl der CFU typischerweise geringer als die Zahl von lebensfähigen Organismen, weil die Beschichtung überzogen Wirkungsgrad deutlich unter 100%. Zum Beispiel kann Plattieren Wirkungsgrade für die dimorphic Pilzpathogen Histoplasma capsulatum so hoch wie 90%, aber routinemäßig so niedrig wie30% und sogar noch niedriger (10%) für die entsprechende Pilz dimorph Paracoccidioides brasiliensis 1, 2. Plating Effizienz für Candida albicans ist auch Gegenstand Variabilität 3. Schließlich entfallen CFU Analyse für nur leben und sich aktiv teilenden Zellen, die zur Festlegung Wachstum auf festen Medien, wobei in vielen Situationen, wäre es nützlich, um die Anwesenheit und Konzentration von toten und / oder metabolisch inaktiven Zellen zu bestimmen.
Zuvor hat durchflusszytometrischen Methoden zur Quantifizierung von mehreren pathogenen Pilzarten wurden 4-6 beschrieben. Doch aufgrund Biosicherheit Containment Probleme bei der Verwendung Sicherheitsstufe 2 (BSL2) oder BSL3-Level Krankheitserreger auf gemeinsamen Durchflusszytometern beteiligt hat Annahme dieser Technik beschränkt. Wie Durchflusszytometrie ist Bildzytometrie eine sensible und schnelle Methode der Zelle Quantifizierung. Allerdings kann Bildzytometrie zu einem Bruchteil der Kosten mit vergleichbaren Ergebnissen 7 durchgeführt werden -11. Hier beschreiben wir Verfahren zur Durchführung Bildzytometrie von pathogenen Pilzen in Verbindung mit Wirtszellen. Wir demonstrieren unsere Methoden mit zwei menschlichen Pilzpathogene: Histoplasma capsulatum und Candida albicans H.. capsulatum ist ein dimorphic Pilzpathogen, die Erkrankung der Atemwege verursacht, in den Menschen, es wächst wie Bäckerhefe und repliziert in Alveolarmakrophagen Candida albicans ist ein Mensch kommensalen Arten, die gelegentlich verursacht Candidiasis.. Wir zeigen, dass Bildzytometrie schnelle Quantifizierung dieser Hefen ermöglicht, zusammen mit Visualisierungsmöglichkeiten.
Protokoll
1. Die Infektion von Makrophagen mit H. capsulatum, C. albicans
- 16 Stunden vor der Infektion, Samen Makrophagen an gewünschten Dichte in 24-Well-Platten. In diesem Protokoll wurde eine Dichte von 3,0 x 10 5 Zellen / Vertiefung verwendet.
- In Pilzzellen in log-Wachstumsphase bei einer gewünschten Multiplizität der Infektion (MOI). Dieses Protokoll bietet Platz für eine Reihe von MOI (0,2-5,0). Für die Infektion von Makrophagen mit RAW264.7 H. capsulatum, verwendeten wir eine MOI von 0,2.
- Nach 1,5 Stunden für Phagozytose erlauben, waschen Makrophagen dreimal mit PBS an extrazelluläre Pilze zu entfernen.
- Inkubieren gewünschte Anzahl von Stunden vor Probenanalyse. Bei niedrigen MOI (0.2 in unserem Experiment), bleiben infizierten Makrophagen für mehrere Tage lebensfähig, und die Proben können etwa alle 12-24 Stunden analysiert werden.
2. Makrophagen Lysis
- Um Pilze aus Makrophagen befreien, entfernen Medien, 3 x waschen mit PBS, und0,5 ml sterilem Wasser. Unter diesen Bedingungen wird Makrophagen lysieren und Pilzzellen bleiben erhalten.
- Inkubieren für 5 min bei Raumtemperatur gerührt.
- Übertragen Lysat sterilen Röhrchen, halten auf dem Eis.
- Übertragen Sie 20 ul Lysat zu einem separaten Röhrchen, dann fügen Sie 20 ul AO / PI-Lösung. Gehen Sie direkt zu Schritt 5: "Probenvorbereitung für Bild Cytometric Analysis"
3. CFU Plating
- Führen zehnfache Verdünnung der Lysate (aus Schritt 2.3) in den Medien.
- Platte 100 ul jeder Verdünnung in zweifacher Ausfertigung auf HMM-Agaroseplatten. Die Platten in einer feuchten Kammer bei 37 ° C mit 5% CO 2 für 7-8 Tage.
- Manuelles zählen Kolonien auf Platten Darstellung von mindestens 100 und maximal 1000 verschiedene Kolonien.
4. Visualization of Fungi innerhalb Live-Makrophagen
- Um Live-Makrophagen sammeln, waschen Sie die Zellen 3 mal mit PBS. 0,5 ml PBS und Inkubation für 30 min bei 4 ° C.
- Um Makrophagen aus Gewebekulturvertiefungen entfernen, sanft und Abpipettieren mehrmals. Übertragen Flüssigkeit sterilen Röhrchen, halten auf dem Eis.
- Übertragen Sie 20 ul Probe auf einem separaten Röhrchen, dann fügen Sie 20 ul AO / PI-Lösung. Gehen Sie direkt zu Schritt 5: "Probenvorbereitung für Bild Cytometric Analysis".
5. Probenvorbereitung für Bild Cytometric Analysis
- Pipettieren Sie die Target-Probe gründlich und übertragen dann 20 ul Probe in die Zelle Einweg-Zählkammer.
- Lassen Sie die Zellen in der Kammer für 30 Sekunden absetzen.
- Legen Zählkammer in das Bild cytometer.
6. Cellometer Instrument Setup
- Legen Sie die Fluoreszenzoptik Module: VB-535 bis 402 und VB-660 bis 502 in das System und sicherstellen, dass sie an Ort und Stelle sind gesperrt.
- VB-535 bis 402 (Anregung bei 475 nm, Emission bei 535 nm) ist für Acridinorange und GFP-Erkennung verwendet.
- VB-660 bis 502 (excitatiauf bei 540 nm wird die Emission bei 660 nm) für Propidiumiodid Detektion verwendet.
- Schalten Sie auf das Bild cytometer und öffnen die zugehörige Software.
7. Cellometer Software Setup
- Wählen Sie die Vorgabe "Assay Type" und "Cell Type" in der Assay Dropdown-Menü.
- Für Lebensfähigkeit wird der Assay für Acridinorange und Propidiumiodid Erkennung optimiert.
- Für den Nachweis von Candida albicans-Infektion wird der Assay für GFP Erkennung optimiert.
- Wählen Sie "Optionen" auf der Oberseite und klicken Sie auf "Take Background Image", und lassen Sie den Vorgang abgeschlossen ist.
- Klicken Sie auf "Vorschau Hellfeldbild".
8. Image Acquisition Vorgehensweise
- Legen Sie die vorbereiteten Probe Kammer in das Bild cytometer.
- Verwenden Sie den Fokus Knopf und stellen Sie den Fokus.
- Einmal im Fokus, auf "Count" klicken, und lassen Sie die Bildaufnahme Vorgang abgeschlossen ist.
- Remove die Einweg-Zählkammer und entsorgen.
9. Bild Datenanalyse
- Konzentration und Lebensfähigkeit Messung
- Sobald die Zählung abgeschlossen ist, werden die Konzentration und die Lebensfähigkeit der Zielzellen in der Ergebnis-Seite angezeigt.
- Klicken Sie auf "Export", um die Daten in FCS Express 4 exportieren für Zellpopulation Analyse der GFP in der Candida albicans-Infektion Experiment.
- FCS Express Analyse von Candida albicans-infizierten Zellen
- Importieren Sie die ". NXDAT" Datei in FCS Express 4 und dann das Ergebnis in einem Fluoreszenz-Histogramm.
- Bewerben Zellpopulation Tor zum Histogramm, um die Bevölkerung Prozentsätze der Candida albicans-infizierten Zellen zu bestimmen.
Ergebnisse
Wir nutzten die Cellometer Bild Anblick cytometer um das Wachstum von H. überwachen capsulatum in Makrophagen. RAW 264.7 Zellen wurden mit H. infiziert capsulatum Hefezellen und zu verschiedenen Zeitpunkten wurden Proben AO / PI-Färbung von bildbasierten durchflusszytometrische Analyse, unterzogen. Parallel dazu wurden Proben von traditionellen CFU Aufzählung analysiert. Zu jedem Zeitpunkt wurden die Proben in Wasser inkubiert, um Makrophagen zu lysieren und die freigesetzte Hefezellen wurden durch die Cellometer Vision-Software (1a und 1b) identifiziert. Wir beobachten sehr vergleichbar Trends in der Konzentration von lebensfähigen Hefen wie bildbasierten durchflusszytometrische Analyse und CFU Aufzählung zu jedem Zeitpunkt (1c) festgestellt. Wie erwartet, war die absolute Zahl der lebenden Hefen AO / PI-Färbung nachgewiesen durchweg höher als die Anzahl von Hefen in der Lage Koloniebildung auf einem festen Medium durch CFU-Analyse untersucht, Hervorhebung Zeichenificant Anzahl lebensfähiger, aber nicht kultivierbaren Zellen.
Visualisierung von Pilzzellen in Live-Makrophagen kann unter Verwendung von GFP-exprimierenden Hefestämme werden. Knochenmark-Makrophagen (BMDM) wurden mit C infiziert albicans Hefezellen, grün fluoreszierendes Protein (GFP) unter der Kontrolle des ADH1-Promotors 12 (Fig. 2a). Die Infektion von BMDM mit GFP-C albicans Hefen bei Vielheiten der Infektion (MOI) im Bereich von 0,1 bis 10 entsprach inkrementelle Erhöhung der GFP Intensität in infizierten Makrophagen (Abbildung 2a) sowie Gesamtanteil von infizierten Makrophagen (Abbildung 2b).
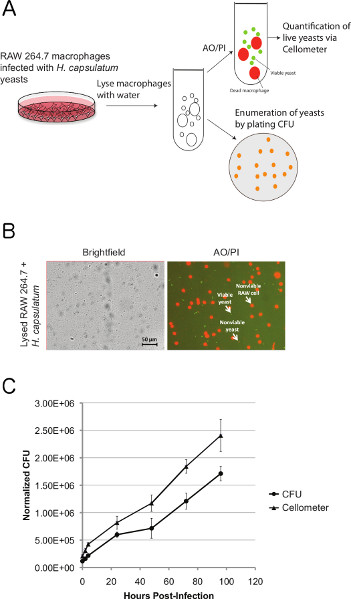
Abbildung 1. Vergleich der bildbasierten cytometric Analyse CFU Aufzählung von H. capsulatum während der in vitro Infektion von RAW 264.7 Makrophagen. (a) RAW 264.7 Makrophagen wurden mit H. infiziert capsulatum Hefen bei einer MOI von 0,2. Bei 24 Stunden-Intervallen nach der Infektion wurden Makrophagen lysiert und lebensfähigen Hefen durch AO / PI-Färbung parallel CFU Aufzählung beurteilt. (B) Vertreter Hellfeld (BR) und FL1/FL2 (grün / rot) kombinierten Bilder von AO / PI gefärbten H . capsulatum aus lysierten RAW 264.7 Makrophagen freigesetzt. Eine Segmentierung Algorithmus gezählt nur AO und PI positive Hefen in den fluoreszierenden Bildern, während die große PI-gefärbten RAW 264.7 Makrophagen (rot) sind ausgeschlossen. Die Bilder wurden unter 10-facher Vergrößerung aufgenommen. (C) Der direkte Vergleich von Hefe Proliferation von AO / PI-Färbung und CFU Aufzählung beurteilt. Lineare Regressionsanalyse der Wachstumstrends wie CFU und Cellometer Analyse Ausbeute R 2 = 0,9927 gemessen./ Files/ftp_upload/50599/50599fig1large.jpg "target =" _blank "> Klicken Sie hier, um eine größere Abbildung anzuzeigen.

Abbildung 2. Quantifizierung BMDM Infektion mit einem GFP-exprimierenden Stamm von C. albicans. (a) Captured Hellfeld (BR) (oben) und Leuchtstofflampen (Mitte) Bilder von GFP-C albicans infiziert BMDMs bei steigender MOI. Histogramme der Fluoreszenzintensitäten zeigen einen Anstieg der GFP-Fluoreszenz Intensität wie die MOI erhöht, was einer Steigerung in der Anzahl C stellt albicans mit BMDM Zellen (unten) verbunden. Die Software ermittelt wurden BMDMs auf Anwendung einer linearen Markierung an der Grundlinie Fluoreszenzintensität von der nicht infizierten Kontrollgruppe (MOI = 0) angezeigt basiert. (B) Prozent infection als Funktion der MOI, die zeigt einen Anstieg in infizierten BMDMs als MOI erhöht. Klicke hier, um eine größere Abbildung anzuzeigen .
Diskussion
Bildzytometrie ermöglicht es dem Benutzer, um qualitativ hochwertige Bilder zu erfassen und mit spezieller Software, führen Sie eine schnelle Quantifizierung von Zellen. Eine mögliche Herausforderung für die Annahme Bildzytometrie im Bereich der mikrobiellen Pathogenese ist, dass die Mikroorganismen zu zählen, die in einer gemischten Population von Zellen, einschließlich Säuger-Wirtszellen sind. Hier zeigen wir, dass Bildzytometrie zur Quantifizierung von lebensfähigen pathogenen Hefen während der in vitro-Makrophagen-Infektion verwendet werden. Unsere Methodik nicht nur treu rekapituliert Trends in Pilzwachstum und Lebensfähigkeit während der in vitro-Assays Makrophagen Infektion beobachtet, sondern auch zeigt verbesserte Empfindlichkeit im Vergleich zu den traditionellen CFU-Assay 13.
Bildzytometrie bietet mehrere praktische Vorteile, wenn sie mit beiden CFU Aufzählung oder Durchflusszytometrie zur Quantifizierung von Hefezellen verglichen. Wenn Galvanisieren CFUs, müssen Laboranten Platte mehrere DILUTIonen, um eine zählbare Anzahl von Zellen auf jeder Platte, die sehr arbeitsintensiv sicherstellen können. Bildzytometrie eliminiert die Notwendigkeit für mehrere Verdünnungen. Zuvor haben wir gezeigt, dass Koloniebildung auf festen Medien oft ist variabel und bei sehr niedrigen Konzentrationen Pilze möglicherweise nicht Kolonien überhaupt bilden, während Bildzytometrie ermöglicht Aufzählung der Zellen unabhängig von Konzentration 13. Die Fähigkeit zu erkennen und zu zählen Pilze in sehr geringen Konzentrationen kann sehr nützlich sein während der frühen Phasen einer low-dose-Infektion, oder beim Abschalten, wenn Zahlen und Lebensfähigkeit kann am Rand der Nachweis sein. Darüber hinaus sind Daten, die von Bildzytometrie erzeugt sofort zur Verfügung, während die Kolonien können mehrere Tage dauern, um sichtbar zu werden. Während viele Pilzarten mittels Durchflusszytometrie nachgewiesen werden kann, ist die Möglichkeit einer Kreuzkontamination ein Anliegen in gemeinsamen Einrichtungen. Die Zählkammer in dieser Studie verwendeten bietet einen signifikanten Vorteil in Bezug auf die biologische Sicherheit, wie es ist disposable und geschlossene. Darüber hinaus bietet Bildzytometrie die praktischen Vorteile von niedrigeren Preisen und einem geringeren Platzbedarf gegenüber herkömmlichen Durchflusszytometrie, die wichtig sein können für kleinere Forschungslabore 11 verglichen.
Neben den praktischen Vorteilen, bietet Bildzytometrie von Hefezellen Informationen, CFU Aufzählung kann nicht. Erstens ist CFU Aufzählung nur in der Lage, Pilze, die in der Lage, Kolonien bewachsene auf dem Medium gewählt wird, die nicht genau darstellen kann die Anzahl lebensfähiger Pilze in einer Infektionsexperiment in Echtzeit festzustellen sind zu erkennen. Auch kann die Fähigkeit zur Kolonie Wachstum auf festen Medien etablieren unterscheiden beim Vergleich von Wildtyp und Mutante Stämme von Pilzen, die eine Quelle der experimentellen Bias 14 sein kann, und diese versteckten Vorurteile können durch Vergleich der Bildzytometrie und CFU Quantifizierung enthüllt werden Wildtyp und Mutanten. Wenn das Verhalten eines bestimmten Stammes durch bo charakterisiertth CFU Aufzählung und Bildzytometrie, glauben wir, dass Bildzytometrie als Alternative mittels Pilz-Quantifizierung verwendet werden.
Eine Einschränkung Bildzytometrie im Allgemeinen ist, dass es nicht möglich Sammlung von so vielen Datenpunkten Durchflusszytometrie. Da das Bild Zytometer leitet quantitative Daten aus einer begrenzten Anzahl von Aufnahmen, ist es schwierig, das System Daten über hundert bis zu mehreren Millionen Zellen pro Probe zu sammeln. Jedoch kann diese Begrenzung durch Gruppieren mehrerer Proben in einem Satz von Daten für die Analyse, um ähnliche Datenpunkte als Durchflusszytometer erreichen umgangen werden. Wir möchten auch betonen, dass die AO / PI-Färbung hier verwendete Methode eine Methode zur Messung der zellulären Lebensfähigkeit darstellt und nicht unbedingt auf zelluläre Vitalität und / oder die Fähigkeit zum Wachstum auf festem Medium in einer bestimmten Probe zu etablieren. Während also unsere Methode bietet eine einfache Methode des Live / Dead Quantifizierung von Hefen, eswird auch interessant sein, die Verwendung von Bildzytometrie in Kombination zu untersuchen mit Zellstoffwechsel Indikatoren wie FUN-1 15.
Bildzytometrie Software identifiziert und zählt Zellen von Interesse basierend auf Größe und Form, und daher ist es unbedingt erforderlich, dass diese Parameter sorgfältig während jedem Experiment eingestellt. Bei der Durchführung Bildzytometrie mit einem neuen Hefestamm, schlagen wir vor, dass eine erste Vergleichsdaten aus einer reinen Hefekulturen, um Daten von der Zählung einer Mischung aus Hefen sowie Wirtszellen erzeugt werden, um sicherzustellen, dass die Parameter so eingestellt werden, dass die Software zwischen unterscheiden erzeugt zwei Zelltypen.
In Zukunft wird diese Arbeit die Grundlage für die weitere Entwicklung von Methoden zur Detektion und Quantifizierung von Mikroorganismen über Bildzytometrie sein. Zum Beispiel wird die Entwicklung von Methoden, um Bild cytometric Pilze innerhalb Orgel Homogenate erkennen von großem Nutzen auf dem Feld sein. Eine weitere Herausforderung für die Zukunft wird sein, dEvelop und verfeinern Bild cytometric Methoden und Software, so dass es für die Quantifizierung von Bakterienzellen verwendet werden. Mit dem Fortschritt in der optischen Systeme, Digitalkameras, und eine Vielzahl von fluoreszierenden Flecken auf dem Feld, die Fähigkeit, Bild und analysieren Bakterienpopulation können in Bild Zytometern umgesetzt werden, die eine schnellere Methode zur Konzentration und Lebensfähigkeit oder Vitalität Messung liefern kann im Vergleich zu der CFU oder optische Dichte Methoden. Zusammenfassend ist die hier vorgestellte Arbeit ein Proof-of-Konzept zeigt, dass Bildzytometrie eine empfindliche Methode zur Quantifizierung von pathogenen Hefen ist. Die Raffinesse der bildbasierten cytometric Analyse-Software bietet die Möglichkeit, Wechselwirkungen zwischen Wirtszellen und pathogene Pilze in einem stark quantitativ zu analysieren. Diese Technik kann für den Bereich von Fragestellungen im Bereich der Pathogenität von Pilzen zu adressieren.
Offenlegungen
Die Autoren Leo Li-Ying Chan und Benjamin Paradis sind Mitarbeiter von Nexcelom Bioscience LLC.
Materialien
| Name | Company | Catalog Number | Comments |
| REAGENTS | |||
| DMEM | Life Technologies | 11965-084 | |
| Fetal Bovine Serum | Life Technologies | 16000044 | |
| AO/PI Solution | Nexcelom Bioscience | CSK-0102 | |
| Disposable Counting Chamber | Nexcelom Bioscience | CHT4-SD100 | |
| EQUIPMENT | |||
| Cellometer Vision | Nexcelom Bioscience | ||
| Cellometer Vision Software | Nexcelom Bioscience |
Referenzen
- Worsham, P. L., Goldman, W. E. Quantitative plating of Histoplasma capsulatum without addition of conditioned medium or siderophores. J. Med. Vet. Mycol. 26, 137-143 (1988).
- Goihman-Yahr, M., Pine, L., Albornoz, M. C., Yarzabal, L., de Gomez, M. H., San Martin, B., Ocanto, A., Molina, T., Convit, J. Studies on plating efficiency and estimation of viability of suspensions of Paracoccidioides brasiliensis yeast cells. Mycopathologia. 71, 73-83 (1980).
- Bhatti, M. A., Hjertstedt, J., Hahn, B. L., Sohnle, P. G. Inefficient delivery of yeast cells as an explanation for reduced plating efficiency of Candida albicans. Med. Mycol. 40, 465-469 (2002).
- Chang, W. L., vander Heyde, H. C., Klein, B. S. Flow cytometric quantitation of yeast: a novel technique for use in animal model work and in vitro immunologic assays. Journal of Immunological Methods. 211, 51-63 (1998).
- Green, L., Petersen, B., Steimel, L., Haeber, P., Current, W. Rapid determination of antifungal activity by flow cytometry. Journal of Clinical Microbiology. 32, 1088-1091 (1994).
- Kirk, S. M., Callister, S. M., Lim, L. C., Schell, R. F. Rapid susceptibility testing of Candida albicans by flow cytometry. Journal of Clinical Microbiology. 35, 358-363 (1997).
- Chan, L. L. -. Y., Lai, N., Wang, E., Smith, T., Yang, X., Lin, B. A rapid detection method for apoptosis and necrosis measurement using the Cellometer imaging cytometry. Apoptosis. 16, 1295-1303 (2011).
- Chan, L. L. -. Y., Shen, D., Wilkinson, A. R., Patton, W., Lai, N., Chan, E., Kuksin, D., Lin, B., Qiu, J. A novel image-based cytometry method for autophagy detection in living cells. Autophagy. 8, 1371-1382 (2012).
- Chan, L. L., Wilkinson, A. R., Paradis, B. D., Lai, N. Rapid Image-based Cytometry for Comparison of Fluorescent Viability Staining Methods. Journal of Fluorescence. 22, 1301-1311 (2012).
- Chan, L. L., Zhong, X., Pirani, A., Lin, B. A novel method for kinetic measurements of rare cell proliferation using Cellometer image-based cytometry. J. Immunol. Methods. 377, 8-14 (2012).
- Chan, L. L., Zhong, X., Qiu, J., Li, P. Y., Lin, B. Cellometer Vision as an alternative to flow cytometry for cell cycle analysis, mitochondrial potential, and immunophenotyping. Cytom. Part A. 79, 507-517 (2011).
- Hull, C. M., Johnson, A. D. Identification of a mating type-like locus in the asexual pathogenic yeast Candida albicans. Science. 285, 1271-1275 (1999).
- Berkes, C. A., Chan, L. L., Wilkinson, A., Paradis, B. Rapid Quantification of Pathogenic Fungi by Cellometer Image-Based Cytometry. J. Micro. Meth. 91, 468-476 (2012).
- Nguyen, V. Q., Sil, A. Temperature-induced switch to the pathogenic yeast form of Histoplasma capsulatum requires Ryp1, a conserved transcriptional regulator. Proc. Natl. Acad. Sci. U.S.A. 105 (12), 4880-4885 (2008).
- Wenisch, C., Linnau, K. F., Parschalk, B., Zedtwitz-Liebenstein, K., Georgopoulos, A. Rapid susceptibility testing of fungi by flow cytometry using vital staining. Journal of Clinical Microbiology. 35, 5-10 (1997).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten