Method Article
Em Assembléia in vitro de Semi-artificial máquina molecular e sua utilização para detecção de danos no DNA
Neste Artigo
Resumo
Nós demonstramos a montagem e aplicação de um dispositivo em escala molecular alimentado por uma proteína topoisomerase. A construção é um sensor de bio-molecular que rotula dois principais tipos de quebras de DNA em cortes de tecido, anexando dois fluoróforos diferentes para os seus fins.
Resumo
Naturalmente bio-molecular máquinas de trabalho em cada célula viva e exibir uma variedade de modelos 06/01. No entanto, o desenvolvimento de centros artificial molecular máquinas em dispositivos capazes de movimento direcional, ou seja, motores moleculares, e sobre a sua escala reduzida partes mecânicas (rodas, eixos, etc pingentes) 7-9. Este imita as máquinas macro-, mesmo que as propriedades físicas essenciais para estes dispositivos, tais como a inércia ea conservação do momento, não são utilizáveis em ambientes nanoworld 10. Projetos alternativos, que não seguem os esquemas mecânicos macromachines e usar mecanismos desenvolvidos na evolução de moléculas biológicas, pode aproveitar as condições específicas do mundo nano. Além disso, adaptando reais de moléculas biológicas para fins de nano-design reduz perigos em potencial dos produtos da nanotecnologia pode representar. Aqui demonstramos a montagem e aplicação de uma construção bio-enabled tais, comoemi-artificial dispositivo molecular que combina uma máquina molecular de ocorrência natural com componentes artificiais. Do ponto de vista enzimologia, a nossa construção é um designer complexo enzima-substrato fluorescente colocados juntos para executar uma função específica útil. Este conjunto é, por definição, uma máquina molecular, uma vez que contém um 12. No entanto, sua integração com a parte de engenharia - hairpin dupla fluorescentes - re-direciona para uma nova tarefa de rotulagem danos no DNA 12.
Nossa construção reúne a partir de um DNA 32-mer e uma enzima topoisomerase I vaccinia (VACC TOPO). A máquina então usa seu próprio material para fabricar duas unidades detector fluorescente etiquetado (Figura 1). Uma das unidades (fluorescência verde) carrega VACC TOPO covalentemente ligado a seu 3'end e outra unidade (fluorescência vermelha) é um hairpin livre com uma 3'OH terminal. As unidades são de curta duração e rapidamente montar de volta para a construção original, que posteriormente recleaves.Na ausência de DNA quebra estas duas unidades separadas de forma contínua e religate de uma forma cíclica. Em cortes de tecidos com danos no DNA, a unidade de transporte de detector topoisomerase seletivamente atribui a quebra ponta e DNA com 5'OH (DNase II tipo-breaks) 11,12, fluorescente rotulá-los. O segundo, hairpin enzima livre formado após clivagem oligonucleotídeo, vai a uma ligadura 5'PO 4 quebrar blunt-ended (DNase I tipo-breaks) 11,12, se ligase T4 DNA está presente na solução 13,14. Quando ligase T4 DNA é adicionado a uma secção de tecido ou uma solução contendo DNA com 4 5'PO ponta e breaks, a ligase reage com 5'PO quatro extremidades do DNA, formando semi-estável da enzima DNA-complexos. Os grampos de cabelo sem corte terminou irá interagir com estes complexos liberando hairpins ligase e covalentemente ligados a DNA, assim rotulagem 5'PO quatro breaks ponta e DNA.
Este desenvolvimento exemplifica uma nova abordagem prática para podesign e de máquinas moleculares e fornece um sensor de útil para a detecção de apoptose e dano de DNA em células fixas e tecidos.
Protocolo
As seções para a detecção baseada em máquina molecular deve ser preparado em primeiro lugar porque a sua preparação leva mais tempo do que a montagem do dispositivo molecular. A construção funciona bem com 5-6μm de espessura, corte de paraformaldeído fixo, blocos de parafina do tecido. Use marcas de slides que retêm seções bem, como ProbeOn Além disso slides carregada e precleaned (Fisher Scientific) ou similar. Recomendamos em primeiro lugar usando um tecido com um padrão conhecido de dano ao DNA, que contém tanto DNase I e tipo II DNase-breaks, como a dexametasona tratadas com ratos apoptóticas timo 13,14.
1. Preparação de seções
- Coloque as seções em um rack de slides e dewax em xilol por 15 min, transferir para um banho de xileno fresco por mais 5 minutos.
- Reidratar passando por concentrações de etanol classificados: 96% de etanol 2x5min; Etanol 80% - 5min; água - 2x5 min.
- Digest seção com Proteínase K. Use 100μL de uma solução de 50μg/mL por seção. Incubar 15 'à temperatura ambiente (23 ° C) em câmara úmida. O tempo pode precisar de ajustes, dependendo do tipo de tecido. Tecidos duros pode exigir mais tempo de digestão. Vezes de 15-25 min são normalmente utilizados. Digestão insuficiente pode resultar no sinal mais fraco. Overdigestion na mão resulta no desaparecimento de sinais e interrupção seção.
- Enxágüe em água destilada por min 2x10.
- Adicionar 100 mL por seção da BSA 2% para preblocking. Incubar por 15 min à temperatura ambiente (23 ° C). Durante esse tempo, montar a máquina molecular.
2. Montagem máquinas moleculares
Todos os reagentes são escalados para 25 mL de volume total, que é suficiente para uma detecção única em uma seção média dimensão do tecido (10x10mm). O volume pode ser ampliado conforme a necessidade.
- Combine em um pequeno tubo de plástico na seguinte ordem:
|
- Misture suavemente por pipetagem. A construção molecular quase instantaneamente auto-monta nesta solução e pode ser usado imediatamente em temperatura ambiente (23 ° C). Aumentando a temperatura para 37 ° C bloqueia a T4 DNA ligase componente baseado em matéria de rotulagem.
3. Utilização de máquinas moleculares em seções de tecido para 5'OH etiqueta dupla e 5'PO4 breaks DNA
- Aspirar a solução preblocking e aplicar 25μL da mistura de reacção completa contendo 70 pmoles de Oligonucleotídeos 1, 53-215 pmoles (1,76-7,1 mg) VACCTOPO e 10 unidades de T4 DNA ligase (500 U / mL) em solução de 66 mM Tris-HCl, pH 7,5, MgCl 5mM 2, 0,1 mM Ditioeritritol, 1 mM ATP, e polietileno glicol 15%-8000. O oligonucleotídeo mesma sequência carregando um único rótulo FITC, Oligonucleotide 2, pode ser usado para a detecção em vez de um único tipo de quebras de DNA. Em tal caso, omitir ligase a partir da reação rotulagem. Também nesta situação uma solução de 50 mM Tris-HCL, pH 7,4, 15% PEG-8000 pode ser usado em vez de tampão T4 DNA ligase.
- Incubar por 1 hora em temperatura ambiente (23 ° C) em câmara úmida com uma lamela de plástico. Proteger da luz. Diminuição da temperatura para 16 ° C reduz o sinal ligase baseado, o aumento da temperatura a 37 ° C elimina completamente o sinal ligase-based. A inibição parcial da ligase às vezes ocorre na mistura de reação, possivelmente devido a contaminantes introduzidos com os preparativos topoisomerase. Portanto, mais de incubação (2-4 horas) pode ser necessária, especialmente quando snúmeros ignificant de ligase marcado breaks estão presentes. Embora ambos os ligase e sinais de topo pode ser observado nesta fase, em muitos casos o sinal ligase pode ser reforçada pela re-aplicação da mistura de reação sem VACC TOPO e Oligonucleotide dual-rotulados, mas contendo um oligonucleotídeo forma hairpin (Oligonucleotide 3) (35 mg / mL) e T4 DNA ligase (250 U / mL). Para melhorar sinal vá para a Etapa 3, para ver a reação, sem realce vá para o Passo 5.
- Remove delicadamente lamínulas imergindo as lâminas verticalmente em uma jarra de Coplin contendo água à temperatura ambiente. Aspirar o excesso de água.
- Melhorar sinal ligase através da aplicação de 25 mL de mistura de reação sem VACC TOPO e Oligonucleotide 1, mas contendo Oligonucleotide 3: 66 mM Tris-HCl, pH 7,5, 5 mM MgCl 2, 0,1 mM Ditioeritritol, 1 mM ATP, e polietileno glicol 15% 8000, 5 unidades de T4 DNA ligase, 35 de Oligonucleotídeos mg / mL 3 (blunt hairpin terminou). O volume total da solução de rotulagem can ser ampliados para acomodar as seções maiores. Incubar por 18 horas (overnight) em temperatura ambiente (23 ° C) em câmara úmida com uma lamela de plástico.
- Remove delicadamente lamínulas imergindo as lâminas verticalmente em Coplin jar contendo água à temperatura ambiente. Em seguida, lave min seção 3x10 em água destilada.
- Enxaguar com tampão bicarbonato de sódio. Solução alcalina lavar aumenta fluorescência FITC, que é sensível e pH é significativamente reduzido em pH abaixo de 7.
- Cobrir seção com uma solução antifading (Vectashield com DAPI), lamela e analisar o sinal usando um microscópio de fluorescência. Quebra dupla vertente-DNA com 5'OH ficam verde-fluorescentes, 5'PO quatro breaks - vai fluorescência vermelha (Figura 2).
4. Resultados representante
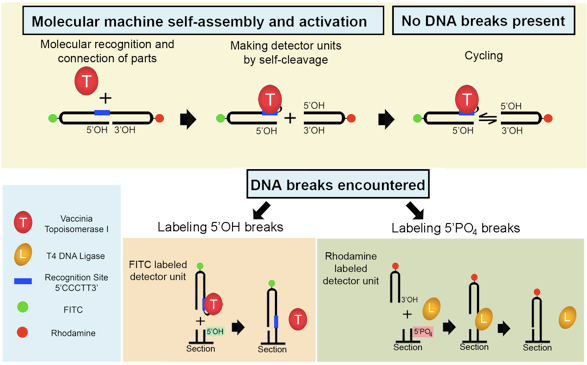
Figura 1. Semi-artificial máquina molecular detecta dois tipos deDanos ao DNA in situ. A máquina de auto-fluorescente quando monta VACC TOPO liga-se à dupla hairpin-32-mer. A máquina começa a operação, dividindo-se em duas unidades detector via topoisomerase-made corte na extremidade 3 'da seqüência de reconhecimento. Isso resulta em um processo cíclico onde a unidade de FITC-rotulados de forma contínua separa e volta para o religates rodamina marcado unidade. Este persiste até uma pausa DNA detectável é encontrado. Quando um receptor de tal alternativa (quebra de DNA 5'OH blunt end) está presente na seção de tecido, a parte FITC vai ligadura a ele. A parte restante rodamina serão anexados a 5 'PO 4 DNA quebra extremidade sem corte com a ajuda do DNA ligase T4. Conseqüentemente, ambos os tipos de quebras de DNA são simultaneamente detectado.
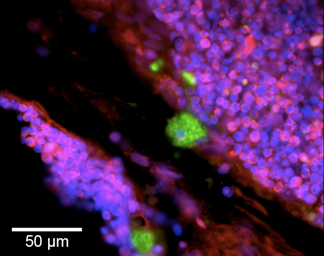
Figura 2. Molecular rótulos máquina dual dois tipos de quebras de DNAem uma seção de tecido de dexametasona tratadas com timo. Blunt quebra-ended DNA de DNase I-II-e DNase tipo são detectados nas áreas cortical do timo em apoptose. Fluorescência citoplasmática verde (5'OH quebra de DNA) marca macrófagos cortical digerir material nuclear de timócitos apoptóticos. Este sinal é produzido por VACC TOPO, e localiza-se fagolisossomos com DNA contendo 5'OH dupla vertente-breaks 11,12. Fluorescência vermelha (5'PO 4 breaks) rótulos de núcleos de timócitos apoptóticos não engolida pelos macrófagos. Um enorme número de timócitos em apoptose simultaneamente acompanhada pela geração de 4 5'PO fita dupla-breaks, visualizado por ligase baseado rotulagem. Estas pausas estão localizadas na periferia nuclear, formando padrões em forma de anel. Todos os núcleos celulares são visualizados através da contracoloração com DAPI (azul fluorescente).
Discussão
Neste vídeo, demonstramos como montar e usar um dual-rotulagem sensor de danos ao DNA. O sensor é uma máquina molecular impulsionada por bio-molecular do motor, um vírus proteína codificada TOPO VACC ligados com componentes artificiais. O desenvolvimento apresentado exemplifica uma abordagem bio-enabled que defende a adaptação das estruturas biológicas, arquiteturas e peças e componentes reais de células para o projeto de não-tóxico dispositivos à escala molecular 12,15. Esta abordagem resolve duas questões inerentes à área do nanosensores vivo: 1. a dificuldade de fazer verdadeiramente uniforme e reprodutível nano-constrói usando nanomateriais tradicionais; e 2. A toxicidade potencial, e reactividade biológica elevado de nanossondas, partículas e outros nanomateriais altamente dispersos. O sensor integra uma máquina de ocorrência natural com componentes moleculares artificiais que re-orientá-la para uma nova função de rotulagem de danos ao DNA. O produto dessa integração é um simi-artificial máquina molecular direcionados para uma nova tarefa. Enquanto topoisomerases, polimerases e outras máquinas enzimática são freqüentemente utilizados nas pesquisas bioquímicas, eles não estão integrados com componentes projetados em assembléias individuais molecular. Conseqüentemente, seu uso rotineiro, não são empregadas como semi-artificial dispositivos 12.
Aqui nós mostramos como usar o sensor no formato de secção de tecido para a rotulagem simultânea de DNase I e tipo II DNase-breaks. Atualmente não existem outros métodos disponíveis para realizar a detecção simultânea como dual.
DNase I e tipo II DNase-breaks são importantes marcadores da progressão da morte celular, especificamente rotulagem as fases de auto-apoptóticos autônoma e fagocíticas 11,16. O sensor é uma adição útil para a pesquisa biomédica arsenal lidar com a detecção e caracterização detalhada da apoptose.
Divulgações
Não há conflitos de interesse declarados.
Agradecimentos
Esta pesquisa foi suportada por subvenção R01NS062842 do Instituto Nacional de Distúrbios Neurológicos e Derrame, National Institutes of Health (VVD) e por doações R21 NS064403 do Instituto Nacional de Distúrbios Neurológicos e Derrame, National Institutes of Health através ARRA (VVD) e R21 EB006301 National Institute of Biomedical Imaging e Bioengenharia, National Institutes of Health (VVD).
Materiais
- Xileno
- 80 e Etanol a 96%.
- Solução 2% de albumina bovina (BSA) em água destilada.
- Oligonucleotídeo 1: um grampo duplo, dual marcado com fluoresceína e tetrametilrodamina:
5'-GGA AAG CCT GC F GGT GCA CCC ATT ACG CAT R AT GCG TT-3 '; F - FITC-dT; R - tetrametilrodamina-dT. Outros vermelho deslocado fluoróforos, tais como BODIPY TR, rodamina ou TAMRA pode ser usado em vez de R tetrametilrodamina como.
Alternativamente, você pode usar 2 Oligonucleotide -. Hairpin uma única dupla marcada com fluoresceína (para a detecção de um único tipo de quebras de DNA (DNase II tipo apenas) A sonda single-fluoróforo de transporte é consideravelmente menos caro e é conveniente sempre que um único tipo de quebra de DNA deve ser rotulado: 5'-GGA AAG CCT GC F GGT GCA CCC ATT ACG CAT ATG CGT T-3 '; F - FITC-dT
- Oligonucleotídeo 3. Blunt-ended rodamina marcado hairpin para o realce do sinal de ligadura situ com DNA ligase T4: 5'-GCG CTA GAC C G R GTC TAG CGC-3 '; R - tetrametilrodamina-dT
- Vaccinia DNA topoisomerase l (VACC TOPO) - 3000 U / mL (Vivid Technologies). Nos experimentos iniciais foram utilizados 215 pmoles (7,1 mg) do TOPO VACC por cada mL 25 da mistura de reacção. No entanto, a concentração topoisomerase pode ser significativamente reduzido sem perda de sensibilidade. Mais tarde, usou uma quantidade quatro vezes menor de VACC TOPO por seção (1,76 g em 25 mL da mistura de reação por seção) com resultados semelhantes. Reduzir a quantidade da enzima a 880 ng (em 25 mL da mistura de reação) resultou em um sinal mais fraco e 8,8 ng de VACC TOPO não produziu nenhum sinal detectável.
- DNA ligase T4 5 U / mL (Roche). Esta preparação altamente concentrada ligase dá o melhor sinal em nossas condições experimentais.
- 10 x tampão de reação para a T4 DNA ligase: 660 mM Tris-HCl, 50 mM MgCl 2, 10 Ditioeritritol mM, 10 mM ATP, pH 7,5 (20 ° C) (Roche). ATP em tampão de reação é facilmente destruído em ciclos repetitivos de descongelamento-congelamento. Alíquota do buffer em pequenas porções 15-20 mL e armazenar a - 20 ° C. Use uma vez.
- 30% (w / v) solução de PEG-8000 (Sigma) em água bidestilada. 15% PEG-8000 na mistura de reacção fortemente estimula a detecção, aumentando as concentrações eficazes dos seus constituintes, por exclusão de volume.
- Proteinase K (Roche) 20 solução estoque mg / mL em água destilada. Armazenar a - 20 ° C. Na solução use reação 50mg/ml em PBS, preparado a partir do estoque. Não reutilize.
- Vectashield com DAPI (Vector Laboratories).
- Tampão de bicarbonato de sódio: 50m M NaHCO 3, 15m M NaCl, pH 8,2.
- Vidro 22x22mm ou 22x40mm ou lamelas de plástico. Lamínulas de plástico são preferíveis durante a reação, já que umre mais fácil de remover da seção.
Referências
- Meacham, G. C., Patterson, C., Zhang, W., Younger, J. M., Cyr, D. M. . Nature. Cell. Biol. 3, 100-105 (2001).
- Schuldt, A. Dynamic Pol expeditions. Nature. Cell. Biol. 4, E279-E279 (2002).
- Urry, D. W. Molecular machines: how motion and other functions of living organisms can result from reversible chemical changes. Angew. Chem. Int. Ed. Engl. 32, 819-841 (1993).
- Schneider, T. D. Theory of molecular machines I. Channel capacity of molecular machines. J. Theor. Biol. 148, 83-123 (1991).
- McClare, C. W. Chemical machines, Maxwell's demon and living organisms. J. Theor. Biol. 30, 1-34 (1971).
- Spirin, A. S. Ribosome as a molecular machine. FEBS Lett. 514, 2-10 (2002).
- Fabbrizzi, L., Foti, F., Licchelli, M., Maccarini, P. M., Sacchi, D., Zema, M. Light-emitting molecular machines: pH-induced intramolecular motions in a fluorescent nickel(II) scorpionate complex. Chemistry. 8, 4965-4972 (2002).
- Drexler, K. E. . Nanosystems: Molecular Machinery, Manufacturing and Computation. , (1992).
- Freitas, R. A. . Nanomedicine: basic capabilities. , (1999).
- Didenko, V. V. DNA probes using fluorescence resonance energy transfer (FRET): designs and applications. BioTechniques. 31, 1106-1121 (2001).
- Minchew, C. L., Didenko, V. V. Fluorescent probes detecting the phagocytic phase of apoptosis: enzyme-substrate complexes of topoisomerase and DNA. Molecules. 16, 4599-4614 (2011).
- Didenko, V. V., Minchew, C. L., Shuman, S., Baskin, D. S. Semi-artificial fluorescent molecular machine for DNA damage detection. Nano. Letters. 4, 2461-2466 (2004).
- Didenko, V. V., Hornsby, P. J. Presence of double-strand breaks with single-base 3' overhangs in cells undergoing apoptosis but not necrosis. J. Cell Biol. 135, 1369-1376 (1996).
- Didenko, V. V., Tunstead, J. R., Hornsby, P. J. Biotin-labeled hairpin oligonucleotides. Probes to detect double-strand breaks in DNA in apoptotic cells. Am. J. Pathol. 152, 897-902 (1998).
- Jones, R. A. L. . Soft Machines: Nanotechnology and life. , (2007).
- Samejima, K., Earnshaw, W. C. Trashing the genome: Role of nucleases during apoptosis. Nat. Rev. Mol. Cell. Biol. 6, 677-688 (2005).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados