Method Article
In-vitro-Assembly von Semi-künstliche molekulare Maschine und ihre Verwendung zum Nachweis von DNA-Schäden
In diesem Artikel
Zusammenfassung
Wir zeigen die Montage und Anwendung eines molekularen Maßstab Gerät durch eine Topoisomerase Protein versorgt. Das Konstrukt ist ein bio-molekularen Sensor, der im Wesentlichen zwei Arten von DNA-Brüchen in Gewebeschnitten Etiketten durch Anbringen von zwei unterschiedlichen Fluorophoren an ihren Enden.
Zusammenfassung
Natürlich vorkommende bio-molekulare Maschinen arbeiten in jeder lebenden Zelle und zeigt eine Vielzahl von Designs 1-6. Doch die Entwicklung von künstlichen molekularen Maschinen konzentriert sich auf Geräte, die gerichtete Bewegung, dh molekularen Motoren, und auf deren abgespeckte mechanische Teile (Räder, Achsen, Anhänger usw.) 7-9. Dies ahmt die Makro-Maschinen, auch wenn die physikalischen Eigenschaften essentiell für diese Geräte, wie Trägheit und Impulserhaltung, nicht verwendbar sind in der Nanowelt Umgebungen 10. Alternative Entwürfe, die sich nicht an die mechanische macromachines Systeme und Einsatz von Mechanismen in der Evolution von biologischen Molekülen entwickelt, können die Vorteile der spezifischen Bedingungen der Nanowelt zu nehmen. Außerdem Anpassung tatsächlichen biologischen Molekülen für die Zwecke der Nano-Design reduziert mögliche Gefahren der Nanotechnologie-Produkte darstellen können. Hier zeigen wir die Montage und Anwendung eines solchen Bio-fähigen konstruieren, wieemi-künstlichen molekularen Gerät, das ein natürlich vorkommendes molekulare Maschine kombiniert mit künstlichen Komponenten. Von der Enzymologie Sicht ist unser Bau eines Designer-Fluoreszenz-Enzym-Substrat-Komplex zusammen, um eine bestimmte nützliche Funktion zu erfüllen. Diese Versammlung ist per Definition eine molekulare Maschine, wie es ein 12 enthält. Doch die Integration mit dem entwickelten Teil - leitet wieder an eine neue Aufgabe, Kennzeichnung DNA-Schäden 12 - Leuchtstofflampen zwei Haarnadel.
Unsere Konstruktion montiert aus einem 32-mer DNA und ein Enzym Vaccinia-Topoisomerase I (VACC TOPO). Die Maschine nutzt dann sein eigenes Material zu zwei fluoreszenzmarkierten Detektor-Einheiten (Abbildung 1) zu fertigen. Eine der Einheiten (grüne Fluoreszenz) führt VACC TOPO kovalent an seiner 3'-Ende und eine andere Einheit (rote Fluoreszenz) befestigt ist ein kostenloser Haarnadel mit einem Terminal 3'OH. Die Geräte sind kurzlebig und schnell wieder zusammenbauen in den ursprünglichen Bau, der anschließend recleaves.In Abwesenheit von DNA-Brüchen dieser beiden Einheiten kontinuierlich zu trennen und religate in zyklischer Weise. In Gewebeschnitten mit DNA-Schäden, die Topoisomerase-tragenden Detektoreinheit selektiv beimisst blunt-ended DNA-Brüchen mit 5'OH (DNase II-Typ Pausen) 11,12, Fluoreszenzmarkierung sie. Das zweite Enzym-freien Haarnadel nach Oligonukleotid-Spaltung gebildet wird, eine 5'PO 4 blunt-ended break (DNase I-Typ Pausen) 11,12 ligieren, wenn T4 DNA-Ligase ist in der Lösung vorhanden 13,14. Wenn T4 DNA-Ligase zu einem Gewebeschnitt oder einer Lösung mit DNA mit 5'PO 4 blunt-ended Pausen hinzugefügt wird, reagiert der Ligase mit 5'PO 4 DNA Enden, bilden semi-stabilen Enzym-DNA-Komplexe. Die stumpfen Enden Haarnadeln wird mit diesen Komplexen freigesetzt Ligase und kovalente Verknüpfung Haarnadeln, um DNA interagieren, damit die Kennzeichnung 5'PO 4 blunt-ended DNA-Brüche.
Diese Entwicklung verdeutlicht eine neue praktische Annäherung an the-Design von molekularen Maschinen und bietet einen nützlichen Sensor zur Detektion von Apoptose und DNA-Schäden in fixierten Zellen und Geweben.
Protokoll
Die Abschnitte für die molekulare Maschine-basierte Erkennung sollten zunächst vorbereitet werden, weil ihre Vorbereitung nimmt mehr Zeit als die Montage der molekularen Gerät. Das Konstrukt funktioniert gut mit 5-6μm dicke Abschnitte aus Paraformaldehyd-fixierte, in Paraffin eingebettetes Gewebe Blöcke geschnitten. Verwenden Sie schieben Marken, die Abschnitte zu halten und, wie ProbeOn Plus-geladen und vorgereinigte Objektträger (Fisher Scientific) oder ähnliches. Wir empfehlen zunächst mit einem Gewebe mit einem bekannten Muster von DNA-Schäden, die sowohl DNase I-und DNase II-Typ Pausen enthält, wie zB Dexamethason behandelten apoptotischen Ratte Thymus 13,14.
1. Vorbereitung von Abschnitten
- Setzen Sie die Teile in einen Objektträger-Rack und Entwachsen in Xylol für 15 min, ein frisches Xylol Bad für weitere 5 Minuten übertragen.
- Rehydrieren, indem sie durch abgestufte Ethanolkonzentrationen: 96% Ethanol-2x5min, 80% Ethanol - 5min, Wasser - 2x5 min.
- Digest Abschnitt mit Proteinase K. Mit 100 &mgr; eines 50μg/mL Lösung pro Abschnitt. Inkubieren 15 'bei Raumtemperatur (23 ° C) in einer feuchten Kammer. Die Zeit muss möglicherweise angepasst werden je nach Gewebetyp. Hartgewebe erfordert möglicherweise mehr Aufschluss. Times of 15-25 min sind in der Regel verwendet. Unzureichende Verdauung kann das schwächere Signal führen. Overdigestion auf der anderen Seite ergibt Signal Verschwinden und Abschnitt Störung.
- Spülen in destilliertem Wasser für 2x10 min.
- Apply 100 ul pro Abschnitt 2% BSA für Vorblockierung. Inkubieren für 15 min bei Raumtemperatur (23 ° C). Während dieser Zeit versammeln die molekulare Maschine.
2. Molekulare Maschinen Montage
Alle Reagenzien sind für 25 ul Gesamtvolumen, das reicht für einen einzigen Nachweis in einer durchschnittlichen Größe Gewebeschnitt (10x10mm) skaliert wird. Die Lautstärke kann skaliert werden, wie gebraucht.
- Kombinieren Sie in einem kleinen Plastikschlauch in dieser Reihenfolge:
|
- Vorsichtig mischen. Die molekulare fast sofort selbst montiert Konstrukt in diese Lösung und kann sofort bei Raumtemperatur (23 ° C) verwendet werden. Eine Erhöhung der Temperatur auf 37 ° C blockiert die T4 DNA-Ligase-basierte Komponente der Etikettierung.
3. Mit molekularen Maschinen in Gewebeschnitten zu dual label 5'OH und 5'PO4 DNA-Brüche
- Saugen Sie das Vorblockierung Lösung und gelten 25 &mgr; l des gesamten Reaktionsmischung mit 70 pmol Oligonukleotid 1, 53 bis 215 pmol (1,76 bis 7,1 ug) VACCTOPO und 10 Einheiten T4 DNA-Ligase (500 U / ml) in einer Lösung von 66 mM Tris-HCl, pH 7,5, 5 mM MgCl 2, 0,1 mM Dithioerythrit, 1 mM ATP und 15% Polyethylenglykol-8000. Die gleiche Sequenz Oligonukleotid Durchführung einer FITC-Label, Oligonukleotid-2, kann stattdessen für den Nachweis von einer einzigen Art von DNA-Brüchen verwendet werden. In einem solchen Fall auslassen Ligase von der Kennzeichnungspflicht Reaktion. Auch in dieser Situation eine Lösung von 50 mM Tris-HCL, pH 7,4, 15% PEG-8000 kann anstelle von T4 DNA-Ligase-Puffer verwendet werden.
- Inkubation für 1 h bei Raumtemperatur (23 ° C) in einer feuchten Kammer mit einem Kunststoff-Deckglas. Vor Licht schützen. Die Absenkung der Temperatur auf 16 ° C reduziert die Ligase-basierte Signal, die Temperatur zu erhöhen, um 37 ° C vollständig eliminiert die Ligase-basierte Signal. Eine partielle Hemmung der Ligase tritt manchmal in der Reaktionsmischung, die möglicherweise durch Verunreinigungen mit Topoisomerase Präparate eingeführt. Daher könnte längere Inkubationszeit (2-4 Stunden) benötigt werden, besonders wenn significant Zahl der Ligase-markierten Pausen vorhanden sind. Obwohl beide Ligase und topo-Signale in dieser Phase beobachtet werden kann, in vielen Fällen die Ligase-Signal weiter verbessert werden kann durch wiederholtes Auftragen des Reaktionsgemisches ohne VACC TOPO-und Dual-Oligonukleotid, aber mit einer Haarnadel geformt Oligonukleotid (Oligonukleotid 3) (35 mg / mL) und T4 DNA-Ligase (250 U / mL). Zur Verbesserung der Signal mit Schritt 3 fortfahren, um die Reaktion ohne Vergrößerung zu sehen fahren Sie mit Schritt 5 fort.
- Entfernen Deckgläser durch leichtes Eintauchen der Objektträger vertikal in einer Coplin Gefäß mit Wasser bei Raumtemperatur. Saugen Sie überschüssiges Wasser.
- Enhance-Ligase-Signal, indem 25 ul Reaktionsansatz ohne VACC TOPO und Oligonukleotid-1, aber mit Oligonukleotid-3: 66 mM Tris-HCl, pH 7,5, 5 mM MgCl 2, 0,1 mM Dithioerythrit, 1 mM ATP und 15% Polyethylenglykol- 8000, 5 Einheiten T4 DNA-Ligase, 35 mg / ml Oligonukleotid 3 (mit stumpfen Enden Haarnadel). Das Gesamtvolumen der Kennzeichnung Lösung can skaliert werden, um die größeren Abschnitte aufzunehmen. Inkubieren für 18 h (über Nacht) bei Raumtemperatur (23 ° C) in einer feuchten Kammer mit einem Kunststoff-Deckglas.
- Entfernen Deckgläser durch leichtes Eintauchen der Objektträger vertikal in Coplin Gefäß mit Wasser bei Raumtemperatur. Dann waschen Abschnitt 3x10 min in destilliertem Wasser.
- Spülen mit Natrium-Bicarbonat-Puffer. Alkalischer Lösung spülen verbessert FITC Fluoreszenz, die pH-sensitiv ist und liegt deutlich unter pH 7 reduziert.
- Abdeckprofil mit einem Antifading Lösung (Vectashield mit DAPI), Deckglas und zu analysieren, das Signal mit Hilfe eines Fluoreszenz-Mikroskops. Doppelstrang-DNA bricht mit 5'OH fluoreszieren grün, 5'PO 4 Pausen - fluoreszieren rot (Abbildung 2).
4. Repräsentative Ergebnisse
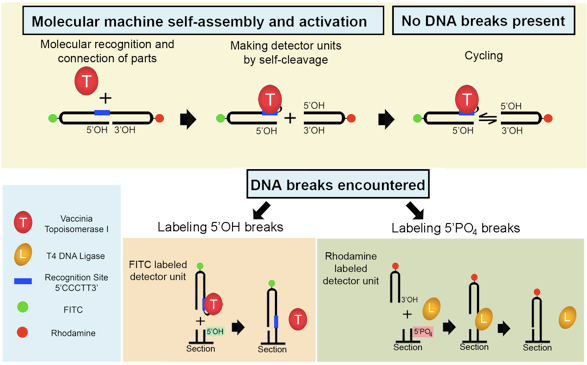
Abbildung 1. Semi-künstliche molekulare Maschine erkennt zwei Arten vonDNA-Schäden in situ. Die fluoreszierenden Maschine selbst montiert, wenn VACC TOPO bindet sich an die Doppel-Haarnadel 32-mer. Die Maschine beginnt den Betrieb durch die Spaltung von selbst in zwei Detektor-Einheiten über Topoisomerase-made Schnitt am 3 'Ende der Erkennungssequenz. Daraus ergibt sich ein zyklischer Prozess, bei dem FITC-markierten Einheit trennt kontinuierlich und religates zurück zu den Rhodamin-markierten Einheit. Dieser bleibt bestehen, bis eine nachweisbare DNA-Bruch aufgetreten ist. Wenn eine solche Alternative Akzeptor (5'OH stumpfen Ende DNA Pause) im Gewebeschnitt wird, wird die FITC Teil, um es zu unterbinden. Die restlichen Rhodamin Teil wird 5 'PO 4 DNA stumpfen Ende bricht mit Hilfe der T4 DNA-Ligase zu befestigen. Folglich sind beide Arten von DNA-Brüchen gleichzeitig detektiert.
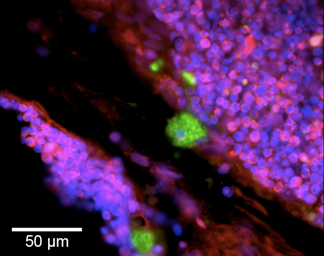
Abbildung 2. Molekulare Maschine Dual-Etiketten zwei Arten von DNA-Brüchenin einem Gewebe Abschnitt Dexamethason behandelten Thymus. Blunt-ended DNA-Brüche von DNase I-und DNase II-Typs sind in der Thymus-kortikalen Arealen Apoptose nachgewiesen werden. Grüne zytoplasmatischen Fluoreszenz (5'OH DNA-Brüche) markiert kortikalen Makrophagen verdauen Kernmaterial von apoptotischen Thymozyten. Dieses Signal wird von VACC TOPO produziert, und lokalisiert zu Phagolysosomen mit DNA enthält 5'OH Doppelstrang-Brüche 11,12. Red-Fluoreszenz (5'PO 4 Pausen) Etiketten Kerne von apoptotischen Thymozyten nicht durch Makrophagen verschlungen. Massive Zahl von Thymozyten gleichzeitig Apoptose durch Erzeugung von 5'PO 4 Doppel-Strangbrüchen begleitet, visualisiert durch Ligase-basierte Kennzeichnung. Diese Brüche sind an der nuklearen Peripherie gelegen, bildet ringförmige Muster. Alle Zellkerne werden visualisiert durch Gegenfärbung mit DAPI (blaue Fluoreszenz).
Diskussion
In diesem Video zeigen wir, wie zu montieren und ein Dual-Kennzeichnung DNA-Schäden Sensor. Der Sensor ist eine molekulare Maschine durch bio-molekularen Motor, ein Virus-kodierte Protein VACC TOPO mit künstlichen Komponenten verbunden angetrieben. Die vorgestellte Entwicklung beispielhaft für eine bio-fähigen Ansatz, der die Anpassung biologischer Strukturen, Architekturen und tatsächlichen Teile und Komponenten von Zellen Fürsprecher der Gestaltung von ungiftigen molekularen Skala Geräte 12,15. Dieser Ansatz behebt zwei Probleme inhärent auf dem Gebiet der in-vivo Nanosensoren: 1. der Schwierigkeit, wirklich gleichmäßige und reproduzierbare Nano-Konstrukte mit traditionellen Nanomaterialien und 2. die potenzielle Toxizität und hohe biologische Reaktivität von Nanosonden, Partikel und andere hochdisperse Nanomaterialien. Der Sensor integriert eine natürlich vorkommende molekulare Maschine mit künstlichen Komponenten, die re-direct es um eine neue Funktion von DNA-Schäden Kennzeichnung. Das Produkt einer solchen Integration ist ein semi-künstliche molekulare Maschine, um eine neue Aufgabe ausgerichtet. Während Topoisomerasen, Polymerasen und andere enzymatische Maschinen häufig in der biochemischen Forschung verwendet werden, sind sie nicht mit technischen Komponenten in einzelne molekulare Baugruppen integriert. Folglich in ihrer Routine zu verwenden, werden sie nicht als semi-künstlichen Vorrichtungen 12 eingesetzt.
Hier zeigen wir, wie man den Sensor in das Gewebe Abschnitt Format für die gleichzeitige Kennzeichnung von DNase I-und DNase II-Typ bricht. Derzeit gibt es keine anderen Methoden zur Verfügung, wie simultanes Dual-Erkennung durchführen.
DNase I-und DNase II-Typ Pausen sind wichtige Marker für Zelltod Progression, speziell die Kennzeichnung der apoptotischen selbst autonom und phagocytic Phasen 11,16. Der Sensor ist eine sinnvolle Ergänzung der biomedizinischen Forschung Arsenal Umgang mit der Erkennung und detaillierte Charakterisierung von Apoptose.
Offenlegungen
Keine Interessenskonflikte erklärt.
Danksagungen
Diese Arbeit wurde durch Grant R01NS062842 vom National Institute of Neurological Disorders and Stroke, National Institutes of Health (VVD) und durch Zuschüsse R21 NS064403 vom National Institute of Neurological Disorders and Stroke, National Institutes of Health durch ARRA (VVD) und R21 unterstützt EB006301 National Institute of Biomedical Imaging and Bioengineering, National Institutes of Health (VVD).
Materialien
- Xylol
- 80 und 96% Ethanol.
- 2% ige Lösung von Rinderserumalbumin (BSA) in destilliertem Wasser.
- Oligonukleotid 1: Doppel-Haarnadel, dual mit Fluorescein und Tetramethylrhodamin gekennzeichnet:
5'-AAG GGA CCT GC F GCA GGT CCC TTA ACG CAT R AT GCG TT-3 '; F - FITC-dT; R - Tetramethylrhodamine-dT. Andere rotverschoben Fluorophore wie BODIPY TR, Rhodamin oder TAMRA kann anstelle von Tetramethylrhodamin als R verwendet werden.
Alternativ können Sie Oligonukleotid 2 -. Doppel-Haarnadel Einzelzimmer mit Fluorescein (für den Nachweis von einer einzigen Art von DNA-Brüchen (DNase II-Typ) Die Einzel-Fluorophor-tragenden Sonde ist wesentlich billiger und ist praktisch, wenn ein einzelnes Art der DNA brechen, um zu kennzeichnen: 5'-AAG GGA CCT GC F GCA GGT CCC TTA ACG CAT ATG CGT T-3 '; F - FITC-dT
- Oligonucleotid 3. Blunt-ended rhodaminmarkierten Haarnadel für die Verbesserung der in-situ-Ligation Signal mit T4 DNA-Ligase: 5'-GCG CTA GAC C R G GTC TAG CGC-3 '; R - Tetramethylrhodamine-dT
- Vaccinia DNA Topoisomerase l (VACC TOPO) - 3000 U / ul (Vivid Technologies). In den ersten Versuchen verwendeten wir 215 pmol (7,1 ug) der VACC TOPO pro jede 25 ul des Reaktionsgemisches. Allerdings kann die Topoisomerase-Konzentration deutlich, ohne den Verlust der Empfindlichkeit reduziert werden. Wir haben später benutzte eine viermal geringere Menge an VACC TOPO pro Abschnitt (1,76 mg in 25 ul der Reaktionsmischung pro Abschnitt) mit ähnlichen Ergebnissen. Reduzierung der Menge des Enzyms bis 880 ng (in 25 ul der Reaktion mix) führte ein schwächeres Signal und 8,8 ng VACC TOPO produziert keine nachweisbaren Signal.
- T4 DNA-Ligase 5 U / ul (Roche). Dieses hochkonzentrierte Ligase Vorbereitung gibt das beste Signal in unseren experimentellen Bedingungen.
- 10 x Reaktionspuffer für T4 DNA-Ligase: 660 mM Tris-HCl, 50 mM MgCl 2, 10 mM Dithioerythrit, 10 mM ATP, pH 7,5 (20 ° C) (Roche). ATP in Reaktionspuffer ist einfach in sich wiederholenden Zyklen Auftauen Einfrieren zerstört. Aliquot der Puffer in kleinen 15-20 ul Portionen und bei - 20 ° C einmal verwenden.
- 30% (w / v) Lösung von PEG-8000 (Sigma) in zweifach destilliertem Wasser. 15% PEG-8000 in der Reaktionsmischung stark stimuliert die Erkennung, die Erhöhung der effektiven Konzentrationen der Bestandteile Vol. Ausgrenzung.
- Proteinase K (Roche) 20 mg / ml Stammlösung in destilliertem Wasser. Lagerung bei - 20 ° C. Bei der Reaktion verwenden 50mg/ml Lösung in PBS, aus dem Lager vorbereitet. Dürfen nicht wiederverwendet werden.
- Vectashield mit DAPI (Vector Laboratories).
- Natriumbicarbonatpuffer: 50m M NaHCO 3, 15m M NaCl, pH 8,2.
- 22x22mm oder 22x40mm Glas oder Kunststoff Deckgläser. Plastic Deckgläser sind während der Reaktion vorzuziehen, da sie einere leichter aus dem Abschnitt zu entfernen.
Referenzen
- Meacham, G. C., Patterson, C., Zhang, W., Younger, J. M., Cyr, D. M. . Nature. Cell. Biol. 3, 100-105 (2001).
- Schuldt, A. Dynamic Pol expeditions. Nature. Cell. Biol. 4, E279-E279 (2002).
- Urry, D. W. Molecular machines: how motion and other functions of living organisms can result from reversible chemical changes. Angew. Chem. Int. Ed. Engl. 32, 819-841 (1993).
- Schneider, T. D. Theory of molecular machines I. Channel capacity of molecular machines. J. Theor. Biol. 148, 83-123 (1991).
- McClare, C. W. Chemical machines, Maxwell's demon and living organisms. J. Theor. Biol. 30, 1-34 (1971).
- Spirin, A. S. Ribosome as a molecular machine. FEBS Lett. 514, 2-10 (2002).
- Fabbrizzi, L., Foti, F., Licchelli, M., Maccarini, P. M., Sacchi, D., Zema, M. Light-emitting molecular machines: pH-induced intramolecular motions in a fluorescent nickel(II) scorpionate complex. Chemistry. 8, 4965-4972 (2002).
- Drexler, K. E. . Nanosystems: Molecular Machinery, Manufacturing and Computation. , (1992).
- Freitas, R. A. . Nanomedicine: basic capabilities. , (1999).
- Didenko, V. V. DNA probes using fluorescence resonance energy transfer (FRET): designs and applications. BioTechniques. 31, 1106-1121 (2001).
- Minchew, C. L., Didenko, V. V. Fluorescent probes detecting the phagocytic phase of apoptosis: enzyme-substrate complexes of topoisomerase and DNA. Molecules. 16, 4599-4614 (2011).
- Didenko, V. V., Minchew, C. L., Shuman, S., Baskin, D. S. Semi-artificial fluorescent molecular machine for DNA damage detection. Nano. Letters. 4, 2461-2466 (2004).
- Didenko, V. V., Hornsby, P. J. Presence of double-strand breaks with single-base 3' overhangs in cells undergoing apoptosis but not necrosis. J. Cell Biol. 135, 1369-1376 (1996).
- Didenko, V. V., Tunstead, J. R., Hornsby, P. J. Biotin-labeled hairpin oligonucleotides. Probes to detect double-strand breaks in DNA in apoptotic cells. Am. J. Pathol. 152, 897-902 (1998).
- Jones, R. A. L. . Soft Machines: Nanotechnology and life. , (2007).
- Samejima, K., Earnshaw, W. C. Trashing the genome: Role of nucleases during apoptosis. Nat. Rev. Mol. Cell. Biol. 6, 677-688 (2005).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten