16S rRNA 시퀀싱: 박테리아 종 식별을위한 PCR 기반 기술
Overview
출처: 에와 부코스카-패니반드1,틸드 앤더슨1,롤프 루드1
1 임상 과학 룬드학과, 감염 의학 학과, 생물 의학 센터, 룬드 대학, 221 00 룬드, 스웨덴
행성 지구는 세균 종의 수백만에 대한 서식지입니다, 각각의 특정 특성을 가지고. 세균성 종의 식별은 감염된 환자를 진단하기 위해 환경 샘플 및 의료 미생물학의 생물 다양성을 결정하기 위해 미생물 생태학에서 널리 사용됩니다. 박테리아는 현미경 검사법과 같은 기존의 미생물학 방법, 특정 매체에 대한 성장, 생화학 및 혈청 술 검사 및 항생제 민감성 분석을 사용하여 분류될 수 있습니다. 최근 수십 년 동안 분자 미생물학 방법은 세균 식별에 혁명을 일으켰습니다. 16S 리보소말 RNA(rRNA) 유전자 염기서열분석이 인기 있는 방법이다. 이 방법은 기존의 방법보다 빠르고 정확할 뿐만 아니라 실험실 조건에서 성장하기 어려운 균주를 식별할 수 있습니다. 더욱이, 분자 수준에서 균주의 분화는 표현형 동일 박테리아 (1-4)의 차별을 가능하게합니다.
16S rRNA는 세균성 리보솜(5)의 30S 하위 단위를 형성하기 위해 19개의 단백질의 복합체와 결합한다. 그것은 16S rRNA 유전자에 의해 인코딩된다, 이는 존재하고 리보솜 조립에 그것의 필수적인 기능때문에 모든 박테리아에 높게 보존; 그러나 특정 종에 대한 지문 역할을 할 수있는 가변 영역도 포함되어 있습니다. 이러한 특징은 16S rRNA 유전자를 박테리아의 식별, 비교 및 계통유전학 적 분류에 사용되는 이상적인 유전 적 단편으로 만들었습니다 (6).
16S rRNA 유전자 염기서열분석은 중합효소 연쇄 반응(PCR) (7-8)를 기반으로 DNA 염기서열 분석(9)을 선행한다. PCR은 다음과 같은 일련의 주기를 통해 DNA의 특정 단편을 증폭시키는 데 사용되는 분자 생물학 방법입니다.
i) 이중 좌초 된 DNA 템플릿의 거부
ii) 템플릿에 보완프라이머 (짧은 올리고 뉴클레오티드)의 안다골
iii) 새로운 DNA 가닥을 합성하는 DNA 폴리머라제 효소에 의한 프라이머 확장
메서드의 회로도 개요는 도 1에표시됩니다.

그림 1: PCR 반응의 회로도 개요. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
성공적인 PCR 반응에 중요한 몇 가지 요인이 있으며, 그 중 하나는 DNA 템플릿의 품질입니다. 박테리아로부터 염색체 DNA의 격리는 표준 프로토콜 또는 상업적 키트를 사용하여 수행될 수 있다. PCR 반응을 억제할 수 있는 오염물질이 없는 DNA를 얻기 위해 특별한 주의를 기울여야 한다.
16S rRNA 유전자의 보존된 영역은 임의의 세균종에서 표적 부위에 결합하고 증폭될 수 있는 범용 프라이머 쌍(1개의 전진 및 1개의 역)의 설계를 허용한다. 대상 영역의 크기가 다를 수 있습니다. 몇몇 프라이머 쌍은 16S rRNA 유전자의 대부분을 증폭할 수 있는 동안, 그 외는 그것의 단지 부분을 증폭합니다. 일반적으로 사용되는 프라이머의 예는 표 1에 표시되며 바인딩 사이트는 그림 2에표시됩니다.
| 프라이머 이름 | 시퀀스(5'→3') | 전진/역방향 | 참조 |
| 8F b) | 아가그트가트CCTGGCTCAG | 전달 | -1 |
| 27F | AGAGTGATCMTGGCTCAG | 전달 | -10 |
| 515F | GTGCCAGCMGCCGCGGGTAA | 전달 | -11 |
| 911R | GCCCCCGTCAATTCMTTTGA | 후진 | -12 |
| 1391R | 가CGGGCGGTGTGTRCA | 후진 | -11 |
| 1492R | GGTTGTTACGACTT | 후진 | -11 |
표 1: 16S rRNA 유전자a)의증폭에 사용되는 표준 올리고뉴클레오티드의 예.
a) 상이한 프라이머 조합을 사용하여 생성된 PCR 제품의 예상 길이는 전방 및 역프라이머(그림 2 참조)에 대한 결합 부위 사이의 거리를 계산하여 추정할 수 있으며, 예를 들어 프라이머 쌍 8F-1492R을 사용하여 PCR 제품의 크기는 ~1500bp, 프라이머 쌍 27F-91R~90bp.90 bp.
b) fD1이라고도 함

그림 2: 16S rRNA 서열 및 프라이머 결합 부위의 대표적인 수치. 보존된 영역은 회색으로 표시되고 가변 영역은 대각선으로 채워져 있습니다. 가장 높은 해상도를 허용하기 위해, 프라이머 8F 및 1492R(rRNA 서열에 근거한 이름)는 전체 서열을 증폭시키는 데 사용되어 유전자의 여러 가변 영역의 시퀀싱을 허용한다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
PCR에 대한 사이클링조건(즉, DNA가 변성되고, 프라이머로 어닐링되고, 합성되는 데 필요한 온도 및 시간)은 사용되는 폴리머라제의 종류와 프라이머의 특성에 의존한다. 특정 폴리머라제에 대한 제조업체의 지침을 따르는 것이 좋습니다.
PCR 프로그램이 완료되면, 제품은 아가로즈 젤 전기포광에 의해 분석된다. 성공적인 PCR은 예상 크기의 단일 밴드를 생성합니다. 제품은 PCR 반응에 존재했던 잔류 프라이머, 데옥시리보뉴클레오티드, 폴리머라제 및 완충물을 제거하기 위해 시퀀싱 전에 정제되어야 한다. 정제 된 DNA 단편은 일반적으로 상업적 시퀀싱 서비스에 시퀀싱을 위해 전송됩니다. 그러나 일부 기관은 자신의 핵심 시설에서 DNA 시퀀싱을 수행합니다.
DNA 서열은 컴퓨터에 의해 DNA 크로마토그램에서 자동으로 생성되며 수동 편집이 때로는 필요하기 때문에 품질을 주의 깊게 검사해야합니다. 이 단계에 이어, 유전자 서열은 16S rRNA 데이터베이스에 증착된 서열과 비교된다. 유사성 영역이 식별되고 가장 유사한 시퀀스가 전달됩니다.
Procedure
1. 설정
- 미생물을 취급하는 동안, 좋은 미생물 사례를 따르는 것이 필요합니다. 모든 미생물, 특히 알려지지 않은 샘플은 잠재적인 병원균으로 취급되어야 합니다. 샘플, 연구원 또는 실험실을 오염하지 않도록 무균 기술을 따르십시오. 박테리아를 취급하기 전과 후에 손을 씻고 장갑을 착용하고 보호복을 착용하십시오.
- 게놈 DNA 격리 및 PCR 제품 정제를 위한 실험 프로토콜에 대한 위험 평가를 수행한다. 일부 시약은 유해할 수 있습니다!
- 순수한 배양은 16S rRNA 시퀀싱에 필수적입니다. 게놈 DNA의 격리를 진행하기 전에, 시작 물질이 완전히 순수한지 확인하십시오. 이것은 개별 식민지를 격리하기 위하여 줄무늬 도금에 의해 행적할 수 있습니다. 이들은 더 개별적으로 접시에 줄무늬, 또는 국물에 줄무늬, 필요한 경우.
- 필요한 실험실 장비:
- PCR용 열 사이클러. 열 사이클러의 기능은 세트 프로그램에 따라 온도를 높이고 낮추는 것입니다. 프로그램을 만드는 동안 모든 PCR 단계의 온도 및 시간 값과 총 사이클 수를 입력하라는 메시지가 표시됩니다.
- 아가로즈 젤 전기 포세이스 시스템. 그것은 그들의 크기와 전하에 근거를 둔 DNA 단편을 분리하는 데 사용됩니다. 이 프로토콜에서, 아가로즈 젤 전기포진은 고립된 게놈 DNA 및 PCR 제품의 품질을 시각화하는 데 사용될 것이다.
2. 프로토콜
참고: 입증된 프로토콜은 박테리아의 순수한 배양에서 16S rRNA 유전자 염기서열분석에 적용됩니다. 그것은 메라지노믹 연구에 적용되지 않습니다.
-
게놈 DNA (gDNA)의 격리를 위한 박테리아를 배양합니다.
- 적합한 매체에서 미생물을 성장시십시오. 이 단계에서액체와 고체 미디어를 모두 사용할 수 있습니다. 최상의 성장을 이수하는 조건을 선택합니다. 실험을 계획 하는 동안, 천천히 성장 하는 박테리아 늦은 로그/고정 성장 단계에 도달 하기 위해 며칠이 필요할 수 있습니다 명심 하십시오. 이 프로토콜에서, 바실러스 서브틸리스(168)는 200rpm, 37°C로 설정된 흔들리는 인큐베이터에서 하룻밤 사이에 리소제니 국물(LB)에서 재배되었다.
-
gDNA의 격리.
- 박테리아가 고체 매체에서 성장한 경우에, 멸균 루프를 사용하여 몇몇 세포를 긁어 내고 증류수의 1 mL에서 그(것)들을 재중단합니다
- 박테리아가 액체 배지에서 재배된 경우, 하룻밤 문화의 약 1.5mL를 사용하십시오.
- 원심분리(1분, 12,000~16,000× g)에 의한 펠릿은 상류체를 제거하고, 상용 키트 또는 표준 프로토콜을 사용하여 gDNA 절연을 위해 세포를 사용한다[예를들어 CTAB 총 DNA 제제(13) 또는 페놀 클로로폼 추출(14)]. 여기서, 상용 키트는 B. subtilis 168 의 1.5 mL에서 gDNA를 분리하는 데 사용되었으며, OD600 = 1.5.
참고 1: 일부 그램 음성 박테리아에 대 한이 단계 생략 하 고 끓는 에 의해 세포에서 DNA의 간단한 릴리스에 의해 대체 될 수 있습니다. 증류수에서 세균 펠릿을 재중단하고 100°C로 설정된 가열 블록에서 10분 동안 배양합니다.
참고 2: 그람 양성 세균세포는 중단하기 어렵다. 따라서 이 박테리아 그룹에서 분리하는 데 전념하는 gDNA 격리 방법 또는 키트를 선택하는 것이 좋습니다.
-
gDNA 품질 검사.
- 아가로즈 젤 전기포에 의해 분리된 gDNA의 품질을 확인하십시오. 먼저, 격리된 gDNA의 5μL을 적재염료(6x)의 1μL과 혼합하고, DNA 염색 시약을 포함하는 0.8% 아가로즈 젤에 샘플을 적재한다.
- 분자 질량 표준을 로드하고 염료 전면이 젤의 바닥에 도달 할 때까지 전기 전도를 실행합니다.
- 전기 전도가 완료되면, 적절한 트랜실루미나이터 (UV 또는 청색 광)에 젤을 시각화합니다. gDNA는 두꺼운 고분자 대역(10kb 이상)으로 나타납니다. gDNA 품질 검사의 예는 도 3에도시된다.
- gDNA가 품질 관리를 통과하는 경우(즉,높은 분자 밴드가 존재하고 gDNA의 거의 - 투 - 더 얼룩이있다), 먼저 다음과 같이 3 미세 원심 분리기 튜브를 라벨하여 연속적으로 gDNA를 희석 : "10x", "100x"와 "1000x".
- 3개의 튜브각각에 멸균 증류수의 파이펫 90 μL.
- gDNA 용액의 10 μL을 가져 와서 "10x"로 표시된 튜브에 추가하십시오.
- 전체부피(즉, 100 μL)를 위아래로 철저히 배관하여 용액이 균일하게 혼합되도록 합니다. 그런 다음 이 튜브에서 용액의 10 μL을 가져 와서 "100x"로 표시된 튜브로 옮습니다.
- 전에 설명한 바와 같이 혼합하고 튜브 "100x"에서 튜브 "1000x"로 용액의 10 μL을 전송하여 동일한 절차를 반복한다. 이러한 희석은 PCR 반응에서 템플릿으로 사용됩니다.

그림 3: 간균 서브틸리스로부터분리된 gDNA의 아가로즈 겔 전기포진. 레인 1 : M - 분자 질량 마커 (위에서 아래로 : 10000 bp, 8000 bp, 6000 bp, 5000 bp, 4000 bp, 3500 bp, 3000 bp, 2500 bp, 2000 bp, 1500 bp, 1000 bp). 레인 2 : gDNA - 바실러스 서브틸리스로부터분리 된 게놈 DNA . 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
-
PCR에 의한 16S rRNA 유전자의 증폭.
참고: 아래의 PCR 프로토콜은 특정 DNA 폴리머라제 및 프라이머 쌍 8F - 1492R에 최적화되어 있습니다(표 1 참조). 프로토콜의 최적화는 각 폴리머라제 및 프라이머 쌍에 대해 요구된다.- 얼음에 모든 시약을 해동.
- 표 2에 표시된 대로 PCR 마스터 믹스를 준비합니다. DNA 중합체는 실온에서 활성화되기 때문에, 반응 설정은 얼음, 즉 PCR 튜브 및 반응 성분을 항상 얼음에 보관해야 한다. 각 gDNA 샘플당 하나의 반응과 음수 제어를 위한 하나의 반응을 준비합니다. 음의 제어는 gDNA 템플릿이 없는 PCR 혼합물이며 반응의 다른 구성 요소가 오염되지 않도록 하는 데 사용됩니다.
참고: 여러 샘플의 경우 마스터 믹스가 일반적으로 준비됩니다. 마스터 믹스는 템플릿을 제외한 모든 반응 구성 요소를 포함하는 솔루션입니다. 반복적인 파이펫팅을 생략하고, 파이프팅 오류를 방지하며, 샘플 간의 높은 일관성을 보장하는 데 도움이 됩니다. 마스터 믹스를 준비하려면 각 성분의 부피를 테스트된 샘플 수(DNA 템플릿 제외)로 곱합니다. 모든 성분을 미세원심분리기 튜브에 혼합하고 전체 볼륨을 여러 번 위아래로 피펫합니다. - 마스터 믹스의 알리쿼트 49 μL은 개별 PCR 튜브에 혼합한다.
- 마스터 믹스와 튜브에 1 μL 템플릿을 추가합니다. 음의 제어를 위해 멸균 물 1 μL을 추가합니다. 구성 요소가 잘 혼합되도록하려면 파이펫을 30-50μL로 설정하여 혼합을 위아래로 ~ 10번 부드럽게 피펫합니다.
- 표 3에 표시된 프로그램으로 PCR 컴퓨터를 설정합니다.
- PCR 기계에 튜브를 넣고 프로그램을 시작합니다.
- 프로그램이 완료되면 아가로즈 젤 전기 포에 의해 PCR 제품의 품질을 검사하십시오.
- 8F-1492R 프라이머 쌍을 이용한 성공적인 PCR 반응은 약 1.5kb(도4)의단일 대역을 산출한다. 다른 대역(예: 비특이제품)이 있는 경우, 어닐링 온도를 조정하여 PCR 프로그램을 최적화합니다. 예상 크기의 단일 밴드가 있는 경우 다음 단계로 진행합니다. 여기서, 100x 희석 gDNA 템플릿을 가진 PCR 반응은 예상 된 크기의 날카로운 밴드를 가지고 특정 제품이 부족으로 최고의 제품을 산출했다. 따라서 그것은 정제및 시퀀싱을 위해 전송하도록 선택되었다.
- 시퀀싱 전에, 제품은 PCR 반응에 존재했던 잔류 프라이머, 데옥시리보뉴클레오티드, 폴리머라제 및 완충제로부터 세척되어야 한다. PCR 제품은 상용 PCR 정제 키트를 사용하여 분리될 수 있다. PCR 반응은 DNA 결합 매트릭스를 포함하는 컬럼에 로드됩니다. PCR 제품은 열에 바인딩되지만 다른 구성 요소는 열을 통해 흐릅니다. 컬럼은 세척 버퍼를 사용하여 세척하고 마지막으로 DNA가 선택의 완충액에 용출됩니다. 키트로 보충되는 용출 버퍼가 시퀀싱과 호환되는지 확인합니다.
- DNA 염기서열분석용 정제PCR 제품을 보냅니다. 선택한 시퀀싱 시설에서 시퀀싱 샘플 제출에 대한 지침을 따르십시오. 최상의 시퀀스 커버리지를 위해 PCR 증폭 프라이머(섹션 2.4.1에 사용된 것과 동일)를 시퀀싱 프라이머로 사용합니다. 여기서 프라이머 8F 및 1492R은 PCR 제품을 시퀀싱하는 데 사용되었습니다.
| 구성 요소 | 최종 농도 | 반응당 볼륨 | x 반응당 볼륨(마스터 믹스) |
| 5x 반응 버퍼 | 1x | 10 μL | 10 μL × x |
| 10mM dNTP | 200 μM | 1 μL | 1 μL × x |
| 10 μM 프라이머 8F | 0.5 μM | 2.5 μL | 2.5 μL × x |
| 10 μM 프라이머 1492R | 0.5 μM | 2.5 μL | 2.5 μL × x |
| 푸시온 폴리머라제 | 1 유닛 | 0.5 μL | 0.5 μL × x |
| 템플릿 DNA * | - | 1 μL | - |
| ddH2O | - | 32.5 μL | 32.5 μL × x |
| 총 볼륨 | 50 μL | 49 μL × x |
표 2: PCR 반응 구성 요소. * 2.3 단계에서 10x, 100배 또는 1000x 희석 gDNA를 사용합니다.
| 걸음 | 온도 | 시간 | 사이클 |
| 초기 변성 | 98°C | 30초 | |
| 변성 | 98°C | 10초 | 25-30 |
| 어 닐 링 | 60°C | 30초 | |
| 확장 | 72°C | 45초 | |
| 최종 확장 | 72°C | 7분 | |
| 들다 | 4°C | ∞ |
표 3: 16S rRNA 유전자의 증폭을 위한 PCR 프로그램.

그림 4: 프라이머 8F 및 1492R 및 gDNA를 템플릿으로 사용하여 증폭된 PCR 제품의 아가로즈 젤 전기전경. B. subtilis에서 gDNA 샘플 (그림 3 참조)는 최상의 결과를 테스트하기 위해 10, 100 및 1000 번 희석되었다. 레인 1 : M - 분자 질량 마커 (위에서 아래로 : 10000 bp, 8000 bp, 6000 bp, 5000 bp, 4000 bp, 3500 bp, 3000 bp, 2500 bp, 2000 bp, 1500 bp, 1000bp, 750 bp, 500 bp5). 레인 2: 10배 희석 템플릿을 가진 PCR 반응. 레인 3: 100x 희석 템플릿을 가진 PCR 반응. 레인 4: 1000x 희석 템플릿을 가진 PCR 반응. 차선 5: (C-) - 음의 제어 (DNA 템플릿없이 반응). 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
3. 데이터 분석 및 결과
참고: PCR 제품은 포워드(여기 8F)와 리버스(여기 1492R) 프라이머를 사용하여 시퀀싱된다. 따라서, 두 세트의 데이터 시퀀스가 생성되고, 하나는 전방용이고 역프라이머에 대해 하나씩 생성된다. 각 서열에 대해 적어도 두 가지 유형의 파일이 생성됩니다: i) DNA 서열을 포함하는 텍스트 파일 및 ii) 시퀀싱 실행의 품질을 보여주는 DNA 크로마토그램.
- 전방 프라이머의 경우 크로마토그램을 열고 시퀀스를 주의 깊게 검사합니다. 품질 시퀀스에 대한 이상적인 크로마토그램은 균일하게 시공간 피크와 거의 또는 전혀 배경 신호(그림 5A)가있어야합니다.
- 크로마토그램이 고품질이 아닌 경우 시퀀스를 폐기하거나 시퀀스 텍스트 파일은 다음에 따라 수정해야 합니다.
- 크로마토그램 전체에 걸쳐 이중 피크의 존재는 여러 DNA 템플릿의 존재를 나타냅니다. 세균 배양이 순수하지 않은 경우의 경우도 마찬가지입니다. 이러한 순서는 폐기해야합니다(그림 5B).
- 모호한 크로마토그램은 같은 위치에 다른 색깔의 봉우리가 있는 것으로 인해 발생할 수 있습니다. 가장 일반적인 오류 중 하나는 시퀀싱소프트웨어(그림5C)에의해 베이스를 동일한 위치에 두 개의 서로 다른 색 피크가 있고 부적절한 할당이 존재한다는 것입니다. 잘못 할당된 뉴클레오티드를 수동으로 수정하고 텍스트 파일에서 편집합니다.
- 저해상도 크로마토그램은 종종 이 지역에서 뉴클레오티드의 잘못 계산을 일으키는 "넓은 피크"를 초래할 수 있습니다(그림 5D). 이 오류는 수정하기 어렵기 때문에 추가 정렬 단계에서 발생할 수 있는 불일치를 신뢰할 수 있는 것으로 취급해서는 안 됩니다.
- 가난한 크로마토그램 판독 품질과 여러 피크의 존재는 일반적으로 시퀀스의 5'와 3'끝에서 볼 수 있습니다. 일부 시퀀서 소프트웨어는 이러한 저품질 단편을 자동으로제거(도 5E)뉴클레오티드는 텍스트 파일에 포함되지 않습니다. 시퀀스가 자동으로 잘리지 않은 경우 끝에 저품질조각(예: 약한 신호, 겹치는 피크, 해상도 손실)을 결정하고 텍스트 파일에서 각 베이스를 제거합니다.
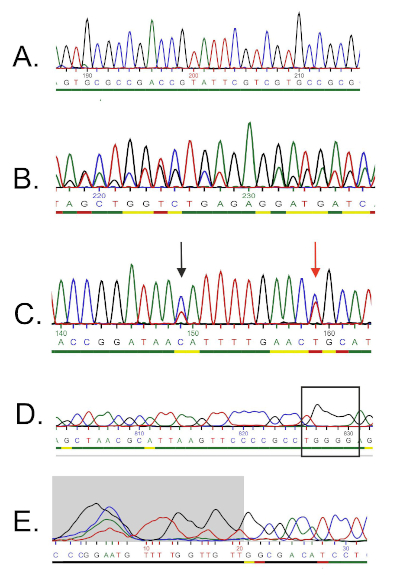
그림 5: DNA 염기서열 분석 문제 해결의 예. A) 품질 크로마토그램 시퀀스(균등 간격, 모호한 피크)의 예입니다. B) 일반적으로 크로마토그램의 시작 부분에서 발생하는 가난한 품질 시퀀스. 회색 영역 영역은 낮은 품질로 간주되며 시퀀싱 소프트웨어에 의해 자동으로 제거됩니다. 더 많은 베이스를 수동으로 다듬을 수 있습니다. C) 이중 피크의 존재 (화살표로 표시). 적색 화살표로 표시된 뉴클레오티드는 시퀀서에 의해 "T"(빨간색 피크)로 읽혀졌지만, 파란색 피크는 더 강하며 또한 "C"로 해석될 수 있다. D) 겹치는 피크는 DNA오염(즉, 하나 이상의 템플릿)을 나타낸다. E) 해상도손실및 신뢰할 수 있는 기본 호출을 방지하는 "넓은 피크"(사각형으로 표시)라고 합니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
- 역 프라이머에 대해 3.1 및 3.2를 반복합니다.
- 마지막으로 전진 및 역방향 시퀀스를 하나의 연속 시퀀스로 조립합니다. 좋은 시퀀싱 실행은 최대 1100 bp의 시퀀스를 생성합니다. PCR 제품이 ~1500bp 길이라는 점을 고려할 때, 전방 및 역 프라이머를 사용하여 얻은 시퀀스는 부분적으로 겹쳐야 한다.
- DNA 서열 조립 프로그램을 이용하여 2개의 서열을 병합한다( 예를 들어 CAP3(http://doua.prabi.fr/software/cap3) (15)와 같은 자유 공구.
- FASTA 형식으로 두 시퀀스를 표시된 상자에 삽입합니다. "제출" 버튼을 클릭하고 결과가 돌아올 때까지 기다립니다.
- 조립된 시퀀스를 보려면 결과 탭에서 "Contigs"를 누릅니다. 정렬의 세부 사항을 보려면 "어셈블리 세부 정보"를 누릅니다.
참고 1: CAP3 소프트웨어가 연속 조립에 사용되는 경우 역프라이머 시퀀스를 역 보완으로 변환할 필요가 없습니다. 그러나 다른 프로그램을 사용하는 경우 이 단계가 필요할 수 있습니다.
참고 2: FASTA 형식은 뉴클레오티드 서열을 나타내는 텍스트 기반 형식이다. FASTA 파일의 첫 번째 줄(설명 줄)은 기호 ">"로 시작하여 시퀀스의 이름 또는 고유 식별자가 표시됩니다. 설명 선에 이어 뉴클레오티드 서열이다. 시퀀스를 다음 형식으로 붙여넣습니다.
>sequence_frw_primer
여기에 텍스트 파일에서 시퀀스를 붙여 넣기
>sequence_rvs_primer
여기에 텍스트 파일에서 시퀀스를 붙여 넣기
- 기본 로컬 정렬 검색 도구(BLAST; https://blast.ncbi.nlm.nih.gov/Blast.cgi)에 대한 웹 사이트를 방문하여 데이터베이스 검색을 수행합니다.
- "뉴클레오티드 BLAST" 도구를 선택하여 시퀀스를 데이터베이스와 비교합니다.
- 시퀀스(3.5로 조립된 연속체)를 "쿼리 시퀀스" 텍스트 상자에 입력한 다음 스크롤 다운 메뉴에서 데이터베이스 "16S rRNA 시퀀스(박테리아 및 Archea)"를 선택합니다.
- 페이지 하단의 "BLAST" 버튼을 누릅니다. 가장 유사한 시퀀스가 반환됩니다. 예제 BLAST 결과는 도 6에표시됩니다. 제시된 실험에서 탑 히트는 블라스트 데이터베이스에서 사용할 수 있는 서열로 100% 정체성을 나타내며, B. 서브틸리스 스트레인(168)이다.
- 상단 히트에 100% ID가 표시되지 않으면 정렬로 이동하여 불일치를 확인합니다. 상단 히트를 클릭하면 정렬의 세부 정보로 이동합니다. 정렬된 뉴클레오티드는 짧은 수직 선에 의해 결합될 것이고 일치하지 않는 뉴클레오티드는 그들 사이의 간격을 갖습니다. 시퀀싱 회사에서 받은 크로마토그램으로 돌아가 일치하지 않는 영역에 초점을 맞추어 시퀀스를 다시 한 번 수정하십시오. 추가 오류가 발견되면 시퀀스를 수정합니다. 보정된 시퀀스를 사용하여 BLAST를 다시 실행합니다.
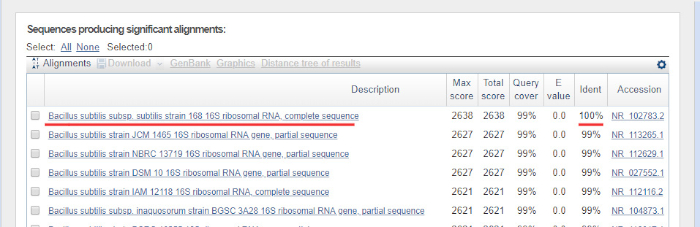
그림 6: 뉴클레오티드 블라스트 결과의 예. 16S rRNA 유전자 서열B. subtilis 168의 순수한 배양으로부터 쿼리 서열로 사용되었다. 최고 히트는 예상대로 B. 서브틸리스 스트레인 168에 100 % ID (밑줄)를 보여줍니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
Application and Summary
세균성 종을 확인하는 것은 다른 연구원을 위해, 뿐만 아니라 헬스케어에 있는 사람들을 위해 중요합니다. 16S rRNA 시퀀싱은 처음에 박테리아 사이의 물리 유전학 관계를 결정하기 위해 연구원에 의해 사용되었다. 시간이 지남에 따라, 그것은 잠재적인 병원균을 식별하는 방법으로 환경 샘플및 임상 실험실의 생물 다양성을 결정하기 위해 메타게노믹 연구에서 구현되었습니다. 그것은 임상 견본에 존재하는 박테리아의 신속하고 정확한 확인을 가능하게 합니다, 초기 진단을 촉진하고 환자의 더 빠른 처리를 촉진합니다.
References
- Weisburg, W.G., Barns, S.M., Pelletier, D.A. and Lane D.J. 16S ribosomal DNA amplification for phylogenetic study. J Bacteriol. 173 (2): 697-703. (1991)
- Drancourt, M., Bollet, C., Carlioz, A., Martelin, R., Gayral, J.P., Raoult D. 16S ribosomal DNA sequence analysis of a large collection of environmental and clinical unidentifiable bacterial isolates. J Clin Microbiol. 38 (10):3623-3630. (2000)
- Woo, P.C., Lau, S.K., Teng, J.L., Tse, H., Yuen, K.Y. Then and now: use of 16S rDNA gene sequencing for bacterial identification and discovery of novel bacteria in clinical microbiology laboratories. Clin Microbiol Infect. 14 (10):908-934. (2008)
- Tang, Y.W., Ellis, N.M., Hopkins, M.K., Smith, D.H., Dodge, D.E., Persing, D.H. Comparison of phenotypic and genotypic techniques for identification of unusual aerobic pathogenic gram-negative bacilli. J Clin Microbiol. 36 (12):3674-3679. (1998)
- Tsiboli, P., Herfurth, E., Choli, T. Purification and characterization of the 30S ribosomal proteins from the bacterium Thermus thermophilus. Eur J Biochem. 226 (1):169-177. (1994)
- Woese, C.R. Bacterial evolution. Microbiol Rev. 51 (2):221-271. (1987)
- Bartlett, J.M., Stirling, D. A short history of the polymerase chain reaction. Methods Mol Biol. 226:3-6. (2003)
- Wilson, K.H., Blitchington, R.B., Greene, R.C. Amplification of bacterial 16S ribosomal DNA with polymerase chain reaction. J Clin Microbiol. 28 (9):1942-1946. (1990)
- Shendure, J., Balasubramanian, S., Church, G.M., Gilbert, W., Rogers, J., Schloss, J.A., Waterston, R.H. (2017) DNA sequencing at 40: past, present and future. Nature. 550:345-353.
- Lane, D.J. 16S/23S rRNA sequencing. (1991) In Nucleic acid techniques in bacterial systematics. (Goodfellow, M. and Stackebrandt, E., eds.) p.115-175. Wiley and Sons, Chichester, United Kingdom.
- Turner, S., Pryer, K.M., Miao, V.P., Palmer, J.D. (1999) Investigating deep phylogenetic relationships among cyanobacteria and plastids by small subunit rRNA sequence analysis. J Eukaryot Microbiol. 46:327-338.
- Fredricks, D.N., Relman, D.A. (1998) Improved amplification of microbial DNA from blood cultures by removal of the PCR inhibitor sodium polyanetholesulfonate. J Clin Microbiol. 36:2810-2816.
- Wilson, K. Preparation of genomic DNA from bacteria. (2001) Curr Protoc Mol Biol. Chapter 2:Unit 2.4.
- Wright, M. H., Adelskov, J., Greene, A.C. (2017) Bacterial DNA extraction using individual enzymes and phenol/chloroform separation. J Microbiol Biol Educ. 18:18.2.48.
- Huang, X., Madan, A. (1999). CAP3: A DNA sequence assembly program. Genome Res. 9:868-877.
Tags
건너뛰기...
이 컬렉션의 비디오:

Now Playing
16S rRNA 시퀀싱: 박테리아 종 식별을위한 PCR 기반 기술
Microbiology
189.0K Views

위노그라드스키 칼럼 생성: 퇴적물 검체에서 미생물 종을 풍부하게하는 방법
Microbiology
129.4K Views

연속 희석 및 플레이팅: 미생물 나열
Microbiology
316.0K Views

농축 배양: 선택 및 차동 매체에서 호기성 및 혐기성 미생물 배양
Microbiology
132.1K Views

순수 배양 및 줄무늬 평판배양: 혼합 검체에서 단일 박테리아 군집 분리
Microbiology
166.2K Views

성장 곡선: 군집 형성 단위 및 광학 밀도 측정을 사용하여 성장 곡선 생성
Microbiology
295.9K Views

항생 물질 감수성 시험: 두 항생제의 MIC 값을 결정하고 항생 시너지를 평가하기 위한 Epsilometer 검사
Microbiology
93.7K Views

현미경 및 염색: 그램, 캡슐 및 내생포자 염색
Microbiology
363.3K Views

플라크 분석: 플라크 형성 단위(PFU)로서 바이러스 역가를 결정하는 방법
Microbiology
186.2K Views

적응 염화칼슘 절차를 이용한 E.coli 세포의 형질변환
Microbiology
86.8K Views

접합: 공여 E.coli에서 수용 E.coli로 암피실린 내성을 전달하는 방법
Microbiology
38.2K Views

파지 형질도입: 공여 E.coli에서 수용 E.coli로 암피실린 내성을 전달하는 방법
Microbiology
29.1K Views
Copyright © 2025 MyJoVE Corporation. 판권 소유