Method Article
除染された喀痰サンプルの標的次世代シーケンシングのためのマトリックスベースのDNA抽出
* これらの著者は同等に貢献しました
要約
本稿では、マトリックスベースの抽出法と磁気ビーズ精製を組み合わせた、除染された喀痰からのDNA抽出に最適化された方法を提示し、ダウンストリームの標的次世代シー ケンシングに取り組みます。
要約
次世代シーケンシング(NGS)は、現在、薬剤耐性結核(DR-TB)をタイムリーかつ正確に診断するための強力なツールとして認識されています。Targeted NGS(tNGS)は、薬剤耐性に関連する特定の遺伝子に焦点を当てることで、数週間から数ヶ月のターンアラウンドタイムを持つ従来の培養ベースの方法の必要性を回避し、合理的なアプローチを提供します。世界保健機関(WHO)は、特にリソースが限られている状況において、結核の診断を改善し、治療の指針となり、治療結果を改善するための貴重な戦略としてtNGSを推奨しています。WHOが推奨するtNGSアッセイの中から、迅速かつ包括的な薬剤感受性試験、系統決定、および株タイピングを提供する方法を選択しました。標準化されたDNA抽出方法が利用可能ですが、時間と労力がかかる場合があります。この課題に対処するために、私たちは、磁気ビーズ精製と組み合わせて、簡略化されたマトリックスベースのDNA抽出プロトコルを最適化しました。この方法は、除染された喀痰沈殿物から直接DNAを抽出するための迅速かつ効率的なアプローチを提供し、迅速なダウンストリームtNGS分析を可能にします。このプロトコルは、喀痰沈底物からのDNA抽出プロセスを合理化することにより、日常的な臨床現場でのtNGSの広範な採用を促進し、最終的には患者の転帰の改善に貢献し、世界的な結核対策の取り組みに役立つ可能性があります。
概要
2023年には、世界で推定370万人の結核患者が診断も治療も受けられず、結核が世界の健康にもたらす大きな脅威が浮き彫りになりました1。WHOは、2023年に約40万人がリファンピシン耐性結核(RR-TB)または多剤耐性結核(MDR-TB)を発症したと推定しています1。結核とDR-TBの迅速な診断と治療は、結核の発生率と死亡率を減らすためのマイルストーンと目標を達成するために不可欠です1。
従来の培養方法や表現型薬物感受性試験(pDST)に頼ると、臨床分離株の耐性プロファイルの決定が遅れ、所要時間は6〜8週間で、複雑な生物学的封じ込めインフラストラクチャが必要になります。定期的な診断検査とDR-TBのNGSを組み合わせることで、包括的な薬剤耐性プロファイルを提供し、MDR-TBレジメンの個別化を改善すると同時に、効果的な治療までの時間を数週間または数か月から2、3、4日に短縮することができます。
2023年と2024年に、WHOは、第一選択薬と第二選択薬の抗結核薬に対する感受性を迅速に判断するための新しい診断クラスとしてtNGSの使用を推奨しました5。これにより、バイオセーフティレベル3(BSL-3)の研究室で 結核菌 (MTB)の培養を必要とせずに、治療の決定を導くための貴重なツールとなります。tNGSアプローチは、シーケンシングの前にポリメラーゼ連鎖反応(PCR)を使用して薬剤耐性を付与する遺伝子標的を増幅するシーケンシングの凝縮された形式です。WHOが推奨するtNGS検査の中から、リファンピシン、イソニアジド、エタンブトール、ピラジナミド、フルオロキノロン、アミカシン、ストレプトマイシン、リネゾリド、ベダキリン、クロファジミンに対する耐性を検出するためのクラスベースの基準を満たすと報告されているDeeplex Myc-TBアッセイを選択しました。このアッセイを使用して、このプロトコルを使用して抽出したDNAの下流tNGSへの適合性を評価します。
さらに、WHOは、MTBにおける薬剤耐性に関連する変異のカタログの第2版を発行し 、全ゲノムシーケンシング(WGS)とtNGSを使用して薬剤感受性を予測し、治療を導くためのロードマップを提供しています6。最近のシステマティックレビューとメタアナリシスでは、tNGSは、pDST7と比較して、MTBゲノムのさまざまな耐性付与領域の23の標的に基づいて、薬剤耐性の検出に対してそれぞれ94.1%と98.1%の感度と特異性を持っていたことが示されました。
しかし、これらの手法の実装は、特に必要なワークフロー、インフラストラクチャ、および機器に関連する複雑さとコストのために、依然として困難です。重要な課題は、除染された喀痰の沈殿物から十分な高品質のマイコバクテリアDNAを直接単離することであり、これは下流のtNGSアプリケーションにとって重要なステップです。これに対処するために、tNGSに合わせた迅速で簡単なDNA抽出法を紹介します。
選択されたMTB tNGSアッセイの標準化されたDNA抽出法には、社内の手動および自動プロトコール8が含まれています。ここでは、簡略化されたマトリックスベースのDNA抽出プロトコルについて説明します(図1)。この方法では、金属とタンパク質を結合するInstaGene Matrix(IGM)を利用し、除染された喀痰沈殿物から直接高品質の核酸抽出を可能にします。この代替手段は、より迅速なターンアラウンドタイムと、ダウンストリームのtNGSに対する十分なDNA収量を提供します。このプロトコルは、手動および自動の方法の複雑さを克服しながら、MTBに耐性を与えるバリアントの迅速な診断のための高品質のtNGSを確保します。マイコバクテリア学分野でのtNGSの使用への関心が高まる中、このプロトコルは日常的な診断ワークフローへの採用を促進する可能性があります。

図1:マトリックス懸濁液を使用して除染された喀痰沈殿物サンプルからマイコバクテリアDNAを抽出する方法の概略図。 この 図の拡大版を表示するには、ここをクリックしてください。
プロトコル
この研究は、ステレンボッシュ大学の研究倫理委員会:生物学的および環境安全(REC:BES)によって承認されています:BES-2024-25384および人間研究倫理委員会:N21/09/093およびN09/11/296。
1. 喀痰沈殿物サンプル調製
注:次の手順は、DNA抽出の前にバイオセーフティレベル3(BSL3)ラボで実行する必要があります。
- N-アセチル-L-システイン(NALC)および水酸化ナトリウム(NaOH)(NALC/NaOH)を使用して以前に除染した喀痰沈殿物サンプルのアリコートを、500 μL-2 mLを1.5 mLまたは2 mLの低結合チューブに移して調製します。
- 喀痰沈殿物サンプルの入ったチューブをチューブラックに直立させ、サンプルを80°Cのオーブンに入れ、1時間インキュベートして熱処理によりマイコバクテリアを不活化します。
- 1時間後、サンプルが入ったチューブラックを取り外し、チューブの表面を拭き、バイオセーフティレベル2(BSL2)の実験室に輸送するために適切な容器に入れます。
注:サンプルを室温まで冷ましてから、さらに取り扱いまたは保管してください。
2. マトリックス懸濁液と高速ホモジナイザーによるDNA抽出(図2)
- 開始する前に、直径2 mmのガラスビーズ3個を1.5 mLまたは2 mLのスクリューキャップチューブに加え、緩くキャップし、オートクレーブします(理想的にはDNA抽出の少なくとも1日前)。
- マトリックス懸濁液を冷蔵庫から取り出し、マグネチックスターラー(試薬ボトルに磁石が含まれています)に置き、試薬を室温に戻して試薬の混合を開始します。
- 加熱ブロックのスイッチを入れ、56°Cに予熱します(2つの加熱ブロックが利用可能な場合は、2番目の加熱ブロックを100°Cに予熱します。
注:次の手順は、BSL2施設の実験台で実行できます。 - 熱不活化した喀痰沈殿物サンプルを16,800 × g (またはフルスピード)で15分間遠心分離します。20-200 μLのピペットで上清を穏やかに吸引して捨て、ペレットが邪魔されないようにします。
- マトリックス懸濁液を穏やかに振盪して混合し(ボルテックスしないでください)、よく混合したマトリックス200μLを加えてペレットを再懸濁します。
- 全容量を、直径2mmの滅菌済みガラスビーズ3個が入った1.5mLのスクリューキャップチューブにピペットで移します。
- サンプルを加熱ブロックに入れ、56°Cで15分間インキュベートします。
注:加熱ブロックからサンプルを取り出した直後に、ステップ2.7の後に加熱ブロックを100°Cに設定します(使用可能な加熱ブロックが1つしかない場合)。 - ボルテックスを10秒間使用してサンプルを均質化し、細胞を分散させます。
- サンプルを加熱ブロックに入れ、100°Cで10分間インキュベートします。
- 高速ホモジナイザーを使用してサンプルを均質化し、次のパラメータを設定します:4.0 m/sで60秒の1サイクル。
- 12,000 × g で5分間遠心分離します。130 μL の DNA 含有上清を、ペレット (細胞破片を含むマトリックス) を乱さずに、新しい 1.5 mL の低結合チューブに移します。マトリックスと細胞破片を含む最初のチューブを廃棄します。
注:安全な停止ポイント。停止する場合は、DNA精製を続行する準備ができるまで、サンプルを-20°Cで凍結します。 - 磁気ビーズを用いたDNA精製を進めます。

図2:高速ホモジナイザーと組み合わせたマトリックスベースのDNA抽出法。 略語:HSH =高速ホモジナイザー。 この図の拡大版を表示するには、ここをクリックしてください。
3. 磁気ビーズによるDNA精製(図3)
注:DNA精製を進める前に、手順3.1および3.2を実行してください。
- 磁気ビーズを冷蔵庫から取り出し、容器を室温で30分間置いて平衡化します。あるいは、磁気ビーズストックボトルをボルテックスしてすべてのビーズを再懸濁し、1,280 μLのアリコートを新しいチューブに吸引し(21サンプルの精製用)、室温で平衡化します。
- 1.8 mLのろ過滅菌水に7.2 mLの純粋なエチルアルコールを加えて、80%エタノールの9 mLバッチを準備します。
注:次の手順は、BSL2施設の実験台で実行できます。 - 磁気ビーズストックボトルまたは準備されたアリコートを完全にボルテックスして、使用前にビーズが再懸濁されていることを確認します。10サンプルごとにボルテックスを繰り返して、均一な懸濁液を確保します。
- 抽出したDNA(130 μL)に1.2倍容量(156 μL)の磁気ビーズを加えます。
注:大量のDNA(培養)を含むサンプルの場合、サンプルの小さなアリコート(例:50μL)を処理し、1.2倍容量の磁気ビーズで精製できます。 - 10回ピペッティングしてサンプルを混合します。サンプルを室温で5分間インキュベートします。
- サンプルを1.5 mLチューブマグネティックラックに3分間、または液体が透明になるまで置きます。上清はビーズを乱さずに慎重に吸引して捨てます。
- チューブを磁石に取り付けた状態で、200 μLの80%エタノールを加え、ビーズが乱されず、30秒間インキュベートできるようにします。
- エタノールを慎重に吸引し、ビーズを乱さずに廃棄します。
- 手順3.7を繰り返して、合計2回の洗浄を行います。
- 2回目の洗浄後、エタノールを取り出して廃棄します。1〜10μLのピペットで残留エタノールを取り除きます。
- チューブを開いたままにして、ビーズを10分間、またはビーズがつや消しになるまで風乾させます。
注意: ビーズが過度に乾燥してひび割れないようにしてください。 - ビーズがつや消しになったら、すぐにチューブを磁石から取り外し、50 μLのヌクレアーゼフリー水(NFW)を各サンプルのビーズに直接加えます。すべてのチューブにNFWを加えた後、10回ピペッティングして個々のサンプルを混合します。チューブの内側にビーズが付着していないかチューブを検査し、必要に応じて混合手順を繰り返します。サンプルを室温で5分間インキュベートします。
注:インキュベーションは5分から10分に増やすことができますが、インキュベーション時間を5分未満に減らさないでください。 - チューブを磁気ラックに3分間、または液体が透明になるまで戻します。チューブを磁気ラックに載せた状態で、DNA含有上清を、明確にマークされた無菌の低結合チューブに移します。ビーズが上清に移らないようにしてください。
注:移し替え中にピペットチップにビーズが見える場合は、上清をマグネティックラックのチューブにピペットで戻し、さらに3分間分離します。ビーズを新しいチューブに移さないでください。
安全な停止ポイント。停止する場合は、NGSに適した方法を使用して定量を続行し、その後MTB tNGSアッセイを使用してPCR増幅を行う準備ができるまで、サンプルを-20°Cで凍結します。
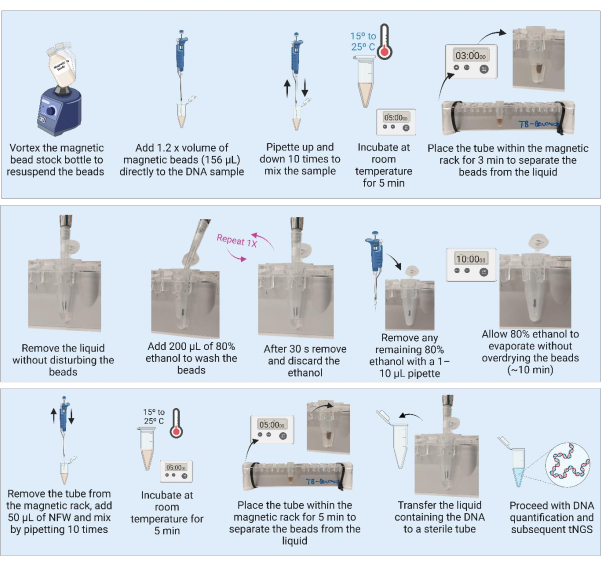
図3:磁気ビーズを使用したDNAの精製と濃縮。 略語:NFW =ヌクレアーゼフリー水;tNGS = ターゲット次世代シーケンシング。 この図の拡大版を表示するには、ここをクリックしてください。
結果
サンプルの説明
細菌負荷が少なくとも1+の顕微鏡検査で酸耐性菌(AFB)陽性の合計165の喀痰沈殿物サンプルは、南アフリカのケープタウンにある国立衛生研究所サービス(NHLS)グリーンポイントによって定期的に収集および処理されました。DNAは、2つの異なる容量[約2 mL(n = 102)と500 μL(n = 63)]を使用して、喀痰沈殿物サンプルから抽出しました。この比較は、堆積物サンプルの量が少ないほど、下流のtNGSに十分なDNAを得られるかどうかを評価するために行われました。
図4は、さまざまな喀痰塗抹グレード(1+、2+、および3+)および喀痰沈殿物量(2 mLおよび500 μL)にわたる総DNA濃度(ng/μL)の箱ひげ図比較を示しています。DNA収量は、抽出に500 μLの沈殿物を使用すると、一般に全体的に減少し、塗抹グレードによってばらつきがあります。したがって、少なくとも2 mLの容量の沈殿物サンプルから抽出された平均DNA濃度は、サンプルのAFBによって層別化された500 μLの沈殿物から抽出されたDNA濃度よりも平均して高かった。

図4:塗抹グレード全体のDNA収量に対する喀痰沈殿物量の影響。この図の拡大版を表示するには、ここをクリックしてください。
図 5 は、2 つの異なるインプット容量 (2 mL と 500 μL) から処理されたさまざまな塗抹グレード (1+、2+、3+) の喀痰沈殿物サンプルのすべてのターゲットの平均シーケンシングリード数 (平均カバレッジ深度) を示す箱ひげ図を示しています。外れ値は個々のポイントで表され、Y 軸はカバレッジの変動に対応するために対数スケールで表示されます。500 μL の沈殿物から抽出された 3+ サンプルの四分位範囲が高いことは、細菌負荷が高いほど変動性が大きくなることを示唆しています。

図5:塗抹標本グレードとサンプルインプット量にわたるすべてのターゲットの平均シーケンシングリード数(カバレッジ深度)の比較。 この図の拡大版を表示するには、ここをクリックしてください。
図6は、関連するWebアプリケーションのバイオインフォマティクスパイプラインを使用したシーケンシング結果の受容性の評価を示しています。シーケンシング品質スコアは、+++ (非常に許容可能)、++ (許容可能)、+ (わずかに許容可能)、-(許容できない)、および ND (未決定) に分類されます。グラフにバーがない場合は、その特定のカテゴリの結果が得られなかったことを示します。塗抹グレード1+は、500μLのインプットボリュームサンプルセットについて、塗抹グレード2+および3+と比較して、許容できない結果とND結果の割合が高いことを示しています。500 μL の喀痰沈底物から抽出したサンプルの平均シーケンシングカバレッジ深度は 4,316 であったのに対し、2 mL の喀痰沈殿物から抽出したサンプルの平均シーケンシングカバレッジ深度は 4,810 でした。このことは、喀痰沈底物から抽出されたDNAが、インプット量に関係なく、下流のtNGSを行うのに適切であったことを示唆しています。
シーケンシング結果の受容性の分布に基づくと、塗抹グレードが3+のサンプルは、1+および2+の塗抹グレードのサンプルと比較して、ほとんどのサンプル配列が++および+++としてスコアリングされ、成功率が最も高かった。500 μL インプットサンプルの場合、- および + カテゴリーのサンプルの割合は、2 mL インプットサンプルと比較して、すべての塗抹グレードで相対的に高くなっています。これは、500 μL インプットのサンプルが低品質で許容範囲が狭いカテゴリに分類されることを示唆しています。2 mL インプットサンプルでは、特に塗抹グレード 3+ の場合、高品質の ++ および +++ カテゴリーのサンプルの割合が高くなります。このことから、2 mL インプットのサンプルは、500 μL インプットのサンプルと比較して、シーケンシング品質スコアが良好である可能性が高いことが示唆されています。

図6:塗抹グレードとサンプル入力量による品質スコアの分布。この図の拡大版を表示するには、ここをクリックしてください。
ディスカッション
マトリックスベースの抽出法と高速ホモジナイザーの組み合わせは、Shea et al.9によって最初に発表されました。この方法は、MTBC培養9,10から抽出したDNAのWGS用に開発および最適化されました。この分析法をMTB tNGSアッセイで使用するために最適化しました。21 サンプルの DNA 抽出と精製には、インキュベーションと遠心分離のステップを含めて、合計 4 時間 40 分でした。記載されているプロトコルの信頼性と、塗抹グレードが1+から3+まで変化するサンプルからのDNA抽出のためのビーズクリーンアップは、平均収量(図4)、組み合わせたすべてのターゲットの全体的なカバレッジ深度(図5)、および関連するWebアプリケーションによって決定されるシーケンシング結果の受容性スコア(図6)に基づいて実証されています。
最近の研究では、塗抹標本のグレードとDNA濃度またはシーケンシングリードの深さとの間に一貫した関係は見られませんでした。著者らは、サンプルの処理とクリーンアップのステップのばらつきが、特に汚染物質が効率的に除去されていない場合に、塗抹標本グレードの高いサンプルの性能に影響を与える可能性があることを示唆しました。さらに、喀痰沈殿物からのシーケンシングは、DNAプールの複雑さのために依然として困難であり、非標的DNAはMTB DNAと競合する可能性があります11。インプットDNA濃度とシーケンシング深度との間に明確な相関関係は観察されませんでしたが、ターゲットPCR前のDNAインプットは、メーカーの指示に従って100 ngを超えてはならないことに注意することが重要です。さらに、細菌量が多く、2+および3+の喀痰沈殿物サンプルには、ヒトDNA、細胞破片、その他の阻害剤などの汚染物質が多く含まれている可能性があり、PCR増幅やライブラリ調製に支障をきたす可能性があります。
精製ステップは濃縮ステップとしても機能し、tNGS前のマイコバクテリアDNAの収量を向上させます。この方法は、細菌負荷の少ないサンプルの処理に効果的であり、500 μLという少量でも信頼性の高い性能を発揮しているため、サンプル量が限られている日常的な設定に適しています。その適用により、クリニックへのフォローアップ訪問の必要性を減らすことができ、それによって患者がフォローアップのために失うリスクを最小限に抑えることができます。さらに、この方法はシンプルで、高度なラボの専門知識を必要としないため、リソースが限られている環境への統合をさらにサポートします。
ただし、プロトコルに関するいくつかの点に注意する必要があります。サンプルは、80°Cでのインキュベーション直後に凍結または冷蔵しないでください。 熱処理直後の急速な冷却により、チューブの内面に結露が発生し、サンプルが希釈され、下流のDNA抽出に影響を与える可能性があります。さらに、突然の冷却は、熱処理中に完全に不活性化されなかった可能性のある核酸の断片化または酵素活性を促進することにより、サンプルの劣化リスクを高める可能性があります。これらの考慮事項は、MTB検出のために喀痰サンプルを処理する場合に特に重要です。WHOのZiehl-Neelsen染色に関する推奨事項によると、塗抹グレード1+は100油浸フィールドあたり10-99 AFBに対応し、2+は少なくとも50フィールドのフィールドあたり1-10 AFBを示し、3+は少なくとも20フィールド12のフィールドあたり10 AFB以上を表します。
この方法の限界は、高速ホモジナイザーまたはビーズ叩き装置を使用した手動均質化の要件による自動化の欠如です。これに対処するために、インキュベーション時間の短縮、インキュベーション温度の低下、高速均質化への代替アプローチをテストすることにより、プロトコルの改良に継続的な取り組みが集中しています。
そこで、このマトリックスベースのDNA抽出法は、高速ホモジナイザーと組み合わせることで、熱とビーズビードの叩きによってゲノムDNAを放出すると同時に、金属やタンパク質などのPCR阻害物質を除去する高速DNA抽出技術です。
開示事項
著者のT.R.は、カリフォルニア大学サンディエゴ校とのサービス契約を通じて、FINDから資金援助を受けています。著者のT.R.は、薬剤耐性結核(R01AI176401)のtNGSソリューションを開発および評価するために、NIHから助成金を受け取りました。著者のT.R.は、Verus Diagnostics Inc.の共同創設者、取締役、および無給株主です。
謝辞
著者らは、技術支援を提供してくれた南アフリカのNHLS-グリーンポイントの結核研究所チームに感謝します。この研究は、国立衛生研究所(NIH)と革新的新診断財団(FIND)、TS ELiOTプロジェクト助成金ID:R01AI153213、Unitaid助成金ID:2019-32-FIND MDRの支援を受けました。フィギュアは BioRender.com を使用して作成しました。
資料
| Name | Company | Catalog Number | Comments |
| Alternative: Benchmark BeadBlaster 24 | Benchmark Scientific | D2400 | |
| AMPure XP Bead-Based Reagent | Beckman Coulter | A63881 | |
| Autoclaved or sterile glass beads (2 mm) | Sigma-Aldrich | Z273627-1EA | |
| Ethyl alcohol, Pure | Sigma-Aldrich | E7023-500ML | |
| Filter pipette tips (10 µL) | Bio-Smart Scientific | FT-10-R | |
| Filter pipette tips (1000 µL) | Bio-Smart Scientific | FT1000-R | |
| Filter pipette tips (20 µl) | Bio-Smart Scientific | FT20-R | |
| Filter pipette tips (200 µL) | Bio-Smart Scientific | FT200-R | |
| Heating-block ranging from 37 °C to 100 °C | Eppendorf | 5382000031 | |
| High-speed homogenizer (HSM) as FastPrep-24 | MP Biomedicals | 116005500 | |
| Incubator 80 °C | Lasec Group | IBLPDHG-9030A | |
| InstaGene Matrix | Bio-Rad Laboratories Inc. | BBRD7326030 | |
| Low-binding microtubes (1.5 mL or 2 mL) | Eppendorf | EP0030108051-250EA | |
| Magnetic rack for 1.5 – 2 mL tubes | Thermo Fisher Scientific Inc. | 12321D | |
| Magnetic stirrer | Merck | Z671886 | |
| Microcentrifuge | Eppendorf | 5406000046 | |
| Milli-Q IX 7015 Pure Water Purification System | Merck | ZIX7015T0C | |
| Nuclease-free water (NFW), molecular grade | Thermo Fisher Scientific Inc. | AM9937 | |
| Pipettes (P10, P20, P200, and P1000) | Eppendorf | EP3123000918-1EA | |
| Qubit Assay Tubes | Thermo Fisher Scientific Inc. | Q32856 | |
| Qubit dsDNA HS Assay Kit | Thermo Fisher Scientific Inc. | Q33231 | |
| Qubit fluorometer instrument | Thermo Fisher Scientific Inc. | Q33226 | |
| Screw cap tubes (1.5 mL) | Scientific Specialties Inc. | P2TUB056C-0001.5ST | |
| Vortex | Lasec Group | WMBB0L0E0216 |
参考文献
- Global Tuberculosis Report 2024. Geneva: WHO. , World Health Organization. https://www.who.int/teams/global-tuberculosis-programme/tb-reports/global-tuberculosis-report-2024 (2024).
- De Araujo, L., et al. Implementation of targeted next-generation sequencing for the diagnosis of drug-resistant tuberculosis in low-resource settings: a programmatic model, challenges, and initial outcomes. Front Public Health. 11, 1204064(2023).
- Murphy, S. G., et al. Direct detection of drug-resistant Mycobacterium tuberculosis using targeted next generation sequencing. Front Public Health. 11, 1206056(2023).
- Kambli, P., et al. Targeted next generation sequencing directly from sputum for comprehensive genetic information on drug resistant Mycobacterium tuberculosis. Tuberculosis. 127, 102051(2021).
- WHO Consolidated Guidelines on Tuberculosis.Module 3: Diagnosis. Rapid diagnostics for tuberculosis detection. Third edition. , World Health Organization. https://iris.who.int/bitstream/handle/10665/376221/9789240089488-eng.pdf?sequence=1 (2024).
- Catalogue of mutations in Mycobacterium tuberculosis. complex and their association with drug resistance. Second edition. , World Health Organization. https://iris.who.int/bitstream/handle/10665/374061/9789240082410-eng.pdf?sequence=1 (2023).
- Schwab, T. C., et al. Targeted next-generation sequencing to diagnose drug-resistant tuberculosis: a systematic review and meta-analysis. Lancet Infect Dis. 24 (10), 1162-1176 (2024).
- Deeplex Myc-TB manual version 5. , GenoScreen. https://acrobat.adobe.com/link/track?uri=urn%3Aaaid%3Ascds%3AUS%3A67c2b53f-c91d-3a73-a6ea-4769e7c8fe57 (2023).
- Shea, J., et al. Comprehensive whole-genome sequencing and reporting of drug resistance profiles on clinical cases of Mycobacterium tuberculosis in New York State. J Clin Microbiol. 55 (6), 1871-1882 (2017).
- Conceição, E. C., et al. Mycobacterium tuberculosis. DNA extraction using InstaGene Matrix and high-speed homogenizer from clinical primary culture. , https://www.protocols.io/view/mycobacterium-tuberculosis-dna-extraction-using-in-dhvs366e (2024).
- Nilgiriwala, K., et al. Genomic sequencing from sputum for tuberculosis disease diagnosis, lineage determination, and drug susceptibility prediction. J Clin Microbiol. 61 (3), e0157822(2023).
- Laboratory Services in Tuberculosis Control Part II: Microscopy. Geneva: WHO. , World Health Organization. https://iris.who.int/bitstream/handle/10665/65942/WHO_TB_98.258_(part2).pdf?sequence=2 (1998).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved