Method Article
מיצוי DNA מבוסס מטריצה לריצוף ממוקד של הדור הבא על דגימות ליחה מטוהרות
* These authors contributed equally
In This Article
Summary
אנו מציגים שיטה אופטימלית למיצוי DNA מליחה מזוהמת באמצעות שיטת מיצוי מבוססת מטריצה בשילוב עם טיהור חרוזים מגנטיים לריצוף ממוקד במורד הזרם של Mycobacterium tuberculosis.
Abstract
ריצוף הדור הבא (NGS) מוכר כיום ככלי רב עוצמה לאבחון בזמן ומדויק של שחפת עמידה לתרופות (DR-TB). NGS ממוקד (tNGS) מציע גישה יעילה על ידי התמקדות בגנים ספציפיים הקשורים לעמידות לתרופות, תוך עקיפת הצורך בשיטות מסורתיות מבוססות תרבית עם זמני אספקה הנעים בין שבועות לחודשים. ארגון הבריאות העולמי (WHO) המליץ על tNGS כאסטרטגיה רבת ערך לשיפור אבחון השחפת (TB) כדי להנחות את הטיפול ולשפר את תוצאות הטיפול, במיוחד בסביבות מוגבלות במשאבים. בין מבחני ה-tNGS המומלצים על ידי ארגון הבריאות העולמי, בחרנו בשיטה המספקת בדיקת רגישות מהירה ומקיפה לתרופות, קביעת שושלת והקלדת זנים. בעוד שקיימות שיטות מיצוי DNA סטנדרטיות, הן יכולות להיות גוזלות זמן ועתירות עבודה. כדי להתמודד עם אתגר זה, ביצענו אופטימיזציה של פרוטוקול מיצוי DNA פשוט ומבוסס מטריצה בשילוב עם טיהור חרוזים מגנטיים. שיטה זו מציעה גישה מהירה ויעילה לחילוץ DNA ישירות ממשקעי כיח מזוהמים, המאפשרת ניתוח tNGS מהיר במורד הזרם. על ידי ייעול תהליך מיצוי ה-DNA ממשקעי כיח, פרוטוקול זה יכול להקל על אימוץ רחב יותר של tNGS במסגרות קליניות שגרתיות, ובסופו של דבר לתרום לשיפור תוצאות המטופלים ולהעניק למאמצי בקרת השחפת העולמיים.
Introduction
על פי הערכות, 3.7 מיליון חולי שחפת לא אובחנו ולא טופלו ברחבי העולם בשנת 2023, מה שמדגיש את האיום המשמעותי שמציב השחפת לבריאות העולמית1. ארגון הבריאות העולמי מעריך כי כ-400,000 אנשים פיתחו שחפת עמידה לריפמפיצין (RR-TB) או שחפת עמידה לתרופות מרובות (MDR-TB) בשנת 20231. אבחון וטיפול מהיר בשחפת ובשחפת DR-TB חיוני להשגת אבני הדרך והיעדים להפחתת שכיחות ותמותה משחפת1.
הסתמכות על שיטות תרבית קונבנציונליות ובדיקת רגישות לתרופות פנוטיפיות (pDST) מעכבת את קביעת פרופיל העמידות של מבודדים קליניים וטיפול, עם זמני אספקה של 6-8 שבועות, ודורשת תשתית מורכבת של הכלה ביולוגית. בדיקות אבחון שגרתיות, בשילוב עם NGS של DR-TB, יכולות לספק פרופיל עמידות מקיף לתרופות ולשפר את ההתאמה האישית של משטרי MDR-TB, תוך צמצום הזמן לטיפול יעיל משבועות או חודשים לימים 2,3,4.
בשנים 2023 ו-2024, ארגון הבריאות העולמי המליץ על שימוש ב-tNGS כסוג חדש של אבחון כדי לקבוע במהירות את הרגישות לתרופות קו ראשון ושני נגד שחפת5. זה הופך אותו לכלי רב ערך להנחיית החלטות טיפול ללא צורך בתרבית Mycobacterium tuberculosis (MTB) במעבדות ברמת בטיחות ביולוגית 3 (BSL-3). גישת tNGS היא צורה מרוכזת של ריצוף המשתמשת בתגובת שרשרת פולימראז (PCR) כדי להגביר מטרות גנים המעניקים עמידות לתרופות לפני הריצוף. מבין בדיקות ה-tNGS המומלצות על ידי ארגון הבריאות העולמי, בחרנו בבדיקת Deeplex Myc-TB, שדווחה כעומדת בקריטריונים מבוססי הקבוצה לאיתור עמידות לריפמפיצין, איזוניאזיד, אתאמבוטול, פירזינמיד, פלואורוקווינולונים, אמיקאצין, סטרפטומיצין, לינזוליד, בדקווילין וקלופזימין. אנו משתמשים בבדיקה זו כדי להעריך את התאמת ה-DNA המופק באמצעות פרוטוקול זה עבור tNGS במורד הזרם.
בנוסף, ארגון הבריאות העולמי פרסםאת המהדורה השנייה של קטלוג המוטציות הקשורות לעמידות לתרופות ב-MTB, המספקת מפת דרכים לשימוש בריצוף גנום שלם (WGS) ו-tNGS כדי לחזות רגישות לתרופות ולהנחות את הטיפול6. סקירה שיטתית ומטא-אנליזה שנערכו לאחרונה הראו כי ל-tNGS יש רגישות וסגוליות של 94.1% ו-98.1%, בהתאמה, לזיהוי עמידות לתרופות, בהתבסס על 23 מטרות באזורים שונים המעניקים עמידות בגנום MTB, בהשוואה ל-pDST7.
עם זאת, יישום שיטות אלו נותר מאתגר בשל מורכבות ועלויות הקשורות במיוחד לתהליכי העבודה, התשתית והציוד הנדרשים. אתגר קריטי הוא בידוד מספיק DNA מיקובקטריאלי באיכות גבוהה ישירות מהמשקעים של ליחה מזוהרת, צעד מכריע ליישומי tNGS במורד הזרם. כדי לטפל בכך, אנו מציגים שיטת מיצוי DNA מהירה ופשוטה המותאמת ל-tNGS.
שיטת מיצוי ה-DNA הסטנדרטית עבור בדיקת MTB tNGS שנבחרה כוללת פרוטוקול ידני ואוטומטי8. כאן אנו מתארים פרוטוקול מיצוי DNA פשוט, מבוסס מטריצה (איור 1). השיטה עושה שימוש ב-InstaGene Matrix (IGM), הקושר מתכות וחלבונים ומאפשר מיצוי איכותי של חומצות גרעין ישירות ממשקעי כיח מזוהמים. חלופה זו מספקת זמן אספקה מהיר יותר ותפוקת DNA מספקת עבור tNGS במורד הזרם. פרוטוקול זה מתגבר על המורכבות של השיטות הידניות והאוטומטיות תוך הבטחת tNGS איכותי לאבחון מהיר של וריאנטים המעניקים עמידות ב-MTB. עם העניין הגובר בשימוש ב-tNGS בתחום המיקובקטריולוגיה, פרוטוקול זה יכול להקל על אימוצו בתהליכי עבודה אבחנתיים שגרתיים.

איור 1: ייצוג סכמטי של השיטות לחילוץ DNA מיקובקטריאלי מדגימות משקעי כיח מטוהרים באמצעות תרחיף מטריצה. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
Protocol
מחקר זה אושר על ידי ועדת האתיקה של המחקר: בטיחות ביולוגית וסביבתית (REC: BES) באוניברסיטת סטלנבוש: BES-2024-25384 וועדת האתיקה של מחקר בבני אדם: N21/09/093 ו- N09/11/296.
1. הכנת דגימת משקעי כיח
הערה: יש לבצע את השלבים הבאים במעבדה ברמת בטיחות ביולוגית שלוש (BSL3) לפני מיצוי DNA.
- הכן מנה של דגימת משקעי הליחה שטוהרה בעבר באמצעות N-אצטיל-L-ציסטאין (NALC) ונתרן הידרוקסיד (NaOH) (NALC/NaOH) על ידי העברת 500 μL-2 מ"ל לצינור בעל קשירה נמוכה של 1.5 מ"ל או 2 מ"ל.
- מניחים את הצינורות המכילים דגימות משקעי ליחה זקופים במתלה צינורות ומכניסים את הדגימות לתנור בטמפרטורה של 80 מעלות צלזיוס ודוגרים למשך שעה כדי לנטרל את המיקובקטריות באמצעות טיפול בחום.
- לאחר שעה, הסר את מתלה הצינור עם דגימות, נגב את משטחי הצינור והנח אותם במיכל מתאים להובלה למעבדה ברמת בטיחות ביולוגית שתיים (BSL2).
הערה: אפשר לדוגמא להתקרר לטמפרטורת החדר לפני טיפול נוסף או אחסון.
2. מיצוי DNA באמצעות תרחיף המטריצה והומוגנייזר במהירות גבוהה (איור 2)
- לפני שמתחילים, הוסף שלושה חרוזי זכוכית בקוטר 2 מ"מ לצינורות מכסה בורג של 1.5 או 2 מ"ל, מכוסים באופן רופף וחיטוי (באופן אידיאלי לפחות יום לפני מיצוי ה-DNA).
- הסר את תרחיף המטריצה מהמקרר והנח אותו על מערבל מגנטי (מגנט כלול בבקבוק המגיב) כדי להביא את המגיב לטמפרטורת החדר ולהתחיל לערבב את המגיב.
- הפעל את בלוק החימום וחמם מראש ל-56 מעלות צלזיוס (אם קיימים שני גושי חימום, מחממים את השני ל-100 מעלות צלזיוס.
הערה: ניתן לבצע את השלבים הבאים על ספסל מעבדה במתקן BSL2: - צנטריפוגה את דגימת משקעי הליחה המומתים בחום ב-16,800 × גרם (או במהירות מלאה) למשך 15 דקות. בעזרת פיפטה של 20-200 מיקרוליטר שאפו בעדינות והשליכו את הסופרנטנט, כדי להבטיח שהכדור יישאר ללא הפרעה.
- מערבבים את מתלה המטריצה על ידי ניעור עדין (לא מערבולת), ומוסיפים 200 מיקרוליטר מהמטריצה המעורבת היטב כדי להשעות מחדש את הגלולה.
- הכניסו את הנפח המלא לתוך צינור פקק בורג של 1.5 מ"ל המכיל שלושה חרוזי זכוכית מעוקרים בקוטר 2 מ"מ.
- מניחים את הדגימות בבלוק חימום ודוגרים בחום של 56 מעלות צלזיוס למשך 15 דקות.
הערה: מיד לאחר הוצאת הדגימה מגוש החימום, הגדר את בלוק החימום ל-100 מעלות צלזיוס לאחר שלב 2.7, אם רק בלוק חימום אחד זמין. - הומוגניזציה של הדגימות באמצעות מערבולת למשך 10 שניות כדי לפזר את התאים.
- מניחים את הדגימות בגוש חימום ומדגרים בחום של 100 מעלות צלזיוס למשך 10 דקות.
- הומוגניזציה של הדגימות באמצעות הומוגנייזר מהיר עם הפרמטרים הבאים: מחזור אחד של 60 שניות ב-4.0 מ'/שנייה.
- צנטריפוגה במשקל 12,000 × גרם למשך 5 דקות. העבירו 130 מיקרוליטר של הסופרנטנט המכיל DNA לצינור טרי בעל קשירה נמוכה של 1.5 מ"ל מבלי להפריע לגלולה (מטריצה עם פסולת תאים). השלך את הצינור הראשון המכיל את המטריצה ופסולת התא.
הערה: נקודת עצירה בטוחה. אם עוצרים, הקפיאו דגימות בטמפרטורה של -20 מעלות צלזיוס עד שהן מוכנות להמשיך בטיהור ה-DNA. - המשך בטיהור DNA באמצעות חרוזים מגנטיים.

איור 2: שיטת מיצוי DNA מבוססת-מטריצה בשילוב עם הומוגנייזר במהירות גבוהה. קיצור: HSH = הומוגנייזר במהירות גבוהה. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
3. טיהור דנ"א באמצעות חרוזים מגנטיים (איור 3)
הערה: לפני שתמשיך בטיהור ה-DNA, בצע את שלבים 3.1 ו-3.2.
- הוציאו את החרוזים המגנטיים מהמקרר והניחו את המיכל בטמפרטורת החדר למשך 30 דקות לשיווי משקל. לחלופין, מערבל את בקבוק מלאי החרוזים המגנטי כדי להשעות מחדש את כל החרוזים ולשאוב 1,280 מיקרוליטר לתוך צינור חדש (לטיהור של 21 דגימות) ולהניח בטמפרטורת החדר כדי לאזן את האיזון.
- הכן אצווה של 9 מ"ל של 80% אתנול על ידי הוספת 7.2 מ"ל אלכוהול אתילי טהור ל-1.8 מ"ל מים מסוננים ומעוקרים.
הערה: ניתן לבצע את השלבים הבאים על ספסל מעבדה במתקן BSL2. - מערבל היטב את בקבוק מלאי החרוזים המגנטי או את האליקוט המוכן כדי להבטיח שהחרוזים מושעים מחדש לפני השימוש. חזור על המערבולת לאחר כל 10 דגימות כדי להבטיח השעיה הומוגנית.
- הוסף נפח פי 1.2 (156 מיקרוליטר) של החרוזים המגנטיים ל-DNA המופק (130 מיקרוליטר).
הערה: עבור דגימות עם כמויות גדולות יותר של DNA (תרבית), ניתן לעבד כמות קטנה יותר של הדגימה (למשל, 50 מיקרוליטר) ולטהר אותה בנפח פי 1.2 של חרוזים מגנטיים. - מערבבים את הדגימות על ידי פיפטינג פי 10. דגרו את הדגימות למשך 5 דקות בטמפרטורת החדר.
- הנח את הדגימות על המתלה המגנטי של צינור 1.5 מ"ל למשך 3 דקות או עד שהנוזל מתבהר. שאפו בזהירות והשליכו את הסופרנטנט מבלי להפריע לחרוזים.
- עם הצינורות על המגנט, הוסף 200 מיקרוליטר של 80% אתנול, וודא שהחרוזים יישארו ללא הפרעה ומאפשרים לדגור למשך 30 שניות.
- שאפו בזהירות והשליכו את האתנול מבלי להפריע לחרוזים.
- חזור על שלב 3.7 בסך הכל שתי כביסות.
- לאחר הכביסה השנייה, הסר והשליך את האתנול. הסר את כל שאריות האתנול בעזרת פיפטה של 1-10 מיקרוליטר.
- השאירו את הצינורות פתוחים לייבוש החרוזים באוויר למשך 10 דקות, או עד שהחרוזים בעלי מראה מט.
הערה: אל תאפשר לחרוזים להתייבש יתר על המידה ולהיסדק. - ברגע שלחרוזים יש מראה מט, הסר מיד את הצינורות מהמגנט והוסף 50 מיקרוליטר של מים נטולי נוקלאז (NFW) ישירות על החרוזים בכל דגימה. לאחר הוספת NFW לכל הצינורות, מערבבים כל דגימה בודדת על ידי פיפטינג פי 10. בדוק את הצינור לאיתור חרוזים שנתקעו בחלק הפנימי של הצינור וחזור על שלב הערבוב במידת הצורך. דגרו את הדגימות למשך 5 דקות בטמפרטורת החדר.
הערה: ניתן להגדיל את הדגירה מ-5 דקות ל-10 דקות, אך אין להפחית את זמן הדגירה לפחות מ-5 דקות. - הנח את הצינורות בחזרה על המדף המגנטי למשך 3 דקות או עד שהנוזל צלול. עם הצינורות על המדף המגנטי, העבירו את הסופרנטנט המכיל DNA לצינור מסומן בבירור, סטרילי ובעל קשירה נמוכה. ודא שהחרוזים לא מועברים לסופרנטנט.
הערה: אם נראים חרוזים בקצה הפיפטה במהלך ההעברה, פיפטה את הסופרנטנט בחזרה לתוך הצינור על המתלה המגנטי כדי להפריד למשך 3 דקות נוספות. אין להעביר חרוזים לתוך הצינור החדש.
נקודת עצירה בטוחה. אם עוצרים, הקפיאו דגימות ב-20 מעלות צלזיוס עד שיהיו מוכנות להמשיך בכימות בשיטה המתאימה ל-NGS והגברת PCR לאחר מכן באמצעות בדיקת MTB tNGS.
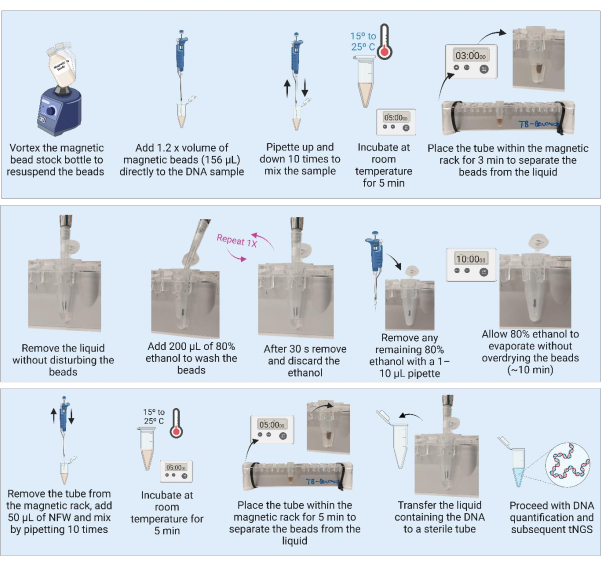
איור 3: טיהור וריכוז דנ"א באמצעות חרוזים מגנטיים. קיצורים: NFW = מים נטולי נוקלאז; tNGS = ריצוף ממוקד של הדור הבא. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
תוצאות
תיאור לדוגמא
בסך הכל 165 דגימות משקעי כיח, חיוביות לבצילי מהיר חומצה (AFB) במיקרוסקופיה עם עומס חיידקי של לפחות 1+, נאספו ועובדו באופן שגרתי על ידי שירותי מעבדת הבריאות הלאומיים (NHLS) גרין פוינט, קייפטאון, דרום אפריקה. DNA הופק מדגימות משקעי כיח באמצעות שני נפחים שונים [כ-2 מ"ל (n = 102) ו-500 מיקרוליטר (n = 63)]. השוואה זו נערכה כדי להעריך אם נפח דגימת המשקעים הקטן יותר יכול להניב מספיק DNA עבור tNGS במורד הזרם.
איור 4 מציג השוואה של תרשים קופסה של ריכוז ה-DNA הכולל (ng/μL) על פני דרגות מריחת כיח שונות (1+, 2+ ו-3+) ונפחי משקעי כיח (2 מ"ל ו-500 מיקרוליטר). תפוקת ה-DNA בדרך כלל יורדת באופן כללי כאשר משתמשים ב-500 מיקרוליטר של משקעים למיצוי, עם שונות בין דרגות המריחה. לפיכך, ריכוזי ה-DNA הממוצעים שחולצו מדגימות משקעים בנפח של לפחות 2 מ"ל היו גבוהים בממוצע מריכוז ה-DNA שהופק ממשקעים של 500 מיקרוליטר, מרובד על ידי AFB של הדגימה.

איור 4: השפעת נפח משקעי הליחה על תפוקת ה-DNA על פני דרגות מריחה. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
איור 5 מציג תרשימי קופסאות הממחישים את המספר הממוצע של קריאות ריצוף (עומק כיסוי ממוצע) של כל המטרות עבור דגימות משקעי כיח עם דרגות מריחה משתנות (1+, 2+ ו-3+) המעובדות משני נפחי קלט שונים (2 מ"ל ו-500 מיקרוליטר). חריגים מיוצגים על ידי נקודות בודדות, וציר ה-y מוצג בסולם לוגריתמי כדי להתאים לשונות בכיסוי. הטווח הבין-רבעוני הגבוה עבור 3+ דגימות שהופקו ממשקעי 500 מיקרוליטר מרמז על שונות גדולה יותר עם עומסי חיידקים גבוהים יותר.

איור 5: השוואה של המספר הממוצע של קריאות רצף (עומק כיסוי) עבור כל המטרות על פני דרגות מריחה ונפחי קלט דגימה. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
איור 6 מתאר את הערכת הקבילות של תוצאות ריצוף באמצעות צינור הביואינפורמטיקה של יישום האינטרנט המשויך. ציוני איכות הרצף מסווגים כ-+++ (מקובל מאוד), ++ (מקובל), + (מקובל בשוליים), - (לא מקובל) ו-ND (לא נקבע). היעדר עמודה בגרף מצביע על כך שלא התקבלו תוצאות עבור קטגוריה ספציפית זו. דרגת מריחה 1+ מציגה שיעור גבוה יותר של תוצאות לא מקובלות ו-ND בהשוואה לדרגות מריחה 2+ ו-3+ עבור ערכת הדגימה של נפח הקלט של 500 מיקרוליטר. לדגימות שחולצו מ-500 מיקרוליטר של משקעי כיח היה עומק כיסוי ריצוף ממוצע של 4,316 בהשוואה ל-4,810 עבור דגימות שחולצו ממשקעי כיח של 2 מ"ל. זה מצביע על כך ש-DNA שהופק ממשקעי כיח היה מספיק לביצוע tNGS במורד הזרם, ללא קשר לנפח הכניסה.
בהתבסס על התפלגות קבילות תוצאות הריצוף, דגימות עם ציון מריחה של 3+ היו בעלות שיעור ההצלחה הגבוה ביותר כאשר רוב רצפי הדגימה קיבלו ציון של ++ ו-+++ בהשוואה לדגימות בדרגת מריחה 1+ ו-2+. עבור דגימות הקלט של 500 מיקרוליטר, שיעור הדגימות בקטגוריות - ו-+ גבוה יחסית בכל דרגות המריחה בהשוואה לדגימות הקלט של 2 מ"ל. זה מצביע על כך שיותר דגימות נופלות לקטגוריות הפחות איכותיות והפחות מקובלות עבור קלט 500 מיקרוליטר. עבור דגימות הקלט של 2 מ"ל, יש שיעור גבוה יותר של דגימות בקטגוריות האיכותיות יותר, ++ ו-+++, במיוחד עבור מריחה בדרגה 3+. זה מצביע על כך שדגימות עם קלט של 2 מ"ל נוטות יותר לייצר ציוני איכות ריצוף טובים יותר בהשוואה לאלו עם קלט של 500 מיקרוליטר.

איור 6: התפלגות ציוני האיכות לפי דרגת מריחה ונפחי קלט מדגם. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
Discussion
השילוב של שיטת המיצוי מבוססת המטריצה יחד עם הומוגנייזר מהיר פורסם במקור על ידי Shea et al.9. שיטה זו פותחה ומותאמת ל-WGS של DNA המופק מתרביות MTBC 9,10. ביצענו אופטימיזציה של שיטה זו לשימוש עם בדיקת MTB tNGS. למיצוי וטיהור ה-DNA של 21 דגימות היה זמן אספקה משולב של 4 שעות ו-40 דקות, כולל שלבי דגירה וצנטריפוגה. האמינות של הפרוטוקול המתואר יחד עם ניקוי חרוזים עבור מיצוי DNA מדגימות עם דרגות מריחה המשתנות בין 1+ ל-3+ מודגמת על סמך תפוקה ממוצעת (איור 4), עומק הכיסוי הכולל של כל המטרות ביחד (איור 5), וציון קבילות תוצאת הריצוף כפי שנקבע על ידי יישום האינטרנט המשויך (איור 6).
מחקר שנערך לאחרונה לא מצא קשר עקבי בין דרגת מריחה לריכוז ה-DNA או עומק קריאת הרצף. המחברים הציעו כי שונות בשלבי עיבוד וניקוי הדגימה עשויה להשפיע על הביצועים בדגימות בדרגת מריחה גבוהה, במיוחד כאשר מזהמים אינם מוסרים ביעילות. בנוסף, ריצוף ממשקעי כיח נותר מאתגר בשל המורכבות של מאגר ה-DNA, שבו DNA שאינו מטרה יכול להתחרות ב-MTB DNA11. אמנם לא ראינו מתאם ברור בין ריכוז ה-DNA הקלט לעומק הריצוף, אך חשוב לציין שקלט ה-DNA לפני ה-PCR המטרה לא יעלה על 100 ננוגרם, בהתאם להוראות היצרן, מכיוון שהדבר עלול להוביל לעיכוב PCR. בנוסף, עומס חיידקים גבוה יותר, דגימות משקעי כיח 2+ ו-3+ עלולות להכיל יותר מזהמים, כגון DNA אנושי, פסולת תאים ומעכבים אחרים, שעלולים להפריע להגברת PCR והכנת הספרייה.
שלב הטיהור מתפקד גם כשלב ריכוז, ומשפר את התפוקה של DNA מיקובקטריאלי לפני tNGS. שיטה זו יעילה לעיבוד דגימות עם עומס חיידקים נמוך והדגימה ביצועים אמינים עם נפחים קטנים עד 500 מיקרוליטר, מה שהופך אותה למתאימה להגדרות שגרתיות שבהן נפח דגימה מוגבל זמין. היישום שלו יכול להפחית את הצורך בביקורי מעקב במרפאות, ובכך למזער את הסיכון לאובדן המטופל למעקב. בנוסף, השיטה פשוטה ואינה דורשת מומחיות מעבדתית מתקדמת, מה שתומך עוד יותר בשילובה במסגרות מוגבלות במשאבים.
עם זאת, יש לציין כמה נקודות לגבי הפרוטוקול. אין להקפיא או לקרר דגימות מיד לאחר הדגירה בטמפרטורה של 80 מעלות צלזיוס. קירור מהיר מיד לאחר טיפול בחום עלול לגרום להיווצרות עיבוי על המשטחים הפנימיים של הצינור, מה שמוביל לדילול הדגימה ועלול להשפיע על מיצוי ה-DNA במורד הזרם. בנוסף, קירור פתאומי עלול להגביר את הסיכון לפירוק הדגימה על ידי קידום פיצול חומצות גרעין או פעילות אנזימטית שאולי לא הושבתו לחלוטין במהלך טיפול בחום. שיקולים אלה רלוונטיים במיוחד בעת עיבוד דגימות ליחה לגילוי MTB. על פי המלצות ארגון הבריאות העולמי לצביעה של Ziehl-Neelsen, דרגת מריחה של 1+ מתאימה ל-10-99 AFB לכל 100 שדות טבילה בנפט, 2+ מציין 1-10 AFB לשדה ב-50 שדות לפחות, ו-3+ מייצג יותר מ-10 AFB לשדה ב-20 שדות לפחות12.
מגבלה של שיטה זו היא היעדר האוטומציה שלה עקב הדרישה להומוגניזציה ידנית באמצעות הומוגנייזר מהיר או מכשיר מכות חרוזים. כדי להתמודד עם זה, מאמצים מתמשכים מתמקדים בחידוד הפרוטוקול על ידי בדיקת זמני דגירה קצרים יותר, טמפרטורות דגירה נמוכות יותר וגישות חלופיות להומוגניזציה במהירות גבוהה.
לפיכך, שיטת מיצוי DNA מבוססת מטריצה זו, בשילוב עם הומוגנייזר במהירות גבוהה, היא טכניקת מיצוי DNA מהירה המשתמשת בחום ובפעימות חרוזים כדי לשחרר DNA גנומי תוך הסרת מעכבי PCR, כגון מתכות וחלבונים.
Disclosures
הסופר T. R. מקבל תמיכה במימון מ-FIND באמצעות חוזה שירות עם UC San Diego. המחבר T. R. קיבל מימון מענק מ-NIH לפיתוח והערכה של פתרון tNGS לשחפת עמידה לתרופות (R01AI176401). המחבר T. R. הוא מייסד שותף, חבר דירקטוריון ובעל מניות ללא תשלום של Verus Diagnostics Inc.
Acknowledgements
המחברים רוצים להודות לצוות מעבדת השחפת ב-NHLS - Green Point, דרום אפריקה על מתן סיוע טכני. עבודה זו נתמכה על ידי המכונים הלאומיים לבריאות (NIH) והקרן לאבחון חדש וחדשני (FIND), מזהה מענק פרויקט TS ELiOT: R01AI153213, מזהה מענק Unitaid: 2019-32-FIND MDR. הדמויות נוצרו באמצעות BioRender.com.
Materials
| Name | Company | Catalog Number | Comments |
| Alternative: Benchmark BeadBlaster 24 | Benchmark Scientific | D2400 | |
| AMPure XP Bead-Based Reagent | Beckman Coulter | A63881 | |
| Autoclaved or sterile glass beads (2 mm) | Sigma-Aldrich | Z273627-1EA | |
| Ethyl alcohol, Pure | Sigma-Aldrich | E7023-500ML | |
| Filter pipette tips (10 µL) | Bio-Smart Scientific | FT-10-R | |
| Filter pipette tips (1000 µL) | Bio-Smart Scientific | FT1000-R | |
| Filter pipette tips (20 µl) | Bio-Smart Scientific | FT20-R | |
| Filter pipette tips (200 µL) | Bio-Smart Scientific | FT200-R | |
| Heating-block ranging from 37 °C to 100 °C | Eppendorf | 5382000031 | |
| High-speed homogenizer (HSM) as FastPrep-24 | MP Biomedicals | 116005500 | |
| Incubator 80 °C | Lasec Group | IBLPDHG-9030A | |
| InstaGene Matrix | Bio-Rad Laboratories Inc. | BBRD7326030 | |
| Low-binding microtubes (1.5 mL or 2 mL) | Eppendorf | EP0030108051-250EA | |
| Magnetic rack for 1.5 – 2 mL tubes | Thermo Fisher Scientific Inc. | 12321D | |
| Magnetic stirrer | Merck | Z671886 | |
| Microcentrifuge | Eppendorf | 5406000046 | |
| Milli-Q IX 7015 Pure Water Purification System | Merck | ZIX7015T0C | |
| Nuclease-free water (NFW), molecular grade | Thermo Fisher Scientific Inc. | AM9937 | |
| Pipettes (P10, P20, P200, and P1000) | Eppendorf | EP3123000918-1EA | |
| Qubit Assay Tubes | Thermo Fisher Scientific Inc. | Q32856 | |
| Qubit dsDNA HS Assay Kit | Thermo Fisher Scientific Inc. | Q33231 | |
| Qubit fluorometer instrument | Thermo Fisher Scientific Inc. | Q33226 | |
| Screw cap tubes (1.5 mL) | Scientific Specialties Inc. | P2TUB056C-0001.5ST | |
| Vortex | Lasec Group | WMBB0L0E0216 |
References
- Global Tuberculosis Report 2024. Geneva: WHO. , World Health Organization. https://www.who.int/teams/global-tuberculosis-programme/tb-reports/global-tuberculosis-report-2024 (2024).
- De Araujo, L., et al. Implementation of targeted next-generation sequencing for the diagnosis of drug-resistant tuberculosis in low-resource settings: a programmatic model, challenges, and initial outcomes. Front Public Health. 11, 1204064(2023).
- Murphy, S. G., et al. Direct detection of drug-resistant Mycobacterium tuberculosis using targeted next generation sequencing. Front Public Health. 11, 1206056(2023).
- Kambli, P., et al. Targeted next generation sequencing directly from sputum for comprehensive genetic information on drug resistant Mycobacterium tuberculosis. Tuberculosis. 127, 102051(2021).
- WHO Consolidated Guidelines on Tuberculosis.Module 3: Diagnosis. Rapid diagnostics for tuberculosis detection. Third edition. , World Health Organization. https://iris.who.int/bitstream/handle/10665/376221/9789240089488-eng.pdf?sequence=1 (2024).
- Catalogue of mutations in Mycobacterium tuberculosis. complex and their association with drug resistance. Second edition. , World Health Organization. https://iris.who.int/bitstream/handle/10665/374061/9789240082410-eng.pdf?sequence=1 (2023).
- Schwab, T. C., et al. Targeted next-generation sequencing to diagnose drug-resistant tuberculosis: a systematic review and meta-analysis. Lancet Infect Dis. 24 (10), 1162-1176 (2024).
- Deeplex Myc-TB manual version 5. , GenoScreen. https://acrobat.adobe.com/link/track?uri=urn%3Aaaid%3Ascds%3AUS%3A67c2b53f-c91d-3a73-a6ea-4769e7c8fe57 (2023).
- Shea, J., et al. Comprehensive whole-genome sequencing and reporting of drug resistance profiles on clinical cases of Mycobacterium tuberculosis in New York State. J Clin Microbiol. 55 (6), 1871-1882 (2017).
- Conceição, E. C., et al. Mycobacterium tuberculosis. DNA extraction using InstaGene Matrix and high-speed homogenizer from clinical primary culture. , https://www.protocols.io/view/mycobacterium-tuberculosis-dna-extraction-using-in-dhvs366e (2024).
- Nilgiriwala, K., et al. Genomic sequencing from sputum for tuberculosis disease diagnosis, lineage determination, and drug susceptibility prediction. J Clin Microbiol. 61 (3), e0157822(2023).
- Laboratory Services in Tuberculosis Control Part II: Microscopy. Geneva: WHO. , World Health Organization. https://iris.who.int/bitstream/handle/10665/65942/WHO_TB_98.258_(part2).pdf?sequence=2 (1998).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved