Method Article
DNA-タンパク質相互作用を研究するためのCD分光法
要約
ATP依存クロマチンリモデラーとDNAリガンドとの相互作用は、CD分光法を用いて説明されている。生成されたピークによって分析された遺伝子プロモーター上の誘導された立体構造変化を使用して、転写調節のメカニズムを理解することができる。
要約
循環二色性(CD)分光法は、生体分子の二次構造と相互作用を調べるのに簡単で便利な方法です。CD分光法の最近の進歩により、さまざまな微小環境におけるDNA-タンパク質相互作用と立体構造ダイナミクスの研究が可能になり 、インビボにおける転写調節の理解が深くなっています。転写が起こるためには、転写領域の周囲の領域を巻き戻す必要があります。これは、ヒストン修飾の調整、DNAへの転写因子の結合、および他のクロマチンリモデリング活動を必要とする複雑なプロセスである。CD分光法を用いて、ATP依存クロマチンリモデル剤などの調節タンパク質によって引き起こされるプロモーター領域における立体構造変化を研究し、転写を促進することができる。タンパク質に生じる立体構造変化もモニターできる。また、標的DNAに対するタンパク質の親和性や配列特異性に関する問い合わせは、標的DNAに変異を組み込むことによって対処することができる。要するに、この敏感で安価な方法のユニークな理解は、クロマチンダイナミクスの変化を予測することができ、それによって転写調節の理解を改善する。
概要
円二色性(CD)は、右利きと左利きの円偏光の差動吸収につながる生体高分子の固有のキラリティに依存する分光技術です。この差動吸収は、円形のジクロリズムとして知られています。したがって、この技術は、キラルセンター1,2を含むタンパク質およびDNAなどの生物学的高分子の立体構造を表現するために使用することができる。
電磁波には、電気と磁気の両方のコンポーネントが含まれています。電気と磁界は、波の伝播方向に対して垂直に振動します。偏光のない光の場合、これらのフィールドは多くの方向に振動します。光が円偏光すると、互いに90°位相差で2つの電磁場が得られます。キラル分子は、右利きの円偏光と左利きの円偏光を異なる範囲に吸収するような円状の光学回転(複屈折)を示す3。得られた電界は、波長の関数である楕円としてトレースされます。このCDスペクトルは、このように、楕円度(q)として記録され、データは波長の関数として平均残基楕円度として提示される。
タンパク質の場合、アミノ酸のCα(グリシンを除く)はキラルであり、これはCD分光法によって利用され、この高分子4の二次構造を決定する。タンパク質分子のCDスペクトルは、通常、遠紫外線範囲で記録されます。αヘリカルタンパク質は、222 nmおよび208 nmで2つの陰性バンドを有し、193 nm4で1つの正のピークを有する。抗平行βシート二次構造を有するタンパク質は、218 nmで負のピークを示し、195 nm4で正のピークを示す。無秩序な構造を有するタンパク質は、210 nm付近の低い楕円性と195 nm4で負のピークを示す。したがって、異なる二次構造の明確なピーク/バンドは、CDを作る変性とリガンド結合の間にタンパク質の二次構造に起こる構造変化を解明するための便利なツールです。
核酸には、糖分子、二次構造のヘリシティ、および環境中のDNAの長期三次配列の3つの源があります5,6。核酸のCDスペクトルは、典型的には190〜300 nm範囲5,6で記録される。DNAの各立体構造は、タンパク質と同様に特徴的なスペクトルを与えますが、ピーク/バンドは溶媒条件とDNA配列の違いのためにある程度変化する可能性があります7。最も一般的な形態であるB-DNAは、260〜280 nm前後の陽性ピークと245 nm6の周りの陰性ピークによって特徴付けられる。B型DNAのピーク/バンドは、塩基対が二重らせんに垂直であるため、一般的に小さく、分子に弱いキラリティを与える。A-DNAは260 nmで優勢な陽性ピークを与え、210 nm6の周りに陰性のピークを与える。左利きのらせんであるZ-DNAは、290nmで陰性バンドを与え、260 nm6の周りに正のピークを与える。このDNAはまた、205 nm6で非常に陰性のピークを与える。
これらの立体構造に加えて、DNAはトリプレックス、四重プレックス、ヘアピンを形成することができ、そのすべてがCD分光法によって区別することができる。並列G-quadruplexは260nmで優勢な正のバンドを与え、反平行G-quadruplexは260 nmで負のバンドを与え、290 nmで正のピークを与え、2つの形態の四重プレックス構造6を区別することを容易にする。トリプレックスは特徴的なスペクトル8を与えない。例えば、Na+ 存在下でG.G.CとT.A.T塩基対を含む分子内三重らせんを形成する可能性のある36ヌクレオチド長DNAのスペクトルは、240nmで強い陰性バンドを示し、広い陽性ピークを示す。広い正のピークは266、273、および286 nmの貢献を示す。Na+ およびZn+ の存在下での同じオリゴヌクレオチドは、4つの陰性バンド(213、238、266、および282 nm)および258nmで陽性のピークを示す。したがって、トリプレックスDNAのスペクトルは、塩条件に応じて変化する可能性がある8。
これらの立体構造に加えて、CDスペクトルはX-DNAと呼ばれる別の形態のDNAの同定を可能にした。X-DNAは、DNA配列に代替アデニンおよびチミン残基が含まれるときに形成される。X-DNAのCDスペクトルは250および280 nmの2つの陰性ピークを含んでいる。X-DNAに関する情報はほとんどないが、陽性スーパーコイル6,9のシンクとして機能すると推測されている。CDスペクトルの変化はまた、リガンドとタンパク質相互作用に関する詳細を明らかにすることができ、したがって、薬物タンパク質相互作用を検出するための分子方法の武器庫に追加された10、11、12、13、14。CDスペクトルは、折り畳みプロセス15中のタンパク質の二次構造の変化を監視するためにも使用されてきた。同様に、CDスペクトルは、リガンドとDNA相互作用の調査にも使用できる16,17。
CD分光法は、このように、DNAの構造の異なる形態を区別する容易で安価な方法であり、それほど安価ではない機器およびソフトウェアへのアクセスがある場合である。この方法は非常に敏感で迅速です。少量のDNAしか必要としないため、核磁気共鳴(NMR)分光法の代替技術に優位性を与えます。リガンドや基質を用いた滴定も容易に行えます。主な制約は、DNAが非常に純粋でなければならないということです。ポリアクリルアミドゲル電気泳動(PAGE)精製DNAを使用することをお勧めします。
CDスペクトルによって得られた情報は、主にタンパク質の構造特徴を推測し、明確なDNA適合体を同定するために使用されてきました。本研究では、イン ビボ クロマチン免疫沈降(ChIP)実験から得られた結果を統合し、目的のタンパク質/予測転写因子がそのエフェクター遺伝子のプロモーター領域に立体構造変化をもたらすかどうかを示すために使用されています。このコラボレーションは、プロモーターの転写開始部位(TSS)とその周辺で予測された転写因子による転写調節機構を予測することにより、従来のCD分光技術の進行を助ける。
クロマチンリモデリングは、密に詰まったクロマチンを転写因子、DNA複製の構成要素、損傷修復タンパク質などの様々な調節因子にアクセスできるようにすることで、DNA代謝プロセスを調節することが知られている明確なメカニズムです。ATP依存クロマチンリモデル剤は、SWI/SNFファミリーのタンパク質とも呼ばれ、真核細胞に存在する主要なリモデルタンパク質である18,19です。系統的クラスタリングは、SWI/SNFファミリーのタンパク質を6つのサブグループ20に分類しました:Snf2のような、Swr1のような、SSO1653のような、Rad54のような、Rad5/16のような、そして遠く離れています。本研究で関心のあるタンパク質であるSMARCAL1は、遠方のサブグループ20に属する。このタンパク質は、CD分光法を用いた転写調節の様式を調べるのに用いられている。
ATP依存クロマチンリモデリングタンパク質のメンバーのほとんどは、核酸を再位置または除去するか、またはATP依存的な様式でヒストン変異体交換を媒介することが示されている21,22。しかし、このファミリーの一部のメンバーは、例えばSMARCAL1のヌクレオソームを改造することが示されていない。SMARCAL1がポリテネ染色体と関連することが研究で示されているにもかかわらず、ヌクレオソームを改造する能力に関する実験的証拠は欠けている23。そこで、SMARCAL1はDNA24の立体構造を変化させることによって転写を調節する可能性があると仮定した。CD分光法は、この仮説を検証するための簡単でアクセス可能な方法を提供した。
SMARCAL1は、主にアニーリングヘリカーゼ25,26,27として機能するATP依存クロマチンリモデリングタンパク質です。DNAコンフォメーション24を改造して転写を変調するように仮定されている。この仮説を検証するために、ドキソルビシン誘発DNA損傷時の遺伝子転写を調節するSMARCAL1の役割を検討した。これらの研究では、SMARCAL1はインビボ分析とADAADのインビトロアッセイ28,29に使用されました。これまでの研究では、ADAADは構造に依存するが配列に依存しない方法でDNAを認識できることが示されています30,31。このタンパク質は、ステムループDNAと同様に、二本鎖を有するDNA分子と一本鎖転移領域に最適に結合し、ATP 30,31を加水分解する。
インビボ実験は、SMARCAL1がプロモーター領域28,29に結合することによってMYC、DROSHA、DGCR8およびDICERの発現を調節することを示した。相互作用の領域は、ChIP実験28,29によって同定された。ChIP技術は、細胞内の同一DNAとタンパク質の相互作用を分析するために使用されます。その目標は、プロモーターまたは他のDNA結合部位上の転写因子などの特定のタンパク質が特定のゲノム領域に結合しているかどうかを判断することです。DNAに結合したタンパク質は、ホルムアルデヒドを用いて最初に架橋される。その後、クロマチンの単離が続きます。分離されたクロマチンは、超音波処理またはヌクレアーゼ消化のいずれかによって500bp断片に切断され、DNAに結合したタンパク質は、タンパク質に特異的な抗体を用いて免疫沈降する。架橋は逆転し、DNAはポリメラーゼ連鎖反応(PCR)または定量的リアルタイムPCRを用いて分析されます。
ChIPの結果は、SMARCAL1がこれらの遺伝子のプロモーター領域の立体構造変化を誘導することによって転写調節を媒介する可能性があるという仮説につながった。QGRSマッパーおよびMfoldソフトウェアは、これらのプロモーター領域が二次構造を形成する可能性を同定するために使用された28,29。QGRSマッパーはG-quadruplexes32の予測に使用され、Mfold33はステムループなどの二次構造を形成するシーケンスの能力を分析します。
二次構造解析の後、さらにインビトロ実験を組換え6X Hisタグ付き活性DNA依存ATPase Aドメイン(ADAAD)、SMARCAL1のウシホモログ、大腸菌から精製したATPaseアッセイは、同定されたDNA配列がエフェクター28,29として作用し得ることを確立するためにADAADを用いて行った。最後に、ADAAD28,29によってDNA分子に誘導された立体構造変化をモニターするためにCD分光法を行った。
タンパク質のATPase活性がDNA分子の立体構造変化を誘導するために不可欠であることを証明するために、エチレンジアミンテトラ酢酸(EDTA)をキレートMg+2または活性DNA依存ATPase Aドメイン阻害剤ネオマイシン(ADAADiN)に添加した。.このCD分光技術は、プロモーターの予測ゲノム領域に結合するために、ChIPまたは他の任意の関連アッセイによって実証された任意の精製タンパク質と利用することができる。
プロトコル
1. 反応成分の働き濃度
- CDやその他の反応成分のバッファーの作業濃度を新たに準備し( 表1参照)、4°Cに保ってから反応を設定します。
注:この論文に記載されているCD反応の場合、成分の働く濃度は次のとおりです: リン酸ナトリウム緩衝液 (pH 7.0) 1 mM, ATP 2 mM, DNA 500 nM, プロテイン 1 μM, MgCl2 10 mM, EDTA 50 mM, ADAADiN 5 μM.
2. ATPase活動
- CD分光法の前に、DNA分子の存在下でタンパク質のATPase活性を確立し、CD分光法で使用されるタンパク質が活性であることを確認し、ATP加水分解を引き出す上で最適なDNA分子を同定する。
- 次の2つの反応からなるNADH結合酸化アッセイにより異なるDNA分子の存在下でのタンパク質のATPase活性を測定する。
- 0.1 μM ADAAD、2 mM ATP、10 nM DNA、1x REGバッファを96ウェルプレートに混合し、最終体積250 μLにします。
注:ピルビン酸キナーゼ酵素は、ADPとPiを使用してホスホエノールピルビン酸をピルビン酸に変換し、ATPを再生します。これにより、ATPは常に反応中の飽和濃度になります。第2の反応において、ピルビン酸キナーゼの作用により形成されたピルビン酸は、乳酸デヒドロゲナーゼによって乳酸に変換される。この反応では、NADH分子の1つがNAD+に酸化される。NADHの消費量は、340nmで分子の吸光度を測定することによって測定される。 - 37°Cで30分間インキュベーターでインキュベートします。
- マイクロプレートリーダーを使用して、340 nmでNAD+ の量を測定します。
- NAD+の量を測定するには、マイクロプレートリーダーと一緒に提供されるソフトウェアを使用してください。
- NADHアッセイをクリックして、340 nmで吸光度を測定します。
- 96ウェルプレートを器具のプレートホルダーに置きます。吸光度を記録するために 読み取りプレート ボタンをクリックします。
注: NAD+ の濃度は、eq (1) を使用して、NADH のモル絶滅係数を 6.3 mM-1 として計算します。
A = εcl (1)
ここで、A = 吸光度
ε = モル絶滅係数
c = モル濃度
l = 光パスの長さ (cm)
- 0.1 μM ADAAD、2 mM ATP、10 nM DNA、1x REGバッファを96ウェルプレートに混合し、最終体積250 μLにします。
3. CDキュベットの選択と準備
- 高透明性のクオーツキュベットでCDスペクトルを収集します。長方形または円筒状のキュベットを使用します。
注:CDクォーツキュベット(公称体積0.4 mL、パス長1mm)は、この論文に記載されているすべての反応に使用されました。 - キュベットの洗浄液を使用して、キュベットを洗浄します。水に1%キュベット洗浄液を加えて400μLの溶液を作り、キュベットに注ぎ、37°Cで1時間インキュベートします。
- キュベットを水で数回洗ってキュベットをきれいにします。キュベット内の水またはバッファーのスキャンを行い、清潔かどうかを確認します。
注: 水またはバッファーは、0 ~ 1 mdeg の範囲で読み取りを行う必要があります。
4. タンパク質とDNAオリゴヌクレオチドの調製
- 反応でタンパク質の体積を50μL以下に抑え、時々あいまいなピークの形成を引き起こす緩衝成分の量を最小限に抑えます。分解を避けるために、実験全体を通してタンパク質を氷の上に置いてください。
- 反応にPAGE精製DNAオリゴヌクレオチドを使用する。
注:ここで説明する反応では、DNAは、ネイティブと熱冷却形態(高速冷却(FC)および低速冷却(SC)の両方で使用されました。高速冷却はDNA中の分子内結合を促進し、より二次構造を生み出す。対照的に、冷却が遅くなるとDNA内の分子間結合が促進され、二次構造が少なくなります。 - 速い冷却のために、熱ブロックの3分の94°CでDNAを熱し、すぐに氷の上でそれを冷却する。ゆっくり冷却するには、94°Cで3分間加熱し、1分間に1°Cの速度で室温まで冷却します。
5. ベースラインスペクトルを記録するための制御実験の設定
- すべての反応で反応量を300 μLに保ちます。1.5 mL遠心分離管に合計5基基反応を設定します。ii) バッファー + MgCl2 + ATP + 水;iii) バッファー + MgCl2 + ATP + タンパク質 + 水;iv) iii + EDTA または ADAADiN;v) バッファー + タンパク質 + 水.
6. CD スペクトルを記録する実験の設定
- 1.5 mL遠心分離チューブに1つずつ5つの反応を設定します。ii) 緩衝液 + DNA + MgCl2 + ATP + 水;iii) バッファー + DNA + MgCl2 + ATP + タンパク質 + 水;iv) iii + EDTA または ADAADiN;v) バッファー + DNA + タンパク質 + 水.
7. スキャンの記録
- ガスの電源を入れ、CD分光器のスイッチを入れます。
- 10~15分後にランプのスイッチを入れて下さい。水浴のスイッチを入れ、ホルダーの温度を37°Cに設定します。
- CDスペクトラムソフトウェアを開きます。
- 温度を37°Cに設定します。
- 波長範囲を180~300nmに設定します。
- ポイントあたりの時間を 0.5 秒に設定します。
- スキャン番号を 5 に設定します。
- プロデータビューアをクリックし、新しいファイルを作成し、実験と日付の詳細を名前を変更します。
- 分解を防ぐために、すべての反応成分を氷の上に保管してください。遠心分離管でベースラインと反応を1つずつ作り、ピペットで混ぜます。反応ミックスを慎重にキュベットに移し、気泡がないことを確認します。
- タイムコース実験を行う場合は、37°Cで必要な時間に反応をインキュベートし、スキャンを行います。DNA、ATP、Mg+2、およびタンパク質を含む緩衝液にEDTAを加え、ATP加水分解を停止する。
- EDTAの濃度を増加し、ATPA活性を完全に阻害するためにそのインキュベーション時間を増加させる。
- ソフトウェア内の対応する反応からベースラインを差し引く(例えば、ベースライン1から反応1を減算する)。CD スペクトラムソフトウェアまたはデータプロットソフトウェアでデータを平滑化します。データプロットソフトウェアでデータをプロットします。
注:対応する反応からベースラインを差し引くと、DNAのみのネットCDスペクトルが与えられます。
8. データ分析と解釈
- eq(2)で与えられた式を使用して、ミリ度で得られた値を平均残基楕円度に変換します。
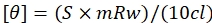 (2)
(2)
ここで、Sはミリ度のCD信号、cはmg/mLのDNA濃度、mRwは平均残基質量、lはパス長をcm単位で表す。 - データプロットソフトウェアを使用して、波長と平均残基楕円度に対してグラフをプロットし、ピークを分析します。
- グラフをプロットするには、Y軸の平均残基楕円度とX軸の波長を選択し、直線グラフをプロットします。
注:このグラフは、DNAの異なる形態の特性ピークを提供します。ピークに対応するDNAの形態は、既存の文献6を用いて同定することができる。
結果
ADAADは、MYCプロモーター上の構造のようなステムループを安定化させる
これまでの実験的証拠は、SMARCAL1がMYC29の陰性調節因子であることを示した。QGRSマッパーによるMYC遺伝子の159bp長いプロモーター領域の分析は、前方鎖がG-quadruplexを形成する可能性を有することを示した(表2)。Mfoldは、MYC DNAの両方の鎖がステムループ状の構造を形成しうる示した(表2)。G-quadruplex(GECE)を含む34bpの長いDNA配列を合成した。GECEオリゴヌクレオチドの前方および逆配列のMfold構造を図1A,Bに示す。
6X His-ADAADを用いたATPaseアッセイは、急速に冷却されたGECEがネイティブおよび低速冷却形態よりも優れたエフェクターであることを示した。したがって、高速冷却GECEは、ATPおよびADAADの存在下でCDスペクトルを記録するために使用された。CDスペクトルは、ADAADが269nmの肩を持ち、DNA内で210nmでより大きなピークを有する258 nmで2つの正のピークを誘導することを示した(図1C)。240 nm前後の陰性に向かうディップも観察された。このスペクトルは、ADAADの最適なエフェクタである合成ステムループDNAをタンパク質とATPと共にインキュベートした場合に得られたスペクトルと類似していた(図2A、B)。Triplex DNAは同様のスペクトルを与えることができる37、タンパク質はこの場合、このような構造を誘導することができるという仮説につながる。ATPは、Mg+2との配位錯体を形成し、このカチオンはATP加水分解に不可欠です。EDTAの添加は、Mg+2をキレートし、ATP加水分解38の阻害につながる。そこで、ADAADによるATP加水分解が立体構造変化に重要であったかどうかを理解するために、反応ミックスにEDTAを添加した。この反応にEDTAを添加すると、このコンフォメーションがアブロメートされます。CDスペクトルは現在、負の210 nmのピークと230と250 nmのピークを持つ広い正のバンドを持っています(図1C)。
ATPA活性の重要性は、ADAADのATPase死んだ変異体を用いても確認された。K241A変異は、モチーフIの保存されたGKTボックスに生じ、この変異体はDNA31の存在下でATPを加水分解する能力を欠いていることが以前に示されている。変異タンパク質をGSTタグで発現し、グルタチオン親和性クロマトグラフィーを用いて精製した。この変異体によって MYC DNAに誘導された立体構造変化は、野生型ADAADによって誘導されたものとは異なっていた。変異タンパク質の存在下で のMYC DNAのCDスペクトルは、陽性210nmピークおよび陰性260nmピークを有していた(図1D)。
ADAADは、DROSHAプロモーターにおけるA形の立体構造を誘導する
DROSHA、DGCR8、およびダイカーのプロモーター領域もQGRSマッパーおよびMfoldソフトウェアによって分析された。QGRSおよびMFoldはいずれも、プロモーター領域がG-quadruplexおよびステム様構造を形成する可能性を有することを示した(表2)。フォワードオリゴヌクレオチドおよび逆オリゴヌクレオチドのMfold構造を図3A,Bに示す。ATPase活性は、天然および熱冷却されたDNAが同様に振る舞っていることを示した。従って、これらのDNA配列の緩冷形態をCD研究に用いた。CDスペクトルは、ADAADがDROSHAプロモーターにおいて210nmで陰性ピークと260nmの正のピークを誘導することを示した(図3C)。このスペクトルはA-DNA6の特徴です。
ADAADはDGCR8プロモーターにおけるB-X転移およびG-四肢プレックス形成を誘導する
使用するオリゴヌクレオチドの前方および逆鎖のMfold構造を図4A,Bに示す。DGCR8ペア1では、210nmで陽性のピーク、260nmでの広い負のピークが観察された(図4C)。このスペクトルはB-X遷移6の特徴である。使用するオリゴヌクレオチドの前方および逆鎖のMfold構造を図4D,Eに示す。DGCR8ペア7のCDスペクトルは、210nmおよび270nmで強い陽性ピークを示し、250nmで負のピークを示した(図4F)。このスペクトルは、並列G-Quadruplex DNA構造6の特徴である。
ADAADは、ダイサープロモーターでA-X遷移を誘導 する
フォワードオリゴヌクレオチドおよび逆オリゴヌクレオチドのMfold構造を図5A,Bに示す。210 nmで陽性のピークを、230 nmで2つの負のピーク-1、260 nmのピークでもう1つを観察した(図5C)。これらのピークはA-X DNA遷移の特徴である6,9。全てのCDスペクトルピークと、転写過程において特定の役割を有するDNAの形態を表3にまとめた。
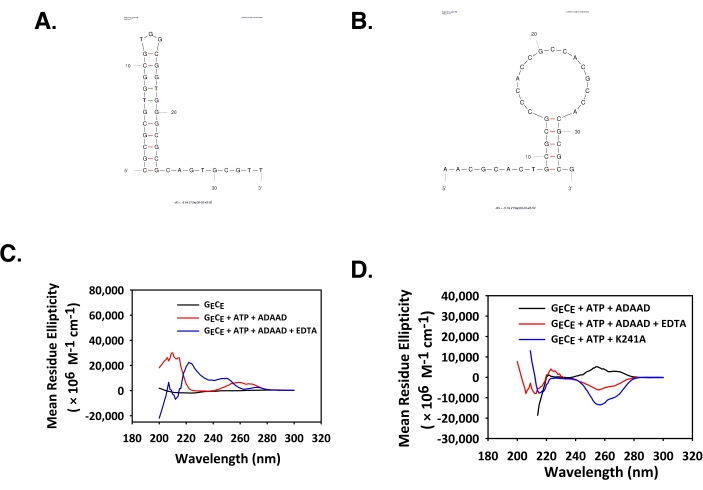
図1:ADAADはGECE DNAの立体構造を変化させる。(A)前方ストランドと(B)逆鎖について、Mfold構造を予測した。(C)GECE単独(黒)のCDスペクトル、GECEは、EDTA(青)を添加する前(赤色)および添加後にATPおよびADAADでインキュベートした。(D)GECEのCDスペクトルは、ATPとGSTタグ付きADAADを前(黒)およびEDTA(赤)を添加した後、ならびにATPおよびGSTタグ付きK241A変異体(青)でインキュベートされたGECEのCDスペクトルをインキュベートした。この数字は29から修正されています。略語: ADAAD = 活性DNA依存性 ATPase A ドメイン;CD = 循環性のジクロイズム。この図の大きなバージョンを表示するには、ここをクリックしてください。

図2:ADAADは、 ステムループDNAについて予測されるslDNA(A)Mfold構造の立体構造を変化させる。(B)slDNA単独のCDスペクトル(黒)、ATP及びADAAD(赤色)でインキュベートされたslDNA。この図は29から変更されています。略語: ADAAD = 活性DNA依存性 ATPase A ドメイン;slDNA = ステムループDNA;CD = 循環性のジクロイズム。 この図の大きなバージョンを表示するには、ここをクリックしてください。
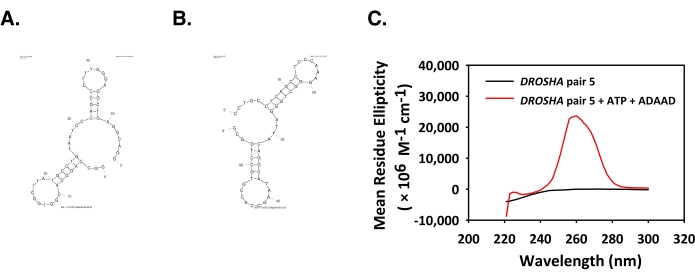
図3:ADAADは 、DROSHA 対5DNAの立体構造を変化させる。 (A)前方ストランドおよび(B)逆鎖について予測されたMfold構造。(C) DROSHA 対5DNA単独のCDスペクトル(黒)、 DROSHA ペア5DNAをATPとADAADと共にインキュベートした(赤色)。この図は28から修正されました。略語: ADAAD = 活性DNA依存性 ATPase A ドメイン;CD = 循環性のジクロイズム。 この図の大きなバージョンを表示するには、ここをクリックしてください。
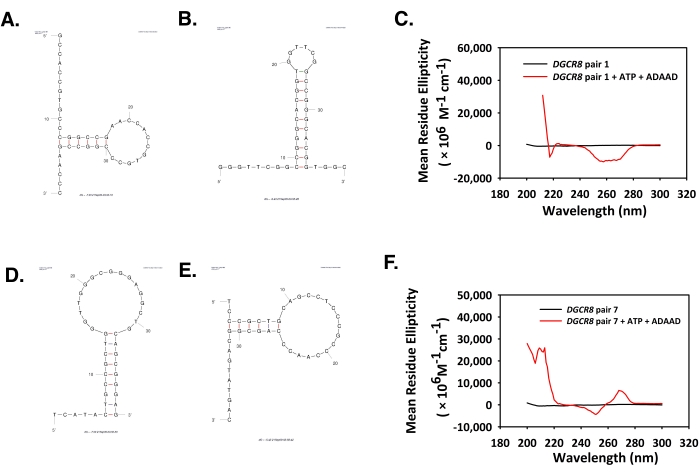
図4:ADAADは 、DGCR8 ペア1および7DNAの立体構造を変化させる。 (A)前方鎖及び(B)DGCR8対1オリゴヌクレオチドの逆鎖について予測されたMfold構造。(C) DGCR8 対1単独のCDスペクトル(黒)、 DGCR8 ペア1をATPおよびADAAD(赤色)とインキュベートした。(D)前方鎖および(E)DGCR8対7オリゴヌクレオチドの逆鎖について予測されるMfold構造。(F) DGCR8 対7単独のCDスペクトル(黒)、 DGCR8 ペア7をATPおよびADAAD(赤色)とインキュベートした。この図は28から修正されました。略語: ADAAD = 活性DNA依存性 ATPase A ドメイン;CD = 循環性のジクロイズム。 この図の大きなバージョンを表示するには、ここをクリックしてください。
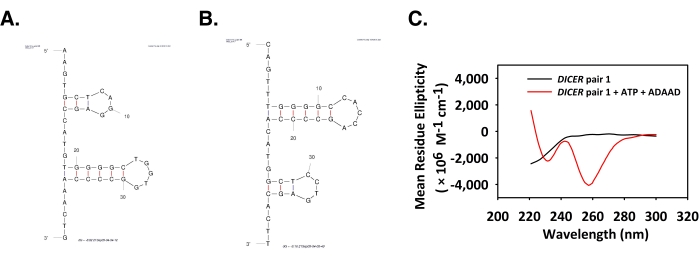
図5:ADAADは 、DICER 対1DNAの立体構造を変化させる。 (A)前方鎖及び(B)DICER対1オリゴヌクレオチドの逆鎖について予測されたMfold構造。(C)CDスペクトルは 、DICER 対1単独(黒)、 DICER 対1をATPとADAAD(赤色)とインキュベートした。この図は28から修正されました。略語: ADAAD = 活性DNA依存性 ATPase A ドメイン;CD = 循環性のジクロイズム。 この図の大きなバージョンを表示するには、ここをクリックしてください。
| 5x REG バッファ | |
| コンポーネント | 作業集中 |
| 乳酸デヒドロゲナーゼ(LDH) | 50単位/mL |
| 酢酸マグネシウム(Mg(OAc)2) | 30mM |
| ホスフェノールピルビン酸 (PEP) | 6.8 mg/mL |
| 酢酸カリウム(KOAc) | 300mM |
| ピルビン酸キナーゼ (PK) | 50単位/mL |
| トリスアセテート (トリス-OAc) | 125 mM |
| β-メルカプトエタノール (β-ME) | 25mM |
表1:バッファコンポーネント
| オリゴヌクレオチド | 順方向シーケンス | ΔG (m-フォールド) Kcal/mol | Gスコア(QGRSマッパー) | 逆シーケンス | ΔG kcal/モル (Mfold予測) | Gスコア(QGRSマッパー) | |||
| slDNA | GCGCAATTGCGCCGA CGATTTタグCGCAA TTGCGC | -16.36 | - | - | - | - | |||
| マイク・ゲセ | CGCGCGTGTGTGG CGGTGGGCGCA GTGCGTT | -8.64 | 19 | アックGCGCGCGCCC カッチクカッチャッカ CGCGCG | -5.74 | - | |||
| ドロシャペア5 | グッカッガカッグ GCTTATGCCTGTAAT CCCAGCCCTTGGGA グクトガグGCAGG | -10.18 | 19, 19 | CCTGCCTCAGCCTCC カアググGCTGGATT アカッカタクカック CGTGCCTGGCC | -10.95 | - | |||
| DGCR8 ペア 1 | グマツグク CCGAACCGTGC CCGGCCGAACCC | -7.9 | - | ググットCGCGGG カッグトッグトック CCGGGCACGGTGGC | -9.4 | 21 | |||
| DGCR8 ペア 7 | トカタクトGCCGCTG GGTGGGGGGGAG GCTGCAGCGGGAG | -7.99 | 33 | TCCCGCGCGCCT CCCGCCCACCCAG CGGCAGTATG | -5.43 | - | |||
| ダイサーペア1 | アクトGCッカグガグ CCATGTGGCTG GTGGCCCCAAACTG | -8.82 | 19 | カクトットッグクカック カグカタカッグ TCCTGAGCACTT | -9.16 | - | |||
表2:オリゴヌクレオチド配列。 すべてのシーケンスは5'-3'方向にあります。略語: slDNA = ステムループDNA。
| オリゴヌクレオチド | NMのCDスペクトルピーク(ATPおよびADAADでインキュベートした後) | DNAの形態 | 転写における役割 |
| slDNA | +215, +250, +272 | トリプレックス | 弾圧 |
| マイク・ゲセ | +210, +258, +269 | トリプレックス | 弾圧 |
| ドロシャペア5 | -210, +260 | A-DNA | 開始/アクティベーション |
| DGCR8 ペア 1 | +210,-260 | B-X トランジション | 正のスーパーコイル/活性化 |
| DGCR8 ペア 7 | +210, -250, +270 | G-クワドルプレックス | アクティベーション |
| ダイサーペア1 | +210, -230, -257 | A-X トランジション | 正のスーパーコイル/活性化 |
表3:CDスペクトルピークは、転写における役割を持つ異なる形態のDNAに対応する。 略語: ADAAD = 活性DNA依存性 ATPase A ドメイン;CD = 循環性のジクロイズム。
ディスカッション
本稿の目的は、ATP依存クロマチンリモデリングタンパク質の存在下でDNAに生じる立体構造変化を研究し、これらの構造変化を遺伝子発現に結びつける手法としてCD分光法を紹介することにある。CD分光法は、DNAの立体構造変化を研究するための迅速かつ容易にアクセス可能な方法を提供します。
この技術に考慮されるべき重要なポイントは、DNAとタンパク質の純度です。DNAとタンパク質の両方が>95%純粋であることを確認することをお勧めします。PAGE精製オリゴヌクレオチドはアッセイに使用する必要があり、かつタンパク質は、好ましくは、95%の純度>まで親和性精製されるべきである。もう 1 つの重要なパラメーターは、ベースライン読み取り値が 1 mdeg を超えないように、キュベットがクリーンであるべきということです。バッファーは、オートクレーブ水を使用して行われるべきであり、バッファーのベースライン読み取り値は 1 mdeg を超えてはならない。プロモーターの立体構造を研究するには、タンパク質が結合する領域を同定することが不可欠である。従って、このプロセスとして目的とするタンパク質を用いてChIP実験を行うことが、このタンパク質に結合するエフェクター遺伝子のプロモーター領域に存在するDNA配列を同定するのに役立つ。領域が特定されると、特定の構造を採用する配列の能力は、利用可能なバイオインフォマティクスツールを使用して分析することができます。これは、ChIPプライマーは通常200 bpの長さであり、複数の立体構造を有する可能性があるため重要である。したがって、バイオインフォマティクスツールを使用して構造を特定することは、オリゴヌクレオチドの長さを1つの構造に短くするのに役立ちます。
最後に、目的のタンパク質がATP依存クロマチンリモデリングタンパク質である場合、オリゴヌクレオチドがエフェクターとして作用する能力は、ATPaseアッセイを用いてチェックする必要がある。CD分光法とATPaseアッセイの両方で、リガンドの飽和濃度が反応に使用されるように注意する必要があります。可能であれば、タンパク質とリガンド相互作用のための解離定数(Kd)は、CD分光法を進める前に計算する必要があります。バインド パラメータの計算には、多数のメソッドが用意されています。ATPaseアッセイを用いて、ミカニス・メンテン定数(KM)は、DNAの濃度を増加させることによって計算することができる。KMは、多くの場合、結合定数に近似することができる。タンパク質が蛍光である場合、結合定数は蛍光分光法を用いて計算することができる。これらの技術のどちらも実現可能ではない場合、電気泳動移動性シフトアッセイ(EMSA)を使用することができます。
CD分光法の主な問題は、キュヴェットが洗浄されていない場合、または試薬が不純な場合に発生します。ベースラインが高すぎる場合は、キュベットをクリーニングすることをお勧めします。多数のキュベットクリーニングソリューションが利用可能です。16-24時間の希薄酸溶液にキュベットを入れることもキュベットをきれいにするのに役立ちます。純粋>95%の試薬を購入し、二重蒸留水とオートクレーブ水を使用することをお勧めします。ベースラインドリフトも潜在的な問題です。長期実験を行う場合は、ベースラインを定期的にチェックすることをお勧めします。タンパク質とDNAの相互作用を研究する際のDNAのピークは、DNAだけで得られるピーク/バンドと正確には一致しない場合があります。タンパク質ピークは、通常、190〜230nmの間の遠紫外線範囲で観察される。したがって、250 nm 未満のピークは、タンパク質ピークからの干渉を持ち、信頼性の高い情報を提供しない可能性があります。DNAは、配列に応じて様々な非Bコンフォメーションを採用することができる。DNA配列が長いほど、DNAオリゴヌクレオチド内に共存する複数の立体構造の可能性が高くなる。これにより、分析が困難になる可能性があります。したがって、バイオインフォマティクスツールによって予測される潜在的な構造に対応する短いオリゴヌクレオチドを使用することをお勧めします。
CD分光法のもう一つの大きな欠点は原子レベルの構造解析が認められていないことであり、得られたスペクトルは唯一の実行可能な構造を特定するのに不十分である。例えば、X線結晶学とタンパク質NMR分光法の両方が原子分解能データを提供するのに対し、CD分光法は構造情報の詳細度が低い。しかし、CD分光法は、膨大な量のタンパク質や大量のデータ処理を必要としない迅速なアプローチです。その結果、CDは、温度、pH、多量、および種々の補因子の存在などの幅広い溶媒変数を調べるのに使用され得る。また、動的系における構造変化(複雑な形成、折り畳み/展開、温度による変性、変性物質、アミノ酸配列/突然変異の変化など)を監視するためにも使用できます。それをストップフロー装置に接続することで、タンパク質/DNA-リガンド相互作用の運動学を研究するためにも使用できます。
適切に特徴付けられるDNA適合者には、A/B/Z DNA、トリプレックス、ヘアピン、G-quadruplexesが含まれる。これらのDNAの形態はすべて、開いたDNAの立体構造、すなわち、陰性超反動のシンクとして機能するアンワインドDNAと関連している。転写は、RNAポリメラーゼの移動の前提条件であるオープン複合体の形成として負の超反動に関連している。したがって、ほとんどの遺伝子の転写は、プロモーター領域における陰性スーパーコイルの増加を伴う。研究は、ヌクレオソームの展開がA-DNAの立体構造につながることを示しています, しかし, 不安定です39.1つの可能性は、関心のあるタンパク質、例えば、SMARCAL1は、そのような構造に結合し、それらを安定させ、したがって 、DROSHA プロモーターの場合に見られるように転写を促進する。グアニン四重体は、フーグスティーン水素結合によって結合されたグアニン四面体に基づいている。 インシリコ解析では 、G4形成配列が遺伝子プロモーターおよび転写開始部位に近位的に特に富化することを確認した。これらのG4配列は、転写を活性化し、再び活性化することができる。
MYCプロモーター40の場合、G-quadruplexの形成はリプレッサーとして働き、一方、ヒト血管内皮成長因子(VEGF)の場合、G-quadruplex構造は転写因子41のドッキング部位として機能し、この遺伝子の発現を活性化させる。SMARCAL1の場合、ADAADがDGCR8対7プロモーター配列と相互作用したときにG-quadruplex構造が観察された。このプライマー対に対するSMARCAL1とRNAPIIの占有率が増加するにつれて、G-quadruplexの形成は、この場合、この遺伝子の転写活性化と相関すると仮定される。DNAは、正にスーパーコイル化することもでき、RNAポリメラーゼの進行は、その前に正のスーパーコイルを発生させることが知られています。この転写生成(+)スーパーコイルは、道路ブロックタンパク質を破壊または排除し、ヌクレオソーム構造を不安定化させ、DNAをRNAポリメラーゼにアクセスしやすくすることができます。X-DNAは、正の超反動のシンクとして機能するDNAの立体構造です。X-DNAの顕著な特徴は、遺伝子のプロモーター上に配列特異的な方法で形成され得る。SMARCAL1の場合、ADAADは、それぞれ、DICERおよびDGCR8プロモーターにおいてATP依存的にA-XおよびB-X遷移を誘導した。ドキソルビシンによるDNA損傷の存在下でこれらのプロモーターにSMARCAL1およびRNAPII占有率が増加したin vivoデータと組み合わせることで、X-DNA形成が障壁/ブロックを除去することによって転写を容易にすると仮定することができる。トリプルDNAヘリックスは特徴的なスペクトルを持っていません。MYCプロモーターに存在するDNA配列のCDスペクトルと合成ステムループDNAは、258nmで269nmの肩を持ち、DNA内で210nmでより大きなピークを有する2つの陽性ピーク1を示した。このタイプのスペクトルは、triplexes37の場合に得ることができる。トリプレックスは、巻き戻しが困難であり、したがって、転写42をブロックすることが知られている。したがって、SMARCAL1によるc-MYCプロモーターにおけるこの構造の形成は、転写の抑制につながると仮定される。
ATPはまた、ATP依存性クロマチンリモデリングタンパク質に結合することに留意すべきである。これらのタンパク質のヘリカゼドメインのモチーフVIに存在する保存されたアルギニンは、タンパク質43のγリン酸との静電相互作用を介して相互作用する。ADAADの場合、タンパク質とATP相互作用のKd は(1.5±0.1)×10-6 M38である。ATPの結合は、DNAの親和性が高くなるようなタンパク質の立体構造変化を誘導する。DNAの結合はまた、ATP31に対する10倍の親和性の増加をもたらすタンパク質の立体構造変化を誘発する。例えば、ADAADの場合、バンド/ピークは-212 nmおよび-222 nmで観察される。ATP はまた 197 nm、+210 nm、-222 nm、-247 nm、および -270 nm のバンドを与える。これらは、リガンドの存在下でDNAの「ネット」の立体構造を得るために、DNA+ ADAAD + ATPのスペクトルから差し引かれなければならない。
本論文では、ATP依存クロマチンリモデリングタンパク質の存在下でDNAに生じる立体構造変化の研究に向けたCD分光法の利便性を示す。DNAの立体構造の変化をChIPデータと相関させることで、DNA適合者がどのように転写を活性化/再プレスする方法に関する情報を研究者に提供することができます。
開示事項
著者は宣言する利害の対立はありません。
謝辞
CD分光度計に対して、JNUの先端計測研究施設に感謝したいと考えています。V.J.とA.D.はCSIRのフェローシップによって支えられました。
資料
| Name | Company | Catalog Number | Comments |
| 2-Mercaptoethanol | Fisher scientific | O3446I-100 | |
| Adenosine 5′-triphosphate disodium salt hydrate | Sigmaaldrich | A2383 | |
| CD Quartz Cuvette | STARNA | 21-Q-1 | |
| Chirascan V100 CD spectrometer | Applied Photophysics | Not available | |
| EDTA Disodium Salt Dihydrate | SRL | 43272 | |
| Glutathione Sepharose 4B | GE Healthcare | 17-0756-01 | Glutathione affinity chromatography |
| Hellmanex III cleaning solution | Hellma | 9-307-011-4-507 | |
| L-Lactic Dehydrogenase | Sigmaaldrich | L2625 | |
| Magnesium Acetate Tetrahydrate | Fisher scientific | BP215-500 | |
| Magnesium Chloride Hexahydrate | Fisher scientific | M33-500 | |
| NADH disodium salt | Sigmaaldrich | 10107735001 | |
| Phosphoenolpyruvate Monocyclohexylammonium Salt | SRL | 40083 | |
| Potassium Acetate | Fisher scientific | P178-3 | |
| Pyruvate Kinase | Sigmaaldrich | P1506 | |
| Sodium Phosphate Dibasic Anhydrous | Fisher scientific | S374-500 | |
| Sodium Phosphate Monobasic Monohydrate | Fisher scientific | S369-500 | |
| Synergy HT microplate reader | BioTek | Not available | |
| Tris Base | Fisher scientific | BP152-500 |
参考文献
- Woody, R. W. Circular dichroism. Methods in Enzymology. 246, 34-71 (1995).
- Kelly, S., Price, N. The Use of Circular Dichroism in the Investigation of Protein Structure and Function. Current Protein & Peptide Science. 1 (4), 349-384 (2000).
- Rodger, A., Marshall, D. Beginners guide to circular dichroism. The Biochemist. 43 (2), 58-64 (2021).
- Greenfield, N. J. Using circular dichroism spectra to estimate protein secondary structure. Nature Protocols. 1 (6), 2876-2890 (2006).
- Kypr, J., Kejnovská, I., Bednářová, K., Vorlíčková, M. Circular Dichroism Spectroscopy of Nucleic Acids. Comprehensive Chiroptical Spectroscopy. , 575-586 (2012).
- Kypr, J., Kejnovska, I., Renciuk, D., Vorlickova, M. Circular dichroism and conformational polymorphism of DNA. Nucleic Acids Research. 37 (6), 1713-1725 (2009).
- Miyahara, T., Nakatsuji, H., Sugiyama, H. Helical Structure and Circular Dichroism Spectra of DNA: A Theoretical Study. The Journal of Physical Chemistry A. 117 (1), 42-55 (2013).
- Khomyakova, E. B. Parallel intramolecular DNA triple helix with G and T bases in the third strand stabilized by Zn2+ ions. Nucleic Acids Research. 28 (18), 3511-3516 (2000).
- Kypr, J., et al. The unusual X-form DNA in oligodeoxynucleotides: dependence of stability on the base sequence and length. Journal of Biomolecular Structure & Dynamics. 13 (6), 999-1006 (1996).
- Zohoorian-Abootorabi, T., Sanee, H., Iranfar, H., Saberi, M. R., Chamani, J. Separate and simultaneous binding effects through a non-cooperative behavior between cyclophosphamide hydrochloride and fluoxymesterone upon interaction with human serum albumin: multi-spectroscopic and molecular modeling approaches. Spectrochimica Acta. Part A, Molecular and Biomolecular Spectroscopy. 88, 177-191 (2012).
- Sharifi-Rad, A., Mehrzad, J., Darroudi, M., Saberi, M. R., Chamani, J. Oil-in-water nanoemulsions comprising Berberine in olive oil: biological activities, binding mechanisms to human serum albumin or holo-transferrin and QMMD simulations. Journal of Biomolecular Structure & Dynamics. 39 (3), 1029-1043 (2021).
- Mokaberi, P., Babayan-Mashhadi, F., Amiri Tehrani Zadeh, Z., Saberi, M. R., Chamani, J. Analysis of the interaction behavior between Nano-Curcumin and two human serum proteins: combining spectroscopy and molecular stimulation to understand protein-protein interaction. Journal of Biomolecular Structure & Dynamics. 39 (9), 3358-3377 (2021).
- Danesh, N., et al. Determining the binding site and binding affinity of estradiol to human serum albumin and holo-transferrin: fluorescence spectroscopic, isothermal titration calorimetry and molecular modeling approaches. Journal of Biomolecular Structure & Dynamics. 36 (7), 1747-1763 (2018).
- Sadeghzadeh, F., et al. Characterizing the binding of angiotensin converting enzyme I inhibitory peptide to human hemoglobin: influence of electromagnetic fields. Protein and Peptide Letters. 27 (10), 1007-1021 (2020).
- Chamani, J., et al. Cooperative alpha-helix formation of beta-lactoglobulin induced by sodium n-alkyl sulfates. Journal of Colloid and Interface Science. 293 (1), 52-60 (2006).
- Dareini, M., et al. A novel view of the separate and simultaneous binding effects of docetaxel and anastrozole with calf thymus DNA: Experimental and in silico approaches. Spectrochimica Acta. Part A, Molecular and Biomolecular Spectroscopy. 228, 117528(2020).
- Dehghani Sani, F., et al. Changes in binding affinity between ofloxacin and calf thymus DNA in the presence of histone H1: Spectroscopic and molecular modeling investigations. Journal of Luminescence. 203, 599-608 (2018).
- Hargreaves, D. C., Crabtree, G. R. ATP-dependent chromatin remodeling: genetics, genomics and mechanisms. Cell Research. 21 (3), 396-420 (2011).
- Morettini, S., Podhraski, V., Lusser, A. ATP-dependent chromatin remodeling enzymes and their various roles in cell cycle control. Frontiers in Bioscience: A Journal and Virtual Library. 13, 5522-5532 (2008).
- Flaus, A., Martin, D. M. A., Barton, G. J., Owen-Hughes, T. Identification of multiple distinct Snf2 subfamilies with conserved structural motifs. Nucleic Acids Research. 34 (10), 2887-2905 (2006).
- Flaus, A., Owen-Hughes, T. Mechanisms for ATP-dependent chromatin remodelling: the means to the end. The FEBS Journal. 278 (19), 3579-3595 (2011).
- Mizuguchi, G., et al. ATP-driven exchange of histone H2AZ variant catalyzed by SWR1 chromatin remodeling complex. Science. 303 (5656), New York, N.Y. 343-348 (2004).
- Elizondo, L. I., et al. Schimke immuno-osseous dysplasia: SMARCAL1 loss-of-function and phenotypic correlation. Journal of Medical Genetics. 46 (1), 49-59 (2009).
- Baradaran-Heravi, A., et al. SMARCAL1 deficiency predisposes to non-Hodgkin lymphoma and hypersensitivity to genotoxic agents in vivo. American Journal of Medical Genetics. Part A. 158 (9), 2204-2213 (2012).
- Bansal, R., et al. SMARCAL1, the annealing helicase and the transcriptional co-regulator. IUBMB life. 72 (10), 2080-2096 (2020).
- Yusufzai, T., Kadonaga, J. T. HARP is an ATP-driven annealing helicase. Science. 322 (5902), New York, N.Y. 748-750 (2008).
- Yusufzai, T., Kong, X., Yokomori, K., Kadonaga, J. T. The annealing helicase HARP is recruited to DNA repair sites via an interaction with RPA. Genes & Development. 23 (20), 2400-2404 (2009).
- Patne, K., et al. BRG1 and SMARCAL1 transcriptionally co-regulate DROSHA, DGCR8 and DICER in response to doxorubicin-induced DNA damage. Biochimica et Biophysica Acta. 1860 (9), 936-951 (2017).
- Sharma, T., Bansal, R., Haokip, D. T., Goel, I., Muthuswami, R. SMARCAL1 negatively regulates c-Myc transcription by altering the conformation of the promoter region. Scientific Reports. 5, 17910(2015).
- Muthuswami, R., Truman, P. A., Mesner, L. D., Hockensmith, J. W. A eukaryotic SWI2/SNF2 domain, an exquisite detector of double-stranded to single-stranded DNA transition elements. The Journal of Biological Chemistry. 275 (11), 7648-7655 (2000).
- Nongkhlaw, M., Dutta, P., Hockensmith, J. W., Komath, S. S., Muthuswami, R. Elucidating the mechanism of DNA-dependent ATP hydrolysis mediated by DNA-dependent ATPase A, a member of the SWI2/SNF2 protein family. Nucleic Acids Research. 37 (10), 3332-3341 (2009).
- Kikin, O., D'Antonio, L., Bagga, P. S. QGRS Mapper: a web-based server for predicting G-quadruplexes in nucleotide sequences. Nucleic Acids Research. 34, 676-682 (2006).
- Zuker, M. Mfold web server for nucleic acid folding and hybridization prediction. Nucleic Acids Research. 31 (13), 3406-3415 (2003).
- Gupta, M., et al. Ligand-induced conformation changes drive ATP hydrolysis and function in SMARCAL1. The FEBS Journal. 282 (19), 3841-3859 (2015).
- Dutta, P., et al. Global epigenetic changes induced by SWI2/SNF2 inhibitors characterize neomycin-resistant mammalian cells. PloS One. 7 (11), 49822(2012).
- Muthuswami, R., et al. Phosphoaminoglycosides inhibit SWI2/SNF2 family DNA-dependent molecular motor domains. Biochemistry. 39 (15), 4358-4365 (2000).
- Gondeau, C. Circular dichroism and UV melting studies on formation of an intramolecular triplex containing parallel T*A:T and G*G:C triplets: netropsin complexation with the triplex. Nucleic Acids Research. 26 (21), 4996-5003 (1998).
- Nongkhlaw, M., Gupta, M., Komath, S. S., Muthuswami, R. Motifs Q and I are required for ATP hydrolysis but not for ATP binding in SWI2/SNF2 proteins. Biochemistry. 51 (18), 3711-3722 (2012).
- Luchnik, A. N. DNA conformational transitions induced by supercoiling control transcription in chromatin. Gene Regulation and Systems Biology. 8, 89-96 (2014).
- Siddiqui-Jain, A., Grand, C. L., Bearss, D. J., Hurley, L. H. Direct evidence for a G-quadruplex in a promoter region and its targeting with a small molecule to repress c-MYC transcription. Proceedings of the National Academy of Sciences of the United States of America. 99 (18), 11593-11598 (2002).
- Uribe, D. J., Guo, K., Shin, Y. -J., Sun, D. Heterogeneous nuclear ribonucleoprotein K and nucleolin as transcriptional activators of the vascular endothelial growth factor promoter through interaction with secondary DNA structures. Biochemistry. 50 (18), 3796-3806 (2011).
- Young, S. L., Krawczyk, S. H., Matteucci, M. D., Toole, J. J. Triple helix formation inhibits transcription elongation in vitro. Proceedings of the National Academy of Sciences of the United States of America. 88 (22), 10023-10026 (1991).
- Dürr, H., Flaus, A., Owen-Hughes, T., Hopfner, K. -P. Snf2 family ATPases and DExx box helicases: differences and unifying concepts from high-resolution crystal structures. Nucleic Acids Research. 34 (15), 4160-4167 (2006).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved