Method Article
Caenorhabditis elegansにおける凝集定量の自動化
要約
以下のプロトコルは、プロテオスタシスの変化の評価として、ワーム培養、蛍光イメージング、およびポリグルタミン凝集体を定量するための自動画像処理のためのハイスループットワークフローの開発と最適化について説明しています。
要約
神経変性タンパク質立体構造疾患(PCD)の有病率の上昇は、長年にわたってこの主題への大きな関心を育んできました。この関心の高まりにより、PCDを有するヒトで観察される疾患表現型を再現することができる動物モデルの多様化および改善が求められている。マウスモデルは非常に貴重であることが証明されていますが、高価であり、面倒で低スループットの方法に関連しています。PCDを研究するための Caenorhabditis elegans 線虫モデルの使用は、メンテナンスの比較的容易さ、低コスト、および迅速な生成時間によって正当化されており、ハイスループットアプリケーションを可能にします。さらに、 C. elegans とヒトゲノムの間の高い保存性により、このモデルは非常に貴重な発見ツールとなっています。蛍光的にタグ付けされた組織特異的ポリグルタミン(polyQ)管を発現する線虫は、蛍光病巣を特徴とする年齢およびpolyQ長さ依存性凝集を示す。このようなレポーターは、組織全体のプロテオスタシスの変化を監視するためのプロキシとしてしばしば使用される。手作業による集計定量は時間がかかり、実験のスループットが制限されます。さらに、手動の病巣定量化は、凝集同定が非常に主観的であり得るので、バイアスを導入する可能性がある。本明細書では、ワーム培養、画像取得、およびデータ処理からなるプロトコルを標準化し、腸特異的polyQを発現する C.elegans を用いたハイスループット凝集定量をサポートする。画像解析ソフトウェアであるCellProfilerを使用して C. elegansベースの画像処理パイプラインを実装することにより、この方法は個々のワームを分離して識別し、それぞれの凝集体を列挙するように最適化されています。自動化の概念は完全に独特ではありませんが、再現性、手動カウントからのバイアスの排除、スループットの向上のために、このような手順を標準化する必要性は高いです。これらの方法は、 C. elegans モデルを用いて、大規模な細菌、ゲノム、または薬物ライブラリーのスクリーニングプロセスを大幅に簡素化できることが期待されています。
概要
アルツハイマー病、パーキンソン病、ハンチントン病などの年齢依存性神経変性タンパク質立体構造疾患(PCD)、または筋萎縮性側索硬化症は、凝集、細胞死、および組織変性につながるタンパク質のミスフォールディングによって特徴付けられる1。タンパク質のミスフォールディングが原因として認識されていますが、これらの疾患の病因は明らかではありません。このように、効果的な治療法の開発は、疾患の発症および進行に寄与する因子および状態に関する知識の欠如によって妨げられてきた。最近の研究は、マイクロバイオームの変化がPCDsの発症、進行、および重症度に影響を与えることを示唆している2,3,4。しかし、ヒト、あるいはネズミのマイクロバイオームの複雑さは、微生物が宿主に及ぼす正確な影響を明らかにする研究を行うことを困難にしている。したがって、Caenorhabditis elegansなどのより単純な生物は、発見ツール5、6、7、8としてよく使用される。最近の研究では、宿主プロテオスタシスおよび疾患病因に対する細菌の影響を調査するためにC.エレガンスを用いている9,10。細菌のコロニー形成、ホルミシス、およびゲノム変化は、ポリグルタミン(polyQ)管9、11、12の凝集に影響を及ぼす例示的な条件の1つである。さらに、これらのミスフォールディングされたタンパク質クラスターは、宿主内でpolyQの長さおよび年齢依存的な蓄積を示し、運動性の障害と関連している9,13。蛍光標識されたパンクタを定量する比較的単純なアプローチは、タンパク質の折り畳みおよび凝集に影響を及ぼす条件、因子、または薬物に関する重要なデータを生成することができる。
蛍光パンクタの定量は信頼性が高く比較的簡単な手順であることが証明されていますが、タンパク質凝集に影響を与える化合物、細菌、または条件の大規模なスクリーニングを容易にするプロトコルを開発することは課題が残っています。自動化されたC. elegans画像処理とプンクタ定量化の概念は、多くの実用的なサポートツールが開発されているため、完全に新しいものではありません14,15。しかし、培養、画像取得、および処理パイプラインの統合は、結果のばらつきを排除し、より高いスループットのスクリーニングを可能にするために不可欠です。
したがって、この原稿の目的は、プロテオスタシスの変化を検出するためのプロキシとして 、C. elegans におけるpolyQ凝集を定量化するために使用される手順を標準化することです。このタスクは、ワームおよび凝集体の識別を自動化することが可能なオープンソースの画像解析ソフトウェア16 であるCellProfilerを採用することによって達成され、ワームの培養、画像の取得、およびデータの処理のためのより大きなプロトコルに統合される。
プロトコル
すべての手順は、フロリダ大学の機関バイオセーフティ委員会によってレビューおよび承認された安全ガイドラインに従っていました。生物学的安全性レベル2の細菌への曝露のリスクを軽減するために、適切なバイオセーフティ対策が講じられた。
注:すべての実験において、 C. elegans は、 エシェリヒ ア・コリOP50を播種した線虫増殖培地(NGM)プレート上で増殖させ、維持しなければならない。
1. 10cmNGMプレートの作製
- 2Lフラスコに3gのNaCl、2.5gのトリプチカーゼ - ペプトン、および17gの寒天を混ぜ合わせ、1Lに二重蒸留水(ddH2O)で満たします。オートクレーブ処理の前に磁気攪拌子を加える。
- 混合物を121°Cおよび21psiの圧力で45分間オートクレーブする。混合物を水浴中で50°Cまで冷まします。
- 無菌技術を使用して、以下の滅菌溶液を加える:1mLの1M CaCl2·2H2O, 1 mL の 1 M MgSO4·7H2O、100%エタノールに溶解した5mg/mLコレステロール1mL(室温まで加温)、および25mLの1MKH2PO4(pH=6.0)。 磁気攪拌盤を用いて混合する。混合は、700RPMで1分間行えばよい。
- 混合物が10cmプレート全体を満たすまで注ぐ。あるいは、目盛り付き血清学的ピペットを使用して、プレートあたり約20mLの混合物を加える。
- 細菌を播種する前にプレートを室温で24時間乾燥させるか、乾燥後にプレーンプレートを4°Cで保存します。
メモ: すべてのメディアコンポーネントは、無菌技術を使用して処理されます。ステップ1.3-1.4は、層流フードで実行する必要があります。
2. 24ウェルプレートにおけるFUDRによるNGM寒天の調製
- 手順 1.1-1.3 に従います。
- NGMに5-フルオロ-2'-デオキシウリジン(FUDR)を補充し、混合して最終濃度100μg/mLを達成します。
注:FUDRはDNA複製を阻害し、その結果、生殖細胞系列および胚発生を標的とすることによって C.エレガンスの 再生をブロックし、最終的に寿命に影響を及ぼす。したがって、FUDR含有プレートに移る前に、ワームが若年成人に完全に発達できるようにすることが重要です。
警告: FUDR は有毒であるため、製造元の安全データシートに従って取り扱う必要があります。 - ピペットガンを用いて、NGM-FUDRを1mLずつ各ウェルに分注する。
注:このプロセスは、自動プレート注ぎシステムを使用することで促進できます。 - プレートを室温で24時間乾燥させてから、細菌を播種するか、プレーンプレートを4°Cで保管します。
3.プレートの播種:OP50および追加の試験細菌
- 一晩 の大腸菌 OP50培養物を調製するために、凍結ストックからの細菌アリコート200μLを、250mLの新鮮で滅菌されたルリアブロス(LB)を含む500mL三角フラスコに加える。
メモ: メディアの容量は、シードする必要があるプレートの数によって異なります。他の細菌培養物を調製するには、滅菌マイクロピペットチップを用いて、凍結ストックからの細菌を5mLの増殖培地を含む16mL培養チューブに接種する。 - 37°Cのインキュベーター内で一晩インキュベートし、220RPM(毎分回転)で振とうする。
注:少なくとも2倍の作動量の媒体を含む滅菌フラスコを使用し、オートクレーブ処理されたアルミニウム箔でシールしてください。無菌技術を用いて接種工程および細菌分注を行う。 - 一晩 大腸菌 OP50培養物を各10cmNGMプレートの中央に1〜2mL分注する。この培養物をNGMプレートの周囲に広げる必要はない。
- 使用/保管前にプレートを室温で乾燥させてください。
注:蓋付きの播種プレートは、乾燥を容易にするために気流のあるフードに入れることができます。
4.プレートの培養と播種:24ウェルプレート
- 所望の細菌株の一晩培養物を調製し、ステップ3.1〜3.2に見られる培養指示に従う。
- 各細菌培養物200 μLをNGM寒天を含む24ウェルプレートの各ウェルに移す。200μLのバクテリア容量が寒天領域全体をカバーし、ワームがバクテリアの芝生を避けないように食物の量を最大化します。
- 乾燥を容易にするために、生物学的安全キャビネット(BSC)でプレートをひび割れたままにしておきます。プレートを定期的にチェックして過度の脱水を防ぎ、プレートの向きを変更して気流と乾燥を促進します。プレートは5時間以内に乾燥する必要があります。
注:生物学的安全レベル2の細菌を用いた作業は、認定BSCで実施し、機関バイオセーフティ委員会によって承認されなければなりません。
5. 年齢同期
メモ:すべての手順は、適切な無菌技術(すなわち、炎の近くまたはBSC内で作業)を使用して実行する必要があります。
- フィルター滅菌M9溶液(5.8gのNa2HPO4·7H2O、3.0gのKH2PO4、5gのNaCl、0.25gのMgSO4・7H2O、1LのddH2O)。
- 滅菌ガラスまたはプラスチック血清学的ピペットを使用してプレート上にM9溶液を数回ピペットし、細菌芝生からワームを持ち上げます。
- ワーム懸濁液を集め、溶液を15mLポリスチレン円錐管に移す。
- 遠心分離機を270 x g、室温(RT、〜23°C)、2分間保持する。
- 真空トラップフラスコを使用して吸引し、上清を捨てて、ワームペレットを邪魔しないままにします。
- ペレットを5〜10mLのM9に再懸濁してワームを洗浄し、ステップ5.2〜5.3を2回繰り返す。
- 5 mLの20%漂白溶液(8.25 mLのddH2O、3.75 mLの1M NaOH、3.0 mLの非殺菌性漂白剤)をチューブに加え、連続的に反転させてワームを溶解する。ワームは、ほぼ完全に溶解したら遠心分離する準備ができています。
注:漂白時間と漂白溶液の量は、サンプルのサイズに依存します。オーバーブリーチングとアンダーブリーチングは一般的なエラーです。そのため、このプロセスでは一般に、サンプルが遠心分離の準備が整う時期を判断するための最適化が必要です。 - 423 x g で2分間遠心分離し、上清を捨てる。
- 10mLの滅菌M9を加えて、卵ペレットを再懸濁する。
- チューブを423 x g で2分間遠心分離して卵をペレット化します。アスピレーターフラスコで上清を除去します。
- 手順 5.6 ~ 5.6.1 を繰り返します。
- 卵ペレットを5mLの滅菌M9に再懸濁し、所望の温度で一晩ヌーテーターの上に置く。
注:年齢同期L1幼虫は、翌日プレートに移す準備ができています。
6. ワームの準備ポストエージング同期
- 年齢同期ワームを270 x g でRT(~23°C)で3分間遠心分離します。
- フローフードやブンゼンバーナーの隣などの清潔な環境で上清を吸引します。約200μLの上清を残し、ワームを再懸濁する。
- マイクロピペットを用いて、濃縮ワーム懸濁液をOP50を予め播種した10cmNGMプレートに移す。
注:各プレートは、食物を使い果たすことなく1,500匹のワームをサポートすることができます。しかしながら、この濃度は、細菌芝生の密度および増殖温度に応じて調整を必要とする場合がある。ワームが飢えるのを防ぐために、複数のプレートを使用することをお勧めします。これらのプレートにFUDRが含まれてはならないことに注意することが重要です。 - プレートを乾燥させます。その後、反転させ、25°Cで48時間保存する。
注:スクリーンの状態に応じて、ステップ6.1のワームを試験プレート(細菌、薬物、または試験化合物を含む)に直接配置することができます。所望の試験条件が発育に影響する場合、ワームを試験条件に曝露する前に、若年成人(〜48時間)までOP50を含むNGM上で培養すべきである。 - 48時間のインキュベーションに続いて、滅菌M9溶液でプレートからワームを洗い流し、円錐形のチューブに入れます。
注:成虫はチューブの底に沈みます。正確な時間は、10cmプレートから回収されたワームの数によって異なります。これらの条件下では、ワームは10分以内に落ち着きます。 - 目視検査を実行して、残りの卵または孵化した幼虫が除去されるように沈降時間の長さを決定する。
- さらに10mLのM9を加えて、ワーム体からの残留細菌をすすいでください。
- ワームを23°Cで270 x g で2〜3分間遠心分離します。
- 洗浄工程をさらに3回行う。最良の結果を得るには、最終洗浄後に約1〜1.5mLのM9溶液をチューブに入れたままにしておきます。
- 10 μLのワーム懸濁液をスライドガラスに移し、ワームの数を数えます。
- ワーム密度を M9 の 10 μL あたり約 150 ワームに調整します。懸濁液中のワームの濃度は、遠心分離後にM9溶液を除去するか、または添加することによって調整することができる。
- いくつかの異なる滴からのカウントを平均することによって、所望の濃度が確立されたことを確認する。少なくとも3滴からカウントを平均することをお勧めします。
- 無菌技術を用いて、約150匹のワームを含む10μLのワーム懸濁液を試験プレートの各ウェルに移す。
- 顕微鏡下で井戸を検査し、それぞれに十分な数のワームがあることを確認します。インキュベーションの前に追加のワームを追加することができます。
- プレートを約10分間乾燥させます。その後、反転させ、25°Cのインキュベーターに72時間移します。
注:最終的なインキュベーション期間は、実験のニーズに合わせて調整できます。72時間のインキュベーション時間は、25°Cの24ウェルプレートで200μLの細菌を摂食させた150匹の動物の増殖をサポートするのに十分である。
7. イメージング用のワームの準備
- より効果的な沈降を促進し、サンプルの損失を最小限に抑えるには、洗浄前にワームを固定して水泳を防止します。
注:少数のサンプルで作業する場合、これはレバミゾール(100μM)への曝露によって達成することができます。しかし、多数のサンプルを扱う場合、ワームは凍結によって固定化することができる。さらに、凍結の延長(18〜24時間)は、調製中のpolyQ凝集体のさらなる発達を妨げる。- マルチウェルプレートを-20°Cで15〜20分間、またはワームが動かなくなるまで置きます。
- 冷凍庫からサンプルを取り出し、5分間放置します。
- マイクロピペットを用いて、M9を1mL加え、4°Cに冷却し、目的のウェルに、プランジャーを4〜6回押し下げて繰り返し、各ウェルのワームを洗浄した。
注:時々、ワームはマイクロピペットチップに固執することがあります。そのため、ワームの混合を防ぐために、各ウェル間で異なるチップを使用する必要があります。 - ワーム懸濁液を微量遠心管に移し、ワームが底に沈むようにします。
- 上清を吸引して捨てる。サンプルを合計3回洗浄する。
- 最終洗浄中にワームが底に落ち着いたら、500 μLの上清を吸引し、500 μLを残してワームを再懸濁させます。
- 残りのウォーム懸濁液を新しい平底24ウェルプレートに移し、-20°Cの冷凍庫に48時間置きます。
注:凍結ワームはバックグラウンド蛍光を低減し、凝集体のより良い視覚化を可能にします。
8. イメージング
- プレートを冷凍庫から取り出して解凍し、余分な結露を拭き取り、フタを取り外してからイメージングを行います。
メモ:画像キャプチャの詳細は、使用する機器やソフトウェアによって異なります。このセクションのプロトコルはガイドとしてのみ役立つべきであり、変更が期待されます。また、tiff ファイル形式で画像をキャプチャすることも必要です。 - 画像キャプチャ中に、次の顕微鏡設定を使用します:露光時間、500ミリ秒。0.63倍のカメラアダプタ(25.2倍)で倍率40倍、GFP強度を100%に設定。
メモ: さまざまな顕微鏡構成およびシステムによって、結果セクションに記載されているものとは異なる画像が取得される可能性があります。必要な画像のより普遍的な記述を達成するために、画像に関するより客観的な詳細が提供される。凝集体は、CellProfiler によって適切に識別されるためには、直径 1.0 ~ 10.0 ピクセルで蛍光強度が 0.10 ~ 1.0 (任意の単位、スケール 0 ~ 1) である必要があります。これらの凝集画像の蛍光バックグラウンドは、一般に0.10閾値未満である。明視野画像の場合、ワームの強度は 0.7 ~ 1.0 の範囲で、背景の強度は 0.1 ~ 0.2 です。明視野画像のワームの平均長さは、頭から尾までの長さが250ピクセルから350ピクセルの範囲です。 - 暗い背景に比べてワームが明るく照らされるまで、透過光コントロールを調整します。露出過多を避けます;ワームのサイズが大きくなります。
- 過度の凝集を防ぐために、井戸内のワームの位置を変更する必要があるかもしれません。凝集したワームをピペットチップを使用して分散させます。
- チャンネルをGFPに設定して、両方の画像の焦点面を確立します。
メモ: 焦点面は GFP チャネルで決定することが不可欠です。明視野画像をキャプチャ中にフォーカスを変更すると、画像解析中に凝集体やワームの位置ずれが生じます。 - 明視野画像をキャプチャし、プレートを乱すことなく対応する蛍光画像をすぐに撮影します。
- パイプラインを介してテストイメージを実行し、手順8.2の後の「NOTE」に従って、イメージ内のオブジェクトの強度とサイズの値を決定します。
注: CellProfiler は、解析前にオブジェクトの強度と長さに関するすべての必要な情報を提供できます。 - 画像を評価するには、まず CellProfiler17 をダウンロードします。ソフトウェアを開き、目的の画像を [画像] ボックスにドラッグ アンド ドロップします。
- ファイルリスト内の画像をクリックして開きます。
- 画面の左上隅にはいくつかのアイコンがあります。オブジェクトのサイズを測定したり、特定の領域を拡大したりするために使用します。拡大鏡ガラスを選択して、関心領域を強調表示します。
- 矢印アイコンを選択して、ワームとアグリゲートの両方の長さを測定します。
- 目的のオブジェクトにカーソルを合わせると、画面の下部に表示される強度値が表示されます。
注: すべての画像はグレースケールで撮影されるため、赤、青、緑のピクセル値はすべて同じになります。
9. 画像解析
- CellProfiler 画像解析パイプラインを利用するには、画像ペアに適切な名前を付けます。次の形式を使用します: P1_A01_S1_C1、P1 は特定のプレートとそのそれぞれの数値指定を参照します。"A" は行を指し、"01" は列を参照します。「S」は、単一のウェルに対する特定の画像ペアを指す。「C」はチャンネルを指し、「1」は明視野画像、「2」は蛍光画像を示します。
- CellProfiler(バージョン4.1.3以上)を公式ウェブサイトからダウンロード17.
- パイプラインをダウンロードします (補足ファイル 1)。パイプライン (パイプライン) を CellProfiler にアップロードするには、[ファイル] > [ファイルからパイプラインをインポート] >選択します。
メモ: モジュール 2 および 3 は、不要とみなされたため無効になっています。画像解析がワームの満足のいく分離と識別をもたらさない場合、それらの機能は回復することができます。ただし、パイプラインの変更が必要です。 - これらのモジュールを組み込むには、各モジュールの左側にあるボックスを選択します。"UntangleWorms" モジュールの入力バイナリ イメージの名前を、"convertobjectstoimage" モジュールの出力イメージに変更します。
- モジュール 2 に関連する初期使用中にエラー メッセージが表示されることがあります。この問題が発生した場合は、分析を続行します。
- ワームの識別に使用するトレーニングセットを「UntangleWorms」モジュールにアップロードします。このトレーニング セットは、 補足ファイル 2 にあります。
- UntangleWorms モジュールを選択して、その設定を開きます。
- トレーニングセットファイル名を特定し、ファイルのアップロードアイコンを選択します。
- 補足ファイル 2 (トレーニング セット) をアップロードします。
- 左上隅にある [画像] モジュールを選択して、画像をアップロードします。手順 9.1 の説明に従って、適切な名前の画像をドラッグ アンド ドロップします。
- 画像を分析する前に、結果の保存に使用する出力フォルダーを選択します。
- プログラムの左下隅近くにある [出力設定] ボタンをクリックします。
- 「デフォルト出力」の右側にあるフォルダアイコンを選択して、目的の出力場所を選択します。
- 「イメージの分析」アイコンを選択して、 イメージ分析 を開始します。
- 解析の完了に時間がかかりすぎて、1 つの画像の処理が行き詰まっている場合は、画像取得の問題が原因である可能性があります。このような場合は、実行を中止し、出力フォルダーを並べ替えて、見つからないイメージ名を書き留めて、未処理のイメージの識別に進みます。
注: パイプラインで処理できない大きなアーティファクトや強烈な照明のアーティファクトを除去するために、イメージに追加の処理が必要になる場合があります。場合によっては、そのような画像を解析から除外する必要があります。 - 完全な分析の後、ソフトウェアは結果を個々のワーム(列N)とそれぞれの集計数(列K)を含むExcelスプレッドシートに整理します。
注: 実行に成功すると、各ウェルの識別されたワームあたりの集計数を含む Excel スプレッドシートが生成されます。これらのデータは、実験者によって決定された方法で操作することができる。 - 補足ファイル 3 (Windows) または補足ファイル 4 (Mac) からメタデータ オーガナイザー (グラフィカル ユーザー インターフェイス) をダウンロードして、CellProfiler からの出力 CSV ファイルからのデータを便利に整理します。
注:このソフトウェアには公式ライセンスがなく、ダウンロード後に自動的に開かれることはありません。 - Windows OS 64 ビット版を使用している場合は、手順 9.14 ~ 9.17 に従います。ソフトウェアは、Windows 32ビット版では動作しません。Mac OS を使用している場合は、手順 9.18 ~ 9.20 を実行します。手順 9.21 ~ 9.22 は、両方のオペレーティング システムで同じです。
- Windows OSの場合は、ダウンロードしたファイルを見つけて目的の場所に解凍します。
- gui_windowsOS_64xという名前の抽出されたフォルダを見つけて開き、「gui」アプリケーションアイコンをクリックしてアプリケーションを起動します。
- 実行の許可を要求するプロンプトが開くことがあります。 [詳細情報 ] を選択し、[ とにかく信頼] をクリックします。
- メタデータ オーガナイザーは、CellProfiler 出力 CSV ファイルをドラッグ アンド ドロップする準備ができました。ステップ 9.21 に進みます。
- Mac OSの場合は、ダウンロードしたファイルを見つけてgui_macOS_64x.zip開きます。この手順では、すべてのファイルが自動的に抽出されます。
- 「ダウンロード」にある抽出されたフォルダを開きます。
- 「gui」アプリケーションを右クリックし、「 開く」を選択します。公式ライセンスがないために、開く許可を求めるプロンプトが表示されます。「 開く 」を選択し、ステップ 9.21 に進みます。
- [ ここにファイルをアップロード ]をクリックするか、目的のCellProfilerCSVファイルをドラッグアンドドロップします。
- [ 整理] ボタンをクリックすると、[ ファイルのダウンロード ] ボタンが付いた新しい画面にユーザーが表示されます。ボタンをクリックして、出力ファイルを保存する場所を選択します。出力ファイルは、ファイル名に「_organized」拡張子が追加された元のファイル名として表示されます。
結果
本明細書に記載するのは、培養およびイメージングプラットフォームとして24ウェルプレートフォーマットを使用して、様々な細菌の存在下でのpolyQ凝集の評価を可能にする培養、画像取得、および処理プロトコルを含む C. elegans ワークフローである(図1)。このプロトコルは、宿主プロテオスタシスに対する細菌、特定の状態、小分子、薬物、またはゲノム操作の効果を研究するために調整することができる。記載された方法は、黄色蛍光タンパク質(vha6p::p olyQ44::YFP)に融合した腸内polyQを恒常的に発現するワームを使用して最適化されている。しかし、筋肉またはニューロンにおけるプロテオスタシスについて報告する他のモデルも、さらなる最適化と共に使用することができる。例えば、予備実験は、筋肉polyQなどの他の組織におけるタンパク質凝集体の定量化におけるこれらの方法の適用を実証する(補足図1)。ただし、セクション 8 NOTE で説明したように、集計サイズと明るさを適切に調整するには、パイプラインを変更する必要があります。

図 1: ワークフローの視覚的表現 プロトコルの主なステップには、ワームの準備と年齢同期(ステップ1〜5)、腸のコロニー形成/ワーム治療(ステップ6)、イメージングのためのサンプル準備(ステップ7)、画像取得(ステップ8)、および画像処理(ステップ9)の5つの異なる段階が含まれます。プロトコルの「セクション」は、図では「ステップ」として参照されています。 この図の拡大版を表示するには、ここをクリックしてください。
最初の最適化実験では、子孫の数が多いため過密状態に関連するさまざまな困難が明らかになり、食物の枯渇が速くなりました。セクション2で説明したNGMプレートにFUDRを補足することで、この問題を解決しました(図2)。さらに、FUDRの存在下では、様々な細菌を与えられたワームは、より一貫した体の大きさを有し、より均一で正確なワーム検出を可能にした。

図2:FUDRを使用すると、子孫が減少して画質が向上します。 FUDRサプリメントプレートは、大腸菌OP50を播種した非FUDR対照NGMプレートで増殖したワームと比較して、C.エレガンスの子孫を排除する。画像は25.2倍の倍率(0.63倍のカメラアダプタで40倍の倍率)で取得されました。スケール バー = 500 μm。この図の拡大版を表示するには、ここをクリックしてください。
バックグラウンド蛍光は、polyQ凝集体の偽陽性検出に寄与した。腸管内のこのような蛍光シグナルを減少させ、凝集体の自動検出を改善するためには、イメージングの前にワームを凍結する必要があった。-20°Cでワームを18~48時間凍結すると、バックグラウンド蛍光を除去することでpolyQ凝集体検出が有意に向上しました(図3A)。人間の目は、凝集体とバックグラウンド蛍光を区別することができます。したがって、フリーズ前後の手動カウントは同じです(図3B)。しかし、自動計数はそれほど正確ではありませんが、凍結すると、手動計数に匹敵する精度で自動計数が大幅に向上しました(図3B)。

図3:凍結により凝集体検出が改善される(A)凍結前後の腸内polyQ44::YFPを発現するC.エレガンスの蛍光画像。挿入は、選択した領域のクローズアップ画像を表します。(B)手動または自動(パイプライン)凝集体定量を使用して凍結前後に緑膿菌MPAO1をコロニー形成したワームにおける腸あたりの平均凝集体数。データは2つの生物学的複製(n=60〜109)を表す。統計的有意性は、スチューデントのt検定(**** p < 0.0001)を使用して計算しました。エラーバーは、平均の標準誤差(SEM)を表す。この図の拡大版を表示するには、ここをクリックしてください。
反転明視野照明を用いて、C.エレガンス全体(図4A)およびGFPチャネルを検出してpolyQ44::YFP凝集体(図4B)を画像化した。各ワームのワーム検出、もつれの解消、および凝集量の定量化は、最適化された CellProfiler 画像処理パイプライン (補足ファイル 1) を適用して行われ、個々のワームあたりの凝集体数を取得できます (図 4C-D)。このアプローチの実現可能性と自動凝集体検出および定量の精度をテストするために、腸特異的polyQ44::YFPを発現するワームを培養し、確立されたプロトコルに従ってイメージング用に調製した(セクション1〜7)。ワームあたりの集計数は、自動パイプライン (セクション 8 ~ 9) または手動カウントのいずれかを使用して評価されました。各実験は、条件ごとに90〜571匹のワームを用いた3つの独立した試験で実施した。2回の試験で得られた腸あたりの平均凝集体数に有意差はなかったが、3回目の試験では、自動アプローチを用いて定量化した場合、ワームの凝集体は有意に少なかった(図5A)。3 回の試行による集計の平均数は、CellProfiler パイプライン (セクション 8 ~ 9) を使用して定量化を行った場合、わずかに減少しましたが、凝集体はわずかに減少しました (図 5B)。それにもかかわらず、2つのアプローチの違いは最小限であり、自動化された方法を大規模な画面に適用できることを示しています。
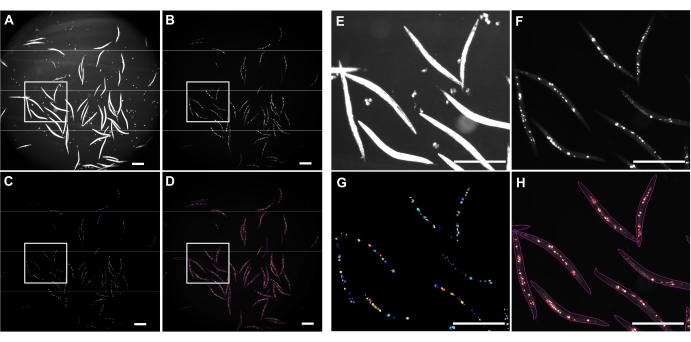
図 4: CellProfiler を使用した凝集検出 (A) ワームの体を識別するために使用される明視野画像。(b)GFPチャネルを用いて取得し、腸内polyQ44::YFP凝集体の総数を同定および定量するために使用した元の蛍光画像。(C) セルプロファイラを使用して識別された集計。(d)同定された凝集体の総数を、元の蛍光画像の上にワームおよび凝集体の輪郭とともに重ね合わせた。画像のキャプチャと処理は、セクション8〜9で説明されている設定を使用して実行されました。パネル E〜H は、画像 A〜Dにおける対応する輪郭を描かれた領域のクローズアップ画像を表す。画像は25.2倍の倍率(0.63倍のカメラアダプタで40倍の倍率)で取得されました。スケール バー = 500 μm 。この図の拡大版を表示するには、ここをクリックしてください。
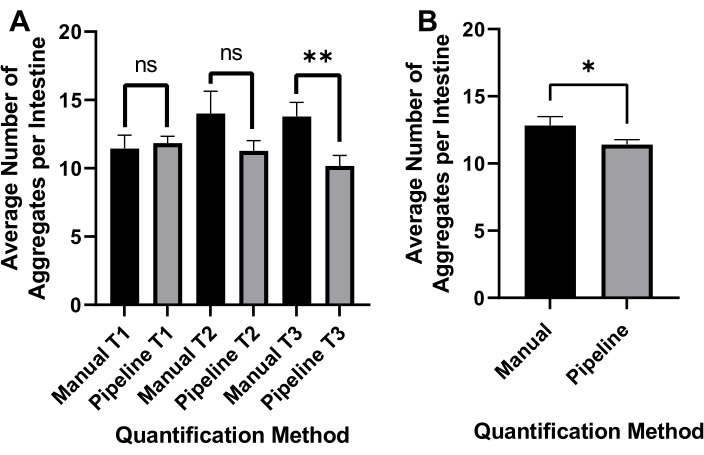
図5:自動凝集体定量の有効性 (A)手動計数(手動)および自動CellProfilerベースの定量化(パイプライン)を使用して、対照 大腸菌 OP50をコロニー形成したワームにおける腸あたりの平均凝集数。結果は、3つの別々の試験(T1−T3)で分析されたデータを表す(n = 90−571)。(B)腸当たりの平均凝集体数は、手動または自動(Pipeline)凝集体定量を用いて得られた。統計的有意性は、スチューデントのt検定(*p<0.05、**p<0.01)を用いて計算した。エラー バーは SEM を表します。 この図の拡大版を表示するには、ここをクリックしてください。
異なる実験者の間での結果の再現性を評価するために、 緑膿菌 MPAO1または 大腸 菌OP50のいずれかを与えられたワームの6つのウェルを含むプレートからの画像を、3人の個人によって取得し、そのうちの2人はこれらのプロトコルを使用してワームをイメージングした経験がなかった。各ウェルから収集された画像には、検出されたワームが30〜115個ありました。凝集の有意でない差は、3人の実験者によって画像化された同じウェルからのワームにおいて検出された。MPAO1とOP50を投与したワームの3人の実験者の間では、腸あたりの平均凝集体数は非常に一貫していましたが、凝集体の平均数には統計的に有意な差がありましたが、MPAO1によってコロニー形成したワームでのみ一致しました(補足図2)。これらの結果は、経験の浅い実験者の間でも結果の再現性を強調しています。
凝集体定量の再現性がワームの位置に大きく影響されないようにするために、15個のワームのセットを選択し、ピペットチップを使用して各画像キャプチャの間に攪拌後に15回別々に画像化しました。 大腸菌 OP50および 緑膿菌 MPAO1を投与したワームにおける凝集体の画像を収集し、CellProfilerを用いて分析した。これらの異なる画像セットのそれぞれからの凝集体の平均数はわずかに異なるが有意ではなく、このアプローチの再現性をさらに支持した(補足図3)。
グラム陰性腸内病原体による C.elegans 腸のコロニー形成は、組織全体のプロテオスタシスを破壊することが示されており、 緑膿菌 はpolyQ凝集の最も強力な誘導因子の1つである9。これらの最適化されたプロトコールが 緑膿菌媒介性凝集の増強を首尾よく検出および定量するかどうかを決定するために、腸内polyQを発現するワームを 大腸菌 OP50(対照細菌)とコロニー形成し、 緑膿菌 MPAO1、セクション1〜8を実施した。取得した画像は、CellProfiler(セクション9、 補足ファイル1)を使用して分析されました。自動定量の結果は 、緑膿菌 MPAO1によって誘導される凝集体数の有意な増加を示し、対照 大腸菌 OP50を給餌したワームと比較して一貫して2倍の増加をもたらす(図6)。

図6:対照 大腸菌 OP50および 緑膿菌 MPAO1でコロニー形成したワームにおける腸当たりの平均凝集体数。 腸あたりの凝集体の数は、CellProfilerを用いて評価した(セクション8〜9)。データは、OP50(n = 1068)およびMPAO1(n = 1557)でコロニー化されたワームにおける腸あたりの凝集体の平均数として表される。統計的有意性は、スチューデントのt検定(**** p < 0.0001)を使用して計算しました。エラー バーは SEM を表します。 この図の拡大版を表示するには、ここをクリックしてください。
最適化されたパイプラインは、プロテオスタシスに影響を及ぼす条件の大規模なスクリーンをサポートするように設計されています。宿主プロテオスタシスに対するそれらの効果について細菌の大規模なライブラリーをスクリーニングする際のこのアプローチの実現可能性を試験するために、本明細書に記載のパイプライン(セクション1〜9)を用いて、polyQ凝集18に対する90P.緑膿菌非必須遺伝子ノックアウト変異株の効果を試験した。このパイロットスクリーンは、すべての緑膿菌非必須変異株を宿主プロテオスタシスに影響を及ぼす能力についてスクリーニングするために設計されたより大きなプロジェクトの一部です。試験した90の細菌株のうち、1つの候補による腸内C.elegansのコロニー形成は、凝集体数の有意な減少を示した(図7)。このアッセイの感度を評価するためのフォローアップ実験は、MPAO1対照と非有意に異なる6つの緑膿菌変異体のランダム選択からの手動凝集カウントを介して実施した。これらの実験は、より伝統的な6cmNGMプレートを用いて実施し、以前に確立された方法9を要約するためにL1としてワームを試験株に移した。手動計数による確認実験により、凝集体の数を有意に減少させたもの(図7)を含む変異体のいずれもpolyQ凝集に影響を与えないことが明らかになった(補足図4)。さらに、90の変異株のスクリーンで観察された凝集の微妙な変化は、選択された候補の手動カウントの間で検出されず、そのような変化が、低いn値などの生物学的および実験的変動性のために起こり得ることを示している。全体として、結果は、私たちの方法が重要な変化を確実に拾うことができるが、微妙な変化は見逃される可能性が高く、すべての潜在的な候補を個別に確認する必要があることを示している。

図7:92の細菌株によってコロニー形成されたワームの代表的なサンプルセットにおける腸あたりの凝集体の数。 データは、MPAO1でコロニー形成されたワームのそれに正規化された腸あたりの凝集体の平均数として表されます。点線は、MPAO1(上、白丸)およびOP50対照(下、白四角)でコロニー化されたワームにおける凝集体の平均数を表す。実線の記号は 、緑膿菌 MPAO1の90個の異なるノックアウト変異株を表す。MPAO1でコロニー形成したワームと単一の変異体との間のワームあたりの平均凝集体数は統計的に有意であった。灰色の円は、手動で確認されたサンプルを表す(補足図4)。統計的有意性は、一元配置分散分析(ANOVA)とそれに続く多重比較ダネットの事後検定(** p < 0.01、**** p < 0.0001)を使用して計算されました。エラー バーは SEM を表します。 この図の拡大版を表示するには、ここをクリックしてください。
CellProfilerによって生成される大量のデータを管理するために、グラフィカル・ユーザー・インターフェース(GUI)が開発され、データ処理と編成が自動化されました(図8)。GUIは、オープンソースのPythonクロスプラットフォームウィジェットツールキットであるTkinterを使用して開発されました。与えられたメタデータから、アプリケーションは、プレート中に存在する各ウェル(カラムJ)から凝集体(カラムK)の数を抽出する。前述のプロセスを実行するために、 "Pandas"と呼ばれるPythonデータ処理ライブラリが使用されました。GUI アプリケーションは、ユーザーがデータ・ファイルをアップロードするためのドラッグ・アンド・ドロップ・サポートを提供します。各ファイル内のデータは、データフレームと呼ばれる2次元の表形式構造の形式で格納されます。空のディクショナリペアは、データフレーム内で見つかった一意のウェルごとに初期化されます。次に、各ウェルで見つかった個別の集計がカウントされ、それぞれの辞書ペアに追加されます。データが少ない列には、各列のサイズが均等になるように、空の値文字列が埋め込まれます。最後に、構造はデータフレームに変換され、スプレッドシートの形式でユーザーが指定したディレクトリにエクスポートされます。

図 8: グラフィカル ユーザー インターフェイス。この図の拡大版を表示するには、ここをクリックしてください。
補足図1:筋肉特異的polyQ凝集体の検出。 筋肉特異的polyQ35::YFPを発現するワームをL1として播種し、OP50上で48時間培養した。ワームが若年成人に発達すると、24ウェルNGMプレートに移し、100μg/mL FUDRを補充し、MPAO1をさらに72時間播種してからイメージングを行った。(A)ワームの体を識別するために使用される明視野画像。(b)GFPチャネルを用いて取得したオリジナルの蛍光画像。(C) セルプロファイラを使用して識別された集計。(d)同定された凝集体の総数を、元の蛍光画像の上にワームおよび凝集体の輪郭とともに重ね合わせた。画像のキャプチャと処理は、セクション8〜9で説明されている設定を使用して実行されました。パネル E〜H は、画像 A〜Dにおける対応する輪郭を描かれた領域のクローズアップ画像を表す。スケール バー = 500 μm 。このファイルをダウンロードするには、ここをクリックしてください。
補足図2:異なる実験者間の凝集定量の再現性。 緑膿菌MPAO1でコロニー形成した6ウェルのワーム(黒いバー)およびコントロール大腸菌OP50でコロニー形成したワームの6ウェル(灰色のバー)について、CellProfilerを使用して定量した凝集体の平均数。各ウェルは、3人の実験者(AVS、DMC、RDH)によって画像化された。データは、腸あたりの平均凝集体数として表される(n=30〜115)。統計的有意性は、一元配置分散分析とそれに続くTukeyの多重比較検定(*p<0.05、**p<0.01)を用いて計算した。エラー バーは SEM を表します。このファイルをダウンロードするには、ここをクリックしてください。
補足図3:凝集体定量の再現性に対するワーム位置の影響。 対照 大腸菌 OP50(灰色のバー)および 緑膿菌 MPAO1(黒いバー)でコロニー形成したワームにおける腸あたりの平均凝集数。結果は、CellProfilerを使用して定量された腸あたりの凝集体の平均数(15≥n≥12)を表す。井戸内のワームの位置は、各取得の間の攪拌によって変化した。いずれの群においても統計的に有意な差は認められなかった。統計的有意性は、一元配置分散分析とそれに続くTukeyの多重比較検定を使用して計算されました。エラー バーは SEM を表します。 このファイルをダウンロードするには、ここをクリックしてください。
補足図4:手動カウントによるパイロット画面の確認。 腸当たりの平均凝集体数を、手動で定量した。データは、野生型MPAO1およびOP50対照(n=30)と比較した6つのMPAO1ノックアウト変異体(灰色の円 図7)でコロニー化されたワームの凝集プロファイルを表す。統計的有意性は、一元配置分散分析とそれに続く多重比較ダネットの事後検定(**** p < 0.0001)を用いて計算された。エラー バーは SEM を表します。 このファイルをダウンロードするには、ここをクリックしてください。
補足図5:腸のpolyQ凝集に対するFUDRの効果。 データは、腸当たりのpolyQ44::YFP凝集体の平均数(n = 20)として表される。ワームを 、大腸菌 OP50上で25°Cで48時間増殖させた後、コントロール(FUDRなし)またはFUDR含有プレート(100μg/mL)上に移した。手動カウントは、さらに48時間後に収集された。統計的有意性は、スチューデントのt検定(ns=有意ではない)を使用して計算されました。エラー バーは SEM を表します。 このファイルをダウンロードするには、ここをクリックしてください。
補足ファイル1:プロテオスタシスパイプライン。CellProfiler で使用するためのダウンロード可能な画像解析パイプライン。申請の手順はセクション9に記載されています。 このファイルをダウンロードするには、ここをクリックしてください。
補足ファイル2:もつれを解くワームのトレーニングセット。 "UntangleWorms" モジュールにアップロードするファイル。この特定のトレーニング セットは、最初のアプローチで使用されたワームに固有です。ワームのサイズと形状を変更すると、識別の精度と品質が変わります。よりパーソナライズされたトレーニングファイルを作成する必要があるかもしれません。新しいトレーニングセットを作成する手順については、CellProfilerの公式ウェブサイト17を参照してください。 このファイルをダウンロードするには、ここをクリックしてください。
補足ファイル3:Windowsオペレーティングシステム用のグラフィカルユーザーインターフェイス。 gui_windowsOS_64x.zip。 このファイルをダウンロードするには、ここをクリックしてください。
補足ファイル4:Macオペレーティングシステム用のグラフィカルユーザーインターフェイス。 gui_MacOS_64x.zip。 このファイルをダウンロードするには、ここをクリックしてください。
ディスカッション
記載されているプロトコルは、オープンソースの画像解析ソフトウェアであるCellProfilerを組み込んだ C. elegans の培養、イメージング、および画像処理の手順を概説しています。代表的な結果は、再現性、バイアスの低減、およびスケーラビリティを実証しています。この標準化された手順は、大規模な細菌、ゲノム、または薬物ライブラリで使用されるスクリーニング戦略を改善します。物体検出の 自動化されたC. elegans メソッドは他にもありますが、ここで説明する手法は、培養、画像取得、および分析を統合する標準化された高スループットのパイプラインを提供します。
ワーム培養のいくつかのバリエーションは、本明細書に記載されるプロトコルを最適化するために試験されなければならなかった。当初、ワームは、年齢同期後(L1段階)に直ちに試料細菌に移された。しかし、そのようなアプローチは、同じ井戸内のワームの間でさえ、可変サイズのワームの集団をもたらした。C. elegansは病原体回避19で知られており、これは観察されたサイズの変動性に寄与し、最終的に下流のイメージングワーム検出に特に影響する可能性がある。このような変動性を排除するために、各ウェル中のNGM領域全体を試験細菌で覆った。さらに、ワームに大腸菌OP50を与え、25°Cで48時間の間、若年成人に完全に発達させた。 ワームを試験細菌に移す前に大腸菌OP50で成虫期に達することを可能にすると、より一貫した体の大きさが得られました。さらに、子孫による過密状態および急速な食物枯渇は、NGM寒天をFUDRで補うことによって排除された。FUDRの実装により、子孫が排除され、子孫が親集団と混ざり合うことによって不明瞭になっていた自動ワーム識別が強化されました。しかしながら、FUDRを利用する際には、化合物がC.エレガンス・プロテオスタシスおよび寿命に影響を及ぼすことが知られているため、慎重であり、適切な対照を使用することが重要である20,21。このプロトコルに記載されている条件下では、FUDRは腸のpolyQ凝集に影響を及ぼさなかった(補足図5)。したがって、その利用は、記載された方法に適切かつ有益であった。
イメージング前のサンプルの凍結は、パイプラインの採用を成功させるための重要なステップであることが判明しました。凍結前の集計カウントは、手動カウントよりも有意に高かった(図3B)。イメージングの前にワームを-20°Cで18~48時間保持すると、バックグラウンド蛍光が減少し、最終的に凝集体検出が改善されました(図3A)。凝集体検出に対する凍結の影響は、polyQについてのみ調査されており、そのような影響のさらなる調査なしに他のモデルに一般化されるべきではありません。
すべての条件が同じに保たれているにもかかわらず、ワーム1匹当たりの平均凝集体数は異なる実行間で変化する可能性があることが観察されたが、OP50とMPAO1をコロニー形成した動物の凝集体数の間の比は一貫していた(図6、補足図2、補足図5)。したがって、すべての実行に大腸菌OP50コントロール、または追加の適切な参照コントロールを常に含めることが不可欠です。実験間の総計数のこのような変動は、環境条件(温度、湿度)22、23または遺伝的背景8によって影響される可能性がある。実際、長期培養後、腸内蛍光が劇的に減少するか完全に失われ、凍結ストックから新しい株を解凍する必要があることが観察された。観察された蛍光の減少は、polyQを発現する遺伝子などの毒性導入遺伝子を抑制する遺伝子変化の結果である可能性がある。それにもかかわらず、異なる実験者間(補足図2)、生物学的複製(図5)、および同じサンプル内(補足図3)内で観察された結果の例外的な再現性は、このアプローチの強さを強調している。
多数の報告が、プロテオスタシス9、11、12、13、24、25を研究するために腸内polyQを使用している。しかし、実験的アプローチと読み出し方法のばらつきにより、結果を直接比較することはできません。それにもかかわらず、以前に公表されたデータからのいくつかの結果は、凝集9、13および同程度の数の凝集体11の細菌誘導を含む、本明細書に記載の自動定量化によって要約される。まとめると、記載されたパイプラインは、プロテオスタシスを研究するための貴重なツールを提供します。
本明細書に記載される方法には、いくつかの固有の課題がある。たとえば、このプロトコルのすべてのコンポーネントを習得するには十分な時間が必要であり、これはプロトコルのセクション8に特に当てはまり、取得した画像がパイプライン解析に適しているかどうかを判断するためにアッセイに精通している必要があります。このプロトコルで使用される画像集録設定からの逸脱は可能です。ただし、設定とワームトレーニングセットの変更が必要になる可能性があります。このパイプラインは、さまざまなサイズの凝集体と接触しているものを区別できるため、凝集体の「ブレンド」が制限され、最終的に検出感度が向上します。ただし、許容サイズ範囲を超える大きなアグリゲートを特定しようとすると、上限サイズのしきい値を拡張すると、タッチしているアグリゲートを区別できないなど、識別不良によるエラーが発生する可能性があるため、問題が発生する可能性があります。画像解析の前に、精度、サイズ、強度のバランスを見つける必要があります。集約識別の効率は、機械学習を組み込んで集合体検出を強化できるニューラルネットワークを作成することによってさらに改善することができる。このような改善は現在検討中であり、異なる焦点面上に存在する凝集体や異常な形状を有する凝集体の検出などの現在の問題に対処するのに大いに役立つであろう。
記載された方法の注目すべき弱点の1つは、異なる細菌株を与えられたワームの手動カウントによって常に反復されるとは限らないため、自動集計カウントの変動性である。例えば、自動カウントに基づいて、 緑膿菌 変異体53(M53)を与えられたワームは、野生型株(MPAO1)と比較して有意に少ない凝集体を有していた(図7)。しかし、ヒットの確認では有意差は見られなかった(補足図4)。一般に、ハイスループット薬物スクリーニングは、偽陽性ヒット検出の高い率を有し、記載された方法も例外ではない26。したがって、すべての潜在的なヒットを確認することはプロトコルの重要な部分です。
このプロトコルは、宿主プロテオスタシスに影響を及ぼす細菌を同定するためのスクリーニング戦略に合うように最適化されましたが、各ステップをさらに変更して、ゲノムRNAiライブラリー、小分子、またはその他の条件の効果を試験することができます。特定のスクリーニング戦略の要件に合わせて、各ステップで追加の変更を行うことができます。さらに、この手法は、特定のモデルに合わせて各ステップの最適化を可能にするレベルの柔軟性を提供します。例えば、このアプローチは、他の組織におけるpolyQ凝集に拡張することができ、または、誘導性蛍光レポーター(例えば、ヒートショック遺伝子)を用いて遺伝子発現をモニタリングする、タンパク質の細胞内局在を評価する(例えば、DAF−16の核局在化)、他の疾患モデルにおける凝集を研究する(例えば、Aβ1−42、α−シヌクレイン、TDP−43など)または生理学的表現型を評価するなど、画像において検出された他の特徴を抽出すること、 ワームのサイズなど。
開示事項
著者らは、利益相反は存在しないと宣言している。
謝辞
この研究は、国立衛生研究所(1RO3AG069056-01)とDMCへの資金提供を受けたアメリカ感染症学会の支援を受けた。資金提供者は、研究デザイン、データ収集と分析、出版の決定、または原稿の準備において何の役割も持っていませんでした。原稿を校正してくれたCzyz Labのメンバーに感謝します。漫画のフィギュアは、BioRenderの有料ライセンスを使用して生成されました。
資料
| Name | Company | Catalog Number | Comments |
| 1.7 mL Microtubes | Olympus Plastics | Cat#24-282 | Microcentrifuge tubes |
| 10 mL Serological Pipettes | GenClone | Cat#12-104 | Plastic pipettes |
| 15 mL Centrifuge Tubes, Racked | Olympus Plastics | Cat#28-101 | Conical tubes |
| 5-Fluoro-2′-deoxyuridine | Fisher Scientific | D2235100MG | FUDR |
| Accu-jet Pro Pipette Controller | Genesee Scientific | 91-600RB | Pipette gun |
| Agar | Fisher Scientific | Cat#BP1423-2 | Granulated agar |
| BioRender | BioRender | Graphical figure generator | |
| Bleach (Regular) | Clorox | Bar# 044600324111, Splash-less Bleach | 4.5% sodium hypochlorite |
| C. elegans: AM140: rmIs132 [unc-54p::q35::yfp] | Morimoto Lab (Northwestern University) | AM140, Q35::YFP | Muscle polyQ |
| C. elegans: AM738: rmIs297[vha-6p::q44::yfp; rol-6(su1006)] | Morimoto Lab (Northwestern University) | AM738, Q44::YFP | Intestinal polyQ |
| CaCl2·2H2O | Fisher Scientific | Cat#C79-500 | Calcium chloride dihydrate |
| CellProfiler | Broad Institute | Image analysis software | |
| Cholesterol | Fisher Scientific | Cat#ICN10138201 | Cholesterol |
| Circulating Water Bath Head | Lauda | 26LE | Lauda E 100 |
| CoolLED pE300lite 365 dir mount STEREO | CoolLED | 8114931 | Fluorescent light control and emitter |
| 16 mL Culture Tubes | Olympus Plastics | 21-129 | Culture tubes, 17 mm x 100 mm |
| Escherichia coli OP50 | Caenorhabditis Genetics Center | OP50 | E. coli control strain |
| Ethanol, 200 Proof | Decon Labs, Inc. | 2701 | |
| Eyepiece 10x/23B, adjustable, 3d gen | Leica Microsystems | 10450910 | Eyepiece set |
| Filter Set ET GFP - MZ10F | Leica Microsystems | 10450588 | Filter cube |
| GraphPad Prism v9.2.0 | GraphPad Software, Inc. | Statistical analysis tool | |
| Heratherm Incubator IMP180 | Thermo | 51031562 | Refrigerated incubator |
| Innova 4000 | New Brunswick Scientific | M1192-0000 | Shaking incubator |
| K5 Camera | Leica Microsystems | 11547112 | Stereomicroscope camera |
| KH2P04 | Fisher Scientific | Cat#P285-3 | Potassium phosphate monobasic |
| LAS X Imaging Software | Leica Microsystems | Microscope imaging software | |
| Leica MZ10 F Optics Carrier | Leica Microsystems | 10450103 | Stereomicroscope |
| Levamisole | Fisher Scientific | Cat#0215522805 | Levamisole hydrochloride |
| Luria Broth (Lennox) | Apex Bioresearch Products | Cat#11-125 | LB |
| Magnetic Stir Plate | Fisher Scientific | 11-100-49S | Stir plate |
| MgSO4·7H2O | Alfa Aesar | A14491 | Magnesium sulfate heptahydrate |
| Microscope Slides | Premiere | 8205 | Single frosted microscope slides |
| Na2HPO4·7H2O | Fisher Scientific | Cat#S373-500 | Sodium phosphate dibasic heptahydrate |
| NaCl | Fisher Scientific | Cat#S671-500 | Sodium chloride |
| NaOH | Fisher Scientific | Cat#S318-3 | Sodium hydroxide pellets |
| Objective Achromat, f = 100 mm | Leica Microsystems | 10411597 | Objective microscope lens |
| Petri Dishes | Genesee Scientific | Cat#32-107G | 100 mm x 15 mm |
| Pseudomonas aeruginosa Mutant Library | Manoil Lab (University of Washington) | P. aeruginosa mutant library | |
| Suspension Culture Plate 24-Well, Flat Bottom | Olympus Plastics | 25-102 | Used for worm growth and imaging |
| Trinocular Tube 100% M-series | Leica Microsystems | 10450043 | |
| Trypticase Peptone | ThermoFisher, Difco | Cat#211921 | |
| TX-400 Rotor | Thermo Scientific | Cat#75003181 | Swing bucket rotor |
| Vacuum Driven Filter System | GenClone | Cat#25-227 | 500 mL, PES Membrane, .22 µm |
| Video Objective with C-Mount | Leica Microsystems | 10447367 | 0.63x camera adapter tube |
参考文献
- Soto, C. Unfolding the role of protein misfolding in neurodegenerative diseases. Nature Reviews Neuroscience. 4 (1), 49-60 (2003).
- Fang, P., Kazmi, S. A., Jameson, K. G., Hsiao, E. Y. The microbiome as a modifier of neurodegenerative disease risk. Cell Host & Microbe. 28 (2), 201-222 (2020).
- Kundu, P., Blacher, E., Elinav, E., Pettersson, S. Our gut microbiome: The evolving inner self. Cell. 171 (7), 1481-1493 (2017).
- Sherwin, E., Dinan, T. G., Cryan, J. F. Recent developments in understanding the role of the gut microbiota in brain health and disease. Annals of the New York Academy of Sciences. 1420 (1), 5-25 (2018).
- Mondal, S., et al. Large-scale microfluidics providing high-resolution and high-throughput screening of Caenorhabditis elegans poly-glutamine aggregation model. Nature Communications. 7 (1), 13023(2016).
- Guisbert, E., Czyz, D. M., Richter, K., McMullen, P. D., Morimoto, R. I. Identification of a tissue-selective heat shock response regulatory network. PLoS Genetics. 9 (4), 1003466(2013).
- Silva, M. C., et al. A genetic screening strategy identifies novel regulators of the proteostasis network. PLoS Genetics. 7 (12), 1002438(2011).
- Nollen, E. A., et al. Genome-wide RNA interference screen identifies previously undescribed regulators of polyglutamine aggregation. Proceedings of the National Academy of Sciences of the United States of America. 101 (17), 6403-6408 (2004).
- Walker, A. C., et al. Colonization of the Caenorhabditis elegans gut with human enteric bacterial pathogens leads to proteostasis disruption that is rescued by butyrate. PLOS Pathogens. 17 (5), 1009510(2021).
- Goya, M. E., et al. Probiotic Bacillus subtilis protects against α-Synuclein aggregation in C. elegans. Cell Reports. 30 (2), 367-380 (2020).
- Kumsta, C., Chang, J. T., Schmalz, J., Hansen, M. Hormetic heat stress and HSF-1 induce autophagy to improve survival and proteostasis in C. elegans. Nature Communications. 8, 14337(2017).
- Prahlad, V., Morimoto, R. I. Neuronal circuitry regulates the response of Caenorhabditis elegans to misfolded proteins. Proceedings of the National Academy of Sciences of the United States of America. 108, 14204-14209 (2011).
- Mohri-Shiomi, A., Garsin, D. A. Insulin signaling and the heat shock response modulate protein homeostasis in the Caenorhabditis elegans intestine during infection. Journal of Biological Chemistry. 283 (1), 194-201 (2008).
- Hakim, A., et al. WorMachine: machine learning-based phenotypic analysis tool for worms. BMC Biology. 16 (1), 8(2018).
- Wählby, C., et al. An image analysis toolbox for high-throughput C. elegans assays. Nature Methods. 9 (7), 714-716 (2012).
- Jones, T. R., et al. CellProfiler Analyst: data exploration and analysis software for complex image-based screens. BMC Bioinformatics. 9 (1), 482(2008).
- CellProfiler: an open-source image analysis software. , Available from: http://www.cellprofiler.org (2021).
- Held, K., Ramage, E., Jacobs, M., Gallagher, L., Manoil, C. Sequence-verified two-allele transposon mutant library for Pseudomona aeruginosa PA01. Journal of Bacteriology. 194 (23), 6387-6389 (2012).
- Schulenburg, H., Ewbank, J. J. The genetics of pathogen avoidance in Caenorhabditis elegans. Molecular Microbiology. 66 (3), 563-570 (2007).
- Feldman, N., Kosolapov, L., Ben-Zvi, A. Fluorodeoxyuridine improves Caenorhabditis elegans proteostasis independent of reproduction onset. PLoS One. 9 (1), 85964(2014).
- Van Raamsdonk, J. M., Hekimi, S. FUdR causes a twofold increase in the lifespan of the mitochondrial mutant gas-1. Mechanisms of Ageing and Development. 132 (10), 519-521 (2011).
- Haldimann, P., Muriset, M., Vígh, L., Goloubinoff, P. The novel hydroxylamine derivative ng-094 suppresses polyglutamine protein toxicity in Caenorhabditis elegans. Journal of Biological Chemistry. 286 (21), 18784-18794 (2011).
- Shinn-Thomas, J. H. Wrapping culture plates with Parafilm M® increases Caenorhabditis elegans growth. BMC Research Notes. 12 (1), (2019).
- Alexander-Floyd, J., et al. Unexpected cell type-dependent effects of autophagy on polyglutamine aggregation revealed by natural genetic variation in C. elegans. BMC Biology. 18 (1), 18(2020).
- Moronetti Mazzeo, L. E., Dersh, D., Boccitto, M., Kalb, R. G., Lamitina, T. Stress and aging induce distinct polyQ protein aggregation states. Proceedings of the National Academy of Sciences of the United States of America. 109 (26), 10587-10592 (2012).
- Sink, R., Gobec, S., Pecar, S., Zega, A. False positives in the early stages of drug discovery. Current Medicinal Chemistry. 17 (34), 4231-4255 (2010).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved