Method Article
Automatisation de la quantification agrégée chez Caenorhabditis elegans
Dans cet article
Résumé
Le protocole suivant décrit le développement et l’optimisation d’un flux de travail à haut débit pour la culture de vers, l’imagerie par fluorescence et le traitement automatisé d’images afin de quantifier les agrégats de polyglutamines en tant qu’évaluation des changements dans la protéostasie.
Résumé
L’augmentation de la prévalence des maladies neurodégénératives de conformation des protéines (PCD) a suscité un grand intérêt pour ce sujet au fil des ans. Cette attention accrue a appelé à la diversification et à l’amélioration des modèles animaux capables de reproduire les phénotypes de maladies observés chez l’homme atteints de PCD. Bien que les modèles murins se soient avérés inestimables, ils sont coûteux et associés à des méthodes laborieuses et à faible débit. L’utilisation du modèle de nématode Caenorhabditis elegans pour étudier les PCD a été justifiée par sa relative facilité d’entretien, son faible coût et son temps de génération rapide, qui permettent des applications à haut débit. De plus, la conservation élevée entre le génome de C. elegans et les génomes humains fait de ce modèle un outil de découverte inestimable. Les nématodes qui expriment des voies de polyglutamine spécifiques aux tissus (polyQ) marquées par fluorescence présentent une agrégation dépendante de l’âge et de la longueur de la polyQ caractérisée par des foyers fluorescents. Ces rapporteurs sont souvent utilisés comme mandataires pour surveiller les changements dans la protéostasie à travers les tissus. La quantification manuelle des agrégats prend beaucoup de temps et limite le débit expérimental. En outre, la quantification manuelle des foyers peut introduire un biais, car l’identification globale peut être très subjective. Ici, un protocole comprenant la culture de vers, l’acquisition d’images et le traitement de données a été normalisé pour prendre en charge la quantification agrégée à haut débit à l’aide de C. elegans qui expriment le polyQ spécifique à l’intestin. En mettant en œuvre un pipeline de traitement d’image basé sur C. elegans à l’aide de CellProfiler, un logiciel d’analyse d’images, cette méthode a été optimisée pour séparer et identifier les vers individuels et énumérer leurs agrégats respectifs. Bien que le concept d’automatisation ne soit pas tout à fait unique, la nécessité de normaliser ces procédures pour la reproductibilité, l’élimination des biais du comptage manuel et l’augmentation du débit est élevée. On s’attend à ce que ces méthodes puissent simplifier considérablement le processus de criblage des grandes bibliothèques bactériennes, génomiques ou de médicaments à l’aide du modèle C. elegans .
Introduction
Les maladies neurodégénératives conformationnelles des protéines (PCD) dépendantes de l’âge telles que les maladies d’Alzheimer, de Parkinson et de Huntington, ou la sclérose latérale amyotrophique, sont caractérisées par un mauvais repliement des protéines qui conduit à l’agrégation, à la mort cellulaire et à la dégénérescence tissulaire1. Bien que le mauvais repliement des protéines soit reconnu comme le coupable, l’étiologie de ces maladies n’est pas claire. En tant que tel, le développement de thérapies efficaces a été entravé par le manque de connaissances concernant les facteurs et les conditions qui contribuent à l’apparition et à la progression de la maladie. Des études récentes suggèrent que les changements dans le microbiome influencent l’apparition, la progression et la gravité des PCD 2,3,4. Cependant, la complexité du microbiome humain, voire murin, rend difficile la réalisation d’études qui révéleraient l’influence exacte des microbes sur leur hôte. Par conséquent, des organismes plus simples, tels que Caenorhabditis elegans, sont souvent utilisés comme outil de découverte 5,6,7,8. Des études récentes ont utilisé C. elegans pour étudier l’effet des bactéries sur la protéostasie de l’hôte et la pathogenèse de la maladie 9,10. La colonisation bactérienne, l’hormèse et les changements génomiques font partie des conditions exemplaires qui affectent l’agrégation des tracts de polyglutamine (polyQ) 9,11,12. De plus, ces amas de protéines mal repliés présentent une accumulation polyQ dépendante de la longueur et de l’âge chez l’hôte et sont associés à une motilité altérée 9,13. L’approche relativement simple de la quantification des ponctuations marquées par fluorescence peut générer des données importantes sur les conditions, les facteurs ou les médicaments qui affectent le repliement et l’agrégation des protéines.
Bien que la quantification de la ponctuation fluorescente se soit avérée être une procédure fiable et relativement simple, le défi reste de développer un protocole qui faciliterait le dépistage à grande échelle des composés, des bactéries ou des conditions qui affectent l’agrégation des protéines. Le concept de traitement automatisé de l’image C. elegans et de quantification puncta n’est pas entièrement nouveau, car un certain nombre d’outils de support pratiques ont été développés14,15. Cependant, l’intégration de la culture, de l’acquisition d’images et d’un pipeline de traitement est essentielle pour éliminer la variabilité des résultats et permettre des écrans à plus haut débit.
En tant que tel, l’intention de ce manuscrit est de normaliser la procédure utilisée pour quantifier l’agrégation polyQ chez C. elegans comme proxy pour détecter les changements dans la protéostasie. Cette tâche a été accomplie en utilisant CellProfiler, un logiciel d’analyse d’images open source16 capable d’identification automatisée des vers et des agrégats, et est intégré dans un protocole plus large pour la culture de vers, l’acquisition d’images et le traitement des données.
Protocole
Toutes les procédures ont suivi les directives de sécurité qui ont été examinées et approuvées par le Comité institutionnel de biosécurité de l’Université de Floride. Des mesures de biosécurité appropriées ont été prises pour atténuer le risque d’exposition aux bactéries de niveau de sécurité biologique 2.
REMARQUE: Pour toutes les expériences, C. elegans doit être propagé et maintenu sur des plaques de milieu de croissance des nématodes (NGM) ensemencées avec Escherichia coli OP50.
1. Préparation des plaques NGM de 10 cm
- Mélanger 3 g de NaCl, 2,5 g de trypticase-peptone et 17 g de gélose dans une fiole de 2 L et remplir à 1 L avec de l’eau distillée double (ddH2O). Ajouter une barre d’agitation magnétique avant l’autoclavage.
- Autoclaver le mélange pendant 45 min à 121 °C et une pression de 21 psi. Laisser refroidir le mélange à 50 °C au bain-marie.
- En utilisant des techniques aseptiques, ajouter les solutions stériles suivantes : 1 mL de 1 M CaCl2· 2H2O, 1 mL de 1 M MgSO4· 7H2O, 1 mL de 5 mg/mL de cholestérol dissous dans de l’éthanol à 100 % (réchauffé à température ambiante) et 25 mL de 1 M KH2PO4 (pH = 6,0). Mélanger à l’aide d’une plaque magnétique à mélanger. Le mélange peut être effectué pendant 1 min à 700 TR/min.
- Verser jusqu’à ce que le mélange remplisse toute la plaque de 10 cm. Vous pouvez également utiliser une pipette sérologique graduée pour ajouter environ 20 mL de mélange par plaque.
- Laisser sécher les plaques pendant 24 h à température ambiante avant l’ensemencement avec des bactéries ou conserver les plaques ordinaires à 4 °C après séchage.
REMARQUE: Tous les composants du support sont manipulés à l’aide de techniques aseptiques. Les étapes 1.3 à 1.4 doivent être effectuées dans une hotte à écoulement laminaire.
2. Préparation de la gélose NGM avec FUDR dans des plaques à 24 puits
- Suivez les étapes 1.1 à 1.3.
- Compléter NGM avec de la 5-fluoro-2′-désoxyuridine (FUDR) et mélanger pour atteindre une concentration finale de 100 μg/mL.
REMARQUE: FUDR inhibe la réplication de l’ADN et, par conséquent, bloque la reproduction de C. elegans en ciblant la lignée germinale et l’embryogenèse, affectant finalement la durée de vie. Par conséquent, il est important de permettre aux vers de se développer pleinement en jeunes adultes avant de les transférer sur des plaques contenant du FUDR.
ATTENTION : Le FUDR est toxique et doit être manipulé conformément à la fiche de données de sécurité du fabricant. - À l’aide d’un pistolet à pipette, distribuer 1 mL de NGM-FUDR dans chaque puits.
REMARQUE: Ce processus peut être facilité par l’utilisation d’un système automatisé de coulée de plaques. - Laisser sécher la plaque pendant 24 h à température ambiante avant d’ensemencer avec des bactéries ou de stocker des plaques ordinaires à 4 °C.
3. Ensemencement des plaques: OP50 et bactéries d’essai supplémentaires
- Pour préparer une culture d’E. coli OP50 pendant la nuit, ajouter 200 μL d’aliquote bactérienne provenant d’un bouillon congelé dans une fiole d’Erlenmeyer de 500 mL contenant 250 mL de bouillon Luria frais et stérile (LB).
REMARQUE: Le volume du support dépend du nombre de plaques qui doivent être ensemencées. Pour préparer d’autres cultures bactériennes, inoculez un tube de culture de 16 mL contenant 5 mL de milieu de croissance avec des bactéries provenant de stocks congelés à l’aide d’une pointe de micropipette stérile. - Incuber toute la nuit dans un incubateur à 37 °C, en agitant à 220 tr/min (rotations par minute).
REMARQUE: Utilisez des flacons stérilisés avec au moins deux fois le volume de travail du support et scellez avec du papier d’aluminium autoclavé. Effectuer l’étape d’inoculation et la distribution bactérienne en utilisant des techniques aseptiques. - Distribuer 1 à 2 mL de la culture D’E. coli OP50 pendant la nuit au centre de chaque plaque NGM de 10 cm. Cette culture n’a pas besoin d’être répandue autour de la plaque NGM.
- Laissez les plaques sécher à température ambiante avant l’utilisation ou le stockage.
REMARQUE: Les plaques ensemencées avec des couvercles peuvent être placées dans une hotte avec un flux d’air pour faciliter le séchage.
4. Culture et ensemencement des plaques: plaques à 24 puits
- Préparez une culture de nuit des souches bactériennes souhaitées, en respectant les instructions de culture trouvées aux étapes 3.1-3.2.
- Transférer 200 μL de chaque culture bactérienne dans chaque puits d’une plaque de 24 puits contenant de la gélose NGM. Un volume de 200 μL de bactéries couvrira toute la zone de gélose, maximisant la quantité de nourriture pour s’assurer que les vers n’éviteront pas la pelouse bactérienne.
- Laissez les plaques fissurées ouvertes dans une armoire de sécurité biologique (ESB) pour faciliter le séchage. Vérifiez périodiquement les plaques pour éviter une déshydratation excessive et changez l’orientation de la plaque pour favoriser une circulation d’air et un séchage uniformes. Les plaques doivent sécher dans les 5 h.
REMARQUE : Tout travail avec des bactéries de niveau de sécurité biologique 2 doit être effectué dans des ESB certifiés et approuvé par le Comité de biosécurité institutionnel.
5. Synchronisation de l’âge
REMARQUE: Toutes les étapes doivent être effectuées à l’aide de techniques aseptiques appropriées (c.-à-d. travailler près d’une flamme ou à l’intérieur d’un ESB).
- Laver les hermaphrodites gravides de 10 cm sur des plaques OP50 à l’aide d’une solution de M9 stérilisée par filtre (5,8 g de Na2HPO4· 7H2O, 3,0 g de KH2PO4, 5 g de NaCl, 0,25 g de MgSO4·7H2O, dans 1 L de ddH2O).
- Pipette M9 solution sur la plaque plusieurs fois en utilisant une pipette sérologique stérile en verre ou en plastique pour soulever les vers de la pelouse bactérienne.
- Recueillir la suspension du ver et transférer la solution dans un tube conique en polystyrène de 15 mL.
- Centrifuger à 270 x g, température ambiante (RT, ~23 °C), pendant 2 min.
- Aspirer à l’aide d’une fiole piège à vide et jeter le surnageant, en laissant la pastille de ver intacte.
- Remettez en suspension la pastille dans 5 à 10 mL de M9 pour laver les vers et répétez les étapes 5.2-5.3 deux fois.
- Ajouter 5 mL de solution de blanchiment à 20% (8,25 mL de ddH2O, 3,75 mL de NaOH 1M, 3,0 mL d’eau de Javel non germicide) au tube et inverser continuellement pour dissoudre les vers. Les vers sont prêts à centrifuger une fois qu’ils sont presque complètement dissous.
REMARQUE: Les temps de blanchiment et le volume de la solution de blanchiment dépendront de la taille de l’échantillon. Le sur-blanchiment et le sous-blanchiment sont des erreurs courantes. En tant que tel, ce processus nécessite généralement une optimisation pour déterminer quand l’échantillon est prêt pour la centrifugation. - Centrifuger pendant 2 min à 423 x g et jeter le surnageant.
- Ajouter 10 mL de M9 stérile pour remettre en suspension la pastille d’œuf.
- Centrifuger le tube pendant 2 min à 423 x g pour granuler les œufs. Retirez le surnageant à l’aide d’une fiole d’aspirateur.
- Répétez les étapes 5.6 à 5.6.1.
- Remettre en suspension la pastille d’œuf dans 5 mL de M9 stérile et la placer sur un nutateur pendant la nuit à la température souhaitée.
REMARQUE: Les larves de L1 synchronisées avec l’âge seront prêtes à être transférées sur des plaques le lendemain.
6. Préparation du ver après la synchronisation de l’âge
- Centrifuger les vers synchronisés avec l’âge à 270 x g pendant 3 min à RT (~23 °C).
- Aspirez le surnageant dans un environnement propre, comme une hotte d’écoulement ou à côté d’un brûleur Bunsen. Laissez environ 200 μL du surnageant et remettez en suspension les vers.
- À l’aide d’une micropipette, transférer la suspension de vis sans fin concentrée sur des plaques NGM de 10 cm qui ont déjà été ensemencées avec OP50.
REMARQUE: Chaque assiette peut supporter 1 500 vers sans manquer de nourriture; cependant, cette concentration peut nécessiter un ajustement en fonction de la densité de la pelouse bactérienne et de la température de croissance. Il est recommandé d’utiliser plusieurs plaques pour empêcher les vers de mourir de faim. Il est important de noter que ces plaques ne doivent pas contenir de FUDR. - Laissez sécher les assiettes; puis inverser et conserver à 25 °C pendant 48 h.
REMARQUE: Selon l’état de l’écran, les vers de l’étape 6.1 peuvent être placés directement sur des plaques d’essai (contenant des bactéries, des médicaments ou des composés d’essai). Si la condition d’essai souhaitée affecte le développement, les vers doivent être cultivés sur NGM contenant OP50 jusqu’à ce que les jeunes adultes (~ 48 h) avant de les exposer à des conditions d’essai. - Après 48 h d’incubation, lavez les vers des plaques avec une solution stérile de M9 et placez-les dans des tubes coniques.
REMARQUE: Les vers adultes couleront au fond du tube. L’heure exacte variera en fonction du nombre de vers récupérés sur des plaques de 10 cm. Dans ces conditions, les vers s’installent dans les 10 minutes. - Effectuer une inspection visuelle pour déterminer la durée du temps de décantation de manière à ce que les œufs résiduels ou les larves écloses soient enlevés.
- Ajouter 10 mL supplémentaires de M9 pour rincer les bactéries résiduelles des corps de vers.
- Centrifuger les vers pendant 2-3 min à 270 x g à 23 °C.
- Effectuez l’étape de lavage 3 fois de plus. Pour de meilleurs résultats, laissez environ 1 à 1,5 mL de solution de M9 dans le tube après le lavage final.
- Transférer 10 μL de la suspension de ver sur une lame de verre et compter le nombre de vers.
- Ajustez la densité du ver à environ 150 vers par 10 μL de M9. La concentration de vers en suspension peut être ajustée en retirant ou en ajoutant une solution de M9 après centrifugation.
- Confirmer que la concentration souhaitée a été établie en faisant la moyenne des comptes de plusieurs gouttes différentes. Il est recommandé de compter en moyenne à partir d’au moins trois gouttes.
- À l’aide de techniques aseptiques, transférer 10 μL de la suspension de vers contenant environ 150 vers dans chaque puits de la plaque d’essai.
- Inspectez les puits au microscope pour vous assurer que chacun a un nombre suffisant de vers. Des vers supplémentaires peuvent être ajoutés avant l’incubation.
- Laisser sécher les plaques pendant environ 10 min; puis inverser et transférer dans un incubateur à 25 °C pendant 72 h.
REMARQUE: La période d’incubation finale peut être ajustée pour répondre aux besoins de l’expérience. Le temps d’incubation de 72 h est suffisant pour soutenir la croissance de 150 animaux se nourrissant de bactéries de 200 μL dans une plaque de 24 puits à 25 °C.
7. Préparation des vers pour l’imagerie
- Pour faciliter une décantation plus efficace et minimiser la perte d’échantillons, immobilisez les vers avant le lavage pour éviter la baignade.
REMARQUE: Si vous travaillez avec peu d’échantillons, cela peut être réalisé par l’exposition au lévamisole (100 μM). Cependant, s’ils travaillent avec un grand nombre d’échantillons, les vers peuvent être immobilisés par congélation. De plus, une congélation prolongée (18-24 h) empêchera le développement ultérieur des agrégats polyQ pendant la préparation.- Placez les plaques multipuits à -20 °C pendant 15 à 20 minutes ou jusqu’à ce que les vers ne bougent plus.
- Retirez les échantillons du congélateur et laissez-les reposer pendant 5 min.
- À l’aide d’une micropipette, ajouter 1 mL de M9, refroidi à 4 °C, à un puits d’intérêt, appuyer et enfoncer à plusieurs reprises le piston 4 à 6 fois pour laver les vers dans chaque puits.
REMARQUE: Parfois, les vers peuvent coller aux pointes de micropipette. En tant que tel, différents embouts doivent être utilisés entre chaque puits pour éviter le mélange des vers. - Transférer la suspension de vis sans fin dans un tube de microcentrifugation et laisser les vers couler au fond.
- Aspirer et jeter le surnageant. Lavez l’échantillon trois fois au total.
- Une fois que les vers se sont déposés au fond pendant le lavage final, aspirer 500 μL de surnageant, en laissant 500 μL pour remettre en suspension les vers.
- Transférer la suspension de ver restante dans une nouvelle plaque à fond plat de 24 puits et la placer dans un congélateur à -20 °C pendant 48 h.
REMARQUE: La congélation des vers réduit la fluorescence de fond et permet une meilleure visualisation des agrégats.
8. Imagerie
- Retirez les plaques du congélateur et laissez décongeler, essuyez l’excès de condensation et retirez le couvercle avant l’imagerie.
REMARQUE: Les détails de la capture d’image varient en fonction de l’équipement et du logiciel utilisés. Les protocoles de cette section ne devraient servir que de guide, et des modifications sont à prévoir. Il est également nécessaire de capturer des images au format de fichier tiff. - Pendant la capture d’image, utilisez les paramètres de microscope suivants : Temps d’exposition, 500 ms ; Grossissement 40x avec adaptateur de caméra 0,63x (25,2x), intensité GFP réglée sur 100%.
REMARQUE: Diverses configurations et systèmes de microscope peuvent acquérir des images différentes de celles fournies dans la section des résultats. Pour obtenir une description plus universelle des images requises, des détails plus objectifs concernant les images sont fournis. Les agrégats doivent avoir un diamètre compris entre 1,0 et 10,0 pixels avec une intensité fluorescente de 0,10 à 1,0 (unités arbitraires, échelle 0-1) pour être correctement identifiés par le CellProfiler. Le fond fluorescent de ces images agrégées est généralement inférieur au seuil de 0,10. Pour les images en champ clair, l’intensité du ver varie de 0,7 à 1,0 avec une intensité d’arrière-plan de 0,1 à 0,2. La longueur moyenne des vers dans les images en champ clair varie de 250 pixels à 350 pixels de la tête à la queue. - Ajustez les commandes de lumière transmise jusqu’à ce que les vers apparaissent brillamment éclairés par rapport à l’arrière-plan sombre. Évitez la surexposition; cela augmentera la taille du ver.
- Il peut être nécessaire de modifier les positions des vers dans le puits pour éviter un agglutination excessive. Dispersez les vers agglomérés à l’aide d’une pointe de pipette.
- Définissez le canal sur GFP pour établir un plan focal pour les deux images.
REMARQUE: Il est essentiel que le plan focal soit déterminé dans le canal GFP. Tout changement de mise au point effectué lors de la capture d’images en champ clair entraînera un désalignement des agrégats et des vers lors de l’analyse de l’image. - Capturez une image en champ clair et prenez immédiatement l’image fluorescente correspondante sans perturber la plaque.
- Exécutez des images de test dans le pipeline pour déterminer les valeurs d’intensité et de taille des objets de l’image conformément à la « NOTE » après l’étape 8.2.
REMARQUE: CellProfiler peut fournir toutes les informations nécessaires concernant l’intensité et la longueur des objets avant l’analyse. - Pour évaluer les images, téléchargez d’abord CellProfiler17. Ouvrez le logiciel et faites glisser et déposez les images d’intérêt dans la zone Images.
- Cliquez sur les images dans la liste des fichiers pour les ouvrir.
- Dans le coin supérieur gauche de l’écran se trouvent plusieurs icônes; utilisez-les pour mesurer la taille des objets ou agrandir une région spécifique. Sélectionnez la loupe pour mettre en évidence une région d’intérêt.
- Sélectionnez l’icône en forme de flèche pour mesurer la longueur des vers et des agrégats.
- Passez le curseur de la souris sur l’objet souhaité pour déterminer sa valeur d’intensité qui peut être vue sur la partie inférieure de l’écran.
REMARQUE: Étant donné que toutes les images sont prises en niveaux de gris, toutes les valeurs de pixels rouges, bleus et verts seront identiques.
9. Analyse d’images
- Pour utiliser le pipeline d’analyse d’image CellProfiler, nommez correctement les paires d’images. Utilisez le format suivant: P1_A01_S1_C1, où P1 fait référence à la plaque spécifique et à sa désignation numérique respective; « A » fait référence à la ligne et « 01 » à la colonne; « S » fait référence à une paire d’images spécifique pour un seul puits ; « C » fait référence au canal, où « 1 » est utilisé pour désigner les images en champ clair et « 2 » pour les images fluorescentes.
- Téléchargez CellProfiler (version 4.1.3 ou supérieure) sur le site officiel17.
- Téléchargez le pipeline (fichier supplémentaire 1). Téléchargez le pipeline (pipeline) dans CellProfiler en sélectionnant Fichier > Importer > pipeline à partir d’un fichier.
REMARQUE : Les modules 2 et 3 ont été désactivés car ils ont été jugés inutiles. Leur fonction peut être restaurée si l’analyse d’images ne permet pas une séparation et une identification satisfaisantes des vers; toutefois, une modification du pipeline est nécessaire. - Pour incorporer ces modules, cochez les cases situées à gauche de chaque module. Renommez l’image binaire d’entrée du module « UntangleWorms » en image de sortie du module « convertobjectstoimage ».
- Un message d’erreur peut apparaître lors de l’utilisation initiale liée au module 2. Si cela se produit, procédez à l’analyse.
- Téléchargez un ensemble d’entraînement utilisé pour identifier les vers dans le module « UntangleWorms ». Cet ensemble de formation se trouve dans le fichier supplémentaire 2.
- Sélectionnez le module UntangleWorms pour ouvrir ses paramètres.
- Identifiez le nom du fichier de l’ensemble de formation et sélectionnez l’icône de téléchargement du fichier.
- Télécharger le fichier supplémentaire 2 (ensemble de formation).
- Téléchargez des images en sélectionnant le module Images dans le coin supérieur gauche. Faites glisser et déposez des images correctement nommées comme décrit à l’étape 9.1.
- Avant d’analyser les images, sélectionnez le dossier de sortie souhaité qui sera utilisé pour stocker les résultats.
- Cliquez sur le bouton Paramètres de sortie situé dans le coin inférieur gauche du programme.
- Sélectionnez l’icône de dossier à droite de Sortie par défaut pour choisir l’emplacement de sortie souhaité.
- Sélectionnez l’icône Analyser les images pour commencer l’analyse des images.
- Si l’analyse prend trop de temps à terminer et est bloquée dans le traitement d’une seule image, cela est probablement dû à des problèmes liés à l’acquisition d’images. Si cela se produit, abandonnez l’exécution et procédez à l’identification des images non traitées en triant le dossier de sortie et en notant quels noms d’image sont introuvables.
REMARQUE : les images peuvent nécessiter un traitement supplémentaire pour supprimer les artefacts volumineux ou intensément éclairés qui ne peuvent pas être traités par le pipeline. Dans certains cas, il peut être nécessaire d’exclure ces images de l’analyse. - Après une analyse complète, le logiciel organisera les résultats dans une feuille de calcul Excel contenant des vers individuels (colonne N) et leur nombre respectif d’agrégats (colonne K).
REMARQUE : Une exécution réussie produira une feuille de calcul Excel contenant le nombre d’agrégats par ver identifié pour chaque puits. Ces données peuvent être manipulées d’une manière déterminée par l’expérimentateur. - Téléchargez l’organisateur de métadonnées (interface utilisateur graphique) à partir du fichier supplémentaire 3 (Windows) ou du fichier supplémentaire 4 (Mac) pour organiser facilement les données du fichier CSV de sortie de CellProfiler.
REMARQUE: Ce logiciel n’a pas de licence officielle et ne sera pas automatiquement ouvert après le téléchargement. - Suivez les étapes 9.14 à 9.17 si vous utilisez Windows OS 64 bits. Le logiciel ne fonctionnera pas sur Windows 32 bits. Suivez les étapes 9.18 à 9.20 si vous utilisez Mac OS. Les étapes 9.21 à 9.22 sont les mêmes pour les deux systèmes d’exploitation.
- Pour le système d’exploitation Windows, localisez le fichier téléchargé et extrayez-le à l’emplacement souhaité.
- Localisez et ouvrez le dossier extrait nommé gui_windowsOS_64x et lancez l’application en cliquant sur l’icône de l’application « gui ».
- Une invite peut s’ouvrir pour demander l’autorisation d’exécuter. Sélectionnez Plus d’informations, puis cliquez sur Faire confiance quand même.
- L’organisateur de métadonnées est prêt à glisser-déposer les fichiers CSV de sortie CellProfiler. Passez à l’étape 9.21.
- Pour Mac OS, recherchez le fichier téléchargé et ouvrez gui_macOS_64x.zip. Cette étape devrait extraire automatiquement tous les fichiers.
- Ouvrez le dossier extrait trouvé dans « Téléchargements ».
- Faites un clic droit sur l’application « gui » et sélectionnez Ouvrir. Une invite apparaîtra demandant la permission d’ouvrir en raison de l’absence d’une licence officielle. Sélectionnez Ouvrir et passez à l’étape 9.21.
- Cliquez sur Télécharger vos fichiers ici ou faites glisser et déposez les fichiers CSV CellProfiler souhaités.
- Cliquez sur le bouton Organiser , qui amènera l’utilisateur à un nouvel écran avec un bouton Télécharger les fichiers . Cliquez sur le bouton et sélectionnez l’emplacement souhaité pour enregistrer le fichier de sortie. Le fichier de sortie apparaîtra comme le nom de fichier d’origine avec l’extension « _organized » ajoutée au nom de fichier.
Résultats
Décrit ici est un flux de travail C. elegans qui comprend des protocoles de culture, d’acquisition d’images et de traitement qui permettent d’évaluer l’agrégation polyQ en présence de diverses bactéries en utilisant un format de plaque à 24 puits comme plate-forme de culture et d’imagerie (Figure 1). Ce protocole peut être ajusté pour étudier l’effet des bactéries, des conditions spécifiques, des petites molécules, des médicaments ou des manipulations génomiques sur la protéostasie de l’hôte. La méthode décrite a été optimisée à l’aide de vers qui expriment de manière constitutive le polyQ intestinal fusionné à une protéine fluorescente jaune (vha6p::p olyQ44::YFP); cependant, d’autres modèles qui rendent compte de la protéostase dans les muscles ou les neurones peuvent également être utilisés avec une optimisation supplémentaire. Par exemple, des expériences préliminaires démontrent l’application de ces méthodes dans la quantification d’agrégats de protéines dans d’autres tissus tels que le polyQ musculaire (figure supplémentaire 1). Toutefois, une modification du pipeline sera nécessaire pour ajuster correctement la taille et la luminosité agrégées, comme mentionné à la section 8 NOTE.

Figure 1 : Représentation visuelle du flux de travail. Les principales étapes du protocole comprennent cinq étapes distinctes : la préparation des vers et la synchronisation de l’âge (étapes 1 à 5), la colonisation intestinale et le traitement des vers (étape 6), la préparation des échantillons pour l’imagerie (étape 7), l’acquisition d’images (étape 8) et le traitement des images (étape 9). Les « Sections » du protocole sont référencées en tant que « Étapes » dans la figure. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Les premières expériences d’optimisation ont révélé diverses difficultés associées à la surpopulation due à un grand nombre de progénitures, entraînant un épuisement plus rapide des aliments. La supplémentation en FUDR dans les plaques NGM décrite à la section 2 a résolu ce problème (Figure 2). De plus, en présence de FUDR, les vers nourris avec diverses bactéries avaient une taille corporelle plus constante, ce qui permettait une détection plus uniforme et plus précise des vers.

Figure 2 : L’utilisation de FUDR améliore la qualité de l’image en réduisant la progéniture. Les plaques supplémentées en FUDR éliminent la descendance de C. elegans par rapport aux vers cultivés sur des plaques NGM non contrôlées FUDR ensemencées avec E. coli OP50. Les images ont été acquises à un grossissement de 25,2x (grossissement de 40x avec un adaptateur de caméra de 0,63x). Barres d’échelle = 500 μm. Veuillez cliquer ici pour voir une version plus grande de cette figure.
La fluorescence de fond a contribué à la détection de faux positifs des agrégats polyQ. Pour réduire ce signal de fluorescence dans le tractus intestinal et améliorer la détection automatisée des agrégats, il était nécessaire de congeler les vers avant l’imagerie. La congélation des vers à -20 °C pendant 18 à 48 h a considérablement amélioré la détection des agrégats polyQ en éliminant la fluorescence de fond (Figure 3A). L’œil humain est capable de différencier les agrégats et la fluorescence de fond; par conséquent, le comptage manuel avant et après le gel est le même (Figure 3B). Cependant, le comptage automatisé n’est pas aussi précis, mais le gel des comptages automatisés a considérablement amélioré avec une précision comparable au comptage manuel (Figure 3B).

Figure 3 : La congélation améliore la détection des agrégats. (A) Images fluorescentes de C. elegans exprimant le polyQ44::YFP intestinal avant et après la congélation. Les insertions représentent des images en gros plan de la zone sélectionnée. Barres d’échelle = 500 μm. (B) Nombre moyen d’agrégats par intestin chez les vers colonisés par P. aeruginosa MPAO1 avant et après la congélation à l’aide d’une quantification manuelle ou automatisée (pipeline) des agrégats. Les données représentent deux réplicats biologiques (n = 60-109). La signification statistique a été calculée à l’aide du test t de Student (**** p < 0,0001). Les barres d’erreur représentent l’erreur-type de la moyenne (SEM). Veuillez cliquer ici pour voir une version agrandie de cette figure.
L’éclairage inversé en champ lumineux a été utilisé pour détecter l’ensemble du canal C. elegans (Figure 4A) et GFP pour imager les agrégats polyQ44::YFP (Figure 4B). La détection des vers, le démêlage et la quantification des agrégats pour chaque ver ont été effectués en appliquant un pipeline de traitement d’image CellProfiler optimisé (fichier supplémentaire 1), ce qui permet d’obtenir le nombre d’agrégats par ver individuel (Figure 4C-D). Pour tester la faisabilité de cette approche et la précision de la détection et de la quantification automatisées des agrégats, des vers exprimant le polyQ44::YFP spécifique à l’intestin ont été cultivés et préparés pour l’imagerie conformément aux protocoles établis (sections 1-7). Le nombre d’agrégats par ver a été évalué à l’aide du pipeline automatisé (sections 8 à 9) ou du comptage manuel. Chaque expérience a été réalisée dans trois essais indépendants utilisant 90 à 571 vers par condition. Alors que le nombre moyen d’agrégats par intestin obtenus avec deux essais n’avait pas de différence significative, les vers du troisième essai avaient significativement moins d’agrégats lorsqu’ils étaient quantifiés à l’aide de l’approche automatisée (Figure 5A). Le nombre moyen d’agrégats des trois essais a donné lieu à un léger mais significativement moins d’agrégats lorsque la quantification a été effectuée à l’aide du pipeline CellProfiler (sections 8-9) (figure 5B). Néanmoins, la différence entre les deux approches était minime, ce qui indique que la méthode automatisée peut être appliquée à des écrans à grande échelle.
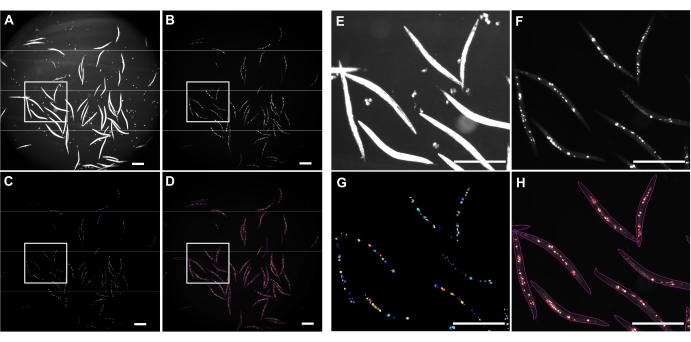
Figure 4 : Détection d’agrégats à l’aide de CellProfiler. (A) Image en champ lumineux utilisée pour identifier les corps de vers. (B) Image fluorescente originale acquise à l’aide du canal GFP et utilisée pour identifier et quantifier un nombre total d’agrégats intestinaux polyQ44::YFP. (C) Agrégats identifiés à l’aide de CellProfiler. (D) Un nombre total d’agrégats identifiés superposés à l’image fluorescente originale avec des contours de vers et d’agrégats. La capture et le traitement de l’image ont été effectués à l’aide des paramètres décrits dans les sections 8 à 9. Les panneaux E-H représentent des images en gros plan des régions soulignées correspondantes dans les images A-D. Les images ont été acquises à un grossissement de 25,2x (grossissement de 40x avec un adaptateur de caméra de 0,63x). Barres d’échelle = 500 μm. Veuillez cliquer ici pour voir une version plus grande de cette figure.
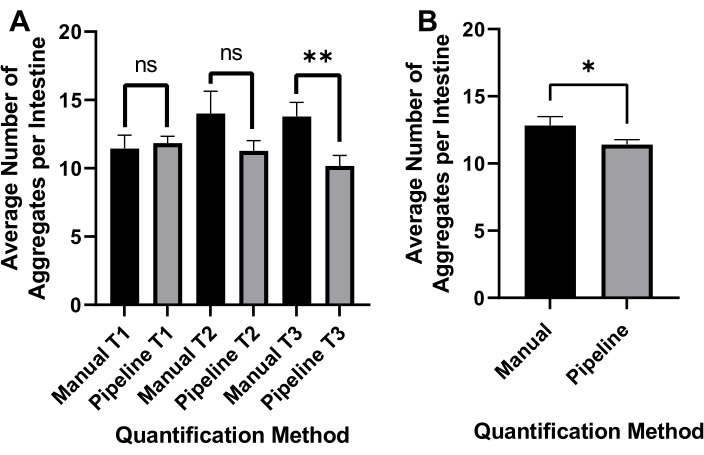
Figure 5 : Efficacité de la quantification automatisée des agrégats. (A) Nombre total moyen par intestin chez les vers colonisés par E. coli OP50 témoin à l’aide du comptage manuel (manuel) et de la quantification automatisée basée sur CellProfiler (Pipeline). Les résultats représentent des données analysées dans trois essais distincts (T1-T3) (n = 90-571). (B) Le nombre moyen d’agrégats par intestin a été obtenu à l’aide d’une quantification manuelle ou automatisée (pipeline) des agrégats. La signification statistique a été calculée à l’aide du test t de Student (* p < 0,05, ** p < 0,01). Les barres d’erreur représentent le SEM. Veuillez cliquer ici pour l’agrandir.
Pour évaluer la reproductibilité des résultats chez différents expérimentateurs, des images provenant d’une plaque contenant six puits de vers nourris avec Pseudomonas aeruginosa MPAO1 ou Escherichia coli OP50 ont été acquises par trois individus, dont deux n’avaient aucune expérience préalable de l’imagerie de vers utilisant ces protocoles. Les images recueillies dans chaque puits contenaient entre 30 et 115 vers détectés. Une différence non significative dans l’agrégation a été détectée chez les vers provenant des mêmes puits qui ont été imagés par les trois expérimentateurs. Bien que le nombre moyen d’agrégats par intestin soit resté très constant entre les trois expérimentateurs pour les vers nourris au MPAO1 et au OP50, il y avait des différences statistiquement significatives dans le nombre moyen d’agrégats, mais seulement chez les vers colonisés par le MPAO1 (figure supplémentaire 2). Ces résultats mettent en évidence la reproductibilité des résultats même entre expérimentateurs inexpérimentés.
Pour s’assurer que la reproductibilité de la quantification agrégée n’est pas influencée de manière significative par la position du ver, un ensemble de 15 vers a été sélectionné et imagé 15 fois distinctes après agitation entre chaque capture d’image à l’aide d’une pointe de pipette. Des images d’agrégats chez des vers nourris avec E. coli OP50 et P. aeruginosa MPAO1 ont été recueillies et analysées à l’aide de CellProfiler. Le nombre moyen d’agrégats de chacun de ces différents ensembles d’images était légèrement mais non significativement différent, ce qui corroborait davantage la reproductibilité de cette approche (figure supplémentaire 3).
Il a été démontré que la colonisation de l’intestin de C. elegans par des agents pathogènes entériques à Gram négatif perturbe la protéostasie dans les tissus, P. aeruginosa étant parmi les inducteurs les plus puissants de l’agrégation polyQ9. Pour déterminer si ces protocoles optimisés permettront de détecter et de quantifier avec succès l’amélioration de l’agrégation médiée par P. aeruginosa, les vers exprimant la polyQ intestinale ont été colonisés par E. coli OP50 (bactérie témoin) et P. aeruginosa MPAO1, rubriques 1 à 8 ont été menées. Les images acquises ont été analysées à l’aide de CellProfiler (section 9 , Fichier supplémentaire 1). Les résultats de la quantification automatisée montrent une augmentation significative du nombre d’agrégats induits par P. aeruginosa MPAO1, ce qui entraîne systématiquement une double amélioration par rapport aux vers nourris avec E. coli OP50 témoin (Figure 6).

Figure 6 : Nombre moyen d’agrégats par intestin chez les vers colonisés par E. coli OP50 et P. aeruginosa MPAO1 témoins. Le nombre d’agrégats par intestin a été évalué à l’aide de CellProfiler (rubriques 8-9). Les données sont représentées comme le nombre moyen d’agrégats par intestin chez les vers colonisés par OP50 (n = 1068) et MPAO1 (n = 1557). La signification statistique a été calculée à l’aide du test t de Student (**** p < 0,0001). Les barres d’erreur représentent le SEM. Veuillez cliquer ici pour l’agrandir.
Le pipeline optimisé a été conçu pour prendre en charge les écrans à grande échelle pour les conditions qui affectent la protéostasie. Pour tester la faisabilité de cette approche dans le dépistage de leur effet sur la protéostasie de l’hôte dans de grandes bibliothèques de bactéries, le pipeline décrit ici a été utilisé (sections 1 à 9) pour tester l’effet de 90 souches mutantes knock-out de gènes non essentielles de P. aeruginosa sur l’agrégation polyQ18. Cet écran pilote fait partie d’un projet plus vaste conçu pour dépister toutes les souches mutantes non essentielles de P. aeruginosa pour leur capacité à influencer la protéostasie de l’hôte. Sur 90 souches bactériennes testées, la colonisation de l’intestin de C. elegans avec un candidat a montré une diminution significative du nombre d’agrégats (Figure 7). Des expériences de suivi visant à évaluer la sensibilité de ce test ont été réalisées à l’aide de comptages d’agrégats manuels à partir d’une sélection aléatoire de six mutants de P. aeruginosa qui différaient de manière non significative du contrôle MPAO1. Ces expériences ont été réalisées à l’aide de plaques NGM plus traditionnelles de 6 cm, transférant des vers sur des souches d’essai comme L1 pour récapituler les méthodes précédemment établies9. Les expériences de confirmation par comptage manuel ont révélé qu’aucun des mutants, y compris celui qui a significativement diminué le nombre d’agrégats (Figure 7), n’a affecté l’agrégation polyQ (Figure supplémentaire 4). De plus, les changements subtils dans l’agrégation observés dans l’écran des 90 souches mutantes n’ont pas été détectés parmi les comptages manuels de candidates sélectionnées, ce qui indique que de tels changements pourraient survenir en raison de la variabilité biologique et expérimentale, telle qu’une faible valeur n. Collectivement, les résultats indiquent que, bien que notre méthode puisse détecter de manière fiable des changements importants, les changements subtils seront probablement manqués et tous les candidats potentiels devront être confirmés individuellement.

Figure 7 : Nombre d’agrégats par intestin dans un échantillon représentatif de vers colonisés par 92 souches bactériennes. Les données sont représentées comme le nombre moyen d’agrégats par intestin normalisé à celui des vers colonisés par MPAO1. Les lignes pointillées représentent le nombre moyen d’agrégats dans les vers colonisés par le contrôle MPAO1 (en haut, cercle ouvert) et OP50 (en bas, carré ouvert). Les symboles solides représentent 90 souches mutantes knock-out distinctes de P. aeruginosa MPAO1. Le nombre moyen d’agrégats par ver entre les vers colonisés par MPAO1 et un seul mutant était statistiquement significatif. Les cercles gris représentent des échantillons qui ont été confirmés manuellement (figure supplémentaire 4). La signification statistique a été calculée à l’aide d’une analyse unidirectionnelle de la variance (ANOVA) suivie d’une comparaison multiple du test post-hoc de Dunnett (** p < 0,01, **** p < 0,0001). Les barres d’erreur représentent le SEM. Veuillez cliquer ici pour l’agrandir.
Pour gérer la grande quantité de données générées par CellProfiler, une interface utilisateur graphique (GUI) a été développée pour automatiser le traitement et l’organisation des données (Figure 8). L’interface graphique a été développée à l’aide de Tkinter, une boîte à outils de widgets multiplateforme Python open-source. À partir des métadonnées données, l’application extrait le nombre d’agrégats (colonne K) de chaque puits (colonne J) présents dans une plaque. Une bibliothèque de traitement de données Python appelée « Pandas » a été utilisée pour effectuer le processus susmentionné. L’application GUI fournit une prise en charge du glisser-déposer permettant aux utilisateurs de télécharger des fichiers de données. Les données de chaque fichier sont stockées sous la forme d’une structure tabulaire bidimensionnelle appelée trame de données. Une paire de dictionnaires vide est initialisée pour chaque puits unique trouvé dans le bloc de données. Ensuite, les agrégats distincts trouvés dans chaque puits sont comptés et ajoutés à leurs paires de dictionnaires respectives. La colonne avec des données inférieures est complétée par des chaînes à valeur vide pour s’assurer que chaque colonne est de taille uniforme. Enfin, la structure est convertie en un bloc de données qui est exporté sous la forme d’une feuille de calcul dans le répertoire spécifié par l’utilisateur.

Figure 8 : Interface utilisateur graphique. Cliquez ici pour afficher une version agrandie de cette figure.
Figure supplémentaire 1 : Détection d’agrégats polyQ spécifiques aux muscles. Les vers exprimant des polyQ35::YFP spécifiques aux muscles ont été plaqués en L1 et cultivés sur OP50 pendant 48 h. Une fois que les vers se sont développés en jeunes adultes, ils ont été transférés sur des plaques NGM à 24 puits, complétés par 100 μg / mL FUDR et ensemencés avec MPAO1 pendant 72 heures supplémentaires avant l’imagerie. (A) Image en champ clair utilisée pour identifier les corps de vers. (B) Image fluorescente originale acquise à l’aide du canal GFP. (C) Agrégats identifiés à l’aide de CellProfiler. (D) Un nombre total d’agrégats identifiés superposés à l’image fluorescente originale avec des contours de vers et d’agrégats. La capture et le traitement de l’image ont été effectués à l’aide des paramètres décrits dans les sections 8 à 9. Les panneaux E-H représentent des images en gros plan des régions soulignées correspondantes dans les images A-D. Barres d’échelle = 500 μm. Veuillez cliquer ici pour télécharger ce fichier.
Figure supplémentaire 2 : Reproductibilité de la quantification agrégée entre différents expérimentateurs. Nombre moyen d’agrégats quantifiés à l’aide de CellProfiler pour six puits de vers colonisés par P. aeruginosa MPAO1 (barres noires) et six puits de vers colonisés par E. coli OP50 (barres grises). Chaque puits a été photographié par trois expérimentateurs (AVS, DMC, RDH). Les données sont représentées comme le nombre moyen d’agrégats par intestin (n = 30-115). La signification statistique a été calculée à l’aide de l’ANOVA unidirectionnelle suivie du test de comparaisons multiples de Tukey (* p < 0,05, ** p < 0,01). Les barres d’erreur représentent le SEM. Veuillez cliquer ici pour télécharger ce fichier.
Figure supplémentaire 3 : L’effet de la position du ver sur la reproductibilité de la quantification agrégée. Nombre total moyen par intestin chez les vers colonisés par E. coli OP50 (barres grises) et P. aeruginosa MPAO1 (barres noires). Les résultats représentent le nombre moyen d’agrégats par intestin (15≥n≥12) quantifié à l’aide de CellProfiler. La position des vers dans les puits a été modifiée par agitation entre chaque acquisition. Aucune différence statistiquement significative n’a été observée dans l’un ou l’autre groupe. La signification statistique a été calculée à l’aide d’une ANOVA unidirectionnelle suivie du test de comparaisons multiples de Tukey. Les barres d’erreur représentent le SEM. Veuillez cliquer ici pour télécharger ce fichier.
Figure supplémentaire 4 : Confirmation de l’écran pilote avec comptage manuel. Le nombre moyen d’agrégats par intestin a été quantifié manuellement. Les données représentent les profils d’agrégation de vers colonisés par six mutants KNOCK-out MPAO1 (cercles gris Figure 7) comparés aux témoins MPAO1 et OP50 de type sauvage (n = 30). La signification statistique a été calculée à l’aide de l’ANOVA unidirectionnelle suivie d’une comparaison multiple du test post-hoc de Dunnett (**** p < 0,0001). Les barres d’erreur représentent le SEM. Veuillez cliquer ici pour télécharger ce fichier.
Figure supplémentaire 5 : L’effet du FUDR sur l’agrégation des polyQ intestinaux. Les données sont représentées comme le nombre moyen d’agrégats polyQ44::YFP par intestin (n = 20). Les vers ont été transférés sur des plaques témoins (pas de FUDR) ou contenant du FUDR (100 μg/mL) après 48 h de croissance à 25 °C sur E. coli OP50. Les comptages manuels ont été collectés après 48 heures supplémentaires. La signification statistique a été calculée à l’aide du test t de Student (ns = non significatif). Les barres d’erreur représentent le SEM. Veuillez cliquer ici pour télécharger ce fichier.
Fichier supplémentaire 1 : Pipeline de protéostasie. Pipeline d’analyse d’image téléchargeable à utiliser dans CellProfiler. Les instructions d’application se trouvent à la section 9. Veuillez cliquer ici pour télécharger ce fichier.
Fichier supplémentaire 2: Jeu d’entraînement démêlant le ver. Fichier à télécharger dans le module « UntangleWorms ». Cet ensemble d’entraînement particulier est spécifique aux vers utilisés dans l’approche initiale. Les modifications de la taille et de la forme des vers modifieront la précision et la qualité de l’identification. Il peut être nécessaire de créer un fichier de formation plus personnalisé. Les instructions pour créer un nouvel ensemble de formation peuvent être trouvées sur le site officiel de CellProfiler17. Veuillez cliquer ici pour télécharger ce fichier.
Fichier supplémentaire 3: Interface utilisateur graphique pour le système d’exploitation Windows. gui_windowsOS_64x.zip. Veuillez cliquer ici pour télécharger ce fichier.
Fichier supplémentaire 4: Interface utilisateur graphique pour le système d’exploitation Mac. gui_MacOS_64x.zip. Veuillez cliquer ici pour télécharger ce fichier.
Discussion
Le protocole décrit décrit les procédures de culture, d’imagerie et de traitement d’images de C. elegans qui intègre CellProfiler, un logiciel d’analyse d’images open source. Les résultats représentatifs démontrent la reproductibilité, la réduction des biais et l’évolutivité. Cette procédure normalisée améliorera les stratégies de dépistage utilisées dans les grandes bibliothèques bactériennes, génomiques ou pharmaceutiques. Bien qu’il existe d’autres méthodes automatisées de détection d’objets de C. elegans , la technique décrite offre un pipeline standardisé à haut débit qui intègre la culture, l’acquisition d’images et l’analyse.
Plusieurs variantes de la culture des vers ont dû être testées pour optimiser le protocole décrit ici. Initialement, les vers ont été transférés à l’échantillon de bactéries immédiatement après la synchronisation de l’âge (stade L1). Cependant, une telle approche a abouti à une population de vers de tailles variables, même parmi les vers dans le même puits. C. elegans est connu pour l’évitement des agents pathogènes19, ce qui aurait pu contribuer à la variabilité observée de la taille et finalement affecter la détection des vers d’imagerie en aval, en particulier. Pour éliminer cette variabilité, toute la zone NGM de chaque puits a été recouverte de bactéries d’essai. De plus, les vers ont été nourris avec E. coli OP50 et ont pu se développer complètement en jeunes adultes pendant 48 h à 25 °C. Permettre aux vers d’atteindre l’âge adulte sous E. coli OP50 avant de les transférer sur des bactéries d’essai a entraîné une taille corporelle plus uniforme. De plus, le surpeuplement et l’épuisement rapide des aliments par la progéniture ont été éliminés en complétant la gélose NGM par FUDR. La mise en œuvre de FUDR a supprimé la progéniture et amélioré l’identification automatisée des vers, qui a été obscurcie par le mélange de la progéniture avec la population parentale. Cependant, il est important d’être prudent et d’utiliser des contrôles appropriés lors de l’utilisation de FUDR, car le composé est connu pour affecter la protéostasie de C. elegans et la durée de vie20,21. Dans les conditions décrites dans ce protocole, le FUDR n’a pas affecté l’agrégation de polyQ intestinale (figure supplémentaire 5); par conséquent, son utilisation était appropriée et bénéfique pour la méthode décrite.
La congélation des échantillons avant l’imagerie s’est avérée être une étape critique dans l’utilisation réussie du pipeline. Les dénombrements agrégés avant la congélation étaient significativement plus élevés que les comptes manuels (figure 3B). Le maintien des vers à -20 °C pendant 18 à 48 h avant l’imagerie a permis de réduire la fluorescence de fond et, en fin de compte, d’améliorer la détection des agrégats (figure 3A). Les effets de la congélation sur la détection des agrégats n’ont été étudiés que pour le polyQ et ne devraient pas être généralisés à d’autres modèles sans une étude plus approfondie de ces effets.
Bien que toutes les conditions soient maintenues les mêmes, il a été observé que le nombre moyen d’agrégats par ver pouvait varier d’une série à l’autre, tandis que le rapport entre le nombre d’agrégats chez les animaux colonisés par OP50 et MPAO1 restait constant (figure 6, figure supplémentaire 2, figure supplémentaire 5). Par conséquent, il est essentiel de toujours inclure le contrôle E. coli OP50, ou tout contrôle de référence approprié supplémentaire, dans chaque exécution. Une telle variabilité du nombre d’agrégats entre les expériences pourrait être influencée par les conditions environnementales (température, humidité)22,23 ou le fond génétique8. En fait, il a été observé qu’après une culture prolongée, la fluorescence intestinale diminuait considérablement ou était complètement perdue, ce qui nécessitait la décongélation d’une nouvelle souche à partir de stocks congelés. La diminution observée de la fluorescence pourrait être le résultat de changements génétiques qui suppriment les transgènes toxiques, tels que ceux exprimant le polyQ. Néanmoins, la reproductibilité exceptionnelle des résultats observés entre différents expérimentateurs (figure supplémentaire 2), entre répliques biologiques (figure 5) et au sein d’un même échantillon (figure supplémentaire 3) souligne la force de cette approche.
De nombreux rapports ont utilisé le polyQ intestinal pour étudier la protéostasie 9,11,12,13,24,25. Cependant, une comparaison directe entre les résultats ne peut être faite en raison de la variabilité entre les approches expérimentales et les méthodes de lecture. Néanmoins, quelques résultats de données publiées précédemment sont récapitulés par la quantification automatisée décrite ici, y compris l’induction bactérienne de l’agrégation 9,13 et un nombre comparable d’agrégats11. Collectivement, le pipeline décrit offre un outil précieux pour étudier la protéostasie.
La méthode décrite ici présente certains défis inhérents. Par exemple, il faut suffisamment de temps pour maîtriser tous les composants de ce protocole, ce qui est particulièrement vrai pour la section 8 du protocole, qui nécessite une connaissance approfondie du test pour déterminer si les images acquises sont appropriées pour l’analyse du pipeline. Des écarts par rapport aux paramètres d’acquisition d’images utilisés dans ce protocole sont possibles ; toutefois, une modification des paramètres et de l’ensemble d’entraînement des vers sera probablement nécessaire. Ce pipeline peut distinguer les agrégats de différentes tailles et ceux qui se touchent, ce qui limite le « mélange » des agrégats et augmente finalement la sensibilité de détection. Cependant, des problèmes peuvent survenir lorsque l’on tente d’identifier de grands agrégats qui dépassent la plage de taille acceptée, car l’élargissement du seuil de taille supérieur peut entraîner des erreurs causées par une mauvaise identification, telles que l’incapacité de différencier les agrégats qui se touchent. Un équilibre entre la précision, la taille et l’intensité doit être trouvé avant l’analyse de l’image. L’efficacité de l’identification des agrégats pourrait être encore améliorée en intégrant l’apprentissage automatique pour créer un réseau neuronal capable d’améliorer la détection des agrégats. De telles améliorations sont actuellement à l’étude et aideront grandement à résoudre les problèmes actuels tels que la détection d’agrégats qui se trouvent sur différents plans focaux ou qui ont des formes anormales.
Une faiblesse notable de la méthode décrite est la variabilité des comptages agrégés automatisés, car ils ne sont pas toujours récapitulés par des comptages manuels chez les vers nourris de différentes souches bactériennes. Par exemple, d’après les dénombrements automatisés, les vers nourris avec le mutant P. aeruginosa 53 (M53) avaient significativement moins d’agrégats que la souche de type sauvage (MPAO1) (figure 7); toutefois, la confirmation de la réponse n’a révélé aucune différence significative (figure supplémentaire 4). En général, les dépistages de drogues à haut débit ont un taux élevé de détection de faux positifs, et la méthode décrite ne fait pas exception26. Ainsi, c’est une partie essentielle du protocole de confirmer tous les résultats potentiels.
Bien que ce protocole ait été optimisé pour s’adapter à une stratégie de dépistage visant à identifier les bactéries qui affectent la protéostasie de l’hôte, chaque étape peut être modifiée pour tester l’effet des bibliothèques d’ARNi génomiques, des petites molécules ou d’autres conditions. Des modifications supplémentaires peuvent être apportées à chaque étape pour répondre aux exigences d’une stratégie de dépistage spécifique. De plus, cette technique offre un niveau de flexibilité qui permet d’optimiser chaque étape en fonction d’un modèle spécifique. Par exemple, cette approche peut être étendue à l’agrégation polyQ dans d’autres tissus ou à l’extraction d’autres caractéristiques détectées dans des images telles que la surveillance de l’expression des gènes à l’aide de rapporteurs fluorescents inductibles (par exemple, les gènes de choc thermique), l’évaluation de la localisation subcellulaire des protéines (par exemple, la localisation nucléaire de DAF-16), l’étude de l’agrégation dans d’autres modèles de maladies (Aβ1-42, α-synucléine, TDP-43, etc.) ou l’évaluation de phénotypes physiologiques, comme la taille du ver.
Déclarations de divulgation
Les auteurs ont déclaré qu’il n’y avait pas de conflits d’intérêts.
Remerciements
Ce travail a été soutenu par les National Institutes of Health (1RO3AG069056-01) et l’Infectious Diseases Society of America qui finance DMC. Les bailleurs de fonds n’ont joué aucun rôle dans la conception de l’étude, la collecte et l’analyse des données, la décision de publier ou la préparation du manuscrit. Nous remercions les membres du Czyz Lab d’avoir relu le manuscrit. Les figurines de dessins animés ont été générées à l’aide de la licence payante BioRender.
matériels
| Name | Company | Catalog Number | Comments |
| 1.7 mL Microtubes | Olympus Plastics | Cat#24-282 | Microcentrifuge tubes |
| 10 mL Serological Pipettes | GenClone | Cat#12-104 | Plastic pipettes |
| 15 mL Centrifuge Tubes, Racked | Olympus Plastics | Cat#28-101 | Conical tubes |
| 5-Fluoro-2′-deoxyuridine | Fisher Scientific | D2235100MG | FUDR |
| Accu-jet Pro Pipette Controller | Genesee Scientific | 91-600RB | Pipette gun |
| Agar | Fisher Scientific | Cat#BP1423-2 | Granulated agar |
| BioRender | BioRender | Graphical figure generator | |
| Bleach (Regular) | Clorox | Bar# 044600324111, Splash-less Bleach | 4.5% sodium hypochlorite |
| C. elegans: AM140: rmIs132 [unc-54p::q35::yfp] | Morimoto Lab (Northwestern University) | AM140, Q35::YFP | Muscle polyQ |
| C. elegans: AM738: rmIs297[vha-6p::q44::yfp; rol-6(su1006)] | Morimoto Lab (Northwestern University) | AM738, Q44::YFP | Intestinal polyQ |
| CaCl2·2H2O | Fisher Scientific | Cat#C79-500 | Calcium chloride dihydrate |
| CellProfiler | Broad Institute | Image analysis software | |
| Cholesterol | Fisher Scientific | Cat#ICN10138201 | Cholesterol |
| Circulating Water Bath Head | Lauda | 26LE | Lauda E 100 |
| CoolLED pE300lite 365 dir mount STEREO | CoolLED | 8114931 | Fluorescent light control and emitter |
| 16 mL Culture Tubes | Olympus Plastics | 21-129 | Culture tubes, 17 mm x 100 mm |
| Escherichia coli OP50 | Caenorhabditis Genetics Center | OP50 | E. coli control strain |
| Ethanol, 200 Proof | Decon Labs, Inc. | 2701 | |
| Eyepiece 10x/23B, adjustable, 3d gen | Leica Microsystems | 10450910 | Eyepiece set |
| Filter Set ET GFP - MZ10F | Leica Microsystems | 10450588 | Filter cube |
| GraphPad Prism v9.2.0 | GraphPad Software, Inc. | Statistical analysis tool | |
| Heratherm Incubator IMP180 | Thermo | 51031562 | Refrigerated incubator |
| Innova 4000 | New Brunswick Scientific | M1192-0000 | Shaking incubator |
| K5 Camera | Leica Microsystems | 11547112 | Stereomicroscope camera |
| KH2P04 | Fisher Scientific | Cat#P285-3 | Potassium phosphate monobasic |
| LAS X Imaging Software | Leica Microsystems | Microscope imaging software | |
| Leica MZ10 F Optics Carrier | Leica Microsystems | 10450103 | Stereomicroscope |
| Levamisole | Fisher Scientific | Cat#0215522805 | Levamisole hydrochloride |
| Luria Broth (Lennox) | Apex Bioresearch Products | Cat#11-125 | LB |
| Magnetic Stir Plate | Fisher Scientific | 11-100-49S | Stir plate |
| MgSO4·7H2O | Alfa Aesar | A14491 | Magnesium sulfate heptahydrate |
| Microscope Slides | Premiere | 8205 | Single frosted microscope slides |
| Na2HPO4·7H2O | Fisher Scientific | Cat#S373-500 | Sodium phosphate dibasic heptahydrate |
| NaCl | Fisher Scientific | Cat#S671-500 | Sodium chloride |
| NaOH | Fisher Scientific | Cat#S318-3 | Sodium hydroxide pellets |
| Objective Achromat, f = 100 mm | Leica Microsystems | 10411597 | Objective microscope lens |
| Petri Dishes | Genesee Scientific | Cat#32-107G | 100 mm x 15 mm |
| Pseudomonas aeruginosa Mutant Library | Manoil Lab (University of Washington) | P. aeruginosa mutant library | |
| Suspension Culture Plate 24-Well, Flat Bottom | Olympus Plastics | 25-102 | Used for worm growth and imaging |
| Trinocular Tube 100% M-series | Leica Microsystems | 10450043 | |
| Trypticase Peptone | ThermoFisher, Difco | Cat#211921 | |
| TX-400 Rotor | Thermo Scientific | Cat#75003181 | Swing bucket rotor |
| Vacuum Driven Filter System | GenClone | Cat#25-227 | 500 mL, PES Membrane, .22 µm |
| Video Objective with C-Mount | Leica Microsystems | 10447367 | 0.63x camera adapter tube |
Références
- Soto, C. Unfolding the role of protein misfolding in neurodegenerative diseases. Nature Reviews Neuroscience. 4 (1), 49-60 (2003).
- Fang, P., Kazmi, S. A., Jameson, K. G., Hsiao, E. Y. The microbiome as a modifier of neurodegenerative disease risk. Cell Host & Microbe. 28 (2), 201-222 (2020).
- Kundu, P., Blacher, E., Elinav, E., Pettersson, S. Our gut microbiome: The evolving inner self. Cell. 171 (7), 1481-1493 (2017).
- Sherwin, E., Dinan, T. G., Cryan, J. F. Recent developments in understanding the role of the gut microbiota in brain health and disease. Annals of the New York Academy of Sciences. 1420 (1), 5-25 (2018).
- Mondal, S., et al. Large-scale microfluidics providing high-resolution and high-throughput screening of Caenorhabditis elegans poly-glutamine aggregation model. Nature Communications. 7 (1), 13023(2016).
- Guisbert, E., Czyz, D. M., Richter, K., McMullen, P. D., Morimoto, R. I. Identification of a tissue-selective heat shock response regulatory network. PLoS Genetics. 9 (4), 1003466(2013).
- Silva, M. C., et al. A genetic screening strategy identifies novel regulators of the proteostasis network. PLoS Genetics. 7 (12), 1002438(2011).
- Nollen, E. A., et al. Genome-wide RNA interference screen identifies previously undescribed regulators of polyglutamine aggregation. Proceedings of the National Academy of Sciences of the United States of America. 101 (17), 6403-6408 (2004).
- Walker, A. C., et al. Colonization of the Caenorhabditis elegans gut with human enteric bacterial pathogens leads to proteostasis disruption that is rescued by butyrate. PLOS Pathogens. 17 (5), 1009510(2021).
- Goya, M. E., et al. Probiotic Bacillus subtilis protects against α-Synuclein aggregation in C. elegans. Cell Reports. 30 (2), 367-380 (2020).
- Kumsta, C., Chang, J. T., Schmalz, J., Hansen, M. Hormetic heat stress and HSF-1 induce autophagy to improve survival and proteostasis in C. elegans. Nature Communications. 8, 14337(2017).
- Prahlad, V., Morimoto, R. I. Neuronal circuitry regulates the response of Caenorhabditis elegans to misfolded proteins. Proceedings of the National Academy of Sciences of the United States of America. 108, 14204-14209 (2011).
- Mohri-Shiomi, A., Garsin, D. A. Insulin signaling and the heat shock response modulate protein homeostasis in the Caenorhabditis elegans intestine during infection. Journal of Biological Chemistry. 283 (1), 194-201 (2008).
- Hakim, A., et al. WorMachine: machine learning-based phenotypic analysis tool for worms. BMC Biology. 16 (1), 8(2018).
- Wählby, C., et al. An image analysis toolbox for high-throughput C. elegans assays. Nature Methods. 9 (7), 714-716 (2012).
- Jones, T. R., et al. CellProfiler Analyst: data exploration and analysis software for complex image-based screens. BMC Bioinformatics. 9 (1), 482(2008).
- CellProfiler: an open-source image analysis software. , Available from: http://www.cellprofiler.org (2021).
- Held, K., Ramage, E., Jacobs, M., Gallagher, L., Manoil, C. Sequence-verified two-allele transposon mutant library for Pseudomona aeruginosa PA01. Journal of Bacteriology. 194 (23), 6387-6389 (2012).
- Schulenburg, H., Ewbank, J. J. The genetics of pathogen avoidance in Caenorhabditis elegans. Molecular Microbiology. 66 (3), 563-570 (2007).
- Feldman, N., Kosolapov, L., Ben-Zvi, A. Fluorodeoxyuridine improves Caenorhabditis elegans proteostasis independent of reproduction onset. PLoS One. 9 (1), 85964(2014).
- Van Raamsdonk, J. M., Hekimi, S. FUdR causes a twofold increase in the lifespan of the mitochondrial mutant gas-1. Mechanisms of Ageing and Development. 132 (10), 519-521 (2011).
- Haldimann, P., Muriset, M., Vígh, L., Goloubinoff, P. The novel hydroxylamine derivative ng-094 suppresses polyglutamine protein toxicity in Caenorhabditis elegans. Journal of Biological Chemistry. 286 (21), 18784-18794 (2011).
- Shinn-Thomas, J. H. Wrapping culture plates with Parafilm M® increases Caenorhabditis elegans growth. BMC Research Notes. 12 (1), (2019).
- Alexander-Floyd, J., et al. Unexpected cell type-dependent effects of autophagy on polyglutamine aggregation revealed by natural genetic variation in C. elegans. BMC Biology. 18 (1), 18(2020).
- Moronetti Mazzeo, L. E., Dersh, D., Boccitto, M., Kalb, R. G., Lamitina, T. Stress and aging induce distinct polyQ protein aggregation states. Proceedings of the National Academy of Sciences of the United States of America. 109 (26), 10587-10592 (2012).
- Sink, R., Gobec, S., Pecar, S., Zega, A. False positives in the early stages of drug discovery. Current Medicinal Chemistry. 17 (34), 4231-4255 (2010).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon