Method Article
参照されていない太平洋カキからの仮想配列cDNAライブラリの生成のための収束戦略
要約
我々は、参照されていない太平洋カキ標本からのRNAサンプルを使用する方法の戦略を説明し、公に利用可能なゲノムデータと比較して遺伝物質を評価し、事実上配列されたcDNAライブラリーを生成する。
要約
新しい細胞株の開発やゲノムシーケンシングプロジェクトの開発など、以前は主要な実験で使用されていた参照種の生物材料へのアクセスは、多くの場合、さらなる研究や第三者に提供することが困難である。サンプルの消費的な性質。現在、アジア、オーストラリア、北米の太平洋沿岸に広く分布しているが、個々の太平洋カキの標本は遺伝的に非常に多様であり、遺伝子ライブラリーの出発材料として直接は適していない。本稿では、地域の水産物市場から得られた参照されていない太平洋カキの標本を用いてcDNAライブラリーを生成する方法を示す。これらのライブラリーを一般に入手可能なカキゲノムと比較し、最も近い関連ライブラリーをミトコンドリア参照遺伝子サイトクロムCオキシダーゼサブユニットI(COX1)およびNADHデヒドロゲナーゼ(ND)を用いて選択した。生成されたcDNAライブラリーの適合性は、UDP-グルクロン酸脱水素酵素(UGD)とUDP-キシロース合成酵素(UXS)をコードする2つの遺伝子のクローニングと発現によっても実証され、UDP-xyloseから生合成を担当する。UDPブドウ糖。
概要
生きた参照生物材料の取得は、長い配達時間、起業家の推論、または国固有の関税規制のために困難な場合があります。代替として、必要な生体材料は、表現非的に同一の標本からも採取されてもよい。しかし、これらのサンプルは遺伝子型のレベルで大きく異なる可能性があるため、同じ種のデジタル保存された参照ゲノムとの比較は、新たに供給された材料の非互換性のためにしばしば困難または無駄にレンダリングされます。既存のDNA増幅方法。個々のサンプルの高度に保存された遺伝子のシーケンシングは、cDNAライブラリーの品質評価のための参照遺伝子として頻繁に使用される保存されたミトコンドリア遺伝子など、種1を同定するための広く使用され、強力なツールです2 ,3,4,5,6.本明細書提示法の根本的な根拠は、参照ゲノムの対応する配列と比較して個々の匿名カキサンプルにおけるミトコンドリア遺伝子配列の高い保存性が、他の遺伝子も示し得ることを示している。核DNA7に対するミトコンドリアDNA進化の一般的に速い速度を考えると、発散のレベルが低く、単に公的に使用するだけで、科学的および産業的に関連する幅広い遺伝子の増幅と分離を可能にする参照として使用可能なシーケンス データ。
本明細書記載法の全体的な目標は、カキ遺伝子のクローニングのためのテンプレートDNAとして使用することができる事実上配列されたカキcDNAライブラリーを生成するための最適化されたワークフローを提示することです。仮想シーケンシングでは、デノボゲノムシーケンシングが回避されます。代わりに、既知のデジタル保存された参照シーケンスは、最終的にライブラリを構成する(または既存のライブラリに追加される)cDNAの生産にプライマーを利用または設計するために直接使用されます。目的は、収束cDNAライブラリーを生成することです, 生成されたcDNA配列と参照配列の間の類似性は、低から高い発散にランク付けできることを意味します.サイトクロムCオキシダーゼサブユニット1(COX1)とNADHデヒドロゲナーゼ(ND)を参照遺伝子として使用する主な利点は、これらのミトコンドリア遺伝子の高い保存性のために、地理的に切断されたカキの標本をプロファイリングできることです。これらの確立されたマーカーとのアプローチを証明した後、我々は、糖ヌクレオチド生合成に関与し、産業関連である可能性がある2つの酵素候補にその応用を実証する8,9,10.太平洋カキのバイオテクノロジーの可能性はまだ未開拓です。したがって、実質的に配列されたcDNAライブラリーを調製するためのこの収束方法は、この関連する生物学的材料からcDNAを生成したい非専門家研究者にも適していると考えています。
プロトコル
注: 回路図の概要を図1に示します。
1. サンプルコレクション
- カキの標本を入手する。収穫後、輸送中に氷の上にカキを保管し、購入後4-7日以内に実験室での使用と処理の前に。
注:このプロトコルのために、カキは南京の中華人民共和国卸売市場(寧徳、福建、中国、梁陽江、江蘇省、中国)、青島のハイジ水製品会社(青島、山東省、中国)、Juchengから購入されました。ヤンタイの水生製品会社(中国山東省山東省山台市出身)と青島の甚西水産物会社(中国山東省青島市出身)。
2. グアニジニウムチオシアネートフェノール抽出によるRNA単離
- カキ組織サンプルの調製
- 殺菌メスで各カキ標本のおおよその幾何学的中心から約100mgの均質な軟部組織を切り取り、サンプルを液体窒素に移す。
- カキの残りの部分を-80°Cで1時間凍結して安楽死させ、生体廃棄物として廃棄する。
- フラッシュ冷凍カキ組織を、50mLの液体窒素で満たされたモルタル(200mL)の微粉末に粉砕します。
- 各標本の凍結組織の75mgを無菌1.5 mL遠心管に計量し、グアニジウムチオシアネートフェノール抽出試薬の1 mLと混合する。15分間4°Cで14,000 x gでサンプルを遠心分離します。
- 新しい1.5 mL遠心管に上清を移し、クロロホルムの200 μLを追加し、混合物が乳白色になるまで10〜15sの渦ミキサーを使用して完全に混合します。
- 4°Cで14,000 x gの遠心分離機を15分間、200 μLピペットで慎重に上水層を新しい1.5mL遠心管に入れ、200 μLピペットで慎重に移します。
- イソプロピルアルコールの500 μLを追加し、反転によって穏やかにサンプルを混合し、その後、氷の上に20分間サンプルを残します。4°Cで14,000 x gで8分間遠心分離機を使用し、上清を除去します。
- 各ペレットを75%EtOHの1mLで再ステーペンし、遠心分離機を4°Cで14,000 x gで5分間5分間取り除きます。
- 手順 2.5 を 1 回繰り返します。ペレットを室温で6分間乾燥させます。長い間乾燥しないでください。さもなければ、次の工程でRNAペレットを溶解することは困難な場合がある。
- 乾燥したRNAペレットを25μLのDEPC(ジエチルピロ炭酸塩)処理水に溶解し、チューブを氷の上に置きます。24時間以内にRNAサンプルを使用してください。
3. 逆転転写によるcDNAライブラリー生成
- 各RNAサンプルについて、10μLピペットを用いた市販の逆転写システムを用いて反応混合物を調出す:MgCl2溶液の4μL、10倍反応バッファーの2μL、dNTP溶液の2μL、RNASe阻害剤の0.5μL、AMVリバースの0.7 μLを添加トランスクリプトーゼ、オリゴ(dT)15プライマーの0.5 μL、抽出されたRNAサンプルの1μL、およびH2 Oの9.3 μLを300μL PCRチューブに入れた。
- PCRサーモサイクラーで混合物を42°Cで60分間インキュベートし、その後5分間95°Cに温度を上げます。
- 生成されたcDNAライブラリを-20 °Cで最大12ヶ月間保存します。
4. ミトコンドリア遺伝子の増幅・精製
- 10 μLピペットを使用してPCR混合物を調作します。高忠実度DNAポリメラーゼの0.25 μL(1.25 U)、dNTP溶液の2μL(各dNTPの2.5mM)、COX1またはNDフォワードプライマーの0.5 μL(100 μM)、対応するCOX1またはNDリバースプライマー(100μM)の0.5 μL、1μLのcDNAライブラリを追加、5xバッファ溶液の5μLと蒸留H2 Oの16μLを300 μL PCRチューブに入れます。
- 次のパラメータを使用してPCR増幅を行う:95°C(持続時間5分)での初期脱率ステップの後、55°C(30s)でのアニーリングステップからなる35個のPCR反応サイクル、72°C(2分)での伸長ステップ、および95°C(30s)での脱彩化ステップ、72 °Cで5分間の1つの確定伸長ステップを実行します。
- 得られたPCR産物の品質をアガロースゲル電気泳動で検証するために、PCR製品の5μLを使用する。増幅されたCOX1またはND遺伝子を、それぞれ759または748塩基対で単一バンドとして観察する。
- PCRの残りの部分をPCRクリーンアップキットで浄化します。
- 100 μLの溶液'PCR-A'(高濃度のカオトロピック塩11を含むDNA結合バッファー)を試料に添加する。内容をミックスするために簡単に渦。
- 精製カラムを2mL遠心管に入れます。4.4.1の反応混合物をピペット。列に移動します。室温で1分間14,000 x gの遠心分離機。
- 遠心管から濾液を捨てます。カラムを2mL遠心管に戻し、700μLの溶液「W2」をカラムに加え、室温で1分間14,000×gの遠心分離機を加えます('W2'は残留カオトロピックを除去するための高濃度のエタノールを含む洗浄液です)精製カラムからの塩)。
- 濾液を捨て、2 mL遠心管にカラムを戻します。カラムに400μLの溶液「W2」を加え、室温で1分間14,000xgの遠心分離機を追加します。
- 金属ブロックヒーターで脱イオン水の予熱1mLを65°Cに予熱する。カラムを新しい1.5 mL遠心チューブに移します。65°Cのピペット25μLの高温予熱脱イオン水を白柱膜の中心に。膜を室温で1分間浸します。
- 室温で1分間14,000 x gの遠心分離機を使用し、カラムを廃棄します。
- 精製したPCR製品を-20°Cで最大12ヶ月間保存してください。
5. ミトコンドリア遺伝子シーケンシングと比較
- 関連するCOX1またはNDフォワードプライマーをシーケンシングプライマーとして使用するサンガーシーケンシングのステップ4.5から精製されたPCRサンプルを送信します。必要に応じて、COX1 または ND リバース プライマーを双方向シーケンスに使用することもできます。
- シーケンシング結果を取得した後、NCBIヌクレオチドBLASTオンラインツール(blast.ncbi.nlm.nih.gov)を用いて、太平洋カキ参照株(NCBI分類法ID:29159)のゲノム配列と配列を比較します(図2および図3)。 ).
6. 遺伝子MgUGDおよびMgUXSのクローニングのためのcDNAライブラリーの適用
- MgUGDおよびMgUXS遺伝子をPCRによるフォワードプライマーとリバースプライマーを使用して、MgUGDおよびMgUXS遺伝子を増幅および精製します。4.4に。
- 消化バッファーの2μL(10x濃縮)と脱イオン水の6μLを1.5mL遠心管に移します。精製されたMgUGDまたはMgUXS PCR産物の10 μLを、Nde IおよびXho I(各1μL、20U)の制限エンドヌクレアーゼと共に加加えます。37 °Cで3時間インキュベートします。
- 浸染めpET-30aベクターを準備する:pET-30aベクターの500 ngを新しい1.5 mL遠心管に移し、脱イオン水を使用して体積を16 μLに上げます。消化バッファーの2 μL(10x濃縮)を、Nde IおよびXho I(各1μL、20U)と共に加えて加える。
- 3時間37°Cで混合物をインキュベートした後、アルカリホスファターゼ(1U)の1 μLを加え、さらに1時間37°Cでインキュベートする。アルカリホスファターゼを予熱した金属ブロックヒーターで10分間75°Cで加熱して不活性化します。
- 消化されたMgUGDまたはMgUXS DNA産物の4 μLを新鮮な1.5 mL遠心管に移し、消化されたpET-30aベクターの4 μLを加えます。ライゲーションバッファー(10x)の1 μL、T4リガーゼ(3U)の1μLを加え、反応混合物を22°Cで3時間インキュベートします。
- ライゲーション製品を用いてエレクトロポレーションにより、電気有能な大腸菌マッハ1管轄細胞を変換します。50 μg/mLカナマイシンを含むLB寒天プレート上に形質転換細胞を広げます。細胞を37°Cで16時間インキュベートする。
- プラスミド特異的T7プロモーターおよびターミネータプライマーを用いてサンガーシーケンシングによって所望の挿入のためのコロニーを確認する。検証済みの細菌クローンからプラスミドを調作します。
7. MgUGDおよびMgUXSの発現および活性試験
- MgUGDおよびMgUXS遺伝子を有するプラスミドを有する大腸菌BL21(DE3)を形質転換し、50μg/mLカナマイシンを含むLB寒天プレート上に形質転換細胞を広げる。細胞を37°Cで16時間インキュベートする。
- 一晩50 μg/mLカナマイシンで5mL LB培地で単一のコロニーを栽培する。培養量を400mL LB培地に移し、600nm(OD600)の波長で光学密度が約0.5度の吸収に達するまで、37°Cの温度で200rpmで連続的に振る。
- インキュベーション温度を20°Cに下げ、イソプロピルβ-D-チオガラクピラノシド(濃度1M)の400μLを加えます。組換えタンパク質の発現を3時間誘導する。
- 4°Cで15分間4,500 x gの遠心分離による細胞の収穫。10mLのリシスバッファー(100mM NaCl、50 mMトリス/HCl、1%トリトンX-100、1mMフェニルメチルスルホニルフッ化物(PMSF)、pH 8.0)でペレットを中断する。
- 20分間の超音波処理によって細胞を破壊する(4°Cで15sの20 μm振幅で40オン/オフサイクル)。20分間4°Cで14,000 x gの遠心分離機を採取し、活動試験のために上清を集める。
- UDP-グルコースの2 μL、NAD+(10mM)、MgCl2の4μL(10mM)、トリス/HClバッファーの2μL(500mM、pH 7.5)、および新しいHonの6μLで細胞液質の2μLをインキュベートすることによりMgUGDの活性アッセイを行います。 mL遠心管と37°Cで30分間インキュベートします。
- UDP-グルクロン酸の2μL(10mM)、トリス/HClバッファーの2μL(500mM、pH 7.5)、および新しい1.5mL遠心分離管で脱イオン化H2 Oの14 μLを30°Cで3μLで細胞液質の2μLをインキュベートしてMgUXSの活性アッセイを行い、30°Cでiイオン化H2 Oを行う。
- 各混合物に20 μLのメタノールと40 μLのクロロホルムを加えて、ステップ7.5および7.6で反応をクエンチします。サンプル混合物を渦状にした後、4°Cで6分間14,000 x gで遠心分離し、各チューブの上部水層を収集します。
- MALDI-TOF質量分析法を用いて、m/z範囲500~700のマイナスイオン化モードで反応産物を分析する。2,5-ジヒドロキシ安息香酸サンプルマトリックスの1 μL(50%水性アセトニトリルで1%w/V)とサンプル混合物の1 μLを混合します。UDP-グルクロロン酸とUDP-キシロースの予想m/z値をそれぞれ579と535で観察します(図4)。
結果
図1は、太平洋カキ個体由来の収束cDNAライブラリーの記載された調製方法の概略的概要を示す。図2は、参照材料のCOX1およびND遺伝子配列から高い発散性を有する遠く関連するカキ標本のCOX1およびND遺伝子の配列を示す。図3は、参照材料のCOX1およびND遺伝子配列からの発散が低い密接に関連するカキ標本のCOX1およびND遺伝子の配列を示す。図4は、産業的に関連する遺伝子MgUGDおよびMgUXSをクローニングするためのcDNAライブラリーの正常な適用を示す。
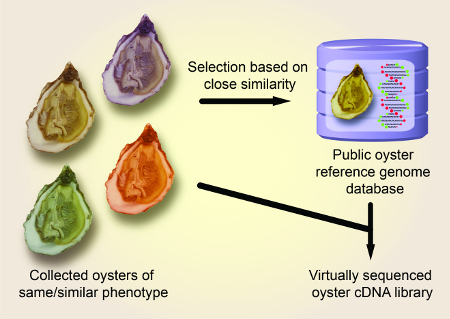
図 1: 説明された分析方法の概略図の分子同定太平洋カキ標本参照遺伝子としてCOX1とNDを用いる。この図のより大きなバージョンを表示するには、ここをクリックしてください。

図 2: 参照からのCOX1およびND遺伝子配列と比較した高度発散標本のCOX1およびND遺伝子配列の配列位置合わせ太平洋カキひずみ。この図のより大きなバージョンを表示するには、ここをクリックしてください。
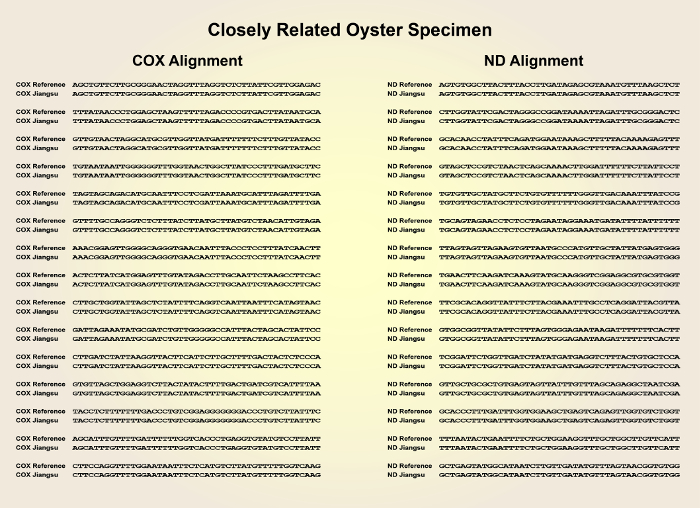
図 3: 参照からのCOX1およびND遺伝子配列と比較して密接に関連する標本のCOX1およびND遺伝子配列の配列位置合わせ太平洋カキひずみ。この図のより大きなバージョンを表示するには、ここをクリックしてください。

図 4: 概略図MgUGDおよびMgUXSの反応産物の分子クローニング、組換え発現および検出。この図のより大きなバージョンを表示するには、ここをクリックしてください。
ディスカッション
提示されたプロトコルは、公的に利用可能なカキDNAゲノムデータベースとCOX1およびND遺伝子を比較することにより、地域のシーフード市場から同様の表現型を持つ参照されていないカキ標本の遺伝的同定を可能にする。この方法の重要性は、仮想cDNAライブラリーの評価に必要なPCR反応が1つだけであるため、その簡略化にあります。2つの保存されたミトコンドリアCOX1およびND遺伝子は、各カキからのRNA抽出物の逆転転写によって生成されたcDNAライブラリーから増幅された。RNA単離の方法(ステップ2.1)は、液体窒素中のカキ組織を直接粉砕することによって簡素化された。各検体のCOX1およびND遺伝子を配列決定した後、配列アライメントは、一部のサンプルが基準株に高い類似性を示すことを明らかにした。最も近い相対は、COX1およびND遺伝子配列の両方の完全な同一性を示した。
この手順の最も重要な手順は、RNA 抽出ステップです。RNAの分解を最小限に抑えるためには、カキ組織の採取とRNA抽出までの時間を短縮することが不可欠です。
成功したクローニングは、最近、カキUGE遺伝子12と本明細書で、生成されたcDNAライブラリーの実用性を検証したMgUGDおよびMgUXS遺伝子13をクローニングすることによって例示され、目的とする任意の数の遺伝子のクローニングを可能にした。退化したプライマーを使用して面倒なクローン作成戦略を必要とせずに。この方法は、COX1およびND遺伝子を増幅して仮想配列cDNAライブラリーを生成する方法で、参照ゲノムの物理サンプルを持たない他の生物学的材料にも将来使用される可能性がある。利用 可能。
開示事項
著者は何も開示していない。
謝辞
この研究は、中国自然科学財団(助成番号31471703、A0201300537、31671854、J.V.とL.L.への助成番号31470435、G.Y.への助成番号31470435)、および100外国人材計画(助成番号JSB2014012.V.)によって一部支援されました。
資料
| Name | Company | Catalog Number | Comments |
| Chemicals: | |||
| 1% Triton X-100 | Solarbio | 9002-93-1 | *Alternative distributors possible |
| 2,5-Dihydroxybenzoic acid | Alfa Aesar | 490-79-9 | *Alternative distributors possible |
| Acetonitrile | Merck | 75-05-8 | *Alternative distributors possible |
| Agarose for molecular biology | Biowest Chemicals | 111860 | *Alternative distributors possible |
| Ampicilin | Solarbio | 69-52-3 | *Alternative distributors possible |
| Chloroform | Lingfeng, Shanghai | 67-66-3 | *Alternative distributors possible |
| DEPC water | Thermo Scientific | R0601 | |
| Ethanol | Jinhuada, Guangzhou | 64-17-5 | *Alternative distributors possible |
| Guanidinium thiocyanate-phenol reagent | Invitrogen | 15596018 | TRIzol reagent |
| Imidazole | Energy Chemical | 288-32-4 | *Alternative distributors possible |
| Isopropyl alcohol | Nanjing Chemical Reagent | 67-63-0 | *Alternative distributors possible |
| Isopropyl β-D-thiogalactopyranoside | Solarbio | 367-93-1 | *Alternative distributors possible |
| Kanamycin | Solarbio | 25389-94-0 | *Alternative distributors possible |
| LB Agar | Thermo Fisher | 22700025 | *Alternative distributors possible |
| LB Broth | Thermo Fisher | 10855021 | *Alternative distributors possible |
| Methanol | Jinhuada, Guangzhou | 67-56-1 | *Alternative distributors possible |
| MgCl2 hexahydrate | Xilong Huagong | 7791-18-6 | *Alternative distributors possible |
| NaCl | Xilong Huagong | 7647-14-5 | *Alternative distributors possible |
| NAD+ | Duly Biotech | 53-84-9 | *Alternative distributors possible |
| Phenyl-methylsulfonyl fluoride | Macklin | 329-98-6 | *Alternative distributors possible |
| Tris | Solarbio | 77-86-1 | *Alternative distributors possible |
| UDP-glucose | Wuhu Nuowei Chemicals | 28053-08-9 | *Alternative distributors possible |
| UDP-glucuronic acid | SIGMA | 63700-19-6 | *Alternative distributors possible |
| Tools/Instruments: | |||
| MALDI-TOF mass spectrometer | Bruker | Autoflex | *Alternative distributors possible |
| Metal block heater | Long Yang Scientific Instruments | Thermoshaker HB20 | *Alternative distributors possible |
| PCR thermocycler | Hema | 9600 | *Alternative distributors possible |
| Enzyme and Kits: | |||
| 10×Ligation buffer | Thermo Scientific | B69 | *Alternative distributors possible |
| 5×PrimeSTAR buffer | Takara | 9158A | |
| Alkaline phosphatase | ThermoFisher FastAP | EF0654 | *Alternative distributors possible |
| COX forward primer | Genscript | ATGTCAACAAATCATTTAGACATTG | |
| COX reverse primer | Genscript | ACTTGACCAAAAACATAAGACATG | |
| Cutsmart Buffer | NEB | B7204S | *Alternative distributors possible |
| dNTP mix | Invitrogen | 18427088 | |
| MgUGD forward primer | Genscript | ACATATGACCCTGTCCAAGATCTGTTGT | |
| MgUGD reverse primer | Genscript | ACTCGAGACTCTGTGAGGCGGTGGAG | |
| MgUXS forward primer | Genscript | CCATATGGCAGAATCCTCACAATCAC | |
| MgUXS reverse primer | Genscript | ACTCGAGCACATTTTTGAATTTGCAGACGT | |
| ND forward primer | Genscript | ATGAGATGGCAATTATTTTTTAAT | |
| ND reverse primer | Genscript | ATGTATTTTGGAAAAATCTCCAC | |
| PCR Cleanup Kit | AxyGen | AP-PCR-250 | *Alternative distributors possible |
| pET-30a(+) vector | Merck Millipore | 69909 |
参考文献
- Blaxter, M. L. The promise of a DNA taxonomy. Philosophical transactions of the Royal Society of London. Series B, Biological. 359 (1444), 669-679 (2004).
- Wen, J., et al. Species identification of dried shellfish (oyster, clam and mussel) products sold on the Chinese market. Food Control. 90, 199-204 (2018).
- Zhang, H., et al. Mitochondrial cob and cox1 genes and editing of the corresponding mRNAs in Dinophysis acuminata from Narragansett Bay, with special reference to the phylogenetic position of the genus Dinophysis. Applied and Environmental Microbiology. 74 (5), 1546-1554 (2007).
- Sell, J., Spirkovski, Z. Mitochondrial DNA differentiation between two forms of trout Salmo letnica, endemic to the Balkan Lake Ohrid, reflects their reproductive isolation. Molecular Ecology. 13, 3633-3644 (2004).
- Karadjian, G., et al. Highly rearranged mitochondrial genome in Nycteria parasites (Haemosporidia) from bats. Proceedings of the National Academy of Sciences of the United States of America. 113 (35), 9834-9839 (2018).
- Morga, B., et al. Identification of genes from flat oyster Ostrea edulis as suitable housekeeping genes for quantitative real time PCR. Fish and Shellfish Immunology. 29 (6), 937-945 (2010).
- Delsuc, F., et al. Molecular systematics of armadillos (Xenarthra, Dasypodidae): contribution of maximum likelihood and Bayesian analyses of mitochondrial and nuclear genes. Molecular Phylogenetics and Evolution. 28 (2), 261-265 (2005).
- Wei, S., et al. Discovery and Biochemical Characterization of UDP-Glucose Dehydrogenase from Akkermansia muciniphila. Protein & Peptide Letters. 24 (8), 735-741 (2017).
- Gu, B., et al. Discovery and Biochemical Characterization of the UDP-Xylose Biosynthesis Pathway in Sphaerobacter thermophilus. Protein & Peptide Letters. 23 (12), 1103-1110 (2016).
- Duan, X. C., et al. Functional characterization of the UDP-xylose biosynthesis pathway in Rhodothermus marinus. Applied Microbiology and Biotechnology. 99 (22), 9463-9472 (2015).
- Vogelstein, B., Gillespie, D. Preparative and analytical purification of DNA from agarose. Proceedings of the National Academy of Sciences of the United States of America. 76 (2), 615-619 (1979).
- Song, H. B., et al. UDP-glucose 4-epimerase and β-1,4-galactosyltransferase from the oyster Magallana gigas as valuable biocatalysts for the production of galactosylated products. International Journal of Molecular Sciences. 19 (6), 1600 (2018).
- Gainey, P. A., Phelps, C. F. Uridine diphosphate glucuronic acid production and utilization in various tissues actively synthesizing glycosaminoglycans. Biochemical Journal. 128 (2), 215-227 (1972).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved