Method Article
Una strategia convergente per la generazione di una libreria cDNA virtualmente in sequenza da ostriche del Pacifico senza riferimenti
In questo articolo
Riepilogo
Descriviamo una strategia su come usare campioni di RNA da campioni di ostriche del Pacifico senza riferimenti e valutiamo il materiale genetico confrontandosi con i dati del genoma pubblicamente disponibili per generare una libreria cDNA virtualmente sequenziata.
Abstract
L'accesso al materiale biologico delle specie di riferimento, che sono stati utilizzati in precedenza in esperimenti chiave come lo sviluppo di nuove linee cellulari o progetti di sequenziamento del genoma, sono spesso difficili da fornire per ulteriori studi o terzi a causa della natura consumiva dei campioni. Anche se ora ampiamente distribuiti sulle coste del Pacifico in Asia, Australia e Nord America, i singoli esemplari di ostriche del Pacifico sono geneticamente molto diversi e quindi non sono direttamente adatti come materiale di partenza per le librerie genetiche. In questo articolo, dimostriamo l'uso di campioni di ostriche del Pacifico senza riferimenti ottenuti dai mercati ittici regionali per generare librerie di cDNA. Queste librerie sono state poi confrontate con il genoma delle ostriche pubblicamente disponibili, e la libreria correlata più vicina è stata selezionata utilizzando i geni di riferimento mitocondriali Cytochrome C Oxidase I (COX1) e NADH Dehydrogenase (ND). L'idoneità della libreria cDNA generata è dimostrata anche dalla clonazione e dall'espressione di due geni che codificano gli enzimi UDP-glucuronic acid dehydrogenase (UGD) e UDP-xylose synthase (UXS), che sono responsabili della biosintesi di UDP-xylose da UDP-glucosio.
Introduzione
L'acquisizione di materiale biologico di riferimento per la vita può essere difficile a causa dei lunghi tempi di consegna, del ragionamento imprenditoriale o delle norme doganali specifiche per paese. In alternativa, il materiale biologico richiesto può essere raccolto anche da campioni fenotipicamente identici. Tuttavia, questi campioni possono variare in modo significativo a livello del genotipo, e quindi i confronti con genomi di riferimento memorizzati digitalmente della stessa specie sono spesso resi difficili o addirittura inutili a causa dell'incompatibilità del materiale appena ricavato con metodi di amplificazione del DNA esistenti. Il sequenziamento dei geni altamente conservati di singoli campioni è uno strumento ampiamente utilizzato e potente per identificare le specie1, come i geni mitocondriali conservati che vengono spesso utilizzati come geni di riferimento per la valutazione della qualità delle librerie cDNA2 ,3,4,5,6. La logica alla base del metodo qui presentato è che l'alta conservazione delle sequenze geniche mitocondriali nei singoli campioni anonimi di ostriche rispetto alle corrispondenti sequenze del genoma di riferimento indica che altri geni possono anche basso livello di divergenza, dato il tasso generalmente più veloce di evoluzione del DNA mitocondriale rispetto al DNA nucleare7, consentendo l'amplificazione e l'isolamento di un'ampia gamma di geni scientificamente e industrialmente rilevanti semplicemente utilizzando semplicemente pubblicamente dati di sequenziamento disponibili come riferimento.
L'obiettivo generale del metodo qui descritto è quello di presentare un flusso di lavoro ottimizzato per generare una libreria cDNA di ostriche virtualmente sequenziata che può essere utilizzata come DNA modello per la clonazione dei geni delle ostriche. Nel sequenziamento virtuale, il sequenziamento del genoma de novo è elusivo; invece, una sequenza di riferimento nota, memorizzata digitalmente viene utilizzata direttamente per utilizzare o progettare primer per la produzione di cDNA che alla fine comprenderanno una libreria (o essere aggiunti a uno preesistente). L'obiettivo è quello di produrre una libreria cDNA convergente, il che significa che le somiglianze tra le sequenze cDNA generate e la sequenza di riferimento possono essere classificate da bassa ad alta divergenza. Un vantaggio chiave dell'uso della sottounità 1 (COX1) e della dehydrogenassi NADH (ND) del citocro c' come geni di riferimento è che anche gli esemplari di ostriche altamente geograficamente disgiunti possono essere profilati a causa dell'elevata conservazione di questi geni mitocondriali. Dopo aver dimostrato l'approccio con questi marcatori ben consolidati, dimostriamo quindi la sua applicazione a due candidati enzimatici che sono coinvolti nella biosintesi dei nucleotidi zuccherini e possono essere di rilevanza industriale8,9, 10. Il potenziale biotecnologico dell'ostrica del Pacifico è ancora inesplorato. Pertanto, riteniamo che questo metodo convergente per la preparazione di una libreria cDNA virtualmente in sequenza sarà appropriato anche per i ricercatori non specializzati che vogliono generare cDNA da questo materiale biologico pertinente.
Protocollo
NOTA: nella Figura 1è illustrata una panoramica schematica.
1. Raccolta di campioni
- Ottenere campioni di ostriche. Conservare le ostriche sul ghiaccio durante il periodo post-raccolta, il trasporto e prima dell'uso in laboratorio e il processo entro 4-7 giorni dall'acquisto.
NOTA: Per questo protocollo, le ostriche sono state acquistate dal mercato all'ingrosso di Nanchino (originario di Ningde, Fujian, Cina e Lianyungang, Jiangsu, Cina), Haijie Aquatic Product Company di Qingdao (originario di Qingdao, Shandong, Cina), Jucheng Aquatic Product Company a Yantai (originario di Yantai, Shandong, Cina) e Jinxiu Aquatic Product Company a Qingdao (originario di Qingdao, Shandong, Cina)).
2. Isolamento dell'RNA per estrazione di tisonio-fenolo di guanidianio
- Preparazione del campione di tessuto ostrica
- Tagliare circa 100 mg di tessuto molle omogeneo dal centro geometrico approssimativo di ogni campione di ostrica con un bisturi sterilizzato e trasferire i campioni in azoto liquido.
- Eutanasia la parte rimanente delle ostriche congelandoandosi a -80 gradi centigradi per 1 h e scartando come rifiuti biologici.
- Macinare il tessuto di ostrica congelati in una polvere fine in un mortaio (200 mL) riempito con 50 mL di azoto liquido.
- Pesare 75 mg di tessuto congelato di ogni esemplare in un tubo di centrifugasterile sterile da 1,5 ml e mescolare con 1 mL del reagente di estrazione del fenidinico-fenolo. Centrifugare il campione a 14.000 x g a 4 gradi centigradi per 15 min.
- Trasferire il supernatante in un nuovo tubo di centrifuga di 1,5 mL, aggiungere 200 l di cloroformina e mescolare accuratamente utilizzando un mixer vortice per 10-15 s fino a quando la miscela diventa bianco latteo.
- Centrifuga a 14.000 x g a 4 gradi centigradi per 15 minuti, e trasferire con attenzione lo strato acquoso superiore con una pipetta da 200 luna senza disturbare l'interfase in un nuovo tubo di centrifuga di 1,5 mL.
- Aggiungere 500 l di alcool isopropile e mescolare delicatamente i campioni per inversione, quindi lasciare i campioni per 20 min sul ghiaccio. Centrifuga a 14.000 x g a 4 gradi centigradi per 8 min e rimuovere il supernatante.
- Risospendere ciascuno dei pellet in 1 mL del 75% EtOH e centrifugare a 14.000 x g a 4 gradi centigradi per 5 min. Rimuovere tutti i supernatali.
- Ripetere il passaggio 2.5 una volta. Asciugare i pellet per 6 min a temperatura ambiente. Non asciugare più a lungo; in caso contrario, potrebbe essere difficile sciogliere il pellet di RNA nella fase successiva.
- Sciogliere il pellet di RNA essiccato in 25 o L di DEPC (diethylpyrocarbonate) -trattamento e mantenere il tubo sul ghiaccio. Utilizzare campioni di RNA entro 24 h.
3. Generazione libreria cDNA per trascrizione inversa
- Per ogni campione di RNA, preparate una miscela di reazione utilizzando un sistema di trascrizione inversa commerciale utilizzando una pipetta da 10-L: Aggiungere 4 ldi di soluzione MgCl 2, 2 L di 10x Reaction Buffer, 2 L di soluzione dNTP, 0,5 l di inibitore di RNase, 0,7 l di AMV Reverse Buffer Trascrizione, 0,5 litri di Oligo(dT) 15 primer, 1 l del campione di RNA estratto e 9,3 l di H2O in un tubo PCR 300 LL.
- Incubare la miscela in un termociclista PCR per 60 min a 42 gradi centigradi, quindi aumentare la temperatura a 95 gradi centigradi per 5 min.
- Memorizzare la libreria cDNA generata per un massimo di 12 mesi a -20 gradi centigradi.
4. Amplificazione e purificazione genica mitocondriale
- Preparare la miscela di PCR utilizzando una pipetta da 10 ll. Aggiungere 0,25 l (1,25 U) di polimerasi del DNA ad alta fedeltà, 2 l di soluzione dNTP (2,5 mM di ogni dNTP), 0,5 l dell'innl di COX1 o dell'innl forwardr ND (100 m), 0,5 di l del COX1 corrispondente o ND reverser (100M), 1 prima della libreria cDNA , 5 ll di soluzione tampone 5x e 16 L di distillato H2O in un tubo PCR 300 ll.
- Eseguire l'amplificazione PCR utilizzando i seguenti parametri: Dopo una fase di denaturazione iniziale a 95 gradi centigradi (durata 5 min), 35 cicli di reazione PCR costituiti da una fase annealing a 55 s (30 s), gradino di allungamento a 72 gradi centigradi (2 min) e gradino di denaturazione a 95 gradi centigradi (30 s) , eseguire un passaggio di allungamento di finalizzazione per 5 min a 72 gradi centigradi.
- Utilizzare 5 L del prodotto PCR per verificare con l'elettroforesi gel agarose la qualità del prodotto PCR ottenuto. Osservare il gene amplificatori COX1 o ND come una singola banda a 759 o 748 coppie di basi, rispettivamente.
- Purificare il resto del prodotto PCR con un kit di pulizia PCR.
- Aggiungere al campione 100 l di soluzione 'PCR-A' (buffer di legame del DNA che contiene alte concentrazioni di sali caotropici11). Vortex brevemente per mescolare il contenuto.
- Collocare la colonna di purificazione in un tubo di centrifuga di 2 mL. Pipette la miscela di reazione di 4.4.1. nella colonna. Centrifuga a 14.000 x g per 1 min a temperatura ambiente.
- Eliminare il filtrato dal tubo di centrifuga. Riportare la colonna al tubo di centrifugazione da 2 mL, aggiungere 700 l di soluzione 'W2' nella colonna e centrifugare a 14.000 x g per 1 min a temperatura ambiente ('W2' è una soluzione di lavaggio che contiene alte concentrazioni di etanolo per la rimozione del chaotropic residuo sali della colonna di purificazione).
- Eliminare il filtrato e riportare la colonna al tubo di centrifuga di 2 mL. Aggiungere 400 l di soluzione 'W2' alla colonna e centrifugare a 14.000 x g per 1 min a temperatura ambiente.
- Preriscaldare 1 mL di acqua deionizzata a 65 gradi centigradi in un riscaldatore a blocchi metallici. Trasferire la colonna in un nuovo tubo di centrifuga di 1,5 mL. Pipette 25 - L del 65 gradi centigradi di acqua preriscaldata al centro della membrana della colonna bianca. Lasciare in ammollo la membrana per 1 min a temperatura ambiente.
- Centrifuga a 14.000 x g per 1 min a temperatura ambiente e scartare la colonna.
- Conservare il prodotto PCR purificato per un massimo di 12 mesi a -20 gradi centigradi.
5. Sequenziamento e confronto genico mitocondriale
- Inviare i campioni PCR purificati dal passaggio 4.5 per il sequenziamento di Sanger utilizzando i relativi primer in avanti COX1 o ND come primer di sequenziamento. Facoltativamente, i primer inversi COX1 o ND possono essere utilizzati anche per la sequenza bidirezionale.
- Dopo aver recuperato i risultati della sequenza, confrontare le sequenze con la sequenza genomica del ceppo di riferimento delle ostriche del Pacifico (NCBI Taxonomy ID: 29159) utilizzando lo strumento online NCBI Nucleotide BLAST (blast.ncbi.nlm.nih.gov) (Figura2 e Figura 3 ).
6. Applicazione della libreria cDNA per la clonazione dei geni MgUGD e MgUXS
- Amplificare e purificare i geni MgUGD e MgUXS utilizzando i rispettivi primer in avanti e inverso di MgUGD e MgUXS da PCR seguendo i passaggi 4.1. a 4,4.
- Trasferire 2 - L di tampone di digestione (10x concentrato) e 6 -L di acqua deionizzata in un tubo di centrifugadi da 1,5 ml. Aggiungere 10 l dei prodotti MgUGD o MgUXS PCR purificati insieme alle endonucles di restrizione Nde I e Xho I (1- l ciascuno, 20 U). Incubare a 37 gradi centigradi per 3 ore.
- Preparare il vettore pET-30a predigerito: Trasferire 500 ng del vettore pET-30a in un nuovo tubo di centrifugazione da 1,5 ml e ricaricare il volume a 16 -L utilizzando acqua deionizzata. Aggiungere 2 L di tampone di digestione (10x concentrato) insieme alla restrizione endonucles Nde I e Xho I (1 L ciascuno, 20 U).
- Dopo aver incubato la miscela a 37 gradi centigradi per 3 h, aggiungere 1 -L di fosforosa (1 U) e incubare a 37 gradi centigradi per un'altra ora. Inattivare la fosfofolasi alcalina riscaldando a 75 gradi centigradi per 10 minuti in un riscaldatore a blocchi metallici preriscaldato.
- Trasferire 4 L dei prodotti di DNA MgUGD o MgUXS digeriti in tubi di centrifuga freschi da 1,5 ml e aggiungere 4 L del vettore pET-30a digerito. Aggiungere 1 l l di Tampone di Ligazione (10x), 1 L L di T4 ligase (3 U) e incubare la miscela di reazione a 22 gradi centigradi per 3 h.
- Trasformare le cellule competenti E. coli Mach1 elettrocompetenti elettrocompetenti mediante elettroporazione utilizzando i prodotti di legatura. Stendere le cellule trasformate su piastre di agar LB contenenti 50 g/mL di kanamycin. Incubano cellule a 37 gradi centigradi per 16 h.
- Verificare le colonie per l'inserimento desiderato da Sanger sequenziamento utilizzando il plasmide-specifico T7 promotore e primer terminator. Preparare i plasmidi dai cloni batterici convalidati.
7. Test di espressione e attività di MgUGD e MgUXS
- Trasformare E. coli BL21 (DE3) Celle competenti con plasmidi recanti i geni MgUGD e MgUXS e diffondere le cellule trasformate su piastre di agar LB contenenti 50 g/mL di kanamicina. Incubano cellule a 37 gradi centigradi per 16 h.
- Coltivare una singola colonia in 5 mL di supporto LB con 50 g/mL di kanamycin durante la notte. Trasferire la coltura in un mezzo LB da 400 mL e agitare continuamente a 200 giri/m m a una temperatura di 37 , fino a quando la densità ottica ad una lunghezza d'onda di 600 nm (OD600) raggiunge un assorbimento di circa 0,5.
- Ridurre la temperatura di incubazione a 20 gradi centigradi e aggiungere 400 l di isopropile-D-thiogalactopyranoside (concentrazione 1 M). Indurre l'espressione delle proteine ricombinanti per 3 h.
- Raccogliere le cellule per centrifugazione a 4.500 x g per 15 min a 4 gradi centigradi. Sospendere i pellet in 10 mL di buffer di lisi (100 mM NaCl, 50 mM Tris/HCl, 1% Triton X-100, 1 mM fenilmethylsulfonyl- fluoruro (PMSF), pH 8.0).
- Interrompere le cellule per sonicazione per 20 min (40 cicli di acconto / spegnimento con 20 m ampiezza per 15 s a 4 gradi centigradi). Centrifuga a 14.000 x g a 4oC per 20 min e raccogliere il supernatante per il test di attività.
- Eseguire il saggio di attività di MgUGD incubando 2 luna con 2 -L di UDP-glucosio (10 mM), 4 -L di NAD (10 mM), 4 L di MgCl2 (10 mM), 2 L di buffer Tris/HCl (500 mM, pH 7,5) e 6 .L di deionizzato2O in un nuovo buffer 1.5 tubo di centrifuga mL e incubare a 37 gradi centigradi per 30 min.
- Eseguire il saggio di attività di MgUXS incubando 2 L L con 2 -L di acido UDP-glucuronico (10 mM), 2 luna di buffer Tris/HCl (500 mM, pH 7,5) e 14 -L di deionizzato H2O in un nuovo tubo centrifuga di 1,5 mL e incubare a 37 min.
- Spegnire le reazioni nei passaggi 7,5 e 7,6 aggiungendo 20: l'L di metanolo e 40 l di cloroformio ad ogni miscela. Dopo aver vortice le miscele del campione, centrifugare a 14.000 x g per 6 min a 4 gradi centigradi e raccogliere lo strato acquoso superiore di ogni tubo.
- Analizzare i prodotti di reazione utilizzando la spettrometria di massa MALDI-TOF in modalità ionizzazione negativa nell'intervallo m/z da 500 a 700. Mescolare 1 o L di miscela campione con 1 : L di 2,5-dihydroxybenzoic matrice campione di acido (1% w/V nel 50% aqueous acetonitrile). Osservare i valori m/z previsti a 579 e 535 rispettivamente per l'acido UDP-glucuronico e UDP-xylose (Figura4).
Risultati
La figura 1 mostra una panoramica schematica del metodo di preparazione descritto della libreria cDNA convergente derivata da individui di ostriche del Pacifico. La figura 2 mostra le sequenze dei geni COX1 e ND di un campione di ostriche distante correlato con elevata divergenza dalle sequenze geniche COX1 e ND del materiale di riferimento. La figura 3 mostra le sequenze dei geni COX1 e ND di un campione di ostrica strettamente correlato con bassa divergenza dalle sequenze geniche COX1 e ND del materiale di riferimento. La figura 4 mostra la riuscita applicazione della libreria cDNA per clonare i geni rilevanti industrialmente MgUGD e MgUXS.
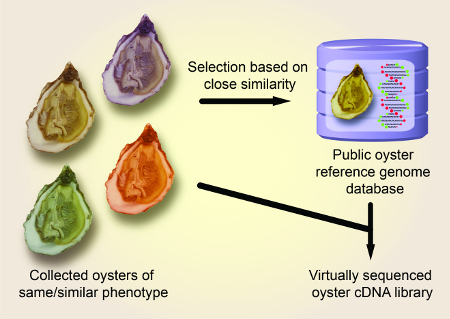
Figura 1 : Panoramica schematica del metodo di analisi descritto per identificazione molecolare di Esemplare di ostriche del Pacifico utilizzando COX1 e ND come geni di riferimento. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 2 : Allineamento della sequenza delle sequenze geniche COX1 e ND di un campione altamente divergente confrontato con le sequenze geniche COX1 e ND del riferimento Ostriche del Pacifico ceppo. Fare clic qui per visualizzare una versione più grande di questa figura.
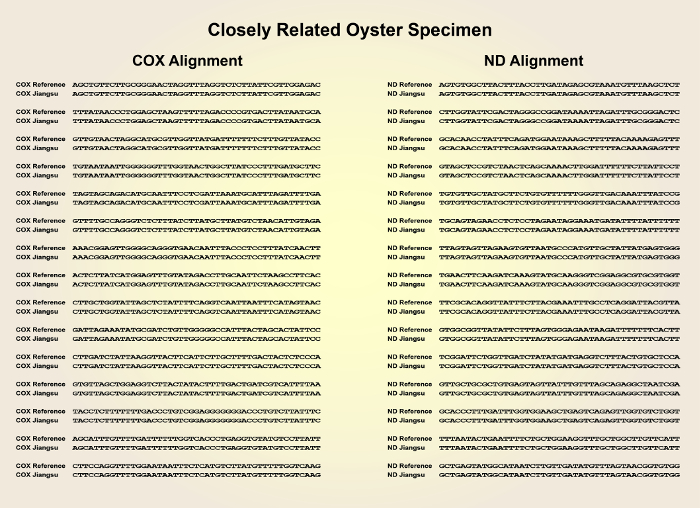
Figura 3 : Allineamento della sequenza delle sequenze geniche COX1 e ND di un campione strettamente correlato rispetto alle sequenze geniche COX1 e ND del riferimento Ostriche del Pacifico ceppo. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 4 : Panoramica schematica di clonazione molecolare, espressione ricombinante e rilevamento dei prodotti di reazione di MgUGD e MgUXS. Fare clic qui per visualizzare una versione più grande di questa figura.
Discussione
Il protocollo presentato consente l'identificazione genetica di campioni di ostriche senza riferimenti con fenotipo simile provenienti dai mercati ittici regionali confrontando i geni COX1 e ND con un database del genoma del DNA delle ostriche disponibile al pubblico. Il significato di questo metodo sta nella sua semplicità, in quanto è necessaria una sola reazione PCR per la valutazione della libreria cDNA virtuale. I due geni coX1 e ND mitocondriali conservati sono stati amplificati da una libreria cDNA che è stata generata dalla trascrizione inversa di estratti di RNA da ogni ostrica. Il metodo di isolamento dell'RNA (passaggio 2.1) è stato semplificato macinando direttamente il tessuto di ostrica in azoto liquido. Dopo aver sequenziato i geni COX1 e ND di ogni campione, i allineamenti di sequenza hanno rivelato che alcuni campioni mostrano un'elevata somiglianza con il ceppo di riferimento. Il parente più vicino ha mostrato un'identità completa sia delle sequenze geniche COX1 che di ND.
Le fasi più critiche di questa procedura sono la fase di estrazione dell'RNA; al fine di ridurre al minimo la degradazione dell'RNA, è essenziale ridurre il tempo che intercela la raccolta del tessuto ostrica e l'estrazione dell'RNA.
La clonazione è stata recentemente esemplificata clonando il gene UGE delle ostriche12 e qui clonando i geni MgUGD e MgUXS13, che hanno convalidato la praticità della libreria cDNA generata, consentendo la clonazione di un numero qualsiasi di geni di interesse senza la necessità di ingombranti strategie di clonazione utilizzando primer degenerati. Questo metodo di identificazione molecolare per amplificazione dei geni COX1 e ND per generare librerie di CDNA virtualmente sequenziate può essere utilizzato anche in applicazioni future per altri materiali biologici che non hanno campioni fisici di genomi di riferimento disponibile.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Questo lavoro è stato sostenuto in parte dalla Natural Science Foundation of China (numeri di sovvenzione 31471703, A0201300537 e 31671854 a J.V. e L.L., numero di sovvenzione 31470435 a G.Y.), e il 100 Foreign Talents Plan (numero di sovvenzione JSB2014012 a J.V.).
Materiali
| Name | Company | Catalog Number | Comments |
| Chemicals: | |||
| 1% Triton X-100 | Solarbio | 9002-93-1 | *Alternative distributors possible |
| 2,5-Dihydroxybenzoic acid | Alfa Aesar | 490-79-9 | *Alternative distributors possible |
| Acetonitrile | Merck | 75-05-8 | *Alternative distributors possible |
| Agarose for molecular biology | Biowest Chemicals | 111860 | *Alternative distributors possible |
| Ampicilin | Solarbio | 69-52-3 | *Alternative distributors possible |
| Chloroform | Lingfeng, Shanghai | 67-66-3 | *Alternative distributors possible |
| DEPC water | Thermo Scientific | R0601 | |
| Ethanol | Jinhuada, Guangzhou | 64-17-5 | *Alternative distributors possible |
| Guanidinium thiocyanate-phenol reagent | Invitrogen | 15596018 | TRIzol reagent |
| Imidazole | Energy Chemical | 288-32-4 | *Alternative distributors possible |
| Isopropyl alcohol | Nanjing Chemical Reagent | 67-63-0 | *Alternative distributors possible |
| Isopropyl β-D-thiogalactopyranoside | Solarbio | 367-93-1 | *Alternative distributors possible |
| Kanamycin | Solarbio | 25389-94-0 | *Alternative distributors possible |
| LB Agar | Thermo Fisher | 22700025 | *Alternative distributors possible |
| LB Broth | Thermo Fisher | 10855021 | *Alternative distributors possible |
| Methanol | Jinhuada, Guangzhou | 67-56-1 | *Alternative distributors possible |
| MgCl2 hexahydrate | Xilong Huagong | 7791-18-6 | *Alternative distributors possible |
| NaCl | Xilong Huagong | 7647-14-5 | *Alternative distributors possible |
| NAD+ | Duly Biotech | 53-84-9 | *Alternative distributors possible |
| Phenyl-methylsulfonyl fluoride | Macklin | 329-98-6 | *Alternative distributors possible |
| Tris | Solarbio | 77-86-1 | *Alternative distributors possible |
| UDP-glucose | Wuhu Nuowei Chemicals | 28053-08-9 | *Alternative distributors possible |
| UDP-glucuronic acid | SIGMA | 63700-19-6 | *Alternative distributors possible |
| Tools/Instruments: | |||
| MALDI-TOF mass spectrometer | Bruker | Autoflex | *Alternative distributors possible |
| Metal block heater | Long Yang Scientific Instruments | Thermoshaker HB20 | *Alternative distributors possible |
| PCR thermocycler | Hema | 9600 | *Alternative distributors possible |
| Enzyme and Kits: | |||
| 10×Ligation buffer | Thermo Scientific | B69 | *Alternative distributors possible |
| 5×PrimeSTAR buffer | Takara | 9158A | |
| Alkaline phosphatase | ThermoFisher FastAP | EF0654 | *Alternative distributors possible |
| COX forward primer | Genscript | ATGTCAACAAATCATTTAGACATTG | |
| COX reverse primer | Genscript | ACTTGACCAAAAACATAAGACATG | |
| Cutsmart Buffer | NEB | B7204S | *Alternative distributors possible |
| dNTP mix | Invitrogen | 18427088 | |
| MgUGD forward primer | Genscript | ACATATGACCCTGTCCAAGATCTGTTGT | |
| MgUGD reverse primer | Genscript | ACTCGAGACTCTGTGAGGCGGTGGAG | |
| MgUXS forward primer | Genscript | CCATATGGCAGAATCCTCACAATCAC | |
| MgUXS reverse primer | Genscript | ACTCGAGCACATTTTTGAATTTGCAGACGT | |
| ND forward primer | Genscript | ATGAGATGGCAATTATTTTTTAAT | |
| ND reverse primer | Genscript | ATGTATTTTGGAAAAATCTCCAC | |
| PCR Cleanup Kit | AxyGen | AP-PCR-250 | *Alternative distributors possible |
| pET-30a(+) vector | Merck Millipore | 69909 |
Riferimenti
- Blaxter, M. L. The promise of a DNA taxonomy. Philosophical transactions of the Royal Society of London. Series B, Biological. 359 (1444), 669-679 (2004).
- Wen, J., et al. Species identification of dried shellfish (oyster, clam and mussel) products sold on the Chinese market. Food Control. 90, 199-204 (2018).
- Zhang, H., et al. Mitochondrial cob and cox1 genes and editing of the corresponding mRNAs in Dinophysis acuminata from Narragansett Bay, with special reference to the phylogenetic position of the genus Dinophysis. Applied and Environmental Microbiology. 74 (5), 1546-1554 (2007).
- Sell, J., Spirkovski, Z. Mitochondrial DNA differentiation between two forms of trout Salmo letnica, endemic to the Balkan Lake Ohrid, reflects their reproductive isolation. Molecular Ecology. 13, 3633-3644 (2004).
- Karadjian, G., et al. Highly rearranged mitochondrial genome in Nycteria parasites (Haemosporidia) from bats. Proceedings of the National Academy of Sciences of the United States of America. 113 (35), 9834-9839 (2018).
- Morga, B., et al. Identification of genes from flat oyster Ostrea edulis as suitable housekeeping genes for quantitative real time PCR. Fish and Shellfish Immunology. 29 (6), 937-945 (2010).
- Delsuc, F., et al. Molecular systematics of armadillos (Xenarthra, Dasypodidae): contribution of maximum likelihood and Bayesian analyses of mitochondrial and nuclear genes. Molecular Phylogenetics and Evolution. 28 (2), 261-265 (2005).
- Wei, S., et al. Discovery and Biochemical Characterization of UDP-Glucose Dehydrogenase from Akkermansia muciniphila. Protein & Peptide Letters. 24 (8), 735-741 (2017).
- Gu, B., et al. Discovery and Biochemical Characterization of the UDP-Xylose Biosynthesis Pathway in Sphaerobacter thermophilus. Protein & Peptide Letters. 23 (12), 1103-1110 (2016).
- Duan, X. C., et al. Functional characterization of the UDP-xylose biosynthesis pathway in Rhodothermus marinus. Applied Microbiology and Biotechnology. 99 (22), 9463-9472 (2015).
- Vogelstein, B., Gillespie, D. Preparative and analytical purification of DNA from agarose. Proceedings of the National Academy of Sciences of the United States of America. 76 (2), 615-619 (1979).
- Song, H. B., et al. UDP-glucose 4-epimerase and β-1,4-galactosyltransferase from the oyster Magallana gigas as valuable biocatalysts for the production of galactosylated products. International Journal of Molecular Sciences. 19 (6), 1600 (2018).
- Gainey, P. A., Phelps, C. F. Uridine diphosphate glucuronic acid production and utilization in various tissues actively synthesizing glycosaminoglycans. Biochemical Journal. 128 (2), 215-227 (1972).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon