Method Article
אסטרטגיה מתכנס לדור של ספריית דנ א בלתי מופנה מצדפות פסיפיק
In This Article
Summary
אנו מתארים אסטרטגיה כיצד להשתמש בדגימות RNA מתוך דגימות הצדפה הפסיפיק, ולהעריך את החומר הגנטי על ידי השוואה עם נתוני הגנום זמין בפומבי כדי ליצור ספריית cDNA כמעט רציף.
Abstract
הגישה לחומר ביולוגי של מינים התייחסות, אשר שימשו בעבר ניסויים מרכזיים כגון בפיתוח של קווי התא הרומן או פרויקטים ברצף הגנום, הם לעתים קרובות קשה לספק למחקרים נוספים או צדדים שלישיים בשל ה consumptive הטבע של הדגימות. למרות שעכשיו נרחב מופץ על חופי האוקיאנוס השקט של אסיה, אוסטרליה וצפון אמריקה, בודדים האוקיינוס השקט דגימות מגוונות מבחינה גנטית ולכן הם לא מתאימים ישירות כמו חומר מתחיל עבור ספריות גנים. במאמר זה, אנו מדגימים את השימוש בדגימות של צדפות האוקיינוס השקט שהתקבלו משוקי פירות ים אזוריים כדי ליצור ספריות cDNA. ספריות אלו הושוו לגנום הצדפות הזמין בציבור, והספרייה הקרובה ביותר נבחרה באמצעות התייחסות מיטוכונדריאלי גנים ציטוכרום C אוקסידאז I (COX1) ו-NADH דהידרוגנאז (ND). ההתאמה של הספרייה cdna שנוצר מומחש גם על ידי שיבוט וביטוי של שני גנים קידוד האנזימים של החומצה udp גלוקוראז (אויח) ו-udp-xylose אבד סטנדרטים (uxs), אשר אחראים על הביוסינתזה של udp-קסיפסיד מ UDP-גלוקוז.
Introduction
הקניית החומר הביולוגי שניתן לטפל בהן, עלולה להיות מאתגרת עקב זמני משלוחים ארוכים, הסקת מסקנות יזמות או תקנות מכס ספציפיות למדינה. כחלופה, החומר הביולוגי הנדרש ניתן גם לאיסוף מדגימות זהות בדרך כלל. עם זאת, דוגמאות אלה עשויות להשתנות באופן משמעותי ברמת ה-גנוטיפ, ולכן השוואות בעלות התייחסות דיגיטלית מאוחסנת מאותו מינים מעובדים לעתים קרובות קשות או אפילו חסרות תועלת עקב אי התאמה של החומר החדש ממקור עם שיטות הגברה קיימות של הדנ א. רצף הגנים שנשמרים במיוחד של דגימות בודדות הוא כלי נפוץ ורב עוצמה לזיהוי מינים1, כגון גנים מיטוכונדריאלי שימור המשמשים לעתים קרובות גנים התייחסות להערכת איכות של ספריות cdna2 ,3,4,5,6 הרציונל הבסיסי של שיטה זו הציגה הוא כי שימור גבוה של רצפי גנים מיטוכונדריאלי בדגימות הצדפה אנונימי בודדים לעומת רצפים המקביל של הגנום התייחסות מעיד כי גנים אחרים עשויים גם להראות רמה נמוכה של סטייה, בהינתן שיעור מהיר בדרך כלל של האבולוציה מיטוכונדריאלי ביחס ל-DNA גרעיני7, המאפשר הגברה ובידוד של מגוון רחב של גנים רלוונטיים מדעית ותעשייתית על ידי שימוש בפומבי נתוני רצף נתונים זמינים כהפניה.
המטרה הכוללת של השיטה המתוארת היא להציג זרימת עבודה ממוטבת ליצירת הצדפה כמעט רציף cDNA הספרייה אשר ניתן להשתמש בה כמו DNA תבנית עבור שיבוט של גנים צדפה. ברצף וירטואלי, רצף הגנום דה נובו הוא שפרקו; במקום זאת, משתמשים ברצף הפניה מוכר ומאוחסן באופן דיגיטלי כדי לנצל או לעצב צבעי יסוד לייצור cDNAs שבסופו של דבר מהווים ספריה (או יתווספו לקיים מראש). המטרה היא לייצר ספריית cDNA מתכנסת, כלומר דמיון בין רצפי ה-Dna שנוצרו ואת רצף ההתייחסות יכול להיות מדורגת מפני התפצלות נמוך עד גבוה. יתרון מרכזי של שימוש בציטוכרום C אוקסידאז משנה 1 (COX1) ו NADH Dehydrogenase (ND) כמו גנים התייחסות היא כי אפילו מאוד גיאוגרפית ופרד דגימות הצדפות ניתן לפרופיל בשל שימור גבוה של גנים אלה מיטוכונדריאלי. לאחר הוכיחה את הגישה עם סמנים אלה מבוססים, אנו לאחר מכן להדגים את היישום שלה לשני מועמדים אנזימים אשר מעורבים בביוסינתזה נוקלאוטיד סוכר ועשויים להיות בעלי רלוונטיות תעשייתית8,9, 10. הפוטנציאל הביוטכנולוגי של הצדפה באוקיינוס השקט עדיין לא נחקר. לפיכך, אנו מאמינים כי שיטה מתכנס זו להכנת ספריית cDNA כמעט ברצף יהיה גם מתאים לחוקרים שאינם מומחים המעוניינים ליצור cDNA מחומר זה ביולוגי רלוונטי.
Protocol
הערה: מבט סכימטי מוצג באיור 1.
1. אוסף דוגמאות
- . השיגו דגימות צדפה שמרו צדפות על הקרח במהלך התקופה החקלאית, ההובלה ולפני השימוש במעבדה והתהליך בתוך 4-7 ימים לאחר הרכישה.
הערה: עבור פרוטוקול זה, צדפות נרכשו מהשוק הסיטונאי Zhong Cai לנאנג (שמקורם בנינגנדה, פוג'יין, סין Lianyungang, ג'יאנגסו, סין), החברה מוצר ימיים Haijie בצ (שמקורם צ'ינגדאו, שאנדונג, סין), Jucheng חברת מוצרים ימיים ביאנטאי (שמקורם יאנטאי, שאנדונג, סין) ו Jinxiu מוצר החברה המוצר צ'ינגדאו (שמקורם צ'ינגדאו, שאנדונג, סין)).
2. בידוד רנ א על ידי גואנידיניום-הפקת פנול
- הכנת דגימת רקמת צדפה
- גזור כ 100 מ"ג של רקמה רכה הומוגנית מהמרכז הגיאומטרי משוער של כל דגימה צדפה עם אזמל מעוקר, ולהעביר את הדגימות לתוך חנקן נוזלי.
- המתת חסד את החלק הנותר של הצדפות על ידי הקפאת ב-80 ° c עבור 1 h ולהשליך כמו פסולת ביולוגית.
- לטחון את רקמת הצדפה הקפואה בהבזק לאבקה משובחת בחומר מליטה (200 mL) המלא ב 50 מ ל של חנקן נוזלי.
- שוקלים 75 מ"ג של הרקמה הקפואה של כל מדגם לתוך שפופרת סטרילי 1.5 mL ולערבב עם 1 מ ל של guanidinium החילוץ-פנול מגיב. צנטריפוגה את המדגם ב 14,000 x g ב 4 ° צ' עבור 15 דקות.
- להעביר את supernatant לצינור חדש 1.5 mL של צנטריפוגה, להוסיף 200 μL של כלורופורם ולערבב ביסודיות באמצעות מערבל מערבולת עבור 10-15 s עד התערובת הופך חלבי-לבן.
- צנטריפוגה ב 14,000 x g ב 4 ° צ' עבור 15 דקות, ולהעביר את שכבת מימית העליון בזהירות עם הצינור 200 μl מבלי להפריע את השלב הפנימי לתוך שפופרת צנטריפוגה של 1 מ ל 1.5 חדש.
- הוסף 500 μL של אלכוהול איזופרופיל וערבב את הדגימות בעדינות על-ידי היפוך, ולאחר מכן השאר את הדגימות עבור 20 דקות על הקרח. צנטריפוגה ב 14,000 x g ב 4 ° צ' עבור 8 דקות ולהסיר את הסופרנטאנט.
- השהה מחדש את כל כדורי ב 1 מ ל של 75% אטוח, וצנטריפוגה ב 14,000 x g ב 4 ° c עבור 5 דקות. הסר את כל הסופרנטאנט.
- חזור על שלב 2.5 פעם אחת. יבש את כדורי 6 דקות בטמפרטורת החדר. אל תייבש למשך זמן רב יותר; אחרת, זה עלול להיות קשה לפזר את הגלולה RNA בשלב הבא.
- לפזר את הגלולה מיובש RNA ב 25 μL של DEPC (diethylפחמתי)-מטופלים מים ולשמור על הצינור על הקרח. השתמש בדגימות RNA בתוך 24 שעות.
3. cDNA הדור בספרייה על ידי שעתוק הפוכה
- עבור כל לדוגמה RNA, להכין תערובת התגובה באמצעות מערכת מסחרי הפוכה שעתוק באמצעות הצינורות 10 μL: להוסיף 4 μL של פתרון MgCl2 , 2 μl של מאגר התגובה 10x, 2 μl של פתרון dntp, 0.5 μl של מעכב RNase, 0.7 μl של היפוך amv טראנסקריפט, 0.5 μL של Oligo (dT) 15 פריימר, 1 μL של דגימת RNA שחולצו ו 9.3 μL של H2O לתוך שפופרת 300 μl-PCR.
- התערובת מודקרת ב-PCR 60 דקות ב-42 ° c, ולאחר מכן הגדילו את הטמפרטורה ל-95 ° c עבור 5 דקות.
- אחסן את ספריית cDNA שנוצרה עד 12 חודשים ב-20 ° c.
4. גן מיטוכונדריאלי הגברה וטיהור
- הכינו את תערובת ה-PCR באמצעות הפיפטה 10 μL. הוסף 0.25 μL (1.25 U) של באיכות גבוהה DNA פולימראז, 2 μL של פתרון dNTP (2.5 mM של כל dNTP), 0.5 μL של COX1 או ND בהילוך קדמי (100 μM), 0.5 μL של היפוך COX1 או ND פריימר הפוכה (100 μM), 1 μL של ספריית cDNA , 5 μL של פתרון מאגר 5x ו -16 μL של מזוקק H2O לתוך צינור 300 μl.
- בצע את הגברה ה-PCR באמצעות הפרמטרים הבאים: לאחר צעד התחלתי ראשוני ב-95 ° צ' (משך 5 דקות), 35 מחזורי התגובה של ה-PCR המורכב משלב ריפוי ב55 ° צ' (30), צעד התארכות בשעה 72 ° צ' (2 דקות) וצעד הדנטורציה ב-95 ° צ' (30) , בצע צעד התארכות אחד עבור 5 דקות ב-72 ° c.
- השתמש 5 μL של מוצר ה-PCR כדי לוודא על-ידי אגנה ג'ל האיכות של מוצר ה-PCR המתקבל. לצפות את הגן COX1 או ND מוגבר כמו להקה אחת בכל אחד 759 או 748 זוגות בסיס, בהתאמה.
- לטהר את שאר מוצר ה-PCR עם ערכת ניקוי של ה-PCR.
- הוסף 100 μL של פתרון ' PCR-A ' (מאגר כריכת DNA המכיל ריכוזים גבוהים של מלחי chaotropic11) למדגם. מערבולת לזמן קצר כדי לערבב את התוכן.
- מקם את עמודת הטיהור לתוך צינורית צנטריפוגה של 2 מ ל. מפיפטה את תערובת התגובה של ה4.4.1. לתוך הטור. צנטריפוגה ב 14,000 x g עבור 1 דקות בטמפרטורת החדר.
- השמט את הפילטרט מצינור הצנטריפוגה. להחזיר את העמודה 2 mL צנטריפוגה שפופרת, להוסיף 700 μL של פתרון ' W2 ' לתוך העמודה צנטריפוגה ב 14,000 x g עבור 1 דקות בטמפרטורת החדר (' W2 ' הוא פתרון כביסה אשר מכיל ריכוזים גבוהים של אתנול להסרת שיורית chaotropic מלחים מעמודת הטיהור).
- התעלם מהfiltrate והחזר את העמודה לשפופרת הצנטריפוגה של 2 מ"ל. הוסף 400 μL של פתרון ' W2 ' לעמודה ולצנטריפוגה ב 14,000 x g עבור 1 דקות בטמפרטורת החדר.
- טרום חום 1 מ ל של מים מיוהים עד 65 ° c בתנור מתכת לחסום. העבר את העמודה לשפופרת צנטריפוגה חדשה 1.5 mL. פיפטה 25 μL של 65 מעלות צלזיוס מחומם לפני המים המחוממים והחמים למרכז קרום הטור הלבן. תנו לקרום להשרות במשך 1 דקות בטמפרטורת החדר.
- צנטריפוגה ב 14,000 x g עבור 1 דקות בטמפרטורת החדר ולמחוק את העמודה.
- אחסן את מוצר ה-PCR הטהור למשך עד 12 חודשים ב-20 ° c.
5. רצף מיטוכונדריאלי גנטי והשוואה
- שלחו את דגימות ה-PCR המטוהרים משלב 4.5 עבור רצפי שלבים באמצעות COX1 או ND לפני התחל ברצף התחל. באופן אופציונלי, ניתן להשתמש בCOX1 או ND התחל הפוך גם עבור רצף דו-כיווני.
- לאחר אחזור התוצאות ברצף, להשוות את רצפי עם רצף הגנום של השמש צדפה התייחסות זן (מזהה טקסונומיה של NCBI 29159) באמצעות הפיצוץ NCBI ק בכלי מקוון (blast.ncbi.nlm.nih.gov) (איור 2 ואיור 3 ).
6. החלת ספריית cDNA לשכפול הגנים MgUGD ו MgUXS
- להגביר ולטהר את הגנים MgUGD ו MgUXS באמצעות התחל קדימה והפוכה הפוך של MgUGD ו MgUXS על ידי ה-PCR לאחר השלבים 4.1. ל4.4.
- העברה 2 μL של מאגר העיכול (10x מרוכז) ו 6 μL של מים מוזים לתוך שפופרת צנטריפוגה 1.5 mL. הוסף 10 μL של מוצרים MgUGD או MgUXS PCR מטוהרים יחד עם end, ההגבלה Nde I ו-Xho I (1 μL כל אחד, 20 U). מודטה ב 37 ° צ' צלזיוס במשך 3 שעות.
- להכין את pET-30a מתעכל וקטור: להעביר 500 ng של pET-30a וקטור לתוך שפופרת צנטריפוגה חדש 1.5 mL ו למעלה את אמצעי האחסון כדי 16 μL באמצעות מים מוהים. הוסף 2 μL של מאגר העיכול (10x מרוכז) יחד עם ההגבלה end, הגבלת Nde I ו-Xho I (1 μL כל אחד, 20 U).
- לאחר הדגירה של התערובת ב 37 ° c עבור 3 h, להוסיף 1 μL של פוספספטאז אלקליין (1 U) ו דגירה ב 37 ° c עבור שעה נוספת. הפוך את פוספספטאז אלקליין על ידי חימום ב 75 ° c עבור 10 דקות בתנור מתכת מחומם מראש לחסום.
- העברה 4 μL של MgUGD או MgUXS DNA מוצרים לתוך טריות 1.5 mL לצינורות צנטריפוגה ולהוסיף 4 μL של וקטור pET-30a מתעכל. הוסף 1 μl של מאגר לחיץ (10x), 1 μl של T4 ליגאז (3 U) ו הדגירה תערובת התגובה ב 22 ° c עבור 3 h.
- שינוי צורה אלקטרומוסמכת E. coli Mach1 תאים מוסמך על ידי אלקטרופורציה באמצעות מוצרים ליפטיים. הפיצו את התאים שעברו שינוי לוחות LB אגר המכיל 50 μg/mL kanamycin. התאים הדגירה ב 37 ° צ' צלזיוס עבור 16 h.
- לאמת מושבות עבור ההכנסה הרצויה על ידי רצף Sanger באמצעות היזם T7 ספציפי הספציפית ושליחות קטלנית התחל. הכינו פלסטלינה מהשיבוטים החיידקיים המאומתים.
7. ביטוי ובדיקות פעילות של MgUGD ו-MgUXS
- המרה E. coli BL21 (DE3) תאים מוכשרים עם פלמידים הנושאים את הגנים MgUGD ו MgUXS ולהפיץ את התאים שעברו שינוי לוחות LB אגר המכיל 50 μg/mL kanamycin. התאים הדגירה ב 37 ° צ' צלזיוס עבור 16 h.
- לטפח מושבה אחת ב 5 מ"ל ליברות בינונית עם 50 μg/mL kanamycin לילה. להעביר את התרבות לתוך 400 mL ליברות בינונית ולטלטל ברציפות ב 200 סל"ד בטמפרטורה של 37 ° צ' עד הצפיפות האופטית באורך הגל של 600 ננומטר (OD600) מגיע לספיגה של כ 0.5.
- הפחת את טמפרטורת הדגירה ל-20 ° c והוסף 400 μL של איזופרופיל β-D-תאיוגליוןצד (ריכוז 1 מ'). לגרום לביטוי של חלבונים רקומביננטי במשך 3 שעות.
- לקצור את התאים על ידי צנטריפוגה ב 4,500 x g עבור 15 דקות ב 4 ° c. להשעות כדורי ב 10 מ ל של מאגר הליזה (100 מ"מ, 50 mM טריס/HCl, 1% טריטון X-100, 1 מ"מ פנילמתיל-פלואור (PMSF), pH 8.0).
- לשבש את התאים על ידי sonication עבור 20 דקות (40 הופעל/off מחזורים עם משרעת 20 יקרומטר עבור 15 s ב 4 ° c). צנטריפוגה ב 14,000 x g ב 4 ° צ' עבור 20 דקות ולאסוף את supernatant עבור בדיקת הפעילות.
- בצע את שיטת הפעילות של MgUGD על-ידי דגירה 2 μL של התא ליפוסט עם 2 μL של ה-UDP-גלוקוז (10 מ"מ), 4 μL של NAD+ (10mm), 4 Μl של MgCl2 (10 מ"מ), 2 μl של מאגר טריס/HCl (500 מ"מ, pH 7.5), ו-6 μl של H מועברו2O ב חדש 1.5 שפופרת הצנטריפוגה של mL ו-דגירה ב 37 ° c עבור 30 דקות.
- לבצע את שיטת הפעילות של MgUXS על-ידי מארג 2 μL של תא ליפוסט עם 2 μL של חומצה UDP-גלוקורונית (10 מ"מ), 2 μL של מאגר טריס/HCl (500 מ"מ, pH 7.5), ו 14 μL של H2O בתוך הצינור החדש של 1.5 mL ו-דגירה ב 37 °
- כיבוי התגובות בשלב 7.5 ו 7.6 על ידי הוספת 20 μL של מתנול ו 40 μL של כלורופורם לכל תערובת. לאחר vortexing את תערובות לדוגמה, צנטריפוגה ב 14,000 x g עבור 6 דקות ב 4 ° צ' ולאסוף את השכבה העליונה מימית של כל צינור.
- לנתח את מוצרי הריאקציה באמצעות מאלדי-תוף ספקטרומטר מסה במצב אינון שלילי בטווח m/z מ500-700. מערבבים 1 μL של תערובת לדוגמה עם 1 μL של 2, 5-דיהידרוטסטוסטרון מטריצה לדוגמה חומצה (1% w/V ב 50% מימית). שימו לב לערכי m/z הצפויים ב-579 ו-535 עבור חומצות UDP-גלוקוריוני ו-UDP-xylose בהתאמה (איור 4).
תוצאות
איור 1 מציג סקירה סכמטית של שיטת ההכנה המתוארת של ספריית cdna המתכנסת הנגזרת מאנשי הצדפות באוקיינוס השקט. איור 2 מראה את רצפי הגנים COX1 ו-ND של דגימה ממרחק הקשורים הצדפה עם התפצלות גבוהה מתוך COX1 ו nd רצפי גנים של חומר הייחוס. איור 3 מראה את רצפי הגנים COX1 ו-ND של מדגם צדפה הקשורים היטב עם חריגה נמוכה מרצפי הגנים COX1 ו-nd של חומר הייחוס. איור 4 מציג את היישום המוצלח של ספריית cdna כדי לשכפל את הגנים הרלוונטיים תעשייתי MgUGD ו MgUXS.
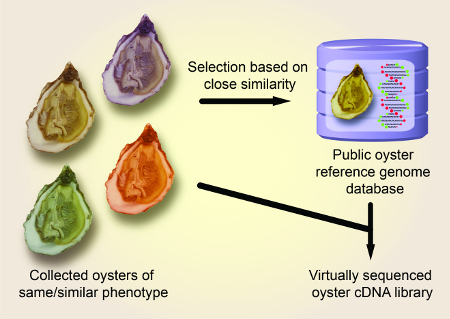
איור 1 : סקירה סכמטית של שיטת הניתוח המתוארת עבור זיהוי מולקולרי של הצדפה הפסיפי באמצעות COX1 ו-ND כמו גנים התייחסות. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.

איור 2 : יישור רצף של רצפי הגנים הCOX1 והnd של הדגימה המפוצלים בהשוואה לרצפי הגנים הCOX1 והnd מההפניה צדפה באוקיינוס השקט מתח. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
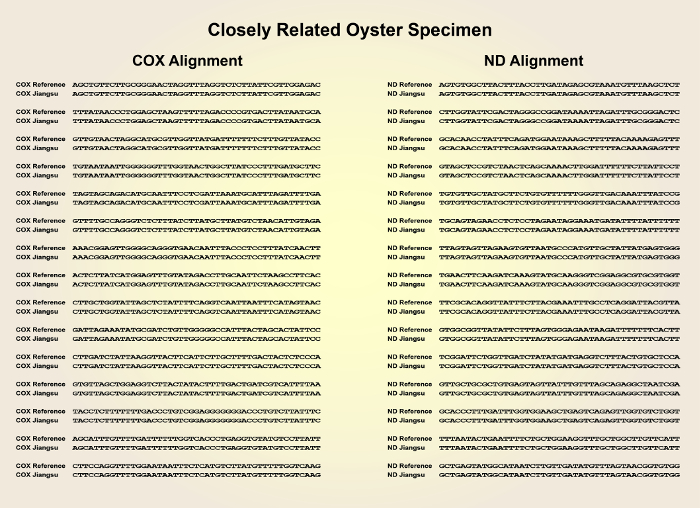
איור 3 : יישור רצף של רצפי גנים COX1 ו-nd של מדגם קשור היטב בהשוואה לרצפי הגנים הCOX1 והnd מההפניה צדפה באוקיינוס השקט מתח. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.

איור 4 : סקירה סכימטית של שיבוט מולקולרית, רקומביננטי ביטוי וגילוי של מוצרי התגובה של MgUGD ו MgUXS. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
Discussion
הפרוטוקול המוצג מאפשר זיהוי גנטי של דגימות צדפה ללא הפניה עם פנוטיפ דומה משוקי פירות ים אזוריים על ידי השוואה של הגנים COX1 ו-ND עם מאגר הגנום ה-DNA צדפה זמין בפומבי. המשמעות של שיטה זו טמונה בפשטותו, כמו רק תגובת PCR אחת נדרשת להערכת הספרייה cDNA וירטואלי. שני COX1 מיטוכונדריאלי שמרו וגנים ND הוהוגדל מספריית cDNA אשר נוצר על ידי שעתוק הפוכה של תמציות RNA מכל צדפה. השיטה של בידוד RNA (שלב 2.1) היה פשוט יותר על ידי שפשוף הרקמה הצדפה בחנקן נוזלי. לאחר ברצף את הגנים COX1 ו-ND של כל אחד מהדוגמה, מספר הרצף גילה כי כמה דגימות להראות דמיון גבוה למתח התייחסות. הקרוב ביותר הראה זהות מלאה הן של COX1 והן רצפי גנים ND.
הצעדים הקריטיים ביותר של הליך זה הם צעד החילוץ RNA; כדי למזער השפלה RNA, חיוני כדי להקטין את הזמן בין קצירת רקמת הצדפה ואת החילוץ RNA.
שיבוט מוצלח לאחרונה לידי ביטוי על ידי שיבוט הצדפה UGE גן12 ובזאת על ידי שיבוט MgUGD ו MgUXS גנים13, אשר אימת את המעשיות של ספריית cdna שנוצר, המאפשר שיבוט של כל מספר של גנים עניין ללא צורך באסטרטגיות שיבוט מסורבלת באמצעות תחל מנוונת. שיטה זו של זיהוי מולקולרי על ידי הגברה של הגנים COX1 ו-ND כדי ליצור ספריות cdna כמעט רציף עשוי לשמש גם יישומים עתידיים עבור חומרים ביולוגיים אחרים שאין להם דגימות פיזיות של גנום ההפניה זמינים.
Disclosures
. למחברים אין מה לגלות
Acknowledgements
עבודה זו היתה נתמכת בחלקו של הקרן המדע הטבעי של סין (להעניק מספרים 31471703, A0201300537 ו 31671854 ל J.V. ו זמנהוף, גרנט מספר 31470435 G.Y.), ואת תוכנית כשרונות זרים 100 (מענק מספר JSB2014012 ל J.V.).
Materials
| Name | Company | Catalog Number | Comments |
| Chemicals: | |||
| 1% Triton X-100 | Solarbio | 9002-93-1 | *Alternative distributors possible |
| 2,5-Dihydroxybenzoic acid | Alfa Aesar | 490-79-9 | *Alternative distributors possible |
| Acetonitrile | Merck | 75-05-8 | *Alternative distributors possible |
| Agarose for molecular biology | Biowest Chemicals | 111860 | *Alternative distributors possible |
| Ampicilin | Solarbio | 69-52-3 | *Alternative distributors possible |
| Chloroform | Lingfeng, Shanghai | 67-66-3 | *Alternative distributors possible |
| DEPC water | Thermo Scientific | R0601 | |
| Ethanol | Jinhuada, Guangzhou | 64-17-5 | *Alternative distributors possible |
| Guanidinium thiocyanate-phenol reagent | Invitrogen | 15596018 | TRIzol reagent |
| Imidazole | Energy Chemical | 288-32-4 | *Alternative distributors possible |
| Isopropyl alcohol | Nanjing Chemical Reagent | 67-63-0 | *Alternative distributors possible |
| Isopropyl β-D-thiogalactopyranoside | Solarbio | 367-93-1 | *Alternative distributors possible |
| Kanamycin | Solarbio | 25389-94-0 | *Alternative distributors possible |
| LB Agar | Thermo Fisher | 22700025 | *Alternative distributors possible |
| LB Broth | Thermo Fisher | 10855021 | *Alternative distributors possible |
| Methanol | Jinhuada, Guangzhou | 67-56-1 | *Alternative distributors possible |
| MgCl2 hexahydrate | Xilong Huagong | 7791-18-6 | *Alternative distributors possible |
| NaCl | Xilong Huagong | 7647-14-5 | *Alternative distributors possible |
| NAD+ | Duly Biotech | 53-84-9 | *Alternative distributors possible |
| Phenyl-methylsulfonyl fluoride | Macklin | 329-98-6 | *Alternative distributors possible |
| Tris | Solarbio | 77-86-1 | *Alternative distributors possible |
| UDP-glucose | Wuhu Nuowei Chemicals | 28053-08-9 | *Alternative distributors possible |
| UDP-glucuronic acid | SIGMA | 63700-19-6 | *Alternative distributors possible |
| Tools/Instruments: | |||
| MALDI-TOF mass spectrometer | Bruker | Autoflex | *Alternative distributors possible |
| Metal block heater | Long Yang Scientific Instruments | Thermoshaker HB20 | *Alternative distributors possible |
| PCR thermocycler | Hema | 9600 | *Alternative distributors possible |
| Enzyme and Kits: | |||
| 10×Ligation buffer | Thermo Scientific | B69 | *Alternative distributors possible |
| 5×PrimeSTAR buffer | Takara | 9158A | |
| Alkaline phosphatase | ThermoFisher FastAP | EF0654 | *Alternative distributors possible |
| COX forward primer | Genscript | ATGTCAACAAATCATTTAGACATTG | |
| COX reverse primer | Genscript | ACTTGACCAAAAACATAAGACATG | |
| Cutsmart Buffer | NEB | B7204S | *Alternative distributors possible |
| dNTP mix | Invitrogen | 18427088 | |
| MgUGD forward primer | Genscript | ACATATGACCCTGTCCAAGATCTGTTGT | |
| MgUGD reverse primer | Genscript | ACTCGAGACTCTGTGAGGCGGTGGAG | |
| MgUXS forward primer | Genscript | CCATATGGCAGAATCCTCACAATCAC | |
| MgUXS reverse primer | Genscript | ACTCGAGCACATTTTTGAATTTGCAGACGT | |
| ND forward primer | Genscript | ATGAGATGGCAATTATTTTTTAAT | |
| ND reverse primer | Genscript | ATGTATTTTGGAAAAATCTCCAC | |
| PCR Cleanup Kit | AxyGen | AP-PCR-250 | *Alternative distributors possible |
| pET-30a(+) vector | Merck Millipore | 69909 |
References
- Blaxter, M. L. The promise of a DNA taxonomy. Philosophical transactions of the Royal Society of London. Series B, Biological. 359 (1444), 669-679 (2004).
- Wen, J., et al. Species identification of dried shellfish (oyster, clam and mussel) products sold on the Chinese market. Food Control. 90, 199-204 (2018).
- Zhang, H., et al. Mitochondrial cob and cox1 genes and editing of the corresponding mRNAs in Dinophysis acuminata from Narragansett Bay, with special reference to the phylogenetic position of the genus Dinophysis. Applied and Environmental Microbiology. 74 (5), 1546-1554 (2007).
- Sell, J., Spirkovski, Z. Mitochondrial DNA differentiation between two forms of trout Salmo letnica, endemic to the Balkan Lake Ohrid, reflects their reproductive isolation. Molecular Ecology. 13, 3633-3644 (2004).
- Karadjian, G., et al. Highly rearranged mitochondrial genome in Nycteria parasites (Haemosporidia) from bats. Proceedings of the National Academy of Sciences of the United States of America. 113 (35), 9834-9839 (2018).
- Morga, B., et al. Identification of genes from flat oyster Ostrea edulis as suitable housekeeping genes for quantitative real time PCR. Fish and Shellfish Immunology. 29 (6), 937-945 (2010).
- Delsuc, F., et al. Molecular systematics of armadillos (Xenarthra, Dasypodidae): contribution of maximum likelihood and Bayesian analyses of mitochondrial and nuclear genes. Molecular Phylogenetics and Evolution. 28 (2), 261-265 (2005).
- Wei, S., et al. Discovery and Biochemical Characterization of UDP-Glucose Dehydrogenase from Akkermansia muciniphila. Protein & Peptide Letters. 24 (8), 735-741 (2017).
- Gu, B., et al. Discovery and Biochemical Characterization of the UDP-Xylose Biosynthesis Pathway in Sphaerobacter thermophilus. Protein & Peptide Letters. 23 (12), 1103-1110 (2016).
- Duan, X. C., et al. Functional characterization of the UDP-xylose biosynthesis pathway in Rhodothermus marinus. Applied Microbiology and Biotechnology. 99 (22), 9463-9472 (2015).
- Vogelstein, B., Gillespie, D. Preparative and analytical purification of DNA from agarose. Proceedings of the National Academy of Sciences of the United States of America. 76 (2), 615-619 (1979).
- Song, H. B., et al. UDP-glucose 4-epimerase and β-1,4-galactosyltransferase from the oyster Magallana gigas as valuable biocatalysts for the production of galactosylated products. International Journal of Molecular Sciences. 19 (6), 1600 (2018).
- Gainey, P. A., Phelps, C. F. Uridine diphosphate glucuronic acid production and utilization in various tissues actively synthesizing glycosaminoglycans. Biochemical Journal. 128 (2), 215-227 (1972).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved