Method Article
その場で測定と細胞密度の相関、発光細菌の発光
要約
発光細菌クオラムセンシングなどのメカニズムのさまざまな光の生産を調節します。この手法では、生物発光における原位置調査および細胞密度に発光の相関をことができます。人工の発光性大腸菌システムはluxオペロン、ルクスのタンパク質及びその相互作用の特性をことができます。
要約
発光可能菌種のかなりの数があります。それらのすべては、同じ遺伝子クラスター、すなわちluxオペロンを共有します。この類似性にもかかわらずこれらの細菌は成長挙動、発光量や発光の規制のような特性の極度な変化を示します。ここで紹介した方法は、プレート リーダーを活用した時間の経過とともに細胞の成長と発光の発光強度の記録を組み合わせた新開発試金です。結果として得られる成長と発光特性は、クオラムセンシング規制など、それぞれの菌株の重要な機能にリンクできます。発光細菌の範囲の栽培は、特定の媒体 (例えば、人工海水媒体) を必要とし、温度を定義します。非発光の標準研究細菌エシェリヒア属大腸菌(E. 大腸菌)、扱いやすい一方で培養できます安価実験室規模の高い量。全体のルクスを含むプラスミドを導入することで大腸菌を利用するオペロン実験条件を簡素化することができます、さらに将来のアプリケーションの多くの可能性を開きます。大腸菌式を利用したすべてのlux遺伝子の発現クローニング ギブソンを介して発現プラスミドの建設と 7 lux遺伝子との 3 つのリブの遺伝子を含む 4 つのフラグメントの挿入によって達成されました。pET28a ベクトルにリブ luxオペロン。大腸菌によるlux遺伝子発現を誘導し、イソプロピル-β-D-thiogalactopyranosid (IPTG) また発光エシェリヒア属大腸菌の結果を介して制御することができます細胞。このシステムの利点は、クオラムセンシング規制制限と非標準成長条件と共に複雑な培地成分などを避けるために温度が定義されます。このシステムにより、 luxオペロンからそれぞれの遺伝子の排除もしくは、別の 1 つの細菌の緊張からluxAB遺伝子を交換、新規遺伝子のも添加によるlux遺伝子との相互作用の解析または蛋白質の複合体、 luxCDEなどを分析します。
概要
生物 (生物発光) による光の放射は、細菌、菌類、昆虫、線虫、魚、イカ1魅惑的なプロセスです。生物発光の発光は、発光反応を光エネルギー (「冷や光」) に化学エネルギーの変換が (部分的に)、から出ています。長鎖、490 nm2,で最大発光を伴う対応する酸への tetradecanal などの脂肪族アルデヒドの monooxygenation を触媒するヘテロ二量体の酵素ルシフェラーゼ発光細菌3。

図 1: 細菌のルシフェラーゼの一般的な反応スキーム。細菌のルシフェラーゼ (LuxAB) を触媒する長鎖の monooxygenation を用いたアルデヒド (CH3(2CH)n町) 削減フラビンモノヌクレオチド (FMNH2) と分子酸素 (O2) 製品を降伏酸 (CH3(2CH)nCOOH)、フラビンモノヌクレオチド (FMN)、水 (H2O)、490 を中心とした光の放射を長鎖 nm。この図の拡大版を表示するのにはここをクリックしてください。
この酸化時に放出されるエネルギーにより励起状態 FMN-4 a-水酸化、発光ルシフェリン4となります。細菌の生物発光、すなわち LuxCDABEG,に関係するタンパク質luxオペロンでエンコードされ、様々 な細菌の緊張2,5以上強く保存されています。遺伝子ルクサとluxBエンコード ヘテロ二量体型ルシフェラーゼ;ラックスのluxDのluxCの遺伝子産物、脂肪酸の還元酵素複合体でのコンポーネントフラビン還元酵素6 luxGエンコードします。生物発光の発光(e.g.,Photobacterium mandapamensis 27561) の数は、追加luxF遺伝子を運ぶ。LuxF がバインド珍しいフラビン誘導体 6 の-(3'-(R) ミリスチル量タンパク質である報告された)-FMN (myrFMN)7,8,9,10,11 ,12。リボフラビンの合成 (例えば、ribEBH) に責任がある遺伝子が識別されているし、さらに遺伝子発現が報告されているビブリオの特に発光のクォーラム センシング制御の役割を果たすこと・ フィシェリと採6,13。非常に節約された遺伝子順序にもかかわらず発光細菌は成長挙動、光発光の強度や発光2,5,14 の規則のような特性の高バリエーションを表示します。.
いくつか変更系統または部分を含んでいるプラスミッドまたはレポーター系としての生物発光を利用した全体luxオペロンが知られています。プロモーター活性を決定するなどのさまざまな環境や食品サンプル、発光共鳴エネルギー伝達 (ブレット)、細菌汚染の監視真核生物における感染症の生体内イメージングパイロシークエンスなどが確立された15,16,17だった。興味深いことに、3 人は最もよく使用されるシステムは、北米のホタル (Photinus pyralis)、線虫 (Photorhabdus luminescens)、腸管病原体と海のパンジー (Renilla に由来する発光レポーターニセフクロセンチュウ)。細菌の起源は、これらのシステムのどれもが、 lux遺伝子やオペロン細菌起源からの使用は応用研究16のより多くの関心を得ています。細菌由来生物発光蛋白質のより少なく豊富なアプリケーションは低く安定性のために主に、細菌の長寿派生発光蛋白質、海洋の生息地に関連することができます。海洋の生息地の生物発光バクテリアは標準的な実験室条件下耕作ではありません。これらの細菌には、特定の成長媒体と人工海水中と低成長/孵化温度 (例えば、 28 ° C) などの条件が必要です。
Luxオペロン特性または単一ルクスの比較を簡単に異なる発光菌株、 luxオペロンの発現と解析を標準化するためのメソッドの範囲の遺伝子が必要です。したがって、全体リブ luxオペロンを標準研究細菌エシェリヒア属大腸菌(E. 大腸菌) に統合するというアイデアが浮上しました。この目的のためギブソン アセンブリを特定の制限のサイトを必要とせず 1 つの発現ベクターに複数の線形、重複フラグメントを統合する便利なツールを証明しました。DNA 挿入が大きすぎる場合、このメソッドは適しても (e.g.,P mandapamensis 27561 luxCDABFEG ribEBH; ~オペロン サイズは 9 kb) を PCR によって増幅されます。最後にシーケンス検証するアセンブリ製品は適切なエシェリヒア属大腸菌に直接変換できるとluxオペロン複数の重なり合う部分に分けることができますし、1 つの発現プラスミドに組み立てられる高収率のシステムタンパク質発現18,19,20。大腸菌によるlux遺伝子発現を処理する簡単なだけでなく細胞の成長と発光の発光の記録を組み合わせた簡単な方法は確立に残った。ここで説明したメソッドには、その場で測定し、細胞密度の相関と発光細菌の発光ができます。
一方で、人工の発光エシェリヒア属大腸菌、様々 な発光細菌の規制とluxオペロン順序lux遺伝子の解析システムp. の全体リブ luxオペロンを含むmandapamensis 27561 と他の手、細胞密度と発光、その場で記録を組み合わせた新開発のプレート リーダー アッセイ様々 な細菌ルクスシステムについての詳細を得るために役立ちます。この基礎的特性とルシフェラーゼと関連酵素の比較は、安定性の向上と活動の既に確立されたレポーター系の代替可能性があります。
プロトコル
1. 設計、準備、およびluxオペロン大腸菌での発現
注: このセクションで使用される商業キットについて材料の表を参照してください。
- エシェリヒア属大腸菌にluxオペロンを転送する適切な制限のサイトおよび目的 (例えばpET28a; の抗生物質耐性遺伝子を持つ標準的なペット ベクターを選択します。NcoI XhoI、カナマイシン)。
- フラグメント デザインし、重複Photobacterium mandapamensis 27561 の DNA シーケンスに基づいてギブソン アセンブリのためのプライマー (GenBank: DQ988878.2)。
- テンプレートとして設計されたプライマーとPhotobacterium mandapamensis 27561 のゲノム DNA の分離と標準 PCR 反応を設定 (プライマーと条件の補足資料を参照してください)。
注: それぞれの菌株のゲノム DNA の単離は、PCR の有効性を向上させます。 - 回転カラム精製で PCR の製品を浄化します。
- 45 分の 37 ° C で NcoI XhoI と分離 pET28a ベクターの制限の消化力を実行します。
- 一直線に並べられたベクトルを浄化し、アガロースで、PCR のフラグメントのゲルの電気泳動およびそれに続く回転カラム精製します。
- 各フラグメントの一直線に並べられたベクトル DNA 濃度を決定し、プロトコル20,21に従ってアセンブリの最適量を計算します。
注: アセンブリの有効性フラグメント サイズと数に依存、製造元のプロトコル20,21によると調整することに。 - すべてのフラグメントと PCR チューブ内のバッファーを結合した後 1 h の 50 ° C で PCR 機でアセンブリ混合物をインキュベートします。
- 適切なエシェリヒア属大腸菌にエシェリヒア属大腸菌細菌プラスミドの変換のための標準的な変形のプロトコルに従って組み立てられたベクトル積を変換高収率プラスミド複製 (例えば、エシェリヒア属大腸菌TOP10 のシステムまたは XL 1)。
- 変換プレートと DNA の隔離のための新しい板の縞からコロニーを拾います。
- 標準的なプロトコルによるとプラスミド DNA を分離します。
- すべてのフラグメントを含むプラスミドの正しい組み立ての確認、すべての組み立てられたフラグメントの特異的なプライマーを使用して標準のプロトコルに従ってコロニー PCR をまず実行します。
- さらにコロニー PCR と後続 agarose のゲルの電気泳動に、適切なアセンブリと正しい DNA シーケンスを確認する DNA 塩基配列にすべての分離アセンブリ ベクトルを準備します。
- 適切なエシェリヒア属大腸菌にエシェリヒア属大腸菌細菌プラスミドの変換のための標準的な変形のプロトコルに従って検証されたプラスミドを変換高収率生産タンパク質 (e.g.,E 大腸菌BL21) システム。
注: は、表現のプロトコルの下に直接進みます。長期保管のグリセロール ストックの準備をお勧めします。
2. 変更されたエシェリヒア属大腸菌株の式
- LB 培地の適切なボリュームの接種、式の準備を一晩かけて培養 (ONC) (例えば、 100 mL) 組み立てられたプラスミッドまたはから直接、エシェリヒア属大腸菌BL21 細胞の事前に準備されたグリセロール ストックと、変換プレート。カナマイシン (50 mg/mL; pET28a の抗生物質耐性遺伝子) の 100 μ L を追加し、37 ° C でインキュベーター シェーカーで 120 rpm ONC を一晩インキュベートします。
- 主な表現文化を (例えば、 800 mL の LB 培地の)、ONC の 8 ml を接種し、カナマイシン (50 mg/mL) の 800 μ L を追加します。
- 37 ° C とインキュベーター シェーカーで 120 rpm で主な文化をインキュベート細胞密度 0.6 - 外径600に達するまで 0.8 (約 2.5 時間)。
- 28 ° C の孵化温度を下げる
- 最終濃度 0.1 mM の IPTG を追加することでタンパク質の発現を誘導します。
注: 実証テストは、28 ° C への温度の低減が最も高い光強度を与えたことを示した。 - 彼らは輝く (約 1 h) を開始するまでは、細胞を観察します。
注: 式の目的に応じてセルが次の日まで栽培されて、その後、収穫され、やセルをすることができます (最大 48 h) 輝いている限り、揺れ続けた。細胞とその蛋白質の浄化の収穫は、標準的な手順に従って行うことができます。
3. 細菌の発光式
メモ: 発光菌株の成長と光の生産の特定の成長媒体/人工海水水媒体が必要です。
- 人工海水水媒体、2 つ別々 に用意された培地成分の構成を準備します。
注: 人工海水水媒体の作製は、元プロトコル22から適応されました。次に掲げる金額は、1 L の液体培地または寒天培地 1 L 用です。- 人工海水水媒体のための次の塩で重量を量る: 28.13 g NaCl、KCl、0.77 g 1.60 g CaCl2 ·2 H2O、4.80 g MgCl2 ·6 H2O、0.11 g NaHCO3、および 3.50 g MgSO4 ·7 H2o.
- 蒸留水 1 L を追加し、すべてのコンポーネントを溶解します。
- LB 培地の以下の成分で重量を量る: 10 g 酵母エキス、ペプトン 10 g、寒天、追加 20 g 寒天。
- 250 mL の水道水を追加し、コンポーネントを溶解します。
- 両方のオートクレーブは、20 分の 121 ° C で個別にメディアを準備しました。
- 寒天、オートクレーブから出した後直接人工海水水媒体の 750 mL と 250 mL の LB 培地を組み合わせるし、プレートを準備します。
- 液体培地オートクレーブ後に直接、または冷却したとき人工海水水媒体の 750 mL で 250 mL の LB 培地を組み合わせます。
注意: 人工の海の水の中は塩の沈殿物によって濁ったことがあります。
- 人工海水水培地寒天培地プレート上の発光菌株を連勝し、24-30 ° C で一晩インキュベート
注: 菌株の長い時間の記憶は通常細菌文化のグリセロールを凍結を実現します。系統の縞が融解後の生育の遅れ位相のための液体文化のため使用する前に、すべての制服の開始条件を保証するためにまず寒天プレート上にする常に必要があります。 - 100 mL 人工海水水培地プレートから単一コロニーを接種して、ONC を準備します。24-30 ° C そしてインキュベーター シェーカーで 120 rpm で ONC を一晩インキュベートします。
- 人工海水水媒体 ONC の 8 mL と 800 mL を接種します。
- 24-30 ° C そしてインキュベーター シェーカーで 120 rpm で細菌の細胞を孵化させなさい。
メモ: 発光細菌の光強度分布は温度に強く異なります。それぞれの菌株の軽い生産の調節機構、によって発光は約 1-6 h 後始めるかもしれない。 - 彼らは輝く (約 1-6 h) を開始するまでは、細菌の細胞培養を観察します。
注: 式の目的に応じてセルが次の日まで栽培されて、その後、収穫され、やセルをすることができます彼らが輝いている限り、揺れ続けた。細胞とその蛋白質の浄化の収穫は、標準的な手順に従って行うことができます。
4. 発光菌株と変更されたエシェリヒア属大腸菌株の活性生体内で
注: 系統の長い時間の貯蔵は通常細菌文化のグリセロールを凍結からそれらです。系統の縞が融解後の生育の遅れ位相のための液体文化のため使用する前に、すべての制服の開始条件を保証するためにまず寒天プレート上にする常に必要があります。
- 連勝目的生物発光菌株または変更されたエシェリヒア属大腸菌寒天プレートのひずみし、28 ° C で一晩インキュベートします。
注: インキュベーション温度ひずみに変わることができるし、経験的評価されるが。発光菌株を比較することがあり、変更された大腸菌、成長条件が一致している必要。 - 寒天版から単一コロニーをそれぞれのひずみと媒体の 3 mL を接種して 28 ° C で約 1-2 時間のインキュベーター シェーカーで 180 rpm セルを孵化させなさい。
- 1:10 の細胞密度を測定 650 に液体培養の希釈 nm。比と 0.05 の OD650 1 mL 文化の体積を計算します。
注意: 後続版リーダーの試金は 650 で細胞密度を決定系統の発光によって干渉を避けるために nm。 - 文化および媒体の計算された容積のピペット ガラス底の 24 ウェル黒壁板に。変更されたエシェリヒア属大腸菌の系統、カナマイシン (pET28a ベクトルの抗生物質耐性) の 1 μ L および IPTG (遺伝子発現の誘導) の 1 μ L をサンプルに追加します。測定中に蒸発を避けるためにプレートに蓋を配置します。
注:エシェリヒア属大腸菌培養による全体luxオペロンを含む pET28a ベクトルを失わなかった取得しないことには、カナマイシンに追加しなければならない各大腸菌サンプル プレートの井戸と大腸菌の光生産を確保するために細胞を測定することができます、IPTG によって遺伝子発現を誘導する必要があります。測定やクロストーク干渉を避けるためには、ガラス板、透明蓋黒壁も板は、最良の結果を示した。しかし、クロストークが見られる、よく位置は慎重に選択する必要があります。 - プレート リーダーで測定を開始します。
注: プレート リーダー プロトコルは専用に開発されたスクリプトに基づいてこの試金のため (補足資料を参照してください) を組み合わせた 2 つの測定、吸光度、発光。データ ポイントが永久的な振動測定と 28 ° c. の一定した温度の間 10 分毎を集め
結果
- LuxCDABFEG - luxオペロンの遺伝子順序は、さまざまな系統2,5,14で保存性が高い。プラスミッドの設計、シーケンス情報発光菌株Photobacterium mandapamensis 27561 から撮影された」とその遺伝子の順序が同じに保たれた、また、単一の遺伝子間の非コード シーケンスが考慮されました。応用ギブソン クローニング法の概要は、図 2で描かれています。シーケンスを重複 20 40 塩基対で合計、 luxCDAB、 luxF、 luxEG、 ribEBHの 4 つのフラグメントが生成されました。ギブソン アセンブリ20のすべての手順を実行した後は、DNA の配列は、すべてのフラグメントを含むプラスミドの適切なアセンブリを確認しました。リブ luxオペロンを含む最終組立製品 pET28a のベクトル マップは、図 3に描かれています。この変更された pET28a ベクターの重要な利点の使用率は、大腸菌の成長条件を標準化、IPTG と誘導を制御します。
それぞれのセル密度と発光細菌の発光を測定するには、プレート リーダーによる方法を開発しました。光強度と細胞密度の単一測定スクリプトを組み合わせるプレート リーダーのメソッドが生成されました。この新しいスクリプト有効外径650の測定、光強度のユーザーのすべての 10 分定義時間フレームは、それぞれの分析 (例えば、 10 h) に使用される細菌の世代時間を調整することに。光学濃度の測定は、650 で行われた nm 発光との干渉を避けるために。概念の健康と大腸菌の細胞の正しい成長挙動を保証するために、証拠として参照の測定を行った。図 4 大腸菌BL21 細胞、大腸菌BL21 セル空 pET28a ベクトルとリブ luxオペロンと pET28a ベクターを含む大腸菌BL21 細胞の比較で挿入が掲載されています。後者のひずみの T7 プロモーターのため、水漏れしたため発光を分析する IPTG は追加されません。すべて 3 参照測定は、3 つの成長段階 (遅れ、指数、および固定相) と s 字状の成長曲線を示します。発光にルクス リブオペロン挿入開始、測定とは対照的に、pET28a ベクターを含む大腸菌BL21 セルのみ発現が光と IPTG の添加による誘導は、非誘発細胞の 30 分後が出力されます。約 5 時間後に照らし、はるかに低い発光を示すのみで開始 (約 4 倍) 誘導システムと比較して。
図 5は、エシェリヒア属大腸菌および発光菌株P. mandapamensis 27561、LB 培地または人工的な海の水の中のいずれかで表されるluxオペロンの光強度と成長曲線の比較小説を使用してその場で法を確立しました。これらの細菌を比較するには、28 ° C の孵化の温度で測定を行った変更されたエシェリヒア属大腸菌の伸び歪 LB 培地、低温で発光菌株が、人工海水水媒体だけでなく、この温度低下は重大であります。この温度依存性は、 P. mandapamensis 27561大腸菌よりもはるかに高い細胞密度に示す図 5Aに表示されます。さらに、大腸菌、しばらく LB 培地生成可能高い細胞密度、自然発光細菌株の人工の海の水の中は最寄りと発光のために不可欠です。記録された細胞の密度は図 5Bに示すように、それぞれの光の強さと相関します。注目すべき、エシェリヒア属大腸菌の生物発光の細胞に達するような異なる時点で記録された最高の強度が両方 LB 培地として人工海水媒体の排出マキシマの光します。この観察と対照をなしてP. mandapamensis 27561 は LB 培地での非常に減らされた成長率で実行可能が細菌のセルがすべて (図 5B) の光を出さない。人工的な海の水の中は、 P. mandapamensis 27561 は大腸菌より低い 200 のほぼ要因である、1 秒あたり約 1 × 104カウントで発光の最大を示しています。図 5Cは、OD で生物発光を正規化する、相対のライト ユニットを表します。これらの結果の確認だけでなくプラスミドの挿入だったE. 大腸菌成功し、機能にluxオペロンを含む、大腸菌を変更、これも歪みがさらに高い発光を有効な代替手段得られ、複雑な海水などのマリーナ菌の細菌の生物発光の制限なく中型、低い温度。
さらに、発光 (図 6) の長寿を分析するエシェリヒア属大腸菌によるルクス肋骨遺伝子発現 24 時間以上の長期的な測定を行った。発光は、19.5 時間続いたが徐々 に減少 (例えば、P. mandapamensis 27561) 菌株より長く非常に低い発光 10 時間後の結果が観察されました。
開発分析の限界を示すためには、図 7に示します 3 つの発光細菌の緊張、すなわちPhotobacterium mandapamensis S1 Photobacterium mandapamensis TH1、ビブリオの測定結果harveyi 14126。最初のひずみ (S1)、メソッドが非常によく動作し、約 2 × 106の最大発光強度を示しています。他の 2 つの系統 (TH1 および 14126) 両方によって生成される光の強度は、使用機器の設定の検出の制限を超えたために、極大の光の強度を決定できません。これらの 2 つの系統の開発メソッド (スクリプト) に定義されているゲイン値は高すぎる設定されました。それにもかかわらず、生物発光活性の発症を相互に比較できます。P. mandapamensis TH1 とP. mandapamensis S1 開始後約 1 時間で、OD650値 0.1 が輝く - 0.2、それぞれ。対照的に、 harveyi V. 14126 1.0 の OD650値で約 5.5 時間後に光を放出し始めます。発光の観測の発症の生物発光と同様、OD の急激な増加と一緒に伴われます。V. harveyi 14126 の発光クオラムセンシング規制の基礎となる、したがって特定の細胞の密度図 713で明らかに観察することができますluxオペロンのアクティベーションを許可する知られています。この結果は、この小説はその場でプレート リーダー試験それは、発光細菌を簡単に比較しても約クオラムセンシング規制かどうかを決定することでこれらの系統の調節機構を定義があることを示してください。かどうかを観察します。
IPTG 誘導後luxオペロンを含む組み立てられた pET28a プラスミド大腸菌BL21 細胞の表現文化の例を図 8に示します。導入後約 1 時間後、大腸菌の細胞は、青緑色に輝くを開始します。図 8A 大腸菌式を示しますは、光の中で撮影した文化と図 8Bは、暗闇の中で同じ文化を与えます。図 8Cは、 P. mandapamensis S1 細菌生物発光の同じ青緑色特性の暗闇の中で光ると人工海水水媒体の寒天プレートを示しています。
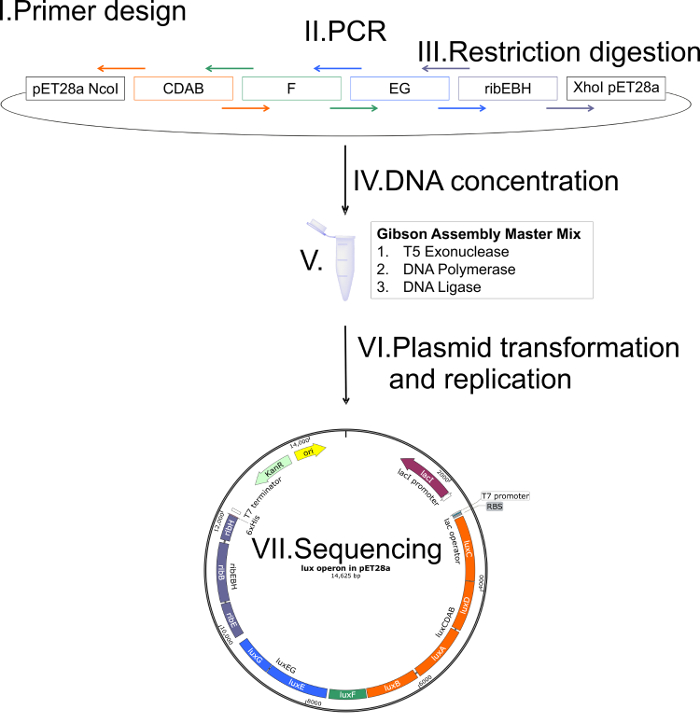
図 2: クローニング戦略応用ギブソンの模式図。ステップ (I):重複プライマー (色付きの矢印; ca. 20 40 塩基対が重なっている)、設計されています。重複するプライマーは、1 つのフラグメントのそれぞれ 5' と 3' の地域と隣接するセグメントのそれぞれの 3' と 5' の領域から成る焼鈍のシーケンスを含みます。ステップ (II):標準 PCR 反応を介してアセンブリの設計のフラグメントが生成されます。ステップ (III):制限の消化力 (例えばNcoI、XhoI) によって目標のベクトルが線形化されます。ステップ (IV):すべてのフラグメントの一直線に並べられたベクトル DNA 濃度は、(製造元のプロトコル) によるとギブソン アセンブリの適切な濃度を調整する決定する必要があります。ステップ (V):すべてのフラグメントと最適化された DNA 濃度の一直線に並べられたベクトルのギブソン アセンブリ マスター ミックス (T5 エキソヌクレアーゼ、DNA ポリメラーゼおよび DNA のリガーゼ) が組み合わされて 1 h. 50 ° C で培養したのステップ (VI):アセンブリ製品の変換適切なエシェリヒア属大腸菌に標準プロトコルによると高収率プラスミド複製 (例えば、エシェリヒア属大腸菌TOP10 や XL 1) のひずみ。ステップ (VII):プラスミッドの正しい組み立ての確認、組み立てられたプラスミドの DNA の配列は、実行しています。この図の拡大版を表示するのにはここをクリックしてください。

図 3:ルクス リブオペロンを含む pET28a のベクター マップ。P. mandapamensis 27561 のリブ luxオペロンは、元の遺伝子順序 (luxCDABFEG ribEBH) で pET28a の多重クローニング サイトに挿入されます。XhoI と NcoI クローニングに使用制限のサイトです。オペロンのギブソン組立のフラグメントがオレンジでluxCDAB緑のluxF 、 luxEGブルー、ラベンダー; ribEBH遺伝子フラグメント内では、個別のボックスとして表示されます。各遺伝子オペロンの間非コード シーケンスは、応用のクローニング法に含まれます。全体リブ luxオペロンを含む pET28a の最終的なプラスミド サイズは 14,625 塩基対です。この図の拡大版を表示するのにはここをクリックしてください。
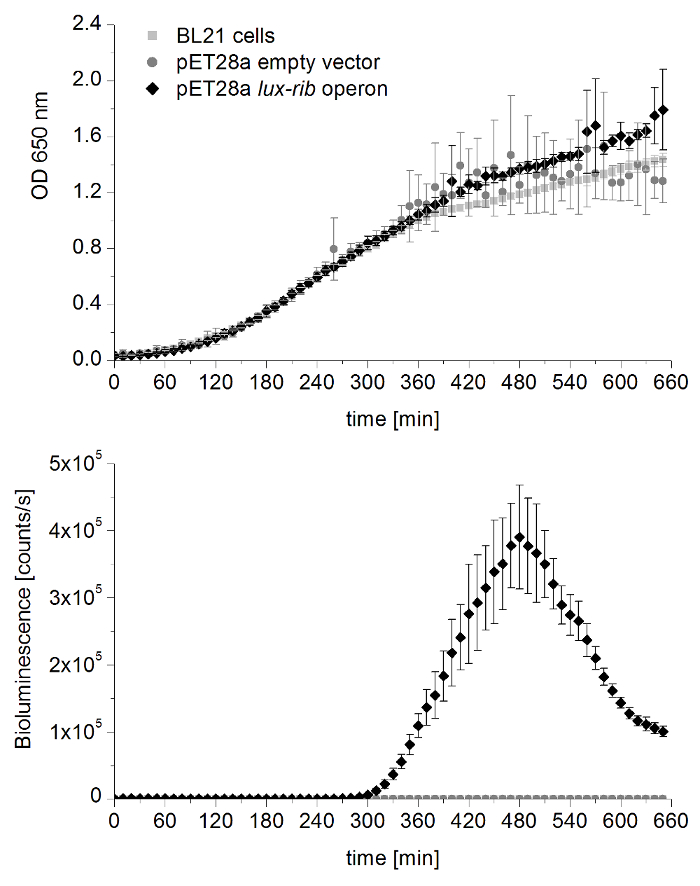
図 4:成長曲線と指令系統の光強度の比較。OD 650 nm 1 秒あたり数の発光強度測定 28 ° C で 10 時間以上 10 分毎すべての測定は、各 4 つの技術的な複製と 3 つの生物学的複製の平均値です。エラーバーは標準偏差を表しています。エシェリヒア属大腸菌BL21 細胞 (灰色の正方形)、エシェリヒア属大腸菌BL21 セル空 pET28a ベクトル (灰色の円) および大腸菌BL21 セル pET28a ベクタールクス リブオペロン挿入 (ブラック ダイヤモンド) に行ったエシェリヒア属大腸菌細胞の正しい成長挙動を保証します。この図の拡大版を表示するのにはここをクリックしてください。
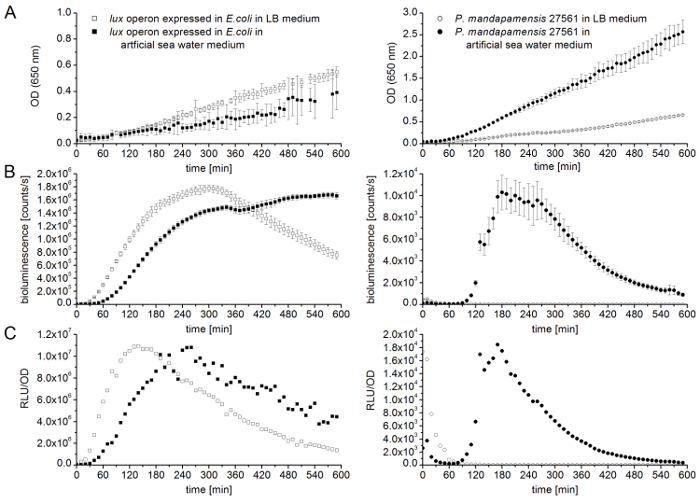
図 5:エシェリヒア属大腸菌(正方形)、 P. mandapamensisでluxオペロンの光強度と成長曲線の比較表現 27561 (円) LB 培地 (開いているシンボル) または人工海水水媒体 (塗りつぶされたシンボル).すべての測定は、各 4 つの技術的な複製と 3 つの生物学的複製の平均値です。エラーバーは標準偏差を表しています。28 ° C の孵化の温度ですべての実験を行った(A)光学濃度 (OD) 観測結果 650 nm は LB 培地と人工海水培地に 10 h. E. 大腸菌 luxオペロン式 (左側のパネル) P. mandapamensis 27561 (右側のパネル) と比較の 10 分毎を行った。セル密度が 650 に決定される nm 発光干渉を避けるために。(B)測定光強度 (発光 [カウント/s]) の行った 10 分毎 10 h. E. 大腸菌 luxオペロン式 (左のパネル)、LB のP. mandapamensis 27561 (右側のパネル) と比較しての中・人工の海水媒体。(C)相対光強度 (RLU/OD)エシェリヒア属大腸菌(左のパネル)、 P. mandapamensis 27561 (右側のパネル) で表されるluxオペロンの細胞密度に発光を正規化することで決定されます。この図の拡大版を表示するのにはここをクリックしてください。

図 6:エシェリヒア属大腸菌の光強度と成長曲線の比較による 24 時間のlux遺伝子発現OD 650 nm、1 秒あたりの数の発光強度は 10 分毎に 28 ° C で 24 時間以上を測定しました。すべての測定は、各 4 つの技術的な複製と 3 つの生物学的複製の平均値です。エラーバーは標準偏差を表しています。さらに、細胞密度によって発光の正規化、相対光強度 (RLU/OD) が表されます。この図の拡大版を表示するのにはここをクリックしてください。
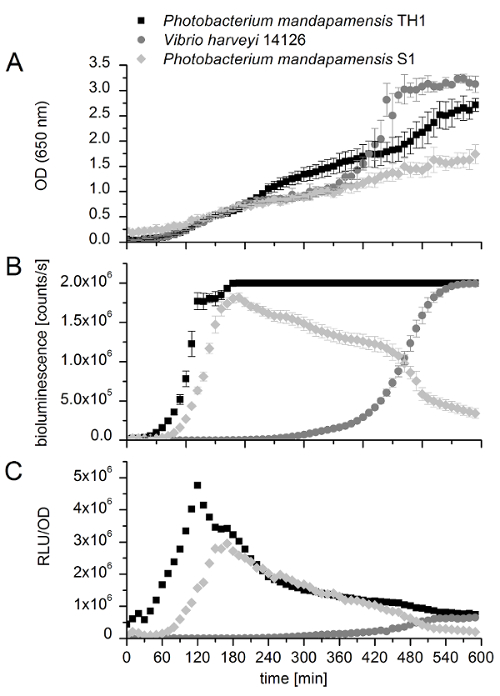
図 7: 潜在的なクオラムセンシングで規制を評価する発光細菌の比較。発光と細胞密度 10 時間 10 分を測定し、各 4 つの技術的な複製と 3 つの生物学的複製の平均値を表します。エラーバーは標準偏差を表しています。Photobacterium mandapamensis th1 細胞の測定 (黒の正方形)、採14126 (灰色円) と (グレー ダイヤモンド) を互いに比較したPhotobacterium mandapamensis S1(A) 650 の光学濃度 (OD) を示しています nm、 (B)光強度 (発光 [カウント/s])、および(C)相対光強度 (RLU/OD)。この図の拡大版を表示するのにはここをクリックしてください。
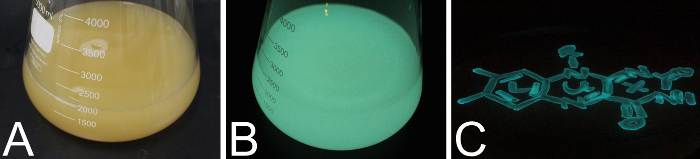
図 8: 発光液体メディアと寒天。(A)光で撮影 pET28a luxオペロン プラスミドを発現する大腸菌BL21 細胞を接種した 2 L LB 培地 5 L フラスコ。(B)同じカルチャ (A) のように暗闇の中で撮影します。写真 A と B の発現誘導後約 2 時間に撮影されました。(C)水培地寒天培地の暗闇の中で撮影したP. mandapamensis S1 の縞の文化人工海水。この図の拡大版を表示するのにはここをクリックしてください。
ディスカッション
P. mandapamensis 27561 の全体リブ luxオペロンを構成する 4 つの断片を含む発現プラスミドの建設は、ギブソンのクローニングを介して達成されました。この大腸菌基づくlux遺伝子発現により光を放出するエシェリヒア属大腸菌のセル。このシステムの大きな利点は、クオラムセンシング規制および標準成長の状態を回避します。
ギブソン クローニング法を使用しての利点は、複数の線形 DNA のフラグメント、高い柔軟性と特定の制限サイト18,19,20は不要の簡単な組み立てです。このメソッドはluxオペロン、単一の遺伝子または遺伝子群を除く新しい遺伝子導入または別の 1 つの緊張から遺伝子の交換を簡単に変更することができます。この方法の適用の前提条件は、それぞれluxオペロンの遺伝子の塩基配列の可用性です。発光細菌の数が少ない場合のみ知られているおよび/または利用可能なそれぞれluxオペロンの完全な DNA 順序です。これらのシーケンスの多くが散弾銃の配列を使用して生成されたので断片化しています。調査P. mandapamensis 27561 場合luxオペロンの完全な DNA 配列が知られており、NCBI 遺伝子データベースから利用できる (GenBank: DQ988878.2)。
ギブソン アセンブリのプロトコルの 1 つの重要なステップは、単一の断片の DNA 濃度の計算です。0.2 - を使用することが推奨されたメーカーのアセンブリ プロトコルで 0.5 pmols と 4-6 片20,21の 20 μ L の容量。どこluxCDABFEG ribEBHは 4 断片で構成されて、私たちのケースで濃度と総ボリュームあった 0.1 pmol 45 μ L、PCR 産物の収量によって。一方で濃度が減少することと、他の一方で、ボリュームが増加しなければならなかった。それにもかかわらず、アセンブリは非常によく働いたし、DNA の配列が最初の試行で正しい挿入を確認しました。この発見は、修正18,19,20をすることができます簡単に私たちの調査のための堅牢で適切な方法としてギブソン アセンブリを確認しました。
新しく設立されたプレート リーダー アッセイは、メソッドを扱いやすいです。それによりの簡単な主要な解析、新しい (非) 既に知られている発光菌株と与える光生産 (例えば、低細胞密度で発光 lag) の調節機構に関する最初のヒントや。さらに、光との組み合わせでの成長条件は、成長培地や温度を変更するだけで簡単に評価できます。
プレート リーダーで測定は 28 ° C でこの異常な設定の理由は、30 ° C の上の温度が成長や発光の少ない、またはまったくもつながる、発光細菌の温度感度です。プレート リーダーがアクティブな冷却システムの行方不明の制限時間の経過とともに定義された温度を保つことができることに注意してください。したがって、周囲温度は、測定の温度以下でなければなりません。
概念の証拠として発光株大腸菌構築 1 つの手と参照の測定 (図 4) P. mandapamensis (図 5) その一方で行ったし、の比較確認この新しく設立されたシステムの信頼性。さらに、長期的な測定は、発光 (図 6) の寿命を保証しました。OD 値が適切な希釈で測定の必要が使用される測定装置 (現在の場合で約 15 時間) の特性に応じて、特定の光学密度上信頼性の高いではないことを考慮する 1 つが、検出器の線形範囲は。OD 値のこれらの高い差異は、信頼できないこれらの長い時間の測定を行います。したがって、10 h などの短い測定は、ここで報告した実験のセットアップを使用して推奨されます。
図 7に確立されたプレート リーダー アッセイの限界を見ることができます。難易度は、測定の適切な設定を見つけることです。'価値 'を得る写真増倍管 (PMT) の感度を調整します。ゲインが高利得要因が信号を増加することを意味、光電子増倍管の信号の増幅です。ゲインが低すぎる設定されて、信号対雑音比が大きくなり、「低」の輝く発光細菌の光強度ができない場合表示されないデータ ゼロに近いより (信号) を測定します。生物発光アッセイの課題は、すべての菌株の計測結果は器械の範囲内にとどまるので、ゲインを設定することです。さらに、発光の測定範囲測定間隔時間によって異なります (例えば1 のための 2,000,000 の最大 s)。
ゲインは、経験的テストし、この手法の確立に使用する特定のプレート リーダー用に選択された、2800 に設定されました。使用ゲイン設定により最大放射光の記録、発光P. mandapamensis S1、オーバーフローすることがなく、緊張のP. mandapamensis th1 細胞および、 P. mandapamensis 27561、エシェリヒア属大腸菌システムHarveyi V. 14126 ゲインが高すぎます。したがって、これらの後者の系統は検出限界を超える、本当に最大の光強度を測定できません。この技術的な制限は、成長条件と細胞密度に匹敵するでしょうが、最大発光高バリエーションを表示発光細菌の比較をできない可能性があります。
経験的評価が分析された菌株使用のウェル プレート内の位置します。ガラス底を持つ黒のウェル プレートを使用した、サンプル間のクロストークが観察されました。特定の系統の光の強度は非常に高い、すべて近隣の井戸が偽肯定的な発光を (例えば、空白) が表示されます。したがって、2 つの異なる系統を個別に、または互いにある空間的な分離と測定することが重要です。
多くはluxオペロンの部分を含む知られているエシェリヒア属大腸菌株を変更、主にアプリケーション指向15,16,17が既にあります。ここで説明する方法は、基礎研究、たとえば各lux遺伝子を別々 に分析の可能性を目指しています。生物発光の研究には、長い歴史がありますが、まだ多くの未解決の問題があります。大腸菌システムとプレートのそれ以上の適用を処理するは簡単で埋め込まれた複合体を除く、 luxオペロンの遺伝子を導入または別株の遺伝子をluxAB遺伝子を交換やタンパク質の分析リーダーの試金、それは規制プロセスとlux遺伝子の機能に関する詳細を得ることが可能かもしれない。
開示事項
著者が明らかに何もありません。
謝辞
プレート リーダーの自己書かれたスクリプトの確立に彼のサポートのため Wladislaw ・ マイヤー (BMG Labtech GmbH) に感謝したいです。この作品はオーストリア人によって支えられた「フォン ツア青少年 der wissenschaftlichen 上海虹橋」(FWF) 午後 (P24189) と博士後期課程の「DK 分子酵素学」(W901) 午後に。
資料
| Name | Company | Catalog Number | Comments |
| NEBuilder High-Fidelity DNA Assembly Cloning Kit | New England Biolabs Inc. | E2621S | for 10 rxn dx.doi.org/10.17504/protocols.io.cwaxad |
| Phusion Polymerase | Thermo Scientific | F530S | |
| Q5 High Fidelity DNA Polymerase | New England Biolabs Inc. | M0491S | |
| GeneJet Genomic DNA Purififcation Kit | Thermo Scientific | K0721 | for 50 rxn |
| Restriction enzymes and buffer (NcoI, XhoI) | New England Biolabs Inc. | R3193S/R0146S | |
| Wizard SV Gel and PCR Clean-Up kit | Promega | A9282 | for 250 rxn |
| Monarch DNA Gel Extraction Kit | New England Biolabs Inc. | #T1020S | for 50 rxn |
| GeneJet Plasmid Miniprep Kit | Thermo Scientific | K0503 | for 250 rxn |
| GoTaq G2 DNA Polymerase | Promega | M7841 | |
| 24-well black sensoplate with glass bottom | Greiner Bio One | 662892 | |
| FLUOStar Omega plate reader | BMG Labtech | ||
| OPTIMA/Mars Analysis Software | BMG Labtech | Version 2.20 | |
| Microsoft Excel 2010 | Microsoft | ||
| Multitron Standard Incubator Shaker | Infors AG |
参考文献
- Widder, E. A. Bioluminescence in the Ocean: Origins of Biological Chemical, and Ecological Diversity. Science. 328 (5979), 704-708 (2010).
- Dunlap, P. Bioluminescence, Microbial. Encycl Microbiol. , 45-61 (2009).
- Ulitzur, S., Hastings, J. W. Evidence for tetradecanal as the natural aldehyde in bacterial bioluminescence. Proc Natl Acad Sci USA. 76 (1), 265-267 (1979).
- Kurfürst, M., Ghisla, S., Hastings, J. W. Characterization and postulated structure of the primary emitter in the bacterial luciferase reaction. Proc Natl Acad Sci USA. 81, 2990-2994 (1984).
- Dunlap, P. Biochemistry and genetics of bacterial bioluminescence. Biolumin Fundam Appl Biotechnol. 1, 37-64 (2014).
- Meighen, E. A. Bacterial Bioluminescence: Organization, regulation, and application of the lux genes. FASEB J. 7, 1016-1022 (1993).
- Moore, S. A., James, M. N. G., O'Kane, D. J., Lee, J. Crystallization of Photobacterium leiognathi non-fluorescent flavoprotein with limited sequence identity to bacterial luciferase. J Mol Biol. 224, 523-526 (1992).
- Moore, S. A., James, M. N. G., O'Kane, D. J., Lee, J. Crystal structure of a flavoprotein related to the subunits of bacterial luciferase. EMBO J. 12 (5), 1767-1774 (1993).
- Moore, S. A., James, M. N. G. Common structural features of the luxF protein and the subunits of bacterial luciferase: Evidence for a (βα)8 fold in luciferase. Protein Sci. 3, 1914-1926 (1994).
- Moore, S. A., James, M. N. G. Structural refinement of the non-fluorescent flavoprotein from Photobacterium leiognathi at 1.60 Å resolution. J Mol Biol. 249, 195-214 (1995).
- Kita, A., Kasai, S., Miyata, M., Miki, K. Structure of flavoprotein FP390 from a luminescent bacterium Photobacterium phosphoreum refined at 2.7 Å resolution. Acta Crystallogr Sect D Biol Crystallogr. 52 (1), 77-86 (1996).
- Bergner, T., et al. Structural and biochemical properties of LuxF from Photobacterium leiognathi. Biochim Biophys Acta - Proteins Proteomics. 1854 (10), 1466-1475 (2015).
- Defoirdt, T., Boon, N., Sorgeloos, P., Verstraete, W., Bossier, P. Quorum sensing and quorum quenching in Vibrio harveyi: Lessons learned from in vivo work. ISME J. 2 (1), 19-26 (2008).
- Meighen, E. Genetics of bacterial bioluminescence. Annu Rev Genet. 28, 117-139 (1994).
- Kelkar, M., De, A. Bioluminescence based in vivo screening technologies. Curr Opin Pharmacol. 12 (5), 592-600 (2012).
- Waidmann, M. S., Bleichrodt, F. S., Laslo, T., Riedel, C. U. Bacterial luciferase reporters: The Swiss army knife of molecular biology. Bioeng Bugs. 2 (1), 8-16 (2011).
- Wilson, T., Hastings, J. W. . Bioluminescence living lights, lights for living. , (2013).
- Gibson, D. G., Young, L., Chuang, R. Y., Venter, J. C., Hutchison, C. A., Smith, H. O. Enzymatic assembly of DNA molecules up to several hundred kilobases. Nat Methods. 6 (5), 343-345 (2009).
- Gibson, D. G., Smith, H. O., Hutchison, C. A., Venter, J. C., Merryman, C. Chemical synthesis of the mouse mitochondrial genome. Nat Methods. 7 (11), 901-903 (2010).
- Gibson, D. G. Enzymatic assembly of overlapping DNA fragments. Methods Enzymol. 498, 349-361 (2011).
- . . NEB NEBuilder HiFi DNA Assembly Reaction. , (2015).
- Atlas, R. M. . Handbook of Microbiological Media, Third Edition. , (2004).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved