Method Article
の世代
要約
エンテロバクター属 。 YSUは、グルコース最小塩培地中で成長する。栄養要求株は、ランダムに宿主ゲノムに自身を挿入するトランスポでそれを変換することによって生成される。突然変異体は最小培地への複合培地からレプリカプレーティングによって発見されています。中断された遺伝子は、遺伝子のレスキューおよび配列決定により同定される。
要約
栄養細菌は、炭素及びエネルギー源として使用される培地グルコース(M-9培地)を補充したM-9最小塩、上で成長する。栄養要求株は、トランスポを用いて生成することができる。このプロトコルで使用される市販のTn 5由来のトランスポはR6Kγ複製起点を含むDNAの線形セグメント、カナマイシン耐性遺伝子から成る二つのモザイク配列は、トランスポザーゼ結合部位として機能する、終了する。 DNA /トランスポザーゼタンパク質複合体として提供されるトランスポは、原栄養株、 エンテロバクター属にエレクトロポレーションによって導入される。 YSUし、無作為に、このホストのゲノムに自身を内蔵しています。形質転換体をカナマイシン(LB-館)を含む寒天プレートルリア - ベルターニ上にプレートレプリカとカナマイシン(M-9館)を含むM-9培地寒天プレート上にある。 M-9館プレート上LB-館プレート上で増殖ではなく、形質転換体は、栄養要求株であると考えられている。精製したゲノム栄養要求株からのDNAを部分的に消化されたライゲーションおよびpir +大腸菌(E. coli)株に形質転換する。 R6Kγ複製起点は、プラスミドがPIR + E.内で複製することができますcoli株 、およびカナマイシン耐性マーカープラスミドの選択を可能にする。各形質転換体は、中断された染色体領域により隣接トランスポゾンを含む新たなプラスミドを保有する。サンガー配列決定および基本的なローカル配列検索ツール(BLAST)が中断された遺伝子の推定アイデンティティを示唆している。このトランスポ突然変異誘発戦略を使用して、3つの利点がある。まず、ホストによってトランスポザーゼ遺伝子の発現に依存しない。第二に、トランスポはエレクトロポレーションによってではなく、コンジュゲーションによって、または形質導入によって、標的宿主に導入されるため、より効率的である。第三に、R6Kγ複製起点は、それが簡単に突然変異したグラムを同定することを可能にする部分的組み換えプラスミドにおいて回収されるエン。この技術は、 エンテロバクター属の他の特性に関与する遺伝子を調べるために使用することができる。 YSUまたは細菌株の幅広い種類の。
概要
栄養細菌は、培地中の生合成1は 、アミノ酸、核酸、ビタミンのような前駆体を生成するために、中心炭素代謝の経路を介してグルコースに変換し、グルコース(M-9培地)を含有するM-9最小塩で成長する。 M-9培地は、炭素源およびエネルギー源としての硫黄源、およびグルコースなどの緩衝液およびリン源としての窒素源、ナトリウム、カリウム、リン酸アンモニウム、塩化マグネシウム、硫酸を含んでいる。ルリア - ベルターニ(LB)培地トリプトンからビタミン及び酵母抽出物からの成長因子のアミノ酸が豊富である。これは、M-9培地上で生育に必要なアミノ酸、ビタミンおよび他の成長因子を合成することができない栄養要求株の増殖を支持する。栄養要求株は、M-9培地中でLB培地ではなく、成長するのに対し、このようにして、原栄養は、LBとM-9培地中で成長します。細菌の栄養集団に変異を導入し、栄養要求性表現型を引き起こす変異した遺伝子を同定することにより、経口であるバクテリア株中での代謝の理解を得るためにssible。
トランスポゾン突然変異誘発は、M-9培地中のグルコース上での増殖に必要な遺伝子の多くを同定するために用いることができる。トランスポゾンは宿主ゲノム2に無作為に自分自身を挿入します。 M-9培地寒天プレート上にLB寒天プレートおよびそのめっきレプリカ上のトランスポゾン形質転換体をスポットすることにより、栄養要求株をスクリーニングすることが可能である。中断の遺伝子は、遺伝子の救助を介して同定されている。この研究は、市販のTn 5はトランスポゼース蛋白質と混合線形トランスポゾンDNAセグメントのソリューションとして出荷されているトランスポを由来使用しています。 DNAセグメントは、トランスポザーゼ遺伝子を欠くが、カナマイシン耐性遺伝子、R6Kγ複製起点とセグメント3,4の各端に結合トランスポザーゼのためのDNA配列である2モザイクモチーフを含む。トランスポザーゼタンパク質は、DNA、単独のDNAセグメントに直接添加されているのでトランスポゾンとして定義され、DNA /トランスポザーゼタンパク質複合体は、トランスポとして定義される。トランスポはカナマイシン感受性ホスト( 図1A)にエレクトロ5によって変換される。カナマイシン(LB館)を含有するLB寒天プレート上で増殖したコロニーを、カナマイシン(M-9館)栄養要求株であるを含むM-9培地寒天プレート上で増殖しなかっトランスポゾン挿入( 図1B)、レプリカプレーティングし、形質転換体(Figireを持つ図1C)。変異体からのゲノムDNAを精製し、部分的に、4塩基切断制限エンドヌクレアーゼ、BFU CI( 図1D)を用いて消化される。連結したDNAはそれはpir遺伝子 ( 図1E)を含有する大腸菌(E. coli)株に形質転換する。この遺伝子は、新たなプラスミド、トランスポゾンを含有するE.において複製する宿主の染色体領域に隣接することができ大腸菌 6。カナマイシン耐性遺伝子は、として機能新しいプラスミドについてelectableマーカー。最後に、中断された遺伝子の同一性を決定するために使用されるトランスポゾンおよび得られた配列の基本ローカルアラインメント検索ツール(BLAST)分析7,8の各端部に相補的なプライマーを用いて配列決定する。
このトランスポ突然変異誘発戦略は3つの利点3を提供しています。トランスポゼース蛋白質がトランスポゾンに直接結合されているのでまず、挿入は宿主内のトランスポザーゼ遺伝子の発現に依存しません。トランスポゾンは、宿主ゲノム中にそれ自体を組み込むと、トランスポザーゼはトランスポゾンのさらなる移動を防止する、分解される。ホストは、内因性のTn 5 transpositional要素を有している場合は、追加の動きを防止することができない。第二に、エレクトロポレーションによってトランスポの導入は、多種多様な宿主で使用することが可能となる。また、細菌接合によりやviによってトランスポゾンを導入する必要がなくなりRAL感染。両方のプロセスは、ホストの感受性を必要とする。第三に、カナマイシン耐性遺伝子およびトランスポでR6Kγ複製起点を含めることは、簡単に中断された遺伝子を同定することを可能にする。トランスポゾン中断領域が格納され、遺伝子同定のための逆ポリメラーゼ連鎖反応(PCR)技術を使用する必要がなくなり、プラスミドのように配列決定することができる。
このビデオで提示プロトコルは、 エンテロバクター属のトランスポゾン突然変異誘発のための各ステップを説明しています。それが中断された推定遺伝子の同定に細菌細胞への導入からトランスポを使っYSU 9。以前に公開されたプロトコル-3,4,10-に加えて、栄養要求株をスクリーニングするために、レプリカ平板法を使用するための詳細な方法が提示される。この変異誘発技術は、identのために、細菌の異なるタイプ、例えば抗生物質や金属抵抗など他の表現型を調査するために使用することができる合成生物学研究における定義された培養条件下で増殖に必要な遺伝子の数を最小限に抑えifying、または遺伝学の実験用コンポーネントまたは微生物生理学コースを教えるために。
プロトコル
コンピテントセル5,11の1.エレクトロポレーション
- エンテロバクター属のO / NのLB培養を希釈する。 YSU新鮮なLB培地50mlに1/20と0.4と0.6との間のOD(600 nm)に120rpmで、30℃で振盪しながら成長する。必要に応じて、それらの最適な成長温度で、他の細菌株を成長させる。
- 5分間の遠心分離を4℃で5分間7,000×gで氷上で細胞を冷やす。
- 、上清を捨て、4℃で滅菌氷冷水および遠心分離機50ml中の細胞を再懸濁し、5分間7000×gで。この手順を繰り返します。
- 上清を捨て、細胞ペレットの体積に等しい氷冷水の量で細胞を懸濁します。長期保存のため、-80℃、融解氷冷10%(v / v)グリセロール、店舗の等しいペレット体積の氷冷10%(v / v)グリセロール、再懸濁し、10 mlの細胞を洗浄エレクトロポレーションの前に氷の上。
- 40μlにトランスポの0.5μlを添加する細胞。市販のトランスポ液/μlの0.33μgのDNAの濃度で供給される。 0.33μgのが推奨されていますが、0.165μgのDNAが十分です。
- 氷冷、0.2ミリメートル、エレクトロポレーションキュベット中の細胞/トランスポ混合物を配置し、キュベットの底に混合物をタップします。 25μF、200Ω、および2.5 kVので細胞に衝撃を与える。細胞は冷やし残ることを保証するために、使用前に-20℃の冷凍庫でエレクトロポレーションキュベットを格納します。
- すぐに、カタボライト抑制(SOC)媒体とスーパーの最適な培養液を滅菌フィルターの960μlを添加する。上下にピペッティングして混合し、滅菌1.5mlマイクロチューブに細胞を移す。
注:細胞がショック後に死に始めるが、SOC培地中の塩は、それらを回復することができます。節約するためには、トランスポを追加したり、陰性対照細胞を衝撃する必要はない。ちょうどそれに無菌のSOC培地960μlを添加する。 - 30℃fで細胞をインキュベートまたは120rpmで振盪しながら45〜60分。これは、ホストゲノムと、細胞は、カナマイシン耐性を表現するために再結合するトランスポのための時間を提供します。
- LB-館寒天プレート上に細胞100μlを広げて、30℃でプレートO / Nインキュベートする。 4℃の冷蔵庫内の残りの形質転換混合物を保管してください。必要であれば、最初のエレクトロポレーション後2週間に後日アップ時の追加金額を広める。
形質転換体の2グリッディング
- 空の100×15 mmのペトリ皿の蓋にテープグリッドを。 「細菌遺伝学におけるショートコース"12からグリッドをコピーします。
- LB-館寒天プレートの底面に線を引きます。グリッドが寒天を通して見えるようにグリッド蓋に固着させるために、テープの小片を使用してください。プレートの底上の線がグリッドの上、中央に整列されていることを確認。テープでプレートを固定することはあってもグリッド化を可能にし、滑りからそれを保つ。ラインは、レプリカめっき後のコロニーの識別を容易にします。
- 無菌のつまようじを持つ単一の形質転換体を選択し、グリッド内の正方形の中心でそれを見つける。ただコロニーとスポットをタッチします。寒天につまようじを掘るしないでください。プレートを汚染回避するために、一端のみで爪楊枝を扱う。
- ステップ2.3のように隣接する広場で別の形質転換体を発見。板が一杯になると、30℃でO / Nをインキュベートする。これは、マスタープレートである。
栄養要求性表現型12を決定するために3.レプリカメッキ
- LB-館のためのM-9館のためのLB-館のための1、2および3:グリッド化された各プレートについて、1から3の番号3の新鮮な寒天プレートのスタックを作る。使用前に37℃で、逆さまに、O / Nでプレートを乾かします。
- ステップ2.2のように各プレートの上に線を引きます。
- 70%エタノールでレプリカ平板ツールを拭き、にレプリカ平板の上に無菌の別珍の正方形を配置オール及び別珍の正方形を取り締まる。手がさえ、それらを洗浄した後に微生物が生息している。その縁によって別珍の正方形を処理することにより、夾雑微生物の導入を防止。
- クランプに描かれた線とマスタープレート上のラインを合わせ、別珍の正方形の上にプレートを置く。別珍の正方形にプレートから細菌を転送するために穏やかな圧力を適用します。細菌を塗抹しないように注意してください。別珍広場からマスタープレートを外します。
- クランプ上のラインとプレートの番号1上に引かれた線の位置を合わせ、別珍の正方形の上に置きます。プレートに別珍の正方形から細菌を転送するために穏やかな圧力を適用します。プレート番号1を削除してください。
- 水のビーカーに別珍の正方形を破棄し、ステップ3.3のようにレプリカプレーティングツール上でクリーン、無菌の別珍の正方形を配置します。このステップでは、画期的な成長と偽陽性の結果を引き起こす過剰接種を防ぐことができます。
- 整列するクランプ上のラインとプレートの番号1上のラインと新別珍の正方形の上に置きます。別珍の正方形にプレート番号1から細菌を転送するために穏やかな圧力を適用します。プレート番号1を削除してください。
- クランプ上のラインとプレートの番号2に描かれたラインの位置を合わせ、別珍の正方形の上に置きます。プレート番号2に別珍の正方形から細菌を転送するために穏やかな圧力を適用します。プレート番号2を削除します。
- プレート番号3を繰り返しステップ3.8第3プレートは、他の二つのプレートのマスタープレートからの完全な転送のための制御である。水のビーカーに別珍の正方形を破棄。 、それらを乾燥、それらを洗ってアルミホイルでラップし、それらを再オートクレーブ、汚染された別珍の正方形を含むビーカーをオートクレーブ、別珍の正方形を再利用するには。
- 30°CO / Nでプレートをインキュベートする。両方LB館寒天プレート上で増殖するが、M-9館プレート上で増殖しなかったコロニーを栄養要求(あると考えられている図2)。
- ストリーク外の新鮮なLB-館寒天プレート上にプレート番号1から栄養要求を、30°CO / Nでプレートをインキュベートする。
- 単離するためのストリーク外の新鮮なLB-館プレート上にステップ3.11においてストリークプレートから3コロニー。 30°CO / Nでプレートをインキュベートする。これは、変異体がよく絶縁されていることを保証します。一つのセクションで各コロニーから3分の連勝にプレートを分割。
- ステップ2.1から2.4のように、グリッドを形成するために、新鮮なLB-館プレートにステップ3.12から派生画線コロニーからのスポットコロニー。各変異体を三重に再テストされている。
- 栄養要求の表現型を確認するためのステップ3.1から3.10のように、再びプレートを複製。
4.遺伝子レスキュー
- 30℃で液体LB館培地でステップ3.13から単離された変異コロニーの3ミリリットルO / N培養を成長。市販のゲノムDNA精製キットを用いて培養液1mlからゲノムDNAを精製する。
- 0.025 Uの混合物をセットアップするBFU CI制限エンドヌクレアーゼ及び氷上で20μlの反応中/μlのゲノムDNAの1μgの14μlの。 BFU CIは12の異なる部位にトランスポゾンを切断するので、制限エンドヌクレアーゼ、BFA Iまたはそれぞれ二つの3つの異なる部位でトランスポゾンを切断するのHaeIIIを使用することがより効率的である。
- 25分間37℃で反応をインキュベートし、次に80℃で20分間、酵素を不活性化する。 0.8%アガロースゲル( 図3)上で部分的に消化されたDNAの3μLを分析します。ゲルの底部まで10キロバイト上から塗抹標本で成功した部分的な消化の結果。
- 500μlの反応でT4 DNAリガーゼ800単位を使用して部分的に消化されたゲノムDNAを15μlを連結。反応のO / Nにおける4℃をインキュベートする。大きい反応容量は、それ自体、単一の断片のライゲーションを最大化し、二つ以上のDNA断片間の相互作用を最小化する。
- Precipitateの3 M酢酸ナトリウム、pH 5.5、1mlの95%エタノールを50μl添加し、20分間-20℃でインキュベートすることによって連結されたDNA。
- 、70%のエタノールでペレットを洗浄、10分間、4℃でDNA及び13500×gでの遠心フュージ真空濃縮器でそれを乾燥させ、およびヌクレアーゼを含まない水10μlにそれを再懸濁します。あるいは、DNAは、空気は、室温で15分間乾燥させることができる。
- Eを変換するために再懸濁させたDNA4μlのを使用してください部1 R6Kγ複製起点のようにエレクトロポレーションにより大腸菌菌株ECD100DのPIRまたはECD100Dのたpir116は 6を複製するpir遺伝子を持つ細菌菌株を必要とします。低コピープラスミドにおける菌株pir結果、およびたpir116株は高コピープラスミド結果のpir変異遺伝子が含まれています。各形質転換体は、トランスポゾンと、中断染色体( 図1E)の領域で新たなプラスミドが含まれています。
- Inocul単一コロニーを含むLB館培地5mlを食べ、120 rpmで37℃で振盪しながらO / Nを成長し、市販のキットを使用して全体のO / N培養物からプラスミドDNAを精製する。
- 制限エンドヌクレアーゼのXho Iを用いてプラスミド14μlのダイジェスト反応の最終容量を20μlのあることを確認します。この酵素は、カナマイシン耐性遺伝子の5 '末端におけるトランスポゾンの中央に部位を認識する。未消化10μlのを分析し、0.8%アガロースゲル( 図3)上でプラスミドを消化し た。プラスミドは、2キロバイトトランスポゾンプラスに隣接する宿主DNAから構成されています。
5. DNAシーケンシング
- 市販のシークエンスキット、トランスポゾンの各末端に相同なプライマー、およびキャピラリーシークエンシング解析システムを用いてプラスミドを配列決定する。代わりに、プラスミドは、シークエンシングのために外部機関に出荷される場合があります。
- 分子量標準(1キロバイトラダー)でを使用してくださいゲルは、プラスミドの大きさを推定する。その後、50 fmolのために必要とされるプラスミドのナノグラム(ng)を数を推定。
- 260nmの(260)および280nm(A 280)の波長で分光光度計を用いてプラスミドDNAの濃度を測定する。キュベットを通る光路長を確保することは1cmである。 50 ng /μLで260 nmの吸光度を乗じて濃度を決定する。高品質のプラスミドDNAは、1.8と2.0の間で、A 260 / A 280比を持つことになります。
- 各配列決定反応のために必要なDNAの量は、プラスミドの濃度で割っ50 fmolのために必要とngの数である。 10μlに全量をもたらすために水で必要なプラスミドの量を混ぜる。
- 1分間96℃、プラスミドDNA /水の混合物を加熱し、次いでRTに冷却する。
- シーケンシングキットからマスターミックス8μlのを追加し、配列決定プライマーの1の1.6μMの2μlの。
- FOLサーマルサイクラープログラムと反応クリーンアップのためのシーケンシングキットからの低プロトコル。
- キャピラリーDNA解析システムを用いて、各サンプルの分析。
- DNA配列を表示するには、市販のソフトウェアパッケージを使用してください。トランスポゾン( 図4)の最後の7塩基対である配列を、「5'-GAGACAG-3 '、」を検索してください。この7 bpのセグメントの5 '末端の前にシーケンスをトランスポゾンに属します。この7 bpのセグメントの3 '末端の後にシーケンスが中断された遺伝子に属します。
- 基本的なローカル配列検索ツール(BLAST)7,8を使用して中断された遺伝子の可能な機能を決定する。以下を使用link: http://blast.ncbi.nlm.nih.gov/Blast.cgi?PROGRAM=blastn&BLAST_PROGRAMS=megaBlast&PAGE_TYPE=BlastSearch .割り込みのシーケンスを貼り付けクエリボックスにED遺伝子は、データベースの「その他」を選択し、ページの下部にある「BLAST」をクリックしてください。結果が表示されたら、アライメント結果( 図5)を表示するには、ページを下にスクロールします。
結果
エンテロバクター属の形質転換。トランスポとのエレクトロポレーションによるYSUは、宿主ゲノム( 図1A、B)にランダムゲノム挿入を開始した。成功したエレクトロポレーションは、LB-館のプレート上で増殖した数千の形質転換体が得られた。 300-400を得るためには、プレートあたりコロニーをよく間隔、各LB-館寒天プレート上で形質転換混合物スプレッドの量が最適化された。各形質転換体は、少なくとも一つのトランスポゾンの挿入が含まれていますが、M-9培地上で成長するための重要な遺伝子がレプリカ平板法によってスクリーニングすることなく、中断された場合、それは明らかではなかった。形質転換体は、M-9培地上にプレート88のグリッドとレプリカで新しいLB館プレートに移した。トランスポゾンは、プロトコル部は、M-9培地プレートにカナマイシンを追加することをお勧めした形質転換体で維持されることを保証するために、注意してください。 M-9培地は、この場合のカナマイシンを添加したが、それなしで栄養要求株を得ることが可能であっていなかった。で図2は、黒四角は、M-9最少培地プレート(2B)上の2つのLB館プレート(2A)上で増殖したコロニーではなく、を示している。これらの栄養要求株は、おそらくアミノ酸、核酸またはLB培地中ではなく、M-9最小培地( 図1C)に見出されるビタミンの合成に関与する遺伝子の中断を含んでいた。他の86のコロニーは、M-9最少培地上での増殖のために必要とされる遺伝子の中断を持っていませんでした。
いくつかのコロニーをレプリカ培養中に隣接したコロニーで汚染されている可能性があります。純粋培養として突然変異体を単離するために、それは新鮮なLB館寒天プレート上にステップ3.1でLB-館プレート番号1からストリークし、30℃でO / Nを成長させた。成長した三つのコロニーは、第二の新鮮なLB-館プレート上に画線し、30℃でO / Nを成長させた。次に、3個のコロニーのそれぞれから生じた1コロニーは、サード新しいLB館プレート上にスポットした。レプリカは、M-9最小限MEDIに戻しメッキええとプレートは三重に図2で観察された栄養要求の表現型を確認した。2013微生物生理学講座においてスクリーニング1760トランスポゾン形質転換体のうち、23は栄養要求だった。
栄養要求から中断された遺伝子は、次に遺伝子レスキューを用いて同定した。上記第ストリークプレート(ステップ3.13)から、栄養要求性コロニーをLB館培地中でO / N増殖させ、ゲノムDNA、ゲノムDNA精製キットを用いて培養物から精製した。アガロースゲル電気泳動は、精製されたDNAのサイズが10kb以 上( 図3、レーン2)であることを示した。トランスポゾンを含有し、ホスト領域( 図1D)を中断フランキングより小さな断片に、ゲノムDNAを切断するために、部分的に37℃で25分間BFU CI 0.025単位で消化し た。ほぼ毎200〜500塩基対を生じる5'-GATC-3 '部位でこの制限エンドヌクレアーゼ切断する。でも、そこでもトランスポゾン内の12 5'-GATC-3 '部位である、フラグメント無傷のトランスポゾンを含有し、宿主ゲノムに隣接する多数の、まだ部分的に消化されたDNA中に存在するであろう。 図3に示すのレーン3は、部分的0.5kbの下のすべての方法ゲルの底に、10キロバイトの上に始まり、DNAのスメアでゲノムDNAを消化。のDNAスミアが2キロバイト、トランスポゾンのサイズより始まる場合、DNAはあまり消化されています。これは、酵素の添加量は、37℃のインキュベーション時間、またはその両方を減少させることが必要であろう。スミアおよび消化されたDNAとのレーンは、未消化のDNAとDNAと同じサイズがされていない場合は、何消化は発生しなかった。これは、37℃のインキュベーション時間またはその両方を追加した酵素の量を増やすことが必要であろう。代わりに、BFU CIを使用すると、制限二、三の異なる部位にトランスポゾンを切断BFA Iまたはヘジン IIIを、エンドヌクレアーゼ使用することが可能であるそれぞれ。これらの酵素のいずれかの使用はE.を増加させることができる遺伝子救助中に大腸菌PIR +変態利回り。
部分的に消化されたDNAを、500μlの反応で連結した。この増加した容量は、他のDNA断片間の相互作用を最小化し、それ自身と連結し、各断片が環状分子を形成する確率を最大化する。ライゲーションしたDNAを大腸菌にエレクトロポレーションにより沈殿させ、形質転換した大腸菌菌株ECD100Dのたpir116。形質転換混合物をLB館プレート上に広げた。増殖したコロニーは、トランスポゾンと、中断宿主遺伝子( 図1E)の領域からなる新たなプラスミドを含んでいた。コロニー形成単位の数は、形質転換混合物の1mlあたり0〜2,000の範囲であった。隣接する染色体領域がEに毒性であったタンパク質をコードしている場合大腸菌宿主の形質転換効率が低いまたはクロモのほんのセグメントであっいくつかは新たなプラスミドに関連していた。プラスミドDNAは、単一の形質転換体を接種し、カナマイシン耐性遺伝子の5 '末端付近トランスポゾンの中心に単一の部位を認識し、制限エンドヌクレアーゼ、 をXho Iで消化し 、培養物から精製した。 図3のレーン4は、未消化のプラスミドを含有し、レーン5は消化されたプラスミドを含んでいた。精製したプラスミドDNAは、スーパーコイル化される傾向にあるので、線形の同じ鎖のDNAを消化よりも速い速度で移動した。このプラスミドの正確なサイズは3 kbのだった。これは、トランスポゾンの2キロバイト、プラス中断された宿主遺伝子の約1キロバイトを含んでいた。隣接する宿主DNAは、1つ以上のXho I認識部位を含んでいる場合、二つ以上のプラスミドのバンドは、IがDNAを消化をXhoとレーンにおいて観察される。
図4は栄養要求から単離されたプラスミドについてシーケンシング結果を示している。証を除去した後太字で室温トランスポゾン配列、宿主DNA配列を、基本的なローカル配列検索ツール(BLAST)分析7,8-ために提出した。 BLASTの結果の一つは、中断したシーケンスは、 エンテロバクター·クロアカエ亜種に類似していることを示唆した。クロアカエNCTC 9394(EMB | FP929040.1 |)L-チロシンおよびL-フェニルアラニン( 図5)13の生合成に関与しているコリスミ酸ムターゼ、遺伝子。時々 、部分的に消化されたゲノムDNAのライゲーションの間、線形トランスポゾンは、1つ以上の他のBFU CIフラグメントにライゲーションする。これは、遺伝子の正体が急激に変化するため、BLASTは混乱をもたらすことができます。例えば、他の栄養要求の最初の41塩基対は、プロリン生合成に関与する1グルタメート-5-セミアルデヒドデヒドロゲナーゼ遺伝子にマッチ。脂質A-disaccための62塩基対のセグメントに続くリボ二リン酸還元酵素サブユニットの遺伝子に合わせたそして、次の219塩基対、 harideキナーゼ。最後の295塩基対セグメントは再びグルタミン酸-5-セミアルデヒドデヒドロゲナーゼに一致。 BFU CI 5'-GATC-3 '部位の存在は、2つのより小さなDNA断片を救出プラスミドに連結していることを示唆した。このように、連結反応するため、部分的に消化されたDNAの希釈は完全に小さな断片のライゲーションによって引き起こされるバックグラウンドがなくなるわけではありません。これらの結果から、この遺伝子の配列は、トランスポゾンに直接隣接したためにトランスポグルタミン酸-5-セミアルデヒドデヒドロゲナーゼ遺伝子に自身を挿入することが結論付けられた。他の栄養要求株は、メチオニン生合成に関与するシスタチオンベータリアーゼの遺伝子の中断を含有していた。ヒスチジン生合成に関与するヒスチジノールデヒドロゲナーゼ。セリン、グリシン、およびシステインの生合成に関与するホスホセリンホスファターゼ。ロイシン、イソロイシンおよびバリン生合成に関与して1ケトール酸レダクトイソメラーゼのため。
NT "FO:キープtogether.withinページ="常に ">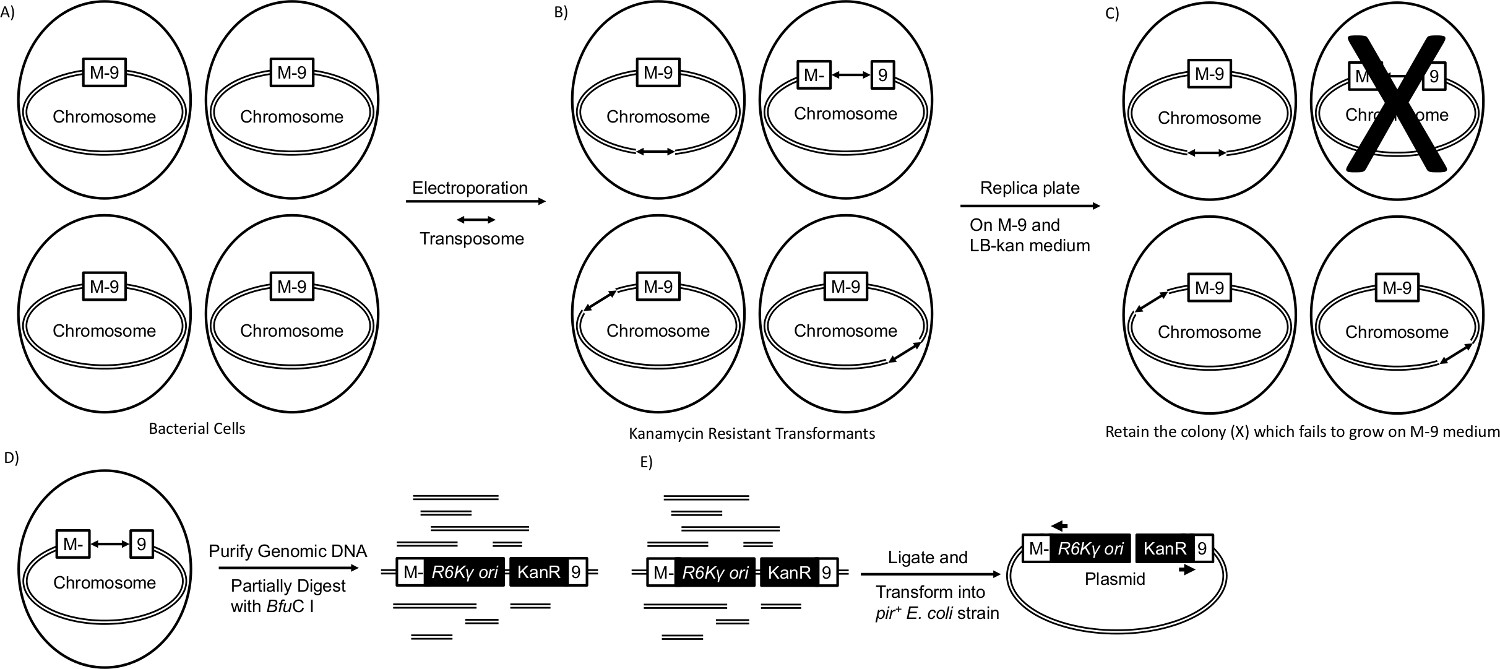
図1.トランスポゾン突然変異誘発。 (A)培地のグルコースを含むM-9最小塩上での増殖のために必要とされる遺伝子を含む細菌細胞。箱入りM-9は、栄養要求性遺伝子を表し、(B)トランスポは、エレクトロポレーションによる細菌株に導入され、各モザイク端に結合されたトランスポザーゼタンパク質(矢印)は、ランダムに宿主染色体3,4にトランスポゾンを挿入する。唯一のトランスポゾン含有する形質転換体をLB館培地プレート上で増殖します。(C)に形質転換体がグルコースを含むM-9培地上にし、LB館培地にプレートのレプリカです。グルコースを含むM-9培地上(クロスアウト)成長に失敗する形質転換体は、栄養要求である。(D)ゲノムDNAを要求株から精製され、部分的に4塩基cuttで消化されている小胞体、DNA部位を認識するBFU CI、5'-GATC-3 '(E)は、部分的に消化されたDNAは、T4 DNAリガーゼで処理し、そしてE.へエレクトロポレーションによって形質転換され大腸菌株のECD100DのPIRは、トランスポゾンおよびM-9培地での増殖に必要とされる中断染色体遺伝子を含む新たなプラスミドを形成する。 pir遺伝子は、新たなプラスミド6の複製起点として機能するトランスポゾンでR6Kγ複製起点を含む環状DNA断片を可能にする。カナマイシン耐性遺伝子は、新たなプラスミドのための選択マーカーとして働く。プラスミドを精製して、中断された遺伝子の〜500 bpの配列は、トランスポゾンの各末端に相同なプライマー(矢印)を使用して決定される。 この図の拡大版をご覧になるにはこちらをクリックしてください。
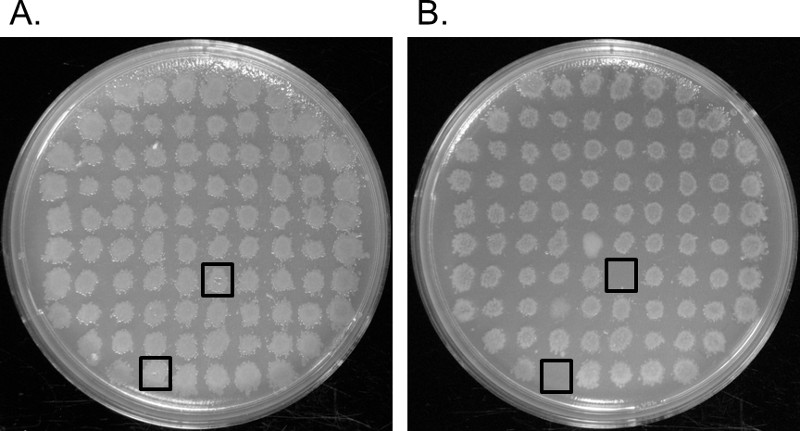
図2.レプリカメッキ。 LB館寒天プレートに移した形質転換体(A)のグリッド(B)M-9培地プレートに移し、同じ形質転換体のグリッド。栄養要求をLB館プレート上ではなく、M-9培地プレート上で増殖した。これらは黒い四角で識別されます。 この図の拡大版をご覧になるにはこちらをクリックしてください。
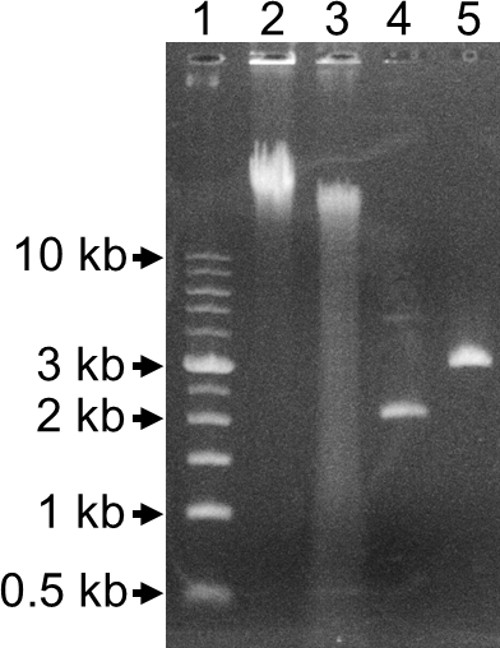
図3. 0.8%アガロースゲルレーン1:1キロバイトラダー。レーン2:未消化のゲノムDNA。レーン3:BFU C I部分消化ゲノムDNA。レーン4:未消化救出血漿ID。レーン5:Xho I では救出プラスミドを消化し た。

プラスミドの図4.部分的なDNA配列。太字のシーケンスは、トランスポゾンの一部であり、BLAST解析のために提出されていませんでした。シーケンスの残りの部分は、ホストから救出される遺伝子のセグメントである。

図5. BLAST分析 7,8 は一塁(クエリ)から出発して、中断された遺伝子は、フェニルアラニンおよびチロシン生合成に関与しているエンテロバクター·クロアカエコリスミ酸ムターゼをコードするように思われる。
ディスカッション
トランスポを使用したトランスポゾン突然変異誘発は、 エンテロバクター属に栄養要求株を生成するための効率的なツールです。 YSU及びグラム陰性菌およびグラム陽性菌3,4の他のタイプ。プロセスは、エレクトロポレーションによって宿主へのTn 5由来のトランスポを導入することにより開始した。挿入物を有するコロニーを同定するために、形質転換されていない標的株は、トランスポゾンによって運ば耐性マーカーを選択するためにカナマイシンに感受性でなければならなかった。いくつかのホストは、それが宿主ゲノムと再結合する前に、トランスポゾンDNAを分解する制限エンドヌクレアーゼを生産する。エレクトロポレーションミックスに制限エンドヌクレアーゼ阻害剤の添加は、分解を最小限に抑え、変換収率3を増加させることができる。エレクトロポレーションコンピテント細菌を調製するが、それは塩の存在によって引き起こされる細菌を死滅させる電気アークまたはスパークを防止することが可能な限り多くの塩を除去することが重要であった。さえの不在下で電気アークは、細胞は、それらを衝撃的な後に急速に死に始めた。 SOC増殖培地を含有する塩を添加すると、すぐに細胞死及び形質転換効率の増加を最小化5浸透圧バランスを回復させた。その後、カナマイシン耐性形質転換体を、M-9最小培地寒天へメッキ新しいLB館プレートおよびレプリカ上にスポットした。プレートの残りの部分は前に接種した上で接種可能な栄養要求を欠場偽陽性の結果を引き起こす可能性がないようにするには、別珍の正方形は、ステップ3.6で切り替えた。その後、レプリカプレーティング時に、ちょうど十分な圧力が、隣接するコロニー間の汚れや汚染を引き起こすことなく、コロニーを転送するために適用した。
栄養要求株における変異遺伝子は、遺伝子の救助を介して同定された。各栄養要求からのゲノムDNAを、TRをカットBFA Iまたはヘソン IIIを用いBFU C Iに部分的な消化カッター頻繁なベースで部分消化し 、使用して剪断したansposon少ない頻度で、トランスポゾン変換効率を向上させることができる。これは、トランスポゾン内の部位を欠く他の制限エンドヌクレアーゼ、6塩基、8塩基カッターでの完全消化を使用することも可能である。消化されたDNAは、その後連結し、E。に形質転換した大腸菌菌株ECD100DのPIRまたはECD100D たpir116 6。 E.ものの大腸菌たpir116株はステップ4.8でエレクトロポレーションにより形質転換し、それが化学的コンピテント細胞を用いることも可能である。この場合、全体の変換が救出されたプラスミドの大規模な選択を取得するために分散させる必要があります。 pir遺伝子は低コピー数プラスミドとして複製する環状化トランスポゾンを可能にし、 たpir116変異遺伝子は、それが高コピープラスミドとして複製することができます。 たpir116株を用いて、低変換収率は、元のホストからの毒性遺伝子産物の発現に起因する可能性がある。毒性は、低コピーを使用することによって低減することができる PIR株。しかし、PIR株からの低プラスミド収量は、シークエンシングがより困難にすることができます。
ゲノム配列決定10およびPCR配列14は、遺伝子救助に代替方法である。ゲノム配列決定法は、精製したゲノムDNAから直接隣接する染色体領域をシーケンス。標的生物のゲノムが既に配列決定されている場合は、この戦略は有効です。変異領域は、ゲノム配列決定およびBLASTによって同定されたならば、全領域をPCRによって増幅し、さらなる分析のためにクローン化することができる。 PCR法は、2つのPCR反応と四つの異なるプライマーを使用しています。短いDNA、リンカー配列(プライマー2)が含まれて縮重プライマーと対トランスポゾン相同なプライマー(プライマー1)最初の反応。第二のPCR反応は、相同なリンカー配列プライマー(プライマー4)を有する別のトランスポゾン相同のプライマー(プライマー3)をペアリングする、ネストされたPCR反応である。プライマー3のためのプライミング部位である次にプライマー1.プライミング部位の3 '側に位置し、第二のPCR反応からのamplicandがクリーンアップされ、配列決定される。この自動配列決定法は、ゲノムDNAの精製およびプラスミドレスキューの両方の必要性を排除し、変異体細胞から直接DNAを増幅する。
代わりに、栄養要求性に関与する遺伝子の同定により、トランスポゾン突然変異誘発はまた、他の容易にアッセイの表現型を研究するために使用することができる。 エンテロバクター属YSU耐性を100μg/ mlアンピシリン(未発表データ)もある多金属耐性細菌株である。のHgCl 2のためのその最小発育阻止濃度(MIC)は70μmで、のZnCl 2が AgNO 3を80μMで、NaAsO 2は 14 mMの9で、800程度である。この株にトランスポを導入し、LB館プレート上で形質転換体をグリッディングした後、グリッドコロニーはこのbacteのMIC未満の濃度で金属を含有する培地上にレプリカをメッキすることができるリアル菌株。低下MICを救助及び変異から中断遺伝子の配列決定は、これらの金属のいずれかに耐性を付与する遺伝子を明らかにすることができる。
変異体を作製することに加えて、トランスポは、抗生物質または金属抵抗として機能する遺伝子のゲインを識別するために、インビトロまたはインビボでベクターとして使用することができる。 in vitroでの戦略、 エンテロバクター属からのゲノムDNAのために。 YSUトランスポゾンの外側部位を認識する制限エンドヌクレアーゼで消化する。 DNAをライゲーションし、Mg 2+を含有する緩衝液中でトランスポと混合される。トランスポゾンとランダムゲノムフラグメント間の組換えの後、E。大腸菌株のECD100DのPIRは DNAで形質転換し、アンピシリンまたは金属塩を含有するプレート上に広げている。成長するコロニーは、ホストの耐性遺伝子の隣に位置してトランスポゾンを含む新しいプラスミドを保有します。 in vivoでの STのためのrategy、 エンテロバクター属 。 YSUはトランスポで形質転換される。そして、全体の変換反応は、ランダムトランスポゾン挿入を有する大きな集団を選択するためにLB館液体培地で増殖させる。ゲノムDNAは、トランスポの外側部位を認識する制限エンドヌクレアーゼで消化し 、連結し、そしてE.に変換される大腸菌菌株ECD100DのPIR。インビトロの方法と同様に、形質転換体は、アンピシリンまたは金属プレート上に広げている。再び、得られたプラスミドは耐性遺伝子の隣に挿入されたトランスポゾンで構成されます。これら2つの戦略は、標的耐性遺伝子が発現E.において活性であることができる場合にのみ成功する大腸菌菌株。
トランスポゾン突然変異誘発細菌株は、成長のために必要と15,16遺伝子の最小数を識別するために使用することができる。 loxP部位と異なる抗生物質耐性マーカーを含む二transposomesはこの戦略のために必要とされるI既知のゲノム配列とNAの細菌株。 2トランスポゾンが統合した後、発現されたCreリコンビナーゼは、トランスポゾン挿入の間の染色体領域を排除し、2つのloxP部位の間の組換え反応を触媒する。ゲノム配列の知識は、除去された遺伝子に識別することが可能となる。増殖する能力を排除した遺伝子は、成長のために必要とされないことを示している。この技術は労働集約的であり、この戦略を使用して成長のための最小のゲノムは、まだ完了していない。しかし、PCR配列決定を使用して、 緑膿菌の自動化されたトランスポゾン突然変異誘発研究は、300〜400の遺伝子が富栄養培地14中でこの細菌の増殖に必要であったと推定した。この研究は、loxP配列 / Creリコンビナーゼ ·システムを使用していませんでした。
最小限のゲノムを有する微生物は、合成生物学17のために有用である。最小限のゲノムを有する細菌株はespeciallです望ましくない副生成物の生成を排除するためのy便利。例えば、 クロストリジウム·アセトブチリカム(C.アセトブチリカム ) はバイオ燃料、ブタノール18を生成するために使用される。その成長中に、この株は、定常期に入る直前に、その指数増殖期の終わりに、ブタノールを生成する。この時点で、ブタノールおよび他の発酵生成物に敏感になり、止め製品を合成するための休眠胞子を形成する。 transposomesはウェルシュ菌 3を研究するために使用されているので、それは望ましくない発酵製品および胞子形成のための遺伝子を欠くC.アセトブチリカム株を構築するための類似のトランスポ/ のloxP / Creリコンビナーゼ系を使用することが可能かもしれないが、より大きな遺伝子を含みブタノール公差。この菌株は、最小限の汚染物質と生成物の収率を増加させる。
要約すると、このビデオジャーナルの記事は、完全なスイートを提供していますトランスポゾン突然変異誘発による栄養要求株を作成するために使用される手順のステップの説明によりp。それは、DNA配列決定により中断された技術や遺伝子の同定をクローニングすることにより変異した遺伝子を救出、レプリカ平板によって変異をスクリーニングする、エレクトロポレーションによりトランスポを導入するカバーしています。この技術は、未知の遺伝子の機能を同定するため、またはおそらく工業プロセスで使用することができる細菌株を構築するために使用することができる。
開示事項
著者は、開示することは何もありません。
謝辞
著者は、2010年から2014年春学期の間、私のトランスポゾン突然変異誘発のアイデアをテストし、私の微生物生理学大学院生の私の学部独立研究生のすべてとすべてに感謝したいと思います。この作品は、ヤングズタウン州立大学の生物科学専攻によって資金を供給された。
資料
| Name | Company | Catalog Number | Comments |
| EZ-Tn5 R6Kγori/KAN-2 Tnp Transposome Kit | Epicentre (Illumina) | TSM08KR | |
| EC100D pir+ Electrocompetent E. coli | Epicentre (Illumina) | ECP09500 | Capable of replicating plasmids with an R6Kγ replication origin at a low copy number |
| EC100D pir-116 Electrocompetent E. coli | Epicentre (Illumina) | EC6P095H | Capable of replicating plasmids with an R6Kγ replication origin at a high copy number |
| 5X M-9 Salts | Thermo Fisher | DF048517 | |
| Lennox LB Broth | Thermo Fisher | BP1427-2 | |
| Agar | Amresco, Inc. | J637-1KG | Solid media contained 1.6% (w/v) agar |
| Kanamycin Sulfate | Amresco, Inc. | 0408-25G | When required, media contained 50 μg/ml kanamycin sulfate |
| D-Glucose Monohydrate | Amresco, Inc. | 0643-1KG | |
| Yeast Extract | Amresco, Inc. | J850-500G | |
| Tryptone | Amresco, Inc. | J859-500G | |
| KCl | Sigma-Aldrich | P4504-500G | |
| NaCl | Amresco, Inc. | X190-1KG | |
| MgCl2 | Fisher Scientific | BP214-500 | |
| MgSO2 | Fisher Scientific | BP213-1 | |
| Super Optimal Broth with Catabolite Repression (SOC) medium | 0.5% (w/v) yeast extract, 2% (w/v) tryptone, 10 mM NaCl, 2.5 mM KCl, 10 mM MgCl2, 20 mM MgSO4 and 20 mM Glucose | ||
| BfuC I | NEB | R0636S | Partial digestion of genomic DNA |
| Xho I | NEB | R0146S | Plasmid digestion |
| T4 DNA Ligase | NEB | M0202S | |
| Nuclease Free Water | Amresco, Inc. | E476-1L | For dissolving precipitated DNA and restriction endonuclease reactions |
| GenomeLab DTCS - Quick Start Kit | Beckman Coulter | 608120 | DNA sequencing |
| Wizard Genomic DNA Purification Kit | Promega | A1120A | |
| Wizard Plus SV Minipreps DNA Purification System | Promega | A1460 | Plasmid purification |
| Replica Plating Block | Thermo Fisher | 09-718-1 | |
| Velveteen Squares | Thermo Fisher | 09-718-2 | Replica plating |
| PetriStickers | Diversified Biotech | PSTK-1100 | Grid for replica plating |
| Petri Dishes | Thermo Fisher | FB0875712 | |
| Gene Pulser II | BioRad | Electroporation | |
| Electroporation Cuvettes – 2 mm | BioExpress | E-5010-2 | |
| CentriVap DNA Vacuum Concentrator | Labconco | 7970010 | Drying DNA |
| CEQ 2000XL DNA Analysis System | Beckman Coulter | DNA sequencing | |
| Vector NTI Advance 11.5.0 | Life Technologies | 12605099 | DNA sequence analysis |
参考文献
- Kim, B. H., Gadd, G. M. . Bacterial physiology and metabolism. , (2008).
- Hayes, F. Transposon-based strategies for microbial functional genomics and proteomics. Annual review of genetics. 37, 3-29 (2003).
- Hoffman, L. M. Random chromosomal gene disruption in vivo using transposomes. Methods in molecular biology. 765, 55-70 (2011).
- Goryshin, I. Y., Jendrisak, J., Hoffman, L. M., Meis, R., Reznikoff, W. S. Insertional transposon mutagenesis by electroporation of released Tn5 transposition complexes. Nature biotechnology. 18 (1), 97-100 (2000).
- Dower, W. J., Miller, J. F., Ragsdale, C. W. High Efficiency Transformation of E. coli by High Voltage Electroporation. Nucleic Acids Research. 16 (13), 6127-6145 (1988).
- Metcalf, W. W., Jiang, W., Wanner, B. L. Use of the rep technique for allele replacement to construct new Escherichia coli hosts for maintenance of R6Kγ origin plasmids at different copy numbers. Gene. 138 (1-2), 1-7 (1994).
- Morgulis, A., Coulouris, G., Raytselis, Y., Madden, T. L., Agarwala, R., Schäffer, A. a Database indexing for production MegaBLAST searches. Bioinformatics. 24 (16), 1757-1764 (2008).
- Zhang, Z., Schwartz, S., Wagner, L., Miller, W. A greedy algorithm for aligning DNA sequences. Journal of computational biology a journal of computational molecular cell biology. 7 (1-2), 203-214 (2000).
- Holmes, A., Vinayak, A., et al. Comparison of two multimetal resistant bacterial strains: Enterobacter sp. YSU and Stenotrophomonas maltophilia OR02. Current microbiology. 59 (5), 526-531 (2009).
- Hoffman, L. M., Jendrisak, J. J., Meis, R. J., Goryshin, I. Y., Reznikoff, W. S. Transposome insertional mutagenesis and direct sequencing of microbial genomes. Genetica. 108 (1), 19-24 (2000).
- Ausubel, F., Brent, R., et al. . Short Protocols in Molecular Biology. , (1997).
- Miller, J. H. . A short course in bacterial genetics: a laboratory manual and handbook for Escherichia coli and related bacteria. 2, (1992).
- Gibson, M. I., Gibson, F. Preliminary studies on the isolation and metabolism of an intermediate in aromatic biosynthesis: chorismic acid. The Biochemical journal. 90 (2), 248-256 (1964).
- Jacobs, M. A., Alwood, A., et al. Comprehensive transposon mutant library of Pseudomonas aeruginosa. Proceedings of the National Academy of Sciences of the United States of America. 100 (24), (2003).
- Reznikoff, W., Winterberg, K. Transposon-Based Strategies for the Identification of Essential Bacterial Genes. Microbial Gene Essentiality: Protocols and Bioinformatics SE. 2, 13-26 (2008).
- Suzuki, N., Inui, M., Yukawa, H. Random genome deletion methods applicable to prokaryotes. Applied Microbiology and Biotechnology. 79 (4), 519-526 (2008).
- Malley, M. A., Powell, A., Davies, J. F., Calvert, J. Knowledge-making distinctions in synthetic biology. BioEssays news and reviews in molecular, cellular and developmental biology. 30 (1), 57-65 (2008).
- Zheng, Y. -. N., Li, L. -. Z., et al. Problems with the microbial production of butanol. Journal of industrial microbiolog., & biotechnology. 36 (9), 1127-1138 (2009).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved