Method Article
の遺伝的操作
要約
ここでは、タイプIとタイプIIを使用するための方法を報告するΔku80株トキソプラズマ効率的に機能的なゲノム解析のためのターゲット遺伝子欠失、遺伝子置換を生成する。
要約
相同組換えを利用した標的遺伝子操作は、遺伝子機能と表現型(単数または複数)の詳細図を得るための機能的なゲノム解析のための選択方法である。標的遺伝子の欠失、標的突然変異、補完遺伝子の機能、および/またはタグ付けされた遺伝子を有する変異株の開発は、これらの遺伝子操作を効率的に二重によって媒介積分を用いて、目的の遺伝子座を標的とすることができる場合は特に、遺伝子機能に対処するための強力な戦略を提供する相同組換えを渡る。
非相同的組換えの非常に高いレートの変動により、 トキソプラズマ原虫のゲノム解析の機能は、以前は、特定の遺伝子座に遺伝子欠失、遺伝子の交換を標的とする効率的な方法が存在しないことによって制限されている。最近、我々はI型とII型Tの株で非相同組換えの主要な経路を廃止遺伝子encodinを削除してトキソプラズマG Ku80がタンパク質1,2。 Δku80株は、in vitroおよびin vivoでタキゾイト(急性)とブラディゾイト(慢性)段階で正常に動作し、実質的に相同組換えの100%の頻度を示す。 ΔKu80が株は単一の遺伝子で同様のゲノムスケール1-4で機能的なゲノム研究が実現可能に。
ここでは、T.でのアプローチを対象とした遺伝子を進めるためにタイプIとタイプIIΔku80Δhxgprt株を使用する方法を報告するトキソプラズマ 。私たちは、ヒポキサンチン-グアニンホスホリボシルトランスフェラーゼ(HXGPRT)選択マーカーの標的挿入や欠失により遺伝子欠失、遺伝子の交換、およびタグの遺伝子を生成するための効率的な方法を概説する。プロトコルを対象の遺伝子は、寄生虫のゲノムの機能解析を進めるために、持ち運び単一菌株を開発するΔku80株では、様々な方法で使用することができる複数のターゲット遺伝子操作。この遺伝的方法とその後の表現型アッセイのアプリケーションがTの生物学の基本的かつユニークな側面を明らかにするトキソプラズマやマラリア( マラリアのsp。)とクリプトスポリジウム症( クリプトスポリジウム ) を引き起こす関連の重要なヒト病原体。
概要
トキソプラズマは、しばしば一般的な偏性細胞内寄生原虫であり、慢性的に動物と人間5の広い範囲に感染します。これは病原体によって10億以上の人間が現在であることを推定して慢性的に感染している。 T.によって引き起こされる疾患の重要性に加えてトキソプラズマ感染症、実験的なツール、強力なゲノム資源6の可用性の向上は、in vitroでの成長と優れたマウスモデルでの容易T.を作ったトキソプラズマ細胞真核生物病原体や、マラリア( マラリアのsp。)とクリプトスポリジウム( クリプトスポリジウム )5,7として壊滅的な病気を引き起こす他の重要アピコンプレクサ原虫の広範な研究のための主要なモデルシステム。モデル生物としてのトキソプラズマの重要な制限は、遺伝子操作をターゲット運ぶ子孫の非効率的な回復となっています。これのproble遺伝子ターゲティングでmは、T.、野生型株における非相同的組換えの非常に高い周波数に対して相同組換えの低周波によるものである広範なDNA相同性が遺伝学的研究2で使用したDNA標的分子に設けられていても原虫 。
我々は最近、遺伝子組み換え型の非相同組換えの主要な経路をブロックKu80がタンパク質1,2をコードする遺伝子を削除して、IとII型トキソプラズマの株。 I型およびIIΔKu80が菌株はin vitroおよびin vivoでタキゾイトとブラディゾイトの段階でインビトロおよびインビボの両方の正常な成長率、大きさ及び挙動を示す結果の型が、これらの株は、相同組換えの本質的に100%の周波数を示し、この表現型は、いくつかのカントーに数百の標的に遺伝子操作の急速に分離希望の子孫の可能性が高くなりますusand倍1,2,4。最近のコミュニティ全体のIとII型ΔKu80が株がペースアップする大幅上昇持つ型の使用、様々ならびにT.におけるターゲット遺伝子アプローチの成功率トキソプラズマ1-4,8-13。ここでは、TのΔKu80が株で、標的遺伝子欠失、遺伝子置換、遺伝子タギングのための包括的なプロトコルを記述するトキソプラズマ 。我々はデザインと確実にトキソプラズマのΔKu80が株で特定の遺伝子または遺伝子座に遺伝子操作をターゲットにする方法を示します。
プロトコル
1。目的の遺伝子を削除するために遺伝子ターゲティング
このプロトコルが定義遺伝子座における標的遺伝子の欠失を有する変異株の効率的な生成のために設計されている。前述のT.トキソプラズマヒポキサンチン-グアニンホスホリボシルトランスフェラーゼ(HXGPRT)選択マーカーは、ミコフェノール酸とキサンチン(MPA + X)選択14-16以下の安全で信頼性の高いマーカーの挿入のために、このプロトコルで使用されています。このプロトコルは、酵母組換えクローニング17,18に基づいて分子を標的とするDNAを構築するために検証され、費用対効果的な方法を使用しています。いくつかの代替プロトコルは、 インビトロ組換えクローニング方法、または融合PCR において 、細菌の組換えクローニング方法を用いて、組換えターゲッティング分子を構築するための商業的に入手可能である。以下に記述されたプロトコルは、クローン化された回復最高全体的な効率性と信頼性を提供パラグラフTのΔKu80が株で、標的遺伝子欠失を有するITES トキソプラズマ 。
DNAを標的1.1準備
- ToxoDB 6(アクセスhttp://www.toxodb.orgを目的の遺伝子(GOI)のためにゲノム配列を取得する)。
注:ゲノム配列が操作される歪みに最も近いToxoDBデータベース内のタイプI、IIまたはIIIゲノム配列から得られるべきである。たとえば、次のプルデンシャルのためにRHとME49のためのGT1。
- 酵母シャトルベクターpRS416(あるいはpRS426)と選択マーカーと33 bpのオーバーラップを組み込む(と33 bpのオーバーラップを取り入れた目的の遺伝子(GOI)の5 'および3'ゲノム標的脇腹を増幅する設計特定の重複プライマーHXGPRT)( 図1A-B)。 5 'の両端に各プライマーでユニーク制限酵素部位を追加する33 bpのオーバーラップとT.の間に置かND 3 'ゲノム標的脇腹、 トキソプラズマ特異的プライマー配列(単数または複数)( 図1A-B)。
注:30 bpのオーバーラップよりも、効率的な酵母組換えクローニングするために必要です。 5 'および3'ゲノムターゲティングフランクは、標的欠失を画定800と1,400 bpの間の特定のDNA断片を増幅するように設計されている。短い脇腹が大幅ΔKu80をΔhxgprt背景 2の効率をターゲットに削減することに注意してください。
- PCRは、5 'プライマーF1とR1を使用してターゲット·脇腹をし、PCR増幅は3'増幅ゲノムDNA 3からプライマーF2とR2を( 図1A-C)を使用してターゲット·脇腹を。これとは別に、PCRは、関連する5 'および3'プライマーHXFとHXRを用いてcDNAをHXGPRT pminiHXGPRTカセット14のdhfr遺伝子のフランキング領域を含む〜2 kbpのHXPGRT遺伝子を増幅する( 図1B-C)。
注:トランスフェクトすることが親株由来のゲノムDNAを用いて標的を増幅フランク。
注:5 'および3'など混乱軌跡からHXGPRTの目標除去など、その後の効率的な遺伝子操作を容易にするために脇腹を標的DNAに対して前方向きに置きHXGPRTマーカーを。
- アガロースゲル電気泳動により正しいPCR産物のサイズを確認し、アガロースゲル標準を使用してDNA断片をDNA濃度または濃度を決定するための別の方法を推定する。
- としてSchiestly Gietz及び17に記載されて有能な酵母アリコートを生成します。
- ゲノム標的脇腹'3 50ngの、ゲノム標的脇腹'、HXGPRT選択マーカー100ngの、そして10の最終容量を達成するための滅菌H 2 Oとの線形化されたシャトルベクター50ngの5の50 ngのを結合-20μlの。 3 PCR産物と酵母E.と有能な酵母を変換GietzとSchiestly 17で記述されたプロトコルを使用して組換えクローニング用大腸菌シャトルベクター。プレートはウラシルマイナス最少培地寒天プレート上で酵母を形質転換し、2のために30℃でインキュベート - 3日間。
注:プラスミドの順序付けられた組立体、5 'ターゲットフランク、HXGPRTマーカー 、および3'標的フランクは、DNA断片の間に操作された33塩基対の重複によって促進される( 図1A-B)との相同組換えによって媒介される酵母。
- ウラシルマイナスプレートに2倍YPAD 2 mlを追加し、優しく寒天を中断せずにコロニーを取り除くためにこすることによって収穫酵母。 5,000×gで5分間酵母液を遠心し、上清を捨てる。
- RNaseAをを含む再懸濁バッファー250μlの酵母でペレットを再懸濁し、50を追加 - 100μlのソリューションにCID-洗浄したガラスビーズ。酵母細胞を粉砕するために最高速度で5分間ボルテックス。
- ガラスビーズをチューブの底に沈殿し、2ミリリットルエッペンドルフチューブに上清を転送することができます。
- 〜100μlの最終容量に再懸濁し、ミニプレップDNA分離キットを用いて酵母DNAを単離する。
- DH10Bエレクトロコンピ 40μlのと2μlの酵母DNA(〜50 ngの)を混ぜ大腸菌は、氷上で保存。
- チルド2ミリメートルギャップエレクトロキュベットにソリューションを転送し、2.4キロボルト及び129Ωで細菌細胞をエレクトロ。
- 14ミリリットルスナップキャップファルコンチューブに各キュベットおよび転送ソリューションに0.8ミリリットルSOC培養液を追加することによって、細胞を救出。 60分 - 40のためのローラードラムに37℃で細胞をインキュベート。
- プレートE. 2XYT +アンピシリン(AMP)プレート上の大腸菌とは、37℃で一晩培養する。
- 〜6を選択 - 8単一コロニーを、37℃で〜16時間3ミリリットル2XYT + AMPで成長
- 各Eの30%グリセロール冷凍庫ストックを準備大腸菌クローン。
- E.からのプラスミドpΔGOIを分離ミニプレップキットを使用して大腸菌クローン、〜100μlの最終容量に再懸濁する。
- 制限酵素を用いたpΔGOIは、DNAプラスミドサイズを測定し、パターンをバンディング予想さpΔGOIDNAを確認するためにダイジェストを検証します。
- におけるゲノム配列データに基づいて、100%のDNA配列を確認するために5 'および3'ゲノムを目標フランクのDNA配列決定によりpΔGOIの検証を確定しhttp://www.ToxoDB.org 。
注:近完全な配列相同性は、それぞれの塩基対の差が完全な相同性の長さが対応する切りを構成しているので、効率3を対象とした遺伝子を最大化するために不可欠である。
注:プルデンシャル親世紀に基づくタイプIIΔku80株のゲノム配列雨はこの時点では利用できません。この作品は、II型ΔKu80が株の代理ゲノムとしてII型ME49ゲノム配列を使用しています。遺伝子座の数で配列データに基づいて、ME49ゲノムプルデンシャルゲノムは1〜10,000ヌクレオチドあたり、以下の周波数で一塩基多型を示すことが推定される。
- 検証E.からグリセロールストックで〜250ミリリットル2XYT + AMP一晩培養を接種することによりpΔGOI株式の> 200μgのを生成します大腸菌ミニプレップクローン。
- 〜1,000μlの最終容量に再懸濁し、maxiprepのDNA単離キットを使用して大規模な培養からpΔGOIプラスミドDNAを分離します。
- トランスフェクションの正しい標的DNA分子を検証するpΔGOImaxiprep DNAの配列決定、制限酵素消化、またはDNAを繰り返します。
- ユニークな制限酵素X(RE.X)を使用して、5 '末端で〜15μgののpΔGOIを線形5 'ターゲット側面( 図1B)に組み込まれているダイジェストサイト。
注:Tで標的遺伝子トキソプラズマは、目的2の遺伝子座をターゲットイベントを効率的に周波数を得るために、DNAを標的〜10μgのの最小値を必要とします。
- pΔGOIの線形化を検証し、アガロースゲル電気泳動または別の方法でDNA濃度を決定する。
注:5で制限酵素消化'脇腹が完了線形化が得られない、設計された3'の場合ユニークRE.YダイジェストサイトはpΔGOIの線形化のための代替サイトとして使用することができます。
注:完全に線形化標的DNA分子はΔKu80が株で相同組換えにより標的遺伝子の成功のために不可欠です。
- incubatにより制限酵素を中和15のために68℃でり - 20分。
- 無菌H 2 O100μlの線形化プラスミドの少なくとも15μgのを準備トランスフェクションの時まで-20℃で保存して線形化pΔGOI。
Tの1.2準備トキソプラズマ原虫 、トランスフェクション、選択、サブクローニング、検証、アーカイブおよび遺伝子操作系統のメンテナンス
Tの文化や操作のための一般的な方法ヒト包皮線維芽細胞(HFF)細胞における原虫は 19-21に記載されている。 Ku80がΔΔhxgprt株は、1,2(1%ウシ胎児血清(FBS)および100倍希釈した抗真菌ストックから、1%抗生物質を補充したイーグル最小必須培地(EMEM)増殖培地)寄生虫感染媒体で正常に複製する。 トキソプラズマですべての作業は、バイオセーフティレベル2の手順を使用して実行する必要があります。 T.トキソプラズマRHΔKu80が2パレntal株はルース·ラボ14からRHΔhxgprt株から生成されました。 PruΔku801親株を安定的に統合されたCAT選択マーカーとブラディゾイト期特異的プロモーターのLDH2の制御下にある緑色蛍光タンパク質(GFP)レポーターが含まPruΔhxgprtPRU(gniaudひずみBSG-4 22)から生成されました。
- 1×10 6現実的なI型RHΔKu80をΔhxgprt寄生虫 、または2×10 6現実的なII型Prugniaud PRU()ΔKu80をΔhxgprtの寄生虫を持つ2つの25cm 2のフラスコに接種してコンフルエントHFF細胞を含む25cm 2のフラスコに接種。
注:生存寄生虫が新たに出た溶解細胞外寄生体として定義されています。
注: このスケールには、3つのトランスフェクション実験のための実行可能な寄生虫の十分な数が用意されています。
- 72時間感染後- ΔKu80がΔhxgprt感染文化〜68を点検。トランスフェクションの正確なタイミングを計画するHFF細胞の95%溶解 - 実行可能な寄生虫や〜90の存在を確認します。
注:低寄生虫の生存率は、遺伝子ターゲティング効率を廃止するので、新たにegressed寄生虫の分離は、このプロトコルの成功のために不可欠です。
- 25cm 2のフラスコにプラグシール蓋を閉じることにより溶液中に実行可能な寄生虫を攪拌し、精力的に蓋の内側またはフラスコの首に水などの液体を飛散することなく、成長表面の寄生虫をオフに攪拌する前後にフラスコを振る。
注意:寄生虫の収穫は、タイプIのために注射針のリリースプロトコルを必要としませんかIIΔKu80が菌株を入力します。
- 3μmの膜を含むフィルターホルダーに装着を10mlのシリンジに寄生虫ソリューションを移し、オープン15ミリリットルスクリューキャップチューブの上にフィルターユニットを配置。注射器は、15ミリリットルのスクリューキャップチューブに寄生虫をフィルタリング。
注:膜を通して寄生虫をフィルタリングすると、感染した細胞や細胞破片を除去します。
- 血球計数器を用いた寄生虫濃度(1ml当たりタキゾイトフォーム)を決定します。
注:オプション:寄生虫のソリューションを使用して、プラーク形成単位を設定する(PFU)アッセイ21 7を読んでされる- 8日後にトランスフェクションされた寄生虫(PFUが≥0.2の比率をタキゾイトため)の十分な実行可能性を判断する。
- ペレットは、1400×gで7分間寄生虫や寄生虫ペレットを乱すことなく〜0.4ミリリットルに上清を吸引。 1,40で2分間スピン0 XGと寄生虫ペレットを乱すことなく〜0.01ミリリットルに上澄み残り吸引。
- チューブの底をフリックで寄生虫ペレットを再懸濁して、すぐに4の寄生虫濃度を得るために、寄生虫をペレットにcytomix 23バッファー(1.33倍濃度)を追加- 5×10 7寄生虫/ mlのcytomix。
注:これらの追加機能は、遺伝子ターゲティングの効率を改善しないのでcytomixにATPとグルタチオンの追加を省略します。
- プロトコル1.1(ステップ26)から線形化pΔGOI100μlのアリコートを解凍。
- 寄生虫cytomix溶液(〜1.3から1.6×10 7寄生虫)0.3mlの転送プロトコル1.1(ステップ26)、からの線形化pΔGOIを100μlにし、すぐに冷やした2ミリメートルギャップ全体を0.4ミリリットル寄生虫+pΔGOIミックスを転送エレクトロポレーションキュベット。
- 1.4 kVの、24Ωで寄生虫をエレクトロ。
- 解像度トン〜5分の後、30ミリリットルの感染培地でコンフルエントHFF細胞を含む150センチメートル2フラスコに、トランスフェクションキュベットの内容全体を転送し、一晩感染した培養物をインキュベート室温でトランスフェクションキュベット。
- ミコフェノール酸の選択を開始+キサンチン(MPA + X)〜20時間の感染媒体を置き換えることにより、トランスフェクション後( 図2A)MPA + X選択培地(MPA(25μgの/ ml)およびキサンチン(50μgの/ ml)の中で感染培地)。
注:インキュベーションMPA + X選択フラスコを邪魔しないでください。
- 視覚的に単分子層を調べることによってMPA + X選択フラスコ〜8日、トランスフェクション後にPFUsの合計数を見積もります。光学顕微鏡による感染のPFUsと健康ゾーンの開発の存在を確認します。
注:プラーク日8により表示されていない場合は、感染の兆候選択およびモニタを維持する変異株加入したイオンが還元複製速度を有することができる。
注:ΔKu80 がΔhxgprt寄生虫株はかなり厳しいと変異株の正常な分離が可能になり、望ましくない非標的事象の極めて低いバックグラウンドを持っているが、致命的ではない成長の欠陥。遺伝子が必須であり、それを削除することはできません、主トランスフェクション、またはその後寄生虫人口を渡された場合、人口がターゲットノックアウトに解決として寄生虫が選択時に複製しなく表現型を明らかにするかもしれません。
- 単層から細胞外の寄生虫を取り除くために、ステップ3のようにフラスコを振る[見えるプラークが開発した後]寄生虫解を生成する。転送〜から寄生虫溶液0.5ml MPA + 25cm 2のへX選択フラスコMPA +コンフルエントHFF細胞(この文化パス1Aを指定する)を含むXセレクションフラスコ。
- 寄生虫を取り除くオリジナルの3日後に150センチメートル2 MPA + X選択フラスコ〜とコンフルエントHFF細胞(この文化パス1Bを指定する)を含む第二の25cm 2の MPA + X選択フラスコへの転送〜0.5ミリリットル寄生虫溶液中のS。
- 感染のゾーンが合格1Aに開発および/または〜5日間1Bフラスコを渡すことができます。視覚的にパス1Aと1Bフラスコ感染の健康ゾーンを持つ25実行可能なPFUsの最小が含まれていることを光学顕微鏡で確認します。
- 25cm 2のフラスコ中MPA + X選択培地の継続的な通過のための通過1Aまたはパス1Bフラスコのいずれかを選択します。 MPA + X選択の下の通路を維持する。
注意:寄生虫の前サブクローニングに母集団から非統合永続エピソームを削除する世代に十分な数の培養されなければならない。 I型RHΔKu80がΔhxgprtための20日後、トランスフェクションの前にサブクローニング、またはそれ以前の25日後にトランスフェクションのために実行しないでください非統合エピソーム1,2を希釈するのに十分な時間を確保するタイプIIプルデンシャルΔKu80をΔhxgprt寄生虫 。
- MPA + X選択培地でコンフルエントHFF細胞を含む96ウェルトレイ内のサブクローン寄生虫。 1寄生虫/ウェルと2寄生虫/ウェルの濃度で第2トレイの濃度で1トレイを準備します。
注:最近(〜90〜25センチメートル2フラスコ中HFF細胞の95%)を溶解したサブクローン寄生虫寄生虫が高い生存能力を持っていることを確認する。
注:サブクローニングのための最適な時間枠トランスフェクションが正常にターゲットに寄生虫は、選択した人口1で高頻度で発生するどのように迅速に測定することにより設立されました。
- 通路に続ける感染毎週通路スケジュールを使用してMPA + X選択培地中でトランスフェ寄生虫の主な人口寄生虫溶液10μlと25cm 2の HFFフラスコ。
注:ターゲット遺伝子欠失を(ノックアウト)を運ぶクローンをステップ18、resubclone連続維持集団から寄生虫の最初のサブクローニングから取得していない場合は〜10から12週間後にトランスフェクション。
注:遺伝子欠失が得られない理由の一つは、いくつかのpΔGOIプラスミドのエピソーム永続性から生じる。エピソーム永続性は、5 'および3'ゲノム標的脇腹とHXGPRT遺伝的要素から発生していないように含まれている配列によって決定されます。約5 - 標的プラスミドの10%が持続エピソームを希釈し、主に安定した組込みに人口を解決するために寄生虫の人口の世代十分な回数を可能にするためにサブクローニングの後の時間を必要とする重要なエピソーム永続性を示す。
- スコア96ウェルトレイ6 - で光顕微鏡で感染の単一ゾーンとして識別される単一のPFUを、含まれている井戸のために8日後サブクローニング(II型寄生虫) - 7日後サブクローニング(タイプI寄生虫)または7 40Xや60Xパワーのどちらか。井の中のPFUの場所を指定するために、96ウェルトレイ蓋の上に小さな点をマーク。
- 井戸に寄生虫を分散させるためにPFU以上流体の流れを導くことによって50μlの(200μlの先端)に設定ピペットを使用して単一のPFUを含む各の内容を混ぜる。
注:よく混合するHFF単層の寄生虫溶解を加速します。タイプI RH株がよく溶解します〜4日混合した後、II型するPRU寄生虫はよく溶解します〜5日、混合後。
- 同時に寄生虫の溶液6μlのアップ描きながら10μlのピペットチップ - 0.5を使用して、これらの溶解の各ウェルの下部に井戸(クローン)とスクラッチを溶解ダースを選択してください。トランス1ミリリットルMPA + X選択培地でコンフルエントHFF細胞を含む24ウェルトレイ内のウェルに寄生液6μlのFER。
注:開口部は井戸の底に存在する寄生虫と一定の接触にピペットチップのボア維持する井戸の底に傷をすることが不可欠である。
- HFF細胞の溶解のために、24ウェルトレイを監視します。
注:タイプI RH寄生虫は、通常3〜4日で、24ウェルフォーマットの井戸を溶解します。タイプIIするPRU寄生虫は通常3〜5日でよく溶解します。
- ウェルあたりコンフルエントHFF細胞と1ミリリットルMPA + X選択培地を含む新しい24ウェルトレイの対応するウェルに24ウェルフォーマットで各ウェルの底から傷実行可能な寄生虫溶液2μLを移します。
注:すべての寄生虫のクローンと線が連続してメインすることができますこの24穴トレイ形式で7日ごとに実行可能な寄生虫溶液2μLを渡すことによってtained。
- その寄生虫が正常に視覚的に光学顕微鏡〜18時間後に通路で検査することによって新たな24ウェルトレーに移したことを確認します。
- ステップ23から24ウェルトレーの溶解井戸から収穫寄生虫 - 24のどちらか1ミリリットルまたは10ミリリットルピペットを使用し、エッペンドルフチューブに寄生虫ソリューションを転送します。
- ペレット7分間、一回、1mlのリン酸リンス1,400×gで寄生虫は3分間1,400×gで再び生理食塩水(PBS)、ペレットを緩衝さ。
- ペレットからPBSを吸引し、寄生虫を再懸濁するためにペレットに200μlのPBSを追加します。 DNA単離するまで-80寄生虫ソリューション℃で凍結。
- 組織DNA分離ミニキットを用いて、各クローンから寄生虫DNAを単離する。
- 検証プライマー( 図2A-C)を用いたPCRによって標的遺伝子の欠失(ノックアウト)を検証。のためのテスト対象の遺伝子の機能的なコード領域の有無及び上流側と下流側CXF CR積ゲノムDNAプライマーを使用して、正しい5 'および3を実証するために、ゲノムターゲティングフランクとHXGPRTにまたがるPCR産物の存在について( 図2Aは-B)試験'削除された遺伝子座にHXGPRT遺伝子の統合を目標と。
注:検証用のプライマーは、目的の遺伝子に固有のまたは偽陽性の結果を得ることが可能でなければなりません。プライマー設計で検証することができますhttp://www.toxodb.orgプライマーが一意であることを確認するためにゲノム(複数可)にプライマー配列をブラスト。
- ステップ24で説明したように週単位のスケジュール上の通路寄生虫クローン。ターゲット削除が確認されていたら、MPA + Xの継続選択はオプションです。
- 冷凍庫の在庫を準備してアーカイブ寄生虫クローン。ペレット外寄生虫25cm 2の培養フラスコで溶解HFF細胞から、1mlあたり寄生虫濃度> 4×10 7寄生虫で細胞培養凍結培地中でメディアと優しく再懸寄生虫ペレットを削除します。液体窒素で無期限にクライオバイアルや店舗寄生虫にアリコートを転送、または-80℃で
2。 HXGPRTの削除
このプロトコルは、遺伝子操作されたΔKu80が株のゲノム中の統合サイトからHXGPRTマーカーを除去するために設計されています。 HXGPRTの除去は、マーカーの回復とHXGPRT 1-3に基づいてのみ選択を使用して、複数の遺伝子操作と株の生成が可能になります。以下に詳述するプロトコルはHXGPRTを削除する再標的遺伝子座方法が記載されているが、本目的の遺伝子座におけるHXGPRTの除去はまた、野生型遺伝子(cと同時再統合を可能にすることに留意すべきであるomplementation)、遺伝子操作のさまざまなことができる変異遺伝子の再統合と同様に、タグ付けされた遺伝子(N-またはC末端GFP、HAタグ、等)の再統合、。 6 -チオキサンチンの選択を使用して、アクション24のメカニズムとターゲティングプロトコルは、以前1-3,15に記載されている。
2.1削除HXGPRT
- HXGPRTします( 図1B)隣接するユニークな制限酵素部位で切断するRE.Zで消化によってpΔGOIから物品HXGPRT選択マーカー。
- アガロースゲル電気泳動によるDNAの完全な消化を確認する。アガロースゲルからの二つのバンドのうちの大きい方を隔離する。
注:二つのバンドの大きい方はHXGPRT遺伝子5 'および3' DNA標的脇腹とともにプラスミドではなく、含まれています。
- スピンカラム·レインインより大きなDNAバンドを含むアガロース部を配置gの膜上のフラットゲル。室温で4分間、13,000×gで列をスピン。 100μlのにボリュームを持ってフロースルーに滅菌H 2 Oを追加します。
注:他の商業の方法はアガロースからDNAを単離することが可能です。
- 18μlの滅菌H 2 Oの最終体積中のDNAを再懸濁し、エタノール沈殿によりDNAを濃縮する
- 7ミリリットルH 2 O、1μlの10×ライゲーション緩衝液および1μlのT4 DNAリガーゼ(5単位)で濃縮されたDNAの1μLを混合し、4℃で反応を配置°CpΔGOIc(pΔGOIclean)( 図3A)を生成するために一晩。
注:RE.Z含むDNA末端を有するDNA断片を標的と同様に変異遺伝子の再統合標的野生型遺伝子(相補性)の再統合標的化するのに適したプラスミドを作成するには、この段階で設計されており、含まれ得るタグ付けされた遺伝子の再統合(N-又はC-末端GFP、HAタグ、等)。
- エレクトロポレーションの前に無菌のH 2 Oとの反応の2倍に希釈する。
- E.にpΔGOIcを変換プロトコル1.1のように、 大腸菌 、サブクローニング分離、および標的プラスミドを検証する。次に(ステップ1.1.11に1.1.26を参照)をトランスフェクションの準備のためにpΔGOIc線形。
- プロトコル1.2(手順1〜11)で説明したように寄生虫のトランスフェクションプロトコルを繰り返します。
注:前のステップ1〜8を実施し、HXGPRTの目標除去が変異株で実現可能であることを確認します。 6 -チオキサンチン(6TX)選択培地(感染培地で200μgの/ mlの6TX)を使用して、変異株の1×10 6タキゾイトでコンフルエントHFF細胞150 cm 2とフラスコに感染し、PFUアッセイでフラスコを置きます。ない(または非常に少ない、<10)ことを確認するために、8日後にフラスコ点検を確立することが不可欠であるPFUが表示されていないが、その潜在効率を対象とした動遺伝子が低減さHXGPRT式 (6TX耐性表現型)を用いて表現型変異株のいずれかの潜在的な自然復帰を超える。
注:約5 - HXGPRT統合サイトの10%がHXGPRTマーカーがまだ標的部位(説明のために代表的な結果の項を参照)に統合されているにもかかわらず6TX抵抗の重要かつ自発的な周波数を示す。
- 6TX選択培地、150 cm 2とフラスコ内の培地を変更することにより、6TX選択〜20時間後、トランスフェクションを開始します。
注:〜10日後にトランスフェクションのためのフラスコを邪魔しないでください。
- 12トランスフェクション(タイプI寄生虫)または日- 10 - 16トランスフェクション(II型寄生虫)10日目でのPFUの形成のためのフラスコを点検。
注:ParasiHXGPRTの削除の対象とされているTESはHXGPRT遺伝子とmRNAが削除されるまで6TX選択下成長し始めることはありません、と残留HXGPRTタンパク質が不活性化される。
- HFFコンフルエント細胞を5mlの6TX選択培地を含む新しい25cm 2のフラスコに寄生虫溶液1.0mlを加え-寄生虫溶液、および転送0.5を作成するために、選択フラスコを振る。さらに2日間のために一日一回、一次150センチメートル2から新しい25cm 2のフラスコへの転送を繰り返します。
注:複数のサンプリングがHXGPRT遺伝子を失っている現実的な目標と寄生虫を取り込むチャンスを増加します。
- 健康複製寄生虫のゾーンでPFUs開発の存在を確認するために〜5日感染後25cm 2のフラスコを点検。感染のゾーンを含む、1つまたは2つのフラスコを選んでください。
注: 寄生虫は6TX選択で正常な成長率でHXGPRT遺伝子複製の削除。
- 6TX選択培地内の通路寄生虫をし続けています。
- 選択の30日 - 25後に寄生虫人口サブクローニング。 〜1寄生虫/ウェルと〜2寄生虫/ウェルとを別の96ウェルトレイを設定します。
- プロトコル1.2(ステップ19から29)によると孤立したクローンから寄生虫DNAを調製し、24ウェルフォーマットの文化の中でクローンを維持する。
- 図3A-Bに概説された戦略を用いてPCRによりHXGPRTの削除を検証します。
3。タンパク質のC-末端タギング
このプロトコルは、MPAを用いた遺伝子のゲノム遺伝子座に相同組換えを介して二重断面を介して統合のためのタグを標的タンパク質のC-末端タグ付けのために設計されて+ X選択2,4。このプロトコルは、効率的に動作するための5 'DHFRシーケンス HXGPRTマーカーが他の遺伝子2,4のために完全に検証機能の3 '非翻訳領域である。
内因性遺伝子座におけるタンパク質の3.1直接C末端タギング
- 組換え酵母クローニング( 図4)とプロトコル1.1に記載された方法を用いて構築したプラスミドpΔGOItagターゲティング作成する。選択したタグ(HAタグ、Mycのタグ、Hisタグに5 '終止コドンを位置3へ移動されることを除いて、ゲノム標的脇腹は、GOIのコード領域の最後の800〜1,200塩基対(またはゲノムDNA)が含まれています'など)
- ( 図4)で概説戦略を使用してプロトコル1.1と1.2の手順に従って、タンパク質をコードする遺伝子の内因性遺伝子座にC末端タグの標的挿入を作成します。
- PCRストラテジーを使用して内因性遺伝子座におけるC末端タグの挿入を確認する。
- a〜dを1プロトコルとプロトコル2に記載の方法を用いてリターゲット遺伝子座タグ付けされたタンパク質を発現正確規制内因性遺伝子座を作成するHXGPRT選択マーカーelete。このプロトコルは、タグ付けされた株(プロトコル1参照)内の別の遺伝子座を標的とするために再び用いることができるHXGPRT選択マーカーを回復する。
結果
詳細なテンプレートは、制限酵素部位の配置とでHXGPRTその後の削除のための遺伝子ターゲッティング、遺伝子の検証が目標とする、ならびにプラスミド構築を促進するプライマーの生成を含む、遺伝子ターゲティングを削除するプラスミドを構築するために設けられている単一ステップのプロセス( 図1A-C、図2Aは 、 図3A)。一般的な概略図は、例えば、標的遺伝子の欠失( 図2A)、ノックアウトの削除を検証するために使用されるプライマーペアを作るために提示され、タイプI rop18( 図2B)、およびPCR検証の代表的な結果が示されている( 図2C)。この代表的な結果は、ターゲットが比較的困難である遺伝子座で得ることができる結果の範囲を例示するために示されている。正常標的遺伝子欠失ラーニング遺伝子の有無をもたらすterest PCR産物(PCR 1)、3の存在'ゲノム標的脇腹(PCR2)、そして適切に5の間に統合HXGPRT選択可能なマーカーの存在'と(PCR3とPCR4)削除を定義する3 'ゲノム標的脇腹。クローン3、4、5、6、8、9、および11はHXGPRTは GOI( 図2C)を置き換えて標的遺伝子の欠失(ノックアウト)が検証される。
遺伝子欠失を含まないクローンは、様々なバンドパターンで表すことができる。クローン1及び2( 図2C)が見られるように一般的には、遺伝子欠失なしのクローンはPCR1とPCR2について観察されたバンドではなく、PCR3またはPCR4ため親株(親のパターン)をミラーします。この "親のパターンは、"いくつかの潜在的なメカニズムから生じる。時折、MPA耐性選択寄生虫はプラスミドターゲティングpΔGOIの非統合永続エピソームを運び、我々は継続的な選択は、多くの場合、これらのエピソームを強制することに注意してください統合することができます。我々はまた、私はΔはpΔGOIの意図しない統合のためにKu80が株はDHFR 5 'および3'プロモーターエレメント、DHFR遺伝子座のどちらかに、または部分的に削除されたHXGPRTに介して表現HXGPRTのcDNAを含むプラスミドターゲティング型のいくつかの珍しい背景を観察する軌跡14。このまれなイベントはターゲットの統合ですが、それは意図された統合はなかったし、任意の遺伝子置換戦略を設計する際に覚えておくべき重要なポイントとして機能します。プラスミドターゲットとするpΔGOIで運ば120塩基対よりもDNAの相同性は、望ましくないバックグラウンドをバック与えることができるΔKu80が株2組換えのための代替サイトを提供することがあります。 HXGPRT選択マーカーは、タイプII型DNAと比較した場合、塩基多型を持って、私は配列に基づいているため、II型プルデンシャルΔKu80が株ではこのような背景が減少または存在しない私を入力する比較される大幅II型プルデンシャルΔKu80がひずみ1を用いた実験では、この希少なバックグラウンドが減少。遺伝子座が不可欠である(削除できません)、または遺伝子座頻度をターゲティング極めて低い遺伝子を持っている、または永続[統合されていない]エピソームを簡単に成長し、選択を介して排泄されていない場合、この親のパターンは、MPAで観察されたパターンを支配する場合耐性クローン。また、標的DNA分子は、時には唯一の5 'または3'ゲノム標的脇腹とクローン10(5のために見られるようなバンドがPCR1とPCR2とともにPCR3またはPCR4(両方ではない)のどちらかのために観察さで統合することが観察される'統合)とクローン7(3'統合)( 図2C)。これらのパターンはHXGPRTを統合したが、この目標との統合は、目的の遺伝子を削除しません座で標的プラスミドの統合上の単一のクロスのまれな発生を示唆している。このパターン( 図2Cにおけるレーン7&10)EMその目標と統合が代わりに発生したかどうかを確認するためのPCRデータを報告する必要がphasizesシングルは相同渡ると標的分子の非相同第2の積分。まれに、私たちは親のパターンとターゲット削除パターンの両方を表しているクローン12( 図2C)で表される遺伝子混合物を観察していません。このパターンは、最も可能性が高いだけでなくクローニングで同じ場所に存在している2寄生虫の遺伝子型からの機会に発生します。
Δku80ΔgoiからHXGPRTの除去のための一般的な回路図:: HXGPRT株からHXGPRTを削除するには( 図3A)が示されている。クローン4、図7、図9、図11、図12( 図3C)に見られるように、ΔKu80がΔGRA2 :: HXGPRT( 図3B)からHXGPRTの除去を検証するために使用されるプライマー対は一意でPCR5〜1.2 kbpの帯域を増幅する。 HXGPRTは軌跡〜3から削除されていない場合。4 kbpのバンドは(クローン1、2、3、6、8、10、およびペアレンタルコントロール)が観察される。いくつかのメカニズムはHXGPRTの統合(MPA + X選択)よりHXGPRTの除去(6TX選択)がより困難の少ない効率的にするために行動する。 MPAの選択は6TXセレクション14,16より単により効率的です。また、HXGPRT発現のより高いレベルは、MPAの選択16,24よりも6TX選択のために必要とされる。したがって、HXGPRT酵素活性又は突然変異または遺伝子座でのHXGPRTの発現レベルを減少させるエピジェネティックな変化を低減することができる突然変異は、潜在的に6TX選択の効果を廃止することができる。さえ6TX選択、これらの課題に、ΔKu80が株で6TX選択の成功率は1-3の最初の試みで、90%以上である。
タンパク質をコードする遺伝子の直接的なC-末端タグ付けのための一般的なスキームは、( 図4)が示されている。このスキームはHXGPRT 3 'タグ付けされた遺伝子のも上記と同様の検証戦略を採用して相同組換えを介して二重のクロスを介して直接統合を使用しています。遺伝子は、これは独立して単離されたプラスミド標的DNAと第二のトランスフェクションを使用して検証することができます必須遺伝子であることが疑われる場合。このような調節遺伝子発現のための制度などの代替の方法は、遺伝子が25必要不可欠である場合、さらに検証するために利用可能です。
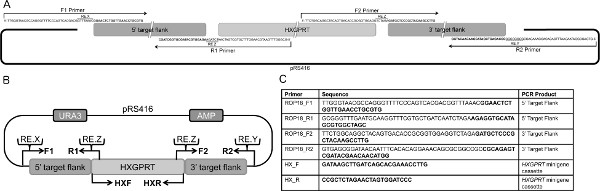
図1。プラスミド標的DNAの設計の概要。 PCRに使用される重複プライマーを設計する。回路図pRS416シャトルベクターとHXGPRTのミニ遺伝子カセットのための33 bpのオーバーラップで5 'および3'ターゲット脇腹を増幅。回路図に描かれたプライマーは5 'および3'pΔROP1810の 目標フランク。ターゲティングDNA分子を設計するためB.総合的な戦略を生成するために使用される。 pΔGOIのバックボーンはpRS416シャトルベクター含むウラシル(URA)とアンピシリン(AMP)選択マーカーである。 〜1 kbpの5 pRS416に挿入'F1及びR1プライマーを用いてHXGPRTミニ遺伝子カセット及びpRS416ための重なりがゲノムDNAから増幅されたDNA標的フランクは、約1 kbpの3' DNA標的フランクはについての重なりで、ゲノムDNAから増幅されるHXGPRTのミニ遺伝子カセット及びF2とR2プライマーとHXGPRTのミニ遺伝子カセットを使用pRS416。次のユニークな制限酵素消化サイトはプライマーに追加されますF1プライマー中RE.X(制限酵素Xカットサイト)R2ではRE.Y、DNA標的横腹5 '5'末端にプラスミドをカットする消費税HXGPRTの minにR1とF2プライマーでDNA標的横腹とRE.Z 3 '3'末端にプラスミドを削減するプライマーrop18の削 除のためのターゲティング構築物を生成するために使用igeneカセット℃のプライマー配列。 大胆な領域がTに対応原虫ゲノム配列、そして太字以外の地域はpRS416とHXGPRTのミニ遺伝子カセットと重複制限酵素部位と配列に対応する。 HX_FとHX_Rプライマー対はHXGPRTミニ遺伝子カセット14を増幅する。プライマーは、5 'から3'へ読み。フェントレスら 10から適応表は大きい数字を表示するには、ここをクリックしてください 。

図2。 HXGPRTを用いた遺伝子の欠失のためのプロトコルの概要。 。Disruptionは1〜KBP 5 'DNA標的脇腹と〜1 kbpの3'プラスミドpΔGOIにDNA標的横腹を使って二重交差相同組換え事象によってΔku80Δhxgprt株におけるGOIの。 PCRのPCR1を使用して戦略、PCR2、PCR3、およびPCR4は(ノンスケール)GOI座の標的とHXGPRTの統合と削除をクローンを検証するために遺伝子型の検証を可能にすることが示されている。B.描写プライマー対rop18の削 除を検証するために設計された。ROP18_DFとROP18_DRはPCR1、ROP18_ExFとROP18_CxRはPCR2、ROP18_CxFとDHFR_CxRがPCR4を( 図2Aを参照)を増幅PCR3とDHFR_CxFとROP18_CxRを増幅する増幅する増幅する。プライマーは、5 'から3'へ読み。表はフェントレスらからHXGPRTを使っ標的遺伝子欠失(rop18)の検証の10℃の代表的な結果を適応した。MPAにおけるΔku80Δhxgprtと選択にプラスミドpΔROP18のトランスフェクション+X、MPA + X耐性クローンはrop18の削 除についてアッセイした。親株コントロールPCR 3(〜1,200 BP)およびPCRのためのPCR 1(〜400塩基対)およびPCR 2(〜650 bpの)製品と負のために肯定的である4(〜1,300 BP)製品。ターゲットGOIノックアウトはPCR2、PCR3とPCR4製品の正であり、PCR 1製品に対して否定的である( 図2Aを参照)。示さPCR1とPCR2( トップパネル)、PCR3( 中央パネル)、およびPCR4( 下パネル)の結果の代表パネルです。クローン3、4、5、6、図8、図9及び図11は、クローン内の標的遺伝子の削除イベントを定義するPCR 1、PCR2、PCR3 PCR4との正確なバンドパターンを示す。クローン1、図2、図7、図10および図12は、遺伝子欠失はなく、他の潜在的な代表的な結果に対応する。 (C =ペアレンタルコントロールひずみΔku80Δhxgprt、M =サイズマーカー) より大きい数字を表示するには、ここをクリックしてください 。

図3。 HXGPRTを削除するには、興味のある再遺伝子ターゲティングのためのプロトコルの概要。ひずみΔku80Δgoiで二重交差相同組換え事象によってHXGPRT選択マーカーの除去のためのA.戦略 ::〜1 kbpの5 '側DNA標的脇腹と〜1 kbpの3'プラスミドpΔGOIc上のDNA標的横腹を使用HXGPRT。遺伝子型を検証するためのPCR戦略は(正確な縮尺ではない)削除をまたがるPCR産物(PCR5)するためのアッセイにプライマー対を用いて示されている。B. GRA2からHXGPRT選択マーカーの除去を検証するために設計された代表的なプライマー対ひずみΔku80Δgra2における軌跡:: HXGPRT。プライマーGRA2 _CLFとGRA2_CxRはPCR5を増幅。プライマー 5 'から3'から読み取る。Δku80ΔgoiプラスミドpΔGOIcのトランスフェクション:: 6TXでHXGPRTと選択後に取得することができる6TX耐性クローンのC.描写パネルが。クローン4、7、9、11、12展示HXGPRTマーカーの対象と削除(〜1.2 KBPの横腹5 'と若干重なると脇腹' 3にまたがる製品と3外'に対応PCR5の正しいバンドパターン脇腹)。クローン1、2、3、6、8、10(このバンドは光である)、これらのクローンが許容6TXに対する抵抗性の程度を示したにもかかわらず、親クローンの遺伝子型に対応する3.4 kbpの〜周りの予想されるバンドパターンを表す彼らの選択(この表現が時折(プロトコル2.1(ステップ8)でノートと代表的な結果の項を参照 )シャットダウンHXGPRTの発現により、自発的に起こることができます(C =ペアレンタルコントロール株Δku80Δgoi:: HXGPRT、M =サイズマーカー)。w.jove.com/files/ftp_upload/50598/50598fig3large.jpg "ターゲット=" _blank ">より大きい数字を表示するには、ここをクリックしてください。

図4。タンパク質のC末端にタグを対象としたDNA分子の設計。戦略はまた、3 '下流調節領域として機能するHXGPRTマーカーの能力に基づいている。 pΔGOItagのバックボーンはpRS416シャトルベクター含むウラシル(URA)とアンピシリン(AMP)選択マーカーである。 DNA標的横腹ため重なってゲノムDNAから増幅された約1 kbpの5 'Fgoiとrtagのプライマー、〜1 kbpの3使用HXGPRTのミニ遺伝子カセット及びpRS416ため重なってゲノムDNAから増幅したDNA標的横腹'はpRS416挿入F2とR2を使用してHXGPRTミニ遺伝子カセット及びpRS416プライマーとHXGPRTのミニ遺伝子カセット。 5 'DNA標的横腹、交換終止コドンに続くタグ(HA、Mycの、彼の、など)と終止コドンを置き換えます。 MPA + X選択は、C末端タグと下流HXGPRTを統合し、HXGPRTマーカーに続く位置に3 'UTRに移動します。次のユニークな制限酵素消化サイトはプライマーに追加されます。FgoiプライマーでRE.X(制限酵素Xカットサイト)DNA標的横腹5 '5'末端にプラスミドを切断し、RE.Yにするプラスミド構築に使用する再標的にタグ付けされた遺伝子座削除するためのHXGPRTのミニ遺伝子カセット消費税にrtagのとF2プライマーでDNA標的逃げ3 '3'末端にプラスミド、及びRE.ZをカットするR2プライマー3 'UTRの配置を復元しますHXGPRTマーカー 。
ディスカッション
ここでは、対象となる遺伝子欠失、遺伝子の交換、および/ またはタグ付けされた遺伝子を持っている遺伝子操作子孫の効率的な回収を可能にするためにΔKu80が寄生虫株にターゲティング効率的な遺伝子のためのプロトコルを提供する。これらのメソッドの逐次実行は1-3単一または複数の標的遺伝子操作を含む寄生虫変異体を単離するための信頼性のあるアプローチを提供します。ターゲット削除の世代が複数の要因に依存しながら、提示戦略とプロトコルΔKu80が株の使用が大幅にTの正確に定義された変異株の作製効率と使いやすさを向上させ機能的ゲノム研究のためのトキソプラズマ 。
Tのゲノム無性段階でトキソプラズマは半数体である。したがって遺伝子を削除することを計画したときにするための考慮事項は、遺伝子がin vitroで不可欠であってはならないということです。それにもかかわらず、厳しく監督の効率Ku80が Δ株における影の遺伝子ノックアウトは非常に高く、これが著しく損なわ複製速度を有する変異株の単離を可能にする。標的遺伝子が不可欠である場合には、寄生虫はしばしば選択時に複製することをやめる。標的遺伝子操作で別の重要な要因は、ゲノム標的側面 の少なくとも620塩基対とその完璧な相同性は2検出ターゲット統合を得るために必要とされています。同性の長い地域約1,000塩基対の標的DNAの脇腹を標的とし、使用して、より高い効率を生み出す信頼性と効率的なアプローチ1-4です。このような理由からゲノムターゲティングフランクがT.の同じ株から生成されることが重要である遺伝的に操作されている株とトキソプラズマ (RH、プルデンシャル)。十分な相同性の欠如は、しばしば1ゲノム標的脇腹の目標と統合になりますが、他のことはできません。不完全な統合やノックアウトMAを取得するために失敗yはまたエピソーム永続に起因する。すぐにトランスフェクション後、標的分子のすべてがエピソームを非統合され、時には標的分子は、多くの世代のためにエピソームとして存続することができます。 KBP再サブクローニングする前に選択の下に寄生虫を渡すために継続〜1の標的DNAの両サイドを使用すると、頻繁にエピソーム永続に関連する問題を解決します。
ノックアウトが検証されるとHXGPRTマーカーが削除されると、遺伝子ターゲティング戦略は1-3単一の寄生虫株で複数の遺伝子欠失を生成する第二GOIに繰り返すことができます。欠失、置換、またはタグ遺伝子の発生は、異なる選択可能なマーカー( ブレオマイシン、CAT、ピリメタミン耐性DHFR等)を使用することによって達成することができる。 CAT選択可能なマーカーは、現在Ⅱ型ΔKu80がプルデンシャルゲノム1で存在しているが、このマーカーはHXGPRTを使用して除去することができますを選択プロトコルは、ここで説明する。私たちの手で、HXGPRT選択が単一コピー対象と挿入がMPA + X培地。HXGPRTで堅牢な選択のための十分である最高ターゲティング効率が最も安全で効率的なマーカーでは、またのターゲット削除のため、現在有用なアプローチを提供しています6 -チオキサンチン(6TX)選択2,3を使用して選択可能なマーカー( 図3)。したがって、このプロトコルは、複数のターゲット遺伝子操作で定義された変異株を開発するための唯一の現在のアプローチを提供します。
このプロトコルは、 トキソプラズマのΔKu80が株で標的遺伝子操作のための貴重かつ効率的な方法を提供し、単一の遺伝子、遺伝子の家族、またはゲノムワイドな機能ゲノム研究の解析に適用することができる。 ΔKu80が株の可用性1,2に先立ち、これらのアプローチが原因伊根に広く現実的ではありませんでしたこれらのメソッドのfficiency。したがって、このプロトコルは、 トキソプラズマの標的遺伝子操作のために広く適用可能であり、寄生虫学、ホストの応答、および機能ゲノミクスに関する質問の範囲に対処するに興味を持ってすべての研究者にアクセス可能である。
開示事項
著者は、彼らが競合する経済的利益を持っていないことを宣言します。
謝辞
この作品はDJBにNIHから助成金(AI041930、AI073142、AI075931及びAI091461)によってサポートされていました。
資料
| Name | Company | Catalog Number | Comments |
| Name of Reagent/Material | Company | Catalog Number | Comments |
| Overlap Primers | Integrated DNA Technologies | 4 nmole Ultramer DNA oligos | |
| Validation Primers | Integrated DNA Technologies | 100 nmole DNA Oligo | |
| Yeast Strain #90845 | ATCC | Designation FY834 | |
| Shuttle Vector pRS416 | ATCC | 87521 | |
| DH10B E. coli | Invitrogen | 12033-015 | SOC broth in kit with E. coli |
| Resuspension Buffer | Qiagen | Buffer P1 in QIAprep Spin Miniprep Kit | |
| Miniprep Kit | Qiagen | 27104 | QIAprep Spin Miniprep Kit |
| Glass Beads | Scientific Industries | SI-BG05 | 0.5 mm acid-washed |
| Qiacube Automated Robotic Work Station | Qiagen | ||
| Electroporation Cuvette | USA Scientific | 9104-5050 | 2 mm-gap |
| BTX600 electroporator | BTX | ||
| Maxiprep Kit | Qiagen | 12662 | QIAprep Spin Maxiprep Kit |
| 25 cm2 Canted neck plug seal flask | Corning | 430168 | |
| 150 cm2 Canted Neck plug seal flask | Corning | 430823 | |
| Human Foreskin Fibroblasts (HFF) cells | ATCC | SCRC1041 | |
| Swin-lok Filter Holder | Whatman | 420200 | 25-mm-diameter |
| Membrane | Whatman | 110612 | Nucleopore Track-etched Membrane 3 mm pore size, 25-mm-diameter |
| Mycophenolic acid (MPA) | Sigma | ||
| Xanthine (X) | Sigma | ||
| 96-well Tissue Culture Tray | Corning Costar | ||
| 24-well Tissue Culture Tray | Corning Costar | ||
| MEM Eagle Media | Lonza Biowhittaker | 12-611F | |
| Fetal Bovine Serum | Gibco | 26140-111 | |
| Antibiotic-Antimycotic (Anti-Anti) | Gibco | 15240-062 | |
| Spin Column | Primm Labs | PAE-100 | Easy Clean DNA Extraction Spin Kit |
| T4 DNA Ligase | New England Biolabs | ||
| 6-thioxanthine | Acros Organics | ||
| Tissue DNA Minikit | Qiagen | 51104 | QIAamp DNA Blood Mini Kit |
| Cell Culture Freeze/Recovery Media | Gibco | 126-48-010 | |
| Phosphate Buffered Saline | Hyclone | SH30028.02 | minus calcium, minus magensium |
参考文献
- Fox, B. A., et al. Type II Toxoplasma gondii KU80 knockout strains enable functional analysis of genes required for cyst development and latent infection. Eukaryot. Cell. 10, 1193-1206 (2011).
- Fox, B. A., Ristuccia, J. G., Gigley, J. P., Bzik, D. J. Efficient gene replacements in Toxoplasma gondii strains deficient for nonhomologous end joining. Eukaryotic Cell. 8, 520-529 (2009).
- Fox, B. A., Bzik, D. J. Avirulent uracil auxotrophs based on disruption of orotidine-5'-monophosphate decarboxylase elicit protective immunity to Toxoplasma gondii. Infection and Immunity. 78, 3744-3752 (2010).
- Hortua Triana, M. A., et al. Biochemical and molecular characterization of the pyrimidine biosynthetic enzyme dihydroorotate dehydrogenase from Toxoplasma gondii. Molecular and Biochemical Parasitology. 184, 71-81 (2012).
- Kim, K., Weiss, L. M. Toxoplasma gondii: the model apicomplexan. Int. J. Parasitol. 34, 423-432 (2004).
- Gajria, B., et al. ToxoDB: an integrated Toxoplasma gondii database resource. Nucleic Acids research. 36, 553-556 (2008).
- Kim, K., Weiss, L. M. Toxoplasma: the next 100 years. Microbes and infection / Institut Pasteur. 10, 978-984 (2008).
- Butcher, B. A., et al. Toxoplasma gondii rhoptry kinase ROP16 activates STAT3 and STAT6 resulting in cytokine inhibition and arginase-1-dependent growth control. PLoS pathogens. 7, e1002236 (2011).
- Daher, W., Klages, N., Carlier, M. F., Soldati-Favre, D. Molecular characterization of Toxoplasma gondii formin 3, an actin nucleator dispensable for tachyzoite growth and motility. Eukaryot. Cell. 11, 343-352 (2012).
- Fentress, S. J., et al. Phosphorylation of immunity-related GTPases by a Toxoplasma gondii-secreted kinase promotes macrophage survival and virulence. Cell Host Microbe. 8, 484-495 (2010).
- Musiyenko, A., Majumdar, T., Andrews, J., Adams, B., Barik, S. PRMT1 methylates the single Argonaute of Toxoplasma gondii and is important for the recruitment of Tudor nuclease for target RNA cleavage by antisense guide RNA. Cellular microbiology. 14, 882-901 (2012).
- Straub, K. W., Peng, E. D., Hajagos, B. E., Tyler, J. S., Bradley, P. J. The moving junction protein RON8 facilitates firm attachment and host cell invasion in Toxoplasma gondii. PLos Pathog. 7, e1002007 (2011).
- Szatanek, T., et al. Cactin is essential for G1 progression in Toxoplasma gondii. Molecular Microbiology. 84, 566-577 (2012).
- Donald, R. G., Carter, D., Ullman, B., Roos, D. S. Insertional tagging, cloning, and expression of the Toxoplasma gondii hypoxanthine-xanthine-guanine phosphoribosyltransferase gene. Use as a selectable marker for stable transformation. J. Biol. Chem. 271, 14010-14019 (1996).
- Donald, R. G., Roos, D. S. Gene knock-outs and allelic replacements in Toxoplasma gondii: HXGPRT as a selectable marker for hit-and-run mutagenesis. Molecular and Biochemical Parasitology. 91, 295-305 (1998).
- Pfefferkorn, E. R., Borotz, S. E. Toxoplasma gondii: characterization of a mutant resistant to 6-thioxanthine. Exp. Parasitol. 79, 374-382 (1994).
- Gietz, R. D., Schiestl, R. H. Frozen competent yeast cells that can be transformed with high efficiency using the LiAc/SS carrier DNA/PEG method. Nature Protocols. 2, 1-4 (2007).
- Oldenburg, K. R., Vo, K. T., Michaelis, S., Paddon, C. Recombination-mediated PCR-directed plasmid construction in vivo in yeast. Nucleic Acids Res. 25, 451-452 (1997).
- Fox, B. A., Gigley, J. P., Bzik, D. J. Toxoplasma gondii lacks the enzymes required for de novo arginine biosynthesis and arginine starvation triggers cyst formation. Int. J. Parasitol. 34, 323-331 (2004).
- Roos, D. S. Molecular genetic tools for the identification and analysis of drug targets in Toxoplasma gondii. Curr. Top. Microbiol. Immunol. 219, 247-259 (1996).
- Roos, D. S., Donald, R. G., Morrissette, N. S., Moulton, A. L. Molecular tools for genetic dissection of the protozoan parasite Toxoplasma gondii. Methods in Cell Biology. 45, 27-63 (1994).
- Singh, U., Brewer, J. L., Boothroyd, J. C. Genetic analysis of tachyzoite to bradyzoite differentiation mutants in Toxoplasma gondii reveals a hierarchy of gene induction. Molecular Microbiology. 44, 721-733 (2002).
- vanden Hoff, M. J., Moorman, A. F., Lamers, W. H. Electroporation in 'intracellular' buffer increases cell survival. Nucleic Acids Research. 20, 2902 (1992).
- Pfefferkorn, E. R., Bzik, D. J., Honsinger, C. P. Toxoplasma gondii: mechanism of the parasitostatic action of 6-thioxanthine. Exp. Parasitol. 99, 235-243 (2001).
- Mital, J., Meissner, M., Soldati, D., Ward, G. E. Conditional expression of Toxoplasma gondii apical membrane antigen-1 (TgAMA1) demonstrates that TgAMA1 plays a critical role in host cell invasion. Mol. Biol. Cell. 16, 4341-4349 (2005).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved