Method Article
Test dei biomarcatori sintetici delle urine multianalitiche mediato da CRISPR-Cas per la diagnostica portatile
In questo articolo
Riepilogo
Questo protocollo descrive un test di biomarcatori urinari sintetici multianalita mediato da CRISPR-Cas che consente la diagnostica del cancro point-of-care attraverso l'analisi ex vivo delle attività della proteasi associata al tumore.
Abstract
La creazione di biomarcatori sintetici per lo sviluppo di strumenti diagnostici di precisione ha consentito di rilevare le malattie attraverso percorsi diversi da quelli utilizzati per le tradizionali misurazioni dei biofluidi. I biomarcatori sintetici generalmente fanno uso di reporter che forniscono segnali leggibili nel biofluido per riflettere le alterazioni biochimiche nel microambiente locale della malattia durante l'incidenza e la progressione della malattia. La concentrazione farmacocinetica dei reporter e l'amplificazione biochimica del segnale della malattia sono fondamentali per ottenere un'elevata sensibilità e specificità in un test diagnostico. Qui, una piattaforma diagnostica del cancro è costruita utilizzando un formato di biomarcatori sintetici: nanosensori basati sull'attività che trasportano reporter di DNA stabilizzati chimicamente che possono essere liberati da firme proteolitiche aberranti nel microambiente tumorale. Il DNA sintetico come reporter di malattie offre capacità di multiplexing attraverso il suo uso come codice a barre, consentendo la lettura di più firme proteolitiche contemporaneamente. I reporter di DNA rilasciati nelle urine vengono rilevati utilizzando le nucleasi CRISPR tramite ibridazione con RNA CRISPR, che a loro volta producono un segnale fluorescente o colorimetrico al momento dell'attivazione enzimatica. In questo protocollo, vengono costruiti nanosensori basati sull'attività con codice a barre del DNA e la loro applicazione è esemplificata in un modello murino preclinico di cancro colorettale metastatico. Questo sistema è altamente modificabile in base alla biologia della malattia e genera più segnali di malattia contemporaneamente, consentendo una comprensione completa delle caratteristiche della malattia attraverso un processo minimamente invasivo che richiede solo la somministrazione di nanosensori, la raccolta delle urine e un test cartaceo che consente la diagnostica point-of-care.
Introduzione
Nonostante lo sforzo significativo per identificare i biomarcatori tumorali come le proteine e il DNA, il campo diagnostico del cancro è stato messo a dura prova dalla loro bassa abbondanza o dalla rapida degradazione incircolazione. Come strategia complementare, i biomarcatori sintetici bioingegnerizzati che rispondono selettivamente alle caratteristiche della malattia per generare segnali amplificati rappresentano nuove strade verso una diagnostica accurata e accessibile 2,3. Per facilitare il rilevamento, questi biomarcatori sintetici sfruttano i meccanismi di attivazione dipendenti dal tumore, come l'amplificazione enzimatica, per produrre analiti con un migliore rapporto segnale/rumore4. In questo caso, una classe di enzimi associati al cancro, le proteasi, viene sfruttata per attivare sensori iniettabili su scala nanometrica per rilasciare reporter di malattia rilevabili dai biofluidi come sangue o urina 5,6. Alla luce dell'eterogeneità del tumore, lo sviluppo di un pannello di sensori attivati dalla proteasi consente di eseguire test multianalita che combinano diversi eventi di scissione della proteasi in una "firma della malattia" per valutare l'incidenza e la progressione del cancro in modo più specifico e multiplexato.
Sono stati sviluppati biomarcatori sintetici attivati dalla proteasi che comprendono substrati peptidici coniugati alla superficie di un carrier inerte7. Quando iniettati in vivo, questi peptidi vengono trasportati al tumore, dopodiché la scissione enzimatica da parte delle proteasi tumorali rilascia reporter nel sangue o nelle urine per il rilevamento. Il rilevamento multiplexato con biomarcatori sintetici attivati dalla proteasi richiede che ogni biomarcatore sintetico all'interno di un cocktail sia etichettato con un codice a barre molecolare univoco. A tal fine, sono stati sviluppati vari approcci, tra cui i codici a barre di massa e i reporter codificati con ligando 8,9,10. A differenza di questi metodi di multiplexing, che possono essere limitati a poche diverse possibilità di segnale, il DNA barcoding offre molte più combinazioni in conformità con l'elevata complessità ed eterogeneità degli stati patologici umani. Per espandere la multiplexità dei biomarcatori sintetici, i sensori sono codificati a barre etichettando ogni reporter con una sequenza di DNA univoca per il rilevamento tramite nucleasi CRISPR-Cas per amplificare il segnale biofluidico ex vivo. Questi codici a barre di DNA a singolo filamento (ssDNA) sono progettati per legarsi agli RNA guida CRISPR complementari (crRNA), attivando l'attività nucleasica collaterale innescata dal bersaglio di CRISPR-Cas12a11. Questa attività nucleasica può essere impiegata per scindere un filamento di DNA reporter rilevato attraverso la cinetica di fluorescenza o utilizzando strisce di carta.
Oltre all'amplificazione molecolare tramite proteasi (in vivo) e CRISPR-Cas (ex vivo), un'altra caratteristica chiave della progettazione dei biomarcatori sintetici attivati dalla proteasi riguarda lo sfruttamento della farmacocinetica dei nanomateriali per aumentare la concentrazione del segnale diagnostico nei biofluidi10. Un approccio è l'uso di un vettore di nanoparticelle per aumentare il tempo di circolazione dei substrati peptidici coniugati in superficie. Un dendrimero di polietilenglicole (PEG) viene selezionato come nanovettore con diametro idrodinamico e multivalenza relativamente piccoli per aumentare la consegna ai tumori. Sebbene sia abbastanza piccolo da promuovere la somministrazione del tumore, la dimensione del vettore PEG è maggiore del cut-off di ~5 nm della barriera di filtrazione glomerulare renale in modo che solo i substrati peptidici scissi possano essere eliminati nelle urine, sfruttando la filtrazione dimensionale da parte dei reni12. In questo protocollo, viene delineato il flusso di lavoro a più fasi per la sintesi e l'applicazione di nanosensori basati sull'attività con codice a barre del DNA in un modello murino preclinico, evidenziando la configurazione del test del biomarcatore urinario sintetico multianalita mediato da CRISPR-Cas, che è stato impiegato da questo gruppo per classificare lo stato della malattia in modelli murini di più tipi di cancro13. Grazie al principio di progettazione versatile, tutti e tre i componenti funzionali del nanosensore - il nanocarrier (polimero PEG), il modulo reattivo agli stimoli (substrato attivato dalla proteasi) e il reporter biofluidico (codice a barre del DNA) - possono essere scambiati con precisione in base alle esigenze specifiche dell'applicazione, consentendo la modularità adattando le specificità del target e del rilascio.
Protocollo
Tutti gli studi sugli animali sono approvati dall'Institutional Animal Care and Use Committee (IACUC) presso l'istituto degli autori. Per eseguire correttamente questi esperimenti, sono necessarie strutture standard per la cura degli animali, tra cui camere di stabulazione, cappucci sterili, anestesia ed eutanasia etica. Tutti gli esperimenti sono condotti in conformità con le linee guida istituzionali e nazionali e supervisionati dal personale veterinario dell'istituto. I topi femmina BALB/c, utilizzati per gli esperimenti, sono ottenuti da una fonte commerciale (vedi Tabella dei materiali) e avevano un'età compresa tra 6 e 8 settimane all'inizio dello studio. Le sequenze per DNA sintetizzato su misura, crRNA, sonde del substrato peptidico basato su FRET e peptidi sensore sono fornite nella Tabella supplementare 1.
1. Selezione del substrato peptidico attivato dalla proteasi
- Raccogliere e preparare campioni di tessuto da polmone sano o tessuto polmonare con noduli tumorali a seguito di un rapporto precedentemente pubblicato13.
- Omogeneizzare i tessuti in soluzione salina tamponata con fosfato pre-raffreddato (PBS, pH 7,4) (200 mg di tessuto/mL di PBS) con provette di dissociazione tissutale per la dissociazione automatizzata dei tessuti in sospensioni unicellulari.
- Centrifugare gli omogeneizzati di tessuto a 6.000 x g per 5 minuti a 4 °C. Conservare il surnatante in una nuova provetta.
- Centrifugare il surnatante a 14.000 x g per 25 minuti a 4 °C.
- Misurare la concentrazione proteica utilizzando il kit per il test delle proteine dell'acido bicinchoninico (BCA) (vedere la tabella dei materiali).
- Aggiungere PBS al campione per preparare una soluzione 0,33 mg/mL.
- Valutare l'attività proteolitica seguendo i passaggi seguenti.
- In una piastra da 384 pozzetti, aggiungere sonde per substrato peptidico a base di FRET da 5 μL e 6 μM a ciascun pozzetto. Per ogni sonda, eseguire la reazione in triplice copia.
NOTA: La sonda del substrato peptidico a base di FRET contiene una breve sequenza peptidica (6-8 amminoacidi, progettata per essere specifica per una proteasi bersaglio o un gruppo di proteasi) terminata con una coppia di fluorofori e quencher. Due sonde FRET sono descritte nella Tabella supplementare 1 come esempio. La libreria completa di sonde può essere trovata in Hao et al.Ore 13. - Centrifugare la piastra del pozzetto per 10 s a temperatura ambiente a 180 x g per assicurarsi che le sonde si trovino sul fondo della piastra.
- Aggiungere 25 μL di campione di tessuto 0,33 mg/mL o 40 μM di proteasi ricombinante13 in ciascun pozzetto.
NOTA: Potrebbe essere necessario ottimizzare la concentrazione finale del campione di tessuto o della proteasi ricombinante in base alla loro attività proteolitica intrinseca. Ad esempio, i tessuti altamente proteolitici come l'intestino possono richiedere fattori di diluizione più elevati per consentire il monitoraggio della scissione enzimatica iniziale. - Centrifugare brevemente la piastra del pozzetto a 180 x g (a temperatura ambiente) per miscelare le sonde e i lisati tissutali.
- Iniziare immediatamente a rilevare la sfaldatura della sonda misurando la fluorescenza con il lettore di piastre a 37 °C ogni 2 minuti per 1 ora (λex: 485 nm; λem: 535 nm) per monitorare la scissione.
NOTA: Utilizzare le lunghezze d'onda di eccitazione ed emissione appropriate per specifiche coppie di fluorofori e quencher. Nel caso di questo studio, i parametri (λex: 485 nm e λem: 535 nm) sono fissati per il fluoroforo FAM. - Per analizzare i dati di misurazione della fluorescenza, utilizzare il pacchetto Python (vedere la tabella dei materiali) per l'analisi della cinetica enzimatica disponibile all'indirizzo https://github.com/nharzallah/NNanotech-Kinetic. Questo script calcola la velocità di reazione iniziale (V0) utilizzando la pendenza dell'adattamento lineare dei primi 8-10 punti temporali iniziali.
- In una piastra da 384 pozzetti, aggiungere sonde per substrato peptidico a base di FRET da 5 μL e 6 μM a ciascun pozzetto. Per ogni sonda, eseguire la reazione in triplice copia.
2. Formulazione e caratterizzazione dei sensori
- Sintetizzare il coniugato di DNA e peptide attivato dalla proteasi (PAP).
- Incubare reporter di DNA fosforotioato 20-mer con gruppo 3'-DBCO (1,1 eq., vedere Tabella dei materiali) con PAP con terminazione azidica con estremità cisteina C-terminale in tampone fosfato 100 mM (pH 7,0) a temperatura ambiente per >4 ore. In caso di lasciare incubare per una notte a 4 °C.
- Purificare il prodotto su un sistema di cromatografia liquida ad alta prestazione (HPLC) dotato di una colonna ideale per le biomolecole (vedi Tabella dei Materiali). Impostare il gradiente HPLC in modo che parta dal tampone 5% A (0,05% TFA in H2O), mantenerlo isocratico per 20 minuti e raggiungere l'80% tampone B (0,05% TFA, 99,95% acetonitrile) a 65 minuti con una portata di 0,3 mL min-1 e raccogliere il prodotto coniugato con tempo di ritenzione a ~31 min.
- Convalidare i coniugati purificati mediante spettrometria di massa MALDI-TOF (Matrix-Assisted Laser Desorption/Ionization-Time of Flight) utilizzando l'acido α-ciano-4-idrossicinnamico come matrice14 (vedi Tabella dei materiali).
- Sintetizzare i nanosensori basati sull'attività codificati dal DNA.
- Sciogliere 2 mg di PEG multivalente (40 kDa, 8 bracci, vedere Tabella dei materiali) con gruppo maleimmide-reattivo in 1 mL di tampone fosfato 100 mM (pH 7,0) e filtro (cutoff: 0,2 μm).
- Aggiungere i coniugati DNA-peptide terminati con cisteina (2 eq., vedi Tabella dei materiali) al PEG e reagire a temperatura ambiente per >4 ore. In caso di lasciare incubare per una notte a 4 °C.
- Rimuovere i materiali non coniugati utilizzando la cromatografia ad esclusione dimensionale con una colonna a matrice composita destrano-agarosio disponibile in commercio su una cromatografia liquida proteica veloce (FPLC) (vedere Tabella dei materiali). Eseguire i campioni in PBS e monitorare l'assorbanza a 260 nm per il DNA e 280 nm per il peptide.
- Concentrare i nanosensori sintetizzati con tubi filtranti centrifughi (MWCO = 10 kDa) secondo la velocità consigliata dal produttore (vedi Tabella dei materiali).
- Quantificare la concentrazione di DNA utilizzando il kit del saggio ssDNA (vedere la tabella dei materiali) e un lettore di piastre a λex: 485 nm e λem: 535 nm. Conservare i nanosensori a 4 °C.
- Caratterizzare i nanosensori basati sull'attività codificati dal DNA.
- Misurare la dimensione delle particelle idrodinamiche di nanosensori codificati nel DNA mediante diffusione dinamica della luce (DLS).
NOTA: L'intervallo di dimensioni previsto dei nanosensori è di 15-50 nm, con una dimensione media di 20-30 nm. Se si desidera un intervallo di dimensioni limitato, è possibile utilizzare FPLC (nel passaggio 2.3) per isolare frazioni più strette con pesi molecolari diversi. - Concentrare i nanosensori a 0,5 mg/mL (per concentrazione di DNA) con tubi filtranti centrifughi (MWCO = 10 kDa) e caricare i campioni su una griglia di rame rivestita con film di carbonio montata su un crioholder. Osservare la morfologia utilizzando la microscopia elettronica a trasmissione criogenica (200 kV, ingrandimento di 10.000-60.000)14.
- Misurare la dimensione delle particelle idrodinamiche di nanosensori codificati nel DNA mediante diffusione dinamica della luce (DLS).
3. Iniezione del sensore e raccolta delle urine
- Raccogliere l'urina per la misurazione del basale.
- Posizionare il mouse in una camera di alloggiamento personalizzata (vedere la Figura 1 supplementare) con una piastra a 96 pozzetti come base.
- Trattieni il mouse e applica una leggera pressione sulla vescica per eliminare l'urina residua sulla piastra.
- Pipettare l'urina raccolta (~100-200 μL) dalla piastra a 96 pozzetti in una provetta da 1,5 mL dopo aver riposizionato il topo nell'alloggiamento normale.
- Stabilire un modello preclinico di tumore murino.
- Inoculare topi femmina BALB/c di età compresa tra 6 e 8 settimane mediante iniezione endovenosa con linea cellulare MC26-Fluc esprimente luciferasi (100k cellule/topo) (vedere Tabella dei materiali). Monitorare settimanalmente la progressione del tumore utilizzando un sistema di imaging a fluorescenza in vivo .
NOTA: Il carico tumorale visibile indicato dal segnale luminescente si verifica approssimativamente nella settimana 2 di iniezione di questa particolare linea cellulare. Controllare attentamente gli animali portatori di tumore durante la progressione del tumore su base regolare.
- Inoculare topi femmina BALB/c di età compresa tra 6 e 8 settimane mediante iniezione endovenosa con linea cellulare MC26-Fluc esprimente luciferasi (100k cellule/topo) (vedere Tabella dei materiali). Monitorare settimanalmente la progressione del tumore utilizzando un sistema di imaging a fluorescenza in vivo .
- Iniettare nanosensori in diversi momenti dopo l'impianto del tumore.
- Preparare una soluzione iniettabile (volume massimo di 200 μL) contenente nanosensori a una concentrazione di 1 nmol mediante codice a barre del DNA in PBS sterile.
- Iniettare 200 μL di soluzione del sensore in PBS in ciascun topo sperimentale per via endovenosa.
- Raccogliere campioni di urina da topi sani di controllo e portatori di tumore 1 ora dopo l'iniezione del sensore, come descritto nei passaggi 1.1-1.3.
NOTA: I campioni di urina fresca possono essere processati per l'analisi del codice a barre del DNA direttamente o congelati immediatamente sul ghiaccio.
4. Rilevamento CRISPR di codici a barre del DNA: basato sulla fluorescenza
- Utilizzare campioni di urina freschi o scongelare i campioni congelati sul ghiaccio. Centrifugare i campioni di urina a 800 x g per 5 minuti a temperatura ambiente.
- Combinare i reagenti nella Tabella supplementare 2, aggiungere per ultimo l'enzima Cas12a (vedere Tabella dei materiali) e mescolare delicatamente la reazione pipettando su e giù. Incubare la reazione a 37 °C per 30 min.
- Eseguire la reazione reporter, come mostrato nella Tabella supplementare 3, in triplice copia utilizzando il prodotto del passaggio 2. Aggiungere la reazione del passaggio 2 per ultima e portarla rapidamente al lettore di piastre.
- Rilevare l'attivazione di LbaCas12a misurando la fluorescenza con un lettore di piastre a 37 °C ogni 2 minuti per 3 ore (λex: 485 nm e λem: 535 nm) per monitorare la cinetica di clivaggio del reporter del DNA.
- Per analizzare i dati di misurazione della fluorescenza, utilizzare il pacchetto Python per l'analisi della cinetica enzimatica disponibile all'indirizzo https://github.com/nharzallah/NNanotech-Kinetic. Questo script calcola la velocità di reazione iniziale (V0) utilizzando la pendenza dell'adattamento lineare dei primi 8-10 punti temporali iniziali.
5. Rilevamento CRISPR di codici a barre del DNA: su carta
- Centrifugare i campioni di urina a 800 x g per 5 minuti a temperatura ambiente.
NOTA: Eseguire i campioni di urina per il rilevamento CRISPR basato su fluorescenza e su carta in parallelo. - Combinare i reagenti indicati nella Tabella supplementare 2. Incubare a 37 °C per 30 min.
NOTA: Questa fase di incubazione è identica a quella per la rilevazione CRISPR basata sulla fluorescenza. - Eseguire la reazione reporter utilizzando il reporter di DNA marcato con FAM-biotina per il saggio a flusso laterale su una striscia di carta (vedere Tabella dei materiali). Combinare i reagenti della Tabella supplementare 4 in una piastra a 96 pozzetti utilizzando il prodotto della fase 2. Coprire con un foglio di alluminio e incubare a 37 °C per 1 h.
NOTA: Selezionare il tempo di incubazione ottimale in base al monitoraggio cinetico in tempo reale nel test di rilevamento CRISPR basato sulla fluorescenza descritto sopra. - A un nuovo pozzetto di una piastra a 96 pozzetti, aggiungere 80 μL di PBS. Aggiungere 20 μL di campione della fase 3 a questo pozzetto.
- Posizionare una striscia di carta a flusso laterale su ciascun pozzetto e attendere che il liquido raggiunga la parte superiore della striscia (<5 min). Cercare l'aspetto delle bande di controllo e/o dei campioni sulla striscia di carta.
- Scatta una foto della striscia di flusso laterale e quantifica l'intensità della banda utilizzando ImageJ.
Risultati
Nomina di substrati peptidici attivati da proteasi
Per progettare sensori che riflettano i cambiamenti nell'attività proteolitica del tessuto, l'attività della proteasi nel tessuto viene innanzitutto caratterizzata utilizzando una libreria di sonde peptidiche13 (Figura 1). I campioni di tessuto freschi e congelati possono fornire informazioni sostanziali sull'attività proteolitica del microambiente tumorale combinando campioni di tessuto con sonde FRET progettate per rilevare la scissione del substrato. Una libreria di sonde FRET con un fluoroforo FAM e un quencher CPQ-2 viene incubata con campioni di tessuto. Le sonde con la maggiore differenza nel tasso di scissione tra tessuto fittizio e tessuto tumorale, misurate mediante spettrofotometria di fluorescenza, sono selezionate come linker peptidici per l'uso nei nanosensori in vivo (Figura 1A). Per comprendere meglio la fisiologia dell'ambiente tumorale e confrontare efficacemente i profili di espressione della proteasi con i profili di attività, le sonde FRET possono essere incubate con proteasi ricombinanti per associare la scissione del substrato all'attività specifica della proteasi, come dimostrato con due sonde di esempio nella Figura 1B.
Formulazione e caratterizzazione dei sensori
I nanosensori sono composti da un nucleo polimerico, peptidi e codici a barre di DNA a singolo filamento, come illustrato nella Figura 2A. I nuclei polimerici sono dendrimeri PEG da 40 kDa che fungono da vettori per un massimo di 8 coniugati peptide-DNA (Figura 2B). I peptidi, ~1,4 kDa, collegano il nucleo e il DNA con una sequenza di amminoacidi progettata per essere scissa attraverso l'attività proteolitica. Il codice a barre del DNA a singolo filamento è lungo 20 coppie di basi, ~6,8 kDa, e modificato chimicamente per una migliore stabilità in vivo. Dopo la coniugazione, il costrutto peptide-DNA viene purificato tramite HPLC, consentendo la separazione dei costituenti coniugati e liberi (Figura 2C). L'analisi di spettrometria di massa indica che il coniugato ha un peso molecolare di 8,283 kDa, che è previsto dati i pesi molecolari costituenti il peptide e il codice a barre del DNA rispettivamente di 1,4 kDa e 6,8 kDa. Il coniugato peptide-DNA è ulteriormente coniugato al dendrimero PEG. Il sensore risultante viene purificato utilizzando FPLC per rimuovere il peptide-DNA libero che presenta un tempo di ritenzione prolungato rispetto a quelli PEGilati (Figura 2D). Le dimensioni del sensore possono essere caratterizzate tramite diffusione dinamica della luce e microscopia elettronica a trasmissione criogenica, che indicano un aumento del diametro in seguito all'aggiunta di peptide-DNA al nucleo da ~8 nm a ~20 nm (Figura 2B). La varianza nella dimensione delle particelle può essere dovuta alla flessibilità delle catene costituenti e a un numero variabile di bracci peptide-DNA coniugati a ciascun dendrimero PEG.
Modello preclinico di malattia murina
Il lavoro in vivo per testare i sensori prevede l'instaurazione di tumori per 3 settimane nei polmoni dei topi BALB/c, l'iniezione dei nanosensori e la raccolta dell'urina 1 ora dopo l'iniezione del sensore (Figura 3A). Un campione di urina di base viene anche prelevato prima dell'inoculazione del tumore per confrontarlo con i campioni dopo l'induzione della malattia. Al fine di valutare l'efficacia del sensore nel rilevare la malignità, la linea cellulare di cancro colorettale MC26 (MC26-Fluc) ad espressione luciferasi viene iniettata per via endovenosa, con conseguenti noduli tumorali polmonari visibili nell'imaging di luminescenza in vivo . La colorazione con ematossilina ed eosina13 rivela l'istopatologia del tessuto polmonare di topi di controllo portatori di tumore e di finti controlli, rispettivamente, e l'evidenza della formazione di tumori a 11 e 21 giorni dopo l'iniezione, come indicato dalle frecce nere nella Figura 3B.
Attivazione di CRISPR da parte del DNA stabilizzato chimicamente
Per ottimizzare l'attivazione di Cas12a tramite codici a barre di ssDNA e appaiamento complementare di crRNA, sono state testate diverse sequenze e lunghezze di oligonucleotidi. L'attivazione di Cas12a viene valutata misurando l'aumento della fluorescenza nel tempo dovuto alla scissione di Cas12a del reporter del DNA degli astanti con un accoppiamento FRET. Un aumento del tasso di scissione è associato a concentrazioni più elevate di codice a barre ssDNA modificato chimicamente, come mostrato con una coppia rappresentativa crRNA/codice a barre (Figura 4B). È importante sottolineare che una relazione lineare tra concentrazione e attivazione, come mostrato nella Figura 4B, consente alla lettura di riflettere la presenza del codice a barre del DNA nelle urine e indirettamente l'attività proteolitica in vivo. Inoltre, è possibile utilizzare più sequenze di crRNA diverse, come descritto nella Tabella supplementare 1, per l'attivazione di Cas12a, che è fondamentale per consentire il multiplexing. Gli attivatori del DNA sono stati selezionati per la costruzione di sensori in vivo in base alla loro somiglianza nelle prestazioni del saggio. L'ssDNA chimicamente modificato, che è fondamentale per la stabilità in vivo del biomarcatore sintetico, ha dimostrato l'attivazione di Cas12a, rispetto al dsDNA e all'ssDNA non modificati (Figura 4C).
Rilevamento urinario multiplexato di codici a barre di DNA stabilizzati chimicamente
Sono state verificate più coppie di attivatori di DNA a singolo filamento (ssDNA) modificati con crRNA per la loro ortogonalità tra diverse sequenze. Ciò consente la lettura simultanea in più saggi a pozzetto, come illustrato nella Figura 5A. Inoltre, le molecole di DNA modificate nelle urine non trattate possono essere rilevate utilizzando una lettura colorimetrica su strisce di carta a flusso laterale, come mostrato nella Figura 5B. La presenza della "banda del campione" principale indica il rilascio di FAM rilevabile attraverso la scissione del reporter dopo che Cas12a è stato innescato dal DNA urinario. Quando Cas12a viene attivato dall'attivatore del DNA nell'urina di topo, scinde il reporter oligonucleotidico accoppiato con fluoresceina (FAM)-biotina, rilasciando la molecola FAM, che è quindi rilevabile sulla "banda del campione". I reporter non autorizzati vengono catturati nella "banda di controllo" a causa del legame della biotina con la streptavidina. I nanosensori sono selezionati per esperimenti in vivo e costruiti utilizzando peptidi attivabili dalla proteasi e codici a barre distinti del DNA. I peptidi vengono nominati attraverso la profilazione tissutale ex vivo (Figura 1A) e i codici a barre del DNA vengono selezionati in base a un'attivazione simile di Cas12a (Figura 4B).

Figura 1: Identificazione di substrati peptidici attivati da proteasi disregolate nel cancro del colon-retto per la costruzione di sensori. (A) I substrati peptidici accoppiati a FRET, ciascuno costituito da una sequenza peptidica affiancata da un fluoroforo FAM e da un quencher CPQ-2, vengono sottoposti a screening contro enzimi proteolitici ricombinanti o omogenati tissutali. (B) Cinetica rappresentativa del clivaggio della proteasi di substrati peptidici accoppiati a FRET (Sonda 1 e 2). La figura è adattata da Hao et al.13. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 2: Caratterizzazione di un nanosensore basato sull'attività basato su DNA con codice a barre con un nucleo in PEG polimerico. (A) I nanosensori con codice a barre del DNA comprendono nanocarrier polimerici (PEG a 8 bracci) funzionalizzati con peptidi attivati dalla proteasi codificati a barre con oligonucleotidi. (B) L'analisi dinamica dello scattering della luce mostra un aumento della dimensione delle particelle da 8,3 nm (solo nucleo PEG) e 13 nm (sensore funzionalizzato). (C) Purificazione HPLC del coniugato peptide-DNA. Il coniugato viene analizzato in spettrometria di massa e mostra il peso molecolare atteso. (D) La purificazione FPLC del sensore mostra la separazione del sensore funzionalizzato e del coniugato peptide-DNA illimitato. La figura è adattata da Hao et al.13. Fare clic qui per visualizzare una versione più grande di questa figura.
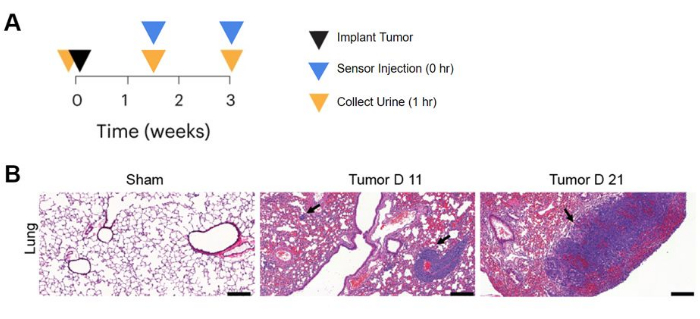
Figura 3: Induzione della malattia in vivo e utilizzo del sensore. (A) Cronologia del monitoraggio longitudinale del tumore con nanosensore in diversi punti temporali dello sviluppo del tumore. (B) Colorazione istologica del polmone di topi BALB/c portatori di tumori polmonari CRC a 11 e 21 giorni dopo l'inoculazione del tumore e topi di controllo Sham iniettati con soluzione salina. Barra della scala = 200 μm. Gli organi sono stati fissati, incorporati in paraffina e colorati con ematossilina ed eosina. Lo studio è stato condotto con n = 3 topi per punto temporale e sono state mostrate immagini di un animale rappresentativo. Le frecce indicano la presenza di noduli tumorali nel polmone. La figura è adattata da Hao et al.13. Fare clic qui per visualizzare una versione più grande di questa figura.
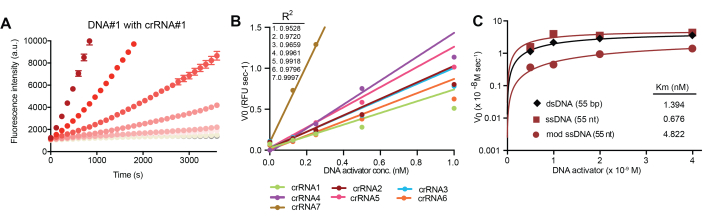
Figura 4: Attivazione di Cas12a da parte di attivatori del DNA stabilizzati chimicamente. (A) L'attivazione di Cas12a da parte di un codice a barre rappresentativo di ssDNA attraverso il legame con il crRNA complementare provoca la scissione trans-scissione del DNA degli astanti in modo dose-dipendente, misurato dall'intensità della fluorescenza tramite spettrofotometro. (B) La velocità di reazione iniziale (V0) è determinata dalla pendenza della curva all'inizio di una reazione in (A) e tracciata per determinare l'intervallo lineare delle prestazioni del saggio. (C) Attivazione di Cas12a da parte di attivatori del DNA a doppio filamento, a singolo filamento e chimicamente modificati. La figura è adattata da Hao et al.13. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 5: Lettura del codice a barre del DNA mediato da CRISPR-Cas12 multiplexato con due opzioni di rilevamento. (A) I tassi di trans-scissione di Cas12a all'attivazione di diverse coppie attivatore ssDNA-crRNA modificato sono determinati nel saggio di scissione fluorescente Cas12a. I test vengono eseguiti con campioni di urina raccolti da topi a cui è stato iniettato 1 nmol di attivatore di ssDNA modificato dopo 1 ora di somministrazione endovenosa. La figura è adattata da Hao et al.13. (B) La lettura cartacea di Cas12a attivata dal campione di urina con variazioni colorimetriche appare in diverse posizioni della striscia di carta. La banda superiore è di un reporter FAM DNA scisso e la banda inferiore è di un reporter FAM-biotina non rilasciato. Fare clic qui per visualizzare una versione più grande di questa figura.
Figura supplementare 1: Camera di raccolta dell'urina da posizionare su una piastra a 96 pozzetti. Il topo viene temporaneamente posizionato all'interno del cilindro per la minzione nella piastra a 96 pozzetti. Fare clic qui per scaricare il file.
Tabella supplementare 1: Sequenze oligotidiche e peptidiche per la formulazione del sensore e il saggio CRISPR. Fare clic qui per scaricare il file.
Tabella supplementare 2: Reagenti per la reazione a base di fluorescenza e a base di carta Cas12a e campione di urina. Fare clic qui per scaricare il file.
Tabella supplementare 3: Reagenti della reazione reporter per la rilevazione CRISPR basata sulla fluorescenza. Fare clic qui per scaricare il file.
Tabella supplementare 4: Reagenti della reazione reporter per la rilevazione CRISPR su carta. Fare clic qui per scaricare il file.
Discussione
Qui viene presentata una piattaforma altamente personalizzabile per il rilevamento multiplexato del cancro con un test delle urine portatile che valuta l'attività proteolitica associata alla malattia utilizzando un sensore iniettato minimamente invasivo. Quando viene attivata dalle proteasi tumorali, la scissione del substrato peptidico viene amplificata tramite il rilascio del codice a barre del DNA nelle urine. I reporter di DNA sintetico in un campione di urina possono essere letti da un'amplificazione enzimatica secondaria mediata da CRISPR-Cas utilizzando il rilevamento fluorometrico o un semplice test cartaceo. Il codice a barre del DNA è un metodo interessante per etichettare i biomarcatori sintetici in modo da consentire la multiplexità. Migliorare la stabilità dei codici a barre del DNA attraverso la modifica chimica è fondamentale per mantenere la stabilità dei nanosensori in vivo. In particolare, il rilevamento dei codici a barre del DNA tramite CRISPR-Cas12a può essere attivato utilizzando codici a barre del DNA completamente modificati con fosforotioato con una spina dorsale stabilizzata. Tuttavia, la modifica chimica riduce la velocità di attivazione di CRISPR-Cas12a rispetto alla sua controparte nativa del DNA, a causa della sua maggiore stabilità. Inoltre, l'inserimento di modifiche terminali al crRNA o l'analisi di diversi oligonucleotidi modificati nei codici a barre del DNA possono aumentare ulteriormente la sensibilità del rilevamento del codice a barre del DNA mediato da CRISPR15, migliorando così il limite di rilevamento (LOD) della diagnostica del cancro.
Oltre all'amplificazione molecolare, un'altra strategia chiave per raggiungere il LOD richiesto per la diagnosi precoce del cancro prevede di sfruttare la filtrazione dimensionale da parte dei reni per concentrare i reporter di DNA sintetico nelle urine. A tal fine, è fondamentale ottimizzare le dimensioni dei codici a barre del DNA. I DNA complementari al crRNA a 20 mer hanno mostrato una lettura ottimale del codice a barre del DNA mediato da CRISPR-Cas12a dai campioni di urina. Questa lunghezza ottimale può differire se applicata a diverse nucleasi CRISPR o modelli animali preclinici.
Per mantenere la riproducibilità di questo sistema di rilevamento, è importante condurre una caratterizzazione approfondita dei nanosensori iniettabili per ridurre al minimo le variazioni da lotto a lotto. La variazione del numero di coniugati peptide-DNA per nucleo altererà la quantità di codici a barre del DNA rilasciati nelle urine e quindi influenzerà la lettura finale. Allo stesso modo, un'attenta iniezione del sensore per via endovenosa aiuterà la coerenza nel dosaggio del sensore. Al fine di garantire un'adeguata raccolta di urina di almeno 50 μL di volume di urina da ciascun animale in un determinato momento, è fondamentale mantenere l'idratazione degli animali portatori di tumore. Ciò può essere supportato mantenendo i topi su gel d'acqua non bagnante o iniettando PBS per via sottocutanea un'ora prima della raccolta delle urine16.
La tempistica della raccolta delle urine e la durata della lettura del codice a barre del DNA mediato da CRISPR-Cs sono fattori critici per la coerenza del test. I codici a barre in circolazione subiscono un caratteristico decadimento della concentrazione a singolo esponenziale dopo iniezione endovenosa e filtrazione renale dipendente dalle dimensioni dal sangue, con un picco di 1 ora dopo la somministrazione nei modelli murini13. Questo punto temporale può variare in un modello animale diverso. Per i due formati di lettura dei saggi di attivazione CRISPR-Cas (saggio a flusso laterale fluorescente vs. saggio a flusso laterale basato su carta), è importante monitorare la cinetica basata sulla fluorescenza prima del flusso laterale, consentendo l'endpoint ottimale per leggere il prodotto di scissione CRISPR-Cas.
Questo approccio è vantaggioso grazie all'incorporazione di due fasi di amplificazione del segnale dall'attivazione enzimatica proteolitica e mediata da CRISPR-Cas e alla capacità di multiplexing di catturare la complessità della malattia. L'espansione della multiplexità dei sensori richiederà un attento dosaggio del materiale e l'espansione della produttività della lettura per mantenere la portabilità e la facilità d'uso. Questa strategia richiede un'accurata caratterizzazione dell'attività della proteasi in più modelli e della specificità del profilo proteolitico verso una particolare malattia o stadio della malattia. Inoltre, mentre l'iniezione del sensore è meno invasiva di molte delle alternative diagnostiche come la biopsia, l'iniezione endovenosa può fare affidamento su flebotomi addestrati per la somministrazione del sensore, limitando potenzialmente il caso d'uso al monitoraggio della malattia piuttosto che al rilevamento iniziale in un ambiente clinico. Oltre all'iniezione sistemica, i nanosensori con codice a barre del DNA possono essere ulteriormente ingegnerizzati con formulazioni che possono essere applicate tramite somministrazione orale, inalatoria e topica non invasiva, per consentire il rilevamento portatile della malattia e un facile automonitoraggio della progressione della malattia e della valutazione della terapia.
Divulgazioni
S.N.B., L.H. e R.T.Z. sono elencati come inventori in una domanda di brevetto relativa al contenuto di quest'opera. S.N.B. detiene azioni di Glympse Bio, Satellite Bio, Lisata Therapeutics, Port Therapeutics, Intergalactic Therapeutics, Matrisome Bio ed è direttore di Vertex; è consulente per Moderna e riceve finanziamenti per la ricerca sponsorizzati da Johnson & Johnson, Revitope e Owlstone.
Riconoscimenti
Questo studio è stato sostenuto in parte da una sovvenzione di sostegno dell'istituto Koch numero P30-CA14051 dal National Cancer Institute (Swanson Biotechnology Center), da una sovvenzione P30-ES002109 del National Institute of Environmental Health Sciences, dal Marble Center for Cancer Nanomedicine dell'Istituto Koch, dal programma di ricerca di frontiera dell'Istituto Koch tramite il Kathy and Curt Marble Cancer Research Fund, e il Fondo Virginia e D. K. Ludwig per la ricerca sul cancro. A.E.V.H. è supportato da una borsa di studio di formazione pre-dottorato finanziata dal NIH (T32GM130546). S.N.B. è un ricercatore dell'Howard Hughes Medical Institute. L.H. è supportato da un K99/R00 Pathway to Independence Award del National Cancer Institute e dal finanziamento iniziale della Boston University.
Materiali
| Name | Company | Catalog Number | Comments |
| 10x NEB Buffer 2.1 | New England Biolabs | B6002SVIAL | |
| 20-mer phosphorothioated DNA reporters with 3’-DBCO group | IDT | Custom DNA | |
| Agilent 1100 High Performance Liquid Chromatography system with Vydac 214TP510 C4 column | Agilent | HPLC | |
| ÄKTA fast protein liquid chromatography (FPLC) | GE Healthcare | FPLC | |
| Amicon ultracentrifuge tubes (MWCO = 10 kDa) | EMD millipore | Various volumes available | |
| Azide-terminated PAPs with C-terminus cysteine | CPC Scientific | Custom peptide | |
| crRNAs | IDT | See Supplementary Table 1 | |
| Cryogenic transmission electron microscopy | JEM-2100F | JEOL | cyroTEM |
| Cysteine terminated DNA-peptide conjugates | CPC Scientific | Custom peptide | |
| Dynamic light scattering (DLS) | DLS | ||
| EnGen LbaCas12a (Cpf1), 100 µM | New England Biolabs | M0653T | |
| Experimental animals | Taconic Biosciences | BALB/cAnNTac | 6–8 weeks of age |
| gentleMACS C tubes | Miltenyi Biotec | 130-093-237 | tissue homogenization |
| HybriDetect Universal Lateral Flow Assay Kit | Miltenyi Biotec | MGHD 1 | |
| Matrix-assisted laser desorption/ionization–time of flight (MALDI–TOF) mass spectrometry | Bruker | Microflex MALDI–TOF | |
| MC26-Fluc cell line | Kenneth K. Tanabe Laboratory, Massachusetts General Hospital | ||
| multivalent PEG (40 kDA, 8-arm) with maleimide-reactive group | JenKem | A10020-1 / 8ARM(TP)-MAL-40K,1 g | |
| Python, Version 3.9 | https://www.python.org/ | ||
| Quant-iT OliGreen ssDNA Assay Kit and Quant-iT OliGreen ssDNA Reagent | Invitrogen | O11492 | ssDNA assay kit |
| ssDNA FAM-T10-Quencher and FAM-T10-Biotin reporter substrates | IDT | Custom DNA | |
| Superdex 200 Increase 10/300 GL column | GE Healthcare | GE28-9909-44 | For FPLC |
| Tecan Infinite Pro M200 plate reader | Tecan | ||
| ThermoFisher Pierce BCA Protein Assay Kit | ThermoFisher Scientific | 23225 |
Riferimenti
- Soleimany, A. P., Bhatia, S. N. Activity-based diagnostics: an emerging paradigm for disease detection and monitoring. Trends Mol Med. 26 (5), 450-468 (2020).
- Muir, R. K., Guerra, M., Bogyo, M. M. Activity-based diagnostics: recent advances in the development of probes for use with diverse detection modalities. ACS Chem Biol. 17 (2), 281-291 (2022).
- Heitzer, E., Haque, I. S., Roberts, C. E. S., Speicher, M. R. Current and future perspectives of liquid biopsies in genomics-driven oncology. Nat Rev Genet. 20 (2), 71-88 (2019).
- Dudani, J. S., Warren, A. D., Bhatia, S. N. Harnessing protease activity to improve cancer care. Annu Rev Canc Biol. 2, 353-376 (2018).
- Hanahan, D. Hallmarks of cancer: new dimensions. Cancer Discov. 12 (1), 31-46 (2022).
- Quail, D. F., Joyce, J. A. Microenvironmental regulation of tumor progression and metastasis. Nat Med. 19 (11), 1423-1437 (2013).
- Kwong, G. A., et al. Synthetic biomarkers: a twenty-first century path to early cancer detection. Nat Rev Cancer. 21 (10), 655-668 (2021).
- Kirkpatrick, J. D., et al. Urinary detection of lung cancer in mice via noninvasive pulmonary protease profiling. Sci Transl Med. 12 (537), eaaw0262 (2020).
- Warren, A. D., et al. Disease detection by ultrasensitive quantification of microdosed synthetic urinary biomarkers. J Am Chem Soc. 136 (39), 13709-13714 (2014).
- Kwong, G. A., et al. Mass-encoded synthetic biomarkers for multiplexed urinary monitoring of disease. Nat Biotechnol. 31 (1), 63-70 (2013).
- Chen, J. S., et al. CRISPR-Cas12a target binding unleashes indiscriminate single-stranded DNase activity. Science. 360 (6387), 436-439 (2018).
- Choi, H. S., et al. Renal clearance of quantum dots. Nat Biotechnol. 25 (10), 1165-1170 (2007).
- Hao, L., et al. CRISPR-Cas-amplified urinary biomarkers for multiplexed and portable cancer diagnostics. Nat Nanotechnol. 18 (7), 798-807 (2023).
- Hao, L., et al. Microenvironment-triggered multimodal precision diagnostics. Nat Mater. 20 (10), 1440-1448 (2021).
- Ghanta, K. S., et al. 5'-Modifications improve potency and efficacy of DNA donors for precision genome editing. Elife. 10, e72216 (2021).
- Bekkevold, C. M., Robertson, K. L., Reinhard, M. K., Battles, A. H., Rowland, N. E. Dehydration parameters and standards for laboratory mice. J Am Assoc Lab Anim Sci. 52 (3), 233-239 (2013).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon