Method Article
Un saggio di fluorescenza del calcio "a doppia aggiunta" per lo screening ad alta produttività dei recettori accoppiati a proteine G ricombinanti
In questo articolo
Riepilogo
In questo lavoro, viene descritto un saggio di fluorescenza di calcio intracellulare ad alto rendimento per piastre a 384 pozzetti per lo screening di piccole librerie di molecole su recettori accoppiati a proteine G ricombinanti (GPCR). Il bersaglio, il recettore della chinina dalla zecca della febbre bovina, Rhipicephalus microplus, è espresso nelle cellule CHO-K1. Questo test identifica agonisti e antagonisti che utilizzano le stesse cellule in un test di "doppia addizione".
Abstract
I recettori accoppiati a proteine G (GPCR) rappresentano la più grande superfamiglia di recettori e sono i bersagli di numerosi farmaci umani. Lo screening ad alta produttività (HTS) di librerie casuali di piccole molecole rispetto ai GPCR viene utilizzato dall'industria farmaceutica per la scoperta di farmaci specifici per bersaglio. In questo studio, un HTS è stato impiegato per identificare nuovi ligandi a piccole molecole di neuropeptidi GPCR specifici per invertebrati come sonde per studi fisiologici di vettori di patogeni umani e veterinari mortali.
Il recettore della chinina specifico per gli invertebrati è stato scelto come bersaglio perché regola molti importanti processi fisiologici negli invertebrati, tra cui la diuresi, l'alimentazione e la digestione. Inoltre, la farmacologia di molti GPCR invertebrati è scarsamente caratterizzata o non caratterizzata affatto; pertanto, la farmacologia differenziale di questi gruppi di recettori rispetto ai GPCR correlati in altri metazoi, in particolare nell'uomo, aggiunge conoscenza alle relazioni struttura-attività dei GPCR come superfamiglia. È stato sviluppato un test HTS per cellule in piastre a 384 pozzetti per la scoperta di ligandi del recettore della chinina dalla zecca della febbre bovina, o zecca bovina meridionale, Rhipicephalus microplus. Il recettore della zecca chinina era stabilmente espresso nelle cellule CHO-K1.
Il recettore della chinina, quando attivato da neuropeptidi endogeni della chinina o da altri agonisti di piccole molecole, innesca il rilascio di Ca2+ dalle riserve di calcio nel citoplasma. Questo saggio di fluorescenza del calcio combinato con un approccio di "doppia addizione" può rilevare molecole "hit" funzionali agoniste e antagoniste nella stessa piastra di analisi. Ogni test è stato condotto utilizzando piastre di farmaci contenenti una serie di 320 piccole molecole casuali. È stato ottenuto un fattore Z' affidabile di 0,7 e sono state identificate tre molecole colpite agonista e due antagoniste quando l'HTS era ad una concentrazione finale di 2 μM. Il saggio di fluorescenza del calcio qui riportato può essere adattato per lo screening di altri GPCR che attivano la cascata di segnalazione Ca2+ .
Introduzione
I recettori accoppiati a proteine G (GPCR), che sono presenti dal lievito all'uomo, rappresentano la più grande superfamiglia di recettori in molti organismi1. Svolgono un ruolo fondamentale nella regolazione di quasi tutti i processi biologici negli animali. Ci sono 50-200 GPCR nel genoma degli artropodi, il che significa che rappresentano la più grande superfamiglia di recettori di membrana2. Sono classificati in sei classi principali, A-F, in base alla loro somiglianza di sequenza e alle funzioni3. I GPCR trasducono vari segnali extracellulari, come quelli di ormoni, neuropeptidi, ammine biogene, glutammato, protone, lipoglicoproteine e fotoni4. I GPCR si accoppiano alle proteine G eterotrimere (Gα, Gβ e Gγ) per trasmettere segnali a valle. I GPCR accoppiati alle proteine Gαs o Gα i/o aumentano o diminuiscono, rispettivamente, i livelli intracellulari di adenosina monofosfato (cAMP) ciclico 3', 5' attivandoo inibendo l'adenilil ciclasi. I GPCR accoppiati a Gαq/11 inducono il rilascio di calcio dalle riserve di calcio del reticolo endoplasmatico attivando la via della fosfolipasi C (PLC)-inositolo-1,4,5-trifosfato (IP3). I GPCR accoppiati a Gα12/13 attivano i fattori di scambio nucleotidico della RhoGTPasi 5,6. I GPCR sono il bersaglio di oltre il 50% dei farmaci umani e di un acaricida, amitraz4. Poiché i GPCR trasducono segnali così diversi, sono obiettivi promettenti per lo sviluppo di nuovi pesticidi che interrompono le funzioni fisiologiche specifiche degli invertebrati.
L'obiettivo di HTS è identificare molecole colpite in grado di modulare le funzioni dei recettori. HTS comporta lo sviluppo di saggi, la miniaturizzazione e l'automazione7. I GPCR neuropeptidici artropodi sono coinvolti nella maggior parte delle funzioni fisiologiche, come lo sviluppo, la muta e l'ecdisi, l'escrezione, la mobilitazione di energia e la riproduzione4. La maggior parte dei GPCR neuropeptidici di artropodi e metazoi segnalano attraverso la cascata di segnalazione del calcio 2,6,8,9,10, come nel peptide mioinibitorio e nei recettori SIFamide della zecca dalle zampe nere Ixodes scapularis; i loro ligandi sono antagonisti nei saggi di motilità dell'intestino posteriore, con SIF che provoca contrazione e MIP che lo inibisce11,12. Un recettore NPY-simile della zanzara della febbre gialla, Aedes aegypti, regola l'ospite femminile in cerca di13. Rispetto ad altri saggi alternativi di mobilizzazione del calcio come il saggio di bioluminescenza del calcio aequorina14, il test di fluorescenza del calcio è facile da eseguire, non richiede la trasfezione di altre proteine ricombinanti di rilevamento del calcio ed è conveniente. Il saggio di fluorescenza del calcio produce un segnale prolungato rispetto al segnale cinetico veloce ottenuto nel saggio di bioluminescenza del calcio dell'aquorina14,15.
Nell'esempio qui, il recettore della chinina dalla zecca della febbre bovina, Rhipicephalus microplus, è stato espresso in modo ricombinante nella linea cellulare CHO-K1 e utilizzato per il saggio di fluorescenza del calcio. C'è solo un gene del recettore della chinina trovato in R. microplus; il recettore segnala attraverso una via di segnalazione dipendente dalla proteina Gq e innesca l'efflusso di Ca2+ dalle riserve di calcio nello spazio intracellulare16. Questo processo può essere rilevato e quantificato da un fluoroforo, che suscita un segnale di fluorescenza quando si legano gli ioni calcio (Figura 1).
Il recettore della chinina è un GPCR specifico per invertebrati, che appartiene ai recettori simili alla rodopsina di classe A. La kinina è un antico neuropeptide di segnalazione presente in molluschi, crostacei, insetti e Acari 4,17,18. I coleotteri (coleotteri) mancano del sistema di segnalazione della chinina; nella zanzara Aedes aegypti, c'è solo un recettore della chinina che lega tre aedeskinine, mentre Drosophila melanogaster ha un recettore della chinina con drosochinina come unico ligando 19,20,21. Non ci sono chinine omologhe o recettori della chinina nei vertebrati. Sebbene l'esatta funzione della chinina sia sconosciuta nelle zecche, le femmine silenziate dal recettore della kinina RNAi di R. microplus mostrano una capacità riproduttiva significativamente ridotta22. Le chinine sono peptidi pleotropici negli insetti. In Drosophila melanogaster, sono coinvolti sia nel sistema nervoso centrale che perifericoregolatore 23, pre-ecdisis24, alimentazione25, metabolismo 26 e modelli di attività del sonno26,27, così come locomozione larvale28. Le chinine regolano la contrazione dell'intestino posteriore, la diuresi e l'alimentazione nella zanzara A. aegypti 29,30,31. I peptidi di chinina hanno un pentapeptide C-terminale conservato Phe-X1-X2-Trp-Gly-NH2, che è la sequenza minima richiesta per l'attività biologica32. La specificità degli artropodi, le piccole dimensioni del ligando endogeno, che li rende suscettibili di interferenze di piccole molecole, e le funzioni pleiotropiche negli insetti rendono il recettore della chinina un bersaglio promettente per il controllo dei parassiti4.
Il saggio di "doppia addizione" (figura 2) consente l'identificazione di agonisti o antagonisti nello stesso saggio HTS15. È adattato da un test di "doppia addizione" comunemente usato nell'industria farmaceutica per la scoperta di farmaci33. In breve, la prima aggiunta di farmaci nella piastra cellulare consente l'identificazione di potenziali agonisti nella libreria chimica quando viene rilevato un segnale di fluorescenza superiore rispetto all'applicazione del controllo del solvente. Dopo 5 minuti di incubazione con queste piccole molecole, un noto agonista (peptide di chinina) viene applicato a tutti i pozzetti. Quei pozzetti che hanno ricevuto casualmente un antagonista dalla piastra del farmaco mostrano un segnale di fluorescenza inferiore all'aggiunta dell'agonista rispetto ai pozzetti di controllo che hanno ricevuto il solvente nella prima aggiunta. Questo test consente quindi l'identificazione di potenziali agonisti e antagonisti con le stesse cellule. In un progetto HTS standard, queste molecole colpite sarebbero ulteriormente convalidate attraverso saggi dose-risposta e da ulteriori saggi di attività biologica, che non sono mostrati qui.

Figura 1: Illustrazione del meccanismo di analisi della fluorescenza del calcio. La proteina Gq innesca la via di segnalazione intracellulare del calcio. Il recettore della chinina (recettore accoppiato alla proteina G) è stato espresso in modo ricombinante nelle cellule CHO-K1. Quando il ligando agonista si lega al recettore, la proteina Gq associata al recettore della chinina attiva PLC, che catalizza la conversione di una molecola PIP2 in IP3 e DAG. L'IP 3 si lega quindi all'IP3R sulla superficie del reticolo endoplasmatico, portando al rilascio di Ca 2+ nel citoplasma, dove gli ioni Ca 2+ si legano ai fluorofori e suscitano un segnale di fluorescenza. Il segnale di fluorescenza può essere ottenuto per eccitazione a 490 nm e rilevato a 514 nm. Abbreviazioni: GPCR = G protein-coupled receptor; PLC = fosfolipasi C; PIP2 = fosfatidilinositolo 4,5-bisfosfato; IP3 = inositolo trisfosfato; DAG = diacilglicerolo; IP3 R = recettore IP3. Creato con BioRender.com. Fare clic qui per visualizzare una versione ingrandita di questa figura.

Figura 2: Il flusso di lavoro per lo screening ad alta produttività di piccole molecole su un recettore accoppiato a proteine G espresso in cellule CHO-K1. (A) Le cellule CHO-K1 ricombinanti che esprimono stabilmente il recettore della chinina sono state aggiunte alla piastra a 384 pozzetti (10.000 cellule/pozzetto) utilizzando un sistema di manipolazione dei liquidi (25 μL/pozzetto) e incubate in un incubatore di CO2 umidificato per 12-16 ore. (B ) Il tampone di analisi contenente il colorante fluorescente (25 μL/pozzetto) è stato aggiunto nella piastra cellulare utilizzando un sistema di manipolazione del liquido. La piastra è stata incubata per 30 minuti a 37 °C per 30 minuti ed equilibrata a RT per altri 30 minuti. (C) Il segnale di fluorescenza di fondo delle cellule in ciascun pozzetto è stato misurato con un lettore di piastre. (D) Soluzioni farmacologiche da una piastra di libreria a 384 pozzetti e solvente bianco (tutti a 0,5 μL / pozzetto) sono stati aggiunti nella piastra di analisi cellulare utilizzando un sistema di manipolazione dei liquidi. (E) Le risposte cellulari alla fluorescenza del calcio sono state misurate con il lettore di piastre immediatamente dopo l'aggiunta delle soluzioni farmacologiche; I composti che hanno suscitato segnali di fluorescenza superiori alla media sono stati individuati come hit agonisti. I colpi antagonisti che bloccano il GPCR (icona sotto) sono stati rivelati dopo l'aggiunta dell'agonista del peptide durante la fase G. (F) Nella stessa piastra di analisi, dopo 5 minuti di incubazione delle cellule con composti di screening, un peptide agonista endogeno Rhimi-K-1 (QFSPWGamide) del recettore della chilina delle zecche è stato aggiunto a ciascun pozzetto (1 μM). (G) Le risposte di fluorescenza cellulare dopo l'aggiunta di peptidi agonista sono state misurate immediatamente dal lettore di piastre. I composti che inibiscono il segnale di fluorescenza sono stati selezionati come hit antagonisti. Abbreviazioni: GPCR = G protein-coupled receptor; RT = temperatura ambiente; RFU = unità di fluorescenza relativa. Creato con BioRender.com. Fare clic qui per visualizzare una versione ingrandita di questa figura.
Protocollo
1. Manutenzione delle celle
NOTA: UNA LINEA CELLULARE CHO-K1 che esprime stabilmente il recettore della chinina da R. microplus, chiamata BMLK3, è stata sviluppata da Holmes et al.16. I dettagli dello sviluppo della linea cellulare sono presentati altrove14. Tutte le seguenti fasi vengono eseguite in condizioni sterili in un armadio di biosicurezza di classe II.
- Far crescere la linea cellulare ricombinante in mezzi selettivi (terreno F-12K contenente il 10% di siero bovino fetale [FBS] e 800 μg/mL di solfato G418) per garantire l'espressione del recettore bersaglio. Conservare le cellule che esprimono stabilmente il recettore in 1 mL di terreno di congelamento (90% FBS e 10% dimetil solfossido [DMSO]) in un crioviale da 2 mL in un congelatore a -80 °C.
NOTA: Per la conservazione a lungo termine delle cellule congelate, conservarle in azoto liquido. - Preriscaldare tutti i terreni prima della coltura cellulare. In un armadio di biosicurezza, trasferire 13 mL di mezzo selettivo preriscaldato (mezzo F-12K contenente il 10% di FBS e 800 μg/mL di solfato G418) in un matraccio T-75 e conservarlo nell'armadio di biosicurezza. Scongelare un flaconcino di BMLK3 cellule congelate (~1,5 × 106 cellule) a bagnomaria a 37 °C per 2-3 minuti. Trasferire le cellule scongelate nel pallone T-75. Mantenere le celle in un incubatore umidificato a 37 °C e al 5% di CO2 se non diversamente specificato.
- Quando le cellule raggiungono il 90% di confluenza (1-2 giorni), preriscaldare tutti i mezzi a 37 °C ad eccezione della soluzione salina tamponata fosfato di Dulbecco (DPBS), che viene mantenuta a temperatura ambiente. In un armadio di biosicurezza, rimuovere il mezzo esaurito dal pallone T-75, lavare le cellule per 5 s con 10 ml di DPBS ruotando delicatamente il pallone, quindi rimuovere il DPBS con una pipetta sierologica.
NOTA: Un'immagine delle cellule CHO-K1 al 90% di confluenza è mostrata in Lu et al.14. - Staccare le cellule dal matraccio T-75 aggiungendo 2 ml di tripsina-EDTA allo 0,25% e incubare per 3-5 minuti a 37 °C nell'incubatore. Quindi, aggiungere 8 ml di mezzo selettivo e mescolare bene pipettando e rilasciando delicatamente 2x-3x con la stessa pipetta sierologica.
- Trasferire 2 mL della sospensione cellulare (~1 × 106 cellule) dal punto 1.4 in un nuovo matraccio T-75 contenente 10 mL di mezzo caldo selettivo. Fai crescere le cellule per 1-2 giorni nell'incubatore fino a raggiungere il 90% di confluenza.
- Utilizzare le celle per il test seguendo i passaggi successivi o ripetere i passaggi 1.4-1.5 una o due volte prima di utilizzare le celle nel test.
NOTA: Non superare i tre o quattro passaggi, poiché il segnale del test con alcune linee cellulari potrebbe indebolirsi con ulteriori passaggi.
2. Saggio di fluorescenza del calcio
- Rivestire la piastra cellulare.
- Utilizzare un sistema di gestione dei liquidi collocato all'interno di un armadio di biosicurezza per tutte le fasi di pipettaggio nelle piastre a 384 pozzetti. Creare programmi personalizzati per il pipettaggio nelle piastre da 384 pozzetti31 (Tabella supplementare S1). Rivestire in anticipo le piastre sterili da 384 pozzetti. In un armadio di biosicurezza, caricare 10 μL/pozzetto di una soluzione acquosa di poli-D-lisina (PDL) a 0,05 mg/ml in ciascuna piastra e incubare per 5 minuti a temperatura ambiente.
- Svuotare il piatto capovolgendo rapidamente il piatto e asciugandolo delicatamente su carta assorbente sterile. Quindi, sciacquare ogni pozzetto con 10 μL di acqua, svuotare la piastra e lasciare asciugare la piastra nell'armadio di biosicurezza per una notte senza coperchio. Chiudere la piastra con il coperchio e conservare a 4 °C in frigorifero.
NOTA: Le piastre rivestite possono essere conservate a 4 °C per un massimo di 6 mesi.
- Giorno 1
- Estrarre la piastra del farmaco (100 μM in 90% DPBS + 10% DMSO, la concentrazione finale nel pozzetto per HTS sarà di 2 μM) conservata nel congelatore a -20 °C e metterla a temperatura ambiente.
NOTA: Layout della piastra del farmaco: Ogni piastra da 384 pozzetti (24 colonne x 16 righe) contiene 320 pozzetti con diversi composti di libreria e 64 pozzetti con solvente bianco (DPBS contenente il 10% di DMSO), che sono disposti in quattro colonne, con due colonne sul bordo di ciascun lato. Vedere la tabella supplementare S2 per il layout delle lastre. - Quando le cellule raggiungono ~ 70% -90% di confluenza nel matraccio T-75, staccare le cellule dal pallone T-75 come descritto di seguito. Preriscaldare tutti i fluidi a 37 °C ad eccezione del DPBS (temperatura ambiente).
- Rimuovere il mezzo esaurito, lavare le celle con 10 ml di DPBS, quindi rimuovere il DPBS. Staccare le cellule dal matraccio T-75 usando 2 mL di tripsina-EDTA allo 0,25% per 3-5 minuti a 37 °C nell'incubatore, aggiungere 8 mL di mezzo selettivo e trasferire la sospensione cellulare in un tubo conico da 15 mL per centrifugare a 1.000 × g per 3 minuti.
- Eliminare il surnatante e risospendere il pellet cellulare in 10 mL di terreno F-12K contenente l'1% di FBS e 400 μg/mL di solfato G418. Conservare la sospensione nell'armadio di biosicurezza mentre si determina il numero di cellule.
- Determinare la densità cellulare della sospensione per un'ulteriore diluizione: mescolare 20 μL di sospensione cellulare in 20 μL di tripano blu allo 0,4%, quindi caricare 20 μL della miscela in una camera di conteggio delle cellule che deve essere letta da un contatore di celle per la densità cellulare.
- Diluire la sospensione cellulare nello stesso mezzo (terreno F-12K contenente 1% FBS e 400 μg/mL di solfato di G418) fino ad un volume finale di almeno 15 mL ad una densità di 4 × 105 cellule/ml.
- Seminare le cellule nella piastra a 384 pozzetti rivestita di PDL. Trasferire 15 mL della sospensione cellulare di cui sopra (4 × 105 cellule/ml) in un serbatoio di reagente auto-friendly da 150 ml. Erogare 25 μL della sospensione cellulare (~10.000 cellule/pozzetto) in ciascun pozzetto dei 384 pozzetti della piastra utilizzando un sistema di manipolazione del liquido in due fasi. Caricare punte a bassa ritenzione da 384/12,5 μL sulla testa del movimentatore di liquidi, aspirare 12,5 μL dal serbatoio (velocità 5,2 μL/s) ed erogare in ciascun pozzetto (velocità: 3,1 μL/s).
- Ripetere il pipettaggio come nel precedente punto 2.2.5 per raggiungere 25 μL per pozzetto. Quindi, incubare la piastra per una notte (14-16 h) a 37 °C e 5% di CO2 nell'incubatore umidificato.
NOTA: poiché il volume massimo di pipettaggio è di 12,5 μL per questa testa specifica, il pipettaggio per 25 μL viene eseguito in due fasi.
- Estrarre la piastra del farmaco (100 μM in 90% DPBS + 10% DMSO, la concentrazione finale nel pozzetto per HTS sarà di 2 μM) conservata nel congelatore a -20 °C e metterla a temperatura ambiente.
- Giorno 2
- La mattina dopo, controlla la piastra cellulare coperta al microscopio; Se non confluente, attendere che le cellule raggiungano il 90% di confluenza.
- Preparare una soluzione madre di colorante fluorescente: risospendere il colorante fluorescente liofilizzato in 100 μL di DMSO ed evitare la luce diretta sulla soluzione madre. Avvolgere il tubo con un foglio di alluminio per evitare lo sbiancamento fotografico.
NOTA: Il materiale può essere suddiviso in aliquote da 15 μL per ciascun saggio su piastra per evitare il congelamento e lo scongelamento ripetuti; le aliquote possono essere conservate a -80 °C per un massimo di 1 mese. - Nell'armadio di biosicurezza con le luci spente, preparare il colorante di carico (1x) in un tubo conico da 15 mL avvolto con un foglio di alluminio, combinando 15 μL della soluzione madre di colorante fluorescente (dal punto 2.3.2) e il resto dei componenti del kit preriscaldato (37 °C): 13,5 mL di 1x HHBS (tampone di Hank con 20 mM HEPES) e 1,5 mL di reagente B (inibitore dell'efflusso del colorante).
NOTA: la luce della stanza è accesa durante questo passaggio. - Chiudere il tubo e mescolare bene capovolgendo delicatamente il tubo più volte (in genere 3-5 volte).
NOTA: Conservare a temperatura ambiente e utilizzare la miscela di colorante di carico entro 30 minuti. - Quando le cellule raggiungono il 90% di confluenza, rimuovere il mezzo esaurito dalla piastra di dosaggio a 384 pozzetti capovolgendo rapidamente la piastra e asciugando delicatamente su asciugamani di carta sterili; Ripeti questo movimento 2x-3x per rimuovere tutto il liquido dalla piastra. Eliminare gli asciugamani bagnati.
- Spegnere la maggior parte delle luci dirette artificiali nella stanza (lasciando una lampada da scrivania morbida e fioca accesa o simile per consentire condizioni di lavoro visibili) e nell'armadio di biosicurezza per tutti i passaggi fino alla fine del test (circa per 1,5 ore).
- Introdurre 15 mL del colorante di carico (1x) dal punto 2.3.3 in un serbatoio auto-friendly da 150 mL.
- Trasferire 25 μL del colorante di carico (1x) dal serbatoio in ciascun pozzetto utilizzando un sistema di manipolazione del liquido con punte a bassa ritenzione da 384/12,5 μL erogando 12,5 μL in ciascun pozzetto della piastra (velocità di aspirazione e di erogazione: 3,8 μL/s).
- Ripetere la fase di pipettaggio come indicato al punto 2.3.7.1 per raggiungere un volume finale di 25 μL in ciascun pozzetto della piastra. Coprire e avvolgere la piastra con un foglio di alluminio per proteggerla dalla luce ambientale.
- Incubare la piastra cellulare coperta a 37 °C nell'incubatore umidificato CO2 per 30 minuti, rimuoverla dall'incubatore ed equilibrarla a temperatura ambiente all'interno del lettore di piastre o sul banco, mantenendo la piastra coperta e avvolta con un foglio per altri 30 minuti. Le celle sono quindi pronte per lo screening ad alta produttività (HTS).
- Preparazione chimica: Centrifugare la piastra del farmaco dal punto 2.2.1 a 1.200 × g in una centrifuga a piastre per 1 minuto a temperatura ambiente.
- Preparare una soluzione peptidica agonista 10x di Rhimi-K-1 (QFSPWGamide) (10 μM) risospendendo 100 nmoli di peptide liofilizzato in 10 mL di 1x HHBS contenente lo 0,1% di DMSO e trasferire la soluzione in un serbatoio auto-friendly da 150 ml.
- Misurare il segnale di fondo: per l'intero test HTS nel lettore di piastre, in Protocolli, scegliere la modalità di fluorescenza del punto finale.
NOTA: Il segnale fluorescente viene letto dalla parte inferiore della piastra a lunghezze d'onda di eccitazione/emissione di 495 nm/525 nm.- Inserire la piastra cella nel lettore di piastre. Nel cruscotto, selezionare regolare il guadagno, selezionare un pozzetto casuale sulla piastra, assegnarlo come 5% -10% del valore massimo misurabile di fluorescenza e selezionare regolare l'altezza (lettura).
- Fare clic su Avvia misurazione per leggere l'intera piastra per il segnale di fluorescenza di fondo in unità di fluorescenza relativa (RFU).
- Saggio "a doppia addizione"
- Utilizzando il sistema di manipolazione del liquido con punte da 384/12,5 μL, miscelare la soluzione del farmaco in ciascun pozzetto della piastra del farmaco, pipettare 3 volte su e giù 10 μL della soluzione del farmaco (in DPBS contenente il 10% di DMSO) (velocità di pipettaggio: 5,2 μL/s) e "aspirare" 1,5 μL da ciascun pozzetto della piastra del farmaco (velocità di aspirazione: 1,0 μL/s).
- "Erogare" 0,5 μL dei composti nella piastra di analisi cellulare (velocità di pipettaggio: 1 μL/s) per raggiungere una concentrazione finale di 2 μM nello 0,2% di DMSO.
- Posizionare immediatamente la piastra di analisi nel lettore di piastre dopo aver aggiunto i composti di screening. Leggere la stessa piastra in entrambe le direzioni di lettura avanti e indietro. Per ottenere queste letture, definire un programma per leggere da "ben 1-384" e immediatamente da "384-1".
NOTA: la lettura in entrambe le direzioni richiede 2 minuti in totale. Questo design di lettura serve a compensare la diminuzione della potenza del segnale che si verifica durante la lettura della piastra in ogni direzione. Vedere il passaggio 3.1 per le analisi di lettura. - Smaltire il restante 1 μL di soluzione di farmaco nelle punte immergendo le punte in un serbatoio di rifiuti da 150 ml contenente ~50 ml di DPBS (velocità di erogazione: 1,0 μL/s).
- Incubare i composti di screening con le cellule per un totale di 5 minuti (inclusi i 2 minuti di lettura della piastra) a temperatura ambiente nell'armadio di biosicurezza con le luci spente. Aggiungere 3 μL del peptide agonista, Rhimi-K-1 (QFSPWGamide), dal serbatoio (velocità di aspirazione: 3,1 μL/s) nella piastra di analisi utilizzando il sistema di manipolazione del liquido (velocità di erogazione: 3,1 μL/s) con punte da 384/12,5 μL.
- Posizionare la piastra nel lettore di piastre immediatamente dopo l'aggiunta del peptide agonista. Leggere la targa in entrambe le direzioni avanti e indietro utilizzando lo stesso programma del punto 2.3.12.3
3. Analisi dei dati
- Utilizzando il software di analisi associato al lettore di piastre installato su un computer, calcolare le risposte di fluorescenza cellulare (in RFU) per entrambe le letture dopo la prima aggiunta di composti (Prima lettura; RFUfa [Tabella supplementare S2]) e dopo i passaggi di addizione dell'agonista (Seconda lettura = RFUformica [Tabella supplementare S2]). Ogni lettura è ottenuta facendo la media dei due valori ottenuti (non mostrati) dalle letture della piastra diretta e inversa rispettivamente dal punto 2.3.12.3 e dal punto 2.3.12.6.
NOTA: RFU ago, RFUant si riferiscono rispettivamente alle unità di fluorescenza relative dei potenziali colpi agonisti (leggere nel passaggio 2.3.12.3) e dei potenziali colpi antagonisti (letti nel punto 2.3.12.6). - Esporta tutti e tre i set di dati, RFU bg, RFU ago e RFUant, dal software di analisi in tre fogli di calcolo separati. Ogni foglio di calcolo avrà solo due colonne: posizione del pozzo e RFU grezzo (file non mostrati qui).
NOTA: RFU bg si riferisce alla RFU dello sfondo letto nel passaggio 2.3.11.2. - Formattare i dati dai tre fogli di calcolo e organizzare i dati di cui sopra in un unico file csv (vedere l'esempio nella tabella supplementare S2). Sottrarre il segnale di fondo letto rispettivamente dalla RFU ago e dallaformica RFU (colonne G e H nella tabella supplementare S2).
- Importare il file csv in una "piattaforma dati HTS online" disponibile in commercio (vedere la tabella dei materiali) per l'analisi dei dati a valle (tabella 1) e l'archiviazione.
- Calcolare manualmente il fattore Z' per il controllo di qualità di ciascun saggio su piastra utilizzando l'equazione (1):
Equazione (1): (1)
(1)
NOTA: μ c- e μc+ rappresentano le RFU medie delle letture degli stessi pozzetti, che fungono da controlli negativi nella prima aggiunta dopo l'aggiunta del solvente (solvente bianco, n = 64; numeri in blu nella tabella supplementare S2) e servono come controlli positivi dopo l'aggiunta dell'agonista (solvente bianco + agonista, n = 64; numeri in magenta), rispettivamente. Inoltre, σ c- e σc+ rappresentano le loro corrispondenti deviazioni standard (SD). - Selezionare manualmente le molecole hit dalle mappe di calore sulla "piattaforma dati HTS online" e calcolare l'attivazione percentuale normalizzata (NPA) e l'attività inibitoria (Io) per gli hit agonisti e antagonisti utilizzando l'equazione (2) e l'equazione (3):
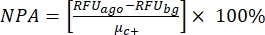 (2)
(2) (3)
(3)
Risultati
Una piastra di farmaco interna (SAC2-34-6170) composta da 320 piccole molecole casuali è stata utilizzata per dimostrare questo test HTS come esempio. L'HTS aveva un'eccellente qualità del saggio con un fattore Z' di 0,7 (Tabella 1). Questo fattore Z' riflette la qualità del saggio indipendente dai composti testati34. Un fattore Z′ pari o superiore a 0,5 indica un buon intervallo dinamico del segnale di analisi tra le RFU dei controlli positivi e dei controlli negativi. I saggi con un fattore Z' inferiore a 0,5 hanno una gamma dinamica del segnale non ottimale, di solito non sono informativi per la selezione dei colpi e sono, pertanto, scartati. La risposta (misurata in unità di fluorescenza relativa, RFU) dai controlli positivi (RFU media di 64 risposte di controllo positive, μc+ = 130.139) è stata in media 19 volte superiore a quella dei controlli negativi (RFU media di 64 risposte di controlli negativi, μc- = 6.675). La fluorescenza di cutoff per la selezione di "colpi agonisti" qui è stata impostata su un rapporto massimo di attivazione percentuale normalizzata (NPA) del >50%. La fluorescenza di cutoff per la selezione di "colpi antagonisti" è stata impostata su un'attività inibitoria osservata (Io) del >50%. Sono state selezionate tre molecole hit agoniste e due hit antagonisti: SACC-0090237 (NPA = 153%), SACC-0036982 (NPA = 118%) e SACC-0006532 (NPA = 152%) come agonisti, e SACC-0103392 (I o = 53%) e SACC-0041280 (I o = 56%) come antagonisti (i dati grezzi sono mostrati nella tabella supplementare S2). Esempi di calcoli di NPA e Io per agonisti (righe verdi) e antagonisti (righe arancioni), rispettivamente, sono mostrati nella tabella supplementare S2.
| Aggiunta composta | μs1 | σs | μc- | σc- | Valore Z' |
| (n = 320) | (n = 320) | (n = 64) | (n = 64) | ||
| 9,212 | 18,130 | 6,675 | 1,895 | 0.7 | |
| Aggiunta di peptidi agonisti | μs | σs | μC+ | σC+ | 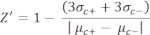 |
| 1,22,322 | 15,987 | 1,30,139 | 11,062 |
Tabella 1: Riepilogo delle risposte cellulari normalizzate dall'intera piastra nel test HTS. La media (μ) e la deviazione standard (σ) sono ottenute dall'analisi del vault CDD. Abbreviazioni: HTS = screening ad alto rendimento; RFUs = unità di fluorescenza relativa; 1s (campioni) = 320 composti testati; μs = valore medio delle risposte cellulari (RFU) da 320 pozzetti; μc- = valore medio delle RFU da controlli negativi; μc+ = valore medio delle RFU da controlli positivi; σs = deviazione standard delle risposte cellulari (RFU) da 320 pozzetti; σc- = deviazione standard delle RFU dai controlli negativi; σc+ = deviazione standard delle RFU dai controlli positivi.
Tabella supplementare S1: Programmi personalizzati del sistema di gestione dei liquidi per le fasi automatiche di pipettaggio. Questa tabella è stata modificata da Xiong et al.31. Clicca qui per scaricare questo file.
Tabella supplementare S2: I dati grezzi dei risultati rappresentativi. Le righe evidenziate in verde rappresentano i colpi dell'agonista e le righe evidenziate in arancione rappresentano i colpi dell'antagonista. Clicca qui per scaricare questo file.
Discussione
L'obiettivo di HTS è identificare le molecole colpite attraverso lo screening di un numero enorme di piccole molecole. Pertanto, i risultati di questo esempio rappresentano solo una piccola parte di un esperimento HTS convenzionale. Inoltre, le molecole hit identificate devono essere convalidate in saggi a valle, come un saggio dose-dipendente sulla stessa linea cellulare ricombinante e su una linea cellulare CHO-K1 che trasporta solo il vettore vuoto, che può essere eseguito simultaneamente per salvare piccole molecole. I saggi di citotossicità aiuteranno a dimostrare che una mancanza di risposta nella seconda aggiunta non è dovuta alla mortalità cellulare. Altri saggi biologici dovrebbero essere eseguiti per convalidare l'attività nei tessuti isolati in vitro, o applicando o alimentando le piccole molecole agli artropodi vivi. La pipeline completa di identificazione del ligando a piccole molecole del recettore della zecca kinina può essere trovata in una precedente pubblicazione31. Attraverso questa pipeline, sono stati identificati un totale di 36 colpi antagonisti del recettore della zecca chinina. Tre di questi antagonisti sono stati ulteriormente testati in un test di contrazione-inibizione tissutale e hanno mostrato attività anti-miotropica sull'intestino posteriore della zanzara, dove i recettori ortologhi della zanzara chinina sono espressi31.
Ogni linea cellulare che esprime GPCR deve essere convalidata per condizioni sperimentali adeguate prima di eseguire HTS. Queste condizioni includono la densità cellulare (10.000 cellule per pozzetto), la concentrazione dei composti schermati (2 μM), la concentrazione finale di DMSO (0,2%) nel test, la concentrazione del ligando agonista utilizzato per la seconda aggiunta (1 μM), la velocità e la profondità del pipettaggio e il tempo di lettura dopo l'aggiunta del peptide agonista (5 min). È fondamentale regolare la profondità e la velocità del sistema di gestione dei liquidi per garantire che le fasi di erogazione non disturbino le celle se le celle sono aderenti. La convalida di queste condizioni prima di intraprendere HTS aiuterà nella risoluzione dei problemi se la qualità del test è scarsa. È importante avere le cellule in condizioni ottimali: le cellule dovrebbero raggiungere il 90% -100% di confluenza ma non essere eccessivamente confluenti nella piastra cellulare quando utilizzate per HTS. Se i segnali di fondo in alcuni pozzetti sono anormalmente bassi o alti, dopo aver confermato al microscopio la densità cellulare ovviamente bassa o alta, i valori anomali devono essere rimossi manualmente durante l'analisi dei dati. Prima di adattare il saggio di fluorescenza del calcio all'HTS, è necessario conoscere la curva cinetica del peptide positivo, poiché la risposta cinetica delle cellule al peptide agonista dovrebbe durare 1-2 minuti per essere adattata in un test HTS e durare abbastanza a lungo da leggere l'intera piastra. Per ottenere il segnale prolungato, deve essere applicata una concentrazione relativamente elevata del peptide agonista o deve essere testata una densità cellulare più elevata. Se la risposta cinetica di determinate cellule recettoriali è troppo breve (dura meno di 1 minuto), si raccomanda di leggere a una velocità maggiore nella lettura del punto finale o di utilizzare solo una parte della piastra per HTS. Certamente, la qualità della linea cellulare ricombinante rispetto al livello di espressione del recettore è cruciale per il successo. Diverse linee cellulari clonali probabilmente variano, poiché l'inserimento del plasmide è casuale nel genoma, e questo potrebbe certamente influenzare l'espressione del recettore. Sarebbe ideale incorporare un test di fluorescenza del calcio precoce del pool trasfettato e durante il processo di selezione delle linee cellulari clonali.
Il limite del saggio di fluorescenza del calcio è che misura l'attività dei messaggeri secondari invece del legame diretto del ligando al recettore. Sebbene sia possibile escludere i colpi agonisti off-target esaminando le molecole colpite sulle cellule che non esprimono il recettore bersaglio, è impossibile escludere completamente le molecole colpite antagoniste off-target usando questo test perché è possibile che alcune molecole possano inibire le vie di rilascio del calcio cellulare a valle della segnalazione GPCR. Pertanto, è necessaria un'ulteriore convalida utilizzando un saggio di legame del ligando o un saggio biologico per la convalida a livello di organismo, come in un test del tessuto delle zecche con un agonista noto, come fatto in precedenza con le zanzare31.
La maggior parte dei saggi funzionali che rilevano l'attivazione GPCR misurano principalmente gli eventi intracellulari in due principali vie di segnalazione: vie dipendenti dalla proteina G e vie indipendenti dalla proteina G. I saggi dipendenti dalla proteina G misurano le concentrazioni di Ca2+, inositolo trifosfato (IP3) o cAMP, mentre i saggi indipendenti dalla proteina G rilevano principalmente il traffico di recettori o il reclutamento di β-arrestina7. Diversi saggi utilizzano tecnologie diverse per le letture, come coloranti sensibili al Ca 2+ (saggi di fluorescenza), fotoproteine come aequorine (saggi di bioluminescenza Ca2+)14, proteine di fluorescenza marcate, saggi basati sul trasferimento di energia di risonanza di fluorescenza (FRET), saggi basati su trasferimento di energia di risonanza di bioluminescenza (BRET) o saggi cellulari basati su biosensori ottici senza etichetta. Questi saggi e le corrispondenti tecnologie di lettura disponibili sono stati esaminati in dettaglio da Zhang e Xie35. Alcuni test sono applicabili a qualsiasi GPCR e alcuni sono specifici a determinati GPCR (ad esempio, il saggio Ca2+ per i recettori Gq-accoppiati, cAMP per i recettori Gi/o- o Gs-accoppiati). Tuttavia, co-esprimendo promiscuamente alcune proteine G con il recettore nelle cellule ricombinanti, la via di segnalazione può essere alterata. Ad esempio, le proteine Gq15/16 possono essere co-espresse con recettori non-Gq-accoppiati per il rilevamento della segnalazione PLC-IP3-Ca 2+ 7,36. In contrasto con i saggi che misurano un singolo evento molecolare, ci sono saggi di analisi ad alto contenuto, che raccolgono una quantità enorme di dati biologici per migliorare la specificità del ligando37. Questo tipo di test è anche più complesso e costoso da eseguire7.
Nella ricerca sugli invertebrati, i saggi funzionali più utilizzati rilevano l'attivazione GPCR analizzando le fluttuazioni delle concentrazioni di secondi messaggeri, come Ca2+ o cAMP. A causa delle differenze nelle vie di segnalazione tra mammiferi e invertebrati, la maggior parte delle altre tecnologie di lettura sopra menzionate non sono state ancora ampiamente adottate per gli studi GPCR sugli invertebrati. Tuttavia, le proteine G sono relativamente conservate in tutto il regno animale. Recentemente, Lismont et al.38 hanno dimostrato che alcuni biosensori di proteine G basati su BRET umani possono anche essere adattati per rilevare l'attivazione di un insetto GPCR quando co-espresso nelle cellule ricombinanti del recettore. Co-esprimendo ciascuno dei diversi biosensori basati sulla proteina G (Gs, Gi/o, Gq/11 o G12/13) con specifici GPCR, è possibile chiarire il meccanismo di accoppiamento della proteina G. Nello studio di Lismont et al.38, Schgr-CRF-DH potrebbe attivare in modo dose-dipendente i biosensori G i/o e G q/11 e non ha attivato i biosensori Gs e G12/13.
Nel complesso, il saggio di fluorescenza del calcio eseguito in una piastra da 384 pozzetti in combinazione con l'approccio della "doppia addizione" qui descritto consente lo screening di decine di migliaia di molecole in un tempo relativamente breve. Questo è un approccio relativamente efficiente in termini di tempo e costi per scoprire molecole hit per le convalide e le applicazioni a valle.
Divulgazioni
Gli autori non hanno alcun conflitto di interessi da rivelare.
Riconoscimenti
Questo lavoro è stato supportato dal premio USDA-NIFA-AFRI Animal Health and Well-Being Award (numero di premio 2022-67015-36336, PVP [Project Director]) e da fondi competitivi del Texas A&M AgriLife Research Insect Vector Diseases Grant Program (FY'22-23) a P.V.P. Il gruppo di facoltà A.W.E.S.O.M.E. del College of Agriculture and Life Sciences, TAMU, è riconosciuto per l'aiuto nella modifica del manoscritto. La tabella supplementare S2 contiene i dati di una libreria interna, casuale, di piccole molecole ottenuta dal laboratorio del Dr. James Sacchettini presso la Texas A & M University e Texas A & M AgriLife Research.
Materiali
| Name | Company | Catalog Number | Comments |
| 0.25% trypsin-EDTA | Gibco Invitrogen | 15050-065 | with phenol red |
| 0.4% trypan blue | MilliporeSigma | T8154 | liquid, sterile |
| 1.5 mL microcentrifuge tubes | Thermo Fisher | AM12400 | RNase-free Microfuge Tubes |
| 5 mL serological pipette | Corning | 29443-045 | Corning Costar Stripette individually wrapped |
| 10 mL serological pipette | Corning | 29443-047 | Corning Costar Stripette individually wrapped |
| 15 mL conical tubes | Falcon | 352196 | sterile |
| 20 µL filter tips | USA Scientifc Inc. | P1121 | sterile, barrier |
| 25 mL serological pipette | Corning | 29443-049 | Corning Costar Stripette individually wrapped |
| 50 mL conical tubes | Corning | 430828 | graduated, sterile |
| 150 mL auto-friendly reservior | Integra Bioscience | 6317 | sterile, individually wrapped for cell seeding in day 1 |
| 150 mL auto-friendly reservior | Integra Bioscience | 6318 | sterile, stacked, for loading dye in day 2 |
| 384/ 12.5 µL low retention tips | Integra Bioscience | 6405 | long, sterile filter |
| 384/ 12.5 µL tips | Integra Bioscience | 6404 | long, sterile filter |
| 384-well plate | Greiner | 781091 | CELLSTAR, clear polystyrene, µClear, Black/Flat |
| Aluminum plate seals | Axygen Scientific | PCR-AS-200 | polyester-based |
| Aluminum foil wrap | Walmart | ||
| Biosafty cabinet II | NuAire | NU-540-300 | |
| Cell counter | Nexcelom | AutoT4 | |
| cell counting slides | Nexcelom | SD-100 | 20 µL chamber |
| CO2 humidified incubator | Thermo Fisher | Forma Series II | |
| Desk Lamp | SunvaleeyTEK | RS1000B | |
| Dimethyl sulfoxide | MilliporeSigma | 276855 | anhydous, >99.9% |
| Drug plate | Corning | 3680 | |
| Dulbecco's phosphate-buffered saline | Corning | 21-031-CV | DPBS, 1x without calcium amd magnesium |
| Ethanol | Koptec | 2000 | |
| F-12K Nutrient Mixture | Corning | 45000-354 | (Kaighn's Mod.) with L-glutamine |
| Fetal bovine serum | Equitech-Bio | SFBU30 | |
| Fluorescent calcium assay kit | ENZO Lifescience | ENZ-51017 | 10x96 tests |
| G418 sulfate | Gibco Invitrogen | 10131-027 | Geneticin selective antibiotic 50 mg/mL |
| Hank's buffer | MilliporeSigma | 55037C | HBSS modified, with calcium, with magnesium, without phenol read |
| HEPES buffer | Gibco Invitrogen | 15630-080 | 1 Molar |
| HTS data storage plateform | CDD vault | https://www.collaborativedrug.com/ | |
| Liquid handling system | Integra Bioscience | Viaflo | 384/12.5 µL |
| Plate centrifuge | Thermo Fisher | Sorvall ST8 | |
| Plate reader | BMG technology | Clariostar | |
| Poly-D-lysine | MilliporeSigma | P6407 | |
| Rhimi-K-1 agonist peptide | Genscript | custom order | QFSPWGamide |
| T-75 flask | Falcon | 353136 |
Riferimenti
- Hanlon, C. D., Andrew, D. J. Outside-in signaling - A brief review of GPCR signaling with a focus on the Drosophila GPCR family. Journal of Cell Science. 128 (19), 3533-3542 (2015).
- Liu, N., Li, T., Wang, Y., Liu, S. G-protein coupled receptors (GPCRs) in insects-A potential target for new insecticide development. Molecules. 26 (10), 2993(2021).
- Pierce, K. L., Premont, R. T., Lefkowitz, R. J. Seven-transmembrane receptors. Nature Reviews Molecular Cell Biology. 3, 639-650 (2002).
- Pietrantonio, P. V., Xiong, C., Nachman, R. J., Shen, Y. G protein-coupled receptors in arthropod vectors: Omics and pharmacological approaches to elucidate ligand-receptor interactions and novel organismal functions. Current Opinion in Insect Science. 29, 12-20 (2018).
- Hilger, D., Masureel, M., Kobilka, B. K. Structure and dynamics of GPCR signaling complexes. Nature Structural & Molecular Biology. 25 (1), 4-12 (2018).
- Liu, N., Wang, Y., Li, T., Feng, X. G-protein coupled receptors (GPCRs): Signaling pathways, characterization, and functions in insect physiology and toxicology. International Journal of Molecular Sciences. 22 (10), 5260(2021).
- Hansen, K. B., Bräuner-Osborne, H. FLIPR® assays of intracellular calcium in GPCR drug discovery. G Protein-Coupled Receptors in Drug Discovery. Leifert, W. , Humana Press. Totowa, NJ. (2009).
- Bauknecht, P., Jekely, G. Large-scale combinatorial deorphanization of Platynereis neuropeptide GPCRs. Cell Reports. 12 (4), 684-693 (2015).
- Frooninckx, L., et al. Neuropeptide GPCRs in C. elegans. Frontiers in Endocrinology. 3, 167(2012).
- Caers, J., et al. More than two decades of research on insect neuropeptide GPCRs: An overview. Frontiers in Endocrinology. 3, 151(2012).
- Šimo, L., Koči, J., Park, Y. Receptors for the neuropeptides, myoinhibitory peptide and SIFamide, in control of the salivary glands of the blacklegged tick Ixodes scapularis. Insect Biochemistry and Molecular Biology. 43 (4), 376-387 (2013).
- Šimo, L., Park, Y. Neuropeptidergic control of the hindgut in the black-legged tick Ixodes scapularis. International Journal for Parasitology. 44 (11), 819-826 (2014).
- Liesch, J., Bellani, L. L., Vosshall, L. B. Functional and genetic characterization of neuropeptide Y-like receptors in Aedes aegypti. PLoS Neglected Tropical Diseases. 7 (10), 2486(2013).
- Lu, H. L., Kersch, C. N., Taneja-Bageshwar, S., Pietrantonio, P. V. A calcium bioluminescence assay for functional analysis of mosquito (Aedes aegypti) and tick (Rhipicephalus microplus) G protein-coupled receptors. Journal of Visualized Experiments. (50), e2732(2011).
- Xiong, C., Baker, D., Pietrantonio, P. V. The cattle fever tick, Rhipicephalus microplus, as a model for forward pharmacology to elucidate kinin GPCR function in the Acari. Frontiers in Physiology. 10, 1008(2019).
- Holmes, S. P., Barhoumi, R., Nachman, R. J., Pietrantonio, P. V. Functional analysis of a G protein-coupled receptor from the Southern cattle tick Boophilus microplus (Acari: Ixodidae) identifies it as the first arthropod myokinin receptor. Insect Molecular Biology. 12 (1), 27-38 (2003).
- Cox, K. J., et al. Cloning, characterization, and expression of a G-protein-coupled receptor from Lymnaea stagnalis and identification of a leucokinin-like peptide, PSFHSWSamide, as its endogenous ligand. Journal of Neuroscience. 17 (4), 1197-1205 (1997).
- Dircksen, H. Chapter 32 - Crustacean bioactive peptides. Handbook of Biologically Active Peptides (Second Edition). Kastin, A. J. , Academic Press. Cambridge, MA. 209-221 (2013).
- Halberg, K. A., Terhzaz, S., Cabrero, P., Davies, S. A., Dow, J. A. Tracing the evolutionary origins of insect renal function. Nature Communications. 6, 6800(2015).
- Pietrantonio, P. V., Jagge, C., Taneja-Bageshwar, S., Nachman, R. J., Barhoumi, R. The mosquito Aedes aegypti (L.) leucokinin receptor is a multiligand receptor for the three Aedes kinins. Insect Molecular Biology. 14 (1), 55-67 (2005).
- Radford, J. C., Davies, S. A., Dow, J. A. Systematic G-protein-coupled receptor analysis in Drosophila melanogaster identifies a leucokinin receptor with novel roles. Journal of Biological Chemistry. 277 (41), 38810-38817 (2002).
- Brock, C. M., et al. The leucokinin-like peptide receptor from the cattle fever tick, Rhipicephalus microplus, is localized in the midgut periphery and receptor silencing with validated double-stranded RNAs causes a reproductive fitness cost. International Journal for Parasitology. 49 (3-4), 287-299 (2019).
- Nässel, D. R. Leucokinin and associated neuropeptides regulate multiple aspects of physiology and behavior in Drosophila. International Journal of Molecular Sciences. 22 (4), 1940(2021).
- Kim, Y. -J., et al. Central peptidergic ensembles associated with organization of an innate behavior. Proceedings of the National Academy of Sciences of the United States of America. 103 (38), 14211-14216 (2006).
- Al-Anzi, B., et al. The leucokinin pathway and its neurons regulate meal size in Drosophila. Current Biology. 20 (11), 969-978 (2010).
- Yurgel, M. E., et al. A single pair of leucokinin neurons are modulated by feeding state and regulate sleep-metabolism interactions. PLoS Biology. 17 (2), 2006409(2019).
- Nässel, D. R., Zandawala, M. Recent advances in neuropeptide signaling in Drosophila, from genes to physiology and behavior. Progress in Neurobiology. 179, 101607(2019).
- Okusawa, S., Kohsaka, H., Nose, A. Serotonin and downstream leucokinin neurons modulate larval turning behavior in Drosophila. Journal of Neuroscience. 34 (7), 2544-2558 (2014).
- Kersch, C. N., Pietrantonio, P. V. Mosquito Aedes aegypti (L.) leucokinin receptor is critical for in vivo fluid excretion post blood feeding. FEBS letters. 585 (22), 3507-3512 (2011).
- Kwon, H., et al. Leucokinin mimetic elicits aversive behavior in mosquito Aedes aegypti (L.) and inhibits the sugar taste neuron. Proceedings of the National Academy of Sciences of the United States of America. 113 (25), 6880-6885 (2016).
- Xiong, C., Baker, D., Pietrantonio, P. V. A random small molecule library screen identifies novel antagonists of the kinin receptor from the cattle fever tick, Rhipicephalus microplus (Acari: Ixodidae). Pest Management Science. 77 (5), 2238-2251 (2021).
- Torfs, P., et al. The kinin peptide family in invertebrates. Annals of the New York Academy of Sciences. 897 (1), 361-373 (1999).
- Ma, Q., Ye, L., Liu, H., Shi, Y., Zhou, N. An overview of Ca2+ mobilization assays in GPCR drug discovery. Expert Opinion on Drug Discovery. 12 (5), 511-523 (2017).
- Zhang, J. -H., Chung, T. D., Oldenburg, K. R. A simple statistical parameter for use in evaluation and validation of high throughput screening assays. Journal of Biomolecular Screening. 4 (2), 67-73 (1999).
- Zhang, R., Xie, X. Tools for GPCR drug discovery. Acta Pharmacologica Sinica. 33 (3), 372-384 (2012).
- Offermanns, S., Simon, M. I. Gα15 and Gα16 couple a wide variety of receptors to phospholipase C. Journal of Biological Chemistry. 270 (25), 15175-15180 (1995).
- Murgia, M. V., et al. High-content phenotypic screening identifies novel chemistries that disrupt mosquito activity and development. Pesticide Biochemistry and Physiology. 182, 105037(2022).
- Lismont, E., et al. Can BRET-based biosensors be used to characterize G-protein mediated signaling pathways of an insect GPCR, the Schistocerca gregaria CRF-related diuretic hormone receptor. Insect Biochemistry and Molecular Biology. 122, 103392(2020).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon