Method Article
Analisi citofluorimetrica delle vescicole extracellulari dal supporto di cella-condizionato
In questo articolo
Riepilogo
Il protocollo descrive un metodo riproducibile progettato per l'utilizzo con i surnatanti di coltura cellulare per rilevare superfici epitopi sulle piccole vescicole extracellulari (EV). Utilizza specifici EV immunoprecipitazione usando i branelli accoppiati con gli anticorpi che riconoscono l'antigene di superficie CD9, CD63 e CD81. Il metodo è ottimizzato per l'analisi di citometria a flusso a valle.
Abstract
Citometria a flusso (FC) è il metodo di scelta per misurazione semi-quantitativa dei marcatori di superficie cellulare antigene. Recentemente, questa tecnica è stata utilizzata per le analisi fenotipiche di vescicole extracellulari (EV) tra cui esosomi (Exo) nel sangue periferico e altri fluidi corporei. Le piccole dimensioni di EV impone l'utilizzo di strumenti dedicati, avendo una soglia di rilevamento intorno 50-100 nm. In alternativa, EV può essere associato a microsfere in lattice che possono essere rilevati da FC. Microsfere, coniugato con gli anticorpi che riconoscono EV-collegata marcatori/Cluster di differenziazione CD63, CD9 e CD81 può essere utilizzato per la cattura di EV. Exo isolato da CM possa essere analizzato con o senza pre-arricchimento di ultracentrifugazione. Questo approccio è adatto per analisi di EV utilizzando strumenti convenzionali di FC. I nostri risultati dimostrano una correlazione lineare tra i valori di intensità di fluorescenza media (MFI) e concentrazione di EV. Perturbare EV tramite sonicazione drammaticamente diminuito MFI, che indica che il metodo non rileva i detriti della membrana. Segnaliamo un metodo accurato ed affidabile per l'analisi degli antigeni di superficie EV, che può essere implementato facilmente in qualsiasi laboratorio.
Introduzione
Le cellule secernono vescicole extracellulari (EV) di diverse dimensioni tra cui microvescicole (MV) ed esosomi (Exo). Quest'ultimo può essere distinto da MV da entrambe le dimensioni e il compartimento subcellulare di origine. MV (200 – 1.000 nm in dimensioni) vengono rilasciati dalle cellule del genitore versando dalla membrana del plasma. Al contrario, Exo (30 – 150 nm) provengono dalle membrane endosomal e vengono rilasciati nello spazio extracellulare quando la MVB (MVB) si fondono con la membrana cellulare1,2.
EV sono sempre più utilizzati come biomarcatori diagnostici così come, potenzialmente, strumenti terapeutici in molti campi compreso oncologia, neurologia, la cardiologia e malattie del sistema muscoloscheletrico3,4,5. Una stragrande maggioranza degli studi in corso utilizzando EV come agenti terapeutici sfruttano l'isolamento delle vescicole dal cellulare-condizionato medium (CM) di cellule coltivate in vitro. Cellule staminali mesenchimali (MSCs) esercitare effetti benefici in vari contesti, e MSC-derivato EV hanno dimostrato benefici in modelli di ischemia/riperfusione miocardica lesione6 e cervello lesioni7. MSC-derivato EV inoltre esibiscono attività modulatory immuni che può essere sfruttata per trattare il rigetto immunitario, come dimostrato in un modello di malattia di terapia-refrattario graft - versus - host8. Le cellule staminali del liquido amniotiche (hAFS) attivamente arricchire CM con MVs ed Exo, eterogeneo nelle dimensioni (50 – 1.000 nm), che mediano gli effetti biologici diversi, come la proliferazione di cellule differenziate, angiogenesi, inibizione di fibrosi, e cardioprotezione4. Abbiamo recentemente dimostrato che EV e particolarmente Exo, secreta dalle cellule progenitrici derivate cardiaco umano (Exo-CPC) ridurre la dimensione di infarto del miocardio in ratti5,9.
Exo Condividi un insieme comune di proteine sulla loro superficie, tra cui tetraspannine (CD63, CD81, CD9) e complesso di istocompatibilità di classe I (MHC-I). Oltre a questo insieme comune delle proteine, Exo contengono anche proteine specifiche per il sottoinsieme di EV del tipo cella produttore. Marcatori di Exo stanno guadagnando importanza fondamentale perché giocano un ruolo cruciale nella comunicazione intercellulare, quindi che regolano molti processi biologici5,10. A causa delle loro piccole dimensioni, trovare un modo semplice per analizzare EV usando il classico flusso cytometry (FC) rimane un compito impegnativo.
Qui, presentiamo un protocollo semplificato per l'analisi di EV con FC, che può essere applicato a campioni pre-arricchiti ottenuti attraverso ultracentrifugazione o direttamente a CM (Figura 1). Il metodo utilizza perline rivestite con un anticorpo specifico che si lega epitopi superfici Exo-collegata canonici (CD63, CD9, CD81) senza ulteriori lavaggi. FC analisi possono essere eseguite utilizzando un citometro convenzionale senza necessità di regolazioni prima di misurazioni. Metodi per la caratterizzazione degli antigeni su singole piccole particelle utilizzando citometri a flusso sono state descritte da altri gruppi rispetto alle varie applicazioni11,12,13. Qui, abbiamo usato microsfere magnetiche funzionalizzate per la cattura di piccole particelle ed Exo, seguita da fenotipizzazione delle particelle catturate dalla FC. Anche se questo metodo può essere utilizzato per caratterizzare la composizione antigenica di piccole vescicole rilasciate da qualsiasi tipo di cellula in vitro, qui abbiamo fornito le condizioni di coltura delle cellule specifiche che si applicano per la coltura delle cellule progenitrici cardiache umane (CPC) e la maggior parte ambiente adatto per la produzione di EV da queste cellule.
Protocollo
1. raccolta ed elaborazione dei Media condizionati
- Cappotto 55 cm2 capsule di Petri con gelatina di pelle porcina 0,02% in PBS.
- Piastra CPC (8.000/cm2) in piatti prerivestiti con 7 mL di per volta Dulbecco di Iscove Medium (IMDM) completati con 20% FBS (siero fetale bovino) e 1% di penicillina/streptomicina (P/S).
Nota: Il termine "CPC" si riferisce alle cellule di espianto umano derivato che sono stati descritti altrove14. CM possono essere raccolti da diversi tipi di cellule coltivati in condizioni di coltura specifici. Indossare guanti e lavorare sotto una cappa biologica. - Una volta che le cellule raggiungono circa 80% confluenza, rimuovere il terreno di coltura, lavare due volte con di Dulbecco PBS (PBS) Ca e Mg-privo di cellule e sostituirlo con 10 mL di medium di Exo-produzione senza siero (dell'Aquila per volta di Dulbecco medie [DMEM] alto glucosio, 4,5 g/mL).
Nota: Procedere con una progressiva diminuzione della concentrazione sierica nel terreno di coltura, di adattarsi gradualmente le cellule alla condizione privo di siero (SF). Per le celle che sono sensibili al medium senza siero preparare un supporto contenente FBS ma impoverito di EV. Preparare supplementato con tutte le sostanze nutritive, oltre a 10 – 20% (v/v) FBS. Centrifugare il mezzo durante la notte a 100.000 × g, 4 ° C. Essere consapevoli del fatto che questa procedura non garantisce la rimozione di 100% di FBS-derivato EV. - Dopo 7 giorni, raccogliere i CM in un provette in polipropilene.
Nota: Il tempo per media condizionata dipende il tipo di cella. Per le celle che sono sensibili al medium senza siero, aumentare il volume iniziale del mezzo e diminuire il tempo per la raccolta di CM per 24-48 h. - Deselezionare CM da detriti cellulari di centrifugare a 3.000 x g per 20 min 4 – 10 ° C. Raccogliere il surnatante in un tubo di filtro centrifugo kDa 100.
- Concentrato deselezionata CM facendo girare il tubo a 2.000 x g per 20 min a 4 – 10 ° C.
Nota: Questo passaggio riduce il volume iniziale di CM permettendo l'uso di tubi di piccolo-volume per la procedura di centrifuga ad alta velocità e contribuirà a eliminare aggregati proteici piccolo. - Raccogliere il concentrato in un tubo del microcentrifuge. Centrifugare a 10.000 x g per 15 min 4 – 10 ° C.
- Raccogliere il surnatante e passare alla sezione 2 (EV arricchimento). Se non è disponibile un'ultracentrifuga passare direttamente alla sezione 3 (quantificazione Nanoparticle Tracking Analysis (NTA)).
Nota: Pre-arricchimento di EV frazione migliora la lettura finale nell'intensità di fluorescenza (Vedi risultati rappresentativi e Figura 4). Sgomberati CM (dal punto 1.8) possono essere memorizzati a 4 ° C per non più di 1-2 giorni o in alternativa a − 80 ° C per diversi mesi. 5 CM deve essere pre-liquidati per rimuovere eventuali detriti cellulari prima di conservazione a-80 ° C al fine di garantire che detriti esosomi e medie non si sono costituito nel processo di congelamento-scongelamento.
2. extracellulare vescicole arricchimento di ultracentrifuga (opzionale)
- Posto il supernatante dal passo 1,8 in un tubo di policarbonato spessore parete ultracentrifuga. Riempire la provetta con la soluzione salina tampone fosfato (PBS): 1x finché raggiunge la massima capacità (3,2 mL).
- Caricare i campioni in un rotore ad angolo fisso di titanio (8 x 3.2 mL, fattore k-13). Caricare il rotore in un'ultracentrifuga da tavolo. Ultracentrifuga a 100.000 x g per 3 h 4 – 10 ° c.
- Eliminare il supernatante e risospendere il pellet in 100 µ l di 1X PBS.
3. Nanoparticle Tracking Analysis (NTA) quantificazione
- Diluire 1 µ l di campione (da passo 2.3 o 1.8) a 999 µ l di PBS 1X. Caricare il campione in una siringa da 1 mL, evitando la formazione di bolle. Caricare la siringa nella porta di ingresso della camera l'esame.
- Accendere il laser. Aprire la fotocamera con il tasto di acquisizione . Regolare il fuoco.
- Registrare almeno 3 diversi telai: 60 s ciascuno.
- Analizzare le 3 diverse acquisizioni utilizzando l'opzione di Elaborazione Batch nel software.
Nota: Se la tecnologia NTA non è disponibile, eseguire una quantificazione della proteina dall'analisi di Bradford (1 x 108 particelle corrispondono a 1 – 2 µ g di proteina totale dopo ultracentrifugazione o a 50 – 60 µ g proteina totale se la quantificazione è effettuata direttamente per CM come Esso comprende contaminanti di proteine differenti da EV; Vedi tabella 1)5.
4. preparazione del campione
- Preparare un pool di 3 tipi di perline di cattura (CD9 perline, perline di CD63 e CD81 perline a un 1: rapporto 1:1; si veda Tabella materiali) e vortex.
Nota: La piscina è stata testata per essere stabile per 1 mese. Può essere utilizzato un solo tipo di cattura della perla, questo permetterà la rilevazione delle sub-popolazioni EV non discriminabili con piscina di perline. Utilizzando singolo antigene cattura perline, sarà possibile al contemporaneo dimostrare la presenza di proteine di studi, quali CD9, CD81 o CD63, e l'antigene di interesse rilevati dall'anticorpo fluorescente. Granuli di lattice alternativi aldeide-solfato sono anche disponibili in commercio. Queste perle presentano superficie idrofoba per l'adsorbimento di MV ed EV. - In un tubo di fondo tondo, aggiungere la quantità di volume corrispondente a 1 x 108 particelle più 1 µ l della piscina della perla, corrispondente a 1,2 x 105 perline in totale (per ciascun test).
- Preparare una provetta contenente 1 µ l di perline senza EV che servirà come controllo negativo, di cui dichiara come "Perle".
- Regolare il volume a 100 µ l con 1x PBS. Collocare la provetta in un termo-mixer e agitare a 400 giri/min durante la notte a 4 – 10 ° C.
- Il giorno successivo, spostare i campioni a una piastra a 96 pozzetti fondo tondo (o tubi FC).
- Aggiungere l'anticorpo (10 µ g/mL di CD9_FITC, 10 µ g/mL di CD63_PE e 5 µ g/mL di CD81_PE).
- Incubare 1h a 4 – 10 ° C.
- Aggiungere 100 µ l di 1X PBS in ciascun pozzetto.
- Procedere all'acquisizione.
5. acquisizione
- Aprire il software di acquisizione e start up dello strumento (citometro > programma di avvio di sistema). Aprire un nuovo esperimento.
- Creare un modello sperimentale, quindi selezionare la piastra e quindi Aggiungi. Selezionare la posizione dei campioni sulla piastra e quindi premere Set come esempio ben.
- Immettere il nome del campione (cioè CPC n. 1_CD9FITC) nelle Regole di denominazione. Aprire la scheda di canale e accendere il FITC e canali di PE.
- Caricare la piastra nello strumento.
- Premere Dot Plot e creare un nuovo grafico di punto (P1). Impostare Forward Scatter Area (FSC-A) contro zona Side Scatter (SSC-A).
- Selezionare inizializzare per iniziare il laser e la fluidica. Premere Acquisisci per avviare l'acquisizione.
- Regolare la scala per visualizzare la popolazione nel mezzo il P1. In P1 disegnare il cancello "Perline" intorno a tutta la popolazione (escluse detriti) (Figura 2A). Premere Stop.
- Premere Dot Plot e creare un nuovo grafico di dot (P2). Impostare Forward Scatter-altezza (FSC-H) versus FSC-A. Gate la nuova trama di punto su "Perline". In P2 disegnare un nuovo cancello intorno alla più grande popolazione e denominarlo "CAMICIOLE" (Figura 2A).
Nota: Questa strategia è stata progettata al fine di evitare il più possibile l'inclusione di aggregati di perline in fasi di analisi e acquisizione. - Premere Dot Plot e creare due punti diversi lotti; uno FITC contro SSC-A e uno PE contro SSC-A. Gate la nuova trama di punto su "Canottiere."
- Selezionare il numero di eventi da registrare (ad es., 20.000). Selezionare Record e iniziare l'esperimento.
6. analisi dei dati
- Caricare i file di acquisizione nel software di analisi.
Nota: I passaggi seguenti devono essere applicati ad ogni campione tra cui perline solo e ogni campione sconosciuto. - Aprire un nuovo protocollo e creare trame puntino seguendo la stessa strategia descritta nel passaggio 5.
- Impostare tutti gli assi di dispersione in scala lineare e tutti gli assi di fluorescenza a scala logaritmica.
- Visualizza la media geometrica di fluorescenza (X-Gmean-MFI) nei canali FITC e PE.
- Calcolare il rapporto X-Gdire dei campioni / X-Gdire delle perline colorate con anticorpo senza esosomi.
- Confrontare la variazione piega MFI di diverse preparazioni di EV.
7. extracellulare vescicola numero titolazione
Nota: Sezioni da 7 a 10 può essere eseguita per impostare il numero di particelle, la concentrazione di anticorpi e la specificità, ma può essere ignorato se il tipo di cella, da cui sono derivati EV, e gli anticorpi rimangono gli stessi.
- Diluire le particelle, ottenute dal passaggio 2.3, in PBS 1X per ottenere una serie di sospensioni che vanno da 5 x 105 a 2,5 x 108 particelle.
- Per ogni sospensione aggiungere in un tubo di fondo tondo 1 µ l di piscina di perlina di cattura.
- Seguire il protocollo dal punto 4.3.
8. titolazione di anticorpi
Nota: Titolazione di anticorpi è di solito eseguita con l'aggiunta di un singolo anticorpo per ogni provetta.
- Mantenere il numero di EV (totale delle particelle o contenuto proteico) costante in tutti i campioni.
Nota: Il numero appropriato è determinato empiricamente come descritto sopra nella sezione 7. - Seguire il protocollo da 4.1 a 4.5 di Spagna.
- Testare una gamma di concentrazioni nell'anticorpo sopra e sotto l'importo consigliato dal fornitore. Ad esempio, per un anticorpo con una concentrazione consigliata di 10 µ g/mL per ogni test, test 1, 2, 5, 10, 20 e 50 µ g/mL. Includere un campione con "solo perline", aggiungendo la più alta concentrazione di anticorpo.
- Incubare per 1 h a 4 – 10 ° C.
- Aggiungere 100 µ l di 1X PBS per ciascun campione.
- Acquisire i campioni.
9. incubazione
- Verificare il tempo di incubazione eseguire acquisizioni in diversi momenti (ad es., 1 h, 2h e 5 h).
- Seguire il protocollo a partire dalla sezione 4.
- Incubare i campioni a 4 – 10 ° C per 1 h.
- Acquisire campioni.
- Incubare i campioni a 4 – 10 ° C per 3 h.
- Acquisire campioni.
10. protocollo convalida
-
Associazione di EV
- In un tubo di fondo tondo, aggiungere la quantità di volume corrispondente a 1 x 108 particelle o la corrispondente quantità di proteine totali.
- Regolare il volume a 100 µ l con 1x PBS.
- Interromperebbe la membrana EV di sonicazione (in alternativa un passo di scossa di calore può essere applicato) secondo le istruzioni del produttore. Impostare la frequenza e l'intensità. Il periodo di sonicazione può essere stabilito empiricamente l'impostazione di un esperimento di corso di tempo (cioè, 0 s, 30 s, 1 min, 5 min, ecc.).
- Aggiungere 1 µ l della piscina della perla.
- Seguire il protocollo a partire dal punto 4.4.
-
Specificità dell'anticorpo
- Preparare una provetta per ogni fluorocromo coniugato IgG e aggiungere il complesso di perline-EV come descritto al punto 4.6.
Nota: L'isotipo fluorocromo-coniugate dovrà corrispondere con le IgG dell'anticorpo utilizzato.
- Preparare una provetta per ogni fluorocromo coniugato IgG e aggiungere il complesso di perline-EV come descritto al punto 4.6.
Risultati
Numero totale di particelle per la colorazione del singolo
Poiché una singola perlina possibile associare più di una particella, abbiamo testato diverse condizioni per impostare la quantità minima di totale EV (singolo anticorpo per provetta) per raggiungere la prima fase esponenziale della curva delle IFM. Una concentrazione fissa di anticorpo è stata utilizzata mentre il numero totale di particelle ha variato da 5 x 105 a 2,5 x 108. Come mostrato in Figura 3A, il numero di particelle che ci permette di garantire che l'anticorpo si esegue all'interno di un'IFM accettabile, evitando l'uso di un eccesso di EV, è 1 x 108 particelle/colorazione.
Titolazione di anticorpi
Abbiamo selezionato la corretta concentrazione di anticorpo conseguente il segnale più alto impedendo il legame non specifico dell'anticorpo. Questo test è stato ottimizzato per le particelle di 1 x 108 , come determinato nell'impostazione precedente. Anti-CD9_FITC, anti-CD63_PE e anti-CD81_PE sono stati testati con concentrazioni che variano da 1 a 50 µ g/mL (Figura 3B). Gli anticorpi anti-CD9_FITC e anti-CD63_PE ha dato una buona risoluzione del segnale (7,5 e 130-fold cambiamento di MFI vs perle da sola, rispettivamente) quando utilizzato alla concentrazione di 10 µ g/mL mentre la concentrazione selezionata per l'anticorpo anti-CD81_PE era di 5 µ g/mL (465.3 Fold Cambia MFI).
Validazione del metodo
Al fine di confermare che il nostro metodo è adatto per analizzare solo "a forma di Coppa" vescicole extracellulari e non membrana detriti, abbiamo applicato passaggi diversi sonicazione, al 10% dell'ampiezza, alla soluzione contenente particelle. 100 µ l di soluzione di PBS contenente particelle di 1 x 108 1x ha subito passaggi di sonicazione diverso da 30 secondi a 5 minuti e la preparazione risultante contenente sia rotto e ben sagomato EV sono stati analizzati come descritto (protocollo sperimentale dal punto 3.3). di conseguenza, abbiamo trovato che 30 secondi di sonicazione diminuire MFI che e ' stato completamente scaricato dopo 1 minuto. Presso questo timepoint, fluorescenza non è rilevabile per uno qualsiasi dei marcatori EV (Figura 3D).
Caratterizzazione di citometria a flusso degli esosomi HEK293 - e CPC-derivati
Questi risultati sono stati generati seguendo il protocollo presentato sopra con il metodo di isolamento ultracentrifuga. Gli esosomi isolati sono quantificati dalla tecnologia NTA e caricati durante la notte con 1 µ l di perline miste (1 x anti-CD9:1 x anti-CD63:1 x anti-CD81). Il complessi perline + Exo era macchiata con la corretta quantità di anticorpo anti-CD9_FITC, anti-CD63_PE e anti-CD81_PE (Figura 3EF e tabella 2).
Inoltre, utilizzando EV-CPC abbiamo confrontato FC analisi con o senza pre-arricchimento di ultracentrifugazione. La figura 4 Mostra che entrambi i metodi sono adatti per gli indicatori di superficie profilo di Exo. Pre-arricchimento della frazione di vescicole extracellulari migliora notevolmente l'intensità di fluorescenza soprattutto per CD63_PE e CD81_PE colorazione (Figura 4 e tabella 3).

Figura 1: protocollo e NTA trame. (A) rappresentazione schematica del protocollo sperimentale. (B) rappresentante NTA trame per passo Media condizionata, pre- ultracentrifuga e post-ultracentrifuga. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 2: acquisizione e analisi dati. (A) flusso cytometry analisi inizia con la creazione di un primo cancello a tutta la popolazione delle perle "perle" (escluse detriti) e poi un secondo cancello per distinguere gli eventi "camiciole". Canottiere sono recintati su un terreno impostato con FSC-H come asse x e FSC-A come asse y. (B-D) Punto rappresentativo appezzamenti risultati di spostamento a destra dell'intensità di fluorescenza per le popolazioni positive di perline-esosomi complessi (verde, CD9 +; rosso CD63 +; marrone CD81 +). Controllo di isotipo (viola) si sovrappongono negativi grigi perline colorate. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 3: Curve di titolazione per il numero di particelle (le frecce indicano l'importo selezionato di particelle, 1 x 108). (A) concentrazioni anticorpali (le frecce indicano la concentrazione selezionata per ognuno usate anticorpo). Concentrazione (B) sia il numero di particelle e l'anticorpo sono grafico vs intensità media di fluorescenza (MFI). (C) curva mostrando diversi 3 acquisizione della preparazione con 1 – 2 h o 5 h di incubazione dell'anticorpo. Curva (D) con il decremento della fluorescenza dopo sonicazione a intervalli di tempo diversi. (E-F) Piegare il cambiamento (media ± SD) di IFM per CD9, CD63 e CD81 vs controllo negativo (perline + anticorpi, non EV) sono indicati per HEK293 EV (n = 3 ripetizioni indipendenti) e per CPC EV (n = 3 linee cellulari primarie da 3 diversi pazienti) per favore clicca qui per visualizzare la versione ingrandita di Questa figura.
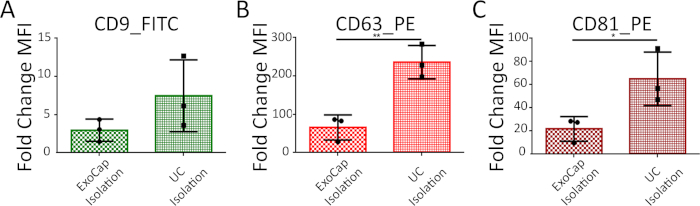
Figura 4: MFI analisi ed il confronto fra due procedure: EV-binding diretto con Perline (catturare perle isolamento) e pre-arricchimento con ultracentrifuga (isolamento ultracentrifuga). I dati sono mostrati come piegare il cambiamento (media ± SD) di IFM per CD9 (A), (B) CD63 e CD81 (C) vs controllo negativo (perline + anticorpi, non EV). N = 3 linee cellulari primarie da 3 diversi pazienti. Clicca qui per visualizzare una versione più grande di questa figura.
| CPC | CONC NTA (parte / µ l) | CONC (µ g / µ l) |
| CPC n. 1 pre-ultracentrifuga | 5.02 + 06 | 2.01 |
| CPC #1 post-ultracentrifuga | 6.10E + 07 | 1.04 |
| N. 2 CPC pre-ultracentrifuga | 5.74E + 06 | 2.30 |
| N. 2 CPC post-ultracentrifuga | 7.43E + 07 | 0.79 |
| N. 3 CPC pre-ultracentrifuga | 2.02 + 06 | 1.90 |
| N. 3 CPC post-ultracentrifuga | 2.91E + 07 | 0,42 |
Tabella 1: Confronto tra la concentrazione di NTA e concentrazione nella proteina per 3 diversi paziente derivato CPC prima e dopo ultracentrifuga.
| PIEGARE IL CAMBIAMENTO DI MFI ± SD | CD9 | CD63 | CD81 |
| HEK293 | 24.44 ± 19,17 | 430,7 ± 344,9 | 535.2 ± 410.3 |
| CPC N. 1 | 14.15 ± 3,72 | 236,05 ± 43.40 | 353.30 ± 452.43 |
| CPC N. 2 | 15.76 ± 1,87 | 983.06 ± 195.63 | 374.45 ± 108,05 |
| CPC N. 3 | 8.94 ± 7,19 | 830.50 ± 184.73 | 60.05 ± 23,18 |
Tabella 2: Valore di piega cambiamento (media ± SD) di IFM per HEK293 EV (n = 3 ripetizioni indipendenti) e CPC EV (n = 3 linee cellulari primarie da 3 diversi pazienti).
| PIEGARE IL CAMBIAMENTO DI MFI ± SD | CD9 | CD63 | CD81 |
| Catturare l'isolamento di perline | 2.96 ± 1,45 | 65.65 ± 18,87 | 21.85 ± 6.12 |
| Ultracentrifuga isolamento | 7,47 ± 2.71 | 236.00 ± 25,06 | 65,05 ± 13,38 |
Tabella 3: Valore di variazione piega della MFI (media ± SD) per CPC EV (n = 3 linee cellulari primarie da 3 diversi pazienti) isolato da cattura perline o ultracentrifuga.
Tabella 4: Specifiche del singolo prodotto.
Discussione
La tecnica convenzionale FC rimane il più semplice metodo analitico per la caratterizzazione di marcatori espressi sulla superficie di EV. A questo proposito, selezionando il protocollo più adatto è fondamentale per ottenere informazioni utili sulle frazioni delle singole particelle di interesse evitando limitazioni a causa di sensibilità dello strumento. Abbiamo descritto un metodo che utilizza particelle magnetiche accoppiate con gli anticorpi che riconoscono Exo e piccoli antigeni di superficie di EV che sono adatti per applicazione FC a valle. Abbiamo validato il metodo utilizzando due diversi tipi di cellule: CPC umano primario che stanno emergendo come una fonte di cellule principali per approcci terapeutici basati su Exo per la malattia cardiaca; e cellule HEK293, una linea di cellule immortalizzate ampiamente usata nella ricerca di biologia della cellula a causa della plasticità e crescita cellulare affidabile.
Il metodo può essere applicato a EV ultracentrifuga-arricchito e, per l'analisi più veloce, anche direttamente su in vitro cellule derivate CM senza pre-arricchimento di ultracentrifugazione. Il materiale di partenza è fondamentale quando si confrontano i campioni. Aggiunta di acquisizione perline direttamente al CM accelererà la procedura ma allo stesso tempo diminuire l'intensità di fluorescenza, come mostrato in Figura 4A. È anche fondamentale utilizzare una quantità appropriata di PBS per mescolare perline ed EV durante la fase di "acquisizione" 4.4. Quando si utilizza un tempo di incubazione costante, un aumento del volume diminuirà l'intensità di fluorescenza dovuta ad accoppiamento EV inefficiente.
Una limitazione del protocollo è che una perlina di cattura singola possibile associare più EV/Exo particelle sulla sua superficie. Ciò limiterà la possibilità di identificare sottogruppi di EV esprimendo la peculiare combinazione di antigeni utilizzando una colorazione più. Il metodo basato sul tallone produce pertanto dati semi-quantitativa. Usando i branelli che trasportano un singolo acquisizione Ab (CD9, CD63 o CD81) permetterà al massimo la caratterizzazione delle particelle che esprimono due epitopi: quello presente sul tallone e riconosciuto dall'anticorpo acquisizione e quello rilevato dall'anticorpo che è successivamente aggiunto.
Il gold standard attuale per Exo analisi utilizzando FC è un protocollo sviluppato da van der Vlist et in 201215. Esso consente un'analisi ad alta risoluzione di EV utilizzando una configurazione ottimizzata del FC High-end disponibili in commercio (ad es., BD afflusso). Questo protocollo è estremamente dettagliato e utile, ma ha ancora bisogno di un ambiente hardware complesso con taratura specifica FC prima dell'uso. Tre anni più tardi, Pospichalova et al.16 proposto un protocollo semplificato per l'analisi FC di Exo utilizzando un citometro dedicato specificamente sviluppato per l'analisi di piccole particelle (ad es., Apogee A50 Micro)17. Per quanto riguarda questo protocollo e altri che hanno usato la soglia speciale impostazione11, qui proponiamo un protocollo di base per eseguire piccoli phenotyping EV usando i branelli magnetici associazione che è adatto per gli strumenti convenzionali di FC e non richiede alcuna ambiente speciale. Diversi protocolli sono descritti metodi basati sul tallone per caratterizzare piccolo EV trovato in fluidi corporei di FC12. Qui, ci mostra la immunocapture di discreti sottopopolazioni delle vescicole positivo per CD9, CD63 e CD81 che sono comunemente usati come marcatori di Exo18. Aldeide-solfato lattice19,20 e polistirolo12perline rimangono valide alternative per l'associazione di EV presente in CM e fluido del plasma sanguigno; Tuttavia, aldeide gruppi esposti sulla superficie della particella polimero consentono accoppiamento delle proteine aspecifiche e altri materiali per le particelle di latex, aumentando così il rischio di contaminazione da organismi della lipoproteina o apoptotici durante il rilevamento e isolamento processo di21,22.
Perline utilizzate nel protocollo associare EV solo intero, ben sagomato. Abbiamo proposto di alterare la struttura di EV di sonicazione per placare il segnale (sezione 4, "protocollo validation"). Infatti, un minuto di risultati di sonicazione in intensità di fluorescenza diminuita, mostrando così che un segnale positivo non può risentire di detriti di membrana adsorbito su perline.
Divulgazioni
Niente da dichiarare.
Riconoscimenti
L.B. è stato sostenuto da borse di ricerca di Helmut Horten Stiftung e Velux Stiftung, Zurigo (Svizzera). G.V. è stato sostenuto da borse di ricerca di Swiss National Science Foundation, la Fondazione Cecilia-Augusta, Lugano e il SHK Stiftung für Herz-und Kreislaufkrankheiten (Svizzera)
Materiali
| Name | Company | Catalog Number | Comments |
| IMDM | Gibco | 12440061 | |
| Amicon Ultra-15, PLHK Ultracel-PL Membran, 100 kDa | Millipore | UFC910024 | |
| CytoFlex, Flow Cytometer Platform | Beckman Coulter | CytoFlex | |
| DMEM, high glucose, HEPES, no phenol red | Gibco | 21063045 | |
| Dulbecco's PBS (PBS) Ca- and Mg-free | Lonza | BE17-512F | |
| ExoCap CD63 Capture Kit | JSR Life Sciences | Ex-C63-SP | |
| ExoCap CD81 Capture Kit | JSR Life Sciences | Ex-C81-SP | |
| ExoCap CD9 Capture Kit | JSR Life Sciences | Ex-C9-SP | |
| Exosome-Depleted FBS | Thermofisher | A2720801 | |
| Exosome-depleted FBS Media Supplement | SBI | EXO-FBS-250A-1 | |
| FBS-Fetal Bovine Serum | Gibco | 10270106 | |
| FITC anti-human CD9 Antibody | Biolegend | 312104 RRID: AB_2075894 | |
| Flow Cytometer analysis software | Beckman Coulter | Kaluza | |
| NanoSight LM10 | Malvern | NanoSight LM10 | |
| NanoSight Software | Malvern | NTA 2.3 | |
| Optima Max-XP | Beckman Coulter | 393315 | |
| PE anti-human CD63 Antibody | Biolegend | 353004 RRID:AB_10897809 | |
| PE anti-human CD81 (TAPA-1) Antibody | Biolegend | 349505 RRID:AB_10642024 | |
| Penicillin-Streptomycin | Gibco | 15140122 | |
| Thermomixer C | Eppendorf | 5382 000 015 | |
| TLA-110 | Beckman Coulter | TLA-110 rotors |
Riferimenti
- Barile, L., Vassalli, G. Exosomes: Therapy delivery tools and biomarkers of diseases. Pharmacology & Therapeutics. , (2017).
- Thery, C. Exosomes: secreted vesicles and intercellular communications. Molecular Biology Reports. 3, 15 (2011).
- Yadav, D. K., et al. Liquid biopsy in pancreatic cancer: the beginning of a new era. Oncotarget. 9, 26900-26933 (2018).
- Balbi, C., et al. First Characterization of Human Amniotic Fluid Stem Cell Extracellular Vesicles as a Powerful Paracrine Tool Endowed with Regenerative Potential. Stem Cells Translational Medicine. 6, 1340-1355 (2017).
- Andriolo, G., et al. Exosomes From Human Cardiac Progenitor Cells for Therapeutic Applications: Development of a GMP-Grade Manufacturing Method. Frontiers in Physiology. 9, (2018).
- Lai, R. C., et al. Exosome secreted by MSC reduces myocardial ischemia/reperfusion injury. Stem Cell Research. 4, (2009).
- Doeppner, T. R., et al. Extracellular Vesicles Improve Post-Stroke Neuroregeneration and Prevent Postischemic Immunosuppression. Stem Cells Translational Medicine. 4, 1131-1143 (2015).
- Kordelas, L., et al. MSC-derived exosomes: a novel tool to treat therapy-refractory graft-versus-host disease. Leukemia. 28, 970-973 (2014).
- Barile, L., et al. Extracellular vesicles from human cardiac progenitor cells inhibit cardiomyocyte apoptosis and improve cardiac function after myocardial infarction. Cardiovascular Research. 103, 530-541 (2014).
- Longatti, A., et al. High affinity single-chain variable fragments are specific and versatile targeting motifs for extracellular vesicles. Nanoscale. 10, 14230-14244 (2018).
- Menck, K., Bleckmann, A., Schulz, M., Ries, L., Binder, C. Isolation and Characterization of Microvesicles from Peripheral Blood. Journal of Visualized Experiments. , (2017).
- Inglis, H., Norris, P., Danesh, A. Techniques for the analysis of extracellular vesicles using flow cytometry. Journal of Visualized Experiments. , (2015).
- Arakelyan, A., et al. Flow Virometry to Analyze Antigenic Spectra of Virions and Extracellular Vesicles. Journal of Visualized Experiments. , (2017).
- Chimenti, I., et al. Isolation and expansion of adult cardiac stem/progenitor cells in the form of cardiospheres from human cardiac biopsies and murine hearts. Methods Molecular Biology. 879, 327-338 (2012).
- van der Vlist, E. J., Nolte-'t Hoen, E. N., Stoorvogel, W., Arkesteijn, G. J., Wauben, M. H. Fluorescent labeling of nano-sized vesicles released by cells and subsequent quantitative and qualitative analysis by high-resolution flow cytometry. Nature Protocols. 7, 1311-1326 (2012).
- Pospichalova, V., et al. Simplified protocol for flow cytometry analysis of fluorescently labeled exosomes and microvesicles using dedicated flow cytometer. Journal of Extracellular Vesicles. 4, 25530 (2015).
- van der Pol, E., et al. Particle size distribution of exosomes and microvesicles determined by transmission electron microscopy, flow cytometry, nanoparticle tracking analysis, and resistive pulse sensing. Journal of Thrombosis and Haemostasis. 12, 1182-1192 (2014).
- Witwer, K. W., et al. Updating the MISEV minimal requirements for extracellular vesicle studies: building bridges to reproducibility. Journal of Extracellular Vesicles. 6, 1396823 (2017).
- Thery, C., Amigorena, S., Raposo, G., Clayton, A. Isolation and characterization of exosomes from cell culture supernatants and biological fluids. Current Protocols in Cellular Biology. , (2006).
- Suarez, H., et al. A bead-assisted flow cytometry method for the semi-quantitative analysis of Extracellular Vesicles. Scientific Reports. 7, 11271 (2017).
- Sodar, B. W., et al. Low-density lipoprotein mimics blood plasma-derived exosomes and microvesicles during isolation and detection. Scientific Reports. 6, 24316 (2016).
- Sluijter, J. P. G., et al. Extracellular vesicles in diagnostics and therapy of the ischaemic heart: Position Paper from the Working Group on Cellular Biology of the Heart of the European Society of Cardiology. Cardiovascular Research. 114, 19-34 (2018).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon