Method Article
Metodi per pH intracellulare Imaging del lignaggio di cellule staminali del follicolo in Live tessuto ovarico di Drosophila
In questo articolo
Riepilogo
Forniamo un protocollo per l'imaging di pH intracellulare di una stirpe delle cellule staminali epiteliali in vivo tessuto ovarico di Drosophila . Descriviamo i metodi per generare mosche transgenici che esprimono un biosensore pH, mCherry::pHluorin, immagine biosensore usando la formazione immagine di fluorescenza quantitativa, generare curve standard e convertire i valori di intensità di fluorescenza per valori di pH.
Abstract
Variazioni di pH intracellulare (pHi) svolgono i ruoli importanti nella regolazione di molte funzioni cellulari, compreso il metabolismo, proliferazione e differenziazione. In genere, pHi dinamiche sono determinati in cellule coltivate, suscettibili di misurazione e manipolando sperimentalmente pHi. Tuttavia, il recente sviluppo di nuovi strumenti e metodologie ha permesso di studiare pHi dinamica all'interno del tessuto intatto, vivo. Per la ricerca di Drosophila , uno sviluppo importante è stata la generazione di una linea transgenica che trasportano un biosensore pHi, mCherry::pHluorin. Qui, descriviamo un protocollo che usiamo abitualmente per formazione immagine dal vivo della drosofila ovarioles per misurare pHi del lignaggio di cellule staminali (FSC) epiteliali del follicolo in mCherry::pHluorin linee transgeniche wild-type; Tuttavia, i metodi descritti qui possono essere facilmente adattati per altri tessuti, compreso i dischi di ala e dell'epitelio dell'occhio. Descriviamo tecniche per esprimere mCherry::pHluorin del lignaggio di FSC, mantenimento del tessuto ovarico durante il live di imaging e acquisire ed analizzare le immagini per ottenere i valori di pHi.
Introduzione
Studi recenti hanno rivelato un ruolo per le modifiche a pHi durante cellulari displasia e differenziazione in vivo1,2. Questi studi hanno trovato che pHi è notevolmente coerenti in cellule dello stesso tipo nella stessa fase di differenziazione, ma che cambia come le cellule di transizione da una fase a altra. In alcuni casi, bloccando le modifiche a pHi parzialmente sconvolge la differenziazione, suggerendo che il cambiamento a pHi non è solo una conseguenza di cambiamenti nel destino della cellula ma invece aiuta a promuovere il cambiamento nel destino della cellula, magari attraverso effetti sulla regolamentazione sensibili al pH proteine o reazioni chimiche necessarie per la differenziazione. Gli studi futuri hanno il potenziale per rivelare più comprensione nei molti ruoli diversi di pHi dynamics in vivo. Tuttavia, una delle sfide di studiare pHi durante il differenziamento in vivo è ottenere misurazioni accurate di pHi. A differenza di altre caratteristiche di differenziazione, quali i cambiamenti nella morfologia cellulare e l'espressione genica, pHi è una proprietà chimica labile della cella che non viene mantenuta in cellule che sono stati corretti e permeabilizzate con metodi standard. Inoltre, pHi può non essere stabile in cellule che sono stressate o morente a seguito di manipolazione sperimentale. Pertanto, è importante mantenere le cellule in vivo e sano come possibile durante la misurazione pHi. Diversi coloranti vitali sono disponibili che funzionano bene per misurare il pHi di cellule in cultura3, ma in molti casi essi non sono adatti per in vivo studi perché non penetrano il tessuto profondamente o uniformemente abbastanza per fornire misurazioni accurate .
Per aggirare il problema della penetrazione del colorante scadente, noi ed altri abbiamo usato una sonda codificata geneticamente, mCherry::pHluorin4,5,6,7, che può essere espressa in particolare nella cella tipi di interesse e imaged in tessuto vivo. pHluorin è una variante del GFP con un pKa più alto (~ 7.0 vs ~ 4.0) che si piega più facilmente a pH più alto; così l'intensità di fluorescenza totale emessa da una popolazione di molecole di pHluorin nella cella aumenta con l'aumentare di pHi8. D'importanza, la fluorescenza è lineare nel range di normalità citosolico di valori di pHi. In contrasto, la fluorescenza della mCherry (pKa ~ 4.5) è insensibile ai cambiamenti di pH all'interno della gamma citosolica. Questi due giornalisti sono covalentemente insieme come una singola proteina chimerica, codificata da un telaio di sola lettura aperta, in modo che sempre sono presenti in quantità uguali. Di conseguenza, il rapporto di pHluorin per l'intensità di fluorescenza mCherry fornisce una misura del pHi che è normalizzato per la concentrazione di sonda in ogni cella. I rapporti possono quindi essere convertiti alle stime dei valori di pHi utilizzando una curva standard che viene generata ottenendo il pHluorin ai rapporti mCherry da tessuti che sono stati equilibrati a valori di pH noto.
Qui, descriviamo i metodi per l'utilizzo di mCherry::pHluorin per misurare il pHi del lignaggio FSC epiteliale nell'ovaia di Drosophila . Questo tessuto ben caratterizzato è stato utilizzato per modellare i diversi aspetti della biologia, epiteliale, quali cellule staminali auto-rinnovamento e differenziazione9,10,11, migrazione cellulare collettiva12 , lo sviluppo e la manutenzione del cellulare polarità13,14. L'epitelio del follicolo è prodotto da due FSCs che risiedono al bordo anteriore del tessuto in una struttura denominata il germarium15,16. Queste cellule si dividono regolarmente durante l'età adulta di auto-rinnovarsi e produrre prole, chiamate cellule prefollicle (PFC), che possono immettere nuovamente la nicchia e diventare un FSC o differenziarsi in uno dei tre tipi di cellule del follicolo diversi: polare cellule, cellule di gambo, o cellule del follicolo di corpo principale. Abbiamo indicato precedentemente che nel tessuto wildtype, il pHi aumenta costantemente durante le prime fasi di differenziazione, da un pHi di circa 6.8 in FSCs a 7.0 in PFC, a 7,3 nel follicolo celle2. Bloccando questo aumento di RNAi atterramento di uno scambiatore sodio/protone espressa ubiquitariamente, DNhe2, severamente altera la differenziazione di pFC, considerando che l'aumento di pHi di sovraespressione di DNhe2 provoca una lieve differenziazione in eccesso fenotipo. Questi risultati dimostrano che pHi è mantenuto stabile nel lignaggio FSC precoce e che può essere sperimentalmente aumentata o diminuita in vivo. I metodi descritti qui possono essere utilizzati per misurare pHi in tessuto wildtype o varie forme di tessuti mutante, compreso atterramento di RNAi o sovraespressione utilizzando un Gal4 di interesse e cloni mitotici.
Protocollo
Nota: per misurare pHi del lignaggio di FSC, noi calcolare il rapporto di intensità di fluorescenza di pHluorin a mCherry in FSCs, PFC e le cellule del follicolo in condizioni fisiologiche e convertire i rapporti in valori di pHi con standard curve di calibrazione per ogni cella tipo 7. Primi, live imaging esperimenti sono effettuati per misurare l'intensità di fluorescenza di pHluorin e mCherry in germaria dissecato in un tampone contenente NaHCO 3, che simula condizioni fisiologiche 1 , 7. prossime, standard curve sono generate dalla misura pHluorin e mCherry intensità di fluorescenza in un Na +-libero, K + tampone contenente la Nigericina ionoforo regolato a due valori di pH diversi, 6.5 e 7.5. In presenza di Nigericina, pHi equilibra con il pH del tampone attraverso la membrana plasmatica, causando pHi abbinare il pH extracellulare. Infine, le curve standard vengono utilizzate per convertire pHluorin ai rapporti mCherry ai valori stimati pHi.
1. pre-Trial: pHi preparazione prima di misurare In Vivo
Nota: per misurare pHi in vivo, il transgene mCherry::pHluorin deve essere espresso nel tipo di cella di interesse. Di seguito sono alcuni dei modi comuni per generare transgenici pHluorin mosche del lignaggio di FSC. Generazione di cloni di mCherry::pHluorin è particolarmente utile per identificare FSCs, che si trovano sul bordo anteriore di un clone FSC. Espressione tessuto specifica mCherry::pHluorin è utile per misurare pHi attraverso l'intero tessuto ed è anche più conveniente quando si combinano con l'espressione di un RNAi o transgene.
- Voli della generazione di transgenici pHluorin
- mCherry::pHluorin-etichettati FSC cloni
- fare mCherry::pHluorin FSC cloni, attraversare UAS-mCherry::pHluorin 1 a un atto-Gal4 flipout o di altro magazzino con un Flp/FRT viscoelastica Gal4.
- Per rendere heatshock cloni inducibili, heatshock F1s adulti per 2 giorni post eclosion in flaconcini vuoti per 1 h a bagnomaria a 37 ° C, quattro volte circa ogni 12 h.
- Per garantire la normale crescita e tassi massimi di oogenesi durante questo periodo, mantenere mosche fornendo lievito fresco bagnato (parti approssimativamente uguali a secco baker ' s lievito e acqua) al giorno per almeno 6 giorni dopo l'induzione di clone.
- Espressione tessuto specifica mCherry::pHluorin
- Cross UAS-mCherry::pHluorin 1 a un follicolo cella driver specifico, y 1 w *; P {GawB} 10930/CyO (Bloomington ID: 7023).
- 3 giorni dopo mosche avviato schiusura, raccogliere e metterli in flaconcini contenenti lievito bagnato, per almeno 24 h prima della dissezione.
- mCherry::pHluorin-etichettati FSC cloni
- Rendendo le soluzioni
Nota: è importante preparare i buffer seguenti il giorno dell'esperimento, perché le concentrazioni di pH e soluto nel buffer possono cambiare nel tempo.- Preparare buffer bicarbonato, Nigericina e dissezione. Aggiungere i componenti nell'ordine elencato per evitare la formazione di precipitati (vedere tabella 1, tabella 2 e tabella 3).
2. Trial: Misurazione pHi del lignaggio di FSC
Nota: la dissezione, montaggio e dal vivo imaging passaggi per la soluzione tampone del bicarbonato e le due condizioni di buffer Nigericina potrebbe essere necessario essere eseguita due volte: una volta per determinare il microscopio impostazioni e una seconda volta per raccogliere i dati sperimentali. Vedere la sezione 2.2 qui sotto per ulteriori informazioni.
- Dissezione e montaggio
Nota: per i materiali necessari per eseguire questa operazione fare riferimento alla Figura 1 e Tabella materiali. Per le camere di montaggio stampato 3D, stampante 3D file vengono forniti come supplementare File 1 e 2 File supplementari.- Dissezione:
- chambers montaggio Prepare applicando un sottile strato di grasso per vuoto al lato più piatto del 3D stampato camera di montaggio. Collocare quel lato su un vetrino coprioggetti 22 x 40 mM per sigillare il coprioggetto alla camera montaggio.
- Sezionare le ovaie da mosche adulte femminile in 500 µ l di tampone di dissezione (buffer di bicarbonato o Nigericina, tabella 3)
- anestetizzare 3-4 mosche usando gas di CO 2 e trasferimento vola su un blocchetto di volare irrorato con CO 2 gas.
- Pick up una Mosca anestetizzata con forcipe e posto nel supporto di dissezione.
- Pizzicare il torace della Mosca con una pinza, mentre tirando sulla punta del suo addome, fino a quando le ovaie sono esposti.
- Dopo aver scollegato le ovaie da altri organi, prendono in giro a pezzi il ovarioles utilizzando 22 1/2 aghi di siringhe. Accuratamente separato il muscolo guaina da ovaie e ovarioles individuale separato dal cluster di ovarioles. Una tecnica efficace per isolare la guaina del muscolo è quello di utilizzare una siringa per tenere il cluster di ovarioles posto alla sua estremità anteriore e corsa verso il basso lungo la lunghezza del singolo ovarioles.
- Incubare le ovaie dissecate nei media di dissezione per esattamente 10 min prima di procedere con il passo di montaggio affinché ci sia tempo sufficiente per il pHi di cellule completamente equilibrare con il pH dei media dissezione Nigericina.
Nota: Assicurarsi che la guaina del muscolo viene rimosso dall'ovarioles. La guaina del muscolo può attenuare il segnale di fluorescenza, che lo rende difficile identificare singole celle utilizzando Concanavilin-A e può causare ovarioles muoversi spontaneamente durante la formazione immagine live.
- Di montaggio
- mettere una piccola goccia di media di dissezione sul vetrino coprioggetti all'interno della camera di montaggio.
- Trasferimento separati usando il forcipe ovarioles.
- Separare più tardi chambers di uovo di fase da germaria associato con gli alloggiamenti di uovo fase precedente e cercare di garantire che il germaria dissecata sono collocati al centro della goccia media dissezione.
- Aggiungere due piccole gocce di smalto ai lati opposti di un tondo 12mm vetrino coprioggetto e lasciarlo asciugare per circa 10 s.
- Posto al turno coprioggetto sul drop di file multimediali di dissezione contenenti separati germaria tale che quel lato con smalto rivolto verso il basso e contatta il supporto di dissezione. Premere verso il basso sulle parti del bordo con smalto per unghie per appiattire e garantire la germaria in posizione. Si consiglia di utilizzare il vecchio smalto perché esso strisci meno sopra la superficie del coprivetrino come è stato fissato in luogo.
- Riempire la camera di montaggio con supporto di ulteriori dissezione. Buffer specifico a condizione che non contenga concanavalina-un colorante può essere utilizzato per riempire la camera.
Nota: Il tempo totale trascorso da dissezione e montaggio non deve superare i 15 min. Questo è importante per ridurre al minimo gli effetti del danno tissutale e morte cellulare.
- Dissezione:
- Live imaging:
Nota: In questo passaggio, prima di raccogliere immagini dalla condizione di bicarbonato e le due condizioni di Nigericina per determinare le impostazioni di microscopio corretto; regolare le impostazioni di microscopio come necessario, e poi raccogliere immagini per i dati sperimentali. Utilizzare un microscopio confocale capace di imaging a 3 canali: GFP (475 eccitazione/509 emissione), mCherry (575 emissione di eccitazione/610) e da' (633 eccitazione/647 emissione).
Nota: Le impostazioni del microscopio Set: le intensità di pixel delle immagini raccolte saranno quantificate per determinare le stime di pHi, quindi è importante che i valori di intensità del segnale in ogni set di immagine è né troppo basso né saturo. La gamma di intensità che può essere catturata in un'immagine è definita dalla profondità di bit di immagine. In molti casi, immagini a 8 bit vengono generati per impostazione predefinita, ma si consiglia di acquisire i dati come immagini a 16 bit, che hanno una gamma dinamica più ampia. È importante garantire che la diafonia tra i canali fluorescente è ridotto al minimo, quindi le impostazioni dovrebbero essere regolate in modo che lo spettro di emissione raccolti da ogni fluoroforo non si sovrappongono. Per verificare queste impostazioni, prendere un'immagine di esempio utilizzando lo spettro di eccitazione per GFP e raccogliere nello spettro di emissione per mCherry e viceversa. Se le impostazioni sono state regolate correttamente, non ci dovrebbe essere nessun segnale in entrambi i casi. Impostare le impostazioni di microscopio (cioè, le impostazioni di tensione su una scansione laser confocale, laser potenza e pinhole dimensioni) affinché le intensità di pixel del segnale in tutti gli insiemi di tre immagini sono all'interno della gamma dinamica del file immagine. Per tutti i nostri esperimenti, abbiamo utilizzato un laser luce bianco microscopio confocale con un obiettivo 40x di acquisire immagini a 16 bit in 1.024 × 1.024 formato, mentre abbiamo ottimizzato il guadagno di tensione per ogni esperimento come necessario. Ad esempio, in un esperimento, abbiamo impostato il guadagno di tensione per GFP, mCherry e da' come 37,8% 72.9% e 260%, rispettivamente.- Immagine ovarioles nel buffer di dissezione Nigericina a pH 6,5 e regolare le impostazioni di tale intensità di pixel delle immagini pHluorin e mCherry sono basse, ma non sotto i limiti di rilevazione della fotocamera.
- Un altro set di ovarioles nel buffer di dissezione Nigericina a pH 7.5 di immagine e regolare le impostazioni in modo che le intensità di pixel delle immagini pHluorin e mCherry sono alti, ma non saturo. Ovaie di immagine
- nella dissezione di bicarbonato nel buffer e garantiscono che le intensità di pixel delle immagini pHluorin e mCherry non sono saturi con le impostazioni scelte. Se i pixel sono saturi, regolare le impostazioni come necessario.
Nota: Microscopio impostazione parametri possono differire basato sulla messa a punto esatta microscopio utilizzato e deve essere ottimizzata per ogni esperimento. Dopo aver impostate le impostazioni microscopio, non cambiare per il resto dell'esperimento. Le impostazioni dovrebbero essere coerenti tra controllo e condizioni sperimentali. Nei nostri esperimenti iniziali pHi, abbiamo generato le curve di calibrazione Nigericina punto 2 e 3 e abbiamo trovato alcuna differenza significativa tra pHi calcolata utilizzando entrambi i metodi. Tuttavia, in generale, l'aggiunta di ulteriori punti della curva di calibrazione dovrebbe migliorare l'accuratezza. Si consiglia pertanto, generando curve con un diverso numero di punti per determinare quanti sono necessari per un determinato insieme di condizioni sperimentali.
- Raccolta dei dati:
- eseguire la dissezione, montaggio e imaging dei campioni nelle condizioni di buffer bicarbonato e Nigericina. Dopo dissezione e montaggio, il tempo massimo trascorso un set di campioni di imaging non deve superare 45 min per garantire che le misure di intensità di fluorescenza sono realizzate in tessuto sano, live.
- Per ogni condizione, acquisizione di immagini per almeno 5 germaria.
3. Post-trial: Analisi delle immagini
- intensità di fluorescenza di misura in FSCs, PFC e le cellule del follicolo
- sfondo sottrazione:
- Open non trasformati immagini in FIJI con ogni canale in una finestra separata ( Figura 4).
- Utilizzare lo strumento rettangolo per disegnare un'area di interesse (ROI) nella finestra del canale pHluorin in una parte dell'immagine senza segnale. Passo
- opzionale: impostare il limite inferiore della soglia in modo che i pixel con valori di intensità sotto sfondo sono esclusi e impostare il limite massimo della soglia massima. Quando la soglia è impostata in questo modo, le aree dell'immagine senza segnale sono per lo più blue e il segnale è chiaramente visibile ( Figura 4A).
- Misura l'intensità media di fluorescenza nel ROI. Se la soglia è stata impostata nel passaggio 3, assicuratevi di controllare il " limite di soglia " casella nella finestra di dialogo impostare misure.
- Sottrarre l'intensità di fondo misurata da ogni canale e la fetta dell'immagine utilizzando la funzione Subtract, trovata nel processo → menu di Math.
- Ripetere passaggi 3.1.1.2-6 per la finestra del canale mCherry tranne che, nel passaggio 3.1.1.2, invece di disegnare un nuovo rettangolo con lo strumento rettangolo, utilizzare la funzione di selezione di ripristino, trovato nella finestra modifica → menu di selezione, per aggiungere un rettangolo con lo stesso dimensioni e posizione sull'immagine.
- Identificare FSCs, PFC e le cellule del follicolo:
- identificare il FSCs, PFC e le cellule del follicolo di morfologia e posizione utilizzando la macchiatura della concanavalina-A trovare limiti di cella ( Figura 2).
- FSCs sono sottili celle triangolari situate sul bordo del germarium al confine di regione 2a/2b. Se mCherry::pHluorin è espresso in un clone FSC, FSC può essere identificata anche come la maggior parte delle cella anteriore del clone.
- PFC sono cellule a forma irregolare nella regione 2b, immediatamente adiacente e a valle del FSCs.
- Le cellule del follicolo sono quadrate o colonnare cellule che circondano la ciste delle cellule di germe in regione 3.
- Ottenuto fluorescenza intensità quozienti
- scegliere una o più sezioni in cui la cella di interesse ha la più alta intensità di fluorescenza nel canale mCherry e assicurarsi che la concanavalina-A colorazione, visto nel canale da', sia nel foc noi all'interno della slice selezionata.
- ROI disegnare intorno ogni FSC e misura l'intensità media di fluorescenza di pHluorin e mCherry in tutte le fette selezionata per misurazioni.
- Dividere l'intensità media di fluorescenza del canale pHluorin dall'intensità media di fluorescenza del canale mCherry per calcolare un rapporto di pHluorin a fluorescenza mCherry.
- sfondo sottrazione:
- PHi derivare valori e generare immagini pseudocolored
- valori pHi Derive
- In tutti i casi, la curva di regressione lineare deve essere generata utilizzando ovaie da mosche dello stesso genotipo per entrambi sperimentale e condizioni di controllo. Generare una curva di regressione lineare per ogni tipo di cellula utilizzando i dati da due condizioni di buffer Nigericina ( Figura 3). In Excel, questo può essere fatto tracciare i dati in un grafico e aggiungendo una linea di tendenza lineare. In alternativa, vedere:
- uso la pendenza e l'intercetta y della curva di regressione lineare calcolate dalle condizioni Nigericina e l'equazione di una retta (y = mx + b) per convertire il pHluorin mCherry rapporto valori calcolati dai campioni nel bicarbonato condizione per valori di pH. In questa equazione, sostituire il modulo di pendenza (l'intercetta y) per b, immettere il valore rapporto per x e risolvere per y.
- Generare un'immagine pseudocolored che riflette i valori di rapporto in FIJI.
- Seguire la procedura descritta sopra per sottrazione di sfondo (passaggio 3.1.1 e Figura 4).
- Divide il canale di pHluorin dal canale mCherry utilizzando la funzione di immagine calcolatrice, trovata nel menu processo. Essere sicuro che il " finestra Crea nuovo " e " risultato a 32 bit (float) " caselle di controllo siano entrambe selezionate. Modificare la tabella di ricerca (LUT), trova nel menu immagine, termico o LUT di scelta. Vedere la Figura 4 per altre opzioni adatte.
- Nella finestra di dialogo Regola luminosità e contrasto (immagine → regolare → luminosità/contrasto), fare clic il " Set " pulsante. Set minimo visualizzato valore su 0 e il massimo valore visualizzato in un rapporto che meglio cattura i dati, solitamente 1.0-2.0 ( Figura 5).
- valori pHi Derive
Risultati
Qui abbiamo descritto il processo di misurazione pHi nell'epitelio del follicolo, che prevede diversi passaggi. In primo luogo, le ovaie vengono sezionate da mosche del genotipo appropriato utilizzando strumenti per dissezione e montaggio (Figura 1). Le ovarioles sono quindi Imaging utilizzando la microscopia a fluorescenza quantitativa e le immagini vengono analizzati per ottenere misurazioni di pHi. Per ogni immagine, i tipi di cellule di interesse vengono identificati come descritto nella sezione 3.1 (Figura 2). Il rapporto tra le intensità di fluorescenza nei canali GFP e mCherry viene convertito in valori di pHi utilizzando una curva standard (Figura 3A) come descritto nella sezione 3.2. Usando questo metodo, abbiamo trovato che pHi aumenta con differenziazione del lignaggio FSC precoce, da 6,8 a FSCs, a 7.0 in cellule di prefollicle, a 7,3 in cellule del follicolo (Figura 3B). I valori di pHi di ciascun tipo di cella possono essere rappresentati graficamente, come in Figura 3B, o come un pseudocolored microfotografia che mostra le differenze in pHluorin ai rapporti mCherry (Figura 4 e Figura 5). In queste immagini, differenze nel pHluorin mCherry rapporti vengono visualizzate come le differenze di colore, come definito da una LUT. È importante selezionare una LUT e valori massimi e minimi per la gamma di colori per produrre un'immagine che è più rappresentativo dei dati. Anche se, in generale, la scelta delle impostazioni di LUT e gamma non darà l'aspetto delle differenze che non sono presenti nell'immagine, una scelta meno adatta di LUTs può oscurare differenze pHi presente nella microfotografia (Figura 5).

Figura 1: Materiali per montaggio e dissezione della drosofila ovarioles. (A), un'immagine che mostra: (1) smalto; (2) vuoto grasso e Becher e dispensare punta utilizzata per l'applicazione di grasso; (3) 23 gauge siringa aghi; (4) Dumont Inox forcipe, dimensione 5; (5) 3D stampato camera di montaggio; (6) 22 x 40 mM vetrini coprioggetti; (7) rotonda vetrini coprioggetti, 12 mm di diametro, 0,13-0,16 mm di spessore; e (8) 9-Pozzo vetro piatto di dissezione. (B), un primo piano immagine della camera 3D montaggio stampato. (C) un'immagine che mostra una coppia dissecata delle ovaie wildtype. (D), un'immagine che mostra ben separati ovarioles. (E), A immagine della camera 3D con le ovaie dissecate montato sotto un coprioggetto in vetro rotondo. I punti neri sono gocce di smalto usato per tenere il coprioggetto rotondo sul posto. (F) un'immagine di dissecata ovarioles sotto un round vetrino coprioggetti dopo il montaggio. La scatola nera indica una regione dell'immagine con un singolo ovariole dissecata. Barre della scala rappresentano circa 500 µm. Clicca qui per visualizzare una versione più grande di questa figura.
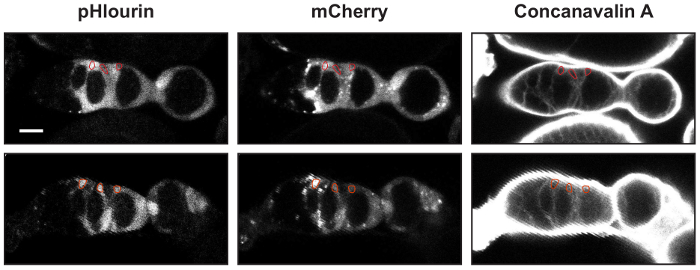
Figura 2: identificazione FSCs, PFC e le cellule del follicolo. Due esempi di germaria con UAS-mCherry::pHluorin e 10930-Gal4 macchiato con Concanavilin-A. Un ROI che delinea un FSC, un pFC e una cella del follicolo è mostrato in ogni immagine. Le intensità di fluorescenza media nei canali pHluorin e mCherry vengono utilizzate per calcolare la pHluorin ai rapporti mCherry. Barre della scala rappresentano circa 10 µm. Clicca qui per visualizzare una versione più grande di questa figura.
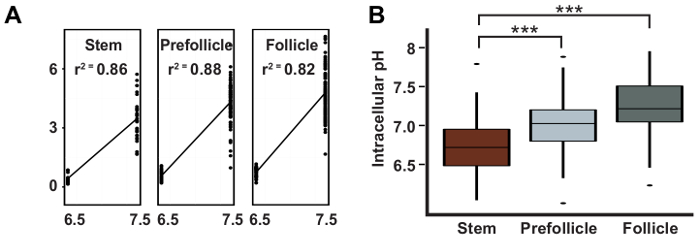
Figura 3: pHi aumenta durante la differenziazione del lignaggio di FSC. Risultati rappresentativi prelevati da Ulmschneider et al. 2 visualizzando: (A), una regressione lineare tipica curva utilizzata per calcolare i valori di pH da pHluorin a mCherry rapporti; e (B), il pHi calcolato valori con intervalli di confidenza del 95% per FSCs, PFC e le cellule del follicolo. Questa figura è stata adattata dopo l'autorizzazione, da Ulmschneider et al. 2. per favore clicca qui per visualizzare una versione più grande di questa figura.

Figura 4: utilizzando FIJI per generare un'immagine raziometrici pseudocolored. (A) immagini che mostrano i risultati di ogni passaggio nel processo di sottrazione di sfondo. A partire da un'immagine non trasformata (pannelli a sinistra), il primo passo è quello di impostare i limiti di soglia, in modo che i pixel con valori di intensità sotto sfondo sono esclusi. Il limite superiore della soglia è impostato al massimo. FIJI coloreranno i pixel esclusi blu, risultante in un'immagine con uno sfondo blu e un germarium chiaramente visibile (pannelli). Si noti che questo passaggio è facoltativo. Il secondo passo è disegnare un ROI in una parte dell'immagine senza misura di segnale (quadrati rossi in pannelli), l'intensità media di fluorescenza del ROI e sottrarre tale importo dall'intera immagine, risultante in un fondo sottratto immagine (pannelli di destra). Il terzo passo è quello di utilizzare la funzione di calcolo di immagine per dividere l'immagine nel canale pHluorin dall'immagine nel canale mCherry. Il risultato sarà un'immagine visualizzata con la tabella di ricerca di grigi e i valori di visualizzazione immagine impostata su abbracciano l'intera gamma dinamica fornita dalla profondità di bit dell'immagine. Il passo finale è quello di regolare le impostazioni di visualizzazione di immagini a un intervallo più appropriato e selezionare una tabella di ricerca. (B) visualizzare immagini di esempio che mostra il risultato di un calcolo di immagine con il minimo valore impostato su 0 e il valore di visualizzazione massimo impostato su 2.5 utilizzando quattro tabelle di ricerca diversi. Il rettangolo accanto a ogni immagine mostra i colori utilizzati in tutta la gamma dinamica per ogni tabella di ricerca. Si noti che per HiLo, 16 colori e termico, vengono utilizzati colori distinti per pixel con un valore di intensità pari a 0 o massimo (ad esempio, blu e rosso, rispettivamente, in HiLo). Questo fornisce un riferimento visivo facile dei limiti della gamma dinamica, consentendo allo spettatore di vedere che il segnale sia all'interno della gamma dinamica. (C) schermo colpo mostrando le opzioni selezionate durante l'importazione di un file in ImageJ utilizzando il plug-in importazione di Bio-Formats. barre della scala rappresentano 10 µm. Clicca qui per visualizzare una versione più grande di questa figura.
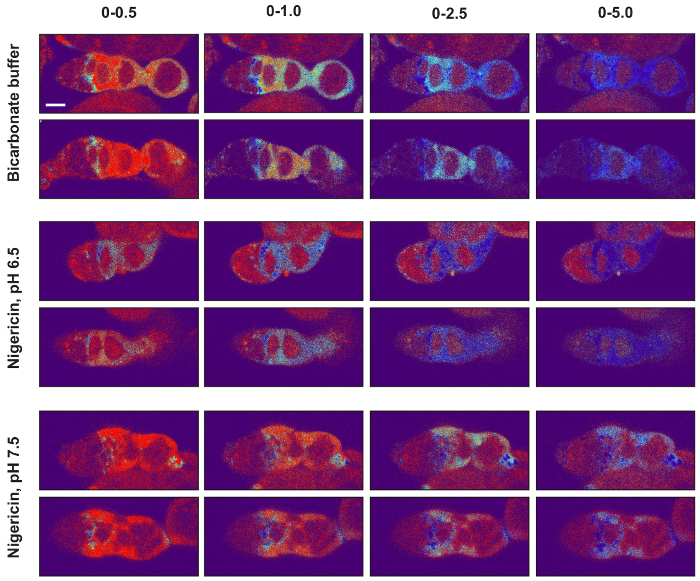
Figura 5: impostazione dell'immagine valori di visualizzazione delle immagini pseudocolored raziometrici. I valori di visualizzazione immagine minima e massima devono essere impostati in modo che il segnale nelle immagini dalla condizione di bicarbonato come pure da entrambe le condizioni Nigericina sono all'interno della gamma dinamica. Per illustrare questo punto, due immagini da ciascuna delle tre condizioni sono indicate con quattro impostazioni di visualizzazione immagine diversa (0-0.5, 0-1, 0-2.5 e 0-5) utilizzando la tabella di ricerca termica. Si noti che, per tutte le tre condizioni sperimentali, quando le immagini vengono visualizzate con il massimo valore visualizzato impostato su 0,5, gran parte del segnale è a o vicino a massimo sulla scala colorimetrica, e quando è impostata a 5.0, gran parte del segnale è a o vicino a minimo sul colore iMetric scala. In entrambi i casi, le differenze nel pHluorin mCherry rapporto attraverso il tessuto non possono essere facilmente apprezzate così queste impostazioni non sono ideali. Al contrario, valori di visualizzazione massima di 1.0 o 2.5 sono molto più appropriati. Con queste impostazioni, le differenze nei rapporti attraverso il tessuto possono essere facilmente apprezzate, e i segnali nelle immagini da tutte le tre condizioni sperimentali vengono visualizzati in colori che sono all'interno della gamma dinamica della scala colorimetrica. Barre della scala rappresentano circa 10 µm. Clicca qui per visualizzare una versione più grande di questa figura.
Discussione
Qui, descriviamo un metodo per misurare il pHi di cellule del lignaggio di FSC all'interno del tessuto wildtype. Questo protocollo è stato sviluppato e perfezionato negli ultimi cinque anni, da quando abbiamo iniziato a studiare pHi in tessuto ovarico di Drosophila . Durante quel tempo, il protocollo è stato utilizzato con successo da più ricercatori nel nostro laboratorio e su almeno quattro diversi filatura disc e microscopi a scansione laser. La riproducibilità della nostra osservazione originale, che pHi aumenti come cellule del lignaggio di FSC si differenziano dalla cellula formativa per il pFC allo stato della cellula del follicolo, attraverso queste prove multiple dimostra sia che questo fenomeno biologico è robusto e che la metodologia è affidabile. Tuttavia, nella nostra esperienza, questa è una procedura difficile da padroneggiare. Richiede attenzione ai dettagli in ogni fase e l'esecuzione rapida, altamente competente della dissezione, montaggio e passi di imaging. Come indicato nel protocollo, la procedura di montaggio e dissezione include un'incubazione di 10 min ma non deve superare i 15 min totale e la procedura di imaging dovrà essere completata in 45 min. In condizioni ideali, ovarioles di Drosophila possono essere mantenute in coltura per fino a 14 h17, ma abbiamo trovato che il tessuto comincia a morire molto più rapidamente nei buffer Nigericina utilizzato per generare le curve standard. Nostre linee guida per assicurare che tutti i dati vengono raccolti mentre il tessuto è ancora ben all'interno della finestra di tempo che sembra sano e morfologicamente normale. Questo non lascia molto tempo per ogni passo, però, quindi è importante pianificare in anticipo per assicurarsi che tutto è pronto per passare da una fase a quella successiva. In realtà, è molto più efficiente se due persone lavorano insieme sulla dissezione, montaggio, e procedure di imaging con una persona che esegue un passo mentre l'altra persona si prepara per il prossimo passo.
Poiché pHi possono essere sensibili alle manipolazioni sperimentali necessari all'immagine il tessuto ex vivo, quali temperatura e buffer di composizione, questo metodo è migliore per identificare le variazioni relative a pHi. Dato che la sonda è costituita da due distinte proteine fluorescenti, loro proprietà di fluorescenza come rendimento quantico, proprietà durata e pieghevole può essere influenzata in modo diverso in diversi tipi cellulari. Pertanto, è essenziale includere campioni trattati con le condizioni di buffer Nigericina e utilizzarli per generare una nuova curva di taratura per ogni tipo di cellula in ogni prova. Al fine di minimizzare ulteriori fonti di variabilità, è dato risalto a mantenere coerente tutte le condizioni durante il corso di formazione immagine. Campioni nelle condizioni di buffer Nigericina dovrebbero essere preparati e ripreso lo stesso giorno come quelli in condizione di buffer di bicarbonato e l'acquisizione immagine e preparazione di tutti i campioni dovrebbero essere tenuti più costanti possibile. Questo è importante perché le misurazioni di pHi sono basate su un confronto tra i set di immagine, così sperimentale differenze che riguardano la rilevazione della mCherry e pHluorin in uno o più dei set di immagine può diminuire la precisione delle stime pHi. Fattori quali modifiche alle impostazioni di tensione dei tubi di fotomoltiplicatore (se si utilizza un laser a scansione confocale) o tempi di esposizione (se si utilizza un disco che gira confocale) o una lente sporca che riduce la quantità di luce che raggiunge la fotocamera, influenzerà chiaramente i risultati . Ma ci sono una miriade di altri fattori più sottili che possono variare di giorno in giorno e potrebbe anche influenzare i risultati, ad esempio se le mosche sono ben nutrite, all'età di mosche, e quanto tempo i laser sono stati su prima di formazione immagine. Preparazione e formazione immagine di tutte le tre condizioni sopra lo stesso giorno minimizza queste differenze e quindi produrrà risultati più coerenti. In particolare, ogni volta che siamo passati a un nuovo microscopio o componenti del microscopio sono stati aggiornati, è stato necessario riottimizzare le impostazioni di nuove attrezzature. Ciò ha provocato spesso un cambiamento nei valori medio delle misurazioni individuali ma, finché nuove curve di calibrazione sono state generate con ogni prova, i cambiamenti nelle apparecchiature ha avuto un impatto minimo sulle stime di pHi. Inoltre, il confronto pertinente è spesso tra diversi tipi di cellule che sono all'interno del tessuto stesso (ad es., il FSCs, cellule del follicolo e PFC) e così sono internamente controllato per variazioni sperimentali.
Anche se questo protocollo si è concentrata su pHi del wildtype tessuto di misura, la metodologia è compatibile con metodi di Drosophila standard per la manipolazione di espressione genica, come espressione di RNAi o un transgene Gal4/UAS e analisi di mosaico. Poiché mCherry::pHluorin è guidato da un promotore di UAS, sarà sempre co-espresso con la RNAi o il transgene e MARCM18 può essere utilizzata per generare cloni omozigoti mutanti con mCherry::pHluorin come l'indicatore clonale. Per i motivi descritti sopra, wildtype tessuto dovrebbe essere analizzato come un controllo accanto a ogni prova con un genotipo mutante. Infine, i principi generali descritti qui possono essere applicati all'uso di mCherry::pHluorin per misurare pHi in altri tessuti. Infatti, questo protocollo è stato adattato da un protocollo per l'utilizzo di mCherry::pHluorin per misurare pHi in Drosophila occhio7, e abbiamo usato la metodologia simile per mCherry::pHluorin nel cervello larvale di immagine. Nel complesso, gli strumenti e i metodi descritti qui forniscono nuove opportunità per indagare le molte diverse funzioni di pHi in vivo all'interno del tessuto vivo, intatto.
Divulgazioni
Gli autori non hanno nulla a rivelare.
Riconoscimenti
Ringraziamo Bryne Ulmschneider per i contributi per il protocollo e Diane Barber per suggerimenti sul manoscritto. Quest'opera è stata finanziata da un Istituto nazionale di sovvenzione di salute GM116384 a T.G. Nystul e D.L. Barber.
Materiali
| Name | Company | Catalog Number | Comments |
| Fly Stocks | |||
| UAS-mCherry::pHluorin[1] | |||
| y1 w*;P{GawB}10930/CyO | Bloomington Stock Center | 7023 | |
| Act-Gal4 flipout stock | Bloomington Stock Center | 4409 | |
| Name | Company | Catalog Number | Comments |
| Chemicals for Buffer preparation | |||
| NaCl | Sigma Aldrich | S5886 | |
| KCl | Sigma Aldrich | P-3911 | |
| glucose | Mallinckrodt | 4912 | |
| HEPES | Thermo Fisher Scientific | BP310 | |
| MgSO4 | Thermo Fisher Scientific | M63 | |
| CaCl2 | Sigma Aldrich | C-5080 | |
| HCO3 | Sigma Aldrich | S-5761 | |
| MgCl2 | Sigma Aldrich | M-9272 | |
| NMDG+ | Sigma Aldrich | M-2004 | |
| K2HPO4 | Mallinckrodt | 7088 | Use to Make KHPO4 pH 7.4 |
| KH2PO4 | Thermo Fisher Scientific | BP362 | Use to Make KHPO4 pH 7.4 |
| Concanavalin A, Alexa Fluor 647 Conjugate | Thermo Fisher Scientific | C21421 | 0.25 mg/ml dilution |
| Nigericin | Thermo Fisher Scientific | N1495 | |
| Name | Company | Catalog Number | Comments |
| Dissection and mounting tools | |||
| 2 Dumont Inox forceps (Size 5) | Thermo Fisher Scientific | NC9473431 | |
| 2 23-gauge syringe needles | Sigma Aldrich | Z192457 | |
| 9-well glass dissecting dish | Thermo Fisher Scientific | 13-748B | |
| Vacuum Grease | Dow Corning | 1018817 | |
| 22 X 40 mM glass coverslips | Thermo Fisher Scientific | 12545C | |
| Round Glass Coverslips, 12mm diameter, 0.13-0.16mm thickness | Ted Pella, Inc. | 26023 | |
| 3-D mounting chamber | custom manufactured | .stl and .ipt files for 3-D printer included as supplemental files | |
| Name | Company | Catalog Number | Comments |
| Other equipment | |||
| pH meter | Thermo Fisher Scientific | 13-620-183A | Model: Accumet AB15 |
| Dissection microscope | Olympus Corporation | 0H11436 | Model: SZ2-ST |
| Confocal Microscope | Leica Biosystems | SP5 or SP8 laser-scanning confocal microscope with a 40× objective with a numerical aperture of 1.3 |
Riferimenti
- Grillo-Hill, B. K., Choi, C., Jimenez-Vidal, M., Barber, D. L. Increased H+ efflux is sufficient to induce dysplasia and necessary for viability with oncogene expression. eLife. 03270, (2015).
- Ulmschneider, B., Grillo-Hill, B. K., Benitez, M., Azimova, D. R., Barber, D. L., Nystul, T. G. Increased intracellular pH is necessary for adult epithelial and embryonic stem cell differentiation. J. Cell Biol. 215 (3), 345-355 (2016).
- Han, J., Burgess, K. Fluorescent Indicators for Intracellular pH. Chemical reviews. 110 (5), 2709-2728 (2010).
- Koivusalo, M., Welch, C., et al. Amiloride inhibits macropinocytosis by lowering submembranous pH and preventing Rac1 and Cdc42 signaling. J. Cell Biol. 188 (4), 547-563 (2010).
- Choi, C. -. H., Webb, B. A., Chimenti, M. S., Jacobson, M. P., Barber, D. L. pH sensing by FAK-His58 regulates focal adhesion remodeling. J. Cell Biology. 202 (6), 849-859 (2013).
- Rossano, A. J., Chouhan, A. K., Macleod, G. T. Genetically encoded pH-indicators reveal activity-dependent cytosolic acidification of Drosophila motor nerve termini in vivo. J. Phys. 591 (7), 1691-1706 (2013).
- Grillo-Hill, B. K., Webb, B. A., Barber, D. L. Ratiometric imaging of pH probes. Methods in Cell Biol. 123, 429-448 (2014).
- Miesenböck, G., De Angelis, D. A., Rothman, J. E. Visualizing secretion and synaptic transmission with pH-sensitive green fluorescent proteins. Nature. 394 (6689), 192-195 (1998).
- Kirilly, D., Xie, T. The Drosophila ovary: an active stem cell community. Cell research. 17 (1), 15-25 (2007).
- Sahai-Hernandez, P., Castanieto, A., Nystul, T. G. Drosophila models of epithelial stem cells and their niches. Wiley interdisciplinary reviews. Dev. Biol. 1 (3), 447-457 (2012).
- Losick, V. P., Morris, L. X., Fox, D. T., Spradling, A. Drosophila stem cell niches: a decade of discovery suggests a unified view of stem cell regulation. Dev. Cell. 21 (1), 159-171 (2011).
- Pocha, S. M., Montell, D. J. Cellular and molecular mechanisms of single and collective cell migrations in Drosophila: themes and variations. Ann Rev. of Gen. 48, 295-318 (2014).
- St Johnston, D., Ahringer, J. Cell polarity in eggs and epithelia: parallels and diversity. Cell. 141 (5), 757-774 (2010).
- Castanieto, A., Johnston, M. J., Nystul, T. G. EGFR signaling promotes self-renewal through the establishment of cell polarity in Drosophila follicle stem cells. eLife. 3, (2014).
- Margolis, J., Spradling, A. Identification and behavior of epithelial stem cells in the Drosophila ovary. Development. 121 (11), 3797-3807 (1995).
- Nystul, T. G., Spradling, A. An epithelial niche in the Drosophila ovary undergoes long-range stem cell replacement. Cell stem cell. 1 (3), 277-285 (2007).
- Morris, L. X., Spradling, A. C. Long-term live imaging provides new insight into stem cell regulation and germline-soma coordination in the Drosophila ovary. Development. 138 (11), 2207-2215 (2011).
- Lee, T., Luo, L. Mosaic analysis with a repressible cell marker (MARCM) for Drosophila neural development. Trends in neuro. 24 (5), 251-254 (2001).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon