Method Article
Métodos para el pH intracelular de la proyección de imagen del linaje de la célula de vástago del folículo en vivo tejido ovárico de Drosophila
En este artículo
Resumen
Proporcionamos un protocolo para pH intracelular de un linaje de células epiteliales en el tejido ovárico de Drosophila en la proyección de imagen. Se describen métodos para generar moscas transgénicas expresando un biosensor pH, mCherry::pHluorin, imagen el biosensor usando proyección de imagen de fluorescencia cuantitativa, generar curvas estándar y convertir valores de intensidad de fluorescencia a valores de pH.
Resumen
Cambios en el pH intracelular (pHi) desempeñan papeles importantes en la regulación de muchas funciones celulares, incluyendo metabolismo, proliferación y diferenciación. Por lo general, dinámica de pHi se determina en las células cultivadas, que son susceptibles de medición y la manipulación experimental de pHi. Sin embargo, el reciente desarrollo de nuevas herramientas y metodologías ha permitido estudiar dinámica pHi dentro del tejido intacto, vivo. Para la investigación de Drosophila , un desarrollo importante era la generación de una línea transgénica con un biosensor de pHi, mCherry::pHluorin. Aquí, describimos un protocolo que habitualmente utilizamos para proyección de imagen en ovariolas de Drosophila para medir pHi en el linaje de la célula de vástago (FSC) epitelial del folículo en líneas transgénicas tipo salvaje de mCherry::pHluorin; sin embargo, los métodos aquí descritos pueden ser fácilmente adaptados para otros tejidos, incluyendo los discos de ala y el epitelio del ojo. Se describen técnicas para expresar mCherry::pHluorin en el linaje FSC, manteniendo el tejido ovárico durante la proyección de imagen, vivo y adquirir y analizar imágenes para obtener valores de pHi.
Introducción
Estudios recientes revelaron un papel para cambios en pHi durante celular diferenciación y displasia en vivo1,2. Estos estudios encontraron que pHi es notablemente consistente en células del mismo tipo en la misma etapa de diferenciación, pero que cambia como las células de transición de una etapa a otra. En algunos casos, bloqueando parcialmente los cambios en pHi altera la diferenciación, lo que sugiere que el cambio en pHi no es sólo una consecuencia de los cambios de destino de la célula pero en cambio ayuda a promover el cambio de destino de la célula, quizás a través de efectos sobre el pH-sensible a la regulación las proteínas o las reacciones químicas necesarias para la diferenciación. Los estudios futuros tienen el potencial para revelar más información sobre las muchas funciones diferentes del pHi dinámica en vivo. Sin embargo, uno de los desafíos de estudiar la pHi durante la diferenciación en vivo es la obtención de medidas precisas de pHi. A diferencia de otras características de diferenciación, tales como cambios en la morfología celular y expresión génica, pHi es una propiedad química lábil de la célula que no se conserva en las células que se han fijado y permeabilized con métodos estándar. Además, pHi puede no ser estable en las células que están estresadas o muere como resultado de la manipulación experimental. Por lo tanto, es importante mantener las células vivas y tan saludable como sea posible cuando mide pHi. Varios colorantes vitales están disponibles que funcionan bien para medir PI de las células en cultura3, pero en muchos casos no son adecuadas para en vivo los estudios porque no penetran el tejido uniformemente o profundamente bastante para proporcionar mediciones precisas .
Para evitar el problema de la penetración de tinte pobre, nosotros y otros hemos utilizado una sonda genéticamente codificada, mCherry::pHluorin4,5,6,7, que puede ser expresado específicamente en la célula tipos de interés y reflejada en tejido vivo. pHluorin es una variante de la GFP con un pKa superior (~ 7,0 vs ~ 4.0) que dobla más fácilmente a pH más alto; así que la intensidad de fluorescencia total emitida por una población de pHluorin de moléculas en la célula aumenta con el aumento del pHi8. Lo importante, la fluorescencia es lineal dentro del rango normal citosólico de los valores de la pHi. En contraste, la fluorescencia de mCherry (pKa ~ 4.5) es insensible a los cambios en el pH dentro de la gama citosólica. Estos dos periodistas son covalente juntos como una sola proteína quimérica, codificada por un marco abierto de lectura único, por lo que siempre están presentes en cantidades iguales. Por lo tanto, el de pHluorin a la intensidad de la fluorescencia mCherry proporciona una medida del pHi que es normalizada a la concentración de la sonda en cada célula. Los cocientes se pueden convertir entonces las estimaciones de los valores de la pHi usando una curva estándar generada por obtener la pHluorin mCherry cocientes de los tejidos que han sido equilibrados a valores de pH conocido.
Aquí, describimos los métodos para el uso de mCherry::pHluorin para medir la pHi del linaje epitelial de la FSC en el ovario del Drosophila . Este tejido bien caracterizado se ha utilizado para modelar diferentes aspectos de la biología epitelial, tales como la célula de vástago auto renovación y diferenciación9,10,11, migración de la célula colectiva12 , el desarrollo y mantenimiento de celulares polaridad13,14. El epitelio del folículo se produce por dos remiten que residen en el borde anterior de los tejidos en una estructura llamada el germarium15,16. Estas células se dividen regularmente durante la edad adulta para la autorrenovación y producir progenie, denominadas células prefollicle (PFC), que pueden volver a entrar en el nicho y convertirse en un FSC o diferenciar en uno de los tres tipos de células del folículo diferentes: las células polares, las células del tallo, o células del folículo del cuerpo principal. Demostramos previamente que en el tejido de tipo salvaje, el PI aumenta constantemente durante las primeras etapas de diferenciación, de una pHi de aproximadamente 6,8 en remiten a 7.0 en PFC, 7.3 en folículo células2. Bloqueo este aumento por la caída de ARNi de un intercambiador sodio/protones ubicuo expresada, DNhe2, severamente deteriora diferenciación de pFC, considerando que el aumento de pHi por la sobreexpresión de DNhe2 causa una leve diferenciación de exceso fenotipo. Estos resultados demuestran que el pHi se mantiene estable en el linaje FSC temprano y que pueden ser experimentalmente aumentado o había disminuido en vivo. Los métodos descritos aquí pueden utilizarse para medir pHi en tejido tipo salvaje o diversas formas de tejidos mutantes, incluyendo caída de ARNi o sobreexpresión usando Gal4 de interés y los clones mitotic.
Protocolo
Nota: para medir la pHi en el linaje FSC, calcular la relación de intensidades de fluorescencia de pHluorin a mCherry en Eve, PFC y las células del folículo en condiciones fisiológicas y convertir la relación en valores de pHi con estándar curvas de calibración para cada célula tipo 7. En primer lugar, energizados experimentos de imagen se realizan para medir intensidades de fluorescencia de pHluorin y mCherry en germaria disecado en un tampón que contiene NaHCO 3, que imita las condiciones fisiológicas 1 , 7. curvas estándar siguiente se generan mediante la medición de pHluorin y mCherry intensidades de fluorescencia en una Na +-K + buffer que contiene el ionóforo nigericin libre, ajustado a dos valores de pH diferentes, 6.5 y 7.5. En presencia de nigericin, pHi se equilibra con el pH del buffer a través de la membrana plasmática, provocando pHi para que coincida con el pH extracelular. Por último, las curvas estándar se utilizan para convertir pHluorin a mCherry proporciones pHi Estimado valores.
1. cuestiones preliminares: pHi de preparación antes de la medición En Vivo
Nota: para medir pHi en vivo, el transgen mCherry::pHluorin se expresará en el tipo de la célula de interés. A continuación se muestran algunas maneras comunes para generar transgénico pHluorin vuela en el linaje FSC. Generando clones de mCherry::pHluorin es particularmente útil para identificar remiten, que se encuentra en el borde anterior de un clon FSC. Tejido específico mCherry::pHluorin expresión es útil para la medición de pHi en el tejido entero y también es más conveniente cuando se combina con la expresión de un ARNi o transgén.
- Vuela generación de transgénico pHluorin
- mCherry::pHluorin-con la etiqueta FSC clones
- hacer mCherry::pHluorin FSC clones, Cruz UAS-mCherry::pHluorin 1 a un acto-Gal4 flipout stock u otra la acción con un Gal4 inducible Flp/FRT.
- Para hacer heatshock clones inducibles, heatshock adultos F1s durante 2 días post eclosión en frascos vacíos para 1 h en un baño de agua de 37 ° C, cuatro veces aproximadamente cada 12 h.
- Para asegurar el crecimiento normal y tasas máximas de ovogénesis durante este período, mantener moscas proporcionando levadura húmeda fresca (aproximadamente partes iguales seco baker ' s levadura y agua) diariamente durante al menos 6 días después de la inducción clon.
- Expresión de tejido específico mCherry::pHluorin
- Cruz mCherry::pHluorin UAS 1 a un folículo celular driver específico, y 1 w *; P {GawB} 10930/CyO (Bloomington ID: 7023).
- 3 días después de moscas eclosing, recoger y colocar en frascos que contienen levadura húmeda, durante al menos 24 h antes de la disección.
- mCherry::pHluorin-con la etiqueta FSC clones
- Haciendo soluciones
Nota: es importante preparar los siguientes buffers en el día del experimento porque las concentraciones en el buffer de pH y soluto pueden cambiar con el tiempo.- Búferes de bicarbonato, nigericin y la disección de preparar. Añadir los componentes en el orden listados para evitar la formación de precipitados (consulte la tabla 1, tabla 2 y tabla 3).
2. Prueba: Medición de pHi en el linaje de FSC
Nota: la disección, montaje y vivo la proyección de imagen pasos para el buffer de bicarbonato y las dos condiciones de buffer nigericin deba ser realizado dos veces: una vez para determinar el microscopio ajustes y una segunda vez para recoger los datos experimentales. Vea la sección 2.2 a continuación para obtener más información.
- Disección y montaje
Nota: para materiales necesarios para realizar este paso, consulte figura 1 y Tabla de materiales. Cámaras 3D montaje impresos, archivos de impresora 3D se proporcionan como suplementario archivo 1 y archivo adicional 2.- Disección:
- preparar salas de montaje, aplicando una capa delgada de grasa para vacío en el lado más plano de la 3D impreso cámara de montaje. Coloque ese lado en un cubreobjetos de 22 x 40 mM para sellar el cubreobjetos a la sala de montaje.
- Diseccionar los ovarios de las moscas adultas en 500 μl de tampón de disección (tampón de bicarbonato o nigericin, tabla 3)
- anestesiar a moscas de 3-4 utilizando gas de CO 2 y transferencia vuela sobre una almohadilla mosca perfundido con CO 2 gas.
- Recoger una mosca anestesia con lugar en los medios de comunicación de disección y pinzas.
- Pellizcar el tórax de la mosca con una pinza, mientras tirando de la punta de su abdomen hasta que sus ovarios son.
- Después de extraer los ovarios de los otros órganos, embromar aparte las ovariolas utilizando una aguja de jeringa de 1/2 22. Separe cuidadosamente el músculo de la envoltura de los ovarios y ovariolas individual separado de la agrupación de ovariolas. Una técnica eficaz para aislar la vaina del músculo es utilizar una aguja de jeringa para mantener el racimo de ovariolas en su extremo anterior y movimiento hacia abajo a lo largo de ovariolas solo.
- Incubar los ovarios disecados en medios de disección durante exactamente 10 minutos antes de proceder con el paso de montaje por lo que hay tiempo suficiente para que el PI de las células para equilibrar totalmente con el pH de los medios de comunicación de disección nigericin.
Nota: Asegúrese de que se extrae la vaina del músculo de las ovariolas. La vaina del músculo puede atenuar la señal de fluorescencia, lo que dificulta identificar células individuales con Concanavilin-A y puede causar ovariolas moverse espontáneamente durante la proyección de imagen vivo.
- De montaje
- Coloque una gota pequeña de los medios de disección sobre el cubreobjetos de vidrio dentro de la cámara de montaje.
- Transferencia separada ovariolas usando fórceps.
- Separar posteriormente etapa huevo de chambers germaria asociado con cámaras de huevo de etapas anteriores y tratar de asegurar que el germaria disecada se colocan en el centro de la gota de medio de disección.
- Añadir dos pequeñas gotas de esmalte de uñas a lados opuestos de un cubreobjetos redondo de 12 mm y dejar aire seco durante unos 10 s.
- Lugar la ronda cubreobjetos sobre la gota de medio de disección que contiene separan germaria tal que el lado con esmalte de uñas hacia abajo y entra en contacto con los medios de comunicación de la disección. Presione las piezas del borde con esmalte de uñas para aplanar y asegurar la germaria en posición. Se recomienda utilizar esmalte de uñas mayores porque mancha menos, sobre la superficie del cubreobjetos, como se asegura en lugar.
- Llene la cámara de montaje con medios de disección adicional. Búfer específico de condición que contienen tinte concanavalina-A se puede utilizar para llenar la cámara de.
Nota: El tiempo total gastado de disección y montaje no debe exceder 15 minutos. Esto es importante para minimizar los efectos de daño de tejido y muerte celular.
- Disección:
- Imagen en:
Nota: en este paso, primero recoger imágenes de la condición de bicarbonato y las dos condiciones de nigericin para determinar la configuración del microscopio adecuado, ajustar el microscopio según sea necesario, y luego recoger imágenes de los datos experimentales. Usar un microscopio confocal de imágenes en 3 canales: GFP (emisión 475 excitación/509), mCherry (575 emisión excitación/610) y far-red (emisión excitación/647 633).
Nota: Set Microscopio configuración: las intensidades de los píxeles de las imágenes recogidas se cuantifican para determinar las estimaciones de pHi, por lo que es importante que los valores de intensidad de la señal en cada juego de la imagen es demasiado baja ni saturado. La gama de intensidades que puede capturar en una imagen se define por la profundidad de bits de imagen. En muchos casos, se generan imágenes de 8 bits por defecto, pero se recomienda adquirir los datos de imágenes de 16 bits, que tienen un rango dinámico más amplio. Es importante asegurarse de que se reduce al mínimo la interferencia entre canales fluorescentes, por lo que la configuración debe ajustarse de modo que el espectro de emisión obtenidos cada fluoróforo no se superponen. Para probar estas configuraciones, tomar una imagen de muestra utilizando el espectro de excitación de GFP y recoger en el espectro de emisión para mCherry y viceversa. Si los valores han sido ajustados correctamente, debe ser no hay señal en cualquiera de los casos. Establecer la configuración del microscopio (es decir, configuración de voltaje de un sistema de escaneo láser confocal, laser power y del agujero de alfiler de tamaño) para que las intensidades de los píxeles de la señal en todos los conjuntos de tres imágenes están dentro de la gama dinámica del archivo de imagen. Para todos nuestros experimentos, se utilizó un blanco luz láser confocal microscopio con un objetivo 40 x para la adquisición de imágenes de 16 bits en formato de 1.024 × 1.024, mientras nos habían optimizado ganancia de voltaje para cada experimento, según sea necesario. Por ejemplo, en un experimento, establece la ganancia de voltaje para GFP, mCherry y far-red como 37,8% 72,9% y 260%, respectivamente.- Imagen ovariolas en buffer de disección nigericin a pH 6.5 y ajustar la configuración tal que intensidades de píxeles de las imágenes pHluorin y mCherry son bajos, pero no debajo de los límites de detección de la cámara de.
- Otro conjunto de ovariolas en buffer de disección nigericin a pH 7.5 la imagen y ajustar la configuración de tal manera que las intensidades de los píxeles de las imágenes pHluorin y mCherry están alta, pero no saturado.
- Imagen ovarios en disección de bicarbonato del almacenador intermediario y aseguran de que las intensidades de los píxeles de las imágenes pHluorin y mCherry no están saturadas con las opciones solicitadas. Si los píxeles están saturados, ajuste la configuración según sea necesario.
Nota: Microscopio parámetros de configuración pueden variar basado en la configuración del microscopio exacta se utiliza y debe optimizarse para cada experimento. Después de establecer los ajustes del microscopio, no los cambian para el resto del experimento. Ajustes deben ser consistentes a través de control y condiciones experimentales. En nuestros experimentos iniciales pHi, genera las curvas de calibración de punto nigericin 2 y 3 y no encontró diferencias significativas entre pHi calculado utilizando cualquiera de los métodos. Sin embargo, en general, la adición de más puntos en la curva de calibración se espera mejorar la precisión. Por lo tanto, se recomienda generar curvas con diferentes números de puntos para determinar cuántos son necesarios para un determinado conjunto de condiciones experimentales de.
- Recolección de datos:
- realizar la disección, montaje y proyección de imagen de las muestras en las condiciones de buffer bicarbonato y nigericin. Después de la disección y montaje, el tiempo máximo de un conjunto de muestras de la proyección de imagen no debe exceder 45 minutos para asegurar que se realicen las mediciones de intensidad de fluorescencia en el tejido vivo, sano.
- Para cada condición, adquirir imágenes de al menos 5 germaria.
3. Juicio Posterior: Análisis de imágenes
- intensidades de fluorescencia medida en Eve, PFC y las células del folículo
- fondo resta:
- abierto sin procesar imágenes en FIJI con cada canal en una ventana separada ( figura 4).
- Utilice la herramienta Rectángulo para dibujar un área de interés (ROI) en la ventana del canal pHluorin en una parte de la imagen sin señal. Paso de
- opcional: establece el límite inferior del umbral para que los píxeles con valores de intensidad por debajo del fondo están excluidos y establecer el límite superior del umbral máximo. Cuando el umbral se establece de esta manera, las áreas de la imagen sin señal en su mayoría son de color azules y la señal es visible ( Figura 4A).
- Medir la intensidad de fluorescencia media en el ROI. Si el umbral se establece en el paso 3, asegúrese de comprobar la " límite umbral " en el cuadro de diálogo establecer las medidas de la caja.
- Restar la intensidad de fondo medido de cada canal y de la imagen utilizando la función de restar, en el proceso de → el menú Math.
- Repetir pasos 3.1.1.2-6 para la ventana del canal mCherry salvo que, en el paso 3.1.1.2, en lugar de dibujar un nuevo rectángulo con la herramienta Rectángulo, utilice la función de selección de restauración, en la edición → menú de selección, para añadir un rectángulo con la misma dimensiones y posición en la imagen.
- Identificar remiten, PFC y las células del folículo:
- identificar la Eve, los PFC y las células del folículo por la morfología y ubicación mediante la tinción de concanavalina-A encontrar límites de la celda ( figura 2).
- Eve es delgadas células triangulares situadas en el borde del germarium en la frontera de 2a/2b de la región. Si mCherry::pHluorin se expresa en un clon de la FSC, FSC también puede ser identificado como la célula más anterior del clon.
- PFC es células de forma irregular en la región 2b, inmediatamente adyacente a y de Eve.
- Las células del folículo son células cuadradas o columnares que rodean el quiste de la célula de germen en la región 3.
- Obtención de ratios de intensidad de fluorescencia
- elegir uno o más sectores en el que la célula de interés tiene mayor intensidad de fluorescencia en el canal mCherry y asegúrese de que la tinción de concanavalina-A, visto en el canal far-red, en foc nosotros dentro de la división seleccionada.
- ROI dibujar alrededor de cada FSC y medir la intensidad de fluorescencia media de pHluorin y mCherry en todos rebanadas solicitada para las medidas de.
- Dividir la intensidad de fluorescencia media del canal pHluorin por la intensidad de fluorescencia media del canal mCherry para calcular un cociente de pHluorin fluorescencia mCherry.
- fondo resta:
- PHi Derive valores y generar imágenes pseudocolored
- valores de pHi Derive
- en todos los casos, se debe generar la curva de regresión lineal con ovarios de moscas del mismo genotipo para ambos experimental y control de condiciones. Generar una curva de regresión lineal para cada tipo de célula con los datos de dos nigericin condiciones de buffer ( figura 3). En Excel, esto puede hacerse trazando los datos de un gráfico y añadiendo una línea de tendencia lineal. Alternativamente, vea:
- uso la pendiente y la intersección de la curva de regresión lineal calculadas a partir de las condiciones de nigericin y la ecuación de una línea (y = mx + b) convertir el pHluorin a valores del cociente mCherry calculada a partir de las muestras en el bicarbonato condición para valores de pH. En esta ecuación, b sustituir la forma de la pendiente (la intersección), poner el valor de la relación de x y resolver para y.
- Generar una imagen pseudocolored que refleja los valores del cociente en FIJI.
- Siga el procedimiento anterior para sustracción de fondo (paso 3.1.1 y figura 4).
- Divide el canal de pHluorin del canal mCherry utilizando la función calculadora de la imagen, en el menú de proceso. Asegúrese de que el " crear nueva ventana " y " resultado de 32 bits (float) " casillas de verificación se comprueban. Cambiar la tabla de búsqueda (LUT), encuentra en el menú de la imagen térmica o LUT de elección. Vea la figura 4 para otras opciones adecuadas.
- En el cuadro de diálogo ajustar brillo y contraste (imagen → ajuste → brillo/contraste), haga clic en el " Set " botón. Conjunto mínimo muestra valor a 0 y el máximo valor a una relación que mejor captura los datos, normalmente 1.0-2.0 ( figura 5).
- valores de pHi Derive
Resultados
Aquí hemos descrito el proceso de medición de pHi en el epitelio folicular, que implica varios pasos. En primer lugar, los ovarios son disecados de moscas del genotipo apropiado usando herramientas para disección y montaje (figura 1). Las ovariolas son entonces reflejadas mediante microscopía de fluorescencia cuantitativa y las imágenes se analizan para obtener medidas de pHi. Para cada imagen, se identifican los tipos de células de interés tal como se describe en la sección 3.1 (figura 2). La relación de las intensidades de fluorescencia en los canales de GFP y mCherry se convierte en valores de pHi usando una curva estándar (Figura 3A) como se describe en la sección 3.2. Usando este método, encontramos que su pHi aumenta con la diferenciación en el linaje principios FSC, de 6.8 en Eve, a 7.0 en células prefollicle, 7.3 en las células del folículo (figura 3B). Los valores de la pHi de cada tipo de célula se pueden representar gráficamente, como en la figura 3B, o como un pseudocolored micrografía que muestra las diferencias en pHluorin mCherry proporciones (figura 4 y figura 5). En estas imágenes, las diferencias en la pHluorin mCherry relaciones aparecen como diferencias en color, como se define en una LUT. Es importante seleccionar un LUT y valores máximo y mínimo para la gama de colores para producir una imagen que representa la mayoría de los datos. Aunque, en general, la opción de configuración de LUT y gama se da la aparición de diferencias que no están presentes en la imagen, una opción menos adecuada de LUTs puede ocultar diferencias de pHi en la micrografía (figura 5).

Figura 1: Material para disección y montaje Drosophila ovariolas. (A) una imagen que muestra: esmalte de uñas (1); (2) vacío grasa y vaso de precipitado y pipeta de punta utilizado para aplicar la grasa; (3) calibre 23 jeringa agujas; (4) pinzas Inox Dumont, tamaño 5; (5) cámara de montaje impreso 3D; (6) 22 x 40 mM cubreobjetos de vidrio; (7) redondo vidrio cubreobjetos, 12 mm de diámetro, 0.13-0.16 mm de espesor; y (8) 9-pozo cristal plato de disección. (B) un primer plano imagen de la cámara 3D montaje impreso. (C) una imagen que muestre un par disecado de ovarios de tipo salvaje. (D) una imagen mostrando ovariolas bien separados. (E) A imagen de la cámara 3D con ovarios disecados montados bajo un cubreobjetos de vidrio redondo. Los puntos negros son gotas de esmalte utilizado para fijar el cubreobjetos redondo. Cubreobjetos de vidrio (F) una imagen de ovariolas disecada debajo una ronda después del montaje. La caja negra indica una región de la imagen con un solo ovariole disecada. Barras de escala representan aproximadamente 500 μm. haga clic aquí para ver una versión más grande de esta figura.
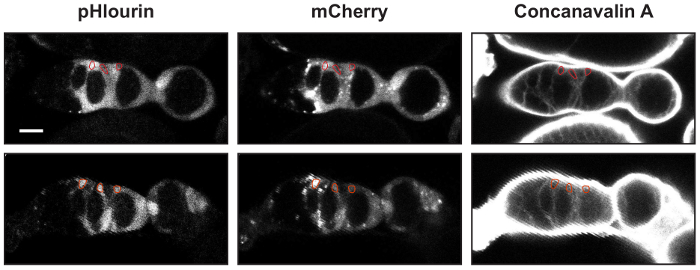
Figura 2: identificación de Eve, PFC y las células del folículo. Dos ejemplos de germaria con UAS-mCherry::pHluorin y 10930-Gal4 teñidas con Concanavilin-A. En cada imagen se muestra un ROI esbozando un FSC, un pFC y un celular del folículo. Las intensidades de fluorescencia media en los canales pHluorin y mCherry se utilizan para calcular el pHluorin mCherry cocientes. Barras de escala representan aproximadamente 10 μm. haga clic aquí para ver una versión más grande de esta figura.
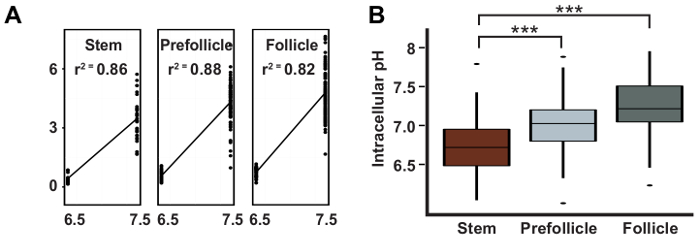
Figura 3: pHi aumenta durante la diferenciación en el linaje FSC. Resultados representativos de Ulmschneider et al. 2 demostración: (A) una regresión lineal típica curva utilizada para calcular los valores de pH de pHluorin mCherry proporciones; y (B) calcula PI con intervalos de confianza del 95% para Eve, PFC y las células del folículo. Esta figura se ha adaptado después del permiso de Ulmschneider et al. 2. haga clic aquí para ver una versión más grande de esta figura.

Figura 4: uso de FIJI para generar una imagen radiométrica pseudocolored. (A) imágenes que muestran los resultados de cada paso en el proceso de sustracción de fondo. A partir de una imagen sin procesar (paneles de la izquierda), el primer paso es establecer los límites de umbral para que excluyen a los píxeles con valores de intensidad por debajo del fondo. El límite superior del umbral se encuentra al máximo. FIJI color de los píxeles excluidos azul, dando por resultado una imagen con un fondo azul y un germarium claramente visible (paneles de mediados). Tenga en cuenta que este paso es opcional. El segundo paso es dibujar un ROI en una parte de la imagen sin señal (cuadrados rojos en mediados paneles), medida la intensidad de fluorescencia media del retorno de la inversión, y restar esa cantidad de toda la imagen, dando como resultado un fondo resta imagen (paneles de la derecha). El tercer paso es usar la función de cálculo de imagen para dividir la imagen en el canal de pHluorin por la imagen en el canal mCherry. El resultado será una imagen que se muestra con la tabla de búsqueda de grises y establecen los valores de la pantalla de imagen para abarcar toda la gama dinámica proporcionada por la profundidad de bits de la imagen. El paso final es ajustar la configuración de visualización de imagen a una gama más apropiada y seleccione una tabla de búsqueda. (B) muestra las imágenes mostrando el resultado de un cálculo de la imagen con el mínimo muestran valor a 0 y el valor máximo de la pantalla a 2.5 usando cuatro tablas de búsqueda diferentes. El rectángulo al lado de cada imagen muestra los colores utilizados en todo el rango dinámico para cada tabla de búsqueda. Observe que para HiLo, 16 colores y termal, diferentes colores se utilizan píxeles con un valor de intensidad de 0 o máximo (e.g., azul y rojo, respectivamente, en el HiLo). Esto proporciona una fácil referencia visual de los límites de la gama dinámica, permitiendo al espectador a ver que la señal está dentro de la gama dinámica. (C) pantalla tiro mostrando las opciones al importar un archivo en ImageJ mediante el complemento de la importación de formatos de Bio. barras de escala representan 10 μm. haga clic aquí para ver una versión más grande de esta figura.
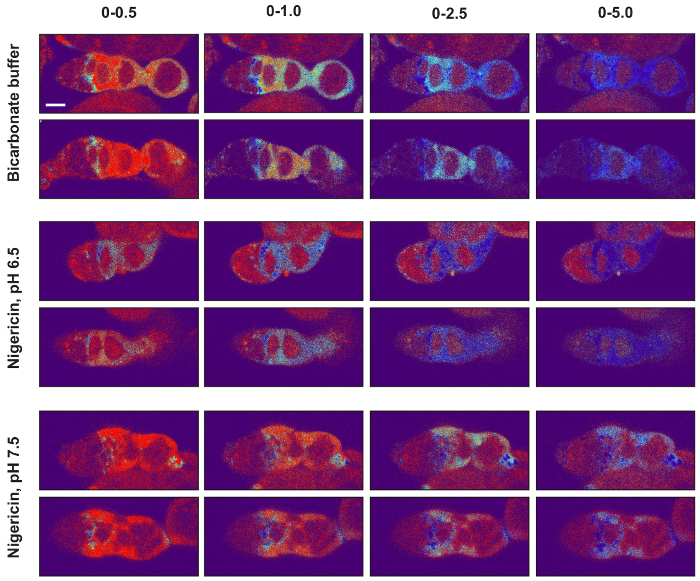
Figura 5: ajuste de la imagen pantalla valores de imágenes proporcionales pseudocolored. Los valores de mínimo y máximo de la imagen pantalla deben establecerse para que la señal en imágenes de la condición de bicarbonato, así como de ambas condiciones de nigericin están dentro de la gama dinámica. Para ilustrar este punto, se muestran dos imágenes de cada una de las tres condiciones con cuatro configuraciones de pantalla de imagen diferentes (0-0.5, 0-1, 0-2.5 y 0-5) utilizando la tabla de búsqueda térmica. Observe que, para todas las condiciones experimentales tres, cuando las imágenes se muestran con el conjunto de valor máximo visualizado a 0.5, gran parte de la señal está al máximo en la escala colorimétrica o cerca, y cuando se encuentra a 5.0, gran parte de la señal está en o cerca de mínimo en el color escala de imetric. En ambos casos, las diferencias en la pHluorin mCherry relación en el tejido no pueden fácilmente apreciarse por lo que estos valores no son ideales. Por el contrario, valores de pantalla máxima de 1.0 ó 2.5 son mucho más apropiados. Con estos ajustes, se pueden apreciar fácilmente las diferencias en las tasas a través de los tejidos, y las señales en las imágenes de todos tres condiciones experimentales se muestran en los colores que están dentro de la gama dinámica de la escala colorimétrica. Barras de escala representan aproximadamente 10 μm. haga clic aquí para ver una versión más grande de esta figura.
Discusión
Aquí, describimos un método para la medición de PI de las células en el linaje FSC dentro del tejido de tipo salvaje. Este protocolo ha sido desarrollado y refinado durante los últimos cinco años, desde que comenzó a estudiar pHi en Drosophila el tejido ovárico. Durante ese tiempo, el protocolo ha sido utilizado con éxito por varios investigadores en nuestro laboratorio y por lo menos cuatro diferentes spinning disco y microscopios de escaneo láser. La reproducibilidad de nuestra observación original, que aumenta de pHi como células en el linaje FSC se distinguen de la célula de vástago para el pFC para el estado de la célula del folículo, a través de estos ensayos múltiples demuestra tanto que este fenómeno biológico es sólida y que la metodología es confiable. Sin embargo, en nuestra experiencia, este es un procedimiento difícil de dominar. Requiere mucha atención al detalle en cada paso y ejecución rápida, altamente peritos de la disección, montaje y proyección de imagen pasos. Como se indica en el protocolo, el procedimiento de disección y montaje incluye una incubación de 10 min pero no debe exceder 15 minutos total y el procedimiento imagenológico se debe completar en 45 minutos. En condiciones ideales, ovariolas de Drosophila se pueden mantener en cultivo para hasta 14 h17, pero encontramos que el tejido empieza a morir mucho más rápidamente en los buffers de nigericin para generar las curvas estándar. Nuestras directrices para garantizan que todos los datos se recogen mientras el tejido está todavía dentro de la ventana de tiempo que aparece morfológicamente normal y saludable. Esto no deja mucho tiempo para cada paso, sin embargo, por lo que es importante planificar con antelación para asegurarse de que todo está listo para proceder de un paso a la siguiente. De hecho, es mucho más eficiente si dos personas trabajan juntas en la disección, montaje, y procedimientos, la proyección de imagen con una persona ejecutando un paso mientras que la otra persona se prepara para el siguiente paso.
Desde pHi puede ser sensible a las manipulaciones experimentales necesarias a la imagen del tejido ex vivo, como la composición del tampón y temperatura, este método es mejor identificar cambios relativos en pHi. Dado que la sonda se compone de dos proteínas fluorescentes distintas, como de sus propiedades de fluorescencia producción de quántum, propiedades de tiempo de la vida y del plegamiento pueden ser afectados diferentemente en diversos tipos celulares. Por lo tanto, es esencial incluir muestras tratadas con las condiciones de amortiguamiento nigericin y usarlas para generar una nueva curva de calibración para cada tipo de célula en cada ensayo. Con el fin de reducir al mínimo las fuentes adicionales de variabilidad, manteniendo todas las condiciones constantes a lo largo de la proyección de imagen se acentúa. Muestras en condiciones de buffer de nigericin deben ser preparadas y reflejadas en el mismo día como ésos en la condición de buffer de bicarbonato y la adquisición de imagen y preparación de todas las muestras deben mantenerse tan constantes como sea posible. Esto es importante porque las mediciones de pHi se basan en una comparación entre los sistemas de imagen, diferencias tan experimentales que afectan la detección de la mCherry y pHluorin en uno o más de los sistemas de imagen puede disminuir la precisión de las estimaciones de pHi. Factores como los cambios en los ajustes del voltaje de los tubos de fotomultiplicador (si está usando un láser de barrido confocal) o tiempos de exposición (si está usando un disco de giro confocal) o una lente sucia que reduce la cantidad de luz a la cámara, claramente afectará a los resultados . Pero hay una miríada de otros factores más sutiles que pueden variar de día a día y también podrían influir en los resultados, tal como si las moscas son bien alimentadas, la edad de las moscas, y cuánto los láseres han sido en antes de la proyección de imagen. Preparación y proyección de imagen todas las tres condiciones en el mismo día reduce al mínimo estas diferencias y así se producen los resultados más consistentes. En particular, cada vez que nos cambiamos a un nuevo microscopio o se han actualizado los componentes del microscopio, es necesario optimizar la configuración de los nuevos equipos. Esto dio lugar a menudo a un cambio en los valores promedio de las mediciones individuales, sin embargo, como se generaron nuevas curvas de calibración con cada ensayo, los cambios en el equipo tuvieron un impacto mínimo en las estimaciones de pHi. Además, la comparación relevante es a menudo entre diferentes tipos de células que están dentro del mismo tejido (por ejemplo, el Eve, PFC y folículo células) y por lo tanto son internamente controlada para las variaciones experimentales.
Aunque este protocolo se ha centrado en medir PI de tejido de tipo salvaje, la metodología es compatible con los métodos estándar de la Drosophila para manipular la expresión génica, como expresión de un transgen usando Gal4/UAS y el análisis de mosaico o de RNAi. Puesto que mCherry::pHluorin es conducida por un promotor UAS, siempre se expresa conjuntamente con el ARNi o transgen, y MARCM18 puede utilizarse para generar clones mutantes homocigóticos con mCherry::pHluorin como el marcador clonal. Por las razones descritas anteriormente, tejido de tipo salvaje debe analizarse como un control al lado de cada prueba con un genotipo mutante. Por último, los principios generales aquí descritos pueden aplicarse a la utilización de mCherry::pHluorin para medir pHi en otros tejidos también. De hecho, este protocolo fue adaptado de un protocolo para el uso de mCherry::pHluorin para medir la pHi en la Drosophila ojo7y hemos utilizado metodología similar a la imagen mCherry::pHluorin en el cerebro de larvas. En general, las herramientas y los métodos descritos aquí ofrecen nuevas oportunidades para investigar las muchas diversas funciones de pHi en vivo dentro del tejido vivo, intacto.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Agradecemos Bryne Ulmschneider contribuciones al Protocolo y Diane Barber para sugerencias sobre el manuscrito. Este trabajo fue financiado por el Instituto Nacional de donación de salud GM116384 para Nystul T.G. y D.L. barbero.
Materiales
| Name | Company | Catalog Number | Comments |
| Fly Stocks | |||
| UAS-mCherry::pHluorin[1] | |||
| y1 w*;P{GawB}10930/CyO | Bloomington Stock Center | 7023 | |
| Act-Gal4 flipout stock | Bloomington Stock Center | 4409 | |
| Name | Company | Catalog Number | Comments |
| Chemicals for Buffer preparation | |||
| NaCl | Sigma Aldrich | S5886 | |
| KCl | Sigma Aldrich | P-3911 | |
| glucose | Mallinckrodt | 4912 | |
| HEPES | Thermo Fisher Scientific | BP310 | |
| MgSO4 | Thermo Fisher Scientific | M63 | |
| CaCl2 | Sigma Aldrich | C-5080 | |
| HCO3 | Sigma Aldrich | S-5761 | |
| MgCl2 | Sigma Aldrich | M-9272 | |
| NMDG+ | Sigma Aldrich | M-2004 | |
| K2HPO4 | Mallinckrodt | 7088 | Use to Make KHPO4 pH 7.4 |
| KH2PO4 | Thermo Fisher Scientific | BP362 | Use to Make KHPO4 pH 7.4 |
| Concanavalin A, Alexa Fluor 647 Conjugate | Thermo Fisher Scientific | C21421 | 0.25 mg/ml dilution |
| Nigericin | Thermo Fisher Scientific | N1495 | |
| Name | Company | Catalog Number | Comments |
| Dissection and mounting tools | |||
| 2 Dumont Inox forceps (Size 5) | Thermo Fisher Scientific | NC9473431 | |
| 2 23-gauge syringe needles | Sigma Aldrich | Z192457 | |
| 9-well glass dissecting dish | Thermo Fisher Scientific | 13-748B | |
| Vacuum Grease | Dow Corning | 1018817 | |
| 22 X 40 mM glass coverslips | Thermo Fisher Scientific | 12545C | |
| Round Glass Coverslips, 12mm diameter, 0.13-0.16mm thickness | Ted Pella, Inc. | 26023 | |
| 3-D mounting chamber | custom manufactured | .stl and .ipt files for 3-D printer included as supplemental files | |
| Name | Company | Catalog Number | Comments |
| Other equipment | |||
| pH meter | Thermo Fisher Scientific | 13-620-183A | Model: Accumet AB15 |
| Dissection microscope | Olympus Corporation | 0H11436 | Model: SZ2-ST |
| Confocal Microscope | Leica Biosystems | SP5 or SP8 laser-scanning confocal microscope with a 40× objective with a numerical aperture of 1.3 |
Referencias
- Grillo-Hill, B. K., Choi, C., Jimenez-Vidal, M., Barber, D. L. Increased H+ efflux is sufficient to induce dysplasia and necessary for viability with oncogene expression. eLife. 03270, (2015).
- Ulmschneider, B., Grillo-Hill, B. K., Benitez, M., Azimova, D. R., Barber, D. L., Nystul, T. G. Increased intracellular pH is necessary for adult epithelial and embryonic stem cell differentiation. J. Cell Biol. 215 (3), 345-355 (2016).
- Han, J., Burgess, K. Fluorescent Indicators for Intracellular pH. Chemical reviews. 110 (5), 2709-2728 (2010).
- Koivusalo, M., Welch, C., et al. Amiloride inhibits macropinocytosis by lowering submembranous pH and preventing Rac1 and Cdc42 signaling. J. Cell Biol. 188 (4), 547-563 (2010).
- Choi, C. -. H., Webb, B. A., Chimenti, M. S., Jacobson, M. P., Barber, D. L. pH sensing by FAK-His58 regulates focal adhesion remodeling. J. Cell Biology. 202 (6), 849-859 (2013).
- Rossano, A. J., Chouhan, A. K., Macleod, G. T. Genetically encoded pH-indicators reveal activity-dependent cytosolic acidification of Drosophila motor nerve termini in vivo. J. Phys. 591 (7), 1691-1706 (2013).
- Grillo-Hill, B. K., Webb, B. A., Barber, D. L. Ratiometric imaging of pH probes. Methods in Cell Biol. 123, 429-448 (2014).
- Miesenböck, G., De Angelis, D. A., Rothman, J. E. Visualizing secretion and synaptic transmission with pH-sensitive green fluorescent proteins. Nature. 394 (6689), 192-195 (1998).
- Kirilly, D., Xie, T. The Drosophila ovary: an active stem cell community. Cell research. 17 (1), 15-25 (2007).
- Sahai-Hernandez, P., Castanieto, A., Nystul, T. G. Drosophila models of epithelial stem cells and their niches. Wiley interdisciplinary reviews. Dev. Biol. 1 (3), 447-457 (2012).
- Losick, V. P., Morris, L. X., Fox, D. T., Spradling, A. Drosophila stem cell niches: a decade of discovery suggests a unified view of stem cell regulation. Dev. Cell. 21 (1), 159-171 (2011).
- Pocha, S. M., Montell, D. J. Cellular and molecular mechanisms of single and collective cell migrations in Drosophila: themes and variations. Ann Rev. of Gen. 48, 295-318 (2014).
- St Johnston, D., Ahringer, J. Cell polarity in eggs and epithelia: parallels and diversity. Cell. 141 (5), 757-774 (2010).
- Castanieto, A., Johnston, M. J., Nystul, T. G. EGFR signaling promotes self-renewal through the establishment of cell polarity in Drosophila follicle stem cells. eLife. 3, (2014).
- Margolis, J., Spradling, A. Identification and behavior of epithelial stem cells in the Drosophila ovary. Development. 121 (11), 3797-3807 (1995).
- Nystul, T. G., Spradling, A. An epithelial niche in the Drosophila ovary undergoes long-range stem cell replacement. Cell stem cell. 1 (3), 277-285 (2007).
- Morris, L. X., Spradling, A. C. Long-term live imaging provides new insight into stem cell regulation and germline-soma coordination in the Drosophila ovary. Development. 138 (11), 2207-2215 (2011).
- Lee, T., Luo, L. Mosaic analysis with a repressible cell marker (MARCM) for Drosophila neural development. Trends in neuro. 24 (5), 251-254 (2001).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados