Method Article
Misurazione di profili biomolecolari DSC con ligandi termolabile per caratterizzare velocemente pieghevole e associazione interazioni
In questo articolo
Riepilogo
Vi presentiamo un protocollo per la rapida caratterizzazione biomolecolare pieghevole e associazione interazioni con ligandi termolabile mediante calorimetria differenziale a scansione.
Abstract
Calorimetria a scansione differenziale (DSC) è una tecnica potente per quantificare i parametri termodinamici che disciplinano biomolecolari pieghevole e interazione di legame. Questa informazione è fondamentale nella progettazione di nuovi composti farmaceutici. Tuttavia, molti ligandi farmacologicamente rilevanti sono chimicamente instabili alle alte temperature utilizzate nelle analisi DSC. Così, associazione di interazioni di misurazione è difficile perché le concentrazioni di ligandi e termicamente convertiti i prodotti sono in continua evoluzione all'interno della cellula del calorimetro. Qui, presentiamo un protocollo utilizzando ligandi termolabile e DSC per rapidamente ottenere informazioni termodinamiche e cinetiche sulla piegatura, l'associazione e ligando processi di conversione. Abbiamo applicato il nostro metodo per il DNA aptamero MN4 che associa la cocaina ligando termolabili. Utilizzando una nuova analisi di raccordo globale che i conti per la conversione di ligando termolabili, il set completo dei parametri di associazione e pieghevole si ottengono da un paio di esperimenti di DSC. Inoltre, ci mostra che la costante di velocità per la conversione di ligando termolabile può essere ottenuta con un solo set di dati supplementari DSC. Le linee guida per l'identificazione e l'analisi dei dati da diversi scenari più complessi è presentata, compreso irreversibile aggregazione della biomolecola, pieghevole lento, lento l'associazione e lo svuotamento rapido del ligando termolabile.
Introduzione
Calorimetria a scansione differenziale (DSC) è un metodo potente per quantificazione associazione biomolecolari e pieghevole interazioni1,2,3. I punti di forza della DSC includono la capacità di delucidare vincolante e pieghevole meccanismi e per produrre i corrispondenti parametri termodinamici2,3. Inoltre, la DSC può essere eseguita in soluzione in condizioni di quasi-fisiologiche e non richiede etichettatura di biomolecole o legante, ad esempio, con fluorofori, spin-etichette o isotopi nucleari4. Lo strumento esegue la scansione di temperatura, la misurazione della quantità di calore necessaria per denaturare la biomolecola in presenza e in assenza del ligando. I termogrammi risultante vengono utilizzate per estrarre i parametri termodinamici che disciplinano il ligando vincolanti e processi di piegatura. Le informazioni fornite da DSC o altre tecniche termodinamici sono fondamentale per guidare la progettazione di farmaci targeting biomolecole1,5,6,7,8. Tuttavia, la scansione ripetuta ad alte temperature (~ 60-100 ° C) può essere problematico. Ad esempio, molti composti farmacologicamente importanti subiscono riorganizzazione o decomposizione all'esposizione continua a temperature elevate9,10,11, vale a dire, essi sono termolabili. Esame dell'associazione interazioni di DSC in genere richiede più scansioni di avanti e indietro al fine di verificare la riproducibilità del termogramma per analisi termodinamica12. Conversione termica di un ligando iniziale di una forma secondaria con caratteristiche di associazione alterata conduce a pronunciate differenze nella forma e posizione dei termogrammi successivi, poiché la concentrazione del ligando iniziale diminuisce con ogni scansione mentre il conversione termica prodotti si accumulano. Questi set di dati non sono suscettibili di analisi tradizionali.
Recentemente abbiamo sviluppato un metodo di raccordo globale per i DataSet di ligando termolabile DSC che produce il set completo dei parametri termodinamici che disciplinano la piegatura biomolecolari e associazione interazioni da un singolo esperimento-ligando viene fatto riferita per la termogramma necessaria per la biomolecola libero4. L'analisi riduce il tempo sperimentale e il campione richiesto da ~ 10 volte rispetto agli approcci standard DSC. Abbiamo rappresentato per il ligando conversione termica assumendo questo succede durante la parte ad alta temperatura di ogni scansione dove il termogramma non dipende dalla concentrazione del ligando. Di conseguenza, la concentrazione del ligando è una costante all'interno della parte del termogramma che viene utilizzato per estrarre i parametri termodinamici. Abbiamo inoltre dimostrato come la costante di velocità per la conversione termica ligando possa essere ottenuta eseguendo un esperimento supplementare con un periodo più lungo di equilibratura ad alta temperatura. Per sistemi in cui la conversione termica ligando è meno temperatura-dipendente (cioè, che si verificano sensibilmente a tutte le temperature), l'analisi può essere modificato per includere le concentrazioni di ligando variabile. Qui dimostriamo questa procedura per il DNA aptamero MN4 in presenza di cocaina ligando termolabili, che converte rapidamente in benzoilecgonina ad alte temperature (> 60 ° C). Chinina è usata come controllo negativo per thermolability ligando poiché non sottoposti a conversione a queste temperature sperimentale e si lega anche al MN4. Descriviamo l'acquisizione di ligando termolabile DSC DataSet e loro analisi producendo i parametri termodinamici e cinetici del pieghevole, associazione e ligando processi di conversione.
Protocollo
1. preparazione del campione
- Purificare la biomolecola desiderato13.
Nota: Questo protocollo usi acquistati cocaina-associazione DNA aptamer MN4 dopo lo scambio contro 2 M NaCl, tre volte seguito da tre turni di acqua deionizzata utilizzando un filtro centrifugo con una membrana di cut-off di peso molecolare 3 kDa. - Sintetizzare e purificare, oppure acquistare il ligando termolabile desiderato13.
Nota: MN4 associa la cocaina ligando termolabili. MN4 si lega anche la chinina, che viene utilizzato come controllo negativo per thermolability di ligando a queste temperature sperimentale. - Preparare buffer per dialisi di biomolecole purificata e dissoluzione di ligandi (buffer di NaCl di fosfato di sodio e 140 mM 20 mM, pH 7.4, per MN4 e i leganti utilizzati qui).
- Dializzare biomolecola contro almeno 2 L di buffer utilizzando tubi di dialisi con 0,5 - 1,0 kDa cut-off.
- Filtrare il buffer finale (denominato buffer di lavoro) attraverso un filtro da 0,2 µm che è stato accuratamente equilibrato con buffer.
- Pesare le masse desiderate dei ligandi e scioglierli in buffer di lavoro filtrata. Se le concentrazioni di ligando desiderato richiedono masse che sono troppo piccole per pesare con precisione, fare una soluzione di riserva di ligando concentrato (10x per esempio).
Nota: È fondamentale che tutti gli esperimenti di DSC utilizzano lo stesso buffer di lavoro per l'esempio e ligando, vale a dire, mai eseguire un esperimento dove il ligando è dissolto in un batch diverso di buffer di lavoro rispetto la biomolecola ciò causerebbe buffer artefatti di mancata corrispondenza dei dati. - Filtrare la soluzione di riserva di biomolecola attraverso un filtro da 0,2 µm che è stato accuratamente equilibrato con buffer di lavoro.
- Determinare la concentrazione di biomolecole mediante misure di assorbanza (260 nm per gli acidi nucleici come MN4 e 280 nm per le proteine).
- Conservare il preparato biomolecola e ligando in frigorifero 4 ° C (adatto per MN4 e i leganti utilizzati qui), o -20 o -80 ° C se la biomolecola e ligandi tollerano congelamento e a lungo termine di deposito è richiesto. Degas le soluzioni tampone, biomolecole e ligando in una table top Degassatore (Vedi Tabella materiali) prima del caricamento nel DSC.
Nota: Degasaggio aiuta a prevenire la formazione di bolle nel DSC a temperature più elevate. Bolle di causano artefatti di segnale che oscurano la DSC picco forme e linee di base.
2. preparazione DSC
- Svitare la maniglia di pressione dal DSC (Vedi Tabella materiali).
- Eseguire il tubo in silicone da buffer di lavoro e associarlo a flangia anteriore (metallo apertura) del capillare di riferimento.
- Creare un ponte tra i capillari di riferimento e campione collegando la flangia posteriore riferimento alla flangia anteriore del campione.
- Applicare un pezzo di tubo in silicone al bordo posteriore del campione che corre a una boccetta dei rifiuti con una linea del vuoto collegata.
- Girare sulla linea del vuoto per svuotare il DSC con 200 mL di buffer di lavoro.
- Caricare il capillare di riferimento del DSC con buffer di lavoro. Allegare all'incirca sezioni di 3-5 cm di tubo in silicone per le flange capillare di riferimento.
- Inserire una punta di pipetta da 1 mL in tubo in silicone di flangia posteriore. 0,8 mL di buffer di lavoro con una pipetta di disegnare e inserire la punta della pipetta con il tampone nel tubo in silicone della flangia di riferimento anteriore.
- Premere delicatamente lo stantuffo della pipetta fino a passare il buffer di lavoro attraverso il tubo in silicone anteriore nel capillare di riferimento e verso la flangia posteriore collegato punta della pipetta. Premere verso il basso lo stantuffo pipetta fino a quando il livello del buffer di lavoro raggiunge appena sopra il tubo in silicone anteriore, quindi rilasciare lo stantuffo pipetta fino a quando il livello del buffer di lavoro raggiunge appena sopra il tubo in silicone posteriore.
- Ripetere passando il buffer di lavoro avanti e indietro per eliminare il volume nel capillare riferimento di bolle.
Nota: Solitamente, 10 i passaggi della soluzione avanti e indietro sono sufficienti per eliminare eventuali bolle.
- Ripetere passando il buffer di lavoro avanti e indietro per eliminare il volume nel capillare riferimento di bolle.
- Tappare la punta della pipetta posteriore con il pollice e tirare delicatamente la punta della pipetta posteriore e anteriore pipetta per rimuoverli dalle flange di riferimento con il tubo in silicone collegato.
- Caricare il capillare di campione con buffer di lavoro come nei passaggi 2.6-2.9. Mettere un tappo di plastica nero sul riferimento posteriore e flange, lasciando le flange anteriori scoperte di esempio.
- Fissare la maniglia di pressione.
- Aprire il software di DSC (Vedi Tabella materiali) e pressurizzare lo strumento facendo clic su freccia nella parte superiore dell'interfaccia rossa una volta che la lettura di potenza si stabilizza; il potere di DSC è indicato in una casella nella parte superiore destra dell'interfaccia insieme alla temperatura dello strumento e la lettura della pressione.
Nota: Monitor pressurizza il potere leggere come il DSC. Cambiamenti nel potere di più di ~ 10 µW indicare la formazione di bolle nei vasi capillari, che può causare artefatti nei dati. Le soluzioni devono essere rimossi e degassate ulteriore prima di continuare. - Equilibrare il DSC con buffer di lavoro eseguendo una scansione in avanti e indietro. Nella scheda "Metodo sperimentale" sul lato sinistro dello schermo, verificare che è selezionata l'opzione "Scansione" per eseguire il DSC in temperatura modalità di scansione.
Nota: Parametri sperimentali sono la temperatura gamma, velocità di scansione, tempo di equilibramento di bassa ed alta temperatura e numero di scansioni di scansione.- Nella rientranza "Parametri di temperatura" sotto la scheda "Metodo sperimentale", fare clic sul pulsante per il "Riscaldamento". Immettere 1 e 100 ° C per le temperature sperimentale inferiore e superiore, 1 ° C/min per la velocità di scansione e 60 s per il periodo di equilibrazione.
- Clicca sul pulsante "Aggiungi serie" sotto il campo di input per il periodo di equilibrazione. Inserire 2 nel campo "Passaggi per aggiungere" nella finestra pop-up (per un riscaldamento e raffreddamento una scansione) e la casella "alternativo riscaldamento/raffreddamento". Fare clic su "OK"; le scansioni aggiunta vengono visualizzati nella parte inferiore dell'interfaccia. Verificare che i parametri per ogni analisi sono come desiderato.
- Iniziare l'esperimento facendo clic sul pulsante "play" verde nella parte superiore dell'interfaccia. Selezionare la cartella desiderata e immettere un nome di file per il salvataggio l'esperimento nella finestra popup. Visualizzare l'avanzamento di esperimento facendo clic sulla scheda "Dati" a destra della scheda "Metodo di sperimentare".
3. raccolta termolabile ligando DSC Datasets
Nota: La procedura minima consiste di cinque esperimenti: esperimenti di riferimento con e senza ligando del buffer (utilizzato per la sottrazione della linea di base, Vedi discussione), campione esperimenti con la biomolecola gratis, biomolecole-ligando e la -ligando biomolecola con un periodo più lungo di equilibratura ad alta temperatura.
- Eseguire esperimenti di riferimento per la sottrazione della linea di base di dati di esempio. Ricaricare il DSC con buffer di lavoro in entrambi i capillari e raccogliere più scansioni di avanti e indietro su un range di temperatura adatta a 1 ° C min-1 (alta temperatura) alla tomaia con tempo di equilibramento di 120 s.
- Elimina le scansioni di equilibrazione buffer precedente dalla parte inferiore dell'interfaccia evidenziando ognuna singolarmente e facendo clic sulla X rossa per la metà destra dell'interfaccia. Aggiungere le nuove scansioni facendo clic sul pulsante "Aggiungi serie", immettendo 20 nel campo per la "Procedura per aggiungere" e selezionando la casella "alternativo riscaldamento/raffreddamento". Fare clic su OK ed eseguire l'esperimento facendo clic sul pulsante verde play come sopra.
- Ripetere i passaggi da 3.1 - esperimenti di 3.1.1 con buffer di lavoro contenente la concentrazione del ligando in entrambi capillari per ottenere il riferimento per il ligando (raccogliere due esperimenti separati utilizzando 120 s e 600 s ad alta temperatura equilibrazione volte rispettivamente, da utilizzarsi nell'acquisire la costante di velocità per la conversione di ligando termolabile).
Nota: La frequenza di scansione utilizzata qui assicura che la biomolecola negli esperimenti successivi all'equilibrio termico nelle scansioni e inversione (Vedi discussione). Scansione tariffe < 0,1-0,2 ° C min-1 portare a termogrammi rumorosi e non sono applicabili in esperimenti di DSC. Le temperature dovrebbero estendere da ben di sotto della temperatura di fusione della biomolecola gratis per ben di sopra della temperatura di fusione di biomolecola ligando-saturi (~ 20-80 ° C per MN4). Verificare la riproducibilità delle scansioni (per esempio, 10 in avanti e inversione 10 scansioni per 20 totale è sufficiente). - Se utilizzando ligandi più (come la cocaina e chinino), lavare il DSC con 200 mL di buffer di lavoro (ripetere da passo 2.5) tra le esecuzioni al fine di rimuovere ligando dai capillari e prevenire la contaminazione incrociata.
Nota: È utile eseguire un esperimento di replicare su biomolecole libera dopo una corsa-ligando al fine di verificare se il ligando precedente assorbe fortemente per le pareti dei capillari e non viene adeguatamente rimosso con tampone di lavaggio. Se i termogrammi per la biomolecola libero sembrano essere spostato a un ordine di grandezza maggiore e la più alta temperatura di denaturazione dopo il ligando esperimento, è probabile che il ligando precedente è ancora presente nel calorimetro dopo il lavaggio. Rimuovere il ligando adsorbito incubando i capillari con 20% Contrad-70 per 1 h a 60 ° C con i tappi di plastica e la maniglia di pressione fuori. Nel software DSC, cambiare la modalità sperimentale a "Isotermico" sotto la scheda scegliere 3.600 s di metodo sperimentale per la durata e i 60 ° C per la temperatura isotermica, con zero entrata per la volta di equilibrazione. Cliccare su "Aggiungi al metodo sperimentale". L'esperimento isotermico viene visualizzata nella parte inferiore dello schermo. Dopo il completamento, sciacquare lo strumento con acqua 2L deionizzata e ripetere dal passo 2.5.
- Eseguire esperimenti campione utilizzando lo stesso DSC caricamento procedure e parametri sperimentali come il riferimento le scansioni. Per il set di dati gratuito biomolecola, assicurarsi che il capillare di riferimento contiene il buffer di lavoro mentre il capillare di esempio contiene la biomolecola gratuito alla concentrazione desiderata nel buffer di lavoro.
- Per gli esperimenti-ligando, assicurarsi che il ligando è nel buffer di lavoro nel capillare di riferimento, e le biomolecole plus ligando è nel buffer di lavoro nel capillare del campione. Lavare il sistema tra aggiunte di leganti differenti come descritto al punto 2.5.
- Eseguire una ulteriore esperimento con la biomolecola associato al ligando termolabile dove il periodo di equilibratura ad alta temperatura è aumentato a 600 s e tutti gli altri parametri sperimentali sono gli stessi come punto 3.2.1.
Nota: La durata del periodo di equilibratura ad alta temperatura per l'esperimento di secondo-ligando è semplicemente scelto per assicurare che il ligando è più rapidamente impoverito che l'esperimento di equilibrazione breve tempo. Se i picchi-ligando dal primo esperimento decadimento lentamente in funzione del numero di analisi (ad esempio, le differenze in maxima picco successivi sono ≤ 0,5 ° C), stima aumenta di 10 - 20 volte in epoca di equilibratura ad alta temperatura al fine di adeguatamente impoveriscono il ligando durante il secondo esperimento. Il calcolo preciso della costante tasso di conversione di ligando richiede che il secondo esperimento ha più rapido svuotamento del ligando rispetto al primo. Le concentrazioni di ligando estratte dall'analisi globale dei due esperimenti sarà simile e quindi inutilizzabile se lo svuotamento di ligando non è sufficientemente accelerato nel secondo esperimento.
4. elaborazione dei dati
- Aperto il DSC sperimentare i file del software di analisi dati di DSC (Vedi Tabella materiali) ed esportare i dati di potenza pura come fogli di calcolo.
- Importare i fogli di calcolo che contiene i dati di potenza pura nel software per i dati di montaggio.
- Linea di base sottrarre i dati di esempio sottraendo i dati di potenza del buffer del gratuito e -ligando biomolecola esperimenti.
Nota: Per l'esperimento di ligando termolabile, sta diminuendo la concentrazione del ligando iniziale con ogni scansione. Pertanto, è ideale per sottrarre la scansione di buffer 1 dalla scansione del campione 1 e così via. Abbiamo trovato che le scansioni di buffer con la cocaina non cambia sensibilmente come procede la conversione di ligando e quindi una scansione di buffer di ligando termolabile singolo può essere utilizzata per sottrarre tutti i dati-ligando termolabili. - Consente di convertire i dati di potenza del campione basale-sottratto capacità termica.
Nota: La conversione richiede volume specifico parziale di biomolecole, che può essere stimato14,15,16. L'equazione per la conversione di potenza a capacità termica è stato descritto in precedenza17.
5. analisi dei dati
- A livello globale adatta l'equilibrazione breve tempo termolabile-ligando calore capacità dataset con un singolo set di baseline, concentrazione del ligando, pieghevole e ligando associazione di parametri come descritto in precedenza4.
- Ripetere la che misura globale per il dataset di equilibrazione lungo tempo al fine di calcolare la costante di velocità per la conversione di ligando termolabili come descritto in precedenza4.
Risultati
Dati rappresentativi per il ligando termolabile DSC sono mostrati nella Figura 1. La posizione e l'altezza del picco-ligando termolabile sposta successivamente verso quella di biomolecole non associata come il ligando termolabile è esaurito con ogni scansione (Figura 1a). Il profilo di denaturazione gratuito è utilizzato come riferimento per l'endpoint di conversione ligando termolabile (Figura 1b). Dati per MN4 associato a chinina sono mostrati come controllo negativo per la conversione di ligando termolabile (Figura 1b). Le scansioni di ligando termolabile finale sono leggermente più alti punti medi di transizione e altezze dei picchi relativi MN4 non associato, che indica che il prodotto di conversione ligando termolabile (benzoilecgonina) ha una debole affinità per MN4. I parametri termodinamici derivanti dall'analisi globale dei DataSet nella Figura 1 sono elencati nella tabella 1. I parametri pieghevoli per MN4 in presenza di cocaina o chinino sono identici all'interno di errore, come previsto poiché diluire piccole molecole non sono previsti per perturbare la termodinamica pieghevole della biomolecola gratis. I parametri di associazione sono in buon accordo con quelli da isotermica di titolazione calorimetria (ITC)18 e rivelano che preferenza di MN4 per chinino sopra cocaina è guidato da un più favorevole entalpia di legame. Nella Figura 2, l'aumento del periodo di equilibratura ad alta temperatura produce riduzioni più pronunciate della concentrazione termolabile ligando con ogni scansione rispetto il dataset di equilibrazione di breve periodo. Utilizzando i parametri di concentrazione forma globale ottimizzata dei due set di dati, la costante di velocità per la conversione di ligando alla temperatura elevata equilibrazione è calcolata dal versante della linea nella rientranza nella figura 2 .
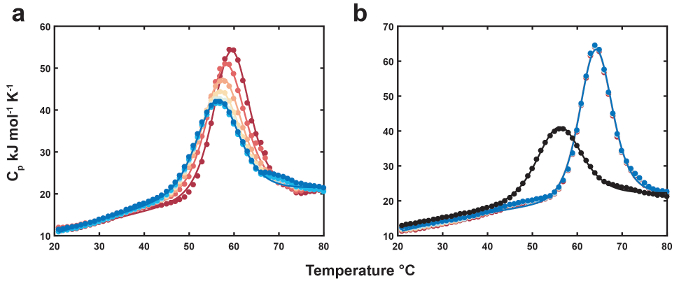
Figura 1. Termolabile ligando DSC. (un) termogrammi di MN4 (83 µM) limite alla cocaina (concentrazione iniziale 778 µM). Scansioni prime e l'ultima di MN4 in presenza di ligando vengono visualizzati come cerchi rossi e blu scuri mentre i PF corrispondenti vengono visualizzati come linee colorate. (b) termogrammi di MN4 gratis (83 cerchi µM, nero) e scansioni successive associato alla chinina (880 µM, cerchi colorati). Si adatta ai DataSet gratis e chinino associato vengono visualizzati come linee nere e colorate, rispettivamente. Riprodotto da riferimento4 con il permesso della Royal Society of Chemistry. Clicca qui per visualizzare una versione più grande di questa figura.
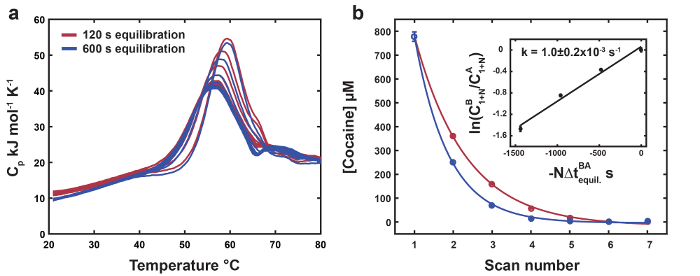
Figura 2. La costante di velocità di misura per la conversione di ligando termolabili. (un) insiemi di termogrammi per MN4 associato alla cocaina con breve (120 s, rosso scuro) e l'equilibratura lungo (600 s, blu scuro) volte a 80 ° C, rispettivamente. (b) le concentrazioni di cocaina estratta da analisi globale dei DataSet in (un) in funzione del numero di analisi. Punti sperimentali e si inserisce esponenziale vengono visualizzati come linee e cerchi colorati rispettivamente. L'inserto mostra una misura lineare al 19 EQ. complementare precedentemente descritti da Harkness et al utilizzando le concentrazioni di cocaina in forma globale ottimizzata per i due set di dati4. La costante di velocità per la conversione termica ligando a 80 ° C è calcolata come la pendenza della retta. L'errore per la costante di velocità per la conversione di ligando termolabile è dato come ± due deviazioni standard. Riprodotto da riferimento4 con il permesso della Royal Society of Chemistry. Clicca qui per visualizzare una versione più grande di questa figura.
| Parametri di misura | Aggiunto di cocaina | Aggiunto il chinino |
| ∆ hUF | 271.3±1.8 | 272.5±4.0 |
| ∆SUF | 824.4±5.1 | 827.9±10.9 |
| un∆GUF | 21.6±0.2 | 21.6±0.9 |
| un∆HB1F | -75.2±1.6 | -101.0±4.0 |
| un∆SB1F | -154.2±5.0 | -213.7±12.0 |
| ∆CpB1F | -1.5±0.1 | -1.2±0.1 |
| un∆GB1F | -28.5±0.2 | -36.2±0.7 |
| un∆HB2F | -33.7±1.8 | - |
| un∆SB2F | -49.9±5.2 | - |
| ∆CpB2F | -2.2±0.1 | - |
| un∆GB2F | -18.6±0.3 | - |
Tabella 1. Parametri termodinamici estratti dall'analisi globale dei DataSet DSC MN4 usando cocaina e ligandi di chinino. i parametri sono stati calcolati a 30 ° C. B1F si riferisce agli stati piegati cocaina - o chinino-bound e B2F alla stato piegato benzoilecgonina associato. ΔH e ΔG sono espressi in kJ/mol, ΔS è espressa in J/mol/K e ΔCp è espresso in kJ/mol/K. gli errori sono stati calcolati secondo il metodo di varianza/co-variance19.
Discussione
Le modifiche e la risoluzione dei problemi
I dettagli dell'analisi raccordo globale utilizzato nella Figura 1 e Figura 2 sono stati descritti in precedenza4. Qui, descriviamo gli aspetti pratici dell'esecuzione e l'analisi di esperimenti di binding DSC con ligandi termolabili. Si noti che una previsione di DSC ottenuta per il ligando termolabile da solo viene sottratto dal ligando + biomolecola dataset, cancellando in modo efficace il calore rilasciato o assorbito dalla conversione termica processo stesso. L'analisi di raccordo globale standard ligando termolabile (Figura 1 e Figura 2) presuppone che il sistema è in equilibrio termodinamico durante la scansione di temperatura e che la concentrazione del ligando termolabile è costante in tutto ogni termogramma, diminuendo esclusivamente durante il periodo di equilibratura ad alta temperatura. Precedentemente abbiamo indicato che questo presupposto riferisce l'associazione a cocaina MN4 e si prevede di tenere per qualsiasi sistema termolabile ligando/biomolecola con cinetiche simili a questi.
Tuttavia, esistono alcune situazioni in cui il sistema non può essere presupposta per essere in equilibrio termodinamico e/o la concentrazione del ligando non può essere considerata costante durante una singola scansione. Questi includono i) quando il ligando termicamente converte rapidamente rispetto alla velocità di scansione di temperatura, ii) quando la biomolecola subisce irreversibile aggregazione ad alta temperatura, iii) quando i tassi di chiusura/apertura sono lenti rispetto alla velocità di scansione, e iv). Quando i tassi di associazione/dissociazione di ligando sono lento rispetto al tasso di scansione. In questi casi, il sistema è sotto il controllo cinetico, piuttosto che la termodinamica e l'analisi fornito nel riferimento 4 non può essere applicata rigorosamente. Dati possono essere simulati quantitativamente in seguito Figura 3, come descritto nel File supplementari 1. In linea di principio, questi calcoli basati su cinetica potrebbero essere utilizzati per inserire dati di DSC non-equilibrio, potenzialmente producendo dati sia cinetici e termodinamici, tuttavia questa analisi esula dallo scopo di questa carta. Invece, vi presentiamo alcuni dati rappresentativi di DSC simulati per aiutare il lettore nell'individuare situazioni di non-equilibrio.
Un esempio ideale di controllo termodinamico è mostrato in Figura 4ab. Termogrammi DSC di biomolecole gratis sono sovrapponibili (Figura 4a) e scansioni con il ligando termolabile non mostrano isteresi, tale che la temperatura di fusione osservato su up-scansione corrisponde la temperatura pieghevole del precedente (a-scan Figura 4b). Quando il ligando termolabile converte rapidamente rispetto alla velocità di scansione, grandi distorsioni appaiono nel termogramma e le equazioni termodinamiche non tengono conto della forma di picco, come mostrato in Figura 4 c, d. Questo può essere alleviato un po ' aumentando la frequenza di scansione. Quando gli aggregati di biomolecole in maniera temperatura-dipendente, tracce di DSC per lo spettacolo gratuito biomolecola successivo diminuisce in grandezza (Figura 4e), durante l'aggiunta di ligand termolabile produce un modello decrescente upshifts termica Analogamente al caso ideale, ma in scala dalla concentrazione biomolecola decrescente (Figura 4f). Quando la chiusura/apertura cinetica sono lenti rispetto al tasso di scansione, isteresi sono ravvisabile nelle tracce di DSC di biomolecole gratis tale che la temperatura di denaturazione apparente fino-Scan è superiore alla temperatura di rinaturazione apparente sul giù-scansione ( Figura 4 g). Aggiunta di un legante termolabile conduce al modello familiare di diminuzione termica upshifts, specialmente per l'up-scansioni (Figura 4 h). Infine, i sistemi con ripiegamento rapido e lento associazione producono privo di isteresi termogrammi DSC per la biomolecola gratis (Figura 4i), tuttavia i dati con il ligando termolabile mostrano isteresi dove è la temperatura di fusione apparente della up-scansione più alta della temperatura apparente pieghevole della giù-scansione precedente (Figura 4j). Tuttavia, il modello tipico di diminuzione termica upshifts sono evidenti sia in up-scansioni che giù-scansioni. Comportamento di non-equilibrio nel caso di lente pieghevole o associazione cinetica può essere alleviato un po ' diminuendo la frequenza di scansione, anche se questo corre il rischio di conversione termica ligando non trascurabile che si verificano durante la scansione. In pratica, la temperatura di equilibrazione scansione tasso e superiore può essere regolata manualmente per ottenere dati simile a Figura 4a, b.
Limiti della tecnica
La nostra analisi termodinamica per DSC esperimenti con ligandi termolabile di associazione richiedono che la piegatura e processi di legame sono relativamente rapidi e che la conversione termolabile ligando è lento prima per la parte ad alta temperatura di ogni scansione. Quando la durata dello stato piegato e/o associato è maggiore di circa 30 s (k,i, ku < 0.03 s-1), isteresi diventa percepibile in scansioni eseguite a 1 ° C min-1. Inoltre, quando la costante di velocità di conversione di ligando è superiore a circa kc = 10-4 s-1 prima della transizione di denaturazione, può esserci lo svuotamento significativo del ligando nel corso di una singola scansione. Applicazione della nostra analisi è inopportuno quando aggregazione irreversibile si verifica. In questi casi, la modellazione più avanzata potrebbe essere applicato ai dati. Nessuna informazione di affinità è disponibile se la conversione di ligando è così rapida che giunge a completamento prima della prima transizione di denaturazione.
Significato per quanto riguarda i metodi esistenti
Il nostro metodo per la prima volta consente DSC per essere usato per misurare la termodinamica di associazione di alta affinità, ligandi termolabili. Eseguendo un'analisi globale e simultanea di tutte le scansioni, parametri termodinamici sono estratte con alta precisione20. Un ulteriore vantaggio è che l'insieme completo dei dati possono essere raccolti in un esperimento se il prodotto di conversione termica non ha alcuna affinità per la biomolecola gratis. Al contrario, producendo una serie di DSC sperimentale tipica per un ligando non termolabili richiede ~ 7-10 totali esperimenti.
Applicazioni future
Questo approccio ha applicazioni dirette per la caratterizzazione di inibitori stretti, termolabili in campagne di scoperta della droga. Diversi composti terapeutici come gli antibiotici e le benzodiazepine sono noti per essere termolabile, in fase di rapida idrolisi o vicino a pH fisiologico e temperature di ~ 60-70 ° C11. Questo metodo DSC è ben posizionato per identificare e caratterizzare molti di più. Pure, modifica del protocollo raccordo per rappresentare sistemi sotto controllo cinetico, piuttosto che termodinamico, come discusso in precedenza, ha il potenziale per aprire la porta a molti altri sistemi di rilevanza biologica.
Fasi critiche all'interno del protocollo
Una delle procedure sperimentali più importanti da considerare è la dialisi o scambio delle biomolecole e ligando in soluzioni tampone di lavoro identici (passaggi di protocollo 1.3-1.6). Mancata corrispondenza di buffer tra le soluzioni di ligando e biomolecola può portare a grandi artefatti nelle scansioni basale e campione che oscurano completamente i dati pertinenti e pieghevoli. Inoltre, è essenziale che il valore di potenza si stabilizzi prima il DSC è pressurizzato in modo che può essere monitorato durante la pressurizzazione (punto 2.3 del protocollo). Se la lettura di potenza cambia di più di ~ 10 µW durante la pressurizzazione, bolle formatesi probabilmente nei capillari e può causare grandi manufatti nei dati. In questo caso, le soluzioni devono essere degassato più a fondo.
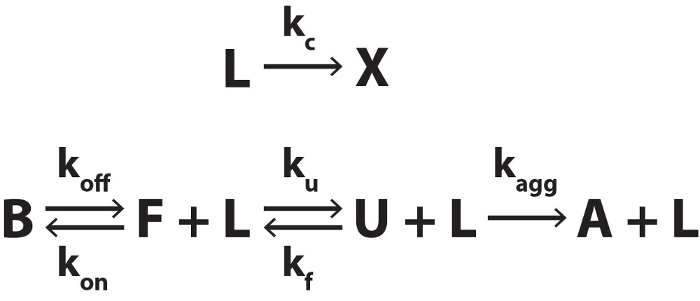
Figura 3. Biomolecolari pieghevole, associazione a un ligando termolabile e aggregazione irreversibile. Il ligando termolabile (L) converte in prodotto di (X) con un tasso costante kc. X non ha alcuna affinità per la biomolecola. Stato associato (B) degli scambi con la biomolecola piegato stato (F) con tasso costanti koff e ksu. Scambi di F con spiegato con tasso costanti ku e kf(U). U irreversibilmente converte allo stato aggregato (A) con il tasso costante kagg. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 4. Esperimenti di simulazione al computer di equilibrio e cineticamente controllato DSC in assenza e in presenza di un ligando termolabile. (un) equilibrio pieghevole biomolecolari. (b) equilibrio pieghevole, ligando termolabile e conversione lenta ligando termolabili. (c) equilibrio biomolecolari pieghevole. (d) equilibrio pieghevole, ligando termolabile e conversione veloce termolabile ligando. (e) equilibrio biomolecolari folding e aggregazione irreversibile lento. (f) equilibrio pieghevole, vincolante, lento conversione ligando termolabile e aggregazione irreversibile lento. (g), Slow biomolecolari pieghevole. (h) Slow pieghevole, l'associazione di equilibrio e conversione lenta ligando termolabili. (io) equilibrio biomolecolari pieghevole. (j) equilibrio pieghevole, ligando termolabile lento e lento ligando termolabile conversione. In tutti i pannelli, la prima e l'ultima scansione simulata sono blu scuro e rosso scuro, rispettivamente. Pannelli che mostrano solo chiare e scure termogrammi blu indicano che tutte le scansioni simulate di sovrapposizione, e solo le ultime due sono visibili nella trama. Esperimenti di DSC sono stati simulati con 20 scansioni (10 di fusione e 10 ricottura) a 1 ° C min-1 velocità di scansione, con una gamma di temperatura di 0-80 ° C. Alcuni pannelli display stretti range di temperatura per consentire la migliore visualizzazione delle tendenze simulazione. Le concentrazioni di biomolecole e ligando nelle simulazioni erano 200 µM e 10 mM, rispettivamente. Ogni esperimento è stato simulato con un 600 tempo di equilibramento s a 0 ° C prima della scansione e un tempo di equilibramento s 60 tra ciascuna delle scansioni successive. Parametri di Arrhenius per equilibrio vincolante e pieghevole erano unasu = 5 x 10-1 M-1 s-1, unoff = s19 1 x 10-1, Eunasu =-20 kJ mol-1, Eun off = 120 kJ mol-1, unapiega = 1 x 10-14 s-1, undispiegarsi = 5 x 1018, Eunapiega =-80 kJ mol-1ed Eundispiegarsi = 120 kJ mol-1 . Parametri di Arrhenius per cineticamente controllato vincolante e pieghevole erano unasu = 5 x 10-3 M-1 s-1, unoff = s16 1 x 10-1, Eunasu =-20 kJ mol-1, E un off = 120 kJ mol-1, unapiega = 1 x 10-16 s-1, undispiegarsi = 5 x 1016, Eunapiega =-80 kJ mol-1ed Eundispiegarsi = 120 kJ mol-1 . Parametri di Arrhenius per ligando termolabile lento e veloce conversione erano unlento = 7.509 x 1010 s-1, Eunlento = 94,65 kJ mol-1e unveloce = 1 s-1, Eun veloce = 10 kJ mol-1. Parametri di Arrhenius per aggregazione irreversibile lento erano unagg. = 5 x 107 s-1 ed Eunagg. = 80 kJ mol-1. Capacità termica in eccesso sono state calcolate con ΔHUF (svolgersi), ΔHBF (associazione) e ΔHAF (aggregazione) = 200 -140 e 50 kJ mol-1, rispettivamente. La descrizione teorica di DSC cineticamente controllato esperimenti con ligandi termolabile e script per l'esecuzione di queste simulazioni (e i parametri necessari) sono disponibili in supplementari File 1. Clicca qui per visualizzare una versione più grande di questa figura.
Divulgazioni
Gli autori non dichiarano conflitti di interesse.
Riconoscimenti
R. W. H. V era sostenuta dalla McGill scienze naturali e ingegneria ricerca Consiglio del Canada (NSERC) programma di formazione in Bionanomachines. A. K. M. e P. E. J. sono stati sostenuti da borse di studio NSERC 327028-09 (A. K. M) e 238562 (E. J.).
Materiali
| Name | Company | Catalog Number | Comments |
| Sodium chloride | Chem Impex | #00829 | |
| Sodium phosphate monobasic dihydrate | Sigma Aldrich | 71502 | |
| Sodium phosphate dibasic | Sigma Aldrich | S9763 | |
| Deioinized water for molecular biology | Millipore | H20MB1001 | |
| 0.2 micron sterile syringe filters | VWR | CA28145-477 | |
| 3 kDa centrifugal filters | Millipore | UFC900324 | |
| Dialysis tubing 0.5-1.0 kDa cutoff | Spectrum Laboratories | 131048 | |
| Silicon tubing | VWR | 89068-474 | |
| Plastic DSC flange caps | TA Instruments | 6111 | |
| DNA aptamer MN4 | Integrated DNA Technologies | https://www.idtdna.com/site/order/menu | |
| Cocaine | Sigma Aldrich | C008 | |
| Quinine | Sigma Aldrich | 22620 | |
| NanoDSC-III microcalorimeter | TA Instruments | http://www.tainstruments.com/nanodsc/ | |
| DSCRun software | TA Instruments | http://www.tainstruments.com/support/software-downloads-support/instruments-by-software/ | |
| NanoAnalyze software | TA Instruments | http://www.tainstruments.com/support/software-downloads-support/instruments-by-software/ | |
| Contrad-70 | VWR | 89233-152 |
Riferimenti
- Bruylants, G., Wouters, J., Michaux, C. Differential scanning calorimetry in life science: thermodynamics, stability, molecular recognition and application in drug design. Curr Med Chem. 12 (17), 2011-2020 (2005).
- Privalov, P. L., Dragan, A. I. Microcalorimetry of biological macromolecules. Biophys Chem. 126 (1-3), 16-24 (2007).
- Brandts, J. F., Lin, L. N. Study of strong to ultratight protein interactions using differential scanning calorimetry. Biochemistry. 29 (29), 6927-6940 (1990).
- Harkness, R. W., Slavkovic, S., Johnson, P. E., Mittermaier, A. K. Rapid characterization of folding and binding interactions with thermolabile ligands by DSC. Chem Commun. 52 (92), 13471-13474 (2016).
- Garbett, N. C., Chaires, J. B. Thermodynamic studies for drug design and screening. Expert Opin Drug Dis. 7 (4), 299-314 (2012).
- Holdgate, G. A., Ward, W. H. J. Measurements of binding thermodynamics in drug discovery. Drug Discov Today. 10 (22), 1543-1550 (2005).
- Plotnikov, V., et al. An autosampling differential scanning calorimeter instrument for studying molecular interactions. Assay Drug Dev Technol. 1 (1), 83-90 (2002).
- Schon, A., Lam, S. Y., Freire, E. Thermodynamics-based drug design: strategies for inhibiting protein-protein interactions. Future Med Chem. 3 (9), 1129-1137 (2011).
- Periánez Parraga, L., G-L, A., Gamón Runnenberg, I., Seco Melantuche, R., Delgado Sánchez, O., Puigventós Latorre, F. Thermolabile Drugs. Operating Procedure In the Event of Cold Chain Failure. Farmacia Hospitalaria. 35 (4), 1-28 (2011).
- Murray, J. B., Alshora, H. I. Stability of Cocaine in Aqueous-Solution. J Clin Pharmacy. 3 (1), 1-6 (1978).
- Waterman, K. C., et al. Hydrolysis in pharmaceutical formulations. Pharm. Dev. Technol. 7 (2), 113-146 (2002).
- Mergny, J. L., Lacroix, L. Analysis of thermal melting curves. Oligonucleotides. 13 (6), 515-537 (2003).
- Neves, M. A., Reinstein, O., Johnson, P. E. Defining a stem length-dependent binding mechanism for the cocaine-binding aptamer. A combined NMR and calorimetry study. Biochemistry. 49 (39), 8478-8487 (2010).
- Bonifacio, G. F., Brown, T., Conn, G. L., Lane, A. N. Comparison of the electrophoretic and hydrodynamic properties of DNA and RNA oligonucleotide duplexes. Biophys J. 73 (3), 1532-1538 (1997).
- Durchschlag, H., Hinz, H. -. J. Chapter 3. Thermodynamic Data for Biochemistry and Biotechnology. , 45-128 (1986).
- Hellman, L. M., Rodgers, D. W., Fried, M. G. Phenomenological partial-specific volumes for G-quadruplex DNAs. Eur Biophys J Biophy. 39 (3), 389-396 (2010).
- Farber, P., Darmawan, H., Sprules, T., Mittermaier, A. Analyzing Protein Folding Cooperativity by Differential Scanning Calorimetry and NMR Spectroscopy. J Am Chem Soc. 132 (17), 6214-6222 (2010).
- Reinstein, O., et al. Quinine binding by the cocaine-binding aptamer. Thermodynamic and hydrodynamic analysis of high-affinity binding of an off-target ligand. Biochemistry. 52 (48), 8652-8662 (2013).
- Tellinghuisen, J. Statistical error propagation. J Phys Chem. A. 105 (15), 3917-3921 (2001).
- Drobnak, I., Vesnaver, G., Lah, J. Model-based thermodynamic analysis of reversible unfolding processes. J Phys Chem B. 114 (26), 8713-8722 (2010).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon