Method Article
Predizione del silenziamento del gene attraverso il controllo spatiotemporale della siRNA rilasciata da nanocarrier polimerici sensibili alla foto
In questo articolo
Riepilogo
Presentiamo un nuovo metodo che utilizza copolimeri a blocchi foto-reattivi per un controllo più efficace spatiotemporale del silencing del gene senza effetti off-target rilevabili. Inoltre, i cambiamenti nell'espressione genica possono essere predittivati utilizzando semplici test di rilascio siRNA e semplice modellizzazione cinetica.
Abstract
Nuovi materiali e metodi sono necessari per meglio controllare il rilascio vs rilascio di acidi nucleici per una vasta gamma di applicazioni che richiedono una precisa regolazione dell'attività genica. In particolare, nuovi materiali sensibili agli stimoli con un miglior controllo spatiotemporale sull'espressione genica potrebbero sbloccare piattaforme traducibili nella ricerca di farmaci e nelle tecnologie della medicina rigenerativa. Inoltre, una maggiore capacità di controllare il rilascio dell'acido nucleico da materiali consentirebbe lo sviluppo di metodi razionalizzati per prevedere a priori l' efficacia del nanocarrier, portando a uno screening accelerato dei veicoli di erogazione. In questo esempio, presentiamo un protocollo per prevedere efficienze di silenziamento del gene e raggiungere il controllo spaziale sull'espressione genica attraverso un sistema nanocarrier modulare fotoreattivo. Il piccolo RNA interferente (siRNA) è complessato con polimeri mPEG- b- poli (5- (3- (ammino) propossi) -2-nitrobenzilmetacrilato (mPEG- b -P (APNBMA))Rm stabili nanocarrier che possono essere controllati con luce per facilitare l'attivazione / disinserzione di siRNA sintonizzabile. Diamo due contesti complementari che impiegano spettroscopia di correlazione alla fluorescenza e elettroforesi gel per la quantificazione accurata del rilascio siRNA da soluzioni che imitano ambienti intracellulari. Le informazioni ottenute da questi saggi sono state incorporate in un modello cinetico di interferenza RNA (RNAi) per predire le risposte dinamiche di silenziamento a varie condizioni di foto-stimolo. A loro volta, queste condizioni ottimizzate di irradiazione permettevano di affinare un nuovo protocollo per controllare spatiotemporalmente il silenziamento del gene. Questo metodo può generare schemi cellulari nell'espressione genica con risoluzione di cellule a cellule e nessun effetto off-target rilevabile. Considerati insieme, il nostro approccio offre un metodo di facile utilizzo per prevedere cambiamenti dinamici nell'espressione genica e controllare esattamente l'attività di siRNA nello spazio e nel tempo. Questo insieme di saggi può essere facilmente adattato per testare un'ampia varietà di otI suoi sistemi sensibili agli stimoli per affrontare le sfide chiave pertinenti a una moltitudine di applicazioni nella ricerca e nella medicina biomedica.
Introduzione
I piccoli RNA interferenti (siRNAs) mediano il silenziamento del gene post-trascrizionale attraverso un percorso catalitico RNAi altamente specifico, potente e adattabile a virtualmente qualsiasi gene bersaglio 1 . Queste caratteristiche promettenti hanno permesso alle terapeutiche siRNA di avanzare negli studi clinici umani per il trattamento di numerose malattie, tra cui il melanoma metastatico e l'emofilia 2 , 3 . Tuttavia, persistono problemi di consegna significativi che hanno ostacolato la traduzione 4 . In particolare, i veicoli di consegna devono rimanere stabili e proteggere gli siRNA dal degrado extracellulare, ma anche rilasciare il carico utile nel citoplasma 5 . Inoltre, molte applicazioni RNAi richiedono metodi migliorati per regolare il silenziamento del gene nello spazio e nel tempo 6 , che ridurrà gli effetti collaterali nelle terapie 7 di siRNA e consentirà di trasformare unDvances in applicazioni che vanno da microarray di cellule per la scoperta di droga 8 alla modulazione delle risposte delle cellule negli scaffolds rigenerativi 9 . Queste sfide evidenziano la necessità di nuovi materiali e metodi per controllare meglio il rilascio rispetto a rilascio nei nanocarrier di siRNA.
Una delle strategie più promettenti per controllare il rilascio di siRNA e migliorare la regolazione spaziale è l'uso di materiali sensibili alle stimoli 10 . Ad esempio, un'ampia varietà di biomateriali sono stati progettati con affinità di legame variabile di acido nucleico in risposta al potenziale alterato del redox o al pH, o campi magnetici applicati, ecografia o luce 11 . Sebbene molti di questi sistemi dimostrano un migliore controllo dell'attività dell'acido nucleico, l'utilizzo della luce come trigger è particolarmente vantaggioso grazie alla sua risposta temporale istantanea, alla precisa risoluzione spaziale e alla facilità di sintonizzazione 12. Inoltre, il potenziale delle tecnologie fotosensibili per la regolazione dell'espressione genica è stato dimostrato da un promotore inducibile all'avanguardia e da sistemi di regolazione ottogenetica; Tuttavia, questi sistemi soffrono di numerose sfide, incluse le capacità limitate per regolare i geni endogeni, i problemi di sicurezza come l'immunogenicità e le difficoltà nell'erogazione di gruppi multi-componente 13 , 14 e 15 . I nanocarrier siRNA rispondenti alla foto sono ideali per superare questi inconvenienti e fornire un approccio più semplice e più robusto per modulare l'espressione genica spatiotemporalmente 16 , 17 , 18 . Purtroppo, i metodi per predire in modo preciso la risposta alla rottura della proteina risultante rimangono inafferrabili.
Una sfida fondamentale è che le valutazioni quantitative di rilascio siRNA sonoRari 19 , 20 , e anche quando queste valutazioni vengono eseguite, non sono state accoppiate ad analisi della dinamica di turnover di siRNA / protein. Sia la quantità di siRNA rilasciata che la sua persistenza / vita sono determinanti importanti delle dinamiche di silenziamento del gene risultanti; Quindi, una mancanza di tali informazioni è una sconnessione principale che preclude una precisa predizione della risposta dose-RNAi 21 . Affrontare questa sfida accelererebbe la formulazione delle relazioni strutturali-funzioni appropriate nei nanocarri e informare meglio i progetti biomateriali 22 . Inoltre, tali approcci consentirebbero lo sviluppo di protocolli di dosaggio più efficaci di siRNA. Nel tentativo di comprendere la risposta dinamica di silenziamento, diversi gruppi hanno studiato modelli matematici di RNAi 23 , 24 , 25 . Questi quadri eranoRiuscito a fornire approfondimenti sulle trasformazioni mediate dal siRNA nell'espressione genica e identificando i passi di limitazione del tasso 26 . Tuttavia, questi modelli sono stati applicati solo ai sistemi di distribuzione genica commerciale ( ad es . Lipofectamine e Polyethylenimine (PEI)) che non sono in grado di controllare il rilascio di siRNA e la complessità dei modelli ha severamente limitato la loro utilità 27 . Queste carenze evidenziano una necessità insoddisfatta di nuovi materiali in grado di ottimizzare il rilascio di siRNA in combinazione con modelli di cinetica predittiva e facile da usare.
Il nostro metodo affronta tutte queste sfide attraverso l'integrazione di una piattaforma nanocarrier sensibile alla luce con metodi accoppiati per quantificare il libero siRNA e la dinamica di modello RNAi. In particolare, la rilascio preciso di siRNA 28 della nostra piattaforma è monitorata da due metodi complementari per la quantificazione accurata di encapsulated vs. unSiRNA legato. I dati sperimentali di questi saggi sono inseriti in un semplice modello cinetico per prevedere efficienze di silenziamento del gene a priori 29 . Infine, la natura on / off dei nanocarrier è facilmente sfruttata per generare modelli cellulari in espressione genica con controllo spaziale sulla scala di lunghezza cellulare. Quindi, questo metodo fornisce un metodo facilmente adattabile per controllare e prevedere il silenziamento del gene in una varietà di applicazioni che trarrebbero vantaggio dalla regolazione spaziale del comportamento delle cellule.
Protocollo
1. Formulazione di siRNA Nanocarriers
- Preparare soluzioni separate di siRNA e mPEG- b- P (APNBMA) con volumi uguali diluiti in tampone di acido 4- (2-idrossietil) piperazina-1-etansolfonico (HEPES) di 20 mM a pH 6,0.
- Aggiungere siRNA ad una concentrazione di 32 μg / mL a 20 mM HEPES soluzione.
NOTA: il siRNA era una sequenza negativa di controllo negativo non mirata; Tuttavia, il siRNA può essere progettato per indirizzare qualsiasi gene di interesse. - Sciogliere i polimeri mPEG- b- P (APNBMA) in una soluzione HEPES da 20 mM. Aggiungere una quantità appropriata di mPEG- b- P (APNBMA) per ottenere una soluzione di 220 μg / mL in modo che il rapporto N / P (N, gruppi amminici su mPEG- b -P (APNBMA); P, gruppi fosfatici su siRNA) È 4.
NOTA: Il protocollo sintetico per i polimeri mPEG- b- P (APNBMA) è riportato altrove 30 .
- Aggiungere siRNA ad una concentrazione di 32 μg / mL a 20 mM HEPES soluzione.
- Aggiungere la soluzione mPEG- b- AP (APNBMA) in goccia a un volume ugualeE della soluzione siRNA mescolando dolcemente su una macchina di vortice. Continuare a vortex per 30 s dopo l'aggiunta di polimero. Incubare i campioni al buio a temperatura ambiente per 30 min.
2. Misurare il rilascio di siRNA utilizzando l'elettroforesi del gel
- Formulare i nanocarrier secondo le fasi 1.1-1.2 e scalare i volumi per soddisfare il numero di campioni desiderati.
- Mescolare il nanocarrier con dodecil solfato di sodio (SDS).
- Preparare una soluzione di SDS da 1 mg / mL in acqua. Aliquota la quantità di soluzione SDS necessaria per produrre soluzioni con un rapporto S / P (S, gruppi di solfati su SDS; P, gruppi di fosfati su siRNA) di 15.
NOTA: Se la soluzione polilaterale contiene 1 μg di siRNA, è necessario aggiungere 13 μg di SDS per ottenere un rapporto di S / P di 15. - Aggiungere la soluzione SDS a ciascuna soluzione nanocarrier in goccia mentre mescolare delicatamente su una macchina di vortice. Continuare a vortice per 30 s dopo l'aggiunta di SDS.
- Centrifugare i campioni a 3.000 xg per 5 s. Incubare i campioni al buio a temperatura ambiente per 30 min.
- Preparare una soluzione di SDS da 1 mg / mL in acqua. Aliquota la quantità di soluzione SDS necessaria per produrre soluzioni con un rapporto S / P (S, gruppi di solfati su SDS; P, gruppi di fosfati su siRNA) di 15.
- Calibrare e impostare un laser UV con un filtro da 365 nm ad un'intensità di 200 W / m. Assicurarsi che l'intensità della luce venga misurata dalla posizione in cui sarà posizionato il fondo della soluzione campione.
- Caricare la soluzione nanocarrier / SDS in una camera di vetro composta da scivoli in vetro separati da una guarnizione di gomma.
- I vetri di pre-lavaggio scivolano in una soluzione di etanolo / acqua 7: 3 (v / v) in acqua e si asciugano completamente. Tagliare un foro (~ 2 x 3 cm rettangolo) in una guarnizione di gomma. Posizionare la guarnizione in gomma su una lastra di vetro.
- Pipettare la soluzione nanocarrier / SDS sullo scivolo in vetro all'interno del foro della guarnizione di gomma. Caricare un eccesso di soluzione (20 μL in eccedenza) sul vetrino, evitando il contatto con la guarnizione di gomma.
NOTA: Alcuni liquidi verranno persi durante i passaggi successivi. - Mettere il secondo bicchiereScivolare sulla parte superiore della guarnizione a scorrimento. Per evitare la formazione di bolle d'aria, posizionare prima un'estremità della diapositiva e poi lentamente abbassare l'altra estremità.
- Fissare i fermi leganti a ciascun lato della camera di vetro per tenerlo chiuso.
- Irradiare i campioni per la durata desiderata ( es . 0-60 min) usando il laser UV con un filtro da 365 nm ad un'intensità di 200 W / m. Rimuovere i fermi leganti e aprire la camera.
- Pipettare 25 μL dei campioni nanocarrier / SDS irradiati in un tubo di microcentrifuga. Incubare le soluzioni al buio a temperatura ambiente per 30 min.
- Preparare un gel agarosico di 2% pre-colorato con 0,5 μg / mL bromuro di etidio in soluzione tampone Tris / Borato / EDTA (TBE) secondo protocolli standard 31 . Preparare un tampone di carico composto da 3: 7 (v / v) glicerolo / acqua.
- Aggiungere 5 μL della soluzione del tampone di caricamento a 25 μl di ciascun campione nanocarrier / SDS. Incubare i campioni al buio aTemperatura ambiente per 10 min.
- Caricare 30 μl di ciascun campione di nanocarrier / SDS nel gel di agarosio del 2%. Eseguire il gel al buio a 100 V per 30 min. Immagine il gel utilizzando un sistema di imaging gel con filtri di bromuro di etidio. Salvare i file immagine di gel e procedere al punto 2.10 per quantificare l'intensità della banda. Assicurarsi che le intensità della banda siano abbastanza luminose per visualizzare chiaramente, ma non troppo luminose, che i segnali sono saturi.
- Quantificare le intensità della banda utilizzando il software ImageJ disponibile 32 .
- Utilizzando lo strumento ROI di ImageJ, determinare l'intensità di fluorescenza delle bande gratuite siRNA in ciascuna corsia disegnando un rettangolo intorno a ciascuna banda. Tracciare le curve di intensità di ogni corsia e integrare l'area sotto le curve disegnando una linea orizzontale attraverso le curve di intensità e facendo clic sulla barra di tracciamento all'interno delle aree chiuse.
- Calcolare l'intensità relativa di ciascuna corsia dividendo l'area sotto la cuRve di ciascun campione per l'area sotto la curva del controllo positivo siRNA (nessun mPEG- b- P (APNBMA) aggiunto e non è stata aggiunta alcuna SDS). Segnala la percentuale di siRNA rilasciata come l'intensità di banda normalizzata di ciascun campione.
3. Misurare la rilascio di siRNA utilizzando la spettroscopia di correlazione alla fluorescenza (FCS)
- Ottenere il siRNA etichettato con un solo fluoroforo all'estremità 5 'del filo di senso.
NOTA: il siRNA può essere acquistato pre-annealed con le etichette coniugate nella posizione desiderata. Il fluoroforo dovrebbe essere fotostrato ed assorbire / emettere tra 450 e 750 nm per evitare lo spegnimento della luce UV e il trasferimento di energia con mPEG- b- P (APNBMA). - Formulare i nanocarrier secondo le fasi 1.1-1.2 usando il siRNA etichettato. Scala i volumi per adattarsi al numero di campioni desiderati.
- Incubare le soluzioni in SDS e irradiare per la durata desiderata secondo le fasi 2.2-2.6.
- preparDella camera di campionamento FCS.
- Lavare una vetrata di vetro con una soluzione di etanolo / acqua 7: 3 (v / v) e asciugare completamente il vetro usando una strofinata e un flusso d'aria.
- Rimuovere i pezzi di carta da un distanziatore adesivo a due lati per esporre il distanziatore adesivo a doppia faccia. Fissare il distanziatore ad una copertura in vetro.
- Pipettare la soluzione nanocarrier / SDS sullo scorrimento del coperchio al centro del foro dal distanziale adesivo.
- Posizionare il vetrino in vetro sulla parte superiore del coperchio. Spingere sul vetrino per assicurare che il vetrino e la copertura siano ben attaccati e formare una tenuta.
- Utilizzare un microscopio confocale per le misurazioni FCS 33 . Utilizzare un obiettivo 40 ° di apocromo di immersione in acqua con un'apertura numerica di 1,2. Utilizzare il giusto canale laser di eccitazione (488 nm) per raccogliere almeno 30 misurazioni di 10 s ciascuna per campione 34 . Assicurarsi che l'intensità laser e l'allineamento del rilevatore rimanganoLo stesso per ogni campione.
- Oltre ai campioni sperimentali, i controlli di misura comprendono: un campione in bianco senza il siRNA etichettato; E un campione libero siRNA con siRNA etichettato ma non mPEG- b- P (APNBMA).
- Analizzare i dati utilizzando il software specifico FCS. Identificare la velocità di conteggio basale di ciascun campione determinando la velocità di conteggio stabile durante un periodo in cui nessun nanocarrier passa attraverso il volume confocale 29 .
- Sottrai la velocità di conteggio del campione in bianco da ogni valore di conteggio del conteggio basale del campione. Normalizzare i valori risultanti al controllo libero di siRNA per calcolare il percento del siRNA libero 35 .
4. Modellazione cinetica per predire il silenziamento del gene
- Creare script in un linguaggio di programmazione matematica utilizzando il semplice set di equazioni differenziali ordinarie per prevedere la silenziazione del gene 29.
NOTA: Gli script possono essere resi disponibili su richiesta.- Scrivi il set di equazioni differenziali ordinarie come:
 (1)
(1)  (2)
(2) 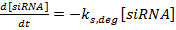 (3)
(3)
NOTA: Per le equazioni 1-3, i termini k mRNA , k siRNA e k prot sono le costanti di velocità per la produzione di mRNA, siRNA e proteine rispettivamente. I termini k m, deg , k s, deg e k p, deg sono le costanti di velocità per il degrado rispettivamente di mRNA, siRNA e proteine. Le costanti di tasso di degradazione sono calcolate sulla base delle metà vite del componente e le costanti di velocità di produzione sono adatte a garantire che i valori di stato stazionario mRNA e proteine siano raggiunti in assenza diF siRNA.- Determinare le metà della mRNA e della proteina per il gene (i) di interesse, sia sperimentalmente come descritto nel riferimento 36 o dalla letteratura (vedi Discussione ). Inoltre, determinare il tempo di raddoppiamento per la linea cellulare. Inserisci questi valori nelle espressioni appropriate di degradazione.
- Regolare le costanti di velocità di produzione in modo che i livelli di espressione genica restino costanti a un valore normalizzato di 100 se non viene introdotto alcun siRNA. In particolare, impostare [siRNA] su zero e variare i valori delle costanti di velocità di produzione k mRNA , k siRNA e k prot. Fino a quando [mRNA] e [proteina] rimangono entro l'1% del valore normalizzato iniziale del 100% La simulazione.
- Utilizzando le relative quantità di siRNA rilasciate dall'elettroforesi del gel e dai test FCS descritti in precedenza come stime, regolare la concentrazione iniziale relativa di siRNA nello script. In particolare, variano [siRNA] per essere proporzionale alla quantità relativa di siRNA rilasciata, con un valore di 100 corrispondente all'importo massimo 29 .
- Scrivi il set di equazioni differenziali ordinarie come:
5. Cultura delle cellule e consegna di siRNA in vitro
- Cultura NIH / 3T3 fibroblasti embrionali murine secondo i protocolli del fornitore.
- Crescere le cellule in mezzo di crescita (Dulbecco's Modified Eagle Medium (DMEM), integrato con 10% di siero fetale bovino inattivato termicamente e 1% di penicillina-streptomicina). Mantenere le cellule a 37 ° C in un ambiente umidificato con 5% CO 2 .
- Seed le cellule in piastre trattate con coltura da 6 pozzetti.
- Seguire la procedura di subcultura consigliata dal fornitore. Conte le cellule usando un emocitometro. Diluire le cellule in supporti di crescita supplementari ad una concentrazione di 75.000 cellule / ml.
- Aggiungere 2 mL di sospensione cellulare (75.000 cellaS / mL) ad ogni pozzetto della piastra a 6 pozzetti. Lasciare che le cellule si aderiscano e si riprendano per 24 h nell'incubatrice.
- Preparare le cellule per la trasfezione lavando con soluzione salina fosforata-tamponata (PBS) e aggiungendo a ciascun pozzetto 1,5 ml di mezzo di trasfezione senza siero e antibiotico (vedi tabella dei materiali ).
- Formulare i nanocarrier siRNA secondo i punti 1.1-1.2. Aggiungere 25 μL di soluzione nanocarrier contenente 30 pmol di siRNA a ciascun pozzetto. Pipettate leggermente i supporti verso l'alto e verso il basso per mescolare. Posizionare le cellule nell'incubatrice per 3 ore.
- Rimuovere il supporto di trasfezione e lavare ogni pozzetto con PBS. Aggiungere 1 ml di supporti di crescita supplementari e posizionare le cellule nell'incubatrice per recuperare per 30 minuti.
- Per preparare le cellule per il trattamento con un foto-stimolo, rimuovere i supporti di crescita supplementari. Aggiungere 1 ml di mezzi di trasfezione (senza fenolo rosso) ad ogni pozzetto.
NOTA: Assicurarsi che il supporto di transfection non contenga fenolo rosso. - Calibrare e impostare un laser UV con un filtro da 365 nm ad un'intensità di 200 W / m. Assicurarsi che l'intensità della luce venga misurata dalla posizione in cui sarà posizionato il fondo della piastra di cella.
- Mettere le celle su una piastra calda impostata a 37 ° C. Rimuovere la copertura delle piastre delle celle. Irradiare le cellule da sopra la piastra per il tempo desiderato (fino a 20 minuti) utilizzando il laser UV con un filtro a 365 nm ad un'intensità di 200 W m -2 .
- Rimuovere il supporto di transfezione e aggiungere 2 ml di supporti di crescita supplementari. Posizionare in incubatore fino a ulteriori analisi ( ad esempio , 24 ore per qPCR e 48 ore per Western blotting).
- Misurare le modifiche nell'espressione genica utilizzando una varietà di tecniche come Western blotting 37 e qPCR. 38 Per i geni con segnali visibili, come il GFP, utilizzare la microscopia a fluorescenza 29 .
NOTA: queste tecniche sono suggerite a causa della loro facilità di utilizzo e precisioneNella quantificazione dell'espressione genica
- Misurare le modifiche nell'espressione genica utilizzando una varietà di tecniche come Western blotting 37 e qPCR. 38 Per i geni con segnali visibili, come il GFP, utilizzare la microscopia a fluorescenza 29 .
6. Controllare il silenziamento del gene in modo spatiotemporale
- Culture, sementi e cellule transfettate secondo i punti 5.1-5.7.
- Preparare una fotomassa che blocca completamente la luce di 365 nm e riduce al minimo le riflessioni.
NOTA: In questo caso, sono stati utilizzati pezzi di foglio di alluminio da 10 x 10 cm e carta da costruzione nera per bloccare la luce e ridurre rispettivamente le riflessioni. Il foglio di alluminio e la carta da costruzione sono stati incollati per formare un'unica unità.- Tagliare / punzonare / modellare la forma desiderata nella fotomassa. Ad esempio, utilizzare una lama affilata e un punzone per formare un reticolo lineare (~ 5 cm di lunghezza) e un motivo circolare (~ 7 mm di diametro), rispettivamente.
- Colla la fotomassa al fondo della piastra a 6 pozzetti con il motivo centrato sotto il pozzo contenente le celle con il lato antiriflesso ( ad es .Carta di montaggio) rivolta verso la piastra. Assicurarsi che la colla non sia posta vicino al bordo (entro ~ 3 mm) del modello.
- Impostare due anelli distanziati di circa 25 cm e collegare una piattaforma a ciascun banco d'anello in modo che le piattaforme siano di uguale altezza. Sospendere la piastra di cella tra i due stand appoggiando la piastra in cima alle piattaforme. Assicurarsi che la piastra sia al livello.
- Irradiare le cellule dal basso del campione per il tempo desiderato (fino a 20 minuti) utilizzando il laser UV con un filtro da 365 nm ad un'intensità di 200 W / m 2 .
- Rimuovere il supporto di transfezione e aggiungere 2 ml di supporti di crescita supplementari. Inserire nell'incubatore per recuperare per almeno 24 h. Immagini le cellule utilizzando la microscopia a fluorescenza come descritto 29 .
Risultati
Dopo la formulazione dei nanocarri, sono stati condotti saggi di rilascio siRNA per informare le condizioni di irradiazione da utilizzare nelle transazioni in vitro . Diversi dosaggi di luce sono stati applicati per determinare il percentuale di siRNA che è stato rilasciato. Il primo saggio ha utilizzato l'elettroforesi gel per separare le molecole siRNA libere dalle molecole siRNA ancora complessate / associate al polimero. I nanocarriers non trattati con la luce sono rimasti stabili e non hanno rilasciato alcun siRNA. Aumentando la durata del tempo di irradiazione, l'intensità di fluorescenza delle bande siRNA libere aumenta. Quantificazione delle intensità della banda mediante software di analisi delle immagini ha dimostrato che ~ 15% del siRNA è stato liberato dopo 20 minuti di esposizione alla luce.
Il secondo test di rilascio siRNA ha utilizzato FCS per misurare il percento delle molecole siRNA che sono state liberamente diffuse in solution. Le percentuali di conteggio basale dei campioni contenenti i nanocarrier non esposti alla luce erano uguali a quelli del conteggio del campione di controllo del buffer vuoto, indicando che non era presente alcun siRNA libero. I tassi di conteggio basale aumentarono aumentando il tempo di irradiazione. Il percentuale del siRNA libero è stato calcolato e trovato essere in accordo con le misurazioni ottenute da elettroforesi gel. Generalmente, le stime delle due tecniche erano all'interno di un errore (p> 0,05 per t-test dello Studente) e indicavano che il percentuale di siRNA rilasciato non aumentava significativamente con più di 20 minuti di irradiazione. È importante notare che l'elettroforesi gel e l'FCS sono stati utilizzati per quantificare il rilascio di siRNA perché sono facili da usare, relativamente veloci da analizzare e fornire risultati accurati.
Le quantità relative di siRNA rilasciate dopo diversi dosaggi di luce sono stati inseriti come siRN inizialeUna concentrazione nel modello cinetico. Come mostrato nella figura 1A , il modello è stato eseguito per prevedere le concentrazioni di mRNA, proteina e siRNA di gliceraldeide 3-fosfato deidrogenasi (GAPDH) in funzione del tempo sotto ogni condizione di irraggiamento. Le previsioni del modello per i livelli di espressione di proteine GAPDH erano in accordo con i livelli di espressione sperimentali determinati ottenuti mediante blotting occidentale ( Figura 1B ). In particolare, il livello di proteina GAPDH è diminuito con l'aumentare del tempo di irradiazione. Le cellule irradiate per il più lungo periodo di tempo (20 min) hanno mostrato i maggiori cambiamenti nell'espressione genica in quanto sono state rilasciate maggiori quantità di siRNA. Importante, le cellule che non sono state esposte alla luce non hanno mostrato alcun ristagno genico, indicando che i nanocarriers hanno mantenuto la dormienza e non hanno rilasciato alcun siRNA, a meno che non venga attivato dal fotostimolo. I test di citotossicità hanno dimostrato che le cellule NIH / 3T3Mantenuto la vitalità ≥ 90% dopo il trattamento con i nanocarrier e 20 min di irradiazione 28 .
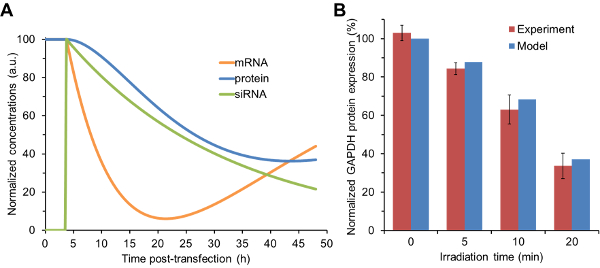
Figura 1 : Predizioni relative alla modellazione cinetica e variazioni sperimentali nell'espressione proteica. ( A ) dati di rilascio siRNA ottenuti da elettroforesi gel sono stati utilizzati per modulare la concentrazione iniziale di siRNA nel modello cinetico. Questi grafici rappresentano l'uscita del modello per le cellule che ricevono 20 minuti di irradiazione. ( B ) Le quantità relative di siRNA da ogni condizione di irraggiamento sono state inserite nel modello cinetico per prevedere i cambiamenti nell'espressione di proteine. Queste previsioni sono state confrontate con i livelli di espressione di proteine GAPDH ottenuti da analisi di blotting occidentale. I risultati sono mostrati come la media ± deviazione standard di dOttenuti da tre campioni indipendenti. I dati sperimentali sono stati ristampati in parte con l'autorizzazione dal riferimento 29 . Clicca qui per visualizzare una versione più grande di questa figura.
Per controllare il silenziamento del gene in modo spatiotemporale, è stato utilizzato un fotomasco per limitare le popolazioni cellulari specifiche al photo-stimolo. Come mostrato in Figura 2 , l'espressione di GFP nelle celle è stata controllata spazialmente in un modello circolare. Le cellule protette dalla luce presentavano intensità di fluorescenza indistinguibili da campioni di controllo non trattati con siRNA e luce. Tuttavia, quasi tutte le cellule all'interno del modello circolare non mostravano alcuna espressione di GFP, indicando un rilascio efficace di siRNA e la rottura del gene. Inoltre, l'uso della luce come un trigger ha permesso l'espressione genica di essere il controlloHa portato a scale di lunghezza cellulare.

Figura 2 : Controllo spaziale sul silenziamento del gene. Le cellule NIH / 3T3 sono state co-trasfettate con lipofere contenenti pFD-GFP e nanopartenti siRNA / mPEG- b- P (APNBMA) targeting GFP. Una fotomassa circolare è stata applicata prima dell'irradiazione con luce di 365 nm per 20 minuti. Le cellule sono state immagate su un microscopio a fluorescenza 48 h post-transfezione. La linea rossa tratteggiata rappresenta il bordo della fotomassa e la barra di scala rappresenta 1 mm. Adattato con permesso di riferimento 29 . Clicca qui per visualizzare una versione più grande di questa figura.
Discussione
Ci sono pochi passi nel metodo che sono particolarmente critici. Nella formulazione dei nanocarri, l'ordine di aggiunta di componenti e la velocità di miscelazione sono due parametri importanti che influenzano l'efficacia 39 . Questo protocollo richiede che la componente cationica, mPEG- b- P (APNBMA), venga aggiunta alla componente anionica, siRNA, in modo goccia mentre si vortexa. A seconda del volume totale di formulazione, questo processo di miscelazione richiede 3-6 s. Per verificare se i nanocarrier sono stati formati correttamente, misurare la distribuzione della dimensione utilizzando una tecnica come la dispersione luminosa dinamica. L'MPEG-b -P (APNBMA) / nanovettori siRNA con un rapporto N / P di 4 aventi un diametro medio di circa 140 nm e polidispersità di 0,2 ~ 28. Inoltre, il rapporto N / P può essere variato per ottenere polilessi con dimensioni / stabilità diverse. Un rapporto N / P di 4 è stato scelto in questi studi perché era il rapporto più basso che ha reso possibile il sequestro completo e il suoMacinazione di bromuro di etidio.
Un altro parametro critico nel metodo prevede l'aggiunta di SDS alle soluzioni nanocarrier nei test di rilascio siRNA. SDS, un tensioattivo anionico, è stato utilizzato per simulare meglio gli ambienti intracellulari contenenti alte concentrazioni di membrane lipidiche e polianioni 29 , 40 . Il rapporto S / P deve essere sufficientemente elevato per simulare l'abbondanza di lipidi anionici presenti nelle cellule, ma non così grande che i nanocarrier disassemblano prima del fotosenso e analizzano ulteriormente. È necessario verificare una gamma di rapporti S / P utilizzando elettroforesi gel per identificare la quantità massima di SDS che può essere aggiunta senza rilasciare il siRNA. Questo protocollo ha utilizzato un rapporto S / P relativamente elevato di 15.
Una limitazione potenziale di questo metodo è legata alla modellazione cinetica. Il modello richiede l'ingresso di mRNA e delle proteine della proteina del gene di interesse. TurnovPer i geni che sono stati ben studiati possono essere trovati in letteratura; Tuttavia, queste informazioni potrebbero non essere disponibili per tutti i geni. Per aggirare questo problema, la metà dei geni simili può essere trovata nella letteratura per fornire stime. In alternativa, i tassi di turnover per il gene specifico di interesse possono essere determinati sperimentalmente 36 . È inoltre importante notare che le informazioni per migliaia di geni possono essere trovate in set di dati compilati 41 .
Un altro parametro chiave in questo metodo è il tipo di cella. I fibroblasti NIH / 3T3 sono stati utilizzati in questi studi come una linea cellulare di modelli perché sono stati ampiamente studiati, sono facili da lavorare e sono applicabili ad una serie di applicazioni di medicina rigenerativa. Tuttavia, il protocollo può essere applicato ad altri tipi di cellule di interesse. La formulazione del nanocarrier potrebbe essere necessario ottimizzare per il tipo specifico di cellule.
In sintesi, questo metodo enConsente la rapida determinazione delle condizioni di irradiazione allo spatiotemporale controllo del rilascio di siRNA e prevede il silenziamento del gene a priori . Questo metodo sarebbe facilmente adattabile ad altri sistemi di erogazione di acidi nucleici fotocompatibili. Ad esempio, i polimeri mPEG- b- P (APNBMA) possono essere modificati sintonizzando le lunghezze di blocco e / o le parti funzionali per adattare il comportamento fotosensibile dei nanocarri 42 . L'impiego di questo protocollo aiuta a chiarire le relazioni di struttura-funzione che governano la stabilità e l'efficacia dei nanocarrier. Queste intuizioni meccaniche possono consentire applicazioni in medicina rigenerativa che richiedono un controllo spaziale sulla silenziazione del gene. Inoltre, a causa della profondità di penetrazione intrinseca di 365 nm di luce nei tessuti acquosi, queste formulazioni sono adatte per il trattamento delle malattie manifestate sulla superficie del corpo come il cancro della pelle e le ferite attuali.
Divulgazioni
Gli autori dichiarano di non avere interessi finanziari concorrenti.
Riconoscimenti
Gli autori ringraziano l'Istituto Nazionale delle Scienze Mediche Generali degli Istituti Nazionali di Salute (NIH) per il sostegno finanziario attraverso un Premio di Sviluppo Istituzionale (IDeA) sotto il numero di sovvenzione P20GM103541 e il numero di sovvenzione P20GM10344615. Le affermazioni qui riportate non riflettono le opinioni del NIH. Riconosciamo inoltre l'Associazione Delaware Biotechnology (DBI) e l'Ufficio per lo Sviluppo economico di Delaware (DEDO) per il sostegno finanziario attraverso il Centro Bioscience per la tecnologia avanzata (Bioscience CAT) (12A00448).
Materiali
| Name | Company | Catalog Number | Comments |
| siRNA | Sigma-Aldrich | SIC001 | non-targeted, universal negative control |
| mPEG-b-P(APNBMA) | synthesized in our lab | N/A | photo-responsive polymer |
| HEPES | Fisher Scientific | BP310-100 | |
| sodium dodecyl sulfate | Sigma-Aldrich | 436143 | |
| rubber gasket | McMaster-Carr | 3788T21 | 0.5 mL thick |
| UV laser | Excelitas Technologies | Omnicure S2000 | collimating lens and 365 nm filter used |
| agarose | Fisher Scientific | BP160-100 | |
| ethidium bromide | Fisher Scientific | BP1302-10 | |
| siRNA labelled with Dy547 | GE Healthcare Dharmacon, Inc. | custom order | fluorophore conjugated to 5’ end of sense strand |
| microscope slide | Fisher Scientific | 12-550-A3 | pre-cleaned glass |
| Secure-Seal Spacer | Life Technologies | S24735 | double-sided adhesive |
| LSM 780 | Carl Zeiss | N/A | confocal microscope |
| ZEN 2010 | Carl Zeiss | N/A | FCS analysis software |
| MATLAB | MathWorks | N/A | programming language |
| NIH/3T3 cells | ATCC | ATCC CRL-1658 | |
| DMEM | Mediatech | 10-013-CV | growth media |
| fetal bovine serum | Mediatech | 35-011-CV | heat-inactivated |
| penicillin-streptomycin | Mediatech | 30-002-CI | |
| 6-well plates | Fisher Scientific | 08-772-1B | |
| Opti-MEM | Life Technologies | 11058021 | transfection media |
Riferimenti
- Forbes, D. C., Peppas, N. A. Oral delivery of small RNA and DNA. J Control Release. 162 (2), 438-445 (2012).
- Davis, M. E., et al. Evidence of RNAi in humans from systemically administered siRNA via targeted nanoparticles. Nature. 464 (7291), 1067-1070 (2010).
- Bouchie, A. Companies in footrace to deliver RNAi. Nat Biotechnol. 30 (12), 1154-1157 (2012).
- Burke, P. A., Pun, S. H., Reineke, T. M. Advancing Polymeric Delivery Systems Amidst a Nucleic Acid Therapy Renaissance. ACS Macro Lett. 2 (10), 928-934 (2013).
- Gooding, M., Browne, L. P., Quinteiro, F. M., Selwood, D. L. siRNA Delivery: From Lipids to Cell-penetrating Peptides and Their Mimics. Chem Biol Drug Des. 80 (6), 787-809 (2012).
- Motta-Mena, L. B., et al. An optogenetic gene expression system with rapid activation and deactivation kinetics. Nat Chem Biol. 10 (3), 196-202 (2014).
- Wang, X., Chen, X., Yang, Y. Spatiotemporal control of gene expression by a light-switchable transgene system. Nat Methods. 9 (3), 266-269 (2012).
- Ziauddin, J., Sabatini, D. M. Microarrays of cells expressing defined cDNAs. Nature. 411 (6833), 107-110 (2001).
- Saltzman, W. M., Olbricht, W. L. Building drug delivery into tissue engineering. Nat Rev Drug Discov. 1 (3), 177-186 (2002).
- Mura, S., Nicolas, J., Couvreur, P. Stimuli-responsive nanocarriers for drug delivery. Nat Mater. 12 (11), 991-1003 (2013).
- Shim, M. S., Kwon, Y. J. Stimuli-responsive polymers and nanomaterials for gene delivery and imaging applications. Adv Drug Delivery Rev. 64 (11), 1046-1058 (2012).
- Kelley, E. G., Albert, J. N. L., Sullivan, M. O., Epps, T. H. Stimuli-responsive copolymer solution and surface assemblies for biomedical applications. Chem Soc Rev. 42 (17), 7057-7071 (2013).
- Weber, W., Fussenegger, M. Emerging biomedical applications of synthetic biology. Nat Rev Genet. 13 (1), 21-35 (2012).
- Konermann, S., et al. Optical control of mammalian endogenous transcription and epigenetic states. Nature. 500 (7463), 472-476 (2013).
- Shimizu-Sato, S., Huq, E., Tepperman, J. M., Quail, P. H. A light-switchable gene promoter system. Nat Biotechnol. 20 (10), 1041-1044 (2002).
- Huschka, R., et al. Gene Silencing by Gold Nanoshell-Mediated Delivery and Laser-Triggered Release of Antisense Oligonucleotide and siRNA. ACS Nano. 6 (9), 7681-7691 (2012).
- Li, H. -J., Wang, H. -X., Sun, C. -Y., Du, J. -Z., Wang, J. Shell-detachable nanoparticles based on a light-responsive amphiphile for enhanced siRNA delivery. R Soc Chem Adv. 4 (4), 1961-1964 (2014).
- Braun, G. B., et al. Laser-Activated Gene Silencing via Gold Nanoshell-siRNA Conjugates. ACS Nano. 3 (7), 2007-2015 (2009).
- Gilleron, J., et al. Image-based analysis of lipid nanoparticle-mediated siRNA delivery, intracellular trafficking and endosomal escape. Nat Biotechnol. 31 (7), 638-646 (2013).
- Wittrup, A., et al. Visualizing lipid-formulated siRNA release from endosomes and target gene knockdown. Nat Biotechnol. 33 (8), 870-876 (2015).
- Roth, C. M. Quantitative measurements and rational materials design for intracellular delivery of oligonucleotides. Biotechnol Prog. 24 (1), 23-28 (2008).
- Mao, S., et al. Influence of polyethylene glycol chain length on the physicochemical and biological properties of poly(ethylene imine)-graft-poly(ethylene glycol) block copolymer/SiRNA polyplexes. Bioconjugate Chem. 17 (5), 1209-1218 (2006).
- Raab, R. M., Stephanopoulos, G. Dynamics of gene silencing by RNA interference. Biotechnol Bioeng. 88 (1), 121-132 (2004).
- Cuccato, G., et al. Modeling RNA interference in mammalian cells. BMC Systems Biology. 5, 1(2011).
- Varga, C. M., Hong, K., Lauffenburger, D. A. Quantitative analysis of synthetic gene delivery vector design properties. Mol Ther. 4 (5), 438-446 (2001).
- Chen, H. H., et al. Quantitative comparison of intracellular unpacking kinetics of polyplexes by a model constructed from quantum Dot-FRET. Mol Ther. 16 (2), 324-332 (2008).
- Bartlett, D. W., Davis, M. E. Insights into the kinetics of siRNA-mediated gene silencing from live-cell and live-animal bioluminescent imaging. Nucleic Acids Res. 34 (1), 322-333 (2006).
- Foster, A. A., Greco, C. T., Green, M. D., Epps, T. H., Sullivan, M. O. Light-Mediated Activation of siRNA Release in Diblock Copolymer Assemblies for Controlled Gene Silencing. Adv Healthc Mater. 4 (5), 760-770 (2015).
- Greco, C. T., Epps, T. H., Sullivan, M. O. Mechanistic Design of Polymer Nanocarriers to Spatiotemporally Control Gene Silencing. ACS Biomater Sci Eng. 2 (9), 1582-1594 (2016).
- Green, M. D., et al. Catch and release: photocleavable cationic diblock copolymers as a potential platform for nucleic acid delivery. Polym Chem. 5 (19), 5535-5541 (2014).
- Lee, P. Y., Costumbrado, J., Hsu, C. Y., Kim, Y. H. Agarose Gel Electrophoresis for the Separation of DNA Fragments. J Vis Exp. (62), e3923(2012).
- Schneider, C. A., Rasband, W. S., Eliceiri, K. W. NIH Image to ImageJ: 25 years of image analysis. Nat Methods. 9 (7), 671-675 (2012).
- Marquer, C., Leveque-Fort, S., Potier, M. C. Determination of Lipid Raft Partitioning of Fluorescently-tagged Probes in Living Cells by Fluorescence Correlation Spectroscopy (FCS). J Vis Exp. (62), (2012).
- Staaf, E., Bagawath-Singh, S., Johansson, S. Molecular Diffusion in Plasma Membranes of Primary Lymphocytes Measured by Fluorescence Correlation Spectroscopy. J Vis Exp. (120), e54756(2017).
- Buyens, K., et al. Monitoring the disassembly of siRNA polyplexes in serum is crucial for predicting their biological efficacy. J Control Release. 141 (1), 38-41 (2010).
- Eden, E., et al. Proteome Half-Life Dynamics in Living Human Cells. Science. 331 (6018), 764-768 (2011).
- Towbin, H., Staehelin, T., Gordon, J. Electrophoretic transfer of proteins from polyacrylamide gels to nitrocellulose sheets: procedure and some applications. Proc Natl Acad. Sci U S A. 76 (9), 4350-4354 (1979).
- Wittwer, C. T., Herrmann, M. G., Moss, A. A., Rasmussen, R. P. Continuous Fluorescence Monitoring of Rapid Cycle DNA Amplification. Biotechniques. 54 (6), 314-320 (2013).
- Cho, S. K., Dang, C., Wang, X., Ragan, R., Kwon, Y. J. Mixing-sequence-dependent nucleic acid complexation and gene transfer efficiency by polyethylenimine. Biomater Sci. 3 (7), 1124-1133 (2015).
- Liu, Y. M., Reineke, T. M. Poly(glycoamidoamine)s for gene delivery: Stability of polyplexes and efficacy with cardiomyoblast cells. Bioconjugate Chem. 17 (1), 101-108 (2006).
- Schwanhausser, B., et al. Global quantification of mammalian gene expression control. Nature. 473 (7347), 337-342 (2011).
- Greco, C. T., Muir, V. G., Epps, T. H., Sullivan, M. O. Efficient tuning of siRNA dose response by combining mixed polymer nanocarriers with simple kinetic modeling. Acta Biomater. 50, 407-416 (2017).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon